JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
리파제 및 포스 후보자에 대한 기질 특이성을 정의
요약
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
초록
미생물 유기체의 외부 기판을 사용할 수 있도록하기 위해 분비된다 (포스) 리파제의 넓은 스펙트럼을 생산하고 있습니다. 또한, 다른 (포스) 리파제는 물리적으로 생산 유기체가 고유 지질의 매출을 일으키는 원인이 자주 세포막의 리모델링에 상승을 제공과 연관 될 수있다. 전위 (포스)은 리파제 유전자 / 단백질 서열 사용할 알고리즘의 번호와 예측 될 수 있지만, 효소 활성, 기질 특이성, 잠재적 생리 기능의 실험적 증거는 종종 수득되지 않았다. 이 원고는 알 수없는 기질 특이성하는 방법과 각각의 (포스) 리파제의 자연 기판에 대한 검색이 최적의 조건을 사용하는 방법과 미래 (포스) 리파제에 대한 분석 조건의 최적화에 대해 설명합니다. 미성년자를 감지하는 데 도움이, 같은 페이지의 -nitrophenyl 유도체 인공 발색 기판을, 수 사용표준 조건에서 예측 (포스) 리파제에 대한 효소 활성. 이러한 보조 효소 활성을 발생하는 데, 효소 분석의 고유 파라미터는 인공 기판의보다 효율적인 가수 분해를 얻기 위해 변화 될 수있다. 효소가 잘 작동되는 조건을 판정 한 후, 자연 전위 다양한 기판은 그들의 열화 구별 크로마토 그래피 방법을 이용 하였다 될 수있는 프로세스를 분석한다. 새로운 효소 기질 특이성의 정의는 종종 후 실험적으로 테스트 될 수있는 이러한 효소의 전위 생리적 역할에 대한 가설을 제공한다. 이 가이드 라인에 따라, 우리는 성장의 인 제한 조건에 따라 세균 Sinorhizobium의 meliloti에서 세포막의 리모델링을위한 중요한 단계에서, 포스 포 콜린과 디아 실 글리세롤하는 포스파티딜콜린을 저하 포스 포 리파제 C (SMc00171)을 식별 할 수 있었다. 두 patatin- 예측을 위해같은 유기체의 포스 포 (SMc00930 및 SMc01003)처럼, 우리는 그들의 기질 특이성을 재정의하고 SMc01003은 디아 실 글리세롤 리파제 있음을 명확히 할 수있다.
서문
이러한 트리 아실 글리세롤과 (글리세) 인지질 등의 글리세롤 기반의 지질이 중요한 구성하고 아마 가장 잘 알려진 지질 클래스 1. 트리 아실 글리세롤 (태그) 지방 또는 일반적으로 저장 지질의 기능 오일, 따라서 에너지 및 탄소 소스로합니다. 태그는 자주 외부 태그를 소화하고 탄소 소스로이를 사용할 수 있도록 생산 미생물에 의해 분비되는 리파제에 의해 저하 될 수 있습니다. 또한, 리파제 널리 그들의 중요한 생명 공학 응용 프로그램을 2 년 동안 연구되어왔다.
때문에 자신의 양친 매성 자연과 가까운 원통 모양, (글리세) 인지질 전시 막 형성 특성과 일반적으로 이중 층 막 3의 주요 지질 구성 요소를 구성합니다. 이러한 박테리아, 대장균, 세 개의 주요 그룹 헤드 변형, 포스파티딜 글리세롤 (PG), 카디오 리핀 (CL) 및 phosphatid 간단한 미생물에서하나는 그들 각자가 서로 다른 분자 종 (4)를 다수로 상기 SN의 다른 지방산 아실 사슬의 상당수 -1 SN -2 위치주는 증가로 치환 될 수 있음을 알아야하지만 ylethanolamine (PE)가 발생하는 . 다른 박테리아는 다른 인지질 외에 또는 대신이있을 수 있습니다. 예를 들어, Sinorhizobium의 meliloti은, 콩과 식물 알팔파 (Medicago 사티)와 질소 고정 뿌리혹 공생 관계를 형성 할 수있는 토양 박테리아, 제 양쪽이 온성 인지질, 포스파티딜콜린 (PC)를 PE 이외에 포함 오. 또한, 지질이 포함되지 인 또는 글리세롤은 세포막의 양친과 형태의 일부가 될 수 있습니다. 예를 들어, 인 - 제한 성장 조건에 따라, S.의 meliloti, (글리세) 인지질은 크게, 즉, sulfolipids, 오르니 틴 지질 및 diacylglyceryl의 trimethylho 인을 포함하지 않는 막 지질로 대체moserine (DGTS) 6. 박테리아, DGTS은 두 단계 경로 7에서 디아 실 글리세롤 (DAG)로 형성되지만 DAG 생성하기위한 소스는 명확하지 않았다된다. 펄스 - 체이스 실험은 PC가 인 제한 조건에서 형성된다 및 DAG 및 포스 포 콜린에 PC를 변환 할 수있는 DGTS 8 전구체하고 우리는 포스 포 리파제 C (PLCP, SMc00171)을 식별 할 수있는이 원고에 기술 된 방법을 사용하여 수 있다는 제안 8.
별도의 연구에서 우리는 발견이 아 닐 -CoA 합성 효소 (FADD) S.의 결핍 돌연변이 성장 9 정지상 들어갈 때 meliloti 또는 대장균 유리 지방산 축적. 이들 지방산은 세포막의 지질로부터 유도 될 듯하지만, 유리 지방산 또는이를 유리화하는 효소 (들)에 대한 정확한 소스는 공지되지 않았다. 다시 말하지만, 전략을 사용하는 것은이 원고에 설명 된 두 파타 틴과 같은 10 (포스) 리파제 (S.에서 유리 지방산의 형성에 기여하고 SMc00930 SMc01003) meliloti (11)는 예측했다. 놀랍게도 SMc01003는 글리세롤 및 유리 지방산 및 모노 아실 글리세롤 마지막 11로 변환 기판으로서 DAG를 이용했다. 따라서, SMc01003는 DAG 리파제 (DGLA)입니다.
알고리즘의 숫자가 전위 (포스)를 예측하는 존재하지만 12, 13는, 리파제 자신의 정확한 기능과 생리 학적 역할은 일반적으로 알려져 있지 않다. 여기에서 우리는 복제, 프로토콜 개요 및 예측 또는 전위 (포스) 리파제를 과발현. 이 원고 효소 분석법 개발 인공 발색 기질을 사용하여 과발현 (포스) 리파제에 최적화 할 수있는 방법을 설명한다. 우리는이 연구 결과는 미생물 생리학에 대한 우리의 이해를 풍부하게하는 방법을 최적화 효소 분석과 실제 (포스) 리파제 기판이 발생할 수있는 방법과 예제를 제공합니다.
프로토콜
예상 리파제 1. 복제 및 과발현 구조 유전자
- 중합 효소 연쇄 반응 (PCR) (14) 및 특정 올리고 뉴클레오티드 (표 1 참조) (15)를 사용하여, 관심 (smc01003, smc00930 또는 smc00171)의 유전자를 증폭 숙주 생물체의 게놈 DNA로부터, 리파아제에 대한 코드 또는 트리스 포스페이트 예측 (즉, , S. meliloti).
- (올리고 뉴클레오티드의 설계 순서에) 특정 제한 사이트를 소개합니다. 상응하는 제한 효소로 증폭 된 DNA 단편을 소화 등의 PET 시리즈 16 플라스미드와 같은 발현 벡터로 복제.
- 클로닝 된 유전자에 대한 정확한 DNA 서열을 확인한 후, 같은 대장균 BL21 (DE3) pLysS를 16 발현 균주로 변환 벡터.
- 식 호스트 E.의 하룻밤 사전 문화를 준비 대장균 BL21 (DE3) PLYSS 100 ml의 문화 루리아 베르 타니 액체 배지 (LB) (17) 20 ㎖를 포함하는 플라스크 플러스 필요한 항생제에, 복제 된 유전자 또는 빈 벡터와 각각의 애완 동물 벡터를 은닉. 배양 30 ° C에서 세포 (또는 리파아제가 유래되는 세균의 일반적인 성장 온도에서).
- 밤새 예비 배양하여, 620 나노 미터 (OD 620) = 0.05에서 초기 광학 밀도를 얻기 위해 2 L 배양 플라스크에서 500 데워진 LB 배지 ㎖ (플러스 필요한 항생제)을 접종한다. , 문화 및 OD 620 = 0.3에서의 성장을 따라 100 μM의 최종 농도 이소 프로필 β-D-티오 갈 락토 시드 (IPTG)를 추가하고 4 시간 동안 30 ℃에서 교반하면서 배양한다.
- 인큐베이션 기간의 마지막에, 30 분 동안 4 ℃에서 5,000 XG에 500㎖의 원심 분리 튜브에서 원심 분리에 각 배양 옮긴다. 정지 완충액 5 ㎖ (예 : S에서 박테리아 세포 펠렛을 재현 탁Mc00930- 50 mM의 디 에탄올 아민 - 염산 pH를 9.8)에 50 mM 트리스 - 염산 pH를 8.0 SMc00171 발현 세포에서 세포가 발현 SMc01003. 사용할 때까지 -80 ° C에서 세포 현탁액을 저장합니다.
2. 무 세포 단백질 추출물을 준비하고 단백질 농도를 결정
- 박테리아 세포 현탁액을 해동 얼음에 저장합니다. 2 당 20,000 파운드에서 세포 현탁액에게 차가운 압력 셀을 통해 세 번 전달합니다. 4 ° C에서 30 분 동안 5,000 XG에서 원심 분리하여 손상 세포와 세포 파편을 제거합니다.
- 원심 분리 후, 후속 분석을 위해 뜨는에서 100, 500 μL의 분취 량을 준비하고 사용할 때까지 -80 ° C에 저장합니다.
- 선택의 방법에 의해 18 바와 같이 별개의 무 세포 추출물의 단백질 농도를 결정하기 위해 100 ㎕의 분취 량 중 하나를 사용한다.
최적화 효소에 대한 3. 인공 기판(포스) 리파제의 활동
- 별개의 효소 활성의 초기 커버리지를 들어, P는 니트로 페놀 (P는 -NP)로 가수 분해에 컬러 제품을 얻을 인공 기판을 사용합니다.
- 인공 피의 -nitrophenyl 에스테르 기판 이미 최적화 효소 분석법 (포스 포 리파제 C PLCP (SMc00171)에 대한 설명뿐 아니라 예측 파타 틴과 같은 포스 포 SMc00930 및 SMc01003 위해) 사용 피펫 팅 방식은 표 2에 기재된.
- 새로운 전위 (포스) 리파제를 탐색하는 경우에 50 mM Tris-HCl, pH 8.5, 100 mM의 염화나트륨, 0.05 % 트리톤 X-100, 0.5 mM의 P는 -nitrophenyl 함유 화합물 (p의 -nitrophenyl 포스페이트를 함유하는 제 표준 효소 분석을 준비 , 비스 피의 -nitrophenyl, 인산 (P)의 -nitrophenyl 카노 에이트, 또는 P -nitrophenyl 팔미 테이트) 및 무 세포 단백질 추출물 (검사 1, 3, 10, 30, 100, 300 및 (1)의 총 부피 1,000 μg의) 용액에 1 ml의 플라스틱 cuvettes.
참고 : 연속 분석에서 P는 -nitrophenyl 에스테르 가수 분해 다음 사용 알칼리성의 pH (그림 1). 대안 적으로, 효소 반응을 정지하고, 모든 P의 -NP가 페놀 형태로 존재하는 것을 보장하기 위해 배양 기간의 끝에서의 NaOH를 첨가, pH 값의 범위에 대해 하나의 시간 점 분석을 사용한다. - 5 분 동안 30 ° C에서 분광 광도계 피의 -NP의 형성 때문에 405 nm에서의 흡광도의 증가를위한 시간 과정을 따른다. 시간 당 흡광도의 상승의 초기 기울기를 결정함으로써 피의 -NP의 초기 선형 형성을 정량화.
- 램버트 - 비어의 법칙 (ΔA = ε ΔC의 d)를 1 사용하여 페이지의 -NP에 대한 농도 (ΔC)의 변화를 계산합니다.
주 : ΔA 결정 흡광도의 선형 변화이고, ε은 M 단위의 각 파장에서의 몰 흡광 계수는 (-1 cm -1), d는 광 경로 (1 cm)의 길이이며, ΔC가 결정되는 단위 M 농도의 변화 ()는 최대.- 분석 부피가 1 mL로하다는 점을 감안하면, P의 형성 -NP의 양을 계산한다.
참고 : 양 = 농도 X 볼륨. - 이 형성되는 시간에 의해 형성된 P의 -NP 량을 분할하여 효소 활성을 계산한다. 이 작업을 생성하는 책임이 마그네슘 (Mg)에서 단백질의 양에 의해 효소 활성을 나누어 특정 효소 활성을 측정.
- 분석 부피가 1 mL로하다는 점을 감안하면, P의 형성 -NP의 양을 계산한다.
- 후보 유전자 (smc00171, smc00930, 또는 smc01003)가 단지 빈 벡터를 항구 추출물로 표현했다있는 단백질 추출물로 자극 흡광도의 변화를 비교.
주 : 다음 단계를 계속하기 위해, 후보 유전자 발현되었다 된 단백질 추출물에 의해 야기되는 특정 활동, 적어도 두번 또는 m이어야단지 빈 벡터를 항구 단백질 추출물에 의한 특정 활동에 대해 얻어진 값보다 광석. - 추가 실험의 경우, P는 -nitrophenyl 함유 화합물의 가수 분해는 무 세포 추출물 (즉, 빈 벡터)와 대한 피의 -NP의 가장 두드러 형성과 p의 -nitrophenolate 음이온 (그림으로 최소 인 그 조건을 선택 단백질 추출물 후보 유전자 발현되었다되는 채용되는 경우 1)을 관찰 할 수있다.
- 3.1 초기 효소 활성을 측정 한 후, 염화나트륨의 농도, 산도, 버퍼의 종류를 변화시킴으로써, 각각의 효소를 분석 조건을 최적화 강도 버퍼, 예컨대 트리톤 X-100 세제, 상기 부재 또는 다른 가의 양이온의 존재.
- 각 변수의 상이한 농도를 들어, (3.1.4.2 참조) 특정 효소 활성을 측정 (얻어진 가장 많은 상태를 정의최대 효소 활성). 각 변수는 최적의 농도로 존재하는 최적화 된 효소 분석을 정의하는 각 변수 발생 최적 조건의 조합을 사용한다.
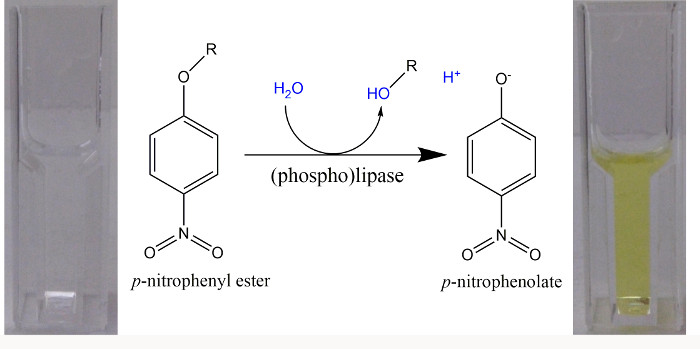
그림 1. P는 -Nitrophenyl 에스테르 분광 분석에서 (포스) 리파제를위한 인공 기판은. P는 -nitrophenyl 에스테르의 가수 분해시, 산 (R-OH) 및 P는 니트로 페놀 (P는 -NP)이 형성된다. 때문에 약동학에 p의 -NP에서 페놀 H의 +의 해리에 대한 = 7.2에> 9.2 99 % 이상은 밝은 노란색 페이지의 -nitrophenolate 양식과 18,000 M -1 cm의 몰 흡광 계수에의 pH에서 - (1)은 파장으로 사용될 수있다 무료 P는 -nitrophenolate (22)의 정량 405 nm 인. 8.5의 pH와 버퍼를 사용했을 때, 흡광도 -1 400 nm의 14,500 M -1 cm의 몰 흡광 계수로 측정 하였다 (23)을 사용 하였다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고 : 관심있는 효소의 활성을위한 최적의 조건을 규정 한 후,이 리파제의 생리 / 실제 기판에 대한 검색에 착수. 원칙적으로, 종종 상보적인 두 취할 이러한 목표, 생체 내 방법 또는 시험 관내 방법을 달성하기 위해 접근한다.
리파제의 생리 기판의 4 생체 식별
"ove_content> 주 : 다른 생체 내 방법에서는, 생성 생체 내 방법에서는, 리파제의 발현이 host's 지질 프로파일을 변경할지 여부를 시간이 지남에 따라 등록하기 위해 숙주 생물 8,11 관심의 리파제를 표현한다. 관심 8,11 유전자의 결실 돌연변이 체 및 지질의 야생형 버전 6,8,11 구별 여부를 연구한다. 유기체의 지질 프로파일의 정량 평가를 얻기 위해서는 간단한 방법은 셀룰러 화합물을 방사성 표지로 이루어져 , 지질 추출 크로마토 그래피로 분리하고, 분리 된 방사성 표지 된 지질 정량.- 지질의 방사성 표지.
- 원하는 배지 (복합 매체 또는 정의 된 최소 배지) 5 ㎖에 관심 (대장균이나 S. meliloti)의 유기체의 하룻밤 예비 배양을 준비하고 30 ° C에서 성장한다.
- 예비 - 배양에서, SA 20 ㎖에 접종100 ml의 배양 플라스크 나 신선한 매체는 초기 OD 620 = 문화 0.3을 얻었다.
- 무균 조건 하에서 문화의 나누어지는 (1 ㎖) 타고 14 ml의 멸균 폴리스티렌 둥근 바닥 튜브에 전송할 수 있습니다.
- 1 ml의 문화에 [1- (14) C] 아세테이트 (밀리몰 60 MCI)의 1 μCi를 추가합니다.
- 24 시간 동안 30 ℃에서 교반하면서 배양액을 부화.
- 인큐베이션 기간의 마지막 5 분 동안 실온에서 12,000 XG에 1.5 ml의 마이크로 원심 튜브를 원심 분리에 배양 옮긴다.
- 물 100 μL에 펠렛을 재현 탁. 이 시점에서, -20 ° C에서 세포 현탁액을 저장하거나 바로 극성 지질 (4.2 절)의 추출을 계속합니다.
- 극성 지질의 추출.
주 : 본질적으로 여기에 설명 된 방법 및 후 Bligh 다이어 (19)에 의해보고 된 절차에 따른다.- 수성 세포 펜던트의 100 μL에(:; 권 / 1 권 2) 클로로포름 용액 : ension는, 메탄올 375 μl를 추가합니다.
- 30 초 동안 와동과 실온에서 5 분 동안 배양한다.
- 원심 분리기 5 분 실온에서 12,000 XG에.
- 새로운 1.5 ML의 microcentrifuge 관에 뜨는을 전송합니다.
- 클로로포름 125 μL 및 물 125 μL, 소용돌이 30 초를 추가합니다.
- 원심 분리기 1 분 실온에서 12,000 XG에.
- 질소 가스의 흐름과 함께 신선한 튜브 및 건조에 낮은 클로로포름 단계를 전송합니다.
- (:; 권 / 1 권 1) 메탄올 용액 : 건조 클로로포름 100 ㎕의 지질을 녹여.
주 :이 시점에서, 지질 용액 5 μL의 분취 량을 액체 섬광 계수에 의해 정량화 될 수있다. - 박층 크로마토 그래피 (TLC) 분석을 위해, 질소 가스의 흐름과 함께, 나머지 95 μL를 건조하고 20 ㎕의 클로로포름 건조 지질 재용 : 메탄올 용액 (1 : 1, 부피 / 부피). 3 μL 나누어지는을 사용하여TLC 분석.
- 박층 크로마토 그래피 (TLC)에 의해 극성 지질의 분리.
참고 : 지질 클래스에 따라하면 분석 할, 고체 및 모바일 단계의 서로 다른 조합의 분리에 이용 될 수 있습니다. 여기서 고체상으로 고성능 박층 크로마토 그래피 (HPTLC) 실리카겔 알루미늄 시트를 사용하여 중성 극성 지질 더 적합 대전 극성 지질 및 서로에 대한 전형적인 분리가 설명된다.- 두 차원 TLC (2D-TLC)에 의해 충전 극성 지질의 분리.
- HPTLC 실리카겔 알루미늄 판 (10 × 10cm), 플레이트의 가장자리에서 2cm의 한 모서리에있는 지질 샘플 3 μL 분취를 적용한다.
- 준비 및 제 차원 분리를위한 이동상 (140 ㎖의 클로로포름 60 ml의 메탄올, 10 ml의 물)를 혼합한다.
- 코트 크로마토 그래피 종이 내부적으로 TLC 현상 실.
주 :이 보장된다는 챔버의 기상제 차원 이동상 후 (30 분 이내) 빠르게 포화된다 챔버에 추가 된 상기 챔버는 유리판으로 폐쇄되었다. - 준비하고 이동상 믹스 (130 ㎖의 클로로포름 50 ㎖의 메탄올과 아세트산 20 ㎖ 빙) 내부 크로마토 종이 코팅 된 초 TLC 현상 챔버 번째 차원의 분리 및 전송을위한 상기 챔버 포화하자.
- 조심스럽게 제 5 차원 밀폐 챔버 내에서 60 분 동안 (예를 들어, 크로마토 그래피를 수행) 판을 상기 제 1 챔버로 건조 된 지질 샘플로 HPTLC 실리카겔 알루미늄 시트를 전송하고 개발한다.
- 챔버에서 접시를 제거하고 용매를 30 분 동안 흐름 후드에서 떨어져 건조 할 수 있습니다.
- 이전 크로마토 그래피에 대해서는 90 ° 판을 켜면 SECON로 지질은 하나의 차원에서 분리 된 된 HPTLC 실리카겔 알루미늄 시트를 전송할D 챔버 및 상기 제 5 치수 60 분 동안 플레이트를 개발한다.
- 챔버로부터 시트를 제거하고, 용매는 적어도 2 시간 동안 흐름 후드에서 마를하자.
- 중성 극성 지질의 분리.
- 플레이트의 가장자리에서 2cm를 시작하는 HPTLC 실리카겔 알루미늄 시트 지질 샘플 3 μL 분취 액을 적용한다. 여러 샘플들은 일차원 크로마토 그래피로 분석하는 경우, 다른 샘플 애플리케이션 스폿 간의 적어도 1.5 cm의 거리를 유지한다.
- 모바일 단계를 준비하고 믹스 (140 ml의 헥산 60 ml의 디 에틸 에테르, 8 ml의 아세트산) 내부 크로마토 그래피 종이 코팅 챔버 포화 (30 분)을 할 수있는 유리판으로 덮여 TLC 개발 챔버 및 전송.
- 챔버에 건조 된 지질 샘플로 HPTLC 실리카겔 알루미늄 시트를 이동 및 밀폐 챔버에서 30 분 동안 플레이트를 개발한다.
- 챔버에서 플레이트를 제거상기 용매는 2 시간 동안 흐름 후드에서 마를하자.
- 두 차원 TLC (2D-TLC)에 의해 충전 극성 지질의 분리.
- 정량 분리 극성 지질의 시각화.
- 개발 된 TLC 시트가 건조되면, 3 일간 폐쇄 카세트에 photostimulable 발광 (PSL) 화면을 품어.
- PSL 스캐너로 배양 화면을 노출하고 분리 된 방사성 표지 지질의 가상 이미지를 획득.
- PSL 소프트웨어 (20)를 사용하여 정량을 수행합니다.
- 시각화 및 개별 극성 지질 클래스의 격리.
- 요오드 결정 1g의 존재 크로마토 실에서 10 분 동안 개발 TLC 시트 인큐베이션.
참고 : 분리 된 지질 화합물은 요오드를 용해 갈색 반점으로 표시됩니다. - 원 연필로 관광 명소, 표준 화합물 (즉, 1,2- dipalmitoyl- SN 글리세롤, 디 팔미 토일-L-α-진료 기관 연합의 상대적인 이동 (R f를)로 비교phatidylcholine, DL-α-monopalmitin, 또는 팔 미트 산), 그들이 속할 수있는 지질 클래스를 식별합니다.
- 흄 후드에서 요오드가 TLC 시트에서 증발 할 수 있습니다.
- 클로로포름 용액 (2 : 주걱의 도움으로, 시트로부터 관심있는 화합물을 함유하는 실리카 겔을 긁어 및 물 100 ㎕과 메탄올 375 μL의 혼합물로 실리카 겔에서 화합물을 추출하는 1; 권 / 부피).
- (이후 4.2.2) 명시된 후 Bligh와 다이어에 따라 추출 계속합니다.
- 사용할 때까지 -20 ° C에서, (권 / 1 권 1) 메탄올 용액 : 클로로포름 100 ㎕에 보관 정제 지질 클래스입니다.
- 요오드 결정 1g의 존재 크로마토 실에서 10 분 동안 개발 TLC 시트 인큐베이션.
리파제의 생리 기판 5. 체외 확인
참고 : 체외 접근 방식에 관심 리파아제가 해당 가수 분해성에 고립 된 지질 또는 개별 순수한 지질의 혼합물을 변환 할 수 있는지 여부를 연구3.2에서 최적으로 정의 된 조건이 Analysis 제품.
- PC 관련 포스 포 리파제 C SMc00171 (5.2 참조), 포스 포 리파제 A (5.3 참조), DAG 리파제 SMc01003 표 3에 따라 효소 분석을위한 피펫 팅 방식을 사용하여 활동 (5.4 참조).
- PC 관련 포스 포 리파제 C 활동 (표 3)의 결정.
- 1.5 ml의 마이크로 원심 튜브로 분당 5000 카운트 총 14 C 표지의 PC (CPM) 및 트리톤 X-100 용액을 추가한다.
- 질소의 흐름에 따라 혼합 및 건조.
- 99.5 ㎕의 최종 부피를 얻기 위해 디 에탄올 아민 - 염산, pH가 9.8 완충액뿐만 아니라 염화나트륨 및 MnCl 2 용액 및 증류수에 추가. 5 초 동안 소용돌이.
- 효소 (5 μg의 단백질) 0.5 μl를 추가하여 반응을 개시하기 위해 (즉, 무 세포 추출액되는 SMc00171가 존재 과발현). 간략하게 섞는다.
- 4 시간 동안 30 ° C에서 품어.
- 바이 반응을 정지메탄올 250 μL 및 클로로포름 125 μL 첨가.
- 전술 한 바와 같이 지질 (4.2 참조)의 압축을 풉니 다.
- 하나의 차원 (1D) -TLC로 구분 지질 (4.3.2 및 4.4 참조), PSL 영상으로 그것들을 분석 할 수 있습니다.
- 포스 A는 활성의 측정 (표 3).
- 1.5 ml의 microcentrifuge 관에, 5,000 총 14 C 표지 인지질의 CPM 및 트리톤 X-100 용액을 추가 할 수 있습니다.
- 질소의 흐름에 따라 혼합 및 건조.
- 최종 100 ㎕의 분석을 위해, 트리스 - 염산, pH가 8.5 버퍼, NaCl 용액과 물을 추가 할 수 있습니다. 5 초 동안 소용돌이.
- 효소의 5 μL (50 μg의 단백질)을 추가 (즉, 무 세포 추출액되는 SMc00930 또는 SMc01003가 존재 과발현).
- 5 시간 동안 30 ° C에서 품어.
- 메탄올 250 μL 및 클로로포름 125 μL를 첨가하여 반응을 정지.
- 별도 전술 한 바와 같이 지질 (4.2 참조) 추출1D-TLC 130 ㎖의 클로로포름 50 ㎖의 메탄올과 이동상으로서 20 ml의 빙초산을 사용하고, PSL 촬상하여이를 분석하여 이들.
- 디아 실 글리세롤 (DAG) 리파제 활성의 결정.
- 14 C 표지 DAG의 준비.
- 방사성 표지 S. meliloti 문화 (4.1 참조) 한 바와 같이 (4.2 참조) 극성 지질의 압축을 풉니 다. 분리 된 S. 메탄올 : 클로로포름 1D-TLC에 의해 meliloti 총 지질 추출물을 아세트산 (130 : 50 : 20; 부피 / 부피) 4.3.1에서 두 번째 차원의 분리에 설명 된 조건을 사용하여.
- 요오드 염색으로 PC를 시각화하고 포스파티딜콜린 (PC)의 현지화를 표시하기 위해 연필을 사용합니다.
- 4.5에 기재된 방사성 표지 된 PC를 분리.
- 섬광 계수에 의해 추출 된 PC를 정량화.
참고 : 약 320,000 CPM의 PC가 예상된다. - 50 mM 트리스 - 염산, pH가 7.2, 0.5에서 클로스 트리 디움 퍼프 린 젠스에서 포스 포 리파제 C의 0.1 U와 PC (25 만 CPM)을 치료% 트리톤 X-100 100 ㎕의 총 부피에서 2 시간 동안 10 mM의 CaCl2를 메탄올 250 μL 및 클로로포름 125 μL를 첨가하여 반응을 정지.
- 1D-TLC (4.3.2 참조) 그들에 의해 이전에 별도의 설명에 따라 지질의 압축을 풉니 다.
- 실리카 플레이트로부터 디아 실 글리세롤을 분리하고 (4.2에 기재된 바와 같이), 섬광 계수에 의해 정량화
- 디아 실 글리세롤 리파제 분석 (표 3).
- 1.5 ml의 microcentrifuge 관에, 5000 14 C 표지 DAG의 CPM 및 트리톤 X-100 용액을 추가 할 수 있습니다.
- 질소의 흐름에 따라 혼합 및 건조.
- 최종 100 ㎕의 분석을 위해, 트리스 - 염산 (산도 9.0) 버퍼하는 NaCl 용액과 증류수에 추가합니다. 5 초 동안 소용돌이.
- 효소의 5 μL (무 세포 추출물 50 μg의 단백질)를 첨가하여 반응을 개시.
- 4 시간 동안 30 ° C에서 품어.
- 메탄올 250 μL를 첨가하여 반응을 정지전술 한 바와 같이 클로로포름 추출물 지질 D 125 μL (4.2 참조).
- 1D-TLC (4.1.3.2 참조) 이후 PSL 이미지로 중성 극성 지질을 분석합니다.
- 14 C 표지 DAG의 준비.
결과
비스 P는 -nitrophenyl 인산와 PC 고유의 포스 C SMc00171의 활동
E. 얻은 무 세포 추출물 대장균 BL21 (DE3)는 smc00171가 형성된 P는 -NP을 측정하는 분광 효소 분석을 이용하여, 비스 페이지 -nitrophenyl 인산 에스테르를 가수 분해하는 ?...
토론
지난 20 년 동안, 많은 생물의 게놈은 서열화되었다 및 게놈 시퀀스 데이터의 재산이 생성되었지만, 기능 해석은 뒤쳐 따라서 게놈 기능에 대한 우리의 이해를 방해한다. 게놈에서 유전자의 기능은 종종 알려진 기능 또는 보존 모티브의 발생 유전자의 유사성을 기준으로 할당됩니다. 그러나, 특정 유전자의 정확한 기능은 대개 알려져 있지 않다. 특히, 효소 구조 유전자를 쉽게 인해 대부분의 효소...
공개
저자는 공개 아무것도 없어.
감사의 말
이 작품은 (Investigación 엉 Fronteras 드 라 Ciencia에서 Investigación Científica BASICA에서 82614, 153998, 253549 및 178359뿐만 아니라 118) 및 Dirección 일반 드에서 Consejo 나시 오날 드를 CIENCIAS Y TECNOLOGIA - 멕시코 (CONACYT 멕시코)에서 보조금에 의해 지원되었다 (DGAPA - UNAM, PAPIIT IN202616, IN203612) Asuntos 드 개인 Académico-대학교 나시 오날 자치시 드 멕시코.
자료
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
참고문헌
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유