JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
생산, 결정화 및 구조 결정의
요약
Proline-proline endopeptidase-1 (PPEP-1) is a secreted metalloprotease and promising drug-target from the human pathogen Clostridium difficile. Here we describe all methods necessary for the production and structure determination of this protein.
초록
New therapies are needed to treat Clostridium difficile infections that are a major threat to human health. The C. difficile metalloprotease PPEP-1 is a target for future development of inhibitors to decrease the virulence of the pathogen. To perform biophysical and structural characterization as well as inhibitor screening, large amounts of pure and active protein will be needed. We have developed a protocol for efficient production and purification of PPEP-1 by the use of E. coli as the expression host yielding sufficient amounts and purity of protein for crystallization and structure determination. Additionally, using microseeding, highly intergrown crystals of PPEP-1 can be grown to well-ordered crystals suitable for X-ray diffraction analysis. The methods could also be used to produce other recombinant proteins and to study the structures of other proteins producing intergrown crystals.
서문
클로스 트리 디움 디피는 원내 항생제 관련 설사 감염 1의 주요 원인 중 하나입니다. 이 그람 양성 혐기성 세균은 분변 - 경구 경로를 통해 그 포자 양식을 통해 전송된다. 지난 10 년, 새로운 '전염병' '또는' 'hypervirulent' '균주 (/ 027 예를 들어, BI / NAP1)는 북미와 유럽이 새로운 감염과 사망률의 급격한 증가가 발생했습니다. C. 디피 -associated 질환 (CDAD)는 높은 사망률 3 생명을 위협하는 대장 염증이다. 증상은 설사 4에서 위 막성 대장염 (5)와 종종 치명적 독성 거대 결장 (6)에 이르기까지 다양합니다.
유독 균주 다제 내성 및 재발 7 높다으로 CDAD 치료가 어렵다. 현재 치료법은 항생제 메트로니다졸, fidaxomicin 또는 반코마이신 또는 repetiti에 포함에서vely 재발하는 경우 분변 미생물 이식. 새로운 치료 전략이 시급히 8 필요하다. 일부 진보가 C. 디피 독소 B (9)을 대상으로, 치료 단클론 항체 Bezlotoxumab로 기록, 최근 성공적으로 임상 3 상을 통과하고 FDA 및 EMA에 승인을 신청했다. 또한, 새로운 항생제 임상 시험 (10)의 다른 단계에서 순간에서 시험되고있다.
새로운 치료 표적을 식별해야 효과적인 치료를 개발합니다. (; CD2830 / Zmp1, PPEP-1 EC 3.4.24.89)가 최근 C. 디피 단백질 분해 효소 프롤린 - 프롤린 엔도 펩 티다 제-1 발견 노크 아웃 변형에 PPEP-1의 부족 등 유망한 대상이되고, C의 독성을 감소 . 생체 내 11 디피. PPEP-1은 C 말단에 두 C. 디피의 adhesins를 절단 분비 메탈로 12, 13 (13), 따라서 부착 bacter를 해제인간의 장내 상피 세포에서 아이오와. 따라서, C. 디피의 정착과 운동성 표현형 사이의 균형을 유지에 관여한다. PPEP-1에 대해 선택적 저해제를 개발하고 그 기판의 입체 구조의 자세한 지식이 필요 불가결 인식하는 방식을 이해하는 것이. 우리는 기질 펩티드 14 PPEP-1 단독 컴플렉스의 제 결정 구조를 해결했다. PPEP-1은 제 1 공지 프로테아제 두 개의 프롤린 잔기 (15) 사이를 선택적으로 절단하여 펩티드 결합. 이것은 이중 꼬임 방식으로 기판에 결합하고, 프로테아제 활성 부위 (14)를 덮는 S 루프에있는 잔기 연장 된 지방족 - 방향족 네트워크를 통해 안정화. 이 기판 결합 모드는 PPEP-1에 고유하며 현재까지 인간의 단백질 분해 효소에서 찾을 수 없습니다. 이것은 유망한 약물 표적하게, 매우 가능성 억제제의 오프 대상 효과.
화면 선택 PPEP-1 INH 개발하려면ibitors은 향후 순수한 단 분산 PPEP-1 단백질의 다량이 필요하다. 또한, 결정되어야 할 것이다 PPEP-1 제 억제제, 공동 결정 구조의 결합 모드를 결정한다. 우리의 손에 PPEP-1은 지속적으로 (intergrown)의 결정을 생성합니다. 따라서 우리는 PPEP-1의 단일 회절 품질의 결정을 생성하는 최적화 과정을 개발 하였다. 이 프로토콜에서 우리는 구체적으로 PPEP-1 (14)의 생산, 정제, 결정화 및 구조 솔루션을 설명합니다. 우리는 분비 신호 서열, 정제 태그의 제거와 친 화성 크로마토 그래피 및 크기 배제 크로마토 그래피 부족한 PPEP-1 변이체의 대장균 세포 내 발현을 사용하여, 아연의 단일 파장 이상 분산을 통해 최적화 화면 구조 결정에 16 microseeding 이어 (아연 SAD) 17. 이 프로토콜은 (다른 단백질의 제조 및 구조 결정에 적용 할 수있다 예를 들어, </ EM> 메탈로) 및 (intergrown)의 결정을 생산하는 단백질 관한 것이다. 요청시 구조물의 플라스미드 DNA는 (는 pET28a-는 NHIS-rPPEP-1)의 회절 데이터는 교육 목적으로 제공 될 수있다.
프로토콜
1. 복제 및 디자인을 구축
- 신호 펩타이드없이 C. 디피 PPEP-1 (대장균) 코돈 최적화 된 시퀀스를 복제 [아미노산 27-220, 이하 재조합 PPEP -1- 이름 (rPPEP-1) 11]을 Nde I을 사용는 pET28a 벡터에 및을 XhoI 제한 부위 3'- 말단 (생성 된 벡터는 pET28a-NHIS-rPPEP-1)에서 종료 코돈과 (도 1). 이는 정제 과정 태그를 제거 가능 트롬빈 절단 부위 (도 1)와 N 말단 6xHis 표지 된 단백질 (NHIS rPPEP-1)를 생성한다. 플라스미드는 선택을위한 카나마이신 저항성 카세트를 포함합니다. 복제에 사용 된 프라이머는 다른 14 설명되어 있습니다.
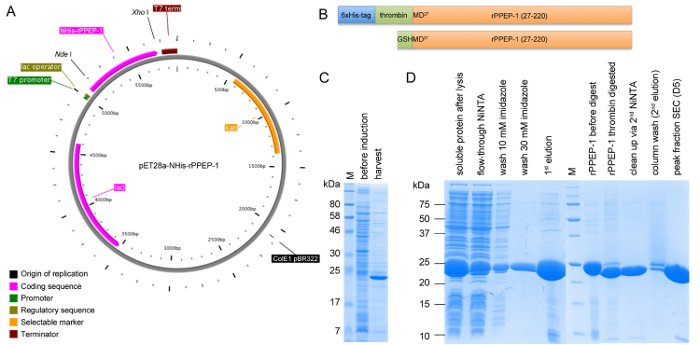
그림 1 : expre의 구성는 pET28a-NHIS-rPPEP-1 및 SDS-PAGE 분석의 도식 표현ssion 모든 정제 단계. NHIS-rPPEP-1 (A) 벡터지도는 내가 PlasMapper로 만든 이니까 I /을 XhoI를 사용하여는 pET28a 벡터에 클로닝. (B)를 NHIS-rPPEP-1 구조 (상판)의 도식 표현과 N 말단 (하단 패널)에서의 결과 추가 GSHM 시퀀스와 6xHis 태그의 트롬빈 - 절단 후 최종 구조. 모든 정제 단계 (: 분자량 마커 M)에서 4 시간과 시료 (D) 37 ℃에서 BL21 (DE3) 별 발현의 SDS-PAGE 분석 (C). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. 발현 및 rPPEP-1의 정제
- NHIS-rPPEP-1의 발현
- 메이크업 및 오토 클레이브 LB (원성 국물) 배지 (10g / L의 트립 톤, 5g / L 효모 추출물, 10g / L의 NaCl, adjuST는) NaOH로 7.5 pH로. 카나마이신 설페이트 (50 μg의 / ㎖) 만 사용 (LB / 칸 매체) 이전에 보충.
- LB / 칸 매체에서 갓 형질 전환 된 대장균에서 200 ml의에게 하룻밤 문화의 예방. 220 rpm으로 진탕 37 ° C에서 하룻밤 성장.
- 다음 날 아침에, 하룻밤 문화의 OD 600 (600 nm 파장에서 광학 밀도)를 확인합니다. 이 2.8 L은 OD 0.1 600에 밤새 문화 1 L LB / 칸 매체 각을 포함하는 플라스크를 당황 접종. 과다한 거품 형성을 방지하기 위해 수성 실리콘 에멀젼 세 방울 보완. (600)이 0.6에 도달 OD 때까지 180 rpm으로 진탕 37 ° C에서 세포를 성장.
- SDS-PAGE 분석을 위해 미리 유도 샘플을 채취 (OD 600에서 1 ㎖의 배양 물로부터의 상당 = 1); NHIS-rPPEP-1의 발현을 유도하기 위해 0.5 mM의 최종 농도 IPTG를 추가합니다. 4 시간 동안 37 ° C / 180 rpm에서 성장을 계속합니다.
- 10 배 디의 OD 600 결정lution는 (OD 600 = 1에서 문화에서 1 ml를 상당)를 수확 샘플을 채취합니다.
- 7,000 XG에 20 분, 4 ° C에 대한 원심 분리하여 세포를 수집합니다. 50 ㎖의 원심 분리기 튜브 및 전송 : 40 ml의 문화의 1 L에서 TBS 버퍼를 잔류 LB 매체에 resuspend 세포 펠렛을 제거하려면 (20 mM 트리스 - 염산, pH가 7.5, 200 mM의 NaCl을 식염수 트리스 버퍼). 사용할 때까지 -80 ° C에서 10,000 XG에 10 분, 4 ° C 및 저장을위한 원심 분리하여 세포를 수집합니다. SDS-PAGE (18)를 통해 표현 (총 해물과 가용성 분획)을 분석합니다.
- 태그 화 rPPEP-1의 정제
- SDS-PAGE 분석을위한 각 정제 단계의 50 μL 샘플을 가져 가라. 10 μg의 / ㎖의 DNaseI 보충 TBS 버퍼에 배양 한 L에서 세포 펠렛을 재현 탁. 세포의 g 당 TBS / DNaseI 5 ㎖를 사용합니다.
- 15 분 (2 초 일시 정지 2 초 펄스) 30 %의 진폭을 이용하여 얼음 / 물에 초음파에 의해를 Lyse는 세포. CENTR에 의해 이물질을 제거초 원심 분리기 튜브 10,000 XG에 10 분, 4 ° C와 전송 상층 액에 대한 ifugation. 165,000 XG, 4 ° C에서 30 분 동안 초 원심 분리기에 걸린 해물.
- 4-6 ° C에서 작업 할 수 있습니다. 연동 펌프 또는 크로마토 그래피 시스템을 사용하여 10 mM의 이미 다졸로 보충 된 pH 7.5의 TBS 버퍼 유리 컬럼에 니켈 니트릴 로트리 아세트산 (NiNTA) 수지 2 mL로 평형화. 대안 적으로, 중력 유동을 사용한다.
- 10 mM의 최종 농도는 1 M 이미 다졸 pH를 7.5로 분해물을 조정한다. 컬럼에 해물을 적용하고 기준에 도달 nm의 280에서 UV 흡수 될 때까지, 10 mM의 각각 30 mM의 이미 다졸, 보충 TBS 버퍼로 단계적으로 씻는다.
- TBS 버퍼 플러스 250 mM의 이미 다졸을 가진 단백질을 용출시켰다. 10 mM의 이미 다졸 보충 TBS에 열을 다시하는 것은-평형 하룻밤 저장합니다.
- 흡광 계수 1을 사용하여 280 nm에서의 단백질 농도를 결정의 효율적인 25900 M -1 -1 cm 또는 다른 방법 (예 : 브래드 포드 방법 19)에 의해. 단백질 mg의 당 트롬빈의 2 대를 추가하고 TBS의 50 배 부피합니다 (NiNTA 용출 부피의 50 배)에 대해 4 ° C에서 하룻밤 단백질 용액을 투석.
참고 : 이미 다졸 280 nm에서 강하게 흡수로, 단백질 농도의 결정에 대한 올바른 빈을 가져 가라. - 비 절단 단백질을 제거 할 수 평형 NiNTA 수지를 통해 단백질 용액을 전달합니다. 다음에, 모든 절단 된 단백질을 회수하여 컬럼 TBS의 동일한 부피의 10 mM의 이미 다졸 보충 적용된다. 열을 청소하려면, 250 mM의 이미 다졸에 남아있는 모든 단백질을 용출. SDS-PAGE (그림 1)를 통해 샘플을 분석 할 수 있습니다.
- 4,000 × g으로 원심 한외 여과 유닛을 사용하여 4 ℃에서 10 분 간격으로 4 용액에 단백질 용액을 농축시킨다. 침전 및 응집을 방지하기 위해 각 구간 후 농축 단백질 섞는다. 가끔이 단계에서LY 일부 침전 혼합 절차에도 불구 rPPEP-1에서 관찰된다.
- 4-6 ℃에서 (TBS 버퍼에) 미리 평형화 크기 배제 크로마토 그래피 칼럼으로 농축 된 단백질을 적용한다. , TBS 버퍼와 열을 용출 1 ml의 분수와 주제 SDS-PAGE 분석에 대한 모든 초 분수의 5 μl를 수집합니다. rPPEP-1은 단량체 (그림 2)에 해당하는 단일 피크로 용출. 때때로 큰 분자량의 작은 피크는 단백질 이량 체에 대응하는 (잘난 피크)이 관찰된다. 수율은 문화의 L 당 순수 단백질의 약 50 mg을해야한다. SDS-PAGE (18)를 통해 모든 샘플 (그림 2)를 분석합니다.
- SDS-PAGE 분석을위한 각 정제 단계의 50 μL 샘플을 가져 가라. 10 μg의 / ㎖의 DNaseI 보충 TBS 버퍼에 배양 한 L에서 세포 펠렛을 재현 탁. 세포의 g 당 TBS / DNaseI 5 ㎖를 사용합니다.
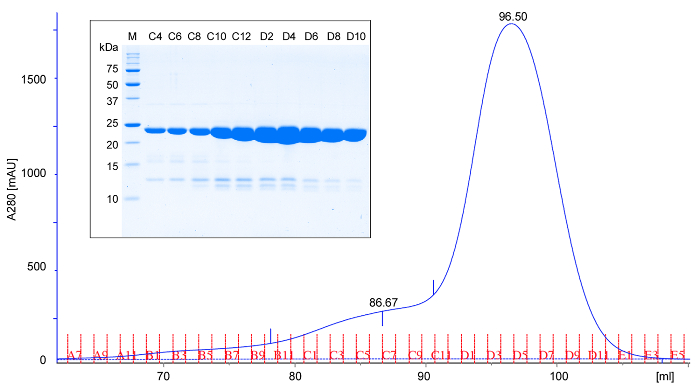
도 2 : rPPEP-1의 대표적인 크기 배제 크로마토 그래피 및 SDS-PAGE 분석. 크기 배제 크로마토 (A280; 6 ° C에서 트리스 - 염산, pH가 7.5, 200 mM의 염화나트륨의 (16/600) 컬럼을 사용하여 정제 된 태그가없는 rPPEP-1의 280 nm에서 흡광도). 용출 부피를 기준으로, 1-rPPEP를 마이그레이션은 우세 단량체임을 시사 22 kDa 단백질로 예상. 드물게 작은 잘난척 피크는이 이량 체에 해당 나타납니다. (삽입) 크기 배제 크로마토 그래피의 분획의 SDS-PAGE 분석 (M; 분자량 마커). 모든 초 부분이 적용됩니다. 주요 rPPEP-1 밴드 아래의 희미한 밴드는 때때로 약간의 불순물을 발생에 대응한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Microseeding를 사용하여 3. 결정화 및 크리스탈 최적화
주 : rPPEP -1- 끊임없이 X 선 회절 분석에 적합하지 않은 높은 (intergrown)의 결정을 제조하는 결정화 조건에서 (도 3). 따라서, 최적화 전략 (도 4)는 고품질의 결정 (도 5)을 획득하기 위해 개발되었다.
- 광고 화면을 사용 rPPEP-1의 초기 스크리닝
참고 : 표준 상업적으로 이용 가능한 스크린 및 결정화 로봇을 사용하여 앉아 드롭 형식으로 결정화 실험을 수행.- 4000 ml의 XG 4 °의 C에서 5 분 간격으로 원심 한외 여과 장치를 사용 / 12 mg의 정제 된 단백질을 농축한다. 침전 및 응집을 방지하기 위해 각 구간 후 농축 단백질 섞는다. 25,900 M -1 cm -1의 흡광 계수를 사용하여 또는 임의의 다른 방식 (예를 들어, 브래드 포드 (Bradford) 방법)에 의해 280 nm에서의 단백질 농도를 결정한다. 20 ℃로 평형 단백질. 16,000 XG에 10 분, 20 ° C에 대해 원심 분리하여 멀리 모든 입자와 먼지를 취소합니다.
- 어느 이미 사전 작성 결정화 판 자체를 사용aled와 4 ° C에서 보관 또는 모든 결정화 조건의 70 μL와 판의 저수지 우물을 입력합니다. 20 ° C까지 모든 결정화 번호판을 평형. 작은 볼륨이 빨리 건조로, 신속하게 작업 할 수 있습니다. 가능한 경우, 로봇의 독 주위 습도 챔버를 사용한다.
참고 : SaltRx, 색인, PEG / 이온, 크리스탈, 마법사, PACT ++, JCSG ++ : 표준 절차로 다음 화면을 사용합니다. - subwells 2-4로 단백질과 저수지를 피펫 팅하여 화면을 설정합니다. 이다 200 : 100 (subwell 2), 150 : 150 (subwell 3), 100 (NL)에서 200 (subwell 4) : 드롭 볼륨 300 NL과 비율 (저장 단백질)입니다. 즉시 20 ° C에서 챔버 플레이트와 장소를 밀봉.
- 셋업 후 즉시 트레이를 검사 한 다음 주간 검사 다음 첫 주 동안 매일 검사합니다.
- 리간드와 rPPEP-1의 공동 결정
- 기질 펩티드 rPPEP-1 복합체의 공동 결정화 rPPEP -1- 믹스1 24 밀리그램 / ㎖ : 1의 비율 (V / V) (12)의 최종 농도를 제공 펩티드 용액 (AC-EVNPPVPD-NH 2) TBS 버퍼에 용해하여 동결 건조 분말)의 7 배 몰 과량과 ㎎ / ㎖ R-PPEP-1 단백질과 PPEP-1을 통해 펩티드의 7 배 몰 과량. 20 ° C에서 30 분 동안 인큐베이션하고 16,000 × g으로 20 ° C에서 10 분 동안 원심 분리하여 모든 입자 및 먼지를 제거하다. 언 바운드 R-PPEP-1 단백질에 기재 한 바와 같이 결정화가 microseeding 절차를 사용하여 진행합니다.

그림 3 : 초기 화면에서 대표 결정. rPPEP-1 12 밀리그램에서에서 (intergrown)의 결정 / ml의 조건에서 재배. 나는 / 38 (1.4 M 구연산 나트륨 탈수 삼 염기, 0.1 M HEPES 나트륨의 pH 7.5, 200 NL : 100 NL) (A) 크리스탈 화면. (B) SaltRx 화면 / 52 (2.4 M 암모늄 포스페이트 DIBASIC, 0.1 M 트리스 산도 8.5; 100 NL 200 NL) 및 (C) (200 NL 100 NL). (D) SaltRx 화면 / 96 (60 % v / V를의 Tacsimate 산도 7.0, 0.1 M BIS-TRIS 프로판의 pH 7.0, 200 NL : 100 NL). 스케일 바 = 0.2 mm. 볼륨 비율은 항상 단백질과 같습니다 저수지. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
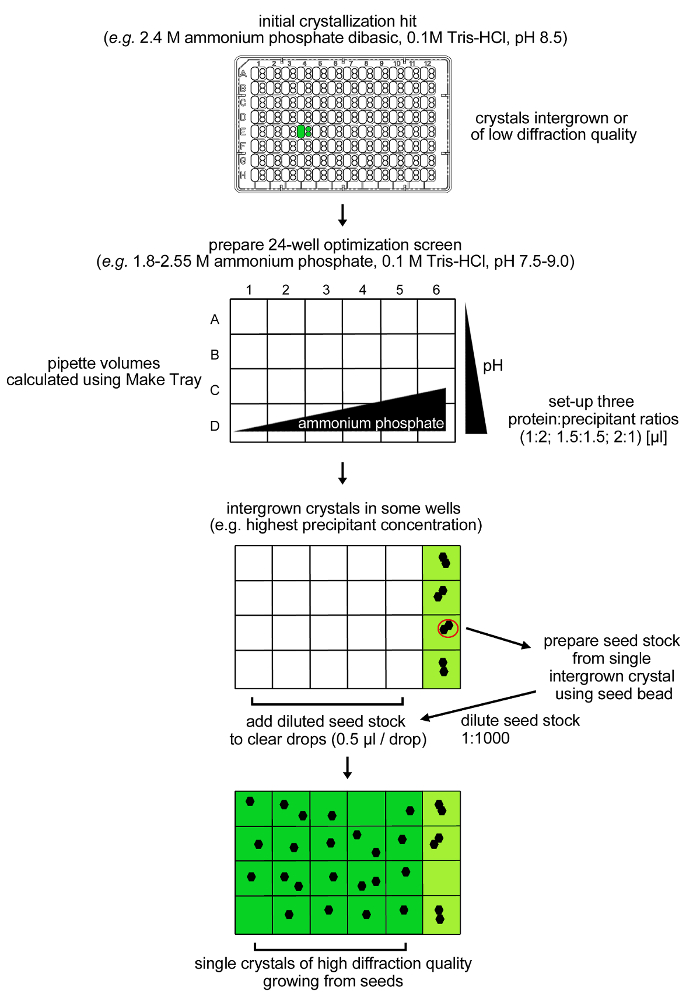
그림 4 : rPPEP-1 결정화를위한 최적화 과정. 여러 격자 ((intergrown)의) 낮은 회절 품질의 12 ㎎ / ㎖에서와 함께 rPPEP-1에서 초기 결정은 24 조건의 최적화 화면에 재현되었다. 또, 단 (intergrown)의 결정을 2.55 M 암모늄, 인산을 함유하는 상태에서 관찰되었다. 시드 주식은 단일 (intergrown)의 결정으로부터 제조 1 희석 : 1,000 동일 CONDIT로이온 (microseeding). 희석 시드 재고 0.5 μL의 체적이 남아 분명 방울로 첨가하고, 단결정의 거의 모든 조건 자랐다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- microseeding를 사용하여 크리스탈 최적화
참고 rPPEP-1의 고도 (intergrown)의 결정 2.4 M 암모늄, 인산, 0.1 M 트리스 -HCl, pH가 8.5 (SaltRx 화면 상태 E4, 세 subwells) (도 3)를 포함하는 조건 이틀 후에 나타난다. microseeding 결합 초기 상태 주변 그리드 스크린을 사용하는 최적화 절차 (도 4)을 적용 하였다.- appropri에서 (0.5 산도 단위의 단계) (0.15 M의 단계) 2.55 M 암모늄 인산 0.1 M 트리스 - 염산 pH를 7.5-9.0 - 1.8 24 조건을 포함하는 그리드 화면 (그림 4)를 준비먹었다 원액 (4 M 인산 암모늄 및 1 M 트리스 완충액).
참고 : 10 최적화 화면을 수행 할 수 있습니다 모든 조건 2 ㎖를 얻기 위해 볼륨과 피펫 팅 방식을 계산하기 위해 만들기 트레이 애플릿 (http://hamptonresearch.com/make_tray.aspx)를 사용합니다. 4 M 암모늄 포스페이트 스톡 용액을 제조하기 어렵다. 물에 완전히 녹여 분말을 교반하는 동안, 용액을 가열한다. - 피펫 (200)은 24 웰 플레이트에 각 격자 화면 용액 μL를 20 ℃에서 평형화.
- 수동으로 결정화 플레이트를 설정합니다. (μL에서) 2 : 1, 1.5 : 1 : 1.5 (: 저장 단백질) 2되는 드롭 체적은 3 μL 및 비율이다. 여기서, 기포 형성을 방지하기 위하여 용적 피펫을 사용한다. 즉시 20 ° C에서 챔버 플레이트와 장소를 밀봉. 공기 방울 형성을 피할 것.
참고 : 1 ~ 4 일 후 고도 (intergrown)의 결정은 2.55 M 암모늄 포를 포함하는 네 가지 조건에 표시sphate 이염 0.1 M 트리스 - 염산 pH를 7.5-9.0 (그림 4). 어떤 결정을 microseeding 절차는 이러한 조건에서 rPPEP-1의 단결정을 얻기 위해 사용되는 2.55 M. 이하 인산 암모늄 농도와 나머지 20 조건에 형성되지 않는다. - 2.55 M 암모늄 인산 0.1 M 트리스 - 염산 pH가 8.0 또는 8.5로 두 조건 중 하나에서 하나의 (intergrown)의 결정을 수확하여 microseed 주식을 준비합니다. 결정은 플라스틱 표면에 부착 될 수 있습니다. 침술 바늘 주위의 플라스틱 조심 변형 결정을 분리하는 데 도움이됩니다.
- 전송 작은 높은 광택 유리 구슬을 포함하는 1.5 ML 튜브에 각각 모액의 50 μL (구슬에 대한-씨). 마운트 나일론 루프를 사용하면 커버 유리 슬라이드 상에 배치 모액 1 μL로 결정 옮긴다.
- 30 초 동안 고속 튜브 및 소용돌이로 결정을 함유하는 액체를 옮긴다. 1합니다 :5 초 동안 완전히 동일한 갓 준비 상태와 소용돌이를 포함하는 새로운 1.5 ML 튜브에 씨앗 주식의 1,000 희석.
참고 : 씨앗 주식은 나중에 사용하기 위해 -80 ° C에서 저장 될 수있다.
- 우물에 (1000 희석 1) 맑은 방울과 피펫 씨 주식의 0.5 μL와 20 조건을 덮고있는 판의 봉인을 제거합니다. 20 ° C에서 챔버 플레이트와 장소를 밀봉. 높은 회절 품질의 단일 결정 2~7일 (그림 5)에 나타납니다.
- appropri에서 (0.5 산도 단위의 단계) (0.15 M의 단계) 2.55 M 암모늄 인산 0.1 M 트리스 - 염산 pH를 7.5-9.0 - 1.8 24 조건을 포함하는 그리드 화면 (그림 4)를 준비먹었다 원액 (4 M 인산 암모늄 및 1 M 트리스 완충액).
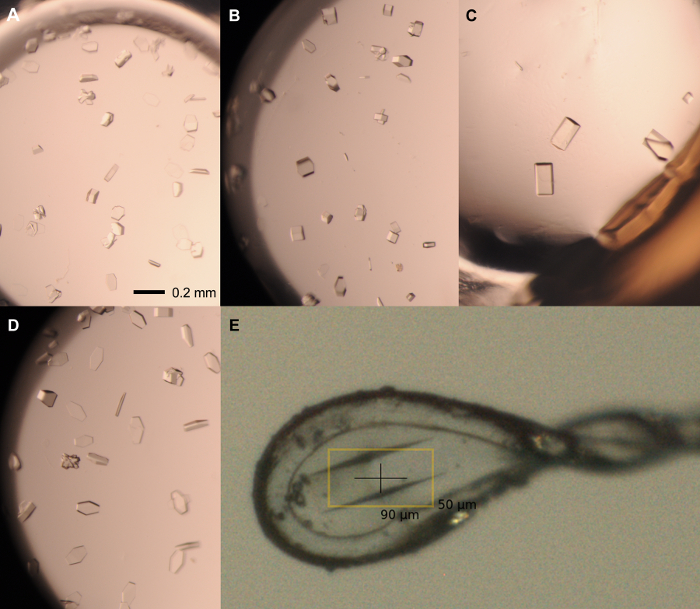
그림 5 : 최적화 화면에서 대표 결정. 12 밀리그램에서 rPPEP-1에서 단결정 / ㎖ 시드 1 : 다음과 같은 조건에서 재배 1,000 희석 종자 재고 : (A) 2.1 M 암모늄 인산, 0.1 M 트리스 산도 7.5; 1.5 μL : 1.5 μL; (B) 2.1 M 암모늄, 인산, 0.1 M 트리스 pH를 7.5; 2 μL 1 μL; (C) 2.25 M 암모늄, 인산, 0.1 M 트리스 pH를 8; 2 μL 1 μL; (D) 2.1 M 암모늄, 인산, 0.1 M 트리스 pH를 8; 1 μL : 2 μL. (E) 2.1 M 암모늄 인산 성장, 0.1-0.2 ㎛의 나일론 루프에서 결정을 개시 0.1 M 트리스 pH를 8 (2 μL 1 μL) 및 냉동 보호, 0.1 M 트리스 pH를 8 2.1 M 암모늄, 인산에서 20 % 글리세롤. 스케일 바 (AD)에서 = 0.2 mm. 볼륨 배급은 항상 단백질과 같습니다 저수지. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 크리스탈 장착 및 데이터 수집
참고 : 회절 데이터 결정의 최고의 품질은 품질과 크기의 피크에 장착해야합니다 얻으려면. 그들은 아칸소까지 결정을 액체 질소에 저장 될 수있다따라서 100 K.에서 X 선 회절 분석을 실시 예는 이들이 유래되는 질병은 극저온 상태로 조정되어야한다. rPPEP -1- 결정 크라이 보호 글리세롤 20 % 또는 30 % 수 크로즈 (크라이 오 - 보호제에 의해 상태의 물 교체)하거나 첨가하여 할 수있다.
- 크리스탈 장착
주 : 모든 결정 조작 단계는 실체 현미경 하에서 수행되어야한다.- 선택한 결정의 최대 길이 나일론 루프의 최적 크기를 선택합니다. rPPEP-1 결정의 전형적인 긴 축이 약 100 ~ 200 μm의 (그림 5)입니다. 커버 슬라이드와 해당 극저온 조건을 준비합니다 (예 : 2.1 M 암모늄 인산, 0.1 M 트리스, 산도 8.0, 20 % 글리세롤).
- 액체 질소로 거품 dewars 채우기 유리 병에 유리 병 클램프를로드하고 액체 질소로 채워진 800 ml의 거품 듀어에서 미리 냉각. 크라이 슬리브와 적절한 식별자로 표시된 크라이 지팡이 홀더를 배치액체 질소 충전이 L 폼 듀어있다. 탑재 된 나일론 루프 자기 지팡이를 넣습니다.
참고 : 액체 질소로 작업 할 때 보호 복 (eyeshield / 안경, 장갑)을 착용 할 것. 따뜻한 객체 유출을 생성 할 수 액체 질소 속으로 뛰어 들었다.
- 액체 질소로 거품 dewars 채우기 유리 병에 유리 병 클램프를로드하고 액체 질소로 채워진 800 ml의 거품 듀어에서 미리 냉각. 크라이 슬리브와 적절한 식별자로 표시된 크라이 지팡이 홀더를 배치액체 질소 충전이 L 폼 듀어있다. 탑재 된 나일론 루프 자기 지팡이를 넣습니다.
- 날카로운 메스로 밀봉 테이프를 열고 집게로 제거 잘라. 피펫 1 커버 슬라이드 상에 극저온 상태의 μL (또는 대안도 같은 접시에 빈에서) 및 장착 된 나일론 루프 (그림 5)와 함께 낚시를하여 드롭 결정을 제거합니다. 첨부 결정은 쉽게 침술 바늘로 주변의 플라스틱을 변형시켜 지상에서 분리 할 수 있습니다.
- 신속하게 냉동 조건의 하락 결정을 전송하고 1 초 동안 평형 수 있습니다. 액체 질소에서 가능하고 플 런지 동결 한 빨리 결정을 물고기.
- 장착 된 루프 주위의 액체 질소가 끓는하지 않을 때, 유리 병에 루프를 배치합니다.크라이 지팡이 홀더에 병을 놓고 6 병로드 할 때 홀더 주위에 냉동 슬리브를 배치합니다. 사용할 때까지 액체 질소로 채워진 탱크에 결정을 저장합니다.
- 선택한 결정의 최대 길이 나일론 루프의 최적 크기를 선택합니다. rPPEP-1 결정의 전형적인 긴 축이 약 100 ~ 200 μm의 (그림 5)입니다. 커버 슬라이드와 해당 극저온 조건을 준비합니다 (예 : 2.1 M 암모늄 인산, 0.1 M 트리스, 산도 8.0, 20 % 글리세롤).
- 데이터 수집
참고 : 데이터 수집이 가능한 경우, 홈 회절에서 수행, 또는 싱크로트론 빔라인에서 할 수 있습니다. rPPEP 1 데이터 하이브리드 광자 카운트 검출기를 사용 스위스 광원 폴 - 셰러 연구소 Villigen, 스위스 빔라인 X06DA 수집 된 내용. 구조 결정에 사용 된 원래의 데이터의 모든 파일 요청에 따라 제공 될 수있다.- 소자 아연의 특성 X 선 흡수 단 에너지 (피크)은 1.282 Å (9667 keV의)에 대한 빔의 파장을 설정한다. rPPEP-1 활성 부위에서 분자 당 하나의 아연을 포함하는 메탈로한다.
- 각 directio가 270 °로, 총 10 ° 웨지의 역 빔 모드에서 100 K에서 데이터를 수집엔. 노출 시간은 이미지 당 0.1 ° 회전에 0.1 초이다. 14 % (0.14)로 전송을 설정합니다.
- 같은 결정화 조건에서 발생하는 두 번째 결정에서 네이티브 고해상도 데이터 세트를 수집하려면 1.00 Å (12,398 케빈)에 빔의 파장을 설정합니다. 노출 시간은 이미지 당 0.1 ° 회전에 0.1 초 100 K.에서 데이터를 수집합니다. 70 % (0.7)로 전송을 설정합니다.
아연 SAD를 통해 5. 구조 결정
주 : 기본적인 결정 기술이 필요 아연 SAD뿐만 아니라 소프트웨어 패키지 XDS 20 피닉스 (21)과 프로그램을 통해 닭 22 rPPEP-1의 구조를 결정하기 위해. 구조의 시각화를위한 프로그램 파이 몰 (23) 또는 키메라 (24)이 필요하다. 소자 아연의 흡수 단에서의 피크에 대응하는 파장에서 수집 된 데이터는 단일 파장 변칙 DISP 사용될 수있다ersion (SAD) (25)는 모두 단백질 원자 연장 될 수 위상 정보를 획득한다.
- 데이터 처리
- 프리델의 메이트 (비정상 데이터)를 분리하는 공간 군 P2 1 2 1 2 1 (공간 군 19) 소프트웨어 XDS (또는 iMosflm 또는 HKL3000)를 사용하여 (정상 및 역) 두 피크 데이터 세트를 처리한다. 단위 셀 파라미터는 A, B, C의 (a)의 주위에 있어야 = 43.17은 71.68, 117.70 및 α = β = γ (°) = (90)이 두 HKL - 파일 (반사 파일)을 제공한다.
- 파일 CORRECT.LP을 검사합니다. 공통 1/2이 적어도 50 % 인 해상도까지 데이터를 사용합니다. 함께 엑스 스케일을 사용하여 두 데이터 세트 / 반사 파일 (HKL-파일)을 확장 할 수 있습니다. 파일 XSCALE.LP을 검사합니다. 변칙 신호 (SigAno) 확장 얼마나 확인하고 여기에 사용되는 1.67 Å에 수집 된 데이터의 경우 2를 약 30 %의 변칙적 인 상관 관계 (Anomal 자료 Corr)와 해상도를 확인합니다. 이것이피닉스 AutoSol에 사용되는 비정상적인 신호의 해상도 차단.
- XDSCONV 5 %의 R 무료 집합을 생성하고 비정상적인 데이터를 (FRIEDEL'S_LAW = FALSE) 유지 사용 (peak_anom.mtz, 예를 들어, 이름)을 CCP4 형식의 반사 파일로 (스케일링) HKL 파일을 변환합니다. 단위 셀 매개 변수 공간 그룹과 R 무료 집합의 존재 (라벨 FreeRflag)과 비정상적인 데이터를 검사 프로그램 mtzdmp과의 일관성에 대한 MTZ-파일을 확인 (단오 / SIGDANO 레이블). 변칙 데이터 추출하지 않고 또한 XDSCONV에 추가 MTZ-파일을 준비합니다 (FRIEDEL'S_LAW = TRUE를, 이름, 예를 들어, peak_native.mtz에 대해) 정제에 대한 나중 단계에서.
- 하부 용액 (상 결정)
- 반사 파일 peak_anom.mtz을 사용하여 피닉스 AutoSol를 실행합니다. 데이터 유형으로 SAD / MAD 피크를 선택하고 (비대칭 유닛 당 두 개의 분자가 있기 때문에) 2 아연 사이트를 선택합니다. 더 정확한 체험관 중 하나를 선택F f를 '/ F' '(빔라인에서 형광 검사에서 결정) 매개 변수 또는 theotetical 값'= -8.245이고 f ''= 3.887위한 정신 값. 또한, 결정화 된 단백질의 아미노산 서열을 포함하는 FASTA 파일을로드.
- 이 경우 2 년, (5.1.2 결정) 약 30 %의 변칙적 인 상관 관계 (Anomal 자료 Corr)와 해상도로 제한을 설정하고 "자동 빌드 모델"옵션을 선택합니다. 전자 밀도로 전체 단백질 위상이 도출 될 수 피닉스 HySS합니다 (피닉스 AutoSol 파이프 라인의 일부)에 의해 발견 된 두 개의 사이트와 아연 (피닉스의 RESOLVE함으로써) 구축 된 모형의 위상 사용. 가장 좋은 모델은 "overall_best.pdb"라고합니다.
- 모델 건물, 정제 및 검증
- 자동 rPPEP-1 모델의 대부분을 구축 할 수있는 옵션이 "자동 빌드 모델"을 선택합니다. 프로그래밍을 사용하여 1.0 σ 형상 레벨에서 전자 밀도를 점검m 닭 (그림 6). 이 결합하고 모델의 원자 주위되어야한다. 이상적으로 일부 물 분자는 (2.5 Å보다 더 나은 해상도) 모델에 내장되어야한다. 대량 물 (분자 사이의 공간)에는 밀도가 포함되어야합니다.
- 전체 모델이 완료 (전자 밀도에 내장 된 모든 아미노산을)인지 확인합니다. 그렇지 않은 경우, 수동으로 닭에서 제공하는 도구를 사용하여 구축 할 수 있습니다. 5 정제와 피닉스 발견의 반복적 인 순찰을 실행하여 구조를 수정하는 것은 각각 overall_best.pdb 모델 파일의 peak_native.mtz 반사 파일과 FASTA 시퀀스 파일을 사용하여 라운드; 그리고 닭에서 수동 모델 건물입니다.
- 닭의 각 도구를 사용하여 구조 모델의 품질을 확인합니다.
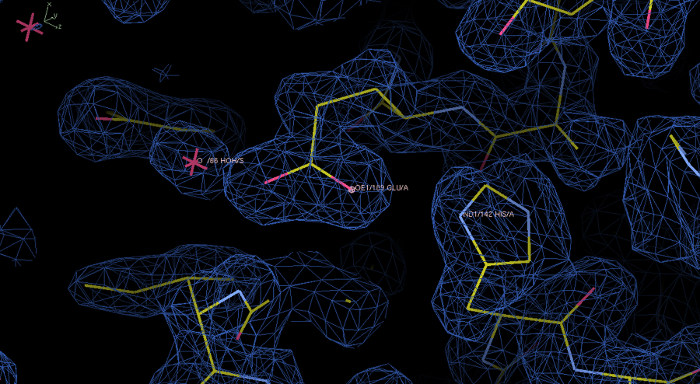
애프터 실험 전자 밀도지도 rPPEP-1 모델 :도 6피닉스 Autosol 실행. 프로그램 닭에 표시 1.0 σ의 윤곽 수준에서 파란색 전자 밀도. 초기지도의 전자 밀도 멋지게 해결되고,이 모델은 전자 밀도에 구축. 줌에서이 잔기와 His142 Glu189뿐만 아니라, 물 분자를 나타낸다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
분자 교체를 통해 고해상도 6. 구조 결정
참고 rPPEP-1 기본 세트에 대해 고해상도의 구조 정보를 얻기 위해 수집된다. 이어서합니다 (피닉스 소프트웨어 패키지 내에서) 소프트웨어 페이저 (26, 27)를 이용한 분자 교환 순서 아연 SAD 모델 등을 통해 결정 구조를 이용하여 채용된다. 소분자와 복합 rPPEP-1의 구조를 풀 때이 절차는 나중에 사용될 수있다.
- (1.4 Å이 경우까지)보다 높은 해상도를 가진 결정 구조를 얻기 위해 공간 군 P2 1 2 1 2 1 (공간 군 19)의 소프트웨어 XDS (또는 iMosflm 또는 HKL3000)를 사용하여 기본 데이터 세트를 처리한다. 단위 셀 파라미터는 A, B, C의 (a)의 주위에 있어야 = 43.17은 71.77, 117.80 및 α = β = γ (°) = (90)이 하나 HKL - 파일 (반사 파일)을 제공한다.
- 파일 CORRECT.LP을 검사합니다. 공통 1/2이 적어도 50 % 인 해상도까지 데이터를 사용합니다. XDSCONV 5 %의 R 무료 일부를 작성, 사용 (native.mtz, 예를 들어, 이름)을 CCP4 형식의 반사 파일로 HKL 파일을 변환합니다. 단위 셀 매개 변수 공간 그룹과 R 무료 집합 (라벨 FreeRflag)의 존재를 검사 프로그램 mtzdmp과의 일관성에 대한 MTZ-파일을 확인하십시오.
- (이전 결정 overall_best.pdb에서 모델을 포함하는 PDB 파일을 준비하고 모든 물 분자와 모든 리간드를 제거 즉,. 아연 원자). 또한, 결정화 된 단백질의 아미노산 서열을 포함하는 FASTA 파일을로드. 반사 파일 native.mtz을 사용하여 피닉스에 페이저를 실행합니다. 비대칭 유닛 당 두 개의 분자를 검색합니다.
- 모델 (이름 native_phaser.1.pdb)와 닭의 전자 밀도지도를 검사, 성공적인 구조 용액 (여기에 10.2 8보다 큰 TFZ 점수) 후. 빌드 5 세련미와 피닉스 발견의 반복적 인 순찰을 실행하여 구조를 수정 각각 native_phaser.1.pdb 모델 파일의 native.mtz 반사 파일과 FASTA 시퀀스 파일을 사용하여 라운드; 그리고 닭에서 수동 모델 건물입니다.
- 닭의 각 도구를 사용하여 구조 모델의 품질을 확인합니다.
결과
rPPEP-1은 대장균 BL21 (DE3) 스타 (도 1C)에서 가장 높은 수율로 여러 대장균 균주에서 과발현된다. 제 NiNTA 친 화성 크로마토 그래피 단계 후 6xHis 태그가 성공적으로 단백질의 대부분으로부터 절단 될 수 있으며, 상기 제 NiNTA 단계에서 소화되지 않은 단백질은 완전히 트롬빈 분해 단백질 (도 1D)로부터 분리 될 수있다. 가끔 대부분의 아마 ...
토론
X 선 결정학은 여전히 단백질 (28)의 입체에 가까운 해상도 원자 구조를 결정하기위한 가장 빠르고 가장 정확한 방법이다. 그러나, 잘 정렬 된 단결정의 성장을 필요로한다. 이들은 종종 얻을 수 어렵고, 결정 상태는 인공이다. 그러나, 다른 방법에 의해 결정된 것과 X 선 결정학에 의해 결정된 구조 단백질의 비교 특히 NMR, 일반적으로 매우 잘 일치 함을 나타낸다. PPEP-1의 경우에, NMR 구조는 ...
공개
The authors have nothing to disclose.
감사의 말
우리는 싱크로트론 데이터 수집시 지원을위한 스위스 광원, 폴 Scherrer의-연구소, Villigen, 스위스의 빔라인의 X06DA에서 직원을 감사드립니다. 우리는 우수한 기술 지원 모니카 Gompert에 감사하고 있습니다. 이 프로젝트는 쾰른 대학에서 지원과 독일 연구 협의회에서 INST 216 / 682-1 FUGG을 부여했다. CP에 개발 건강과 질병의 국제 대학원에서 박사 교제는 인정된다. 이러한 결과로 이어지는 연구는 보조금 계약 번호 283570 (BioStruct-X)에서 유럽 공동체의 일곱 번째 프레임 워크 프로그램 (FP7 / 2007-2013)에서 자금을 받았다.
자료
| Name | Company | Catalog Number | Comments |
| Genes / Vectors / cell strains | |||
| pET28a vector | Merck-Millipore | 69864 | Thrombin cleavable N-terminal His-tag |
| E. coli strain BL21 (DE3) Star | ThermoFisher Scientific | C601003 | RNase H deficient |
| Codon-optimized gene (for E. coli) of PPEP-1 (CD630_28300) | Geneart (Thermo Fisher Scientific) | custom | amino acids 27-220 |
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Yeast extract | any | ||
| Tryptone | any | ||
| Antifoam B | Sigma-Aldrich | A5757 | aqueous-silicone emulsion |
| Agar | any | ||
| Kanamycin | any | ||
| IPTG | AppliChem | A1008 | |
| Tris-HCl | AppliChem | A1087 | Buffer grade |
| NaCl | any | Buffer grade | |
| DNaseI | AppliChem | A3778 | |
| Imidazole | AppliChem | A1073 | Buffer grade |
| Thrombin | Sigma-Aldrich | T4648 | |
| Ammonium phosphate dibasic | Sigma-Aldrich | 215996 | |
| Glycerol 100% | any | purest grade | |
| Sucrose | Sigma-Aldrich | 84097 | |
| Liquid nitrogen | any | for storage and cryocooling of crystals | |
| Name | Company | Catalog Number | Comments |
| Equipment (general) | |||
| Shaking incubator | any | providing temperatures of 20 °C - 37 °C | |
| Glassware | any | baffled Erlenmeyer flasks (50 ml - 2.8 L) | |
| Centrifuge for large culture volumes | any | centrifuge for processing volumes up to 12 L | |
| Sonicator Vibra-Cell VCX500 | Sonics | SO-VCX500 | or any other sonicator / cell disruptor |
| Ultracentrifuge | any | centrifuge providing speeds up to 150,000 x g | |
| NiNTA Superflow resin | Qiagen | ||
| Empty Glass Econo-Column | Bio-Rad | 7371007 | or any other empty glass or plastic column |
| Size exclusion chromatography column HiLoad Superdex 200 16/600 | GE Healthcare | 28989335 | |
| Chromatography system Äkta Purifier | GE Healthcare | 28406264 | or any other chromatography system |
| Dialysis tubing Spectra/Por 3 | Spectrum Labs | 132724 | |
| Dialysis tubing closures | Spectrum Labs | 132738 | |
| Ultrafiltration units (concentrators) 10,000 NWCO | any | ||
| UV-Vis spectrophotometer | any | ||
| Name | Company | Catalog Number | Comments |
| Equipment (crystallography) | |||
| Low volume pipette 0.1-10 µl | any | ||
| Positive displacement pipette Microman M10 | Gilson | F148501 | |
| Crystallization robot | any | ||
| 96-well crystallization plates TTP IQ with three protein wells | TTP | 4150-05810 | or any other 96-well crystallization plate |
| 24-well CombiClover Junior Plate | Jena Bioscience | EB-CJR | |
| Crystal Clear Sealing Tape | Hampton Research | HR3-511 | |
| Siliconized Glass Cover Slides | Hampton Research | HR3-225 | |
| Commercial crystallization screens: SaltRx, Index, PEG/Ion, Crystal | Hampton Research | diverse | |
| Commercial crystallization screens: Wizard, PACT++, JCSG++ | Jena Bioscience | diverse | |
| JBS Beads-for-Seeds | Jena Bioscience | CO-501 | |
| CrystalCap SPINE HT (nylon loops) | Hampton Research | diverse | loop sizes 0.025 mm - 0.5 mm |
| CrystalCap Vial | Hampton Research | HR4-904 | |
| Cryogenic Foam Dewar 800 ml | Hampton Research | HR4-673 | |
| Cryogenic Foam Dewar 2 L | Hampton Research | HR4-675 | |
| Vial Clamp, Straight | Hampton Research | HR4-670 | |
| CrystalWand Magnetic, Straight | Hampton Research | HR4-729 | |
| CryoCane 6 Vial Holder | Hampton Research | HR4-711 | |
| CryoSleeve | Hampton Research | HR4-708 | |
| CryoCane Color Coder - White | Hampton Research | HR4-713 | |
| Scalpel | any | ||
| Straight microforcep | any | for manipulation of sealing tape. etc. | |
| Acupuncture needle | any | e.g. from a pharmacy | |
| Stereo microscope | any | for inspection of crystallization plates and crystal mounting, magnification up to 160X |
참고문헌
- Bouza, E. Consequences of Clostridium difficile infection: understanding the healthcare burden. Clin Microbiol Infect. 18 (Suppl 6), 5-12 (2012).
- O'Connor, J. R., Johnson, S., Gerding, D. N. Clostridium difficile infection caused by the epidemic BI/NAP1/027 strain. Gastroenterology. 136 (6), 1913-1924 (2009).
- Mitchell, B. G., Gardner, A. Mortality and Clostridium difficile infection: a review. Antimicrob Resist Infect Control. 1 (1), (2012).
- George, W. L., Sutter, V. L., Finegold, S. M. Antimicrobial agent-induced diarrhea--a bacterial disease. J Infect Dis. 136 (6), 822-828 (1977).
- George, R. H., et al. Identification of Clostridium difficile as a cause of pseudomembranous colitis. Br Med J. 1 (6114), 695 (1978).
- Bartlett, J. G. Narrative review: the new epidemic of Clostridium difficile-associated enteric disease. Ann Intern Med. 145 (10), 758-764 (2006).
- Kelly, C. P., LaMont, J. T. Clostridium difficile--more difficult than ever. N Engl J Med. 359 (18), 1932-1940 (2008).
- Ünal, C. M., Steinert, M. Novel therapeutic strategies for Clostridium difficile infections. Expert Opin Ther Targets. 20 (3), 269-285 (2016).
- Kelly, C. P., et al. The Monoclonal Antibody, Bezlotoxumab Targeting C. difficile Toxin B Shows Efficacy in Preventing Recurrent C. difficile Infection (CDI) in Patients at High Risk of Recurrence or of CDI-Related Adverse Outcomes. Gastroenterology. 150 (4), S122 (2016).
- Tsutsumi, L. S., Owusu, Y. B., Hurdle, J. G., Sun, D. Progress in the discovery of treatments for C. difficile infection: A clinical and medicinal chemistry review. Curr Top Med Chem. 14 (1), 152-175 (2014).
- Hensbergen, P. J., et al. Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831. FEBS Lett. 589 (24), 3952-3958 (2015).
- Cafardi, V., et al. Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins. PLoS One. 8 (11), e81306 (2013).
- Hensbergen, P. J., et al. A novel secreted metalloprotease (CD2830) from Clostridium difficile cleaves specific proline sequences in LPXTG cell surface proteins. Mol Cell Proteomics. 13 (5), 1231-1244 (2014).
- Schacherl, M., Pichlo, C., Neundorf, I., Baumann, U. Structural Basis of Proline-Proline Peptide Bond Specificity of the Metalloprotease Zmp1 Implicated in Motility of Clostridium difficile. Structure. 23 (9), 1632-1642 (2015).
- Rawlings, N. D., Waller, M., Barrett, A. J., Bateman, A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors. Nucleic Acids Res. 42 (Release 10.0), D503-D509 (2014).
- Bergfors, T. Seeds to crystals. J Struct Biol. 142 (1), 66-76 (2003).
- Dauter, Z., Dauter, M., Dodson, E. Jolly SAD. Acta Crystallogr D Biol Crystallogr. 58 (Pt 3), 494-506 (2002).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr. 66 (Pt 2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D Biol Crystallogr. 66, 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 486-501 (2010).
- . . The PyMOL Molecular Graphics System. , (2002).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25 (13), 1605-1612 (2004).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods Enzymol. 115, 90-112 (1985).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Zwart, P. H., et al. Automated structure solution with the PHENIX suite. Methods Mol Biol. 426, 419-435 (2008).
- Zheng, H., Handing, K. B., Zimmerman, M. D., Shabalin, I. G., Almo, S. C., Minor, W. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Discov. 10 (9), 975-989 (2015).
- Rubino, J. T., et al. Structural characterization of zinc-bound Zmp1, a zinc-dependent metalloprotease secreted by Clostridium difficile. J Biol Inorg Chem. 21 (2), 185-196 (2016).
- Carson, M., Johnson, D. H., McDonald, H., Brouillette, C., Delucas, L. J. His-tag impact on structure. Acta Crystallogr D Biol Crystallogr. 63 (Pt 3), 295-301 (2007).
- Gasteiger, E., Walker, J. M., et al. Protein Identification and Analysis Tools on the ExPASy Server. The Proteomics Protocols Handbook. , 571-607 (2005).
- Dummler, A., Lawrence, A. M., de Marco, A. Simplified screening for the detection of soluble fusion constructs expressed in E. coli using a modular set of vectors. Microb Cell Fact. 4, 34 (2005).
- Stura, E. A., Wilson, I. A. Applications of the streak seeding technique in protein crystallization. J Crys Growth. 110 (1), 270-282 (1991).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유