JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
광 반응성 고분자 나노 캐리어로부터의 siRNA 방출의 시공간 조절을 통한 유전자 침묵 예측
요약
우리는 광 반응성 블록 공중 합체를 사용하는 새로운 방법을 제시한다.이 방법은 검출 불가능한 오프 - 타겟 효과없이 유전자 침묵을보다 효율적으로 공간적으로 제어 할 수있다. 또한 유전자 발현의 변화는 간단한 siRNA 방출 분석 및 간단한 운동 모델링을 사용하여 예측할 수 있습니다.
초록
새로운 물질 및 방법은 유전자 활동의 정확한 조절을 필요로하는 광범위한 적용을위한 핵산의 결합 대 방출을보다 잘 제어하기 위해 필요합니다. 특히, 유전자 발현에 대한 시공간적 제어가 개선 된 새로운 자극 - 반응 물질은 약물 발견 및 재생 의학 기술에서 번역 가능한 플랫폼의 잠금을 해제합니다. 또한, 물질로부터의 핵산 방출을 제어하는 능력이 향상되면 나노 캐리어 효능 을 선험적 으로 예측할 수있는 능률적 인 방법의 개발이 가능 해져 배달 차량의 신속한 스크리닝이 가능해진다. 여기, 우리는 유전자 침묵 효율을 예측하고 모듈 식 광 반응성 나노 캐리어 시스템을 통해 유전자 발현에 대한 시공간 제어를 달성하기위한 프로토콜을 제시한다. 작은 간섭 RNA (siRNA)는 mPEG- b -poly (5- (3- (아미노) 프로 폭시) -2- 니트로 벤질 메타 크릴 레이트) (mPEG- β -P (APNBMA)빛으로 조절되어 조정 가능한 ON / OFF siRNA 방출을 촉진 할 수있는 안정한 나노 캐리어입니다. 우리는 세포 내 환경을 모방 솔루션에서 siRNA 릴리스의 정확한 부량을위한 형광 상관 분광법과 겔 전기 영동을 사용하는 두 보완 assays을 설명합니다. 이 분석에서 얻은 정보는 다양한 광 자극 조건에 대한 동적 침묵 반응을 예측하기위한 간단한 RNA 간섭 (RNAi) 동역학 모델에 통합되었습니다. 이어서, 이러한 최적화 된 조사 조건은 시공간적으로 유전자 침묵을 제어하기위한 새로운 프로토콜의 개선을 가능하게했다. 이 방법은 cell-to-cell 분해능과 off-target 효과가없는 유전자 발현의 세포 패턴을 생성 할 수 있습니다. 종합하면, 우리의 접근 방식은 유전자 발현의 역동적 인 변화를 예측하고 시공간적으로 siRNA 활동을 정밀하게 제어 할 수있는 사용하기 쉬운 방법을 제공합니다. 이 assays 세트는 다양한 ot를 테스트하기 위해 쉽게 적용 할 수 있습니다그녀의 자극 반응 시스템은 생물 의학 연구 및 의학 분야의 수많은 어플리케이션과 관련된 핵심 과제를 해결합니다.
서문
small interfering RNAs (siRNAs)는 사실상 모든 표적 유전자 1에 대해 매우 특이적이고 강력하며 맞춤형 인 촉매 RNAi 경로를 통해 전사 후 유전자 사일런 싱을 매개한다. 이러한 유망한 특성으로 인해 전이성 흑색 종 및 혈우병을 비롯한 수많은 질병의 치료를위한 인간 임상 시험에서 siRNA 치료제가 발전 할 수있었습니다 2 , 3 . 그러나 번역을 방해하는 중요한 배달 문제가 계속 발생합니다 4 . 특히, 운반체는 안정을 유지해야하며 세포 외 분해로부터 siRNA를 보호해야하며, 또한 세포질에 유핵 적재 물을 방출해야합니다 5 . 또한, 많은 RNAi 응용 프로그램은 siRNA 치료법 7 에서 부작용을 줄이고 변형 가능하게하는 공간 및 시간 6 에서 유전자 침묵을 조절하는 향상된 방법을 필요로합니다.약 발견 8 을위한 세포 microarrays에서 재생성 비계 9 에있는 세포 반응의 조음에 배열하는 신청에있는 dvances. 이러한 과제는 siRNA 나노 캐리어에서 결합 대 방출을보다 잘 제어 할 수있는 새로운 물질 및 방법의 필요성을 강조합니다.
siRNA 방출을 제어하고 시공간 조절을 향상시키는 가장 유망한 전략 중 하나는 자극 반응 물질의 사용입니다 10 . 예를 들어, 생체 물질의 다양한 변화는 핵산 변형 산화 환원 전위 나 산도 또는 자장인가, 초음파 또는 광 (11)에 대응하여 결합 친 화성을 함께 설계 하였다. 이들 시스템 중 다수가 핵산 활성에 대한 개선 된 제어를 입증하지만, 트리거로서의 광의 사용은 순간적 시간 반응, 정확한 공간 분해 및 조율 용이성으로 인해 특히 유리하다 12. 또한, 유전자 발현 조절을위한 사진 민감 기술의 잠재력은 최첨단의 유도 성 프로모터 및 광 발생 조절 시스템에 의해 입증되었다. 그러나 이러한 시스템은 내인성 유전자를 규제하기위한 제한된 용량, 면역 원성과 같은 안전성 문제, 다중 부품 조립품의 운반이 어렵다는 것을 비롯하여 수많은 어려움을 겪고 있습니다 13 , 14 , 15 . 광 반응성 siRNA 나노 캐리어는 이러한 결점을 극복하고 유전자 발현을 공간적으로 조절하는보다 간단하고 견고한 접근법을 제공하는데 이상적으로 적합합니다 16 , 17 , 18 . 불행하게도, 결과 단백질의 녹다운 반응을 정확하게 예측하는 방법은 아직 파악하기 어렵습니다.
핵심 과제는 siRNA 방출의 정량적 평가가희귀 19 , 20 및이 평가가 수행 되더라도 siRNA / 단백질 회전율 역학의 분석에 결합되지 않았습니다. siRNA의 방출량과 지속성 / 수명은 생성 된 유전자 침묵 동역학의 중요한 결정 인자입니다. 따라서 그러한 정보가 부족하면 RNAi 21 의 용량 반응의 정확한 예측을 방해하는 주요 단절이 있습니다. 이러한 문제를 해결하기 nanocarriers의 적절한 구조 함수 관계의 형성을 촉진하고 더 바이오 물질 (22)을 설계 알리는 것이다. 또한, 이러한 접근법은보다 효과적인 siRNA 투약 프로토콜의 개발을 가능하게 할 것이다. 역동적 인 사일런스 반응을 이해하기 위해 여러 그룹이 RNAi 23 , 24 , 25 의 수학적 모델을 조사했습니다. 이 프레임 워크는유전자 발현의 siRNA 매개 된 변화에 대한 통찰력을 제공하고 속도 - 제한 단계 (26)를 식별하는 데 성공. 그러나, 이러한 모델은 제어 siRNA를 방출 할 수없는 상업적인 유전자 전달 시스템 (예를 들어, 리포 펙 타민 및 폴리에틸렌 이민 (PEI))에 적용되어, 상기 모델의 복잡성이 크게 그들의 유틸리티 (27)를 한정하고있다. 이러한 단점은 간결하고 사용하기 쉬운 예측 동역학 모델과 결합하여 정확하게 조정할 수있는 새로운 물질에 대한 충족되지 않은 요구를 강조합니다.
우리의 방법은 빛에 민감한 나노 캐리어 플랫폼과 결합 된 방법을 통합하여 자유로운 siRNA와 모델 RNAi 역학을 정량화함으로써 이러한 모든 문제를 해결합니다. 특히, 우리 플랫폼의 정확하게 제어 된 siRNA 릴리스 28 은 캡슐화 된 대 양성자를 정확하게 정량하기위한 두 가지 보완적인 방법으로 모니터링됩니다결합 된 siRNA. 이 분석에서 실험 데이터는 유전자 침묵 효율성 선험적 (29)을 예측하는 간단한 운동 모델로 입력됩니다. 마지막으로, nanocarriers의 온 / 오프 특성은 쉽게 세포 길이 규모에 공간 제어와 유전자 표현에 세포 패턴을 생성하는 데 활용됩니다. 따라서,이 방법은 세포 행동의 시공간 조절로부터 이익을 얻는 다양한 응용에서 유전자 침묵을 제어 및 예측하는 용이하게 적응 가능한 방법을 제공한다.
프로토콜
1. siRNA 나노 캐리어의 제형
- pH 6.0에서 20mM 4- (2- 하이드 록시 에틸) 피페 라진 -1- 에탄 설 폰산 (HEPES) 버퍼로 희석 된 동일한 부피의 siRNA 및 mPEG- b -P (APNBMA)의 개별 용액을 준비한다.
- 20 MM HEPES 솔루션에 32 μg / ML의 농도 siRNA를 추가합니다.
참고 : siRNA는 비 표적화 된 보편적 음성 대조군 시퀀스였다. 그러나, siRNA는 관심있는 모든 유전자를 표적으로하도록 설계 될 수 있습니다. - 20 mM HEPES 용액에 mPEG- b -P (APNBMA) 중합체를 용해시킨다. N / P 비 (N, mPEG- b -P (APNBMA)의 아민 기, P, siRNA상의 인산기)가되도록 220 μg / mL 용액을 만들기 위해 적절한 양의 mPEG- b -P (APNBMA) 4입니다.
참고 : mPEG- b -P (APNBMA) 고분자의 합성 프로토콜은 다른 곳에서보고됩니다 30 .
- 20 MM HEPES 솔루션에 32 μg / ML의 농도 siRNA를 추가합니다.
- mPEG- b -P (APNBMA) 용액을 같은 부피e를 siRNA 용액에 넣고 vortex machine으로 부드럽게 섞는다. 폴리머 첨가 후 30 초 동안 계속해서 볼 텍싱하십시오. 어두운 온도에서 30 분간 상온에서 시료를 품는다.
2. 겔 전기 영동을 이용한 siRNA 방출 측정
- 단계 1.1-1.2에 따라 나노 캐리어를 포 뮬레이션하고 원하는 샘플 수를 수용하도록 부피를 측정하십시오.
- 나노 캐리어를 sodium dodecyl sulfate (SDS)와 섞는다.
- 물에서 SDS의 1 MG / ML 솔루션을 준비합니다. S / P 비 (S, SDS상의 황산염 그룹, P, siRNA의 인산염 그룹)가 15 인 솔루션을 생산하는 데 필요한 SDS 솔루션의 양을 나누십시오.
참고 : 폴리 플렉스 용액에 1 μg의 siRNA가 포함되어있는 경우 13 μg의 SDS를 추가해야 S / P 비율이 15가됩니다. - 소용돌이 기계에서 부드럽게 혼합하면서 각 나노 캐리어 용액에 SDS 용액을 적가한다. SDS 첨가 후 30 초 동안 계속해서 볼 텍싱하십시오.
- 시료를 3,000 xg에서 5 초간 원심 분리하십시오. 어두운 온도에서 30 분간 상온에서 시료를 품는다.
- 물에서 SDS의 1 MG / ML 솔루션을 준비합니다. S / P 비 (S, SDS상의 황산염 그룹, P, siRNA의 인산염 그룹)가 15 인 솔루션을 생산하는 데 필요한 SDS 솔루션의 양을 나누십시오.
- 365 nm 필터를 사용하여 200 W / m의 강도로 UV 레이저를 보정하고 설정하십시오. 빛의 세기는 샘플 용액의 바닥이 고정되는 위치에서 측정해야합니다.
- 고무 개스킷으로 분리 된 유리 슬라이드로 구성된 유리 챔버에 나노 캐리어 / SDS 용액을로드하십시오.
- 사전 세척 유리는 물에서 7 : 3 (v / v) 에탄올 / 물 용액으로 미끄러 져 완전히 건조됩니다. 고무 가스켓에 구멍 (~ 2 x 3cm 직사각형)을 자릅니다. 유리 슬라이드 위에 고무 가스켓을 놓습니다.
- 고무 가스켓의 구멍 안쪽 유리 슬라이드에 나노 캐리어 / SDS 솔루션을 피펫. 고무 가스켓과의 접촉을 피하면서 과량의 용액 (잉여의 20 μL)을 유리 슬라이드 위에 올려 놓습니다.
참고 : 일부 액체는 후속 단계에서 없어집니다. - 두 번째 유리 놓기슬라이드 개스킷 상단에서 미끄러 져 움직입니다. 기포 발생을 방지하려면 슬라이드의 한쪽 끝을 먼저 내린 다음 다른 끝을 천천히 내리십시오.
- 바인더 클립을 유리 챔버의 양쪽에 부착하여 닫습니다.
- 200 W / m의 강도에서 365 nm 필터를 사용하는 UV 레이저를 사용하여 원하는 시간 ( 예 : 0-60 분) 샘플을 조사하십시오. 바인더 클립을 제거하고 챔버를 엽니 다.
- 25μL의 조사 된 나노 캐리어 / SDS 샘플을 마이크로 원심 분리기 튜브에 피펫 팅한다. 어두운 방에 30 분 동안 실온에서 용액을 품는다.
- 표준 프로토콜 31 에 따라 트리스 / 보레이트 / EDTA (TBE) 버퍼 솔루션 0.5 μg / ML 에티 디움 브로마이드로 사전 얼룩이 2 % 아가로 오스 겔을 준비합니다. 3 : 7 (v / v) 글리세롤 / 물로 구성된 로딩 완충액을 준비하십시오.
- 각 nanocarrier / SDS 샘플 25 μL에 로딩 버퍼 솔루션 5 μL를 추가합니다. 어둠 속에서 샘플을 부화 시키십시오.실온에서 10 분.
- 각 nanocarrier / SDS 샘플 30 μL 2 % 아가로 오스 젤에로드합니다. 어둠 속에서 겔을 100V에서 30 분 동안 작동시킵니다. 에 티듐 브로마이드 필터가있는 겔 이미징 시스템을 사용하여 이미지를 만듭니다. 겔 이미지 파일을 저장하고 밴드 강도 정량화를 위해 단계 2.10으로 진행하십시오. 밴드 강도가 명확하게 시각화 할 수있을 정도로 밝지 만 너무 밝지 않아서 신호가 포화되어 있는지 확인하십시오.
- 공개적으로 사용 가능한 ImageJ 소프트웨어 32를 사용하여 밴드 강도를 정량화하십시오.
- ImageJ의 ROI 도구를 사용하여 각 밴드 주위에 직사각형을 그려서 각 레인의 자유 siRNA 밴드의 형광 강도를 결정합니다. 각 차선의 강도 곡선을 플롯하고 강도 곡선을 가로 지르는 수평선을 그리고 둘러싸인 영역 내부의 추적 막대를 클릭하여 곡선 아래의 영역을 통합하십시오.
- cu 아래의 면적을 나누어 각 차선의 상대적 강도를 계산합니다.siRNA 양성 대조군 (mPEG- b -P (APNBMA)을 첨가하지 않고 SDS를 첨가하지 않음)의 곡선 하 면적에 의해 각각의 샘플을 측정 하였다. 각 샘플의 표준화 된 밴드 강도로서 방출 된 siRNA의 백분율을보고하십시오.
3. Fluorescence Correlation Spectroscopy (FCS)를 이용한 siRNA 방출 측정
- 센스 가닥의 5 '말단에 단일 형광 단으로 표지 된 siRNA를 얻습니다.
참고 : siRNA는 원하는 위치에 접합 된 표지를 사용하여 미리 어닐링하여 구입할 수 있습니다. 형광체는 광 안정성이 있어야하며 mPEG- b -P (APNBMA)로 자외선 퀀칭 및 에너지 전달을 피하기 위해 450 ~ 750 nm 사이를 흡수 / 방출해야합니다. - 표지 된 siRNA를 사용하여 1.1-1.2 단계에 따라 나노 캐리어를 배합하십시오. 원하는 샘플 수를 수용하도록 볼륨 크기를 조정하십시오.
- SDS에 솔루션을 품어과 2.2-2.6 단계에 따라 원하는 시간 동안 照射.
- 준비FCS 샘플 챔버의 배치.
- 유리 슬라이드를 7 : 3 (v / v) 에탄올 / 물 용액으로 씻고 닦아내와 공기 흐름을 사용하여 유리를 완전히 말립니다.
- 양면 접착 스페이서에서 종이 조각을 제거하여 양면 접착 스페이서를 노출 시키십시오. 유리 coverslip에 스페이서를 첨부하십시오.
- 접착 스페이서에서 구멍의 중간에있는 커버 슬립에 나노 캐리어 / SDS 솔루션을 피펫.
- coverslip의 위에 유리 슬라이드를 놓습니다. 유리 슬라이드를 밀어서 유리 슬라이드와 커버 슬립이 잘 부착되어 밀봉되도록하십시오.
- FCS 측정 33을 위해 공 촛점 현미경을 사용하십시오. 1.2의 개구 수를 가진 40X 물 침지 아포 크로마토 대물 렌즈를 사용하십시오. 적절한 여기 레이저 채널 (488 nm)을 사용하여 시료 당 각각 10 s의 최소 30 측정을 수집하십시오 34 . 레이저 강도와 검출기 정렬이 유지되는지 확인하십시오각 샘플에 대해 동일합니다.
- 실험 샘플 이외에, 다음을 포함하는 측정 컨트롤 : 표지 된 siRNA가없는 빈 샘플; 그리고 mPEG- b -P (APNBMA)가없는 siRNA 샘플을 가진 무료 siRNA 샘플.
- FCS 특정 소프트웨어를 사용하여 데이터를 분석하십시오. nanocarriers가 공 촛점 볼륨 29를 통과하는 시간 동안 안정적인 카운트 속도를 결정하여 각 샘플의 기본 카운트 속도를 식별합니다.
- 모든 샘플 기준선 계수 값에서 빈 샘플의 계수율을 뺍니다. 결과 값을 자유 siRNA 컨트롤로 표준화하여 유리 siRNA 35 의 백분율을 계산하십시오.
진 진화를 예측하는 운동 모델링
- 유전자 침묵을 예측하기위한 간단한 미분 방정식 세트를 사용하여 수학적 프로그래밍 언어로 스크립트 만들기 29.
참고 : 요청시 스크립트를 제공 할 수 있습니다.- 다음과 같이 상미 분 방정식을 작성하십시오.
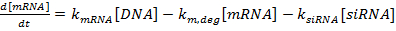 (1)
(1)  (2)
(2) 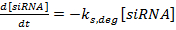 (삼)
(삼)
참고 : 식 1 ~ 3에 대한 용어 k mRNA , k siRNA 및 k prot 는 각각 mRNA, siRNA 및 단백질 생산 속도 상수입니다. 용어 k m, deg , k s, deg 및 k p, deg 는 각각 mRNA, siRNA 및 단백질의 분해 속도 상수입니다. 분해 속도 상수는 성분의 반감기를 기준으로 계산되며, 생산 속도 상수는 mRNA 및 단백질 정상 상태 값이 부재시f siRNA.- 이론적으로 참고 36 또는 문헌 ( Discussion 참조)에서 설명한대로 실험 대상 유전자 (들)에 대한 mRNA와 단백질의 반감기를 결정하십시오. 또한, 세포주의 배가 시간을 결정하십시오. 이 값을 적절한 분해율 식에 입력하십시오.
- 생산 속도 상수를 조정하여 siRNA가 도입되지 않은 경우 유전자 발현 수준이 100의 정규화 된 값에서 일정하게 유지되도록하십시오. 구체적으로 [siRNA]를 0으로 설정하고 [mRNA]와 [단백질]이 100 %의 초기 정규화 값의 1 % 내에서 남아있을 때까지 k mRNA , k siRNA 및 k prot 생성 속도 상수 값을 변경합니다. 시뮬레이션.
- 앞서 설명한 겔 전기 영동 및 FCS 분석에서 방출 된 siRNA의 상대적 양을 추정치로 사용하여 스크립트에서 siRNA의 초기 상대적 농도를 조정합니다. 구체적으로, [siRNA]는 방출 된 siRNA의 상대적 양에 비례하며, 100의 값은 최대 양 29에 상응한다.
- 다음과 같이 상미 분 방정식을 작성하십시오.
5. 세포 배양 및 시험관 내 siRNA 전달
- 배양 물 NIH / 3T3 마우스 배아 섬유 아세포를 공급업자로부터의 프로토콜에 따라 배양 하였다.
- 성장 매체 (10 % 열 불 활성화 태아 소 혈청과 1 % 페니실린 - 스트렙토 마이신으로 보충 된 Dulbecco 수정 된 이글 매체 (DMEM))에서 세포를 성장. 5 % CO 2로 습한 환경에서 37 ℃에서 세포를 유지한다.
- 6 - 웰 조직 배양 접시에 세포를 씨앗 처리.
- 공급 업체의 권장 하위 배양 절차를 따르십시오. hemocytometer를 사용하여 세포를 계산합니다. 보충 성장 미디어에서 75,000 세포 / ML의 농도로 세포를 희석.
- 2 mL의 세포 현탁액 (75,000 세포s / mL)을 6- 웰 플레이트의 각 웰에가 하였다. 세포가 부착하고 인큐베이터에서 24 시간 동안 복구하자.
- 인산염 완충 식염수 (PBS)로 씻고 혈청 및 항생제가없는 형질 전환 배지 1.5 mL를 각 웰에 첨가하여 형질 감염을위한 세포를 준비합니다 ( 표 참조).
- 단계 1.1-1.2에 따라 siRNA nanocarriers를 공식화하십시오. 각 우물에 30 pmol의 siRNA가 들어있는 나노 캐리어 용액 25 μL를 넣으십시오. 부드럽게 미디어를 위아래로 혼합하여 혼합합니다. 3 시간 동안 인큐베이터에 세포를 놓습니다.
- transfection 미디어를 제거하고 PBS로 각 우물을 씻으십시오. 보충 성장 미디어 1 ML을 추가하고 인큐베이터에 30 분 동안 회복 세포를 놓으십시오.
- 광 자극으로 치료를 위해 세포를 준비하려면 보충 성장 매체를 제거하십시오. 각 우물에 1 ML의 transfection 미디어 (페놀 레드 제외)를 추가합니다.
참고 : 형질 감염 배지에 페놀 레드가 포함되어 있지 않은지 확인하십시오. - 365 nm 필터를 사용하여 200 W / m의 강도로 UV 레이저를 보정하고 설정하십시오. 셀 플레이트의 바닥이 고정되는 위치에서 광도를 측정하십시오.
- 37 ° C로 설정된 핫 플레이트에 세포를 놓습니다. 세포의 판 덮개를 제거하십시오. 200W m -2 의 강도에서 365nm 필터가있는 UV 레이저를 사용하여 원하는 시간 (최대 20 분) 동안 플레이트 위의 세포를 조사하십시오.
- transfection 미디어를 제거하고 보충 성장 미디어 2 ML을 추가합니다. 추가 분석 ( 예 : qPCR의 경우 24 시간, Western blotting의 경우 48 시간)까지 인큐베이터에 놓습니다.
- 웨스턴 블 럿팅 37 및 qPCR과 같은 다양한 기술을 사용하여 유전자 발현의 변화를 측정하십시오. 38 GFP와 같이 보이는 신호가있는 유전자의 경우 형광 현미경 검사 29를 사용하십시오 .
참고 :이 기술은 사용의 용이성과 정확성 때문에 제안되었습니다유전자 발현을 정량화 할 때
- 웨스턴 블 럿팅 37 및 qPCR과 같은 다양한 기술을 사용하여 유전자 발현의 변화를 측정하십시오. 38 GFP와 같이 보이는 신호가있는 유전자의 경우 형광 현미경 검사 29를 사용하십시오 .
6. 시공간적 인 방식으로 유전자 침묵 조절
- 단계 5.1-5.7에 따라 세포를 배양하고, 종자를 만들고, 형질 감염시킨다.
- 365 nm의 빛을 완전히 차단하고 반사를 최소화하는 포토 마스크를 준비하십시오.
참고 :이 경우, 빛을 차단하고 반사를 줄이기 위해 10 x 10cm 크기의 알루미늄 호일과 검은 색 건설 용지를 사용했습니다. 알루미늄 호일과 건설 용지는 함께 붙어서 하나의 유닛을 형성했습니다.- 원하는 모양을 절단 / 펀치 / 가공하여 포토 마스크에 넣습니다. 예를 들어 날카로운 칼날과 구멍 펀치를 사용하여 포토 마스크에 직선 패턴 (~ 5cm 길이)과 원형 패턴 (~ 7mm 직경)을 각각 형성하십시오.
- 반사 방지면 ( 예 : 검은 색 c)이있는 셀을 포함하는 웰 아래에 패턴이있는 6-well 플레이트의 바닥에 포토 마스크를 붙입니다onstruction paper)를 판에 대면합니다. 접착제가 패턴의 가장자리 근처 (약 3mm 이내)에 놓이지 않도록하십시오.
- 약 25 cm 간격으로 두 개의 링 스탠드를 설치하고 플랫폼이 동일한 높이가되도록 각 링 스탠드에 플랫폼을 부착하십시오. 플레이트를 플랫폼 위에 놓고 두 개의 스탠드 사이에 셀 플레이트를 매달 십시 오. 판이 평평한 지 확인하십시오.
- 200 W / m 2 의 강도에서 365 nm 필터를 가진 UV 레이저를 사용하여 원하는 시간 (최대 20 분) 동안 샘플 아래에서 세포를 조사하십시오.
- transfection 미디어를 제거하고 보충 성장 미디어 2 ML을 추가합니다. 적어도 24 시간 동안 회복하기위한 인큐베이터에 놓으십시오. 설명 29 형광 현미경을 사용하여 이미지 세포.
결과
나노 캐리어의 제제화 후 siRNA 방출 분석을 수행하여 시험 관내 형질 감염 에 사용되는 조사 조건을 알려 주었다. 방출 된 siRNA의 백분율을 결정하기 위해 다양한 양의 빛을 가했다. 첫 번째 분석은 여전히 폴리머와 복합체 / 결합 된 siRNA 분자에서 유리 siRNA 분자를 분리하기 위해 겔 전기 영동을 사용했습니다. 빛으로 치료되지 않은 나노 캐리어는 안정한 ...
토론
이 방법에는 몇 가지 단계가 특히 중요합니다. nanocarriers을 수립 할 때, 구성 요소 추가 및 혼합 속도의 순서는 효능 (39)에 영향을 미치는 두 가지 중요한 매개 변수입니다. 이 프로토콜은 vortexing하는 동안 음이온 성 구성 요소 인 siRNA에 양이온 성 구성 요소 인 mPEG- b -P (APNBMA)가 첨가되도록 요구합니다. 총 배합량에 따라이 혼합 공정은 3-6 초가 소요됩니다. 나노 캐리어가 ?...
공개
저자는 경쟁적인 금전적 이해 관계가 없다고 선언합니다.
감사의 말
저자들은 보조금 번호 P20GM103541 및 부여 번호 P20GM10344615로 기관 개발 상 (IDeA)을 통한 재정 지원을 위해 국립 보건원 (NIH)의 국립 종합 의학 연구소에 감사를드립니다. 여기에있는 진술은 NIH의 견해를 반영하지 않습니다. 우리는 또한 첨단 기술 생명 과학 센터 (Bioscience CAT) 상 (12A00448)을 통해 재정 지원을 위해 델라웨어 생명 공학 연구소 (DBI)와 델라웨어 경제 개발 사무소 (DEDO)를 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| siRNA | Sigma-Aldrich | SIC001 | non-targeted, universal negative control |
| mPEG-b-P(APNBMA) | synthesized in our lab | N/A | photo-responsive polymer |
| HEPES | Fisher Scientific | BP310-100 | |
| sodium dodecyl sulfate | Sigma-Aldrich | 436143 | |
| rubber gasket | McMaster-Carr | 3788T21 | 0.5 mL thick |
| UV laser | Excelitas Technologies | Omnicure S2000 | collimating lens and 365 nm filter used |
| agarose | Fisher Scientific | BP160-100 | |
| ethidium bromide | Fisher Scientific | BP1302-10 | |
| siRNA labelled with Dy547 | GE Healthcare Dharmacon, Inc. | custom order | fluorophore conjugated to 5’ end of sense strand |
| microscope slide | Fisher Scientific | 12-550-A3 | pre-cleaned glass |
| Secure-Seal Spacer | Life Technologies | S24735 | double-sided adhesive |
| LSM 780 | Carl Zeiss | N/A | confocal microscope |
| ZEN 2010 | Carl Zeiss | N/A | FCS analysis software |
| MATLAB | MathWorks | N/A | programming language |
| NIH/3T3 cells | ATCC | ATCC CRL-1658 | |
| DMEM | Mediatech | 10-013-CV | growth media |
| fetal bovine serum | Mediatech | 35-011-CV | heat-inactivated |
| penicillin-streptomycin | Mediatech | 30-002-CI | |
| 6-well plates | Fisher Scientific | 08-772-1B | |
| Opti-MEM | Life Technologies | 11058021 | transfection media |
참고문헌
- Forbes, D. C., Peppas, N. A. Oral delivery of small RNA and DNA. J Control Release. 162 (2), 438-445 (2012).
- Davis, M. E., et al. Evidence of RNAi in humans from systemically administered siRNA via targeted nanoparticles. Nature. 464 (7291), 1067-1070 (2010).
- Bouchie, A. Companies in footrace to deliver RNAi. Nat Biotechnol. 30 (12), 1154-1157 (2012).
- Burke, P. A., Pun, S. H., Reineke, T. M. Advancing Polymeric Delivery Systems Amidst a Nucleic Acid Therapy Renaissance. ACS Macro Lett. 2 (10), 928-934 (2013).
- Gooding, M., Browne, L. P., Quinteiro, F. M., Selwood, D. L. siRNA Delivery: From Lipids to Cell-penetrating Peptides and Their Mimics. Chem Biol Drug Des. 80 (6), 787-809 (2012).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nat Chem Biol. 10 (3), 196-202 (2014).
- Wang, X., Chen, X., Yang, Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nat Methods. 9 (3), 266-269 (2012).
- Ziauddin, J., Sabatini, D. M. Microarrays of cells expressing defined cDNAs. Nature. 411 (6833), 107-110 (2001).
- Saltzman, W. M., Olbricht, W. L. Building drug delivery into tissue engineering. Nat Rev Drug Discov. 1 (3), 177-186 (2002).
- Mura, S., Nicolas, J., Couvreur, P. Stimuli-responsive nanocarriers for drug delivery. Nat Mater. 12 (11), 991-1003 (2013).
- Shim, M. S., Kwon, Y. J. Stimuli-responsive polymers and nanomaterials for gene delivery and imaging applications. Adv Drug Delivery Rev. 64 (11), 1046-1058 (2012).
- Kelley, E. G., Albert, J. N. L., Sullivan, M. O., Epps, T. H. Stimuli-responsive copolymer solution and surface assemblies for biomedical applications. Chem Soc Rev. 42 (17), 7057-7071 (2013).
- Weber, W., Fussenegger, M. Emerging biomedical applications of synthetic biology. Nat Rev Genet. 13 (1), 21-35 (2012).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Shimizu-Sato, S., Huq, E., Tepperman, J. M., Quail, P. H. A light-switchable gene promoter system. Nat Biotechnol. 20 (10), 1041-1044 (2002).
- Huschka, R., et al. Gene Silencing by Gold Nanoshell-Mediated Delivery and Laser-Triggered Release of Antisense Oligonucleotide and siRNA. ACS Nano. 6 (9), 7681-7691 (2012).
- Li, H. -. J., Wang, H. -. X., Sun, C. -. Y., Du, J. -. Z., Wang, J. Shell-detachable nanoparticles based on a light-responsive amphiphile for enhanced siRNA delivery. R Soc Chem Adv. 4 (4), 1961-1964 (2014).
- Braun, G. B., et al. Laser-Activated Gene Silencing via Gold Nanoshell-siRNA Conjugates. ACS Nano. 3 (7), 2007-2015 (2009).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nat Biotechnol. 31 (7), 638-646 (2013).
- Wittrup, A., et al. Visualizing lipid-formulated siRNA release from endosomes and target gene knockdown. Nat Biotechnol. 33 (8), 870-876 (2015).
- Roth, C. M. Quantitative measurements and rational materials design for intracellular delivery of oligonucleotides. Biotechnol Prog. 24 (1), 23-28 (2008).
- Mao, S., et al. Influence of polyethylene glycol chain length on the physicochemical and biological properties of poly(ethylene imine)-graft-poly(ethylene glycol) block copolymer/SiRNA polyplexes. Bioconjugate Chem. 17 (5), 1209-1218 (2006).
- Raab, R. M., Stephanopoulos, G. Dynamics of gene silencing by RNA interference. Biotechnol Bioeng. 88 (1), 121-132 (2004).
- Cuccato, G., et al. Modeling RNA interference in mammalian cells. BMC Systems Biology. 5, 1 (2011).
- Varga, C. M., Hong, K., Lauffenburger, D. A. Quantitative analysis of synthetic gene delivery vector design properties. Mol Ther. 4 (5), 438-446 (2001).
- Chen, H. H., et al. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum Dot-FRET. Mol Ther. 16 (2), 324-332 (2008).
- Bartlett, D. W., Davis, M. E. Insights into the kinetics of siRNA-mediated gene silencing from live-cell and live-animal bioluminescent imaging. Nucleic Acids Res. 34 (1), 322-333 (2006).
- Foster, A. A., Greco, C. T., Green, M. D., Epps, T. H., Sullivan, M. O. Light-Mediated Activation of siRNA Release in Diblock Copolymer Assemblies for Controlled Gene Silencing. Adv Healthc Mater. 4 (5), 760-770 (2015).
- Greco, C. T., Epps, T. H., Sullivan, M. O. Mechanistic Design of Polymer Nanocarriers to Spatiotemporally Control Gene Silencing. ACS Biomater Sci Eng. 2 (9), 1582-1594 (2016).
- Green, M. D., et al. Catch and release: photocleavable cationic diblock copolymers as a potential platform for nucleic acid delivery. Polym Chem. 5 (19), 5535-5541 (2014).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose Gel Electrophoresis for the Separation of DNA Fragments. J Vis Exp. (62), e3923 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Marquer, C., Leveque-Fort, S., Potier, M. C. Determination of Lipid Raft Partitioning of Fluorescently-tagged Probes in Living Cells by Fluorescence Correlation Spectroscopy (FCS). J Vis Exp. (62), (2012).
- Staaf, E., Bagawath-Singh, S., Johansson, S. Molecular Diffusion in Plasma Membranes of Primary Lymphocytes Measured by Fluorescence Correlation Spectroscopy. J Vis Exp. (120), e54756 (2017).
- Buyens, K., et al. Monitoring the disassembly of siRNA polyplexes in serum is crucial for predicting their biological efficacy. J Control Release. 141 (1), 38-41 (2010).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764-768 (2011).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proc Natl Acad. Sci U S A. 76 (9), 4350-4354 (1979).
- Wittwer, C. T., Herrmann, M. G., Moss, A. A., Rasmussen, R. P. Continuous Fluorescence Monitoring of Rapid Cycle DNA Amplification. Biotechniques. 54 (6), 314-320 (2013).
- Cho, S. K., Dang, C., Wang, X., Ragan, R., Kwon, Y. J. Mixing-sequence-dependent nucleic acid complexation and gene transfer efficiency by polyethylenimine. Biomater Sci. 3 (7), 1124-1133 (2015).
- Liu, Y. M., Reineke, T. M. Poly(glycoamidoamine)s for gene delivery: Stability of polyplexes and efficacy with cardiomyoblast cells. Bioconjugate Chem. 17 (1), 101-108 (2006).
- Schwanhausser, B., et al. Global quantification of mammalian gene expression control. Nature. 473 (7347), 337-342 (2011).
- Greco, C. T., Muir, V. G., Epps, T. H., Sullivan, M. O. Efficient tuning of siRNA dose response by combining mixed polymer nanocarriers with simple kinetic modeling. Acta Biomater. 50, 407-416 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유