Method Article
시퀀스 및 선택적 인식 하 여 단일 가닥 RNAs에 더블-좌초 RNAs의 화학적 펩 티 드 핵 산 수정
요약
우리는 합성에 대 한 프로토콜와 펩 티 드 핵 산 (PNA) 올리고 통합의 정화 수정 잔류물. 수정된 PNAs에 의해 RNA duplexes의 인식 특성에 대 한 생화학 및 생물 방법은 설명 합니다.
초록
RNAs 중요 한 바이오 마커 및 치료 대상으로 떠오르고 있다. 따라서, 화학 프로브와 RNA 순서 및 구조의 인식에 대 한 치료 ligands 개발 잠재력이 있다. 화학적 펩 티 드 핵 산 (PNA) 올리고 최근 개발 된 시퀀스 별로 RNA duplexes 인식할 수 있는 수정. PNAs 중립 펩 티 드와 같은 등뼈와 화학적으로 안정 되어 있다. PNAs 수 수 합성 비교적 쉽게 수동 Boc 화학 단단한 단계 펩 티 드 합성 방법에 의해. PNAs 역 상 HPLC, 분자량 특성 매트릭스 보조 레이저 탈 착/이온화 시간의 비행 (TOF MALDI)에 의해 다음에 의해 정화 된다. 비 변성 시키기 polyacrylamide 젤 전기 이동 법 (페이지) 기술을 용이 하 게 세 겹 형성의 영상 때문에 신중 하 게 설계 된 무료 RNA 이중 구성 및 PNA 바인딩된 triplexes 자주 쇼 다른 마이그레이션 속도. Ethidium 평범한 사람 게시물 얼룩과 비 변성 시키기 페이지 종종 바인딩 친 화력 및 특이성 PNA 올리고의 특성화를 위한 쉽고 유익한 기술입니다. 일반적으로, 여러 개의 동 곳 RNA 또는 단 하나 기본적인 쌍 돌연변이와 duplexes 바인딩 친 화력 및 특이성 등의 PNA 바인딩 속성의 특성을 사용할 수 있습니다. 2 Aminopurine는 아데닌 (6-aminopurine);의 이성질체 2 aminopurine 형광 강도 지역 구조 환경 변화에 민감한 이며 PNA 바인딩 사이트 근처 통합 2 aminopurine 잔류물과 세 겹 형성의 모니터링에 적합. 2-Aminopurine 형광 적정 대상된 더블-좌초 RNAs (dsRNAs)으로 수정된 PNAs 바인딩 선택도 단일 가닥 RNAs (ssRNAs)를 통해 확인 하도 사용할 수 있습니다. 자외선 흡 광도 감지 열 용융 실험 허용 PNA RNA duplexes와 PNA·의 열 안정성 측정 RNA2 triplexes입니다. 여기, 우리는 합성을 설명 하 고 PNA 올리고 통합의 정화 잔류물, 수정 하 고 수정된 PNAs에 의해 RNA duplexes의 인식의 특성에 대 한 생화학 및 생물 방법을 설명.
서문
RNAs는 중요 한 생체 및 규칙에서 RNAs의 역할의 발견과 다양 한 생물 학적 과정1,2,3의 촉매의 최근 발전으로 인해 치료 대상으로 등장 하고있다. 전통적으로, 센스 가닥 왓슨 크릭 이중 형성3,,45,6,7,8, ssRNAs 바인딩할 사용 되었습니다. 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 27. 최근, 펩 티 드 핵 산 세 겹 형성 (TFPNAs) Hoogsteen 수소 결합 (그림 1)3,,2829통해 dsRNAs 바인딩할 설계 되었습니다. dsRNA는 사전 mRNAs와 mRNAs, 사전 또는 pri miRNAs3, 그리고 많은 다른 비 코딩 RNAs1,,2627를 포함 하 여 전통적인 antisense 대상 RNAs의 대부분에 존재. TFPNAs를 사용 하 여 트리플 헬릭스 형성을 통해 dsRNAs 타겟팅의 구조 특이성 때문에 유리한 수 있습니다 고는 dysregulated 질병, 예를 들면 RNAs의 정상적인 기능을 복원에 사용 하기 위해 큰 잠재력입니다.
최근에 출판 된3,,2829,30,31,32,33, Rozners 그 외 여러분, 그리고 우리에 의해 작동 34,35,36,,3738,39,40,41, 개선에 노력을 보고 향상 된 선호도 dsRNAs 향해 수정된 TFPNAs의 선택적 바인딩. Thio-pseudoisocytosine (L) 단위체30 및 guanidine 수정 5-메 틸 시 토 신 (Q) 단위체31를 포함 하 여 합리적으로 설계 된 PNA 단위체 (그림 2)에 대 한 합성 방법을 개발 했습니다. 다양 한 생 화 확 및 생물 특성 방법을 통해 우리는 상대적으로 짧은 PNAs (6-10 잔류물) L과 Q 잔류물 통합 표시 왓슨 크릭 G-C와 C-G 기본적인 쌍의 향상 된 인식 각각 dsRNAs 설명 했다. PNAs L과 Q 잔류물을 포함 하는 또한, ssRNA 이상과 dsDNA dsRNA 향해 더 선택적 바인딩을 표시 수정 되지 않은 PNAs에 비해. Q 자료에 guanidine 기능42 PNAs를 HeLa 세포31입력을 수 있습니다.
우리의 실험실에서 우리는 PNAs 수동 Boc 화학에 의해 합성 (Boc 또는 t-Boc tert-butyloxycarbony에 대 한 서 ( 그림 2참조) 단단한 단계 펩 티 드 합성 방법4. Boc 그룹은 sterically 덜 PNA 모노 머 연결 하는 동안 도움이 될 수 있습니다 그룹을 보호 하는 fluorenylmethyloxycarbonyl (Fmoc) 아민에 비해 부피가 큰 그룹을 보호 하는 아민으로 Boc와 PNA 단량체의 합성은 편리 합니다 단단한 지원입니다. Boc 그룹 산 정한 이며 제거할 수 있습니다 쉽게 고체 지원에 dichloromethane (DCM)에 20-50 %trifluoroacetic 산 (TFA) PNA 합성 하는 동안. 자동된 펩타이드 합성기 PNA 올리고; 합성 채택 될 수 있다 그러나, 3 5 배 초과 PNA 단위체의 자동된 펩타이드 합성기 필요 합니다. 수동 합성 훨씬 적은 PNA 단위체를 (2-3-배 초과), 각 커플링 쉽게 카이 저 테스트43에 의해 모니터링이 필요 합니다. 또한, 많은 자동된 합성기 호환 되지 않습니다 부식성 TFA의 사용으로 인해 Boc 전략 합성 Boc 제거 단계.
PNA 올리고 역 상 고성능 액체 착 색 인쇄기 (라인란트-HPLC) MALDI TOF에 의해 분자량 특성 뒤에 의해 순화 될 수 있다 (그림 3 및 4)30, 31. 우리 고용 세 겹 형성 모니터링, 인해는 사실 무료 RNA 이중 구성 및 PNA 바인딩된 triplexes 종종 표시 다른 마이그레이션 속도 (그림 5)30,31 페이지 비 변성 시키기 . 효율적인 포스트 얼룩 RNA 이중 및 PNA· 둘 다를 위해 달성 될 수 있다 필요 없는 라벨 RNA2 트리플 밴드입니다. 비교적 적은 양의 샘플 페이지 실험 비 변성 시키기 위해 필요 하다. 그러나, 로드 (부 화) 버퍼 및 실행 버퍼 (pH 8.3) 않을 수 있습니다 같은, 비교적 높은 pH 8.3의 크게는 삼박자를 불안정 수 있습니다 때문에 접착 안정적인 triplexes 제한 되 고 측정 결과.
2 Aminopurine는 아데닌 (6-aminopurine);의 이성질체 2 aminopurine 형광 강도 (약 370에서 방출 피크 nm) 지역 구조 환경 변화에 민감한 이며 적합 한 바인딩 사이트 (PNA 근처 통합 트리 2 aminopurine 잔류물 형성의 모니터링에 대 한 그림 6) 31. 많은 다른 염료를 보이는 범위에서 형광 방출 쇼와 달리 2 aminopurine 표시 된 RNA 노출 될 수 있습니다 방에 빛 사진 표백 하지 않고. 페이지 실험 pH 8.3의 실행 버퍼 자주 필요 하다, 달리 2-aminopurine 기반으로 형광 적정 바인딩 지정된 pH에 1 개의 해결책에서 사용 하 고 따라서 측정 및 탐지의 상대적으로 약한 수 고 평형에 운동 불안정 바인딩입니다.
자외선 흡 광도 감지 열 용융 실험 허용 duplexes (그림 7)31 와 트라이의 열 안정성 측정플렉스30,32,,4445. 길이 시퀀스 구성에 따라 triplexes의 용 해 되거나 명확한 전환 표시 되지 않을 수 있습니다. 가 열 및 냉각 곡선 겹치면 열역학 매개 변수를 얻을 수 있습니다. 등온선 적정 열 량 측정 (ITC)32; 여 정확한 열역학 매개 변수를 얻을 수 있습니다. 그러나, 비교적 많은 양의 샘플은 ITC 일반적으로 필요 합니다.
프로토콜
1. 수동 단단한 단계 펩 티 드 종합의 PNAs 사용 하 여 Boc 화학
참고: 성공과 원하는 PNA 올리고 머 합성의 용이성에 대 한 모든 용 매 및 시 약은 무수 이어야 한다. 적절 한 분자 체 (4A, 1-2 m m 직경 펠 릿)를 추가 하 고 가끔 병으로 건조 질소 가스를 퍼지. 수정 된 PNA 단위체의 합성에 대 한 각 참조 30 , 31 보고 된 프로토콜을 사용할 수 있습니다. 수정 되지 않은 PNA 단위체 상업적인 소스에서 구입하실 수 있습니다. 각 세척 단계에 적절 한 양의 용 매에서 물기는 그것 전에 슬러리를 형성 수 지에 추가
- 첫 번째 단량체의 로드 및 수 지에 초과 무료 주 아민 상한
- 무게 30 mg 4-methylbenzhydrylamine 염 산 염 (MBHA·HCl)의 폴리스 티 렌 수 지 (상용; 값 0.7-1.4 로드 m m o l/g; 100-200 메쉬 크기), 및 5 mL에 단단한 단계 펩 티 드 반응 배는 자 지와 유리 스 토퍼 장착.
- 팽창 하 고 아민 (HCl 소금) 노출 수 지를 수 있도록 1 h DCM의 적절 한 금액에 수 지를 담근 다.
참고: 수 지 구슬 해야 항상 완전히 빠져들 수 합성을 통해 용 매에서. - 플라스 크의 정상에 건조 질소 가스의 부드러운 흐름을 적용 하 여 DCM에서 드레인. 50% (v/v) N, N의 1 mL을 추가-DCM에서 diisopropylethylamine (DIPEA) 15 분 동안 그것을 두고. 이 수 지에 자유로운 아민에 연결 된 HCl 소금 중화.
- 반복 단계 1.1.3입니다. 한편, 모노 머의 6 µmol 및 6 µmol (benzotriazol-1-yl-옥 시) tripyrrolidinophosphonium hexafluorophosphate (PyBOP)의 무게 그리고 1.5 mL 튜브에 전송. 200 µ L dimethylformamide (DMF)의 DIPEA의 12 µmol을 추가 합니다. 소용돌이 3-5 분을 위한 커플링 솔루션
참고: 사용 하는 원하는 로드 값은 0.2 m m o l/g. 추가 첫 번째 단량체 원하는 PNA 시퀀스의 C-터미널에 단위체가 이다. 첫 번째 아미노산의 로드 값 46 picric 산 방법 의해 산출 될 수 있다. - DCM에서 DIPEA에서 드레인 솔루션입니다. DMF (x 3), 이어서 DCM (x 3)와 수 지를 세척 하 고는 자 지를 닫습니다. 수 지에 준비 된 커플링 솔루션을 추가 하 고 부드럽게 흔들어. 깨끗 한 스테인리스 주걱의 사용과 결합 솔루션으로 혈관의 내부 벽을 따라 수 지를 밀어. 유리 스 토퍼를 부착 하 고 40에서 3 h는 인큐베이터 흔드는에 선박을 확보 ° c.
참고: 또는, 반응 배 수 수 꾸준한 실 온에서 보관 펩 티 드 결합 반응의 완료를 허용 하려면 6-8 h. - 아세트산 무수 물의 240 µmol DCM의 200 µ L로 DIPEA의 360 µmol을 혼합 하 여 상한 솔루션을 준비 합니다. 커플링 솔루션에서 배수와 (x 3) DCM와 DMF (x 3) 수 지를 세척. 상한 솔루션에 추가 하 고 때때로 배를 부드럽게 흔들어 30 분 동안 배를 떠나. Acetylation에 의해 수 지에 초과 무료 1 차 아민 그룹을 마스크 상한.
- 반복 단계 1.1.6입니다. 상한 솔루션 배수와 DCM (x 3)와 수 지를 세척.
- 얇은 모 세관 튜브를 사용 하 여 수 지 구슬의 작은 약 수를 제거 하 고 1.5 mL 유리 작은 유리병에 넣어. 43의 카이 저 테스트 수행 합니다. 15 µ L 각 카이 저 테스트 솔루션 유리에 유리병과 총 열을 사용 하 여 열을 추가 합니다. 가 열 후 구슬의 색상을 관찰 합니다. 구슬의 색깔 그대로, 수 지에 자유로운 아민 그룹의 부족을 나타내는 유지 한다.
참고: 카이 저 테스트 키트는 상업적으로 사용할 수 또는 보고 된 프로토콜에 따라 준비 될 수 있다. 솔루션 a: ninhydrin 에탄올;에 솔루션 에탄올; 페 놀 b: 솔루션 c: 칼륨 청산가 리 (KCN) pyridine에.
주의: KCN은 매우 독성; 적절 한 보호복을 착용 해야 하 고 난방 가연성 용 매 또는 시 약의 부재에서 환기가 증기 두건에서 수행 되어야 합니다. - 반복 단계 1.1.6 수 지 구슬 파란색 또는 희미 한 파란색 표시.
- N 맨끝 아민 보호 그룹의 제거
- 반응 용기에서 용 매를 배수와 DCM, 수 지는 완전히 빠져들 보장에 trifluoroacetic 산 (TFA)의 50% (v/v)의 해결책을 추가 하십시오. 15 분, 아민 그룹의 deprotection를 촉진 하기 위하여 때때로 흔들어 배를 남겨 주세요. 반복 2 더 많은 사이클.
주의: TFA 매우 부식성입니다. 처리할 때에 적절 한 보호복을 착용 한다. - 는 DCM (x 3), (x 3), DMF 및 DCM (x 3) 수 지를 세척. 추가 5%의 솔루션 DCM에서 DIPEA. 15 분 동안 배를 남겨 주세요. 이 단계는 TFA 카운터 음이온을 중화 하 여 자유 아민을 활성화 합니다. 한 번 더 반복.
- 는 DCM 솔루션에서 DIPEA에 밖으로 플러시. DCM (x 3)와 수 지를 세척. 카이 저 테스트 (단계 1.1.8).
참고: 아민 그룹의 성공적인 deprotection 구슬의 청색 변색을 얻을 것입니다. 일단 deprotection 성공, 펩 티 드 결합에 의해 단위체의 후속 커플링 실시 될 수 있습니다.
- 반응 용기에서 용 매를 배수와 DCM, 수 지는 완전히 빠져들 보장에 trifluoroacetic 산 (TFA)의 50% (v/v)의 해결책을 추가 하십시오. 15 분, 아민 그룹의 deprotection를 촉진 하기 위하여 때때로 흔들어 배를 남겨 주세요. 반복 2 더 많은 사이클.
- 후속 단위체의 커플링
- 의 무게 18 µmol (Boc-PNA-Q-오 단량체, 단위체의 13.2 mg 무게가 있다)에 대 한 모노 머를 원하는 1.5 mL 튜브에 PyBOP의 18 µmol. DMF의 200 µ L 및 1.5 mL 튜브에 DIPEA의 36 µmol을 추가 합니다. 모든 고체 화합물 해산 때까지 소용돌이.
- 는 DMF (x 3)와 수 지를 세척. 반응 용기에 커플링 솔루션을 추가 하 고 부드럽게 흔들어. 깨끗 한 스테인리스 주걱의 사용과 결합 솔루션으로 혈관의 내부 벽을 따라 수 지를 밀어. 유리 스 토퍼를 부착 하 고 40에서 3 h는 인큐베이터 흔드는에 선박을 확보 ° c.
- 드레인 커플링 솔루션와 (x 3) DCM와 DMF (x 3) 수 지를 세척. 1.1.8 단계를 수행 합니다. 구슬 남아의 색상 변경 하는 경우 원하는 PNA 시퀀스 완료 될 때까지 1.2.1-1.3.3 단계를 반복 합니다. 구슬의 청색 변색 모노 머의 커플링 후, 관찰 단계 1.3.1-1.3.3를 반복 합니다. 변색은 여전히 지속 되 면 다시 상한 수행 (단계 1.1.6).
참고: 확장 시간 (3-12 h) 및 모노 머의 과도 상응의 사용 또는 커플링 및 커플링 시 약 권장 연결 문제가 발생 하는 경우. - 원하는 PNA 시퀀스 완료 되 면 (x 3) DCM와 DMF (x 3) 수 지를 세척. 15 분 동안 건조 질소 가스의 지속적인 흐름을 적용 하 여 수 지를 완전히 건조. 이 건조 수 지 분할 및 lysine 또는 형광 태그 cyanine 3 등 (Cy3)와 PNA의 N-말단에 carboxyfluorescein를 연결 하는 데 사용 될 수.
- Lysine 또는 형광 태그 (Cy3, Cy5, 또는 carboxyfluorescein) PNA의 N-말단에 부착
- 원하는 PNA 시퀀스 미리 로드 하는 수 지의 10 밀리 그램 무게. 5 mL 반응 그릇에 전송 합니다. 1 헤 대 한 DCM에 수 지를 담가
- Lysine의 첨부 파일에 대 한 수행 단계 1.2.1-1.3.3, 교체는 단위체 어느 Boc 리스 (Z)-오 또는 Fmoc 리스 (Boc)-오.
- Carboxyfluorescein의 첨부 파일에 대 한 수행 단계 1.2.1-1.3.35 (6)-carboxyfluorescein는 단위체로 서의 사진 초과 및 N, N '-diisopropylcarbodiimide (DIPC) 및 hydroxybenzotriazole (HOBt) DMF에 결합 시 약으로. 밤새 40 인큐베이터 셰이 커에 커플링 반응에 두고 ° c.
참고: carboxyfluorescein는 빛에 민감한, 반응 선박으로 덮어야 한다 알루미늄 호 일. - Cy3 또는 Cy5 염료, 클릭 화학 방법에 의해 라벨의 부착을 위한 수행 단계 1.2.1-1.3.3, N-Boc-2-propargyl-L-glycine는 단위체를 바꿉니다. 이 functionalizes alkyne 그룹과 PNA의 N terminus. 포함 하는 아 지 드 Cy3 연결할 클릭 구리 촉매 반응을 수행 또는 Cy5 형광 염색 12 , , 47 48 , 49
- 고체 지원, 정화, 특성에서 PNA 분열
참고: 아민 그룹 Fmoc 그룹 보호로 보호 되는 경우 20% piperdine DMF에 함께 하 여 아민 그룹을 deprotect 먼저 15 분 (2 주기)에 대 한 솔루션입니다. DMF (x 3) DCM (x 3) 다음으로 철저 하 게 수 지를 세척. 15 분 동안 건조 질소 가스의 지속적인 흐름을 적용 하 여 수 지를 완전히 건조. - 전송 5 밀리 그램 작은 유리병에 건조 수 지의
- . Thioanisole의 10 µ L의 1, 2-ethanedithiol, 수 지는 시 약에 빠져들은 보장 4 µ L를 추가 합니다. 5 분에 대 한 실 온에서 튜브를 두고
- 수 지와 썩은 고기를 포함 하는 관으로 TFA의 추가 100 µ L. 부드럽게 소용돌이 혼합물 그리고 간단한 원심을. 10 분에 대 한 실 온에서 튜브를 두고
주의: TFA 매우 부식성입니다. 처리할 때에 적절 한 보호복을 착용 한다. - 신중 하 게 튜브에 trifluoromethanesulfonic 산 (TFMSA)의 20 µ L를 추가합니다. 부드럽게 실 온에서 간단한 원심 분리를 겪고 전에 반응 혼합물을 교 반 하십시오. 2 헤에 대 한 실 온에서 안정 튜브를 두고
주의: TFMSA 매우 부식성입니다. 처리할 때에 적절 한 보호복을 착용 한다. - 필터 5 mL 라운드 바닥 플라스 크 (RBF) 유리 파스퇴르의 사용으로 칵테일 분열을 피펫으로 면 장착. TFA의 작은 금액을 사용 하 여 수 지를 세척.
- 모든 휘발성 용 제 증발 때까지 수집 된 여과 액을 건조 질소 가스를 제거. RBF로 차가운 diethyl 에테르의 1 mL을 추가.
참고: diethyl 에테르 침전에 PNA를 일으킬 것입니다. 여러 번 1.5 mL 튜브에 흐린 솔루션을 전송 하기 전에- 린스는 diethyl와 RBF 에테르. 정착 PNA 침전 수 있도록 주제를 원심 분리 관. 용을 가만히 따르다와 침전을 압력가 마로 소독 물 300-500 µ L를 추가 합니다. PNA를 해산 하기 위해 철저 하 게 소용돌이.
- RP HPLC를 통해 원유 PNA 샘플 정화 water-acetonitrile-0.1% TFA를 사용 하 여 모바일 상으로. 해당 분수 수집, 압력가 마로 소독 물에 순화 된 PNA를 다시 해산 하기 전에 진공 집중 장치를 사용 하 여 모든 용 매를 증발.
- MALDI TOF 분석 샘플 결정 매트릭스로 α-cyano-4-hydroxycinnamic 산 (CHCA)의 사용을 통해 순화 된 PNA 특징.
- UV 흡 광도 측정 (260 nm) 65에 PNA의 ° C. 계산 방정식와 PNA의 농도:

참고: 여기 c 농도, A는 얻은 흡 광도 읽기, ε는 RNA 순서의 소멸 계수 이며 l 베트 (1의 광학 경로 길이 cm). PNA 시퀀스의 소멸 계수는 개별 단위체 50의 소멸 계수 합계. 15.4, 7.3, 11.7, 8.8 thymine, 구 아닌, 시 토 신, 아데닌의 소멸 계수는 mL/µmol·cm, 각각. 사용 L과 Q 단위체의 소멸 계수는 시 토 신 (C) 기본의 것과 같은 것으로 간주 됩니다.
참고:이 시 약 PNA에서 그룹 보호의 제거 하는 동안 형성 되는 반응성 양이온 종 트랩 청소부 역할을 합니다. 썩은 고기를 적절 한 그룹을 보호 하는 사이드 체인에 따라 선택 될 수 있다. - 수 지와 썩은 고기를 포함 하는 관으로 TFA의 추가 100 µ L. 부드럽게 소용돌이 혼합물 그리고 간단한 원심을. 10 분에 대 한 실 온에서 튜브를 두고
2. 비 변성 시키기 페이지
116.88 mg NaCl (200 mM), 100 mM ethylenediaminetetraacetic 산 (EDTA) (0.5 m m)의 50 µ L, 1 M의 200 µ L를 사용 하 여- 필요한 버퍼 솔루션의 준비
- 준비 보육 버퍼 (10 mL) HEPES (20 m m), 9.75 m H 2 O; pH 7.5에 버퍼 조정.
- 준비 1 트리 스-Borate-EDTA (TBE) 실행 버퍼 (1 L) 100 mL 10를 사용 하 여 x x 트리 스-Borate-EDTA pH 8.3과 900 mL H 2 o.
- 준비 10% 염화 persulfate (AP) 솔루션 (300 µ L)를 사용 하 여 30의 마그네슘 염화 persulfate, 및 300 µ L H 2 o.
- 12 %polyacrylamide 젤의 준비
- 청소 잘 형성 빗, 유리 캐스팅 접시, 그리고 에탄올과 스페이서, 잘 형성 빗과 스페이서는 1 m m 두께. 젤 어셈블리를 설정 하 고 젤 씰링 테이프와 함께 인감.
- 22 cm x 16.5 c m x 1 m m 크기의 polyacrylamide 젤 50ml 젤 솔루션은 적절 한. 5.7 g 아크릴 아 미드와 N, N의 0.3 g으로 무게 '-methylenebisacrylamide (19:1)와 50 mL 원심 분리기로 전송 관.
주의: 아크릴 이다 발암 성입니다. 아크릴에서 먼지 매연에 호흡 하지 않도록 하 고 처리할 때 적절 한 보호복은 착용 하는 확인 하십시오. - 15 분 또는 모든 고체 화합물 해산 할 때까지 50 ° C 물 목욕으로 원심 분리기 튜브를 배치 하 여 50 mL 1 X TBE 실행 버퍼에 고체 화합물을 디졸브. (3000 rpm, 5 분, 25 ° C)는 솔루션 내에서 모든 기포를 제거 하는 원심 분리를 실시 합니다. 실내 온도에 냉각 솔루션을 허용.
- 젤 솔루션으로 10 %APS 솔루션의 250 µ L 및 tetramethylethylenediamine (TEMED)의 50 µ L을 추가 하 고 부드럽게 주걱을 사용 하 여 혼합. 즉시 모든 기포를 소개 하지 않도록 만들어 유리 접시 사이 솔루션을 붓는 다. 잘 형성 빗을 삽입 하 고 젤 설치를 사용할 준비가 될 때까지 4 ° C에서 저장 하기 전에 발생 하는 합 수 있도록 적어도 60 분 동안 실내 온도에 떠날.
- 샘플의 준비
- 깨끗 한 1.5 mL 튜브에 주요 주식에서 RNA 머리 핀 (1 µ M)의 필요한 수량을 제거. 건조 한 진공 집중 장치를 사용 하 여 RNA 솔루션.
참고: 각 샘플에는 20 µ L 보육 버퍼에서 RNA의 1 µ M이 포함 되어 있습니다. 일반적으로, RNA의 13 샘플 실험을 한 페이지에 대 한 준비가 되어 있습니다. 모든 샘플에 대 한 RNA는 단일 튜브에 함께 준비 하실 수 있습니다. - 주요 주식 및 별도 1.5 mL 튜브로 전송 (최대 50 µ M의 최종 농도)와 타겟된 PNA의 필요한 볼륨을 제거합니다. 진공 집중 장치를 사용 하 여 PNA 솔루션의 물을 증발.
- 1.5 mL에 인큐베이션 버퍼의 추가 260 µ L 튜브 포함 된 말린 RNA 및 혼합 철저 하 게 되도록 모든 RNA dissolved입니다. 냉각에 RNA 주제: 장소 5 분 즉시에 대 한 열 블록 (95 ° C에 미리 데워)으로 튜브는 얼음 목욕으로 전송 하 고 10 분 떠나
- 1.5 mL의 각으로 RNA의 추가 20 µ L 튜브 포함 된 PNA 및 혼합 철저 하 게 건조. 어 닐 링 수행: 실내 온도를 천천히 샘플 냉각 열 블록의 힘 떨어져 10 분 회전을 위해 RNA와 PNA 혼합물 (65 ° C에 미리 데워) 열 블록에 포함 된 튜브를 배치. 4 ° C에서 샘플을 밤새 품 어.
- 깨끗 한 1.5 mL 튜브에 주요 주식에서 RNA 머리 핀 (1 µ M)의 필요한 수량을 제거. 건조 한 진공 집중 장치를 사용 하 여 RNA 솔루션.
- 실행 및 젤의 처리
- 유리 접시의 하단 측면에 젤-씰링 테이프를 제거. 탑재 하 고 안전한 플라스틱 클램프를 사용 하 여 수직 젤 스탠드에 유리 접시.
참고: 실행 하는 젤은 실시 약 4 ° c.에 추운 방에 모든 장비, 예제 및 버퍼 젤을 실행 하기 전에 4 ° C에 냉각 된다. - 접시 버퍼를 실행의 대략 1-2 cm에 빠져들 때까지 1 x TBE 실행 버퍼와 낮은 버퍼 저수지를 작성 합니다. 버퍼를 초과할 때까지 버퍼의 수준 젤의 탑 1-2 cm. 천천히 실행 위 버퍼 저수지를 부드럽게 잘 형성 빗, 우물을 채우기 위해 실행 중인 버퍼를 허용 제거.
- 젤 서 전원 공급 장치에 연결합니다. 사전 실행 최적화는 22 cm x 16.5 c m x 1 m m 젤 250 V의 정 전압으로 적어도 30 분 동안 젤.
- 한편, 각 샘플의 35% 글리세롤 용액의 4 µ L (샘플 볼륨의 20%)를 추가 하 고 부드럽게 혼합. 사전 실행 완료 되 면, 신중 하 게 잘 사용 하는 micropipette의 하단에 각 샘플 (를 포함 하 여 하나의 RNA 혼자 샘플 플러스 샘플 RNA와 PNA 혼합물을 포함)의 20 µ L를 로드 하 고 로드 팁, 어떤 기포를 소개 하지 않도록 만드는 젤. 250 V, 사전 실행 비슷한 5 헤의 정 전압에서 젤 실행
- 5 h 후 전원 공급 장치를 중지 하 고 스탠드에서 유리 접시를 제거 합니다. 남은 젤-씰링 테이프를 제거 하 고 유리 접시를 분해. 부드럽게 제거 하 고 이온을 제거 된 물 350 mL 가득한 용기에 젤을 담가. 조심 스럽게 ethidium 평범한 사람 (10mg/mL)의 35 µ L을 추가 하 고 30 분에 대 한 플랫폼 셰이 커 (저속)에 컨테이너를 놓습니다
주의: Ethidium 평범한 사람은 돌연은. 처리할 때에 적절 한 보호복을 착용 한다. - 지정된 폐기물 컨테이너에 ethidium 평범한 사람 솔루션을 삭제합니다. 증류수의 1.5-2 l 젤 린스. 검사는 영상 장치를 사용 하 여 젤 (재료의 표 참조) 532의 녹색 레이저 및 방출 필터 설정 610 nm.
- 유리 접시의 하단 측면에 젤-씰링 테이프를 제거. 탑재 하 고 안전한 플라스틱 클램프를 사용 하 여 수직 젤 스탠드에 유리 접시.
- 젤 분석
- 젤 밴드 농도 무료 소프트웨어, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html)를 사용 하 여 계량. 에 따라 밴드 농도 정상화:

참고: 여기 나 이중 max는 PNA, 및 나의 추가 없이 혼자 RNA 머리 핀의 밴드 강도 삼중 맥스 추가 PNA의 높은 농도 가진 트리 밴드 강도 이다.- 계산에 따라 세 겹 형성의 분수:

- 계산에 따라 세 겹 형성의 분수:
- 플롯 PNA의 농도 대 한 세 겹 형성 (Y)의 일부분 (추가 Μ M)입니다. 분리 상수 (K d)를 방정식에 데이터:
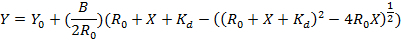
참고: 여기 R 0은 RNA 머리 핀 농도 (1 µ M). 여기 Y 0와 B은 트리 분수의 최대 변화와 초기 각각. Y 다양 한 PNA 농도에서 삼박자의 분수입니다. X는 총 PNA 농도 K d는 분리 상수.
- 젤 밴드 농도 무료 소프트웨어, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html)를 사용 하 여 계량. 에 따라 밴드 농도 정상화:
3. 2-Aminopurine 형광 바인딩 분석 결과
- 샘플 (dsRNA 포함)의 준비
- dsRNA (1 µ M 각을 표시 하는 2-aminopurine (2AP)의 필요한 수량을 제거 물가) 주요 주식에서 깨끗 한 1.5 mL 튜브에. 진공 집중 장치를 사용 하 여 RNA 솔루션에서 물을 증발.
참고: 각 샘플에는 75 µ L 보육 버퍼에서 RNA의 1 µ M이 포함 되어 있습니다. 일반적으로, RNA의 13 샘플 준비 된다. 모든 샘플에 대 한 RNA는 단일 튜브에 함께 준비 하실 수 있습니다. - 주요 주식 및 별도 1.5 mL 튜브에 전송에서 다양 한 농도 대 한 타겟된 PNA의 필요한 볼륨을 제거합니다. 진공 집중 장치를 사용 하 여 PNA 솔루션의 물을 증발.
- 1.5 mL에 인큐베이션 버퍼의 추가 975 µ L 튜브 포함 된 RNA 및 혼합 철저 하 게 모든 RNA 해산 되도록 건조. 짧게 RNA 솔루션 원심과 어 닐 링을 따라: 실내 온도를 천천히 샘플 냉각 열 블록의 힘 떨어져 10 분 회전 튜브 (95 ° C에 미리 데워) 열 블록으로 장소.
- 1.5 mL의 각으로 RNA의 추가 75 µ L 튜브 포함 된 PNA 및 혼합 철저 하 게 건조. 적어도 1 h. 품에 대 한 실 온에서 샘플 샘플 4 ° C에서 하룻밤 둡니다.
- dsRNA (1 µ M 각을 표시 하는 2-aminopurine (2AP)의 필요한 수량을 제거 물가) 주요 주식에서 깨끗 한 1.5 mL 튜브에. 진공 집중 장치를 사용 하 여 RNA 솔루션에서 물을 증발.
- 샘플 (ssRNA 포함)의 준비
- 깨끗 한 1.5 mL 튜브에 필요한 수량 ssRNA 2AP 표시 (1 µ M)의 주요 주식에서 제거. 주요 주식과 ssRNA 포함 하는 각각 1.5 mL 튜브에 전송에서 다양 한 농도 대 한 타겟된 PNA의 필요한 볼륨을 추출 합니다. 진공 집중 장치를 사용 하 여 RNA와 PNA 혼합물 건조.
- 외피의 추가 75 µ L 각 1.5 mL 튜브에 버퍼링 하 고 철저 하 게 혼합. 어 닐 링을 혼합물을 주제: 전원 및 샘플 천천히 실내 온도에 냉각 하자 10 분 회전을 위해 튜브 (95 ° C에 미리 데워) 열 블록으로 배치. 4 ° C에서 샘플을 밤새 품 어.
- 측정 및 분석
- 330-550 nm의 파장 범위에서 방출 측정을 형광 분 광 광도 계를 사용 하 여. 303의 여기 파장을 사용 하 여 nm.
참고: 샘플은 실 온에서과 각 샘플은 측정 3 회, 평균 수행. - 외피의 전송 70 µ L 1cm 평방 베트에 버퍼입니다. 측정 시작.
- 는 베트에서 버퍼를 제거합니다. 증류수와 cuvette 린스와 건조 질소 가스를 제거 합니다. 모든 샘플에 대 한 반복 합니다. 모든 샘플에서 버퍼 측정 빼기.
- 플롯 파장 (nm)에 대 한 형광 강도 (거리). 370에서 형광 강도 기록 모든 샘플에 대 한 nm. 370에서 형광 강도 플롯 nm (거리) PNA의 해당 농도 대 한 추가 (µ M).
- 단계 2.5.2 분리 상수 (K d)를에서 방정식에는 데이터에 맞게: Y = Y 0 + (B / (2R 0)) (R 0 + X + K d-((R 0 + X + K d) 2 -4R 0 X) 1/2), R 0는 dsRNA 2AP 표시 농도 (1 µ M).
참고: 여기에 Y 0와 B는 초기 및 최대 370에서 형광 강도의 변화 nm, 각각. Y는 370에서 형광 강도 다양 한 PNA 농도에서 nm. X는 총 PNA 농도 K d는 분리 상수.
- 330-550 nm의 파장 범위에서 방출 측정을 형광 분 광 광도 계를 사용 하 여. 303의 여기 파장을 사용 하 여 nm.
4. 열 용융 실험 UV 흡 광도 감지
- 샘플의 준비
참고: UV 흡 광도 측정 (260 nm) (되도록 RNA 이차 구조는 중단) 95 ° C에서 RNA의. 방정식으로 RNA의 농도 계산:
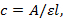
c 농도 인 곳에, A는 얻은 흡 광도 읽기, ε는 소멸 계수 RNA 순서의 l 베트 (1 cm)의 광학 경로 길이입니다. RNA의 소멸 계수 MeltWin , 51 52를 사용 하 여 가장 인접 모델에 따라 계산 됩니다. 프로그램 패키지는 요청 시 제공 될 수 있습니다.- 제거 필요한 양의 깨끗 한 1.5 mL로 주요 주식에서 (5 µ M) ssRNA 튜브. 타겟된 PNA의 필요한 볼륨을 제거 (5 µ M) 주요 주식과 ssRNA 포함 하는 각각 1.5 mL 튜브에 전송에서. 진공 집중 장치를 사용 하 여 RNA와 PNA 혼합물 건조.
- 외피의 추가 130 µ L 각 1.5 mL 튜브에 버퍼링 하 고 철저 하 게 혼합. 어 닐 링을 혼합물을 주제: 전원 및 샘플 천천히 실내 온도에 냉각 하자 10 분 회전을 위해 튜브 (95 ° C에 미리 데워) 열 블록으로 배치. 4 ° C에서 샘플을 밤새 품 어.
- 측정 및 분석
- 260에서 흡 광도 측정 하는 대 일 분 광 광도 계를 사용 하 여 8 microcell cuvette을 사용 하 여 경로 길이 1 cm.의 nm 측정 샘플 ' absorbances 15에서 온도 증가에 95 ° C, 95에서 0.5 ° C/분의 램프 속도에서 15 ° C에 온도 감소 다음 우물의 각 샘플의
- 전송 130 µ L, 확인 하 고 하나 잘 부 화 버퍼를 포함 되어 있습니다. 측정을 시작 합니다. 필요에 따라 반복.
- 통일, 표준화 된 읽기 높은 온도와 흡 광도 값을 정상화 하 고 플롯 정규화 된 흡 광도 온도 (° C)에 대 한 읽기. 곡선의 첫 번째 파생물을 플롯 합니다. 가우스 함수에 첫 번째 파생 커브 피팅 하 여 용융 온도 얻기.
결과
역 상 HPLC PNA 올리고 정화 수 있습니다. 우리는 순수한 PNA 올리고 HPLC 정화 (그림 3)의 2 라운드와 얻을 수 있습니다. MALDI TOF 분석 (그림 4)는 PNAs의 id는 확인할 수 있습니다.
비 변성 시키기 페이지 바인딩 친 화력 및 특이성 PNA 올리고의 특성화를 위한 쉽고 유익한 기술입니다. 우리 일반적으로 사용 하 여 여러 개의 RNA 헤어핀 또는 duplexes 단 하나 기본적인 쌍 돌연변이와 특성 바인딩 속성 (그림 5). 그림 5 에 표시 된 비 변성 시키기 페이지 데이터 명확 하 게 제안는 Q-및 L-수정 PNA dsRNA 지역 C G 쌍 (그림 5B, 하단 패널)을 인식할 수 없습니다 (그림 5B, 상위 C G 쌍 없이 하나 패널)입니다. T·을 통해이 구체적이 고 향상 된 인식이 이다 A-U, L· G-C, 및 Q· C G PNA· RNA2 기본 트리플 (그림 1A, C, D) 형성. 하나 또는 여러 개의 돌연변이와 다양 한 PNAs 수정된 PNA의 향상 된 바인딩 속성을 보여 주기 위해 사용할 수 있습니다. 우리는 2 mM Mg2 + 외피 버퍼에 추가 영향을 주지 않습니다 바인딩을 나타났습니다 크게31.
우리는 Q L 수정 PNA 타겟된 dsRNA 지역 (그림 6A, 6 C 6 D) 하지만 (그림 6B, 6E 6F) ssRNA 하지 2 aminopurine 형광 적정에 의해 증명 하고있다. PNA P3 Kd 값이 0.8 ± 0.1 µ M 2 aminopurine 표시 된 dsRNA에 바인딩합니다. 370에서 형광 강도 2 aminopurine 라는 ssRNA에 대 한 nm는 ssRNA PNA P3의 바인딩의 부족을 나타내는 다양 한 P3 농도로 비교적 일정 하 게 유지.
PNAs 전환 (그림 7)는 ssRNA에 대 한 바인딩을 제안 녹는 아무 열 Q 잔류물 (P2 및 P3) 쇼를 포함 하. 이것은 입체 충돌 왓슨 크릭에서 Q G 쌍 같은 존재입니다. 수정 되지 않은 PNA P1에 비해, PNAs P4 그리고 P5 포함 수정 L 잔류물 하지만 아무 Q 잔류물, 표시 해당 RNA PNA duplexes 입체 충돌 왓슨 크릭에서 L G 쌍 같은 존재 때문에 용융 온도 감소. 자외선 흡 광도 감지 열 녹는 데이터는 2 aminopurine 형광 적정 데이터, 또한 그 질문 및 L 잔류물을 포함 하는 PNA에 바인딩되지 않기 ssRNA appreciably 표시와 일치 (그림 6B, 6E 6F). Q 자료 (그림 1E 왓슨 크릭 같은 L G 쌍에 비해 왓슨 크릭 같은 Q G 쌍 (그림 1F)의 형성에 더 중요 한 입체 충돌 있다 L 자료 보다 더 불안정은 Q 자료 통합 ).
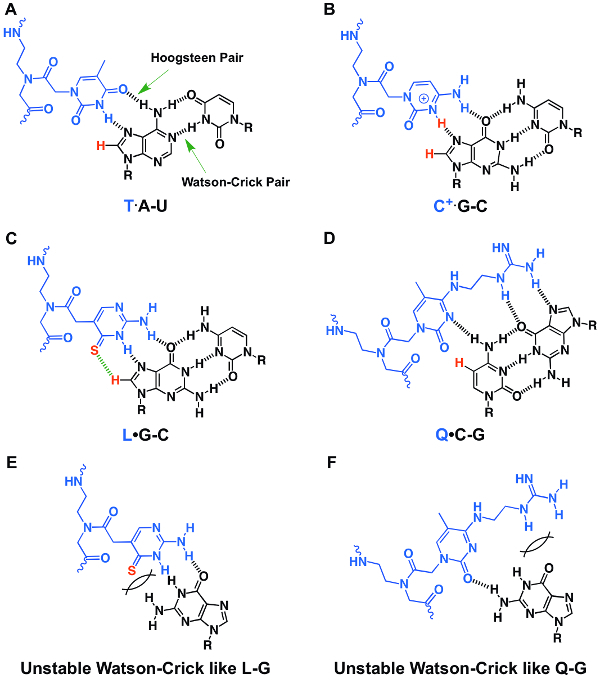
그림 1 : 안정적인 기본 트리플 하 고 불안정 한 기초 쌍 구조의 화학 구조. (A-D) 주요 홈 PNA· T·의 세 배 RNA2 기본 (A) A U, C+· G-C (B), L· G-C (C), 및 Q· C-G (D)입니다. (E, F) L-G (E)와 Q-G (F)의 PNA RNA 기본적인 쌍 같은 불안정 한 왓슨 크릭. 편지 R RNA의 설탕 인산 염 등뼈를 나타냅니다. 수소 결합은 검은색 파선으로 표시 됩니다. 그림 참조31에서 재현 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2 : PNA 단위체의 화학 구조. 4 PNA 단위체 (T, C, L, 및 Q) 표시 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
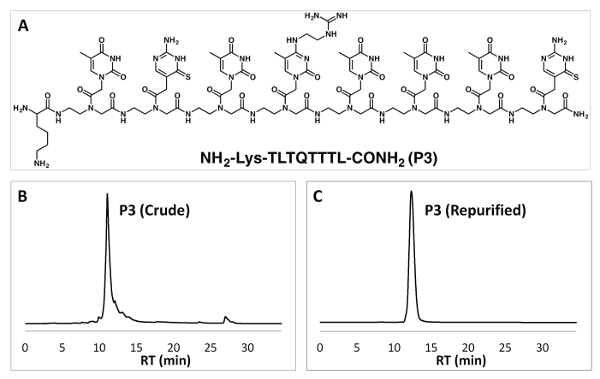
그림 3 : PNA 올리고 머와 RP HPLC에 의해 정화의 화학 구조. (A) PNA 시퀀스 P3의 화학 구조. (B, C) 원유 PNA P3의 RP HPLC 데이터 (B) 다시 정화 PNA P3 (C). 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
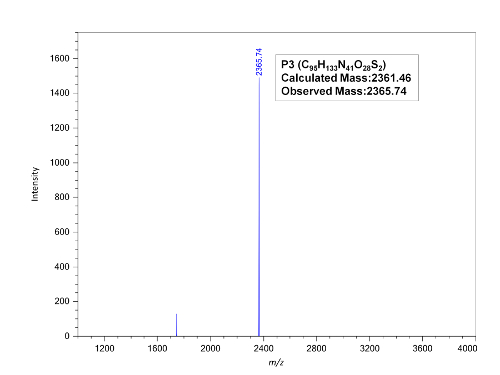
그림 4 : 정화 PNA P3의 MALDI TOF 스펙트럼. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
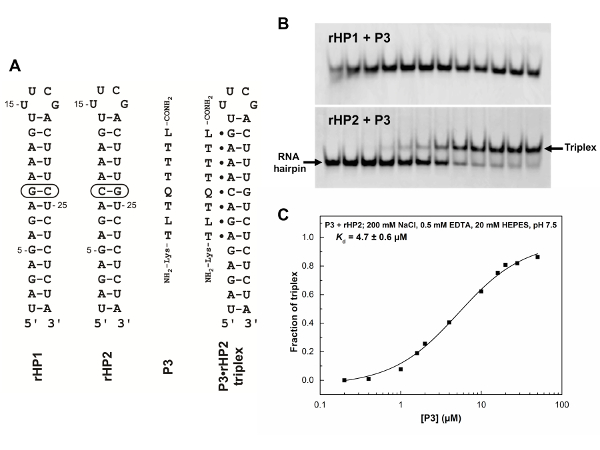
그림 5 : RNA 머리 핀 및 PNA 시퀀스와 비 변성 시키기 페이지 바인딩 특성. (A) RNA 헤어핀 (rHP1 및 rHP2), PNA P3, 그리고는 PNA· RNA2 세 겹 PNA P3 및 rHP2 사이 형성. (B) 비 변성 시키기 페이지 (12%) rHP1 및 rHP2 바인딩 PNA P3에 대 한 결과. 인큐베이션 버퍼가 200 m m NaCl, 0.5 m m EDTA, 20 mM HEPES, pH 7.5입니다. 로드 된 RNA 헤어핀 (rHP1 및 rHP2) 20 µ L에 1 µ M에 있다. 오른쪽에서 왼쪽 차선에서 PNA 농도 0, 0.2, 0.4, 1, 1.6, 2, 4, 10, 16, 20, 28, 50 µ M. PNA P3 않습니다 하지 바인딩합니다 rHP1 하 고 (위 패널) 하지만 rHP2에 바인딩합니다 (하단 패널). P3 rHP2 바인딩을 위해 (C) Kd 결정. 세 겹 형성 (Y)의 분수는 PNA 농도 대 한 플롯은. 그림은 참조31에서 적응. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
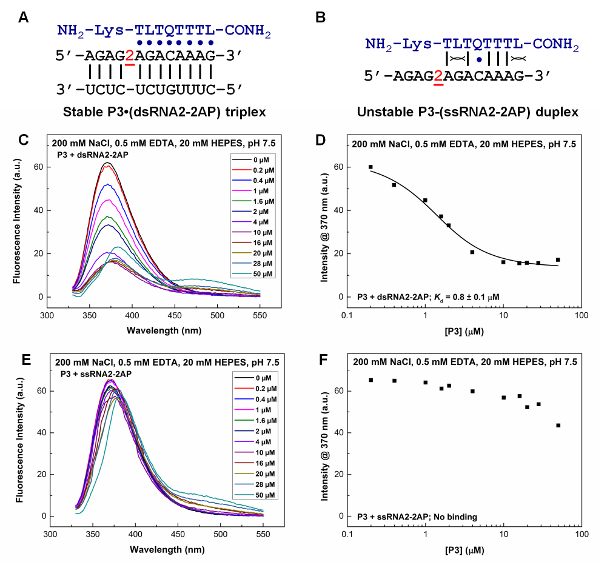
그림 6 : PNA P3 바인딩의 형광 적정 연구2 aminopurine 표시 된 RNAs 하. 2 aminopurine 잔류물 RNA 순서에서 '2'로 지정 됩니다. 인큐베이션 버퍼가 200 m m NaCl, 0.5 m m EDTA, 20 mM HEPES, pH 7.5입니다. (A)는 PNA· RNA2 세 겹 P3 및 2 aminopurine 표시 된 dsRNA (dsRNA2-2AP) 사이 형성. (B) 가상 PNA RNA 이중 P3 및 2 aminopurine 레이블 ssRNA (ssRNA2-2AP) 사이 형성. (C, E) 형광 방출 스펙트럼 2 aminopurine 표시 된 RNA 이중 (1 µ M) 및 ssRNA (1 µ M), 각각,와 P3 농도 pH 7.5에 다양합니다. 약 475에 피크 nm PNA에 L 자료의 약한 형광 방출 예정 이다. (D, F) K d 결정 2 aminopurine 형광 강도 플롯에 따라 (370에서 nm)의 RNA 이중 및 ssRNA, 각각, PNA P3 농도 대. 그림은 참조31에서 적응. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 7 : RNA PNA duplexes에 대 한 결과 용 해 열. 인큐베이션 버퍼 200 m m NaCl, 0.5 m m EDTA, 20 m NaH2포4, pH 7.5 m 이다. 모든 샘플의 단일 가닥 RNA (ssRNA1)와 PNA 130 µ L. (A) 단일 가닥 RNA (ssRNA1)에, PNAs (P1, P2, P3, P4, 및 P5)와 PNA P3 및 평행 방향에 ssRNA1 사이 형성 하는 가상 PNA RNA 이중 5 µ M를 포함. Q-G와 L G 쌍 같은 왓슨 크릭 입체 충돌을 가리킨다. (B) 융해 곡선 다른 PNAs에 대 한 ssRNA1에 바인딩. 녹는 온도 녹는 전환으로 곡선에 대 한 표시 됩니다. 그림은 참조31에서 적응.
토론
RNA 이중 바인딩 PNA 올리고 (예를 들어, 10 메)는 중간 크기의 분자 그리고 따라서 표시할 수 있습니다 바인딩 시 전기 이동 기동성 교대 RNAs를 비교 또는 약간 더 큰 크기 (예를 들어, 50-메 르 또는 더 작은). RNA PNA 보다 상당히 큰 경우에, 제한 된 젤 기동성 교대 때문 RNA에 PNA의 적정 작동 하지 않을 수 있습니다. 따라서, 비 변성 시키기 페이지 분석 실험에 대 한 큰 RNA는 잘릴 수 있습니다. Fluorophore 표시 된 PNA에 큰 RNA의 적정 감시를 위한 세 겹 형성 비 변성 시키기 agarose 젤에 의해 젤40중간 로드 샘플 수 있습니다.
RNA의 일정 총 농도와 비 변성 시키기 페이지 적정 실험을 위해 우리는 일반적으로 효율적인 게시물 ethidium 평범한 사람에 의해 무료 RNA와 트리플 밴드의 얼룩에 대 한 1 µ M의 레이블이 RNA 농도 사용 합니다. RNA 농도 0.2 µ M로 낮은 RNA 구조31에 따라 충분 한 있을 수 있습니다. 레이블이 없는 RNA의 농도 (0.2 µ M)를 정확 하 게 측정 될 수 있다 Kd 값은 0.2 µ M에 대 한 또는 더 큰 이어야 합니다 결정 합니다. 다른 얼룩 염료 착 효율을 향상 시키기 위해 사용할 수 있습니다. 또는 게시 되지 않은 데이터 Cy3 염료-표시 된 RNAs 꽉 바인딩 이벤트 측정 하 비 변성 시키기 페이지 실험에 사용할 수 있습니다 것이 좋습니다.
사실은 2-aminopurine은 적당히 형광만, 2-aminopurine 형광 적정도 Kd 값 0.2 µ M31이상 가까이 바인딩의 측정에 제한 됩니다. RNA 또는 PNA 측정 형광 적정을 통해 솔루션에 상대적으로 꽉 바인딩 바인딩 변경 형광 신호53,54, 경우에 상대적으로 밝은 염료와 함께 표시 될 수 있습니다. 55.
DsRNA 바인딩 PNAs에 의해 RNA 구조를 대상으로 전략 RNAs의 제한 된 수에 대 한 테스트 되었습니다. 바인딩 속성 따라 달라질 수 있습니다 다른 시퀀스와 자료 쌍 작곡으로 dsRNAs 높습니다. 하나는 항상 TFPNAs의 디자인에 대 한 이중의 퓨 린 부유한 가닥을 선택할 수 있습니다. 어떻게 연속 Q·를 이해 하는 것이 중요 C G 세 배는 삼박자의 안정성에 영향을 수 있습니다. 더 광범위 한 시퀀스 종속 연구 명확 하 게 TFPNAs의 시퀀스 종속 바인딩 속성을 이해 하는 것 필요 합니다.
TFPNAs의 바인딩 선호도 길이 증가 및/또는 추가는 기초 및 백본56,57 TFPNAs의 수정 더 향상 될 수 있습니다. 그러나, 연속 이중 지역 수 있습니다 자주 구성 하지 중단 없이 10 개 이상의 연속 기본적인 쌍의 비 왓슨 크릭 구조에 의해. 하나 비 왓슨 크릭 구조 dsRNA 지역에 인접 한의 인식에 대 한 작은 분자와 TFPNAs 켤레 있습니다. 원칙적으로, TFPNA-작은 분자 공액 바인딩 친 화력과 특이성 TFPNA 또는 작은 분자 혼자에 비해 향상 예정입니다. 그러나,는 활용58,59,60,61,,6263,64에 대 한 링커의 화학 및 물리적 특성 최적화 해야 합니다.
TFPNAs 바인딩할 수 있는 선택적으로 dsRNAs ssRNAs와 dsDNAs는 사실 매우 유용한 화학 프로브 및 RNA 구조 역학 및 상호작용의 규칙을 통해 잠재적인 치료 ligands로 TFPNAs를 개발 하는 것을 제안합니다 단백질 그리고 대사 산물입니다. TFPNAs의 세포질 통풍 관 작은 분자, 펩 티 드, 등 나노 입자, 세포 관통 moieties로 활용 또는 리5,6 같은 supramolecular 구조 complexation을 통해 촉진 될 수 있습니다. ,12,17,25,31,41,,6566. 더 fluorophores 등 radioisotopes bioimaging 태그와 TFPNAs의 기능화 용이 하 게 하는 검색, 이미징, 및 살아있는 유기 체에서 기능 RNA 구조를 대상으로 수 있습니다.
공개
여기에서 보고 된 작업에 따라 (팻/179/14/15/PCT) 특허 출원을 제기 했다.
감사의 말
이 작품은 싱가포르 교육 (MOE) 계층 1의 (RGT3/13 및 RG42/15 G.C.)와 모에 계층 2 (MOE2013-t 2-2-024 및 MOE2015-t 2-1-028 G.C.에)에 의해 지원 되었다.
자료
| Name | Company | Catalog Number | Comments |
| Molecular sieves, 4A, 1-2 mm diameter pellets | Alfa Aesar | 87956 | |
| 4-Methylbenzhydrylamine hydrochloride (MBHAŸHCl) | Sigma-Aldrich | 532444 | |
| N,N-diisopropylethylamine (DIPEA) | Alfa Aesar | A11801 | |
| (Benzotriazol-1-yl-oxy)tripyrrolidinophosphonium hexafluorophosphate (PyBOP) | Alfa Aesar | B25251 | |
| Acetic anhyride | Sigma-Aldrich | 320102 | |
| Kaiser Test Kit | Sigma-Aldrich | 60017 | |
| Trifluoroacetic acid (TFA) | Alfa Aesar | L06374 | |
| Unmodified PNA monomers | ASM Research Chemicals GmbH | 5004007, 5004008, 5004009, 5004010 | |
| Boc-Lys(Z)-OH / Fmoc-Lys(Boc)-OH | Sigma-Aldrich | B8389 / 47624 | |
| Thioanisole | Alfa Aesar | A14846 | |
| 1,2-Ethanedithiol | Alfa Aesar | L12865 | |
| Trifluoromethanesulfonic acid | Alfa Aesar | A10173 | |
| LiChrosper® 100 RP-18 endcapped (5 µm) LiChroCART® 250-4 | Merck Millipore | 150838 | |
| RNA Oligos | Sigma-Aldrich | Customized | |
| α-cyano-4-hydroxycinnamic acid (CHCA) | Sigma-Aldrich | 39468 | |
| Ethylenediaminetetraacetic acid (EDTA) | Alfa Aesar | J15694 | |
| HEPES | Lonza | 17-737E | |
| Acrylamide | Sigma-Aldrich | A8887 | |
| N.N'-methylenebisacrylamide | Sigma-Aldrich | 146072 | |
| Ammonium persulfate (APS) | Bio-rad | 161-0700 | |
| Tetramethylethylenediamine (TEMED) | Bio-rad | 161-0800 | |
| 10X Tris-Borate-EDTA (TBE) Buffer, pH 8.3 | 1st Base | BUF-3010-10X1L | |
| Glycerol | Promega | H5433 | |
| Ethidium bromide (10 mg/mL) | Bio-rad | 161-0433 | |
| High Precision Cell (Quartz Suprasil, 200-2500 nm) | Hellma Analytics | 105.250-QS |
참고문헌
- Cech, T. R., Steitz, J. A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157 (1), 77-94 (2014).
- Velagapudi, S. P., Gallo, S. M., Disney, M. D. Sequence-based design of bioactive small molecules that target precursor microRNAs. Nat Chem Biol. 10, 291-297 (2014).
- Patil, K. M., Chen, G., Jurga, S., Erdmann, V. A., Barciszewski, J. . Modified Nucleic Acids in Biology and Medicine. , 299-317 (2016).
- Hyrup, B., Nielsen, P. E. Peptide nucleic acids (PNA): synthesis, properties and potential applications. Bioorg Med Chem. 4 (1), 5-23 (1996).
- Shiraishi, T., Nielsen, P. E. Improved cellular uptake of antisense peptide nucleic acids by conjugation to a cell-penetrating peptide and a lipid domain. Methods Mol Biol. 751, 209-221 (2011).
- Shiraishi, T., Nielsen, P. E. Nanomolar cellular antisense activity of peptide nucleic acid (PNA) cholic acid ("umbrella") and cholesterol conjugates delivered by cationic lipids. Bioconjugate Chem. 23 (2), 196-202 (2012).
- Khoo, B., Roca, X., Chew, S. L., Krainer, A. R. Antisense oligonucleotide-induced alternative splicing of the APOB mRNA generates a novel isoform of APOB. BMC Mol Biol. 8, 3 (2007).
- Stein, C. A., et al. Efficient gene silencing by delivery of locked nucleic acid antisense oligonucleotides, unassisted by transfection reagents. Nucleic Acids Res. 38 (1), 3 (2010).
- Kole, R., Krainer, A. R., Altman, S. RNA therapeutics: beyond RNA interference and antisense oligonucleotides. Nat Rev Drug Discov. 11 (2), 125-140 (2012).
- Peacey, E., Rodriguez, L., Liu, Y., Wolfe, M. S. Targeting a pre-mRNA structure with bipartite antisense molecules modulates tau alternative splicing. Nucleic Acids Res. 40 (19), 9836-9849 (2012).
- Stenvang, J., Petri, A., Lindow, M., Obad, S., Kauppinen, S. Inhibition of microRNA function by antimiR oligonucleotides. Silence. 3 (1), 1 (2012).
- Ma, X., et al. Intracellular delivery of antisense peptide nucleic acid by fluorescent mesoporous silica nanoparticles. Bioconjugate Chem. 25 (8), 1412-1420 (2014).
- Wojtkowiak-Szlachcic, A., et al. Short antisense-locked nucleic acids (all-LNAs) correct alternative splicing abnormalities in myotonic dystrophy. Nucleic Acids Res. 43 (6), 3318-3331 (2015).
- Lenartowicz, E., et al. Antisense Oligonucleotides Targeting Influenza A Segment 8 Genomic RNA Inhibit Viral Replication. Nucleic Acid Ther. 26 (5), 277-285 (2016).
- Avitabile, C., et al. Targeting pre-miRNA by peptide nucleic acids: a new strategy to interfere in the miRNA maturation. Artif DNA PNA XNA. 3 (2), 88-96 (2012).
- Barczak, A. K., et al. RNA signatures allow rapid identification of pathogens and antibiotic susceptibilities. Proc Natl Acad Sci USA. 109 (16), 6217-6222 (2012).
- Das, I., et al. A peptide nucleic acid-aminosugar conjugate targeting transactivation response element of HIV-1 RNA genome shows a high bioavailability in human cells and strongly inhibits tat-mediated transactivation of HIV-1 transcription. J Med Chem. 55 (13), 6021-6032 (2012).
- Fabani, M. M., et al. Efficient inhibition of miR-155 function in vivo by peptide nucleic acids. Nucleic Acids Res. 38 (13), 4466-4475 (2010).
- Riguet, E., et al. A peptide nucleic acid-neamine conjugate that targets and cleaves HIV-1 TAR RNA inhibits viral replication. J Med Chem. 47 (20), 4806-4809 (2004).
- Torres, A. G., et al. Chemical structure requirements and cellular targeting of microRNA-122 by peptide nucleic acids anti-miRs. Nucleic Acids Res. 40 (5), 2152-2167 (2012).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Mol Cell Proteomics. 12 (6), 1539-1552 (2013).
- Wesolowski, D., et al. Basic peptide-morpholino oligomer conjugate that is very effective in killing bacteria by gene-specific and nonspecific modes. Proc Natl Acad Sci USA. 108 (40), 16582-16587 (2011).
- Armitage, B. A. The impact of nucleic acid secondary structure on PNA hybridization. Drug Discov Today. 8 (5), 222-228 (2003).
- Thomas, S. M., et al. Antitumor effects of EGFR antisense guanidine-based peptide nucleic acids in cancer models. ACS Chem Biol. 8 (2), 345-352 (2013).
- Bahal, R., McNeer, N. A., Ly, D. H., Saltzman, W. M., Glazer, P. M. Nanoparticle for delivery of antisense gammaPNA oligomers targeting CCR5. Artif DNA PNA XNA. 4 (2), 49-57 (2013).
- Adams, B. D., Parsons, C., Walker, L., Zhang, W. C., Slack, F. J. Targeting noncoding RNAs in disease. J. Clin. Invest. 127 (3), 761-771 (2017).
- Matsui, M., Corey, D. R. Non-coding RNAs as drug targets. Nat Rev Drug Discov. 16 (3), 167-179 (2017).
- Devi, G., Zhou, Y., Zhong, Z., Toh, D. -. F. K., Chen, G. RNA triplexes: from structural principles to biological and biotech applications. Wiley Interdiscip Rev RNA. 6 (1), 111-128 (2015).
- Rozners, E. Recent Advances in Chemical Modification of Peptide Nucleic Acids. J Nucleic Acids. 2012, 8 (2012).
- Devi, G., Yuan, Z., Lu, Y., Zhao, Y., Chen, G. Incorporation of thio-pseudoisocytosine into triplex-forming peptide nucleic acids for enhanced recognition of RNA duplexes. Nucleic Acids Res. 42 (6), 4008-4018 (2014).
- Toh, D. K., et al. Incorporating a guanidine-modified cytosine base into triplex-forming PNAs for the recognition of a C-G pyrimidine-purine inversion site of an RNA duplex. Nucleic Acids Res. 44 (19), 9071-9082 (2016).
- Li, M., Zengeya, T., Rozners, E. Short peptide nucleic acids bind strongly to homopurine tract of double helical RNA at pH 5.5. J Am Chem Soc. 132 (25), 8676-8681 (2010).
- Gupta, P., Zengeya, T., Rozners, E. Triple helical recognition of pyrimidine inversions in polypurine tracts of RNA by nucleobase-modified PNA. Chem Commun. 47 (39), 11125-11127 (2011).
- Zengeya, T., Li, M., Rozners, E. PNA containing isocytidine nucleobase: synthesis and recognition of double helical RNA. Bioorg Med Chem Lett. 21 (7), 2121-2124 (2011).
- Gupta, P., Muse, O., Rozners, E. Recognition of double-stranded RNA by guanidine-modified peptide nucleic acids. Biochemistry. 51 (1), 63-73 (2012).
- Zengeya, T., Gupta, P., Rozners, E. Triple-helical recognition of RNA using 2-aminopyridine-modified PNA at physiologically relevant conditions. Angew Chem Int Ed. 51 (50), 12593-12596 (2012).
- Muse, O., et al. Sequence selective recognition of double-stranded RNA at physiologically relevant conditions using PNA-peptide conjugates. ACS Chem Biol. 8 (8), 1683-1686 (2013).
- Hnedzko, D., Cheruiyot, S. K., Rozners, E. Using Triple Helix Forming Peptide Nucleic Acids for Sequence-selective Recognition of Double-stranded RNA. Curr Protoc Nucleic Acid Chem. 58, 61-64 (2014).
- Zengeya, T., Gupta, P., Rozners, E. Sequence selective recognition of double-stranded RNA using triple helix-forming peptide nucleic acids. Methods Mol Biol. 1050, 83-94 (2014).
- Endoh, T., Hnedzko, D., Rozners, E., Sugimoto, N. Nucleobase-Modified PNA Suppresses Translation by Forming a Triple Helix with a Hairpin Structure in mRNA In Vitro and in Cells. Angew Chem Int Ed. 55 (3), 899-903 (2016).
- Hnedzko, D., McGee, D. W., Karamitas, Y. A., Rozners, E. Sequence-selective recognition of double-stranded RNA and enhanced cellular uptake of cationic nucleobase and backbone-modified peptide nucleic acids. RNA. 23 (1), 58-69 (2017).
- Wexselblatt, E., Esko, J. D., Tor, Y. On guanidinium and cellular uptake. J Org Chem. 79 (15), 6766-6774 (2014).
- Kaiser, E., Colescott, R. L., Bossinger, C. D., Cook, P. I. Color test for detection of free terminal amino groups in the solid-phase synthesis of peptides. Anal. Biochem. 34 (2), 595-598 (1970).
- Roberts, R. W., Crothers, D. M. Stability and properties of double and triple helices: dramatic effects of RNA or DNA backbone composition. Science. 258 (5087), 1463-1466 (1992).
- Zhou, Y., et al. Recognition of RNA duplexes by chemically modified triplex-forming oligonucleotides. Nucleic Acids Res. 41 (13), 6664-6673 (2013).
- Gisin, B. F. The monitoring of reactions in solid-phase peptide synthesis with picric acid. Anal. Chim. Acta. 58 (1), 248-249 (1972).
- Gogoi, K., Mane, M. V., Kunte, S. S., Kumar, V. A. A versatile method for the preparation of conjugates of peptides with DNA/PNA/analog by employing chemo-selective click reaction in water. Nucleic Acids Res. 35 (21), 139 (2007).
- Shabanpoor, F., Gait, M. J. Development of a general methodology for labelling peptide-morpholino oligonucleotide conjugates using alkyne-azide click chemistry. Chem Commun. 49 (87), 10260-10262 (2013).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A Stepwise Huisgen Cycloaddition Process: Copper(I)-Catalyzed Regioselective "Ligation" of Azides and Terminal Alkynes. Angew Chem Int Ed. 41 (14), 2596-2599 (2002).
- Haaima, G., Hansen, F. H., Christensen, L., Dahl, O., Nielsen, P. E. Increased DNA binding and sequence discrimination of PNA oligomers containing 2,6-diaminopurine. Nucleic Acids Res. 25 (22), 4639-4643 (1997).
- Schroeder, S. J., Turner, D. H. . Methods Enzymol. 468, 371-387 (2009).
- McDowell, J. A., Turner, D. H. Investigation of the Structural Basis for Thermodynamic Stabilities of Tandem GU Mismatches: Solution Structure of (rGAGGUCUC)2 by Two-Dimensional NMR and Simulated Annealing. Biochemistry. 35 (45), 14077-14089 (1996).
- Sato, T., Sato, Y., Nishizawa, S. Triplex-Forming Peptide Nucleic Acid Probe Having Thiazole Orange as a Base Surrogate for Fluorescence Sensing of Double-stranded RNA. J Am Chem Soc. 138 (30), 9397-9400 (2016).
- Cheruiyot, S. K., Rozners, E. Fluorescent 2-Aminopyridine Nucleobases for Triplex-Forming Peptide Nucleic Acids. ChemBioChem. 17 (16), 1558-1562 (2016).
- Sato, T., Sato, Y., Nishizawa, S. Optimization of the Alkyl Linker of TO Base Surrogate in Triplex-Forming PNA for Enhanced Binding to Double-Stranded RNA. Chem Eur J. 23 (17), 4079-4088 (2017).
- Virta, P. M., Tahtinen, V., Granqvist, L., Murtola, M., Stromberg, R. 19F NMR spectroscopic analysis of the binding modes in triple helical PNA/microRNA-complexes. Chem Eur J. , (2017).
- Zengeya, T., Gindin, A., Rozners, E. Improvement of sequence selectivity in triple helical recognition of RNA by phenylalanine-derived PNA. Artif DNA PNA XNA. 4 (3), 69-76 (2013).
- Moses, A. C., Huang, S. W., Schepartz, A. Inhibition of Rev.RRE complexation by triplex tethered oligonucleotide probes. Bioorg Med Chem. 5 (6), 1123-1129 (1997).
- Ben Gaied, N., Zhao, Z., Gerrard, S. R., Fox, K. R., Brown, T. Potent triple helix stabilization by 5',3'-modified triplex-forming oligonucleotides. ChemBioChem. 10 (11), 1839-1851 (2009).
- Grimm, G. N., Boutorine, A. S., Lincoln, P., Norden, B., Helene, C. Formation of DNA triple helices by an oligonucleotide conjugated to a fluorescent ruthenium complex. ChemBioChem. 3 (4), 324-331 (2002).
- Tran, T., et al. Targeting the r(CGG) Repeats That Cause FXTAS with Modularly Assembled Small Molecules and Oligonucleotides. ACS Chem Biol. , (2014).
- Gianolio, D. A., Segismundo, J. M., McLaughlin, L. W. Tethered naphthalene diimide-based intercalators for DNA triplex stabilization. Nucleic Acids Res. 28 (10), 2128-2134 (2000).
- Rumney, S., Kool, E. T. Structural Optimization of Non-Nucleotide Loop Replacements for Duplex and Triplex DNAs. J Am Chem Soc. 117, 5635-5646 (1995).
- Stafford, R. L., Dervan, P. B. The reach of linear protein-DNA dimerizers. J Am Chem Soc. 129 (45), 14026-14033 (2007).
- Gupta, A., Bahal, R., Gupta, M., Glazer, P. M., Saltzman, W. M. Nanotechnology for delivery of peptide nucleic acids (PNAs). J Control Release. 240, 302-311 (2016).
- Avitabile, C., et al. Incorporation of Naked Peptide Nucleic Acids into Liposomes Leads to Fast and Efficient Delivery. Bioconjugate Chem. 26 (8), 1533-1541 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유