Method Article
동시 매핑 및 인간 미토 콘 드리 아 DNA에 Ribonucleotides의 정량
요약
여기 우리는 동시에 quantitate 의무가 메서드와 알칼리 가수분해와 후속 5´-엔드 genomic DNA의 효소 분열을 결합 하 여 단일 뉴클레오티드 해상도 높은 그대로 DNA에 게놈 넓은 지도 ribonucleotides 설명 시퀀싱.
초록
Ribonucleotides는 게놈에 존재의 수를 추정 하 설립된 접근은 제한 법인된 ribonucleotides 템플릿으로 짧은 합성 DNA 파편 또는 플라스 미드를 사용 하 고 전체 결과 추정 하는 다음의 정량 게놈입니다. 또는, 알칼리 젤 또는 남부도 말을 사용 하 여 ribonucleotides는 게놈에 존재의 수를 견적 될 수 있습니다. 최근 비보에 접근 위치와 포함 된 ribonucleotides의 id를 제공 하는 ribonucleotides의 게놈 넓은 매핑 허용 다음-세대 시퀀싱을 사용 합니다. 그러나, 그들은 정량의 게놈으로 통합 되는 ribonucleotides의 수를 허용 하지 않습니다. 여기는 동시에 지도 하 고 차세대 시퀀싱에 의해 인간의 미토 콘 드리 아 DNA에서 에 비보에 통합 하는 ribonucleotides의 수를 quantitate 하는 방법에 설명 합니다. 우리가 매우 그대로 DNA를 사용 하 고 시퀀스 특정 이중 가닥 나누기는 endonuclease, 알칼리 이후에 hydrolyzing 법인된 ribonucleotides와 함께 그것을 소화 하 여 소개. 생성 된 끝은 어댑터와 출혈 그리고이 끝 다음-세대 시퀀싱 컴퓨터에서 시퀀싱 되. Ribonucleotides의 절대 수는 순서 특정 endonuclease 인식 사이트에서 읽기 평균 수 당 인식 사이트 밖에 서 읽기의 수로 계산할 수 있습니다. 이 프로토콜 또한 지도 및 DNA에 무료 흠을 quantitate 활용 될 수 있습니다 및 수 5´ 오를 처리할 수 있는 다른 DNA 병 변 지도를 적응 또는 5´-인산 염 끝. 또한,이 방법은 적합 한 참조 게놈은 사용할 수 있는 모든 유기 체에 적용할 수 있습니다. 이 프로토콜은 따라서 DNA 복제, 5´ 간 처리, DNA 손상, 및 DNA 복구 하는 중요 한 도구를 제공 합니다.
서문
진 핵 세포에서 ribonucleotides (rNTPs)의 농도 deoxyribonucleotides (dNTPs)1의 농도 보다 훨씬 높다. DNA polymerases ribonucleotides, 차별만이 차별 완벽 한 이며, 결과적으로, deoxyribonucleotides 대신 ribonucleotides 수 있습니다 통합할 수 게놈 DNA 복제 하는 동안. Ribonucleotides 가장 일반적인 정식이 아닌 뉴클레오티드 게놈2에 통합 될 수 있습니다. 대부분 이러한 ribonucleotides의 RNase h 2 시작된 ribonucleotide 절단 선 (RER) 또는 Topoisomerase 1 (참조3검토) 오카자키 조각 성숙 하는 동안 제거 됩니다. Ribonucleotides는 제거할 수 없습니다 DNA2,4 에 안정적으로 통합 유지 하 고 (검토5검토) 하는 유해 하 고 유익한 방법으로 그것을 영향을 미칠 수 있습니다. 긍정적인 신호 역할을 할 수 있는, 게다가 예 유형 짝짓기에 스위치 Schizosaccharomyces pombe 6 와 불일치 하는 동안 초기 DNA 가닥을 표시 복구 (MMR)7,8, ribonucleotides에 영향을 미칠는 구조9 그리고 일차 스트레스 및 게놈 불안정성11의 결과로 그들의 ribose10, 2´-수 산 기 그룹이 주변 DNA의 안정성. 게놈 DNA (gDNA)에 ribonucleotides와 복제 및 복구 메커니즘으로 게놈 안정성에 대 한 의미에 그들의 관련성의 풍부한 게놈 넓은 방법에 있는 그들의 정확한 발생과 주파수를 조사 하는 이유를 제공 합니다.
RNase h 2 활동 인간의 미토 콘 드리 아에서 발견 되지 않은 그리고 ribonucleotides는 따라서 미토 콘 드리 아 DNA (mtDNA)에 효율적으로 되지 제거. 여러 경로 인간의 미토 콘 드리 아에 뉴클레오티드의 공급에 참여 하 고 미토 콘 드리 아 뉴클레오티드 풀에서 교란 인간 mtDNA에 ribonucleotides의 높은 수를 발생 시키는 지 여부를 조사, 우리 지도를 quantitate 프로토콜 개발 섬유 아 세포, HeLa 세포, 및 환자 셀 라인12에서 고립 된 인간 mtDNA에 이러한 ribonucleotides.
경쟁 rNTPs 반응에 포함 되어 단일 ribonucleotide 삽입 또는 뇌관 연장 실험을 기반으로하는 대부분의 생체 외에서 (검토 검토13) DNA polymerases' rNTPs에 대 한 선택도 결정 방식 믹스, 식별 또는 ribonucleotide 관 상대 정량 짧은 DNA 템플렛에 허용. 짧은 시퀀스에 양적 접근 하지 dNTP와 rNTP 풀 세포 농도 반영 하 고 따라서 제공 중 합 효소 선택도에 대 한 통찰력 하지만 전체 게놈에 관한 제한 된 의미의 수 있습니다. 그것은 보였다 ribonucleotides는 플라스 미드와 같은 더 긴 DNA 템플렛의 복제 하는 동안 통합의 상대적인 양이 방사선된 dNTPs를 사용 하 여 hydrolyzing14는 알칼리 성 환경 DNA 시퀀싱 젤에 구상 될 수 있다. 또한, gDNA 남쪽 오 점 다음 알칼리 가수분해, 물가 관련 조사 및 ribonucleotide 법인 vivo에서15의 절대 속도의 결정에 분석 되어 있다. 이러한 접근 법인 주파수의 상대 비교를 허용 하지만 위치 또는 법인된 ribonucleotides의 id에 아무 대 한 통찰력을 제공. 더 최근 접근 gDNA에서 vivo에서, 하이든-Seq16, Ribose Seq17, Pu Seq18또는 emRiboSeq19활용 포함 된 ribonucleotides' 같은 ribonucleotide 콘텐츠 분석 감도를 알칼리 또는 RNase h 2 치료, 각각, 다음-세대 시퀀싱 ribonucleotides 게놈 넓은 식별 하 고. 이러한 메서드는 검색 된 ribonucleotides의 절대 법인 주파수에 대 한 통찰력을 제공 하지 않습니다. 하이든-seq 프로토콜 시퀀스 특정 효소 분열의 단계를 추가 하 여 우리가 여기에 편리 하 게 설명 하는 방법은 동시 매핑 수 있도록 시퀀싱 접근에서 얻은 정보를 확장 하 고 정량의 임베디드 ribonucleotides12. 이 방법은 매우 그대로 DNA 추출 생성 될 수 있고 적합 한 참조 게놈은 사용할 수 있는 거의 모든 유기 체에 적용 됩니다. 메서드 quantitate는 nuclease에 의해 소화 될 수 있는 잎 5´ 인산 염 또는 5´ 오 끝을는 병 변의 위치 확인을 적용할 수 있습니다.
지도 및 genomic DNA에서 ribonucleotides을 quantitate, 메서드 시퀀스 특정 endonuclease에 의해 분열을 결합 하 고는 endonuclease에 대 한 특정 인식 순서는 및 5´-5´-인산 염을 생성 하는 알칼리 가수분해 사이트 끝 오 ribonucleotides 있었다 위치에서 끝납니다. 생성 된 무료 끝 어댑터와 출혈 이후에 있기 때문에 차세대 시퀀싱을 사용 하 여 시퀀싱, 그것 매우 그대로 DNA를 사용 하 여 DNA 추출 및 라이브러리 준비 하는 동안 임의의 조각화 방지 중요성 이다. 이러한 읽기 endonuclease 분열에 읽기로 표준화 평가 동시 정량 및 매핑 검색된 ribonucleotides 수 있습니다. 무료 5´ 끝 제어 실험 KCl와 치료에 의해 DNA의 알칼리 가수분해가 대체 어디에서 검색 됩니다. 인수 데이터 ribonucleotide 위치 및 수량에 대 한 통찰력을 제공 하 고 ribonucleotide 콘텐츠 및 법인 주파수에 관하여 분석을 수 있습니다.
프로토콜
이 그림 1에서 설명 하는 프로토콜과 gDNA, ribonucleotides, 치료는에 알칼리 수를 quantitate 수 금지 효소로 소화의 절연 ribonucleotides의 phosphodiester 채권 gDNA, 무료 5´ 오 끝의 인 산화, 어댑터, 두 번째 가닥 합성 및 PCR 증폭 시퀀싱 전에 ssDNA 결 찰에 통합.
1. 어댑터 및 인덱스 뇌관
- 얻을 ARC49 ARC140 oligonucleotides, ARC76/77, 어댑터, ARC78-ARC107 인덱스 뇌관 (표 1 참조).
참고: Oligonucleotides HPLC 정화 해야 합니다. ARC76/77 이중으로 정렬 됩니다. - 트리 스-EDTA (테) 버퍼에 각 oligonucleotide의 준비 100 µ M 재고 솔루션 (재료의 표 참조)-20에서 저장 ° c.
- 차입 버퍼 (EB, 참조 테이블의 재료)에 희석 하 여 ARC67/77의 10 µ M 솔루션 및 ARC49 및 인덱스 뇌관의 2 µ M 솔루션을 준비 합니다. -20 ° c.에 게
2. 성장과 수확의 셀
- 성장 HeLa 세포 70 ml Dulbecco ' s 37에 250 mL 스피너 플라스 크에서 10% 태아 둔감 한 혈 청으로 보완 수정이 글 매체 (DMEM) ° c.
- 셀의 수를 계산 하 고 50 mL 튜브, 200 x g에 5 분 동안 원심 분리기에서에서 5 x 10 6 세포를 수집 하 고는 상쾌한 삭제.
- 1 x 20 mL PBS, 200 x g에 5 분 동안 원심 분리기를 가진 세포를 세척 하 고는 상쾌한 삭제.
- 동결-20 ° C에서 펠 릿 또는 DNA 정화 계속.
3. DNA 정제 및 정량
- gDNA 아래에 설명 된 페 놀-클로 프롬 적 출을 사용 하 여 정화.
- Resuspend 2 mL 세포의 용 해 버퍼의 셀 (테이블의 자료 참조)와 열 블록에 42 ° C에서 30 분 동안 품 어.
주의: 세포의 용 해 버퍼 유해 구성 요소를 포함합니다. SDS 솔루션은 자극, 가수분해 K은 sensitizing, 자극, 및 독성. 보호 옷과 장갑을 착용. - 두 2 mL 튜브에 샘플을 분할 하 고 페 놀-클로 프롬-isoamyl 알콜 (25:24:1)의 1 볼륨 (V) 추가.
주의: 페 놀-클로 프롬-isoamyl 알콜은 독성, 변이 원 성, 부식성, 및 수생 환경에 유해. 증기 두건에서 사용 하 고, 보호 옷과 장갑을, 착용 하 고 특별 한 페 놀-클로 프롬 폐기물에서 폐기.
실 온에서 30-60 s에 대 한 반전 및 15000 x 5 분 원심 분리기 - 믹스 g.
참고: 결과 왜곡할 것 이다 무작위 스트랜드 나누기 소개 피하려고 하지 소용돌이 DNA 할. - 새 2 mL 튜브 위, 수성 단계 전송 및 페 놀-클로 프롬-isoamyl 알콜 (25:24:1)의 V 1.
- 반전 및 원심 분리기에서 15000 x g 4에 5 분 동안 혼합 ° c.
- 전송 20 µ L을 추가 하 고 새로운 2 mL 튜브에 위, 수성 단계 NaCl (5m)과 차가운 소 프로 파 놀의 V 1.
주의: 소 프로 파 놀, 자극, 가연성 및 독성입니다. 통풍이 캐비닛에 그것을 저장, 보호 옷과 장갑, 착용 하 고 화 염에서 멀리 유지. - 반전 하 여 혼합 및 적어도 1 h-20에 대 한 품 어 ° c.
- G, 4 ° C와 폐기 상쾌한 x 15, 000에서 20 분 동안 원심 분리기.
- 세척 DNA 펠 릿 200 µ L 찬 70% 에탄올, 4 ° C에서 15000 x g에서 20 분 동안 원심 및 상쾌한 삭제.
주의: 70% 에탄올은 가연성 및 자극. 통풍이 캐비닛, 보호 옷을 입고, 장갑,-20 ° C, 그렇지 않으면 저장소에서 작업 솔루션을 유지 하 고 화 염에서 멀리 유지. - DNA 펠 릿 20-25 분에 대 한 실 온에서 건조
- 100 µ L 테 펠 릿 분해 DNA 버퍼링 하 고 1 개의 관에 샘플 수영장.
- Resuspend 2 mL 세포의 용 해 버퍼의 셀 (테이블의 자료 참조)와 열 블록에 42 ° C에서 30 분 동안 품 어.
- Quantitate DNA 농도 제조 업체에 따르면 dsDNA 정량 시 약을 사용 하 여 ' s 사양 (재료의 표 참조).
참고: dsDNA 정량 시 약을 사용 하는 spectrophotometric DNA 정량 잔여 페 놀에 의해 영향을 받을 수 있기 때문에. - -20 ° C에 게 DNA HincII 치료를 계속 하거나.
4. HincII 치료와 알칼리 가수분해
5 µ L 10 x 버퍼 3.1, 1 µ L (10 U)를 포함 하는- 반응에서 다이제스트 1 µ g의 DNA 혼합 HincII, 및 nuclease 무료 H 2 O 50 µ L의 최종 볼륨을.
참고: 결 찰에 대 한 최적의 조건을 달성 하기 위해, 두 번째 가닥 합성 및 PCR 증폭, 그것은 필요할 수 있습니다 예상 된다 DNA ribonucleotides의 매우 낮은 수를 포함 하는 경우 입력된 DNA 양을 증가 시키는. 마찬가지로, 그것은 ribonucleotides의 수는 매우 높은 경우 입력된 DNA를 감소 해야 할 수도 있습니다. - 37 ° c.에 30 분 동안 품 어
- 정화 HincII 치료 DNA 상자성 구슬.
참고: 관 뚜껑 열고 튜브를 열어서 펠 렛을 방해 하지 하려면 다음 단계에 계속.- 각 상자성 구슬의 1.8 V 추가 샘플, 신중 하 게 pipetting, 혼합 및 10 분에 대 한 실 온에서 품 어
- 작은 구슬 5 분에 대 한 다음 제거 하 고 상쾌한을 버리고 자기 랙 사용.
- 약 30 s 다음 제거 및 삭제는 상쾌한 70% 에탄올 (실내 온도)의 150 µ L와 펠 릿을 세척.
- 세척 약 30 70% 에탄올 (실내 온도)의 200 µ L와 펠 릿 s 다음 제거 하 고 삭제는 상쾌한.
참고: 잔여 에탄올 10 µ L 피 펫으로 제거할 수 있습니다. 방울 짧게 미리 내려 버리고 수. - 건조 약 15-20 분에 대 한 실 온에서 샘플
참고: 정확한 시간 구슬의 볼륨 및 펠 릿의 모양에 따라 달라 집니다, 산 탄을 시각적으로 확인 한다 따라서. - 자석 선반에서 튜브를 제거 하 고 45 µ L EB, 신중 하 게 pipetting으로 혼합에에서 펠 릿을 elute.
- 5 분 다음 작은 자석 선반에 구슬 하 고 순화 된 DNA의 45 µ L를 사용 하 여 단계 4.4에서에서 품 어.
- 코 (3 M) 또는 50 µ L의 총 볼륨을 만드는 DNA에 KCl (3 M)의 추가 5 µ L.
주의: 3 M 코 솔루션 부식성입니다. 보호 옷과 장갑을 착용. - 품은 교 잡에서 55 ° C에서 2 h 오븐 얼음에 5 분 뒤
참고: 것이 좋습니다 튜브의 균일 한가 열을 유지 하 고 뚜껑에 결로 방지 난방 블록 보다는 오븐에 코 치료를 수행 하. - 침전 DNA 10 µ L 나트륨 아세테이트를 추가 하 여 (3 M, pH = 5.2) 및 125 µ L 감기 100% 에탄올. 5 분 ice에 품 어
주의: 100% 에탄올은 가연성 및 자극. 통풍이 캐비닛에 저장, 보호 옷과 장갑을, 착용 하 고 화 염에서 유지. - Centrifuging 21000 x g, 5 분 동안 4 ° C에 의해 gDNA를 펠 렛 하 고는 상쾌한 삭제.
- 세척 DNA 펠 릿 250 µ L 찬 70 %EtOH, 21000 x g, 5 분, 4 ° C에서 원심 및 삭제는 상쾌한.
참고: 제거 하려면 방울, 튜브 수 수 스핀 다운 짧게 다시 하 고 상쾌한 10 µ l 피 펫 제거 될 수 있다. - 게 보이는 어떤 액체 증발 될 때까지 약 5-10 분 동안 열려 튜브에서 건조 하는 펠 릿.
- 실 온에서 30 분 동안 20 µ L EB에에서 게 DNA 펠 릿 디졸브.
5. 5´ 끝 인 산화
- 각 샘플 미리 죄수
- 반응 혼합 준비T4 polynucleotide 키 니 아 제 반응 버퍼, 1 µ L (10 U) 3´-인산 가수분해 효소-마이너스 T4 polynucleotide 키 니 아 제, 및 2.5 µ L ATP (10mm) x 2.5 µ L 10의 isting.
- 전송 19 µ L 각 DNA의 새로운 200 µ L 관으로 샘플 및 열 cycler에서 85 ° C에서 3 분 변성.
- 멋진 DNA 얼음에 샘플링 하 고 반응 혼합의 6 µ L 각 샘플 추가.
- 30 분 및 중지 20 분 65 ° C에서 샘플을 배양 하 여 반응 하는 37 ° C에서 품 반응 혼합
- 정화 DNA로 4.3에 상자성 구슬의 1.8 V를 사용 하 여 설명 하지만 14 µ L EB에에서 elute.
6. ssDNA 결 찰
- 0.5 µ L ATP (2mm), T4 RNA 리가 반응 버퍼, 5 µ L CoCl 3 (NH 3) 6 (10 m m), x 5 µ L 10의 사전에 구성 된 각 샘플에 대 한 반응 혼합 준비 0.5 µ L ARC140 (100 µ M), 25 µ L 50% 말뚝 8000. Pipetting으로 잘 믹스.
주의: CoCl 3 (NH 3) 6 이며 발암, sensitizing, 수생 환경 유해. 보호 옷과 장갑을 착용. - 전송 13 µ L에서 순화 된 DNA의 새로운 200 µ L 튜브 5.5 단계와 열 cycler에서 85 ° C에서 3 분 변성.
- 차가운 얼음에 DNA 각 샘플, pipetting, 혼합 반응 혼합의 36 µ L을 추가 하 고 간단히 스핀 다운.
- 추가 1 µ L (10 U) T4 RNA 리가의 각 반응에 pipetting, 혼합 하 고 짧게 스핀 다운.
- 어두운 하룻밤에 실 온에서 샘플을 품 어.
7. 두 번째 가닥 합성
4.3에 설명 된 대로- Purify DNA 출혈 하지만 상자성 구슬의 0.8 V를 사용 하 여, 작은 구슬 10 분 및 20 µ L EB에에서 elute.
참고: 결 찰 반응 혼합의 높은 점도 인해 pelleting의 첫 번째 단계는 연장. - 전송 20 µ L 새로운 200 µ L PCR 튜브에 DNA 샘플의. 제조 업체에 따라 상자성 구슬의 0.8 V를 사용 하 여 정화 단계 반복 ' s 사양 및 14 µ L EB에에서 elute.
- 10 x T7 DNA 중 합 효소 반응 버퍼, 2 µ L의 2 µ L의 사전에 구성 된 각 샘플에 대 한 반응 혼합 준비 ARC76/77 (2 µ M), 2 µ L dNTPs (2mm), 및 0.8 µ L BSA (1 mg/mL).
- 새 200 µ L 튜브에 DNA를 정화 하는 전송 12.8 µ L, 열 cycler에서 85 ° C에서 3 분 변성.
- 얼음에 DNA를 냉각 하 고 각 샘플, pipetting으로 혼합, 짧게, 스핀 다운 및 실 온에서 5 분 동안 품 어 반응 혼합의 6.8 µ L 추가.
- 각 반응에 0.4 µ L (4 U) T7 DNA 중 합 효소를 추가 하 고 실 온에서 5 분 동안 품 어.
- 정화 DNA로 4.3에 상자성 구슬의 0.8 V를 사용 하 여 설명과 11 µ L EB에에서 elute.
8. PCR 증폭 및 라이브러리 정량
- 준비 새로운 200에서 각 샘플에 대 한 반응 혼합물 µ L 튜브 7.5 µ L의 미리 구성 된 ARC49 (2 µ M), 7.5 µ L 인덱스 뇌관 (2 µ M, 각 샘플에 대 한 독특한), 그리고 뜨거운 시작 x 25 µ L 2 준비 믹스.
- 각 반응에 DNA 샘플의 추가 10 µ L. 다음 조건을 사용 하 여 라이브러리를 증폭: 95 ° C 45에서 변성 15 98 ° C의 18 주기 다음 s s, 30 위한 65 ° C s, 72 ° C 30에 대 한 s, 72 ° c에서 4 °에서 2 분 대기 샘플에 대 한 최종 신장으로 끝나는 증폭 후 C.
- 4.3, 상자성 구슬의 0.8 V를 사용 하 여에 설명 된 대로 라이브러리를 정화 하 고 20 µ L TE 버퍼에 elute.
- 제조 업체에 따르면 dsDNA 정량 시 약을 사용 하 여 라이브러리를 quantitate ' s 사양 (재료의 표 참조).
- 샘플-20 ° C에서 저장 하거나 라이브러리 분석 계속.
9. 라이브러리 분석 및 풀링은
- 각 라이브러리의 품질을 결정 하 고 디지털 전기 시스템을 사용 하 여 평균 조각 크기를 추정.
참고: 평균 조각 크기는 electropherogram의 곡선 아래의 영역은 절반으로 추정 하 여 평가 무시 하는 표식에서 봉우리. 코 또는 KCl 치료 후 적당 한 라이브러리 프로필의 대표적인 결과 그림 2A에 부여 됩니다. - 계산으로 라이브러리의 농도 (nM):
(c/10 3) /(p*650)] * 10 9
어디 c ng / µ L에서 라이브러리의 농도 이며 p 혈압, 평균 조각 크기에 예상된 9.1로. - 풀 같은 어 금 니 양의 최대 24 라이브러리 시퀀싱에 대 한 다른 인덱스 뇌관으로 증폭. 25 µ L의 최종 볼륨을 10의 농도 테 버퍼를 추가 nM.
참고: 라이브러리 풀링할 수에 따라 각 도서관에서 DNA의 금액은 조정 됩니다. 뇌관 이합체 경우 단계에서 9.1 약 130의 뚜렷한 피크로 정화 단계 4.3, 상자성 구슬, 0.8 V를 사용 하 여에 설명 된 대로 반복 되 고 DNA는 eluted 25 µ L 테 buf에 때문에 bp, 라이브러리 풀의 최종 볼륨 25 µ L를 초과할 수 그다지입니다. - 결정 제조 업체에 따르면 dsDNA 정량 시 약을 사용 하 여 새 라이브러리 풀 농도
- ' s 사양 및 평균 피크 크기 위에서 설명한. 시퀀싱 및 데이터 분석 (섹션 10 및 11) 진행.
10. 시퀀싱
- 풀링된 라이브러리 12 75-자료 쌍-엔드 시퀀싱을 수행.
11. 데이터 분석
- 트림 어댑터 시퀀스를 제거 하려면 모든 읽기 품질에 대 한 필터링 및 길이 읽기.
참고:이 수행할 수 명령으로 cutadapt 1.2.1 20을 사용 하 여 ' cutadapt-f fastq-와일드 카드 일치-읽기--조용한-m 15-q 10-한 NNNNNNN < 파일 >', NNNNNNN 실제 어댑터 시퀀스로 대체 됩니다 및 < 파일 >는 fastq 파일 이름으로 바뀝니다. - 사용자 지정 스크립트를 사용 하 여 이전 단계에서 삭제 된 읽기의 동료를 제거.
- 남은 친구 1 정렬 라이브러리 준비에 사용 되는 모든 oligonucleotides의 시퀀스를 포함 하는 인덱스에 쌍 (예를 들어, Bowtie 0.12.8 21와 명령줄을 사용 하 여 옵션-m1-v2). 성공적인 정렬 모든 쌍 삭제.
- Bowtie 명령줄과 함께 사용 하 여 유기 체 참조 게놈을 나머지 쌍 정렬 옵션-v 2-X 10000-최고.
- 지도 모든 정렬 되지 않은 쌍의 친구 1을 정렬 하 여 미토 콘 드 리아 분자 시작과 끝 사이의 간격을 읽고 (Bowtie 명령줄과 함께 사용 하 여 옵션-v2).
- 모든 단일 및 결합 끝 정렬에 대 한 5´-끝의 수를 결정합니다. 분해 된 ribonucleotides가 있던 위치에 이러한 기본 상류의 위치를 이동.
- Bowtie 파일에서 데이터를 내보낼 게놈 브라우저 공통 시각화에 대 한 사용자 지정 스크립트를 사용 하 여 bedgraph 파일 형식으로 서식을. 각 가닥 당 읽기에 대 한 읽기를 정상화 백만.
- Incorp의 정체성을 결정 하는 유기 체 게놈 시퀀스를 참조 bedgraph 파일에서 위치 및 개수를 사용 하 여ribonucleotides orated.
참고: 인간 미토 콘 드리 아 게놈 16,200-300 및 5,747-5,847에 대 한 영역에서 읽기에 대 한 각 제외 되어야 이후이 지역 많은 무료 5´-끝 ribonucleotide 법인 DNA 중 합 효소 γ에 의해 무관 포함. - 분할 단일 가닥 휴식, (즉, 미토 콘 드 리아 분자 당 ribonucleotides의 수) 당 ribonucleotides의 번호를 HincII 사이트 당 읽기 평균 수와 11 HincII 사이트에서 읽기를 제외한 총 읽습니다.
결과
12를셀 위에서 설명한, 대표적인 데이터 생성 헬러에서 인간의 미토 콘 드리 아 DNA를 분석 하는 방법론을 설명 합니다. 그림 2B 요약된 읽기 모든 HincII 사이트에 보여줍니다 무거운 (HS)와 인간 mtDNA의 가벼운 물가 (LS) KCl 치료 (왼쪽된 패널) 후. 약 70% 모든 검색된 5´ 끝의 HincII 소화의 높은 효율을 보여주는 컷 사이트에 지역화 합니다. 라이브러리 포함 된 ribonucleotides에서 DNA를은 하 코 치료 약 40% (그림 2B, 오른쪽 패널)에 HincII 사이트에서 읽기 수가 감소 합니다. 이 많은 5´ 끝 ribonucleotide 관의 사이트에서 생성 되므로 충분 한 라이브러리 품질 지표입니다. 그림 2C 보여줍니다 지역화 및 5´-끝 (녹색)의 주파수 KCl 치료 및 읽기 생성 후 하이든-seq (마젠타)에 의해 코 치료 후, 무료 5´-끝과 끝에 ribonucleotides 알칼리 가수분해에 의해 생성 된. 무료 5´ 끝 및 ribonucleotides 인간 mtDNA의 HS에 지역화 왼쪽된 패널에 표시 되 고 그는 ls 지역화 오른쪽 패널에 표시 됩니다. Ribonucleotides (2D 그림, 상단 패널) 또는 HincII 사이트 (하단 패널) HS와 LS mtDNA의 원시 읽기의 상대적 숫자 표시, 각각, HS, 기준으로 LS의 14-fold 또는 31-fold 강한 범위 비슷한 편견에 대 한 관찰 하지 핵 한 DNA 이 스트랜드 바이어스 두 가닥의 기본 구성에 뚜렷한 차이 의해 설명 될 수 있습니다 고 HincII 사이트에 읽기에 정상화의 중요성을 보여 줍니다.
읽기를 정상화 미토 콘 드리 아 게놈 (그림 3A) 당 ribonucleotides의 수의 양적 측정 HincII 제공을 계산 합니다. 그림 3B에서 볼 수 있듯이 각 ribonucleotide 코 치료 각 가닥의 시퀀스 구성으로 정규화 후 읽기 표시 비율 1, 뚜렷한 ribonucleotide 제안 읽기의 비 무작위 분포를 나타내는 다른 패턴 및 높은 라이브러리 품질입니다. 그 비율은 이전 소화 효소의 분열 특이성을 확인 하는 HincII와의 영향을 받지 않습니다. 얼마나 많은 각 ribonucleotide 1000 보완 기지 ( 당 통합의 양적 측정을 생성 HincII 분열 사이트에서 그 뿐만 아니라 게놈 뉴클레오티드 내용에 포함 된 ribonucleotides의 사이트에서 읽기를 정상화 그림 3C).
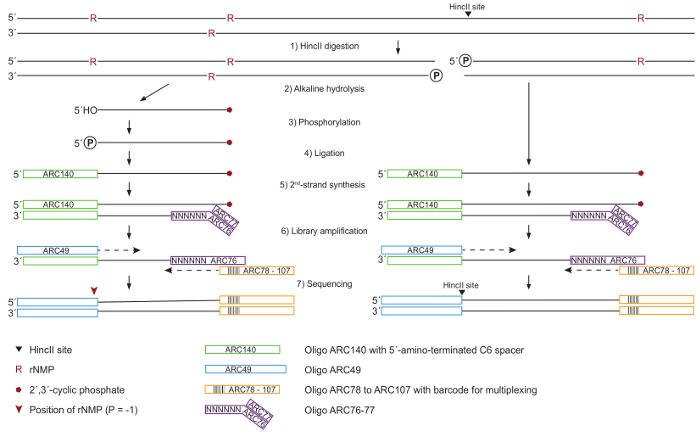
그림 1: DNA 처리 및 라이브러리 준비에 대 한 도식. (1) 전체 게놈 DNA는 죽 습 HincII에 의해 ribonucleotides의 후속 정량에 정규화에 대 한 생성 하는 HincII 사이트 (검은 화살표)에서 무뚝뚝한 끝. (2) DNA 2´, 3´ 끝 및 무료 5´ 오 끝에 3´ 순환 인산 염 (빨간 국방부)로 이어지는 코 ribonucleotide 사이트에서은 하와 함께 처리 됩니다. (3) 5´-오 끝 T4 Polynucleotide 키 니 아 제 3´-인산 가수분해 효소-빼기에 의해 phosphorylated는. (4) 모든 5´ 끝 인산 염 그룹을 들고 T4 RNA 리가 여 ARC140 oligonucleotide를 출혈 됩니다. (5) 두 번째 가닥 T7 DNA 중 합 효소를 사용 하 여 임의의 N6 시퀀스를 포함 하는 ARC76-77 oligonucleotides 합성 됩니다. (6) 도서관은 한 고 충실도 DNA 중 합 효소 ARC49 및 다중화 고유 바코드를 포함 하는 ARC107 인덱스 뇌관을 ARC78 중 하나를 사용 하 여에 의해 증폭 된다. (7) 5´-끝 쌍 간 연속 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2: 유효성 검사 메서드. (A) 대표 electropherograms의 품질을 결정 하는 자동화 된 전기 시스템을 사용 하 여 생성 생성 중 (HS)와 가벼운 물가 (LS) HincII 사이트에서 코 또는 KCl. (B) 요약 신호 처리 라이브러리 인간 mtDNA KCl (왼쪽된 패널) 또는 코 (오른쪽 패널) 치료 후. (C) Circos 무료 5´-끝 (녹색)의 하이든-Seq (무료 5´ 끝 및 ribonucleotides, 마젠타색) HS (왼쪽된 패널)과 LS (오른쪽 패널) 인간 mtDNA에서에서 그림. 봉우리는 백만 읽기 당으로 정규화 하 고 최대 피크 하이든-seq 라이브러리의 읽기의 최대 수를 조정 됩니다. (D) 요약 원시 ribonucleotides (위 패널)와 HincII (하단 패널) 무거운 (H)와 빛 (L) 가닥 인간 mtDNA (미토.) 또는 반전 (RV) 또는 핵 (Nuc.)에서 포워드 (FW) 가닥에서 읽습니다. DNA입니다. 그림 B, C 및 D는 참조12에서 적응. 오차 막대는 뜻의 표준 오류를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3: 대표 결과. (A) 상대 수 코 처리 라이브러리 무거운 (H) 또는 빛 (L) 물가에 대 한 HincII 사이트에서 읽기를 정규화 하는 ribonucleotides입니다. 코에 대 한 mtDNA 게놈에 ribonucleotide 정체성의 (B) 비 (코)를 취급 하 고 HincII 코 취급 (HincII + 코)와 무거운 (H) 또는 mtDNA의 빛 (L) 물가에 라이브러리 죽 습. (C) HincII 및 코 처리 라이브러리 무거운 (H) 또는 mtDNA의 빛 (L) 물가에 대 한 Ribonucleotide 주파수 1000 보완 기지를 정규화. 수치는 참조12에서 적응. 오차 막대는 뜻의 표준 오류를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 이름 | 시퀀스 | |||
| ARC49 | AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT | |||
| ARC76 | GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTNNNN * N * N | |||
| ARC77 | AGATCGGAAGAGCACACGTCTGAACTCCAGTC * A * C | |||
| ARC78 | CAAGCAGAAGACGGCATACGAGATCGTGATGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC84 | CAAGCAGAAGACGGCATACGAGATACATCGGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC85 | CAAGCAGAAGACGGCATACGAGATGCCTAAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC86 | CAAGCAGAAGACGGCATACGAGATTGGTCAGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT | |||
| ARC87 | CAAGCAGAAGACGGCATACGAGATCACTGTGTGACTGGAGTTCAGACGTGTGCTCTTCCGA | |||
표 1: Oligonucleotides. 하이든이 사용 oligonucleotides 같습니다 대담한 얼굴 색인을 나타냅니다. * phosphorothioate 본드를 나타냅니다. ARC140 C6 링커 함께에서 5´ 오 그룹 대신 5´ 아미노 그룹을 포함 되어 있습니다. 이 수정 결 찰 하는 동안 ARC140 concatemers의 형성을 감소 시킨다.
토론
여기 우리는 동시에 지도 하 고 시퀀스 특정 사이트 설립된 하이든-seq 프로토콜에 추가로 게놈에서 DNA 분열의 간단한 소개에 의해 gDNA, 및 mtDNA에서 ribonucleotides 특히, 계량 기법 제시. 이 연구는 인간 mtDNA에 초점을 맞추고, 하는 동안 원래 하이든 seq 메서드 Saccharomyces cerevisiae, 다른 유기 체12,16메서드의 번역 설명에서 개발 되었다.
신뢰할 수 있는 결과 위해,이 접근 방식에서 얻은 몇 가지 중요 한 단계를 주목 해야 한다: (A) 시퀀싱 어댑터가 모든 사용 가능한 5´ 끝에 선 이후, 그것은 매우 손상 DNA와 함께 작동 하도록 중요 한. DNA는 격리 되어야 합니다 라이브러리 DNA 분리 직후 선호 여야 한다 또는-20 ° c.에 DNA를 저장 될 수 있다 오랜 동안 DNA는 냉장고에서 저장 반복적으로 동결 하 고 그것을 재개 하는 권장 하지 않습니다. (B)를이 방법으로 적합 한 라이브러리를 생성, 전체 샘플 및 양적 가수분해의 균일가 열을 보장 하는 난방 블록 보다는 외피 오븐에서 DNA의 코 치료를 수행 하기 위해 결정적 이다. (C) 또한, 그것은 풀링 및 시퀀싱 하기 전에 라이브러리의 품질을 제어 하는 중요 한입니다. DNA 계량 한다 고 적절 한 조각 크기를 확인 하 고 뇌관 이합체 검사 라이브러리 DNA의 적절 한 금액을 보장 하는 자동된 전기 영동 시스템을 사용 하 여 분석.
의미 있는 데이터 분석을 위해 그것은 또한이 방법의 유익한 값은 배경 및 시퀀스 또는 가닥 편견을 평가 하기 위해 적절 한 컨트롤에 의존 하는 것이 중요. 우리는 정기적으로 때만 시퀀스 특정 endonuclease (그림 2B, 왼쪽된 패널)와 소화 KCl 샘플의 70%에서 매핑 효율성 달성. 또한, 그것은 endonuclease 치료 전체에는 영향을 미치지 않습니다 확인 하는 중요 한 HincII를 비교 하 여 법인된 ribonucleotides의 검색 치료 및 치료 샘플 (그림 3B). 이 실험에서 우리는 HincII 소개 사용 특정 사이트 컷, 다른 높은-충실도 제한 효소를 사용 될 수도 있지만.
프로토콜 적응 시킬 수 있는 종료 5´-인산 염 또는 5´ 오를 처리할 수 있는 DNA 장애의 다른 유형을 공부 하. 결과의 정확도 처리의 특이성에 의존 하며 적당 한 컨트롤 (예: 야생 타입 또는 치료) 확인에 대 한. 또한 때이 메서드를 다른 응용 프로그램 또는 다른 유기 체와 함께 사용 하기 위해 적응, 그것의 현재 설치에서 메서드 라이브러리에 처리 되는 DNA의 약 1 µ g 필요 고려해 야 하나. 포함 된 ribonucleotides의 수에 끝 수 이므로,이 따라 유기 체 또는 돌연변이, ribonucleotides의 더 낮은 숫자를 포함 하는 샘플 필요 이상의 DNA 끝에의 충분 한 번호를 입력 합니다 이후 도서관 건설입니다. 마찬가지로, DNA 샘플 ribonucleotides의 훨씬 더 높은 수 있다면, 그것은 요구할 것입니다 또한 결 찰, 두 번째 가닥 합성 및 PCR 증폭에 대 한 최적의 조건을 얻기 위해 덜 입력된 DNA를 사용 하 여. 그것은 주목할 만한 도서관 건축이이 프로토콜에서 설명 된 대로 또한 생성 된 데이터 ( 2D 그림에 표시 된) 대로 핵 게놈을 취재 하 고만 데이터 분석 mtDNA에 집중 했다. 이것은 온건 하 게 낮은 ribonucleotide 주파수로 더 큰 게놈이이 방법으로도 캡처되지는 보여줍니다.
이 메서드를 고려할 경우 특정 제한 한다 고려해:이 방법, 이론, 해야 거의 모든 유기 체에 적용 적합 한 참조 게놈 읽기의 정렬에 대 한 필요는. 또한, 우리의 프로토콜에서 얻은 결과 세포의 많은 수에서 읽기를 나타냅니다. 셀의 하위 집합의 특정 ribonucleotide 설립 패턴이 접근이 방식에 의해 확인 될 수 없습니다. Ribonucleotides ribonucleotides의 매우 낮은 수를 가진 더 큰 게놈에 매핑된, 임의의 닉스에서 ribonucleotides를 구별할 수 어려울 수 있습니다 하 고 적절 한 컨트롤 따라서 필요.
우리가 여기, 설명 방법 하이든-Seq16, Ribose Seq17, Pu Seq18또는 emRiboSeq19같은 기술을 사용할 수 비보에 확장 합니다. 이 접근의 활용 포함 된 ribonucleotides의 감도를 알칼리 또는 RNase h 2 치료, 각각, 그들의 지도의 비교를 허용 하는 ribonucleotides 게놈 넓은 식별 하 다음-세대 시퀀싱을 채용 상대 설립입니다. DNA 순서를 구체적으로 고착 하 여 알칼리 가수분해 포함된 ribonucleotides에서 뿐만 아니라, 위에서 설명한 대로 ribonucleotides에 대 한 읽기 정규화 할 수 있습니다 그 분열 사이트 뿐만 아니라 식별을 허용 하 고의 매핑 ribonucleotides, 뿐만 아니라 각 DNA 분자에 대 한 그들의 정량. DNA 복제와 관련 된 질병의 맥락에서 우리의 기술 응용 DNA 수리, 및 TLS은 분자 메커니즘 및 게놈 무결성을 일반적으로 기본에서 ribonucleotides의 역할에 대 한 깊은 이해를 제공할 수 있습니다.
공개
저자 들은 아무 경쟁 금융 관심사 선언 합니다.
감사의 말
이 연구는 스웨덴 연구 위원회에 의해 지원 되었다 (www.vr.se) 호를 부여 (2014-6466와 전략 연구 (www.stratresearch.se) 호 (ICA14-0060)에 대 한 스웨덴 재단. Chalmers 기술 대학이이 작품 MKME에 재정 지원 제공. Funders 연구 설계, 데이터 수집 및 분석, 결정 게시 또는 원고의 준비에 전혀 역할을 했다.
자료
| Name | Company | Catalog Number | Comments |
| 10x T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| 10x T4 RNA Ligase Reaction Buffer | New England Biolabs | B0216L | |
| 1x PBS | Medicago | 09-9400-100 | dissolve 1 tablet in H2O to a final volume of 1 L |
| 2-Propanol | Sigma-Aldrich | 33539-1L-GL-R | |
| 2100 Bioanalyzer | Agilent Technologies | G2940CA | |
| 50 mL Centrifuge Tube | VWR | 525-0610 | |
| Adenosine 5'-Triphosphate (ATP, 10 mM) | New England Biolabs | P0756S | dilute with EB to 2 mM |
| Agilent DNA 1000 Kit | Agilent Technologies | 5067-1504 | |
| BSA, Molecular Biology Grade (20 mg/mL) | New England Biolabs | B9000S | diltue with nuclease-free H2O to 1 mg/mL |
| Buffer EB | QIAGEN | 19086 | referred to as EB |
| CleanPCR paramagnetic beads | CleanNA | CPCR-0050 | |
| Deoxynucleotide (dNTP) Solution Mix (10 mM each) | New England Biolabs | N0447L | dilute with EB to 2 mM |
| DMEM, high glucose, GlutaMAX Supplement | Gibco | 61965026 | |
| DynaMag 96 Side | Thermo Fisher | 12331D | |
| Ethanol 99.5% analytical grade | Solveco | 1395 | dilute with milliQ water to 70% |
| Ethylenediaminetetraacetic acid solution (EDTA, 0.5 M) | Sigma-Aldrich | 03690-100ML | |
| Fetal bovine serum | Gibco | 10500056 | |
| HEPES buffer pH 8.0 (1 M) sterile BC | AppliChem | A6906,0125 | |
| Hexammine cobalt(III) chloride (CoCl3(NH3)6) | Sigma-Aldrich | H7891-5G | dissolve in nuclease-free H2O for 10 M solution, sterile filter. CAUTION: carcinogenic, sensitizing and hazardous to aquatic environment. |
| HincII | New England Biolabs | R0103S | supplied with NEBuffer 3.1 |
| Hybridiser HB-1D | Techne | FHB4DD | |
| KAPA HiFi HotStart ReadyMix (2X) | Kapa Biosystems | KK2602 | |
| Lysis buffer | 50 mM EDTA, 20 mM HEPES, NaCl 75 mM, Proteinase K (200 µg/mL), 1% SDS | ||
| Micro tube 1.5 mL | Sarstedt | 72.690.001 | |
| Microcentrifuge 5424R | Eppendorf | 5404000014 | |
| Microcentrifuge MiniStar silverline | VWR | 521-2844 | |
| Multiply µStripPro 0.2 mL tube | Sarstedt | 72.991.992 | |
| Nuclease-free water | Ambion | AM9937 | |
| Phenol – chloroform – isoamyl alcohol (25:24:1) | Sigma-Aldrich | 77617-500ML | |
| Potassium chloride (KCl) | VWR | 26764.232 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Potassium hydroxide (KOH) | VWR | 26668.296 | dissolve in nuclease-free H2O for 3 M solution, sterile filter |
| Proteinase K | Ambion | AM2546 | |
| Qubit 3.0 Fluorometer | Invitrogen | Q33216 | |
| Qubit Assay Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA BR Assay Kit | Invitrogen | Q32850 | CAUTION: Contains flammable and toxic components |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | CAUTION: Contains flammable and toxic components |
| Refrigerated Centrifuge 4K15 | Sigma Laboratory Centrifuges | No. 10740 | |
| SDS Solution, 10% | Invitrogen | 15553-035 | |
| Sodium acetate buffer solution, pH 5.2, 3 M (NaAc) | Sigma-Aldrich | S7899 | |
| Sodium chloride (NaCl) | VWR | 27810.295 | dissolve in nuclease-free H2O for 5 M solution, sterile filter |
| T100 Thermal Cycler | Bio-Rad | 1861096 | |
| T4 Polynucleotide Kinase (3' phosphatase minus) | New England Biolabs | M0236L | |
| T4 RNA Ligase 1 (ssRNA Ligase) | New England Biolabs | M0204L | supplied with PEG 8000 (50%) |
| T7 DNA Polymerase (unmodified) | New England Biolabs | M0274S | supplied with 10x T7 DNA Polymerase Reaction Buffer |
| TE Buffer | Invitrogen | 12090015 | |
| ThermoMixer F2.0 | Eppendorf | 5387000013 |
참고문헌
- Traut, T. W. Physiological Concentrations of Purines and Pyrimidines. Mol. Cell. Biochem. 140, 1-22 (1994).
- McElhinny, S. A. N., et al. Abundant ribonucleotide incorporation into DNA by yeast replicative polymerases. Proc. Natl. Acad. Sci. USA. 107, 4949-4954 (2010).
- Williams, J. S., Lujan, S. A., Kunkel, T. A. Processing ribonucleotides incorporated during eukaryotic DNA replication. Nat. Rev. Mol. Cell Biol. 17, 350-363 (2016).
- Clausen, A. R., Zhang, S., Burgers, P. M., Lee, M. Y., Kunkel, T. A. Ribonucleotide incorporation, proofreading and bypass by human DNA polymerase delta. DNA Repair. 12, 121-127 (2013).
- Potenski, C. J., Klein, H. L. How the misincorporation of ribonucleotides into genomic DNA can be both harmful and helpful to cells. Nucleic Acids Res. 42, 10226 (2014).
- Vengrova, S., Dalgaard, J. Z. RNase-sensitive DNA modification(s) initiates S. pombe mating-type switching. Gene. Dev. 18, 794-804 (2004).
- Lujan, S. A., Williams, J. S., Clausen, A. R., Clark, A. B., Kunkel, T. A. Ribonucleotides Are Signals for Mismatch Repair of Leading-Strand Replication Errors. Mol. Cell. 50, 437-443 (2013).
- Ghodgaonkar, M. M., et al. Ribonucleotides Misincorporated into DNA Act as Strand-Discrimination Signals in Eukaryotic Mismatch Repair. Mol. Cell. 50, 323-332 (2013).
- DeRose, E. F., Perera, L., Murray, M. S., Kunkel, T. A., London, R. E. Solution Structure of the Dickerson DNA Dodecamer Containing a Single Ribonucleotide. Biochemistry. 51, 2407-2416 (2012).
- Li, Y. F., Breaker, R. R. Kinetics of RNA degradation by specific base catalysis of transesterification involving the 2 '-hydroxyl group. J. Am. Chem. Soc. 121, 5364-5372 (1999).
- McElhinny, S. A. N., et al. Genome instability due to ribonucleotide incorporation into DNA. Nat. Chem. Biol. 6, 774-781 (2010).
- Berglund, A. K., et al. Nucleotide pools dictate the identity and frequency of ribonucleotide incorporation in mitochondrial DNA. Plos Genet. 13, (2017).
- Brown, J. A., Suo, Z. C. Unlocking the Sugar "Steric Gate" of DNA Polymerases. Biochemistry. 50, 1135-1142 (2011).
- Sparks, J. L., et al. RNase H2-Initiated Ribonucleotide Excision Repair. Mol. Cell. 47, 980-986 (2012).
- Miyabe, I., Kunkel, T. A., Carr, A. M. The Major Roles of DNA Polymerases Epsilon and Delta at the Eukaryotic Replication Fork Are Evolutionarily Conserved. Plos Genet. 7, (2011).
- Clausen, A. R., et al. Tracking replication enzymology in vivo by genome-wide mapping of ribonucleotide incorporation. Nat. Struct. Mol. Biol. 22, 185-191 (2015).
- Koh, K. D., Balachander, S., Hesselberth, J. R., Storici, F. Ribose-seq: global mapping of ribonucleotides embedded in genomic DNA. Nat. Methods. 12, 251 (2015).
- Keszthelyi, A., Daigaku, Y., Ptasinska, K., Miyabe, I., Carr, A. M. Mapping ribonucleotides in genomic DNA and exploring replication dynamics by polymerase usage sequencing (Pu-seq). Nat. Protoc. 10, 1786-1801 (2015).
- Ding, J., Taylor, M. S., Jackson, A. P., Reijns, M. A. M. Genome-wide mapping of embedded ribonucleotides and other noncanonical nucleotides using emRiboSeq and EndoSeq. Nat. Protoc. 10, 1433-1444 (2015).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17, 10-12 (2011).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10, (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유