JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
유사 분열 동안 염색체 분리의 라이브 셀 이미징
요약
이 프로토콜 레이블 및 라이브 염색체 mitotic 세포 Histone2B GFP BacMam 2.0 레이블 및 회전 디스크 confocal 현미경 시스템을 사용 하 여 시각화 하는 간단 하 고 편리한 방법을 설명 합니다.
초록
염색체 해야 안정적이 고 균일 하 게 분리 되어 딸 세포에 mitotic 세포 분열 동안. 염색체 분리의 충실도 스핀 들 어셈블리 검사점 (SAC)을 포함 하는 여러 메커니즘을 통해 제어 됩니다. SAC 모든 염색체 kinetochores microtubules 스핀 들에 첨부 하지 않으면 세포 유사 분열 진행의 예방에 대 한 책임은 복잡 한 피드백 시스템의 일부입니다. 염색체 지체 및 비정상적인 염색체 분리 역 기능 세포 주기 통제 검문소의 표시기 이며 셀 분할의 게놈 안정성을 측정 하는 데 사용할 수 있습니다. SAC의 규제 완화는 염색체 분리 중 오류의 축적을 통해 악성 세포로 정상 세포의 변화에 발생할 수 있습니다. SAC의 구현 및 복잡 한 kinetochore의 형성 단단히 kinases와 단백질 인산 가수분해 효소 2A 등 인산 가수분해 효소 사이 상호 작용에 의해 규제 됩니다 (PP2A). 이 프로토콜 마우스 미 발달 섬유 아 세포 했다 PP2A B56γ 규제 소 단위의 녹아웃 쥐에서 고립에서 염색체를 후행의 라이브 셀 이미징을 설명 합니다. 이 방법은 다른 세포 주기 제어 이미징 기술 cytometry 또는 immunocytochemistry 같은 염색체의 동적 spatiotemporal 시각화 대신 셀 cytokinesis 상태 스냅숏을 제공의 단점을 극복 동안 유사 분열.
서문
다음 프로토콜에서 우리 염색체 분리 및 마우스 미 발달 섬유 아 세포 히스톤 2B-GFP, BacMam 2.0 라벨 및 라이브 셀 이미징 사용 하 여에서 세포 주기 동안 mitotic 진행을 시각화 하는 편리한 방법을 설명 합니다.
세포 주기 통제 검문소 염색체 분리를 모니터링 하 고 셀 1,2,3의 유전 무결성의 유지 관리에 중요 한 역할. 미 분리 된 염색체의 이수성, 가장 단단한 종양 4의 특징은 발생할 수 있습니다. 따라서, 후행 염색체의 염색체 불안정성을 공부 하는 방법으로 사용할 수 있습니다.
붙일 표시 또는 라이브 염색체 분리를 시각화 하는 데 사용 하 고 mCherry 태그의 생성 하지만 후행 염색체 단백질 수 태그 H2B GFP 단백질 유전자 전달 및 분자 생물학 5의 상당한 지식이 필요 합니다. 여기는 CL-HB는 리 젠 트, 편의 위해이 전화 CellLight Histone2B GFP BacMam 2.0 시 약의 사용에 설명 합니다. 이 시는 즉시 사용할 수 있습니다 하 고 따라서 벡터 품질 및 무결성에 대 한 우려를 제거 합니다. 또한,이 시는 잠재적으로 유해한 치료 또는 지질 및 염료 선적 화학 물질의 사용을 필요 하지 않습니다. 기존의 형광 라벨, 달리 CL-HB 리 젠 트 기능에 독립적으로 얼룩 (즉., 막 잠재력). CL HB 리 젠 트를 단순히 셀에 추가 하 고 단백질 식 밤새 껏 알을 품 수 수 있습니다. CL HB 섭정 포유류 세포에서 복제 하지 않습니다 그리고 biosafety 수준 (BSL) 1 실험실 설정에서 사용할 수 있습니다. 또한, 가장 역동적인 세포 분석을 수행 하 후 최대 5 일까지 충분 한 시간 동안 야간 보육이 과도 transfection은 감지할 수 있습니다.
또는, 염색체 이상이 cytometry, immunohistochemistry 또는 형광 제자리 교 잡 (물고기) 6등 다양 한 기법에 의해 공부 될 수 있었다. Cytometry는 이수성, 측정 될 수 있는 DNA 콘텐츠 및 세포 주기 세포의 단계에 따라 공부를 사용할 수 있습니다. Cytometry 이수성을 측정 하는 데 사용할 수 있습니다, 하지만 그것은 염색체 미 분리에 정보를 제공 하지 않습니다. 물고기와 immunohistochemistry 기술 DNA 또는 염색체에 바인딩할 형광 프로브를 사용 합니다. 이러한 기술은 세포의 인구 상태 스냅숏을 제공, 그들은 라이브 셀 이미징 함으로써 특정 셀 시간의 기간 동안 다음에 cytokinesis spatiotemporal 시각화를 통해 얻은 정보를 누락을 허용 하지 않습니다.
이 프로토콜은 지체 염색체 또는 염색체 미 분리 취급 하는 nocodazole 마우스 미 발달 섬유 아 세포 (MEFs)에 PP2A-B56γ-마우스에서 분리 연구 하 사용 되었다. 응용 프로그램 위에 이외에,이 프로토콜 레이블 및 세포 주기 규칙을 공부 하는 데 사용할 수 있는 다양 한 셀 형식에 염색체 분리 또는 종양 세포의 염색체 불안정을 시각화 하는 간단한 도구를 제공 합니다. 또한, 그것은 또한 사용할 수 있습니다 다양 한 약물 치료에 의해 발생 하는 염색체 불안정을 공부 하거나 유전자 염색체 미 분리의 결과로 밖으로 노크의 효과 연구.
프로토콜
이러한 연구에서 실시 한 모든 실험 프로토콜 식품 및 의약품 안전 청 (FDA) 연구 시설에서 기관 동물 관리 및 사용 위원회에 의해 승인에 따라 수행 했다.
1. 격리와 마우스 미 발달 섬유 아 세포 (MEFs)의 문화
- 표준 프로토콜 7,,89마우스 미 발달 섬유 아 세포 (MEFs) PP2A-B56γ-마우스 긴장과 야생 타입 littermates에서를 격리 합니다.
- ExpandMEFs 3 구절에 대 한 동결 하 고 실험 8필요할 때까지 저장 합니다.
2. 2-잘 연 발된 커버 유리에 대 한 자란 Mefs 라이브 영상
- MEF 성장 미디어 Dulbecco 수정이 글 중간 (DMEM/F12)를 포함 하는 10%의 500 mL를 준비 태아 둔감 한 혈 청 (FBS), 1 X 페니실린/스 항생제, L-글루타민 (200 m m) 및 비 본질적인 아미노산 (NEAA) 500 mL 미디어 병에서 X 1.
- 37 ° c.에 미리 데워 진된 물 욕조에 통로 3에서 야생 타입에서 냉동된 MEFs와 PP2A-B56γ-생쥐의 병 동
- 전송 15 mL 튜브에 MEFs를 해 동 하 고 천천히 10 mL 피 펫을 사용 하 여 15 mL 튜브에 DMEM/F12 미디어의 dropwise 20 mL를 추가 합니다.
- T75 플라스 크에서 DMEM/F12 성장 매체의 20 mL와 함께 MEFs를 전송 하 고 셀 70% 합칠 때까지 그들을 확장 합니다. Confluency 4 x 또는 10 x 배율에서 역 현미경을 사용 하 여 추정 된다.
- Aspirate 유리 방목 지를 사용 하 여 성장 매체 후드에 진공 시스템에 부착 된 플라스틱, 0.25 %trypsin / EDTA의 3 mL를 추가 하 고 반응을 중지 DMEM/F12 성장 매체의 5 분 추가 3 ml 37 ° C에서 품 어.
- 낮은 속도 (300 x g) 실 온에서 5 분 동안에 세포 원심 조심 스럽게는 상쾌한을 제거 하 고 다시 물 욕조에 37 ° C에 미리 데워 DMEM/F12 성장 매체의 1 mL에 셀 펠 릿을 중단.
- Trypan 블루 제외 방법 또는 다른 적절 한 셀 계산 방법 8을 사용 하 여 셀을 열거 합니다.
- 2-잘 연 발된 커버 유리에 MEFs/잘 씨 약 20000. 200 µ L/잘 DMEM/F12 성장 매체를 추가 하 고 37 ° C, 5% CO2에서 그들을 하룻밤 배양 하 여 연결할 셀을 허용.
3입니다. 동기화
- DMEM/F12 성장 미디어 0.1%를 포함 하는 세포를 배양 하 여 G0/G1 단계에서 MEFs를 동기화 G0/G1 단계에서 최대 셀 수를 24 h, FBS.
참고: 지금까지 혈 청 기아는 기본 설정의 방법으로 사용 되었다, 비록에 다양 한 다른 방법 mitotic 무대 체포 10을 셀 필요에 따라 사용할 수 있습니다.
4. 라벨
- 1.5 m g/mL DMSO에 nocodazole의 재고 솔루션을 준비 합니다. Nocodazole는 microtubule 형성 10을 억제 하 여 G2/M 단계에서 셀을 체포.
- 3 일 게시물 동기화, 추가 200 µ L (10 %FCS)와 성장 매체의, 200 ng/mL nocodazole와 CL-HB 리 젠 트 (히스톤 2B GFP BacMam 2.0).
- CL HB 리 젠 트 입자 당 셀 (PPC) 셀에 다음과 같이 추가를 계산 합니다.
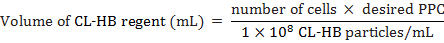
셀 수 라벨의 시간에서 셀의 예상된 총 수는, PPC는, 세포 당 입자의 수 그리고 1 × 108 시의 mL 당 입자의 수입니다. 예를 들어 30의 PPC와 레이블 20000 셀에
참고: CL-HB 섭정은 대부분 세포 유형 10, 50 PPC 사이 잘 작동합니다. 그러나, 30 PPC이이 연구를 위해 잘 일했다. - 37 ° C, 5% CO218 h에 대 한 MEFs를 품 어.
참고: 현재 실험에 대 한 스핀 들 어셈블리 검사점 탈출 mitotic 세포에서 염색체의 시각화 발생 18 h nocodazole와 세포 주기 검거 후.
5입니다. 염색체 후행의 시각화
- 마우스 미 발달 섬유 아 세포 (MEFs)는 환경 챔버와 기름 침수, 63 x 렌즈 회전 디스크 confocal 현미경 시스템을 사용 하 여 시각화.
- 488 nm 및 방출 파장 450 nm의 여기 파장을 사용 하 여 GFP 채널 이미지 수집에 대 한.
- 회전 디스크 confocal 현미경 시스템을 사용 하 여 자동화 된 스캐닝 단계 기능, Z 피 삽입, 무대 상단 부 화 및 사진이이 기술에 대 한 표백 후 직접 형광 복구.
- 이미징, 전에 1 일 환경 챔버 전원 켜기 하 고 37 ° C에서 전체 챔버를 밤새 따뜻하게.
- 현미경 스탠드, 카메라, 회전 디스크 장치, 조명 기, 아르곤 레이저, 컴퓨터와 자동화 된 단계에 대 한 전원 ON을 설정 합니다.
- 시스템을 워밍업 3 분; 점화 키를 켜면 아르곤 레이저를 시작 합니다. "레이저 실행" "대기 모드"에서 아르곤 레이저에 대 한 토글 스위치를 전환 합니다.
- 데이터 수집 및 처리 소프트웨어를 시작 합니다.
- 무대 최고의 보육에 대 한 CO2 컨트롤러를 시작 하 고 5% CO2 의 농도 설정. 이 영상의 시작 하기 전에 수행 되어야 합니다.
- 시각화를 위한 무대에 인큐베이터와 장소에서 연 발된 커버 유리를 제거 합니다. 시각화는 기름 침수 63 x 렌즈 (NA1.4)를 통해 셀.
- 눈 렌즈를 통해 서 봅니다, 그리고 이미지 초점 고 핵 붕괴 (NEBD) 단계에서 셀을 식별 합니다.
- 적절 한 레이저 (Histone2B-GFP를 시각화 하기 위해 488 nm 아르곤 레이저)를 시작 합니다.
- 수집 제어 창을 열고 GFP 채널에 대 한 노출 시간을 설정 합니다. NEBD에 있는 셀을 식별 합니다.
- 수동으로 대상 셀의 위쪽 및 아래쪽 초점면, 결정 하 고 설정 단면 광학 xyz를 입력 합니다.
- 20 분;에 대 한 관찰 세포는 세포 분열을 통해 진행 되지 않습니다, 만약 20 분 후 이미지 수집을 중지 하 고 NEBD에 있는 다음 셀으로 이동.
- 셀 당 약 1 h는 SAC에서 탈출 하는 PP2A-B56γ-셀에 유사 분열 동안 이미지 지체 염색체에 필요 합니다. 영화에 대 한 데이터를 얻기 위해 모든 3 분 사진을 찍을.
참고: 야생 타입 셀 체포 하 고 NEBD nocodazole로 치료 하면 과거 진행 하지 않습니다. - 추가 분석을 위해 zvi 파일 형태로 이미지를 저장 합니다.
6. 이미지 처리 및 분석
참고: 3 차원 영상 처리 및 Axiovision 버전 4.8.2 또는 Imaris 버전 8.2와 같은 사용 가능한 소프트웨어를 사용 하 여 분석을 수행 합니다. 이 연구는 ImageJ 소프트웨어 사용 되었습니다.
- 이미지 시퀀스를 엽니다. 이미지는 이미 스택 형식에서, 경우 다음 단계를 진행 합니다. 그렇지 않으면, 모든 관련 이미지를 결합 하 여 사용 하 여 스택 ' 이미지 > 스택 > 스택 이미지 ' 메뉴 막대에서.
- 밝기/명암 대비와 레벨에 필요한 어떤 조정 든 지를 수행 합니다.
- 영화 타임 스탬프를 추가:
- 로 이동 ' 이미지 > 스택 > 레이블...' 메뉴 막대에서.
- 적절 한 형식, 시작 시간 값 및 각 이미지 사이의 시간 간격을 선택 합니다.
- '미리 보기' 확인란을 선택 하 고 위치 및 형식 설정을 조정 합니다. 타임 스탬프를 적용 하려면 ' 확인'을 누릅니다.
- 영화에 눈금 막대를 추가할:
- 이미지에서 스케일링을 설정 ' 분석 > 비율 설정 ' 메뉴 막대에서.
- '픽셀에서 거리' 분야에서 거리는 알려져 픽셀 수를 입력 하 고 '알려진 거리' 분야에서 거리를 입력 합니다. µ M. 스택에 눈금을 적용 하려면 ' 확인'을 누르십시오 예를 들어, 거리에 대 한 길이 올바른 단위를 설정 합니다.
- 로 이동 ' 분석 > 도구 > 배율... ' 눈금 막대를 추가 하려면. µ M에 '폭'로 바의 크기를 설정 하 고 나머지 적절 한 옵션을 서식 조정 합니다. 눈금 막대에 전체 스택을 추가 하려면 '모든 조각을 라벨' 상자를 확인 하십시오.
- 이미지 창의 가장자리를 왼쪽 하단에 삼각형 플레이 버튼을 클릭 하 여 동영상을 미리 보기. 사용 하 여 프레임 속도 조정 ' 이미지 > 스택 > 애니메이션 > 애니메이션 옵션 ' 메뉴 막대에서.
- 선택 하 여 동영상 파일을 내보내기 ' 파일 > 다른 이름으로 저장 > AVI...' 하 고 프레임 속도 및 압축을 선택 합니다.
- '확인' 선택 저장을 누릅니다 위치와 파일 이름. 동영상 파일을 저장 하려면 ' 저장' 키를 누릅니다.
결과
야생 유형 및 PP2A-B56γ-마우스에서 MEFs 2-잘 챔버 커버 유리에 시드 되었고 첨부할 수 있습니다. 하루에 2, MEFs는 0.1%를 사용 하 여 동기화 했다 FBS 24 h에 대 한. 하루 3, MEFs 미디어에서 200 ng/mL에 nocodazole 및 30 PPC CL-HB regentwere 37에서 18 h incubated° C, 5 %CO2. 하루에 4, 셀은 회전 디스크 confocal 현미경 시스템 (그림 1)를 사용 하 여 몇 군데 있었?...
토론
정확한 염색체 분리를 보장 하는 세포 주기 통제 검문소 방지 이수성 및 셀 변환 1,2,3. 현재 연구에서 우리는 PP2A B56γ의 그 비활성화 발견 약화 스핀 들 어셈블리 검사점 귀착되 었 다. 라이브 셀 이미징을 사용 하 여 nocodazole 9PP2A B56γ MEFs 유사 분열 동안 염색체 미 분리 처리를 관찰할 수 있었습니다.
공개
저자 들은 아무 경쟁 금융 관심사 선언 합니다.
감사의 말
우리는 박사 Guo Chiuan 웅와 박사 Bharatkumar 조시 원고를 개선 하는 소중한 의견에 감사 하 고 싶습니다.
자료
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Gibco | 11320082 | |
| L-Glutamine | Gibco | 25030081 | |
| MEM Non-Essential amino acids solution (100X) | Gibco | 11140050 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Dulbecco’s Phosphate Buffered saline | Gibco | 14190144 | |
| Trypsin-EDTA | Gibco | 25300054 | |
| Fetal bovine serum | Atlanta Biologicals | S11150 | |
| DMSO | EMD Millipore | MX 1458-6 | |
| Cryogenic vial storage boxes | Fisherbrand | 10-500-28 | |
| Cryogenic vials | Corning/costar | 431416 | |
| T75 flasks | Cellstar | 658175 | |
| 2 well chambered cover glass | Nunc | 155380PK | |
| Cellometer Vision Cell Profiler | Nexcelom Bioscience LLC | Cellometer Vision Trio | |
| Nocodazole | Sigma | M1404 | |
| CellLight Histone 2B-GFP, BacMam 2.0 | Thermo Fisher Scientific Inc. | C10594 | |
| Zeiss Cell Observer Spinning Disk Confocal Microscope system | Carl Zeiss Microscopy | Zeiss Cell Observer SD | |
| Water Bath | Thermoscientific | 280 series | |
| Incubator | Sanyo commercial solutions | MCO-18AIC (UV) | |
| Class II Biological safety cabinet | The Baker Company | SterilGard | |
| Axiovision software | Zeiss | Ver.4.8.2 | |
| ImageJ software | National Institute of Health | Ver. 1.51r |
참고문헌
- Funk, L. C., Zasadil, L. M., Weaver, B. A. Living in CIN: Mitotic Infidelity and Its Consequences for Tumor Promotion and Suppression. Dev Cell. 39, 638-652 (2016).
- Etemad, B., Kops, G. J. Attachment issues: kinetochore transformations and spindle checkpoint silencing. Curr Opin Cell Biol. 39, 101-108 (2016).
- Lara-Gonzalez, P., Westhorpe, F. G., Taylor, S. S. The spindle assembly checkpoint. Curr Biol. 22, 966-980 (2012).
- Jallepalli, P. V., Lengauer, C. Chromosome segregation and cancer: cutting through the mystery. Nat Rev Cancer. (2), 109-117 (2001).
- Zhu, L., et al. Mitotic protein CSPP1 interacts with CENP-H protein to coordinate accurate chromosome oscillation in mitosis. J Biol Chem. 290 (45), 27053-27066 (2015).
- Pikor, L., Thu, K., Vucic, E., Lam, W. The detection and implication of genome instability in cancer. Cancer Metastasis Rev. 32 (3-4), 341-352 (2013).
- Varadkar, P., Despres, D., Kraman, M., Lozier, J., Phadke, A., Nagaraju, K., McCright, B. The protein phosphatase 2A B56γ regulatory subunit is required for heart development. Dev Dyn. 243 (6), 778-790 (2014).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of Mouse Embryonic Fibroblast Cells Suitable for Culturing Human Embryonic and Induced Pluripotent Stem Cells. J. Vis. Exp. (64), e3854 (2012).
- Varadkar, P., Abbasi, F., Takeda, K., Dyson, J. J., McCright, B. PP2A-B56γ is required for an efficient spindle assembly checkpoint. Cell Cycle. 18 (12), 1210-1219 (2017).
- Rosner, M., Schipany, K., Hengstschläger, M. Merging high-quality biochemical fractionation with a refined flow cytometry approach to monitor nucleocytoplasmic protein expression throughout the unperturbed mammalian cell cycle. Nat Protoc. 8 (3), 602-626 (2013).
- Jonkman, J., Brown, C. M. Any Way You Slice It-A Comparison of Confocal Microscopy Techniques. J Biomol Tech. 26 (2), 54-65 (2015).
- Versaevel, M., Braquenier, J. B., Riaz, M., Grevesse, T., Lantoine, J., Gabriele, S. Super-resolution microscopy reveals LINC complex recruitment at nuclear indentation sites. Sci Rep. 8 (4), 7362 (2014).
- Wild, T., Larsen, M. S., Narita, T., Schou, J., Nilsson, J., Choudhary, C. The Spindle Assembly Checkpoint Is Not Essential for Viability of Human Cells with Genetically Lowered APC/C Activity. Cell Rep. 1 (8), 1829-1840 (2016).
- Iuso, D., et al. Exogenous Expression of Human Protamine 1 (hPrm1) Remodels Fibroblast Nuclei into Spermatid-like Structures. Cell Rep. 1 (9), 1765-1771 (2015).
- Ratcliffe, E., Glen, K. E., Naing, M. W., Williams, D. J. Current status and perspectives on stem cell-based therapies undergoing clinical trials for regenerative medicine: case studies. Br Med Bull. 108, 73-94 (2013).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유