JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
마찬가지로 효과적인 이진 분류 공연으로 여러 바이오 마커 하위 집합 선택
요약
기존 알고리즘 바이오 마커 검출 데이터 집합에 대 한 하나의 솔루션을 생성합니다. 이 프로토콜의 여러 마찬가지로 효과적인 솔루션의 존재 및 고 생물 의학 연구원 제안된 과제에 대 한 데이터 세트를 조사 수 있도록 사용자 친화적인 소프트웨어를 제공. 컴퓨터 과학자 들은 팬 들은이 기능에 그들의 바이오 마커 검출 알고리즘의 정보를 제공할 수도 있습니다.
초록
바이오 마커 검출 높은 처리량 'omics' 연구자에 대 한 생물 더 중요 한 질문 중 하나 이며 특정된 데이터 집합에 대 한 최적화 된 성능 측정 한 biomarker 하위 집합을 생성 하는 거의 모든 기존 바이오 마커 검출 알고리즘 . 그러나, 최근 연구도 동일 또는 유사 하 게 효과적 분류 공연 여러 바이오 마커 하위 집합의 존재 증명. 이 프로토콜은 이진 분류 공연, 사용자 정의 자르기 보다 더 하위 집합 바이오 마커 검출을 위한 간단 하 고 간단한 방법론을 선물 한다. 데이터 준비 및 로드, 초기 정보 요약, 매개 변수 조정, 바이오 마커 검사, 결과 시각화 및 해석, 바이오 마커 유전자 주석, 및에 결과 및 시각화 수출의 프로토콜 구성 출판 품질입니다. 제안 된 바이오 마커 전략 스크리닝 직관적 이며 바이오 마커 검출 알고리즘 개발에 대 한 일반적인 규칙을 보여 줍니다. 사용자 친화적인 그래픽 사용자 인터페이스 (GUI) 프로그래밍 언어 파이썬, 생물 의학 연구원은 그들의 결과에 직접 액세스할 수 있도록 사용 하 여 개발 되었다. 소스 코드와 kSolutionVis의 설명서는 http://www.healthinformaticslab.org/supp/resources.php에서 다운로드할 수 있습니다.
서문
하나는 가장 일반적으로 이진 분류 조사 하 고 도전적인 데이터 마이닝, 생물 의학 영역에서 문제 샘플 가장 정확한 차별 전원1, 의 두 그룹에 대 한 훈련 분류 모델을 구축 하는 2 , 3 , 4 , 5 , 6 , 그러나 7., 생물 의학 분야에서 생성 하는 큰 데이터는 고유의 "큰 p 작은 n" 패러다임, 기능의 일반적으로 샘플6,,89의 수 보다 훨씬 더 큰 숫자와 함께. 따라서, 생물 의학 연구원 overfitting 문제8,9를 피하기 위해 분류 알고리즘을 활용 하기 전에 기능 치수를 줄일 필요가 있다. 진단 바이오 마커는 건강 한 컨트롤 샘플10,11에서 특정된 질환의 환자를 분리 검색 기능의 하위 집합으로 정의 됩니다. 환자는 일반적으로 긍정적인 샘플으로 정의 되며 건강 한 컨트롤 부정적인 샘플12로 정의 됩니다.
최근 연구는 생물 의학 데이터 집합5에 대 한 동일 또는 유사 하 게 효과적인 분류 공연 하나 이상의 솔루션 존재 제안 했다. 거의 모든 기능 선택 알고리즘은 동일한 데이터 집합에 대 한 하나의 솔루션을 생산 하는 결정적 알고리즘입니다. 유전자 알고리즘을 동시에 비슷한 공연, 여러 솔루션을 생성할 수 있습니다 하지만 그들은 여전히 주어진된 데이터 집합13,14에 대 한 출력으로 최고의 피트 니스 기능을 하나의 솔루션을 선택 하려고 합니다.
기능 선택 알고리즘 필터 또는 래퍼12대략 그룹화 될 수 있습니다. 상위-k 특징 이진 클래스 레이블 기능 가정에 따라 그들의 중요 한 개인 협회에 의해 선정은 서로15,,1617의 독립적인 필터 알고리즘 선택 . 이 가정은 거의 모든 실제 데이터 집합에 대 한 진정한 보유 하지 않습니다, 비록 추론 필터 규칙에서 잘 수행 많은 경우, 예를 들어, mRMR (최소 중복 및 최대 관련성) 알고리즘, Wilcoxon 테스트 기반 기능 필터링 (WRank) 알고리즘, 및 ROC (수신기 작동 특성) 플롯 (ROCRank)를 필터링 알고리즘을 기반 으로합니다. mRMR, 그것은 훨씬 더 작은 문제, 각각의 두 개의 변수를 포함, 최대 종속성 기능 선택 알고리즘 비교의 시리즈와 조합 추정 문제에 근접 하기 때문에 효율적인 필터 알고리즘은 고 따라서 더 강력한18,19는 없음을 공동 확률을 사용 합니다. 그러나, mRMR 수로 관련성, 증가 시킬 수 있으며 따라서 개별적으로 쓸모 있지만 결합 하는 경우에 유용 일부 기능 조합을 그리 워 기능 간의 상호 작용을 측정 하지 않습니다 일부 기능의 유용성 과소 평가. WRank 알고리즘을 어떻게 구별 기능 샘플, 두 클래스 사이의 이며 outliers20,21의 견고성에 대 한 알려져 있다 비패라메트릭 점수를 계산 합니다. 또한, ROCRank 알고리즘은 얼마나 중요 한 특정 기능 영역 ROC에서 곡선 (AUC)은 조사 이진 분류 성능22,23평가 합니다.
다른 한편으로, 래퍼 기능 하위 집합, 반복적으로 추론 규칙에 의해 생성 된 미리 정의 된 분류자의 성능을 평가 하 고 최고의 성능을 측정24기능 하위 집합을 만듭니다. 래퍼는 일반적으로 분류 성능에서 필터를 능가 하지만 느린25을 실행 됩니다. 임의 숲 정례화 (RRF)26,27 알고리즘 각 무작위 포리스트 노드, 누구의 기능 중요성 점수 Gini 색인에 의해 평가에서 학습 데이터의 하위 집합에서 기능을 평가 하 여 욕심이 규칙을 사용 하는 예를 들어 . 새로운 기능에 대 한 선택의 정보 이득 선택 기능을 개선 되지 않으면 처벌 될 것 이다. Microarrays (PAM)28,29 알고리즘 또한 래퍼 알고리즘에 대 한 예측 분석 클래스 레이블의 각 한 중심을 계산 하 고 전체 향해 진 중심을 축소 하는 기능을 선택 하는 또한, 클래스 중심입니다. 팸은 외곽 특징에 대 한 강력한입니다.
상위 분류 성능 가진 여러 솔루션은 어떤 주어진 데이터 집합 필요할 수 있습니다. 첫째, 결정적 알고리즘의 최적화 목표는 반드시 생물 학적 샘플에 적합 하지 않습니다는 수학 공식, 예를 들어, 최소 오류 속도30에 의해 정의 됩니다. 둘째, dataset 여러 비슷한 또는 심지어 동일 공연으로 크게 다른 솔루션을 할 수 있습니다. 거의 모든 기존 기능 선택 알고리즘 무작위로 출력31이러한 솔루션 중 하나를 선택 합니다.
이 연구는 어떤 주어진된 이진 분류 데이터 집합에 대 한 비슷한 공연을 여러 기능 선택 솔루션을 생성 하기 위한 정보 분석 프로토콜을 소개 합니다. 가장 생물 의학 연구원 informatic 기술 또는 컴퓨터 코딩에 익숙하지 않은 고려 하면, 사용 하기 쉬운 그래픽 사용자 인터페이스 (GUI) 생물 의학 이진 분류 데이터의 신속한 분석을 촉진 하기 위하여 개발 되었다. 데이터 로드 및 요약, 매개 변수 튜닝, 파이프라인 실행 및 결과 해석 분석 프로토콜에 의하여 이루어져 있다. 간단한 클릭으로, 연구원은 바이오 마커 하위 집합 및 게시-품질 시각화 플롯을 생성할 수 있게. 프로토콜은 두 개의 이진 분류 데이터 집합의 급성 림프 구성 백혈병 (ALL), 즉, ALL1 및 ALL212의 transcriptomes를 사용 하 여 테스트 되었습니다. ALL1 및 ALL2 데이터 집합 광범위 한 연구소 게놈 데이터 분석 센터에서 http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi에서 다운로드 했다. ALL1에는 12,625 기능 128 샘플을 포함 되어 있습니다. 이 샘플의 95 B-세포는 모두 고 33는 T-세포 모든. ALL2 100 샘플 12,625 기능을 포함합니다. 이 샘플의 있습니다 65 환자는 재발을 고통을 하지 않았다 35 환자. ALL1 4 개의 필터와 4 개의 래퍼 96.7%, 및 10012달성 8 기능 선택 알고리즘의 6의 최소 정확도 쉽게 이진 분류 데이터 집합 했다. ALL2 위의 8 기능 선택 알고리즘 83.7% 정확도12보다 더 나은 달성 더 어려운 데이터 집합 동안. 이 최고의 정확도 56 기능 상관 관계 기반 기능 선택 (CFS) 래퍼 알고리즘에 의해 감지와 함께 달성 했다.
프로토콜
참고: 다음 프로토콜 정보 분석 절차의 세부 사항 및 주요 모듈의 의사 코드를 설명합니다. 자동 분석 시스템은 파이썬 버전 3.6.0 파이썬 모듈 팬더, abc, numpy, scipy, sklearn, sys, PyQt5, sys, mRMR, 수학과 matplotlib를 사용 하 여 개발 되었다. 이 연구에 사용 된 재료는 재료의 테이블에에서 나열 됩니다.
1. 준비 데이터 매트릭스 레이블 클래스
- 그림 1A에서 볼 수 있듯이 탭 또는 쉼표로 구분 된 매트릭스 파일 데이터 매트릭스 파일을 준비 합니다.
참고: 각 행에는 기능, 모든 값 이며 첫 번째 항목 기능 이름. 기능은 microarray 기반 transcriptome 데이터 집합에 대 한 ID를 probeset 또는 methylomic 데이터 집합의 메 틸 화 값 시스테인 잔류물과 같은 다른 값 ID 있을 수 있습니다. 각 열 샘플 이름 되 고 첫 번째 항목으로 주어진된 샘플의 기능 값을 제공 합니다. 행 탭 (그림 1B) 또는 쉼표 (그림 1C)에 의해 열으로 구분 됩니다. 매트릭스 탭 구분 파일 파일 확장자.tsv에 의해 인식 되 고 쉼표로 구분 된 매트릭스 파일 확장.csv 합니다. 이 파일 저장 하 여 행렬으로.tsv 또는.csv 형식 Microsoft Excel과 같은 소프트웨어에서 생성 될 수 있습니다. 데이터 매트릭스 컴퓨터 코딩 또한 생성 될 수 있습니다. - 탭 또는 쉼표로 구분 된 행렬 파일 (그림 1D), 데이터 매트릭스 파일에 유사한 클래스 레이블 파일을 준비 합니다.
참고: 첫 번째 열 제공 샘플 이름 하 고 각 샘플의 클래스 레이블 클래스제목 열에 제공 됩니다. 추가 열을 추가할 수 있도록 최대한 호환성 코딩 과정에서 간주 됩니다. 클래스 레이블 파일을.tsv 또는.csv 파일로 포맷 수 있습니다. 열 클래스 에 이름을 어떤 용어 수 있으며 샘플의 두 개 이상의 클래스가 있을 수 있습니다. 사용자는 다음과 같은 분석에 대 한 클래스의 2를 선택할 수 있습니다.
2. 로드 데이터 매트릭스 레이블 클래스
- 데이터 매트릭스와 클래스 레이블 소프트웨어에 로드 합니다. 부하 데이터 매트릭스 사용자 지정 데이터 매트릭스 파일을 선택 버튼을 클릭 합니다. 부하 클래스 레이블 을 해당 클래스 레이블 파일 선택 버튼을 클릭 합니다.
참고: 두 파일을 로드 하는 kSolutionVis 두 파일 사이의 호환성의 일상적인 화면을 실시 한다. - 기능 및 데이터 매트릭스 파일에서 샘플 요약. 데이터 매트릭스 파일의 크기를 견적 한다.
- 샘플 및 클래스 레이블 파일에서 클래스를 요약 합니다. 클래스 레이블 파일의 크기를 견적 한다.
- 데이터 매트릭스에서 각 샘플은 클래스 레이블 여부를 테스트 합니다. 클래스 레이블 샘플의 수를 요약 합니다.
3. 요약 하 고 데이터 집합의 기준선 통계 표시
- 지정 된 키워드 입력 없이 요약, 버튼을 클릭 하 고 소프트웨어 20 인덱싱된 기능과 해당 기능 이름을 표시 됩니다.
참고: 사용자가 그들의 기준선 통계 및 모든 입력된 샘플 중 해당 값 분포를 볼 수 찾을 하고자 기능 이름을 지정 해야 합니다. - 키워드, 예를 들어 "1000_at", textbox 요약 될 특정 기능을 찾을 수 기능 에 제공 합니다. 이 기능에 대 한 기준선 통계를 요약 버튼을 클릭 합니다.
참고: 키워드는 어디서 나 사용자에 대 한 검색 과정을 촉진 대상 기능 이름에 나타날 수 있습니다. - 버튼 요약 특정된 키워드와 함께 더 이상의 기능을 찾을 수를 클릭 한 다음 하나의 특정 기능을 요약 하는 위의 단계를 진행 하는 독특한 기능 ID를 지정 합니다.
4. 클래스 레이블 및 상위권 기능의 수를 결정
- (중간)에 그림 2 와 같이 긍정적인 ("P (33)") 및 네거티브 ("N (95)") 클래스의 이름을 클래스 긍정적 이 고 부정적인 클래스드롭다운 상자에서 선택 합니다.
참고: 그것은 균형된 이진 분류 데이터 집합, 즉, 긍정적이 고 부정적인 샘플의 숫자 간의 차이 최소화를 선택 하는 것 좋습니다. 샘플 수는 두 개의 드롭다운 상자에서 각 클래스 라벨의 이름 뒤 괄호에 또한 주어진 다. - 최고의 기능 (매개 변수 pTopX)의 수로 10 Top_X (?) 드롭다운 상자에서 선택 기능 하위 집합의 포괄적인 화면에 대 한
참고: 소프트웨어는 자동으로 P-각 기능을 비교 하는 긍정적이 고 부정적인 클래스의 t-검정에 의해 계산 된값 에 의해 모든 기능을 평가 한다. 더 작은 P-기능값 샘플 두 클래스 사이의 더 나은 차별 힘이 있다. 종합 심사 모듈은 컴퓨터로 집중적. PTopX 매개 변수 기본적으로 10입니다. 그들은 만족을 찾을 때까지 사용자가 10 ~ 50의 범위에서이 매개 변수를 변경할 수 있습니다 좋은 분류 공연 하위 집합 기능.
5. 다른 공연에 대 한 시스템 매개 변수 조정
- 드롭다운 상자의 Acc/bAcc (?) 선택된 분류자 극단적인 학습 기계 (느릅나무)에 대 한 성능 측정 (pMeasurement) 정확도 (Acc)를 선택 합니다. 이 매개 변수의 다른 옵션은 측정 정확도 균형 (bAcc).
참고: FN, TN, TP 시키고 FP 수 참 긍정, 거짓 제외, 진정한 제외 하 고 틀린 확실성의 수가 각각. Acc 측정 이란 (TP+TN)/(TP+FN+TN+FP),6균형된 데이터 집합 가장 잘 작동 하는. 그러나 Acc 에 대 한 최적화 된 분류자 부정적인 샘플 수는 긍정적인 것 들의 그것 보다 훨씬 더 큰 하는 경우 모든 샘플 부정적인 클래스에 할당 하는 경향이 있다. BAcc (Sn + Sp)로 정의 됩니다 / 2, 어디 Sn = TP/(TP+FN) 및 Sp = TN/(TN+FP) 샘플를 각각 부정과 긍정에 대 한 잘못 예측 된 가격입니다. 따라서, bAcc 두 개의 클래스를 통해 예측 성과 정규화 하 고 두 개의 불균형된 클래스 균형된 예측 성능으로 이어질 수 있습니다. Acc 는 pMeasurement의 기본 선택입니다. 소프트웨어는 기본적으로 느릅나무 분류자를 사용 하 여 계산 분류 공연. 사용자 또한 SVM (지지 벡터 머신), KNN (k 가까운 이웃), 결정 트리, 또는 Naïve Bayes 분류자를 선택할 수 있습니다. - 입력된 상자에 지정 된 성능 측정에 대 한 구분 값 0.70 (매개 변수 pCutoff) 선택 pCutoff:.
참고: Acc 와 bAcc 0과 1 사이의 범위와 사용자 지정 값 pCutoff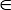 [0, 1] 일치 하는 솔루션을 표시 하는 구분으로. 소프트웨어는 포괄적인 기능 집합, 실시 그리고 pCutoff 의 적절 한 선택 더 직관적이 고 노골적인 3D 시각화를 만들 것입니다. PCutoff는 0.70에 대 한 기본 값입니다.
[0, 1] 일치 하는 솔루션을 표시 하는 구분으로. 소프트웨어는 포괄적인 기능 집합, 실시 그리고 pCutoff 의 적절 한 선택 더 직관적이 고 노골적인 3D 시각화를 만들 것입니다. PCutoff는 0.70에 대 한 기본 값입니다.
6. 파이프라인을 실행 하 고 대화식 시각화 된 결과
- 클릭 분석 파이프라인을 실행 하 여 그림 2 (아래) 시각화 플롯을 생성 합니다.
참고: 왼쪽된 테이블 모든 기능 하위 집합 및 앞에서 설명한5느릅나무, 분류자의 10 교차 유효성 검사 전략에 의해 계산 그들의 pMeasurement 를 제공 합니다. 두 개의 3 차원 분산형 플롯 및 2 선 플롯 현재 매개 변수 설정 가진 기능 하위 집합 심사 절차에 대 한 생성 됩니다. - 최고의 기능 하위 집합 (매개 변수 piFSNum)의 수의 기본 (매개 변수 piCutoff, 입력된 상자 값), pMeasurement 차단의 기본 가치와 10 0.70를 선택 합니다.
참고: 파이프라인 매개 변수 pTopX, pMeasurement, 및 pCutoff를 사용 하 여 실행 됩니다. 그러나 하위 집합 추가 될 수 있습니다 검색된 기능 차단 piCutoff를 사용 하 여, piCutoff 보다 작을 수 없습니다 pCutoff상영. 따라서, piCutoff 는 pCutoff 로 고만 성능 측정 ≥ piCutoff 와 기능 하위 집합을 시각화 됩니다. PiCutoff 의 기본값은 pCutoff입니다. KSolutionVis 많은 솔루션 및만 최고의 piFSNum 를 감지 하는 때로는 (기본값: 10) 기능 하위 집합을 시각화 합니다. 소프트웨어에 의해 감지 하는 기능 하위 집합의 수는 piFSNum보다 작은, 모든 기능 하위 집합 구상 될. - 수집 하 고 그림 3에서처럼 소프트웨어에 의해 감지 하는 기능을 해석.
참고: 왼쪽된 상자에 테이블 검색된 기능 하위 집합 및 그들의 성능 측정을 보여줍니다. 처음 3 개의 열의 이름은 "F1", "F2", 및 "F3"입니다. 한 행에 그들의 순위 순서로 주어진 각 기능 집합에는 세 가지 특징 (F1 < f 2 < F3). 마지막 열 각 기능 부분 집합의Acc ( bAcc) 성능 측정을 제공 하 고 그것의 열 이름 (Acc 또는 bAcc)는 pMeasurement의 값.
7. 3 차원 분산형 플롯 시각화를 해석합니다 하 고 마찬가지로 효과적 이진 분류 공연 3 차원 분산형 플롯을 사용 하 여 기능 하위 집합을 해석
- 3D 산 점도 최고의 분류 공연 (Acc 또는 bAcc)와 상위 10 기능 하위 집합의 그림 3 (중간 상자) 소프트웨어에 의해 감지를 생성 하기 위해 분석 버튼을 클릭 합니다. 그들의 계급의 순서를 오름차순에서 기능 하위 집합에 3 개의 기능을 정렬 하 고 3 개의 특징의 순위를 사용 하 여 F1/F2/F3 축, 즉, F1으로 < f 2 < f 3.
참고: 도트의 색상 해당 기능 하위 집합의 이진 분류 성능을 나타냅니다. Dataset는 마찬가지로 효과적인 성능 측정 여러 기능 하위 집합을 할 수 있습니다. 따라서, 상호 작용 하 고 단순화 된 점도 필요 하다. - 값 입력된 상자에 0.70을 변경 pCutoff: (오른쪽 상자) 그림 3 보듯이 3D 산 점도 성능 측정 ≥ piCutoff와 기능 하위 집합을 생성 하기 위해 분석 단추를 클릭. 새 창 3D 산 점도의 보기 각도 수동으로 조정 하 여 3D 튜닝 을 하는 버튼을 클릭 합니다.
참고: 각 기능 부분 집합 같은 방식으로 위에 점이 표시 됩니다. 기본 각도에서 3D 산 점도 생성 됩니다. 3D 시각화 및 튜닝, 별도 창을 촉진 하기 위하여 3D 튜닝버튼을 클릭 하 여 열 것 이다. - 검색된 기능 하위 집합의 중복을 줄이기 위해 줄이기 버튼을 클릭 합니다.
참고: 경우 사용자가 추가 기능 세 쌍둥이 선택 하 고 기능 하위 집합의 중복을 최소화 하고자, 소프트웨어 또한 제공 합니다 mRMR 기능 선택 알고리즘을 사용 하 여이 함수를. 축소 버튼을 클릭 하면 kSolutionVis 기능 세 쌍둥이에 중복 기능을 제거 하 고 테이블을 다시 생성 그리고 두 분산형 플롯 위에서 언급 한. 기능 세 쌍둥이의 제거 기능은 테이블에서 핵심 단어에 의해 대체 됩니다. F1/F2/F3 축에 None 의 값은 piFSNum (F1/F2/F3의 정상 값의 범위는 [1, top_x])의 값으로 표시 됩니다. 따라서, 점 없음 값을 포함 하는 3D에서 "국외 자" 점 플롯 될 나타날 수 있습니다. 수동으로 조정할 수 있는 3 차원 플롯 보충 자료에 "3 차원 점 작의 수동 조정"에서 찾을 수 있습니다.
8. 찾기 진 주석 및 인간 질병의 그들의 협회
참고: 8 ~ 10 단계는 DNA와 단백질의 시퀀스 레벨에서 유전자에 주석을 추가 하는 방법을 보여주는 것입니다. 첫째, 위의 단계에서 각 바이오 마커 ID의 유전자 상징 데이비드32, 데이터베이스에서 검색 됩니다 하 고 두 명의 대표 웹 서버 각각의 DNA와 단백질 수준에서이 유전자 기호를 분석 하는 데 사용 됩니다. 서버 GeneCard 주어진된 유전자 기호의 포괄적인 기능 주석 하며 남자 데이터베이스 (OMIM)에 있는 온라인 Mendelian 상속 질병 유전자 협회의 가장 포괄적인 변호사를 제공 합니다. UniProtKB 서버는 가장 포괄적인 단백질 데이터베이스 중 하나 이며 서버 그룹 기반 예측 시스템 (GPS)는 신호 인 산화의 매우 큰 목록이 kinases의 예측.
- 복사 하 고 웹 브라우저에 데이터베이스 데이비드의 웹 링크를 붙여이 데이터베이스의 웹 페이지를 엽니다. 유전자 ID 변환 그림 4A 에서 본 링크를 클릭 하 고 기능 Id 38319_at/38147_at/33238_at (그림 4B) ALL1 데이터 집합의 첫 번째 바이오 마커 집합의 입력. 유전자 목록 링크를 클릭 하 고 그림 4B와 같이 제출 목록 을 클릭 합니다. 관심의 주석을 검색 하 고 표시 유전자 목록 (그림 4C)을 클릭 합니다. 유전자 기호 (그림 4D). 의 목록
참고: 여기 검색 유전자 기호 추가 기능 주석을 다음 단계에서 사용 됩니다. - 복사 하 고 웹 브라우저에 데이터베이스 진 카드의 웹 링크를 붙여이 데이터베이스의 웹 페이지를 엽니다. 데이터베이스 쿼리 입력된 상자에 유전자의 이름 CD3D을 검색 하 고 표 1 과 그림 5A와 같이 유전자 카드33,34에서이 유전자의 주석의 찾을.
참고: 유전자 카드 명명법, genomics, proteomics, subcellular 지 방화, 그리고 참여 경로 다른 기능 모듈을 제공 하는 포괄적인 유전자 기술 자료입니다. 그것은 또한 PDB/PDB_REDO35, 기념물과 진36, OMIM37, UniProtKB38같은 다양 한 다른 생물 의학 데이터베이스에 외부 링크를 제공합니다. 기능 이름이 표준 유전자 기호 경우39변환할 데이터베이스 합을 사용 합니다. CD3D는 유전자는 T 세포 수용 체 T3 델타 체인의 이름입니다. - 복사 하 고 웹 브라우저에 데이터베이스 OMIM의 웹 링크를 붙여이 데이터베이스의 웹 페이지를 엽니다. 유전자의 이름 CD3D을 검색 하 고 표 1 과 그림 5B에서 같이 데이터베이스 OMIM37에서이 유전자의 주석의 찾을.
참고: OMIM 지금 인간의 유전자 연결 가능한 질병을 가장 포괄적이 고 신뢰할 수 있는 소스 중 하나로 제공합니다. OMIM 박사 빅터 A. McKusick 질병 관련 유전자 변이40카탈로그에 의해 시작 되었다. OMIM 지금 15000 인간의 유전자와 12 월 현재 이상 8500 고기 1세인트 2017을 다루고 있습니다.
9. 주석이 인코딩된 단백질과 포스트 번역 상 수정
- 복사 하 고 웹 브라우저에 데이터베이스 UniProtKB의 웹 링크를 붙여이 데이터베이스의 웹 페이지를 엽니다. 유전자의 이름 CD3D UniProtKB의 쿼리 입력된 상자에 검색 하 고 표 1 과 그림 5C같이38데이터베이스이 유전자의 주석의 찾을.
참고: UniProtKB 단백질, 명칭 및 기능 정보를 포함 하 여에 대 한 주석의 풍부한 소스를 수집 합니다. 이 데이터베이스는 또한 PDB/PDB_REDO35,37, OMIM Pfam41등 다른 널리 사용된 데이터베이스에 외부 링크를 제공 합니다. - 복사 하 고 웹 브라우저에 웹 서버 GPS의 웹 링크를 붙여이 웹 서버의 웹 페이지를 엽니다. UniProtKB 데이터베이스38 에서 바이오 마커 유전자 CD3D에 의해 단백질 시퀀스를 검색 하 고 표 1 과 그림 5D에서 같이 GPS, 온라인 도구를 사용 하 여 단백질의 포스트 번역 상 수정 (PTM) 잔류물을 예측.
참고: 생물 학적 시스템은 역동적이 고 복잡 한, 그리고만 알려진된 정보를 수집 하는 기존 데이터베이스. 따라서, 생물 의학 예측 온라인 도구 뿐만 아니라 오프 라인 프로그램 가설된 메커니즘을 보완 하기 위해 유용한 증거를 제공할 수 있습니다. GPS는 개발된에 대 한 향상 된 12 년7,42 고 단백질의 특정된 펩 티 드 순서43,44PTM 잔류물을 예측 하는 데 사용할 수 있습니다. 도구는 단백질의 subcellular 위치45 및 녹음 방송 요인 바인딩 모티브 46 다른 사람들의 예측을 포함 하 여 다양 한 연구 주제에 대 한 사용할 수 있습니다.
10. 주석을 단백질 단백질 상호 작용 및 그들의 풍부한 기능 모듈
- 복사 하 고 웹 브라우저에 웹 서버 문자열의 웹 링크를 붙여이 웹 서버의 웹 페이지를 엽니다. CD3D와 P53, 유전자에 대 한 목록을 검색 하 고 찾을 문자열47데이터베이스를 사용 하 여 그들의 조율 된 속성. 동일한 절차 다른 웹 서버, 데이비드32를 사용 하 여 실행 될 수 있습니다.
참고: 개별 유전자에 대 한 상기 주석 외 있다 유전자의 그룹의 속성을 조사 하기 위해 사용할 수 있는 많은 대규모 정보 도구. 최근 연구는 개별적으로 나쁜 마커 유전자 보다 유전자 세트5를 구성 수 있습니다 설명 했다. 따라서, 그것은 더 복잡 한 생체에 대 한 화면에 계산 비용 가치입니다. 데이터베이스 문자열 알려진 또는 예측 상호 작용 연결을 시각화 수 있습니다 그리고 데이비드 서버는 쿼리 된 유전자47,32에서 중요 한 표현 형-연결 기능 모듈을 검색할 수 있습니다. 다양 한 다른 대규모 정보 분석 도구를 사용할 수 있습니다.
11. 생성 된 바이오 마커 하위 집합 및 시각화 플롯 수출
- .Tsv 또는.csv 텍스트 파일로 추가 분석에 대 한 감지 된 바이오 마커 하위 집합을 내보냅니다. 모든 감지 된 바이오 마커 하위 집합의 테이블에 테이블 내보내기 버튼을 클릭 하 고 텍스트 형식을 다른 이름으로 저장을 선택.
- 이미지 파일로 시각화 플롯을 내보냅니다. 각 플롯 아래 저장 버튼을 클릭 합니다 하 고 어떤 이미지 포맷으로 저장을 선택 합니다.
참고: 소프트웨어 픽셀 형식.png 및 벡터 포맷.svg 지원합니다. 픽셀 이미지는 벡터 이미지 저널 게시 목적에 필요한 어떤 해상도로 변환 될 수 있습니다 하는 동안 컴퓨터 화면에 표시 하기 위한 좋은.
결과
이 워크플로 (그림 6)의 목표는 이진 분류 데이터 집합에 대 한 유사한 효율성으로 여러 바이오 마커 하위 집합을 검색 하는입니다. 전체 과정은 두 개의 예제 데이터 집합 ALL1 및 ALL2 최근 출판 바이오 마커 검출에서 추출 연구12,48에 의해 나와 있습니다. 사용자는 보조 자료에 지시에 따라 kSolutionVis를 설치?...
토론
이 연구는 이진 분류 사용자 지정 데이터 집합의 따라 하기 쉬운 멀티 솔루션 바이오 마커 검출 및 특성화 프로토콜을 제공합니다. 소프트웨어는 사용자와 소프트웨어의 GUI를 사용 하 여 쉽게 그들의 데이터 집합을 조사 하는 생물 의학 연구원을 수 있도록 다양 한 파일 형식에 대 한 유연한 가져오기/내보내기 인터페이스에 중점을 둔다. 이 연구는 또한 유사 하 게 효과적인 모델링 공연, 이전 많?...
공개
우리는이 보고서와 관련 된 충돌의 관심 있다.
감사의 말
이 작품은 중국 과학원 (XDB13040400)와 길 림 대학에서 시작 그랜트의 전략적 우선 순위 연구 프로그램에 의해 지원 되었다. 익명 검토자와 생물 의학 테스트 사용자 유용성 및 kSolutionVis의 기능 개선에 그들의 건설적인 의견을 주셔서 감사 했다.
자료
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
참고문헌
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유