Method Article
증강 기능 다중 CRISPR 기반 증강 간섭 셀 라인에서의 해 부
요약
이 프로토콜 설계 및 다중화 비활성화 융해 단백질 SID4X-dCas9-리아로 알려진 또한 증강 간섭 (증강 인자-i)와 강화의 대상으로 수행 하는 데 필요한 단계를 설명 합니다. 이 프로토콜 유전자 발현을 조절 하는 강화의 식별을 가능 하 게 일반적인 대상 유전자 규제 강화 간의 관계의 해 부를 촉진 한다.
초록
여러 강화 종종 주어진된 유전자 조절 아직 대부분 유전자에 대 한 그것은 불분명 남아 있는 강화 유전자 발현에 대 한 필요 하 고 어떻게 이러한 강화 결합 transcriptional 응답. 강화의 수백만 확인 되었습니다로 높은 처리량 도구 게놈 넓은 규모로 증강 기능 확인 필요 합니다. 증강 기능 공부에 대 한 현재 방법은 유전자 삭제 nuclease 실력 Cas9를 사용 하 여 만드는 포함 하지만 여러 연속 클론 셀 라인으로이 기술을 사용 하 여 여러 강화의 조합 효과 공부 하기 어려운 생성. 여기, 선물이 증강-i, 그들의 생 loci에서 동시에 여러 강화의 기능 심문 수 CRISPR 간섭 기반 방법. 증강 인자-i 타겟된 loci에서 히스톤 deacetylation 통해 증강 비활성화 달성 하기 위해 융합 nuclease 불충분 한 두 억압 도메인의 Cas9, SID와 리아, 사용 만든다. 이 프로토콜 가이드 대상된 지역의 일시적인 비활성화 수 있도록 RNAs의 과도 transfection를 활용 하 고 특히 조직 문화 설정에서 자극을 유도할 수 있는 transcriptional 응답을 차단에 효과적 이다. 증강 인자-i은 모두 그것의 게놈을 대상으로 글로벌 유전자 발현에 미치는 영향에 매우 구체적인. 이 프로토콜에서 얻은 결과 증강 유전자 발현, 기여, 규모와 어떻게 기여는 영향을 받는 다른 인근 강화에 기여 하 고 있는지 이해 하는 데 도움이.
서문
시퀀싱 프로젝트 인코딩1, 로드맵 Epigenomics2, FANTOM 등 대규모3 확인 했습니다 인간 게놈 내 상 상속 강화의 수백만 수백 종류의 세포에서. 각 발기인 4.9 강화의 평균 연결 하 고 각 증강 연락처 2.4 유전자3의 평균 유전자 발현은 종종 여러 분산된 규정 상호 작용의 통합의 결과 제안 추정 된다. 중요 한 나머지 과제 정의 개별 강화 뿐만 아니라 유전자 발현에 기여 하는 하지만 어떻게 그들이 결합 하 여 식에 영향을 미칠. 유전 접근 일반적으로 쥐5 초파리4 에서 모형 유기 체에 강화 간의 관계를 식별 하는 데 사용 됩니다. 그러나,이 실험은 어렵고 여러 유전자에서 여러 강화의 연구에 대 한 낮은 처리량.
큰 규모로 증강 기능을 공부 하는 한 가지 방법은 대규모 병렬 기자 분석 실험을 포함 한다. 이 분석 실험 기자 유전자6식 드라이브를 그들의 능력에 대 한 DNA 시퀀스의 수천의 동시 상영에 대 한 수 있습니다. 이 분석 실험 DNA 순서 혼자 유전자 규칙 정보7전달 하기 충분할 수 나타났습니다, 그들은 온 네이티브 chromatin 컨텍스트 외부에서 수행 되 고의 주의 분리의 발기인. 또한, 대규모 병렬 기자 분석에 분석 되 고 DNA 시퀀스의 크기는 일반적으로 관련 주변 시퀀스를 제외 시킬 수 있습니다 미만 200 basepairs. 중요 한 것은, 기자가 분석만 한 번에 하나의 시퀀스의 활동 측정, 그들은 할 하지 고려 강화 사이 존재할 수 있는 복잡 한 관계. 따라서, 대규모 병렬 기자 분석 DNA 시퀀스의 본질적인 활동에 대 한 정보를 제공 될 수 있습니다, 그들은 할 하지 반드시 우리에 게는 게놈의 맥락에서 그 DNA 시퀀스의 기능.
최근 개발 된 CRISPR/Cas9 도구8 있다 그들은 내 생 로커 스에서 강화의 삭제에 대 한 허용 유전자 규칙의 연구를 촉진 했다. 그러나, genomic 불안정성으로 이어질 수 있습니다 여러 강화를 동시에 삭제 하 고 그것은 단일 셀 라인에서 연속 증강 삭제를 생성 하는 시간이 소요. 또한, 새로운 게놈 시퀀스 삭제 복구, 다음의 사이트에서 생성 되 고이 시퀀스 규제 기능을 얻을 수 있습니다. Cas9의 대체 버전9,10 활성화 또는 nuclease 불충분 한 형태의 Cas911,12 도메인을 억압의 융해에 의존 하는 유전자 발현을 변조를 위해 특별히 개발 되었습니다. (dCas9)입니다. 이 융해 단백질 그들은 육체적으로 DNA 순서를 변경 하 고 대신 epigenetics 규제 영역을 심문 하기 위해 변조로 여러 loci를 동시에 공부에 이상적입니다. 가장 널리 사용 되는 억압 적인 퓨전 리아, KAP1 공동 진압 복잡 한 신병이 억압 관련 히스톤 H3 리 진 9 trimethylation (H3K9me3)13의 증 착을 홍보입니다. dCas9-리아, 일컬어 CRISPR 간섭14,16; 유전자의 식15,그들의 공헌에 대 한 대상과 화면 개별 강화를 사용 되었습니다. 그러나, 그것은 하지 최적화 되었습니다 여러 영역을 동시에 대상에 대 한. 강화, 모자이크-seq17, 다중 CRISPR 간섭의 한 버전 사용 단일 셀 RNA-seq는 판독 하지만이 기술은 단일 셀의 낮은 감도 때문에 높게 표현한 유전자의 연구에만 적합 하 고 비싼 RNA-이
우리가 스 트로 겐에 transcriptional 응답의 컨텍스트 내에서 조합 증강 기능 해 부에 대 한 CRISPR 간섭을 기반 방법을 개발 하고자 했다. 스 트로 겐-응답 유전자의 약 절반 2 이상의 강화 근처의18, 에스트로겐 수용 체 알파 (응급실)을 준수할 여러 강화 에스트로겐 응답에 참여 하 고 규제 논리 필요 이해 수 있습니다 제안 포함 동시에 여러 명의 강화를 목표로. 초기 연구에서 발기인 CRISPR 방해를 사용 하 여 모든 발기인은 동등 하 게 리아 중재 억압19응답 제안, 우리 dCas9 별개의 억압 적인 도메인 추가의 비활성화를 촉진 수 있습니다 권유 다양 한 강화입니다. 우리 선택 Sin3a 상호 작용 도메인의 Mad1 (SID)20 히스톤 deacetylases21의 채용에 리드에 transcriptional 활동에 연관 된 히스톤 아 세 틸 그룹을 제거 하는. 중요 한 것은, SID 도메인 dCas922 와 이야기23, 융합 하는 때 유전자 발현을 감소에 효과적 이었고 Sin3a 다양 한 증강 시퀀스 컨텍스트24에 강력한 억압 적인 공동 요인이 될 표시 되었습니다. 우리 응급실, 준수할의 다른 강화 10 개를 대상으로 SID4x-dCas9-리아 (증강 인자-i)를 사용 하 고 응급실 바인딩 사이트 (ERBS) 4 유전자18에 스 트로 겐 transcriptional 응답에 필요한 식별 합니다. 우리는 또한 에스트로겐 transcriptional 응답의 생산에 협력 하는 사이트를 식별 하는 증강의 조합 대상. 우리는 50 개 사이트까지 잠재적으로 타겟팅 할 수 있습니다 동시에 감지 유전자 식 변화 발견. 칩 seq와 RNA-seq 사용 하 여, 우리는 증강-i은 여러 강화를 동시에 공부에 대 한 매우 구체적인 기술 시연 했다.
이 프로토콜에서 우리는 증강-i, 조직 문화 설정에 동시에 여러 강화의 기능 연구를 가능 하 게 하는 유연한 기술 수행에 관련 된 단계를 설명 합니다. 증강 인자-i 유전자 삭제와 상관 매우 하지만 히스톤 deacetylases (HDACs)에 따라 달라 집니다 일시적인 비활성화를 제공 합니다. 바이러스 성 벡터를 통해 안정적인 통합 반대 과도 transfection을 통해 가이드 RNAs를 제공,이 프로토콜 증 착 및 H3K9me3의 잠재적인 확산 방지 합니다. 이 프로토콜 세부 정보 가이드 RNA 디자인 및 깁슨 어셈블리의 transfection 통해 복제 RNAs lipofection를 사용 하 여 가이드와 결과 유전자 발현의 분석 정량 하 여 변경. 우리는 또한 게놈 transcriptome 수준에 증강-i 대상의 특이성을 평가 하기 위한 메서드가 포함 됩니다. 이 기술을 연구 개발 하는 동안 어 유전자 규정 강화 인간 암 세포 라인에서 어떤 포유류 증강의 해 부에 적용 됩니다.
프로토콜
1. 셀의 세대 라인 안정적으로 표현 하는 SID4X-dCas9-리아
참고: transfection 조건 및 약물 농도 여기 있다 최적화 되었습니다 이시카와 셀, 자궁내 막 암 세포 선, 10 %FBS 1% 페니실린/스 (완전 한 RPMI)와 보충 RPMI 1640 미디어에서 성장. 다른 셀 라인 다른 transfection 조건 및 약물 농도 필요할 수 있습니다. 사용자가 또한 야생-타입 셀, SID4X-dCas9-리아 가이드 RNA 플라스 미드; 표현 함께 표현 하는 플라스 미드와 안정 셀 라인을 생성 하는 대신에 과도 transfection 실험을 수행할 수 있습니다. 그러나, 일시적인 transfections에서 결과 transfection에 의해 다를 수 있습니다 SID4X-dCas9-리아 수준으로 재현 하기 어려울 수 있습니다.
- 완전 한 RPMI의 3 mL에 30-50% 합류 (약 300000 이시카와 셀)에서 6 잘 플레이트의 적어도 2 우물에 이시카와 셀 플레이트.
- 셀에서 미디어 발음 셀 1 x PBS (pH 7.4)을 한 번 씻어. PBS를 발음 하 고 트립 신 (4 mL 10 cm 접시 또는 T-75 플라스 크 5 mL)을 추가 합니다.
- 37 ° C에서, 분리 된 셀에 대 일 분 마다 확인 하 고 배를 부드럽게 흔들어 ~ 5 분에 대 한 셀을 품 어.
- 셀을 분리 했으면 일단 피 펫 위아래로 몇 번, 그리고 연결 된 셀 출시를 그릇의 측면 아래로 부드럽게 피 펫 셀 trypsinized.
- 15 mL 원뿔 튜브에 세포를 전송 하 고 250 x g에 5 분에 대 한 셀 아래로 회전.
- 트립 신을 발음 하 고 미디어의 5-10 mL에 셀 resuspend. P1000 피 펫을 사용 하 여 필요한 경우 세포 덩어리를 분리.
- 셀을 계산 하 고 접시 당 3 mL의 총 볼륨에 잘 ~ 300000 셀 하는 데 필요한 볼륨을 결정 합니다. 6 잘 플레이트의 2 별도 우물에 셀을 추가 합니다. 각 채우기 완료 RPMI와 3 mL을 잘.
- 셀은 접시에 고르게 분포 되도록 도금 후 접시 마다 5 분 첫 번째 15 분에 부드럽게 흔들어. 현미경을 사용 하 여 셀 우물의 중간에서 분산가 되도록.
- 도금의 24 시간 안에 다음 transfections 관심의 셀 라인에 대 한 적절 한 transfection 시 약을 사용 하 여 수행 합니다. 이시카와 셀 아래에 설명 된 절차를 사용 합니다.
참고:이 프로토콜 양이온 liposome에 기초를 둔 transfection 시 약의 사용을 가정합니다. Electroporation 종류는 이러한 시 약에 매우 민감한 또는 lipofection와 낮은 transfection 효율을 전시 하는 대체 방법을 제공 합니다. Transfection 조건 증강-i 실험을 시도 하기 전에 관심의 셀 라인에 대 한 낙관 되어야 한다.- 1.7 mL Eppendorf 관에에서 희석 SID4X-dCas9-리아 플라스 미드의 2.5 μ g 및 800 튜브에 최종 볼륨 155 µ L 이며 플라스 미드의 최종 농도 0.020 µ g / µ L를 혈 청 자유로운 미디어에 형광 단백질을 표현 하는 플라스 미드의 ng.
- 다른 튜브에 pCMV GFP 등 네오 마이 신 저항 카세트를 포함 하지 않는, 혈 청 자유로운 매체에 관에서 마지막 볼륨 155 µ L 이며 플라스 미드의 최종 농도 0.020 µ g / µ L 플라스 미드의 3.3 µ g을 희석.
- 소용돌이 각 튜브를 짧게 하 고는 microfuge를 사용 하 여 아래로 회전.
- 각 튜브를 transfection 시 약 (자료 테이블)의 9.9 µ L를 추가 합니다. 짧게 낮은 속도에서 vortexing에 의해 혼합. microfuge와 다운 튜브를 회전 합니다.
- 적어도 5 분, 하지만 20 분 더 이상 실내 온도에 튜브를 품 어.
- Biosafety 캐비닛에 추가할 준비 DNA: 시 약 믹스의 150 µ L dropwise 6 잘 플레이트에 대 한 잘. 시 약 DNA 준비: 믹스의 다른 튜브에 대 한 반복 합니다. 부드럽게 소용돌이 의해 접시를 혼합 하 고 인큐베이터에 접시를 반환 합니다.
- 하루 2 게시물 transfection에 미디어를 변경 하 고 600 ng/μ의 최종 농도에 G418 보충. 이 농도 셀 형식에 대 한 최적화 할 수 있습니다.
- 완전 한 RPMI 미디어 변화와 G418 컨트롤 페 셀은 죽은 포함 하는 SID4X-dCas9-리아 우물 confluent 될 때까지 2-4 주 동안 매일 보충. 정확한 양의 셀 복구 하는 데 필요한 시간 셀의 2 배 시간에 따라 달라 집니다.
- 때, confluent 될 셀 통로 있는 T-25 또는 T-75 배를 완전 한 RPMI G418의 낮은 복용량 (이시카와 셀 300 ng/μ). 이 통로 중 확인 2 aliquots ~ 100, 000 셀의 각 (약 1/10일 6 잘 플레이트의) 2 별도 1.7 mL Eppendorf 관에 RNA와 DNA 분리, 각각. (5 분, 250 x g) 아래로이 관을 회전, pipetting, 여 트립 신을 제거 하 고 나중에 사용-20 ° C에서 튜브를 고정.
- 상업적으로 사용할 수 있는 키트를 사용 하 여 게놈 DNA를 분리 하 고 셀 라인 내의 융해 단백질의 존재를 확인 하는 "pAC95_PCR" 또는 "SID4X_PCR" 뇌관 (표 1)를 사용 하 여 PCR을 수행. 긍정적인 통제로 부정적인 컨트롤로 부모의 선에서 추출 하는 genomic DNA와 SID4x-dCas9-리아 플라스 미드 DNA를 사용 합니다. 게놈 DNA와 자전거 다음과의 50-100 ng 높은 중 합 효소 마스터 혼합 사용: 98 ° C 30에 대 한 s, 25의 사이클 (98 ° C 10 s, 58 ° C 30에 대 한 s, 2 분 동안 72 ° C), 5 분 동안 72 ° C 4 ° c.에서 개최
- RNA 수준에 융해 단백질의 표현을 확인 하려면 상용 키트를 사용 하 여 셀 라인에서 추출한 RNA 정량 수행 합니다. "DCas9_qPCR" 뇌관 (표 1),이 프로토콜의 단계 6.3에서에서 제공 1 단계 정량 Pcr 프로토콜을 사용 합니다.
- 확인 하려면 융합 단백질 수준 표현 수행 하십시오 서쪽 lysates 셀 라인에서에 오 점. 반대로 깃발 또는 안티를 사용 하 여-하 항 체 융합 단백질을 검출 하기 위하여.
2. 가이드 RNA 디자인
참고:이 프로토콜은 U6 가이드 RNA 벡터 교회 연구소에 의해 만들어진된 Addgene (Addgene 41824)에서 사용할 수 있는 복제와 함께 사용 하기 위해 설계. Puromycin 저항 41824로 동일한 복제 전략에 대 한 허용을 포함 하는이 벡터의 버전을 만들려면, 우리 pGL3 U6 sgRNA PGK puromycin 벡터 (Addgene 51133)에이 벡터에서 여러 복제 사이트를 이동. Addgene 41824 또는 puromycin (Addgene 106404) 우리의 버전 아래에 설명 된 복제 전략와 호환 됩니다.
- 600-900 basepairs를 관심의 각 규제 지역에 대 한 DNA 시퀀스를 가져옵니다. 어디 (그림 2A) 관심 영역을 정의 하는 지침에 대 한 전사 인자 바인딩 사이트 또는 chromatin 내게 필요한 옵션을 사용 합니다.
참고: 예제 그림 2A 에서 업스트림 및 다운스트림 기능을 하는 동안 그것은 또한 규제 요소 introns 내에 있는 대상 수 있습니다. - 모든 시퀀스 FASTA 형식을 사용 하 여 단일 텍스트 파일에 배치 합니다.
- 하지 하지 관심의 셀 라인에 표현 되는 유전자의 발기인과 같은 실험 조건에 따라 변경 하는 것으로 예상 하나 이상 부정적인 제어 영역을 식별 합니다. 이 지역에 대 한 DNA 순서를 얻을 FASTA 형태로 텍스트 파일에 추가 합니다.
참고: 우리는 가이드 우리가 타겟팅 하는 모든 지역에 대 한 부정적인 컨트롤로 IL1RN 발기인25 를 대상으로 하는 RNAs를 사용 합니다. 사용자가 또한 부정적인 컨트롤로 전사 인자 바인딩 사이트를 포함 하지 않는 관심 영역 근처 intergenic 시퀀스를 선택할 수 있습니다. 그러나, 여러 loci 동시에 타겟이 되고있다, 만약 하나의 부정적인 제어 영역 실험 설계 및 결과의 해석을 간소화 합니다. 타겟된 증강 intronic 이면 대상 추가 부정적인 컨트롤로, dCas9 퓨전으로 상 상속 규정 요소를 포함 하지 않는 같은 로커 스에서 intronic 지역 전사 방해할 수 유용할 수 있습니다. - 긍정적인 통제 지역, 관심, 규제 영역의 상 상속 목표는 발기인 등 높은 관심의 셀 라인에서 복사할 유전자의 발기인을 식별 합니다. 이 지역에 대 한 DNA 순서를 얻을 FASTA 형식의 텍스트 파일에 추가 합니다.
- E-선명26 (http://www.e-crisp.org/E-CRISP/)와 같은 프로그램을 사용 하 여 찾으려고 가이드 RNAs 낮은 떨어져 목표 (이상적으로 0-3)와 생성 된 DNA 순서에. Protospacer 인접 한 모티브 (PAM), S. pyogenes에서 dCas9에 대 한 폼 "NGG" 소요의 20 뉴클레오티드 상류 가이드 RNAs에 의하여 이루어져 있다.
- E-선명 웹사이트에서 드롭-다운 메뉴를 사용 하 여 관심사의 유기 체를 선택 합니다. 게놈 어셈블리 종 이름 오른쪽에 나타납니다.
- 입력은 FASTA 시퀀스 라디오 버튼을 선택 합니다. 위에서 FASTA 시퀀스를 복사 하 고 대화 상자에 붙여 넣어. FASTA 헤더 각 시퀀스에 포함 되어 있는지 확인 합니다.
참고: 최대 50 시퀀스 쿼리할 수 있습니다 동시에. - 드롭 다운 메뉴에서 단일 디자인과 매체 라디오 버튼을 선택 합니다.
- SgRNA 검색 시작버튼을 클릭 합니다. 새로운 브라우저 소프트웨어 탭, 열리고 결과가 표시 됩니다. 함께 모든 쿼리 시퀀스에 대 한 서식이 지정 된 Excel 테이블 형식 보고서를 다운로드버튼을 클릭 하 여 후보 시퀀스를 다운로드 합니다.
- Excel 또는 텍스트 편집 프로그램을 사용 하 여 테이블 형식 보고서를 엽니다.
- UCSC 게놈 브라우저 게놈을 BLAT 후보 전체 길이 gRNA 시퀀스 (23 basepairs)를 사용 합니다.
- 브라우저에서 UCSC 게놈 브라우저 웹사이트 (http://genome.ucsc.edu)으로 이동. 우리의 도구섹션에서 공격기 단어 찾아서 그것을 클릭 합니다. BLAT 검색 도구가 열립니다.
- BLAT 검색 게놈 텍스트 아래에 있는 드롭-다운 메뉴를 사용 하 여 관심의 유기 체와 게놈 어셈블리 선택.
- E-선명 하 여 생성 된 테이블 형식 보고서에서 가이드 RNA 시퀀스를 복사 및 대화 상자에 붙여 넣어. 각 시퀀스는 독특한 FASTA 헤더, 제출 대화 상자 아래쪽에 클릭 있는지 확인 하십시오.
참고: 25 시퀀스까지 수 검사 한 번에. BLAT 검색 결과 페이지에서 정렬을 나타내는 각 줄과 각 가이드 RNA 시퀀스의 정렬 표시 됩니다. 이상적으로, 각 가이드는 가이드 RNA의 고유성을 나타내는 RNA에 대 한 하나의 정렬 해야 합니다. - 가능 하면 게놈에서 여러 위치에 정렬 가이드를 하지 마십시오.
- 가이드 RNA 지역화 및 관심의 영역 내에서 배포 검사, 쿼리 가이드 RNAs 중 하나에 대 한 작업 섹션에서 브라우저 링크를 클릭 합니다. 게놈 브라우저 표시 됩니다 및 선택된 가이드 RNA에 중심 것입니다. 페이지의 상단에 축소 버튼을 사용 하 여 시각화 RNAs e-선명 관심 영역 내에서 식별 하는 다른 가이드의 배포.
- 4 가급적 이면 겹치지 가이드 RNA 순서 (그림 2B)의 지역에 걸쳐 배포 되는 선택 합니다. 관심 영역 600을 초과 하는 경우 혈압, 1-2 추가 가이드를 추가 하십시오. 가이드를 RNAs homopolymeric 뻗 기 및 극단적인 GC 콘텐츠,이 기능 가이드 RNA 복제 프로세스를 방해 하 고 효율성을 대상으로 하는 가이드 RNA를 줄일 수 하지 마십시오.
- 일단 가이드를 선택 각 원하는 가이드에 대 한 전체 가이드 RNA 순서 (23의 뉴클레오티드)를 포함 하는 파일 만들고 3' 끝에서 PAM (NGG)로 서 5' 뉴클레오티드를 제거 합니다. 이 단계는 oligo 주문 수 있습니다.
- Oligonucleotide 시퀀스의 5' 끝에 다음을 추가: GTGGAAAGGACGAAACACCG.
- 3' oligonucleotide 시퀀스의 끝에 다음 추가: GTTTTAGAGCTAGAAATAGC.
참고: 마지막 시퀀스 해야 59 뉴클레오티드 오랫동안 봐이 같은: GTGGAAAGGACGAAACACCG 대상 (19 nt)-GTTTTAGAGCTAGAAATAGC. - 각 규정 요소 증강 i 타겟이 될 그것을 위한 적어도 4 독특한 oligonucleotides 있는지 확인 하십시오. 표 1에 나열 된 "U6_internal" 뇌관 함께 이러한 시퀀스를 주문.
3. 가이드 RNA 복제
참고: 벡터 유일한 컨트롤에 몇 가지 경우 식민지와 식민지 접시, 당 수백 저조한 우리의 손에 매우 효율적으로 입증 깁슨 어셈블리를 통해 가이드 RNA 복제. 이러한 효율성은 풀링된 복제 하는 동안 복잡성을 유지 중요 합니다. 깁슨 어셈블리 복제의 또 다른 장점은 사용자 제한 효소 절단 가이드 RNA U6 클로닝 벡터에 삽입 하려는 사이트의 존재에 대해 걱정할 필요가 없습니다. 그럼에도 불구 하 고,이 프로토콜은 전통적인 제한 효소 기반으로 원하는 경우 복제에 대 한 적용할 수 있습니다.
- (RNase 무료, DNase 무료) 초순에 100 μ M의 최종 농도에 가이드 RNA oligos를 다시 구성. 적어도 4 별도 가이드 RNA oligos 관심의 각 지역에 대 한 있어야 합니다.
- 관심의 각 규제 지역에 대 한 관심의 영역에 해당 하는 모든 oligos의 풀을 만듭니다. Eppendorf 관에서 각 지역에 대 한 각 개별 재구성된 가이드 RNA 올리고의 5 μ를 결합 한다. 수영장 vortexing, 잘 혼합 후 1 μ를 제거 하 고 초순에이 aliquot 1: 200을 희석.
참고: 원하는 경우 개별 규제 지역 타겟팅이 풀 수 더 결합 될 여러 영역을 대상으로 복잡 한 풀을 생성 하. 50 규제 지역 최대 수 있습니다 대상 동시에 단일 풀 (그림 3C). - 깁슨 조립 전에 oligos에 상 동 영역을 연결할 U6 뇌관으로 짧은 PCR을 수행 합니다. 약 40 기지 각 올리고, 저조한 양쪽 끝에 U6 벡터에 충분 한 상 동에 있는 ~ 100 bp 제품에 추가 됩니다.
- 각 가이드 RNA 수영장, 20 μ PCR 높은 중 합 효소 마스터 믹스와 다음 구성 요소를 설정: 1 μ 단계 3.2, U6 앞으로 뇌관 (10 μ M), U6의 1 μ의 1 μ에서에서 희석된 올리고 풀의 반전 뇌관 (10 μ M) 최대 20 μ를 물.
- 다음 조건 열 cycler에서 품 어: 30 98 ° C의 10의 주기 (98 ° C 10 s, 55 ° C 30에 대 한 s, 2 분 동안 72 ° C), 72 ° C 5 분 및 4 ° c.에서 개최
- 낮은 분자량 사다리와 함께 1-2 %agarose 젤에 반응의 5 μ를 실행 합니다. 최종 제품 ~ 100 basepairs (그림 2C) 해야 합니다.
- 열 기반 DNA 정제 키트, 확장 반응을 청소 하 고 차입 버퍼는 키트에 제공 된의 20 μ에 elute.
참고: 짧은 제품으로, 사용 하지 않도록 비드 기반 청소-ups, 100 보다 작은 파편을 제외 하도록 설계 된 bp. - Fluorometer 또는 분 광 광도 계를 사용 하 여 순화 된 DNA를 계량 (예상 수확량은 10-20 ng/μ). 가이드 RNA 삽입-20 ° C에서 저장 될 수 있다 또는 선형화 U6 벡터와 깁슨 어셈블리에서 즉시 사용할 수 있습니다.
- U6 클로닝 벡터 깁슨 어셈블리에 대 한 받는 사람을 준비 하는 금지 효소 다이제스트를 설정 합니다. 많은 깁슨 어셈블리 반응을 수행할 수 있다면, 컷된 벡터의 충분 한 수익률을 보장 하기 위해 여러 요약을 설정 합니다.
- AflII 효소의 20 단위 및 플라스 미드의 1 μ g를 사용 하 여 적절 한 제한 효소 버퍼 20 μ 반응에서. 1-2 h 37 ° C에서 품 어.
- 구슬 또는 열 기반 DNA 정화 킷 다이제스트를 청소 하 고 차입 버퍼의 20 μ에 elute. Fluorometer 또는 분 광 광도 계를 사용 하 여 순화 된 DNA를 계량. 샘플 나중 사용을 위해-20 ° C에서 동결 될 수 있습니다.
- 준비 된 벡터에 깁슨 어셈블리 수행을 삽입 합니다.
- 얼음에 깁슨 어셈블리 반응 설정. 벡터 및 7의 사용 50 ng ng 20 μ 반응에 삽입의. Pipetting 촉진을 초순에 삽입 1시 10분 희석. 벡터만 깁슨 어셈블리 반응, 50를 사용 하 여 설정 물으로 삽입을 대체 하는 벡터의 ng.
- 깁슨 어셈블리 반응 뒤에 4 ° c.에 보류 하는 50 ° C에서 15 분 동안 품 어
- 얼음 조립된 제품을 전송 합니다. 조립된 제품 1:4 초순 얼음에 희석. 예를 들어 초순의 15 μ를 깁슨 어셈블리 제품의 5 μ를 추가 합니다.
- 변환 희석된 깁슨 어셈블리 제품.
- 얼음에 고효율 유능한 세포를 해 동 하 고 각 변화에 대 한 25 μ aliquots. 여러 사이트를 대상으로 하는 복잡 한 수영장을 원하는 경우 같은 복잡 한 gRNA 풀의 여러 독립적인 변환을 수행 하는 다른 튜브에 충분 한 세포를 녹여.
- 각 희석된 제품에 대 한이 희석의 1 μ 1.7 mL Eppendorf 관 유능한 세포의 25 μ를 포함에 추가 합니다. 짧게 튜브를 터치 하 여 혼합. 30 분 동안 얼음에 튜브를 품 어.
- 열 충격 30 셀 42 ° c, s 다음 2 분 동안 얼음에 즉시 전송.
- 300 μ SOC 미디어 (2 %tryptone, 0.5% 효 모 추출 물, 10 m m NaCl, 2.5 m m KCl, 10mm MgCl2, 10 m m MgSO4, 및 20 m m 포도 당)을 추가 하 고 셀 떨고 (300 rpm) 37 ° C에서 1 시간에 대 한 복구를 하자. 이 시간 동안, 암 피 실린/carbenicillin 인큐베이터에서 37 ° C를 가진 한 천 배지 따뜻한. 한 접시를 사용 하 여 각 변환에 대 한.
- 셀의 50 μ 접시와 격판덮개 37 ° C 배양 기에서 밤새 껏 두기 위하여. 개별 사이트를 대상으로 하는 풀에 대 한 포함 하는 암 피 실린/carbenicillin (1 mg/mL) 파운드 국물 (자료 테이블)의 3-5 mL에 직접 놓고 250 rpm minipreps 37 ° C에서 흔들어와 밤새 품 어.
- 세포를 수확 하 고 DNA를 분리.
- 큰 가이드 RNA 라이브러리 여러 사이트를 대상으로 한 maxiprep (150 mL 액체 문화)으로 각 개별 접시에서 모든 식민지를 수집 플레이트 스 크레이 퍼를 사용 합니다. 이 50 mL 팔 콘 튜브 및 튜브에는 식민지를 근 근이 적절 한 항생제와 파운드의 붓는 ~ 5 mL에 의해 촉진 수 있습니다. 개별 사이트를 대상으로 하는 라이브러리에 대 한 miniprep (3-5 mL 액체 문화)에 번호판을 다쳤어요.
- 37 ° C에서 3-5 h 250 rpm에서 진동에 대 한 적절 한 항생제와 함께 이러한 문화를 품 어.
- DNA 추출도 무료 preps에서 키트를 사용 하 여 수행 합니다.
- Fluorometer 또는 분 광 광도 계를 사용 하 여 DNA를 계량. 플라스 미드 transfection에 즉시 사용 하거나 나중에 사용-20 ° C에서 저장 될 수 있습니다.
- 생어를 사용 하는 단일 사이트를 대상으로 하는 작은 풀 가이드 RNA 순서 U6 벡터 내에서 존재를 확인 하려면 "U6_PCR_R" 뇌관 준비 miniprep에 시퀀싱 표 1에 나와 있습니다. 가이드 RNAs의 풀링 때문 19 basepair gRNA 대상 시퀀스 혼합된 기지를 얻을 것입니다 하지만 U6 발기인 및 가이드 RNA 비 계가이 시퀀스를 둘러싼 그대로 이어야 한다.
4입니다. transfection 증강 인자-i의
참고: 이시카와 셀에 증강-i를 사용 하 여 에스트로겐 응답의 성공적인 봉쇄에 대 한 그것은 페 놀 레드에서 그들을 유지 하 여 transfection 5-7 일전 10% 무료 RPMI 숯-박탈 FBS와 1%에 대 한 에스트로겐의 세포를 박탈 하는 데 필요한 페니실린/스입니다. 셀 해야 될 교양이 미디어에 transfection 전후 동안 에스트로겐 응답을 차단 하려는 경우. 완전 한 페 놀 레드 셀의 무료 RPMI 페 놀 레드 무료 trypsin의 사용을 권장 합니다.
- Transfection, 전날 30-50 %confluency (이시카와 셀 잘 당 ~ 60000 셀)에서 24-잘 접시에 셀 (야생-타입 또는 안정적으로 표현 하는 SID4X-dCas9-리아) 접시. Transfections 복제, 수행할 수 있습니다 및 제어 가이드 RNAs 페 하 웰 스를 포함 되도록 충분 한 세포를 플레이트. 세포 단계 1.1.7 에서처럼 셀 도금 후 접시를 부드럽게 흔들어 잘 걸쳐 고르게 배포 됩니다 확인 하십시오.
참고:이 프로토콜 양이온 liposome에 기초를 둔 transfection 시 약의 사용을 가정합니다. Electroporation 이러한 시 약에 매우 민감한 종류는 대체 방법을 제공 합니다. Transfection 조건 증강-i 실험을 시도 하기 전에 관심의 셀 라인에 대 한 낙관 되어야 한다. - 다음 날, 선택의 transfection 시의 지침에 따라 transfections을 준비 합니다. 이시카와 셀, 24-잘 접시의 각 음에 대 한 총 플라스 미드의 사용 550 ng. 혈 청 무료 미디어 (1.1 μ g의 DNA에 transfecting 2 웰 스에 대 한 전체 볼륨의 52 μ)에서 0.020 μ g/μ의 최종 농도에 플라스 미드를 희석. Transfection 시 약의 3 μ를 사용 하 여 DNA의 모든 1 μ g에 대 한 소용돌이 1.2 단계에서 설명한 대로 품 어. 각 우물에 최종 혼합물의 25 μ를 추가 합니다.
참고: 사이트의 대상 조합에 무게를 사용 동일한 플라스 미드의 각 개별 사이트에 대 한 하 고 컨트롤 플라스 미드 (빈 가이드 RNA 클로닝 벡터 또는 가이드 IL1RN 같은 부정적인 제어 영역을 대상으로 하는 RNAs 남은 무게를 채울 발기인)입니다. 변이 transfections에 대 한 3:2 Cas9 퓨전: 가이드 RNA 플라스 미드의 비율을 사용 합니다. 형광 기자를 포함 하는 플라스 미드 transfection 효율 모니터링을 추가할 수 있습니다. - 36 h 게시물 transfection에 변경 페 놀 레드를 사용 하 여 미디어 RPMI 10% 숯 박탈 FBS와 1% 페니실린/스 (이시카와 셀)에 대 한 무료 및 puromycin 공급 (최종 농도: 1 μ g/mL)과 네오 마이 신 (최종 농도: 300 ng/mL). 세포 transfection 시 약에 민감한 경우, 미디어는 앞으로 바뀔 수 있다 하지만 항생제를 24 h 게시물 transfection 보다 일찍 추가 해야 합니다.
참고: 셀을 수확 하기 전에 항생제를 추가한 후 적어도 24 시간을 기다립니다. 48 h 일찍 transfection를 게시 하 고 최대 5 일 게시 transfection 식 변경 때문에 증강-i를 감지할 수 있습니다. 작업 하는 경우 에스트로겐의 박탈 하는 이시카와 셀 8 h 10 nM 17β-estradiol (e 2) 유도 후 항생제 치료를 수행 하 고 즉시 세포를 수확.
5. 세포 수확 및 RNA 추출
- 1% β-mercaptoethanol (공학은) 세포의 용 해 버퍼를 준비 합니다. 충분 한 세포 공학은 혼합물 (300 μ 각 잘 수확 되 고에 대 한) 인지 확인 합니다.
- 진공 흡 인기를 사용 하 여 미디어 발음
- 1 x PBS의 동등한 양 가진 한 번 세포 세척 (500 μ)와 가능한 많은 PBS를 제거 하는 aspirate.
- 멀티 채널 피 펫을 사용 하 여 각 잘을 300 μ의 세포 공학은 솔루션을 추가 합니다. 세포의 용 해 솔루션을 플라스틱 그리고 8-아래로 10 번, 그리고 깊은 잘 격판덮개 또는 1.7 mL Eppendorf에 얼음에 관. RNA는 즉시, 추출 될 수 있다 또는 lysates 미래 처리를 위한-80 ° C에 얼 수 있다.
- Lysates에서 RNA를 추출, DNase 치료를 포함 하는 상업적으로 사용할 수 있는 키트를 사용 합니다. 초순의 최소 권장된 볼륨에 elute (RNase 무료, DNase 무료) 또는 차입 버퍼 및 RNA를 정량. 샘플의 작은 숫자, fluorometer 또는 분 광 광도 계를 사용 하 여. 샘플의 많은 수를 감지 하는 RNA 측정 플레이트 리더에 형광 프로브를 사용 하 여. 샘플은 정량화 전후-80 ° C에서 동결 될 수 있습니다.
6. 측정 단계 정량 및 RNA-seq를 사용 하 여 유전자 표현 변경
- 정량 Pcr 뇌관의 유전자 및 하나 이상의 내부 관리 유전자는 타겟된 유전자의 수준에 가까운 표현 하 고 실험 조건에서 변경 되지 않습니다 얻을. 이상적으로, 이러한 뇌관을 genomic DNA의 증폭을 피하기 위해 엑손-엑손 접합을 스팬 됩니다.
- RNA의 셀 라인에서 얻은에이 뇌관을 테스트 합니다. 사용 녹아 곡선 분석을 단일 제품의 생산을 확인. 단일 제품 제작 하지 않은 경우 추가 뇌관 쌍을 테스트 합니다.
- 샘플에 대 한 각 증강-i 및 제어 가이드 취급 하는 RNA, 그 샘플에 얼마나 많은 유전자를 분석 해야 합니다 식별 합니다. 유전자의이 세트는 CTCF 등 GAPDH하우스키핑 유전자를 포함 해야 합니다.
- 정량 Pcr 반응을 설정 합니다.
- 물에 동일한 농도에 모든 샘플을 희석 그런 50 총 RNA의 ng pipetted 쉽게 그리고 충분히 각 반응에 대 한 RNA를 희석. 예를 들어 ~16.6 ng/μ에 RNA를 희석 하 고 사용 하는 각 반응에서 RNA의 3 μ. 믹스 마스터를 설정 하는 동안 얼음에 RNA를 유지.
- 상용 1 단계 정량 키트를 사용 하 여 측정 각 유전자에 대 한 별도 마스터 믹스를 준비 합니다. 각 뇌관 (10 μ M 재고 솔루션)의 1 μ와 20 μ 반응 볼륨을 사용 합니다. 얼음에 이러한 반응을 설정 합니다.
- 적합 한 열 cycler는 반응 접시에서 RNA 샘플 뒤에 마스터 믹스를 추가 합니다. 플레이트 마감재와 함께 밀봉 하 고 vortexing 또는 pipetting으로 부드럽게 혼합. 짧게 접시 원심 (140 x 60에 대 한 g s) 우물의 바닥에는 그 액체를 위해.
- 다음과 같이 열 cycler에 접시를 품 어 (또는 키트로 지시): 30 분, 10 분 동안 95 ° C 48 ° C 40의 사이클 (15 95 ° C s, 1 분 동안 60 ° C).
- 각 샘플 내에서 측정 된 각 유전자에 대 한 Ct 값을 가져옵니다. 유전자 발현에 변화를 확인 하는 비교 Ct 메서드를 사용 합니다.
- 정규화 된 Ct 값을 생성 하는 각 샘플에 대 한 관심의 각 유전자의 Ct에서 하우스키핑 유전자의 Ct를 뺍니다.
- 치료 컨트롤 샘플에 대 한 각 유전자에 대 한 정규화 된 Ct 값의 평균 걸릴. 로그 기본 2 규모 배 억압 다음 해당 유전자에 대 한 처리 컨트롤 샘플에 대 한 동일한 값에서 각 유전자에 대 한 정규화 된 증강 인자-i 치료 샘플 Ct를 빼서 계산할 수 있습니다.
- 증강 인자-i 치료 따르는 유전자 발현에서 세계적인 변화를 확인 하려면 사용자의 시퀀싱 기술과 호환 상용 키트를 사용 하 여 RNA 시퀀싱에 대 한 샘플을 준비 합니다. 시작 물자에 대 한 RNA의 사용 ~ 500 ng 이상 2 생물 복제에 대 한 라이브러리를 준비 하는 고.
7. SID4X-dCas9-리아를 사용 하 여 칩 seq 하 여 타겟팅 특정 게놈의 확인
참고: SID4X-dCas9-리아 융해 단백질 플래그 피토 프 태그와 하 피토 프 태그를 모두 포함 하지만 칩 seq에 대 한 최상의 결과 반대로 플래그 항 체와 가져온. 원하는 경우, 사용자 증강-i에 의해 영향을 받을 녹음 방송 요인 또는 H3K27ac, 증강 활동 증강-i에 의해 감소의 마크에 대 한 추가 칩 seq 실험을 수행할 수 있습니다. 그러나, 각 칩 seq 실험 10 x 106 세포, 그래서 계획에 따라 필요 합니다.
- 증강 인자-i 풀 셀 transfect
- 15 cm 조직 문화 접시에 접시 10 x 106 셀. 각 접시의 1 인자에 대 한 1 칩 seq 실험을 나타냅니다.
- 다음 날, transfect 접시 당 총 DNA의 20 μ g을 사용 하 여 셀. Transfections SID4X-dCas9-리아를 안정적으로 표현 하는 셀 라인에,에 대 한 DNA 가이드 관심의 모든 사이트를 대상으로 하는 RNAs의 플라스 미드 풀 그리고 선택적으로 플라스 미드 형광 단백질을 표현 한다. 변이 transfections에 대 한 3:2 dCas9 융합 단백질: 가이드 RNA 풀의 비율을 사용 합니다. 다른 TFs 또는 히스톤 수정 칩 seq, 다른 접시에 하나 이상 추가 제어 가이드 RNA transfection를 수행 합니다.
- 네오 마이 신 (300 ng/mL) 24-48 h 게시물 transfection에 puromycin (1 μ g/mL)와 요리를 취급 합니다. Chromatin 수확 하기 전에 적어도 24 시간을 기다립니다.
- 요리에서 chromatin 수확.
참고: 이시카와 셀에 응급실 게놈 바인딩에 증강 인자-i의 효과 연구, 요리에 1 h 10 nM E2 치료를 수행과 제어 가이드 RNAs 증강-i 전에 수확 페. H3K27ac에 증강 인자-i의 효과 연구, 요리에 8 h 10 nM E2 유도 수행과 제어 가이드 RNAs 증강-i 전에 수확 페.- 각 접시 (1%의 최종 농도)을 37% 포름알데히드의 500 μ를 적용 합니다. 짧게 판을 소용돌이 친다. 10 분 동안 실내 온도에 앉아 접시를 보자.
- 2.5 M 글리신 (125 m m의 최종 농도)의 1 mL를 추가 합니다. 짧게 판을 소용돌이 친다.
- 포름알데히드와 글리신 미디어에서 부 어. 감기 1 x PBS의 동일한 볼륨 (~ 20 mL)를 추가 합니다.
- PBS에서 부 어. 가능한 많은 PBS를 제거 하는 진공 흡 인기와 발음. 얼음에 요리를 놓습니다.
- 3-5 mL PBS 또는 Farnham 세포의 용 해 버퍼 x 감기 1의 추가 (5 m m 파이프 pH 8.0, 85 m m KCl, 0.5 %NP-40) protease 억제제 (그냥 사용 하기 전에 추가) 각 접시에 x 1. 요리 판 스 크레이 퍼로 긁어와 얼음에 15 mL 원뿔 튜브에 솔루션을 전송.
- Chromatin 1000 x g. 폐기에 4 ° C에서 5 분 동안 원심 분리기에 튜브 아래로 회전 하 여 작은 공의 상쾌한 고 차후 사용을 위해-80 ° C에서 펠 릿 저장 하거나 반대로 플래그 항 체 또는 다른 타겟팅 하는 항 체를 사용 하 여 선택의 칩 seq 프로토콜 진행 녹음 방송 요인 또는 관심 (H3K27ac, H3K9me3)의 히스톤 수정.
결과
그림 1 프로토콜에서 설명 하는 워크플로의 회로도 보여준다. 근처에 스 트로 겐 통제 유전자 MMP17, 칩 seq (그림 2A)에 의해 정의 된 인근 3 바인딩 사이트가 어 바운드 강화 기여 가이드 RNAs를 확인 하려면 각 지역에 대 한 설계 되었습니다. 디자인 가이드 RNAs, 시퀀스를 둘러싼 각 응급실의 600-900 bp 창 관심의 바인딩 사이트 선택 하 고 가이드 RNA 디자인 프로그램에 넣어 했다. 가이드 인 RNA 0-2 사이트는 BLAT을 사용 하 여 인간 게놈에 정렬 했다 목표 예측 시퀀스. 및 칩 seq에 의해 정의 된 영역을 스팬 RNAs 가이드 4 겹치지 DNaseI 과민 증 (그림 2B)를 대상으로 선정 됐다. 추가 시퀀스 (표 1) 다운스트림 복제 및 결과 59 뉴클레오티드 조각을 주문 했다 각 끝에 추가 되었습니다. 도착 후, 가이드 RNAs 희석 되었고 사이트, 그리고 짧은 PCR에 의해 풀링된 깁슨 조립 전에 상 동 영역을 추가 하려면 수행 되었다. 그림 2C 59 basepair의 각 끝에 시퀀스의 20 basepairs를 추가할 것입니다 "U6_internal" 뇌관 (표 1)를 사용 하 여 짧은 PCR 후 예상된 가이드 RNA 제품 가이드 RNA 조각, 100 ~ basepair 시퀀스에 결과 보여줍니다. 깁슨 어셈블리에 따라 이러한 가이드 RNA 풀 박테리아로 변형 되었다 그리고 플라스 미드 minipreps 다음 날을 준비 했다. 그림 2D MMP17 근처 여러 강화 혼자 표적으로 증강 해 부 실험, 및 조합 증강-i를 사용 하 여 결과 보여줍니다. 증강-i 대상 사이트는 검은 육각형으로 표시 됩니다. 표시 된 사이트를 대상으로 하는 가이드 RNA 플라스 미드를 박탈 하는 에스트로겐 이시카와 셀 라인으로 안정적으로 표현 하는 SID4X-dCas9-리아 페 했다. 2 일 후, 미디어 변경 되었습니다과 puromycin transfected 세포에 대 한 풍부 하 게 추가 되었습니다. 다음 날, 셀 8 h 10 nM estradiol 치료 따르는 것을 수확 했다. RNA 격리, 고 1 단계 정량 수행 되었다. 이 예제에서 사이트 1과 2는 사이트 3 (그림 2D, 레인 ii-4)이이 조건 하에서 기여 하지 않는 하는 동안 MMP17의 완전 한 estrogenic 응답 필요 하다. 때만 2 또는 3 사이트는 활성 (vi와 vii), 에스트로겐 응답은 때 없는 사이트는 유사한 활성 (viii), 제안 하는이 사이트를 독립적으로 기여할 수 없습니다. 사이트 1 자체 (v), 일부 식을 기여할 수 있지만 가장 큰 활동은 볼 때 사이트 1 및 2 활성 (iv).
4 다른 유전자 근처 10 강화를 동시에 조작 하기 (그림 3A)의 복잡 한 풀 가이드 RNAs 42 증강 가이드 및 16 발기인 가이드 생성 된. 가이드 RNA oligos 초기 가이드 RNA 확장 PCR (단계 3.3), 전에 풀링된 했다 그리고 결과 PCR 제품 정화 깁슨 어셈블리를 사용 하 여 빈 puromycin U6 클로닝 벡터와 결합 했다. 깁슨 어셈블리에 따라 여러 개의 독립적인 변환은 수행 하 고 도금 했다. 플레이트는 파운드에 긁힌 것 이었고 2-4 h maxiprep 이전에 대 한 밖으로 성장할 수 있습니다. 그림 3B 표시 대표 감소 유전자 발현에 정량 하 여 때 이러한 가이드 RNA 풀을 한 스 트로 겐 박탈 이시카와 셀 라인으로 안정적으로 표현 하는 SID4X-dCas9-리아 페 (그림 2D) 위에서 설명한 대로 처리 됩니다. 증강 인자-i에서 감소는 상 상속 대상 유전자의 발기인을 대상으로 얻은 그 비슷합니다. 그림 3C RNAs 증강-i를 사용 하 여 에스트로겐 응답의 감소에 가이드의 희석의 효과 보여줍니다. 1:50 G0S2 아직도 근처 증강을 대상으로 가이드 RNA 수영장의 희석 생성 증강-i 50까지를 대상으로 사용할 수 있습니다 제안 하는 유전자 발현에 있는 뜻깊은 감소는 한 번에 사이트. 그러나, 비활성화 사이트의 수백 더 민감한 탐지 방법을 채용 하지 않는 한 동시에 대상 수 없습니다 것을 나타내는, 희석 수 있습니다.

그림 1입니다. 프로토콜 증강-나를 사용 하 여 다중 증강 해 부에 대 한 도식 가이드 RNAs (빨간색과 파란색)은 e-선명 및 UCSC 게놈 브라우저를 사용 하 여 선택한 사용 하 여 설계 되었습니다. 4 명의 가이드 RNAs는 관심 (전사 인자 바인딩 사이트 칩 seq에 의해 정의 된 대로) 영역에 걸쳐 선택 됩니다. (빨간색과 파란색)의 지역별 풀링된 가이드 RNA oligonucleotides 깁슨 어셈블리 및 변환 전에 상 동 영역 (주황색)을 추가 하는 PCR를 받 다. 안정적으로 표현 하는 SID4X-dCas9-리아 셀 라인 또는 SID4X-dCas9-리아 플라스 미드와 함께에서 야생-타입 셀 결과 플라스 미드 풀 lipofection을 통해 페는. 가이드 RNA 플라스 미드 풀은 한 번에 또는 동시에 여러 사이트를 대상으로 한 사이트를 대상으로 개별적으로 페 수 있습니다. Transfected 세포 가이드 RNAs를 포함 하는 셀에 대 한 풍부 하는 항생제로 처리 됩니다. 에 ~ 72 h 게시물 transfection, 세포 수확. 핵 산 정량, RNA-seq, 또는 칩이 추출 될 수 있다 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
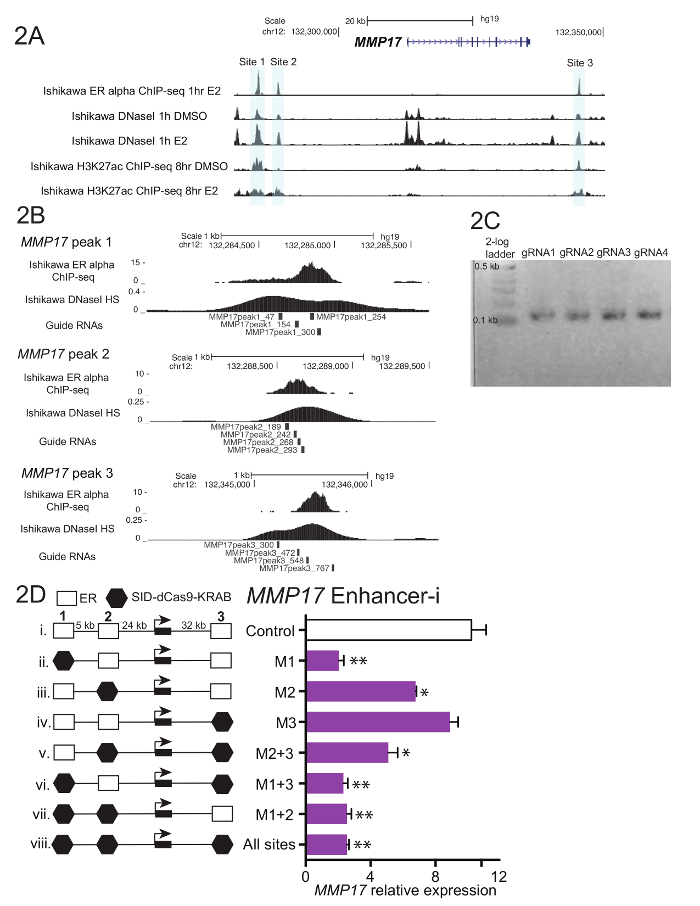
그림 2입니다. MMP17를 위한 RNA 설계 및 증강 해 부 안내. 근처 타겟 응급실 알파 바인딩된 강화 (회색)의 (A) 게놈 브라우저 화면 MMP17. 이 그림에서 칼튼, 외. 수정 되었습니다. 18. (B) 가이드 RNA 바인딩 사이트183에 대 한 설계. ER 칩 seq에 의해 정의 된에 대 한 바인딩 사이트는이 지역에 걸쳐 대상과 4 가이드 RNAs 타일입니다. 바인딩 사이트에 걸쳐 있는 DNaseI 감도 신호 가이드 RNA 디자인에 대 한 대상 시퀀스를 정의 하기 위해 사용할 수 있습니다. 칩 seq와 DNaseI HS 데이터 1 헤 (C) 대표 가이드 RNA 시퀀스 깁슨 어셈블리, 상 동 영역을 추가 하는 짧은 PCR을 받은 것에 대 한 준비가 된 10 nM estradiol로 치료 시 세포에서 얻은 했다. (D) MMP17 의 상대 식 정량 특정 지역 증강-i와 8-h 10 nM estradiol 치료의 대상으로 다음을 통해 측정. 식 세포 estradiol 치료 하지 않을 MMP17 의 CTCF 및 식 수준에 상대적입니다. 제어 가이드 RNAs의 발기인 대상 IL1RN. 모든 오차 막대를 나타내는 SEM, 이중 별표 나타냅니다 p < 0.01 및 단일 별표 나타냅니다 p < 쌍된 t-검정에서 0.05. 이 그림에서 칼튼, 수정 된 외. 18. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
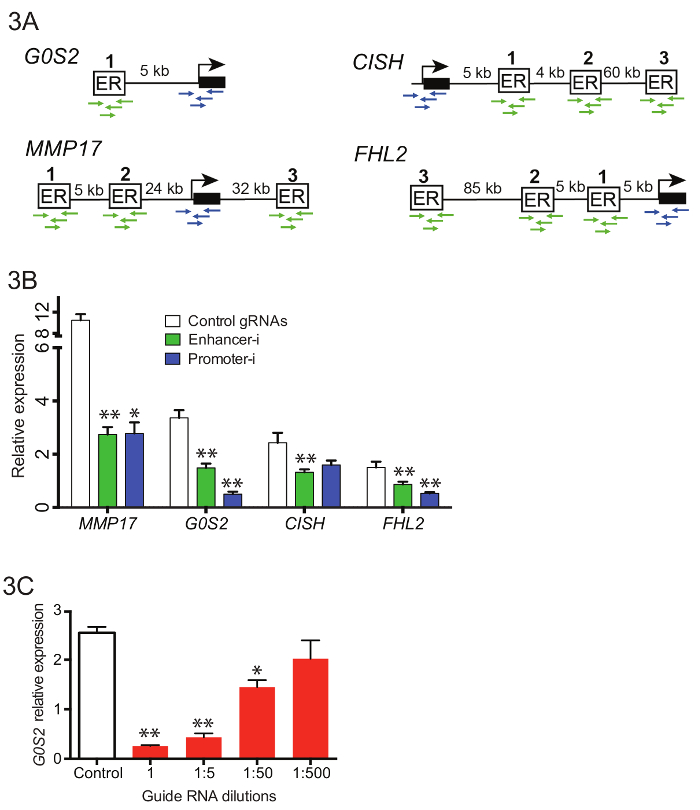
그림 3입니다. 증강-i. 풀링 대상 근처에 다른 유전자를 동시에 여러 강화 바인딩 사이트와 풀링된 증강-i에 타겟이 될 발기인의 (A) 회로도 (B) 이시카와 셀에 E2 치료 증강-i 플라스 미드 풀 (녹색), (파란색) 발기인-i 플라스 미드 풀 또는 제어 gRNAs (화이트)18페 후 정량 Pcr로 측정 된 식에 효과. 모든 유전자에서 중요 한 감소는 증강 i 관찰 됩니다. 이 그림에서 칼튼, 수정 된 외. 18. (C) 가이드 G0S2를 대상으로 하는 RNAs의 다른 양의 페 이시카와 셀에 G0S2 식 레벨 e 2 치료 후에 효과. 상당한 감소 가이드 RNA의 소량으로도 볼 수 있다 (1시 50분 희석), 제안 하는 최대 50 개 사이트 동시에 대상 수 있습니다. 모든 오차 막대를 나타내는 SEM, 이중 별표 나타냅니다 p < 0.01 및 단일 별표 나타냅니다 p < 쌍된 t-검정에서 0.05. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 이름 | 시퀀스 |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
표 1입니다. 가이드 RNA 확장 및 시퀀싱, 정량, 융해 단백질의 탐지 사용 한 뇌관
토론
이 프로토콜 증강 기능 생 genomic 소재 시에 DNA 순서를 실제로 변경 하지 않고 해 부에 대 한 간단 하 고 유연한 방법을 설명 합니다. 반면 비슷한 개념 dCas9 리아27을 사용 하 여 이전에 게시 CRISPR 간섭 프로토콜, 증강 인자-i는 3 가지 주요 방법으로 이러한 프로토콜에서 다릅니다. 첫째, 증강 인자-i MAD120 증강 비활성화 달성 하기의 SIN3A 상호 작용 도메인을 활용 합니다. 증강 비활성화 HDAC 저 해제, 비활성화 하는 기본 메커니즘 HDAC 의존 하자를 사용 하 여 구출 될 수 있다. CRISPR dCas9-리아와 간섭, 달리 증강-i H3K9me3의 증 착에 연결 되지 않습니다. 사실은 RNAs, 3 일에 수확 되 고 세포 transfection 게시 가이드의 변이 도입에 의존 하는 증강 인자-i이 높습니다. CRISPR 간섭, H3K9me3 증가 7 일 게시물 변환12에서 관찰 됩니다. 마지막으로, 증강 인자-i 프로토콜 여러 사이트를 동시에 대상 및 대상의 효율성을 모니터링할 수 있는 전략을 제공 합니다. 모자이크-seq17, dCas9-리아는 여러 강화를 동시에, 대상 하는 데 사용 됩니다 하지만이 기술은 식 변화를 식별 하는 단일 셀 RNA 시퀀싱에 의존 하 고 (스 트로 겐-응답 유전자) 등 많은 유전자가 들 키 지 않고는 난청 단일 셀 RNA이 증강 인자-i의 감도 개별적으로 강화 연구 조합 어떤 유전자에 대 한 신뢰할 수 있는 방법을 제공 합니다.
증강 인자-i의 가장 중요 한 단계가 이다 transfection, 관심의 셀 라인에 대 한 최적화 되어야 합니다. 이 프로토콜 transfected 세포에 대 한 풍부 하 게 puromycin 치료에 의존 하지만 그 공동 transfecting 가이드 RNAs 형광 단백질 및 형광 셀 cytometry 사용 하 여 정렬 일부 셀에 대 한 더 나은 농축 방법으로 증명할 수 있습니다. 유형입니다. 문제 해결 및 확인 transfection 정량 식 가이드 RNAs의 수준과 SID4x-dCas9-리아 모니터링 하는 것이 좋습니다. 가이드 RNA 레벨은 낮은 경우 (사이클 임계값 > 30), 사용자가 고려할 수 있습니다 또한 대체 가이드 녹음 방송 생체 외에서 28등 RNA 생산 전략. 그것은 또한 가능한 높은 gRNA 수준에도 불구 하 고 가이드 RNA는 SID4x-dCas9-리아 단백질의 대상으로 하는 것은 비효율적, 어떤 경우에 다른 선택 가이드 RNA 시퀀스 할 수 있습니다. 증강 인자-i 치료 세포에서 chromatin와 융합 단백질에 칩 seq를 수행 하 여 대상으로의 효율성을 모니터링할 수 있습니다. SID4x-dCas9-리아, 관심 영역에서의 높은 신호는 그것의 상 상속 대상 유전자 식 변경 감지 하는 경우 다음 가능성이 지역 공부 하는 조건 하에서 그 유전자의 표정에 공헌 하지 않는다.
증강 인자-i의 한 가지 잠재적인 한계는 오프 대상 효과 너무 많은 사이트는 동시에 표적으로 하는 경우 누적 될 수 있습니다. 그럼에도 불구 하 고, 최저 CRISPR 방해 전략 있다 대상 효과 RNAi29, 보다 적은 특히 dCas9 리아 표현 polyclonal 셀 라인을 사용할 때. 우리가 본 SID4X-dCas9-리아의 게놈 바인딩 대상에서 동시에 10 개의 사이트를 대상으로 하는 동안 우리가 하지 그 바인딩 이벤트의 결과로 유전자 식 변경 확인 했습니다. 일부 강화 여러 발기인 또는 다른 강화 연락처 수 있습니다, 비록 유전자 규칙의이 형태는 일반적인 경우에 명확 하지 않다 많은 유전자 단일 증강의 대상으로 시 식에 변경 될 수 있음을 가능 하다. 식 변화는 특정 증강을 대상으로 관찰을 확인 하 고 오프-목표는 아니지만 효과, 사용자가 수행할 수 증강-i 겹치지 가이드 같은 지역 타겟팅 하는 RNAs의 두 가지 세트와 함께. 또한, nuclease 유능한 Cas9를 사용 하 여 영역의 유전자 삭제 추가 유전자 발현에 미치는 영향을 확인할 수 있습니다.
히스톤 deacetylation 통해 증강-i 기능으로 그것은 그것의 비활성화 능력 histone acetylation 감지할 수준을 강화 제한 가능. 타겟팅 특정 강화에 더 효과적일 수 있습니다 대체 억압 융해의 다양 한이 있다. DCas9 DNA methyltransferase 융해 유전자 발현 원심 강화30, 대상으로 하는 경우를 줄이기 위해 사용할 수 있습니다 하지만이 억압은 자주 과도. 또 다른 억압 적인 퓨전 히스톤 H3 리 27 trimethylation 리드 하 고 다양 한 세포 선 및 발기인31dCas9-리아와 비슷한 수준에서 유전자 발현을 누르는 GATA1 친구 (FOG1) 도메인을 사용 합니다. 흥미롭게도, 발기인, SID 도메인의 단일 복사본 증강-i에서 현재 사용 하는 4 복사본 보다 더 증강 비활성화를 제공할 수 있습니다 제안에 억압 적인 잠재력을 감소 dCas9 FOG1의 더 많은 복사본을 추가 합니다. 그것은 일부 loci 위의 dCas9 융해의 다른 조합에 의해 듀얼 타겟팅에서 이점을 얻을 수 있습니다. 예를 들어 안정적인 장기 억압 dCas9 DNMT3a 및 dCas9-리아32동시 변환에 의해 얻을 수 있습니다. 이러한 억압 적인 융해의 대부분만 타깃이 되어 단일 장소에 한 번에, 그리고 그것은 불분명 남아 있는 여러 강화를 동시에 조작에 가장 효과적 이다.
증강 인자-i, 유전자의 소수에 대 한 강화의 조합을 공부에 대 한 적합 한 방법 동안 사용자 putative 강화 유전자의 수백을 공부 하고자 하는 경우 처리량에 여전히 다소 제한 됩니다. 이 기술의 미래 응용 프로그램 이미징 기반 기술을 여러 샘플에서 여러 유전자를 동시에 계량을 통합할 예정 이다. 중요 한 것은, 이러한 기술은 시간이 걸리는 RNA 격리에 대 한 필요성을 제거, lysate에서 RNA 분자의 직접적인 탐지와 호환 됩니다. 이러한 적응 강화의 더 큰 세트의 심문을 촉진 한다.
공개
저자는 공개 없다.
감사의 말
이 작품은 NIH/NHGRI R00 HG006922 및 J.G.에 NIH/NHGRI R01 HG008974 그리고 사냥꾼 Cancer 학회에 의해 지원 되었다. J.B.C.는 유전자 T32GM007464에 NIH 연수 프로그램에 의해 지원 되었다.
자료
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
참고문헌
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유