JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
재구성 가능한 DNA 아코디언 랙의 설계 및 합성
* 이 저자들은 동등하게 기여했습니다
요약
우리는 디자인, 시뮬레이션, 젖은 실험실 실험 및 아코디언 랙 6으로 6 메시의 DNA의 재구성에 대 한 분석에 대 한 상세한 프로토콜을 설명합니다.
초록
DNA nanostructure 기반 기계 시스템 또는 ångström 해상도를 나노미터 2D 및 3D의 복잡 한 나노 모션 생성, DNA 빠져나가기 약물 전달, 분자 원자로 등 나노기술의 다양 한 분야에서 큰 잠재력을 표시 그리고 nanoplasmonic 시스템입니다. 재구성 가능한 DNA 아코디언 랙 수 총칭 조작 요소, 2D 또는 3D 나노 네트워크 응답 DNA 입력으로 여러 단계에서를 설명 합니다. 플랫폼은 DNA 빠져나가기 재구성의 여러 단계와 네트워크 규모에 몇 가지 요소에서 제어할 수 있는 요소 수를 늘릴 가능성이 있다.
이 프로토콜에 우리는 6에 의해 6 메쉬 재구성 가능한 DNA 아코디언 랙 전체 실험 과정을 설명합니다. 프로토콜 구조와 합성 및 재구성에 대 한 젖은 실험실 실험의 설계 규칙 및 시뮬레이션 절차 포함 되어 있습니다. 또한, 가장 (전송 전자 현미경 검사 법)과 무서 워 (형광 공명 에너지 전달)를 사용 하 여 구조 분석 프로토콜에 포함 됩니다. 이 프로토콜에서 설명 소설 설계 및 시뮬레이션 방법을 연구원 추가 응용 프로그램에 대 한 DNA 아코디언 랙을 사용 하 여 도움이 됩니다.
서문
그들은 생산을 나노미터 2D 및 3D의 복잡 한 나노 모션 때문에 DNA nanostructures 또는 DNA 빠져나가기1,2,3,,45 에 따라 기계 시스템 독특합니다. ångström 해상도, 다양 한 바이오 자극2,,36에 따르면. 이러한 구조에 기능성 소재를 연결 하 고 그들의 위치를 제어,이 구조는 다양 한 분야에 적용할 수 있습니다. 예를 들어 DNA 빠져나가기를 분자 원자로7, 마약 배달8및 nanoplasmonic 시스템9,10에 대 한 제안 되었습니다.
이전에 우리는 재구성 가능한 DNA 아코디언 랙 요소11 (그림 1A)의 2D 또는 3D 나노 네트워크를 조작할 수 있는 도입. 단지 몇 가지 요소를 제어 하는 다른 DNA 지지, 달리 플랫폼 공동으로 다양 한 단계에 주기적으로 기준점과 2D 또는 3D 요소 조작할 수 있습니다. 우리는 프로그래밍 가능한 화학 및 생물 반응 네트워크 또는 분자 컴퓨팅 시스템 건축 될 수 있다 우리의 시스템에서 관리할 수 있는 요소의 수를 늘려 예상. DNA 아코디언 랙 여러 DNA 광선의 네트워크가 단일 가닥 DNA (그림 1B)의 구성 된 관절에 연결 되어 있는 구조입니다. 아코디언 랙 DNA 빔에 의해 생성 된 DNA 자물쇠 광선의 스티커 부분에 교배 하 고 브리징 부분의 잠금 (잠금된 상태)의 길이 따라 빔 사이의 각도 변경 하 여 다시 구성 됩니다. 또한, 다단계 재구성은 DNA 가닥 발 기반 변위12,13자물쇠를 분리 하 여 자유 상태의 형성 후 새로운 자물쇠를 추가 하 여 보여 줍니다.
이 프로토콜에서 우리는 재구성 가능한 DNA 아코디언 랙 전체 설계 및 합성 과정을 설명합니다. 프로토콜 설계, 시뮬레이션, 젖은 실험실 실험, 및 6에 의해 6 메시의 DNA 아코디언 랙 및 이러한 재구성의 합성에 대 한 분석을 포함 한다. 프로토콜에서 설명 하는 구조는 이전 연구11 의 기본 모델 이며 65 nm 65 여 14 빔으로 구성 된 크기에 nm. 설계 및 시뮬레이션, 아코디언 랙의 구조 설계는 기존의 DNA 종이 접기14,15 (즉, 단단히 포장)에서 다르다. 따라서, 설계 규칙 및 분자 시뮬레이션 전통적인 방법에서 수정 되었습니다. 설명 하기 위해, 우리는 caDNAno14 의 수정 된 접근 및 아코디언 랙 추가 스크립트 oxDNA16,17 를 사용 하 여 시뮬레이션을 사용 하 여 설계 기법을 보여줍니다. 마지막으로, 구성 된 아코디언 랙 구조 분석에 대 한 가장 및 무서 워 두 프로토콜 설명 되어 있습니다.
프로토콜
1. 6 DNA 아코디언 랙 caDNAno14 여 6의 디자인
- 다운로드 하 고 설치 하는 것은 DNA의 아코디언 랙 디자인 caDNAno 2.0 소프트웨어14 (caDNAno 2.5는 또한 https://github.com/cadnano/cadnano2.5에서 사용할 수). CaDNAno14 을 사각형 격자와 새로운 부품 추가 하려면 사각형 도구 를 클릭 합니다.
- 각 광선의 아코디언 랙 번호 고 caDNAno14 (그림 2)의 왼쪽된 격자 패널에 그립니다.
- 연필 도구를 클릭 하 고 각 빔 caDNAno14에 바로 편집 패널에 그립니다. 휴식 모든 32 광선 bp, 인접 한 광선 사이 관절입니다. 스테이플 크로스 관절와 같은 위치에 배치 합니다. 추가 단일 가닥 크로스는 관절을 caDNAno14 삽입 도구 와 연필 도구 를 사용 합니다.
- 연필 도구 를 클릭 하 고 연결 하는 관절. 각 광선 7 개의 관절 있다.
- 라우팅 알고리즘11이전에 보고 된 비 계를 사용 하 여 단일 루프에는 건설 기계를 병합 하 비 계 크로스 오버를 생성 합니다. 미만 8 비 계 및 스테이플 가닥 사이 최소 바인딩 도메인 못하게 bp (그림 3).
- 그림 3에서 보듯이 아코디언 랙의 반대편에 있는 정점에 있는 어셈블리에서 사용 되지 않는 건설 기계를 놓습니다.
- 휴식 도구를 클릭 합니다. 스테이플 물가 원형 또는 이상-60 가닥 휴식 혈압.
- 디자인 DNA 잠금 가닥.
- 휴식 도구를 클릭 합니다. 스티커 부분을 삭제 8 주식 DNA 영역의 휴식 8 bp 주식 DNA 영역의 혈압. 6 여 6 아코디언 랙에서 18 스티커 부분 (그림 1)가 있다.
- 역방향 시퀀스 스티커 부분에 보완 잠금 가닥의 양쪽 끝에 놓고 원하는 길이 (그림 1B)의 폴 리 T 가닥으로 구성 된 브리징 지역별 그들을 연결 합니다.
- 재구성, 추가 8 bp DNA의 끝 발 시퀀스의 가닥 변위에 대 한 잠급니다. 표 2에 사용 하는 발 순서가입니다.
- 브리징 지역에 무료 리버스는 폴 리 A 가닥을 준비 합니다.
- 디자인 가닥을 반대로 재구성 실험에 대 한 DNA 자물쇠를 보완.
- 시퀀스 도구 를 클릭 하 고 비 계 DNA를 클릭 합니다. 선택 발판으로 표준 M13mp18. 내보내기 도구 를 클릭 그리고 csv 형식 (표 1) 시퀀스를 저장 합니다.
2. 구조는 oxDNA 시뮬레이션
- 다운로드 및 설치 oxDNA16,17. 최신 소스 코드는 https://sourceforge.net/projects/oxdna/files/에 있습니다.
- CaDNAno14 파일 'cadnano_interface.py', oxDNA,1617 패키지에서 제공 되는 파이썬 스크립트를 사용 하 여 시작 구성 파일을 확인 합니다. 사용법은 다음과 같습니다: '파이썬 cadnano_interface.py cadnano_file.json 스퀘어'. 토폴로지 파일 및 구성 파일 이제 생성 됩니다.
참고: 토폴로지 파일 포함 얼마나 많은 가닥 그리고 뉴클레오티드 뉴클레오티드 사이 백본 백본 채권에 관한 정보와 구조에 있습니다. 구성 파일 timestep, 에너지, 및 크기와 같은 일반 정보를 포함합니다. 위치 벡터, 백본 기반 벡터, 법선 벡터, 속도, 뉴클레오티드의 각 속도 등 방향 정보 포함된 (그림 4) 이기도합니다. - 아코디언 랙 진짜 구조 정보를 반영 수 있도록 caDNAno14 에서 토폴로지 및 구성 파일에서 정보를 변경 합니다. 모든 빔 caDNAno14 에서 토폴로지 및 구성 파일 시각화 때 병렬 정렬 됩니다. 그러나, 아코디언 랙 이므로 격자 구조 보 세 뉴클레오티드 사이의 거리는 훨씬 시뮬레이션 (그림 5).
- 회전 하 고 원하는 격자 구조에 각 광속을 이동 합니다. 구성 파일에서 왼쪽에 9 열은 위치 벡터, 백본 기반 벡터와 법선 벡터 (그림 4). 광선을 회전, 회전 위치의 모든 회전 변환을 사용 하 여 백본 기반 및 정상적인 벡터. 다음 그림 5에서 보듯이 그것을 찾을 위치 벡터를 변경 하 여 광선을 이동 합니다.
- OxDNA 패키지에서 제공 하는 스크립트를 사용 하 여 구조를 이완 ($oxDNA에서 예를 참조 하십시오/예/RELAX_INITIAL_CONFIGURATION에 대 한 추가 정보).
- 편안 하 게 구성 파일을 사용 하 여 10 백만 단계에 대 한 분자 동역학 시뮬레이션을 실행 합니다. 사용법은 다음과 같습니다: '. / oxDNA < 입력 >' 5000 또는 10000 단계 모든 데이터를 저장.
- 시각화
참고: 구조는 cogli를 사용 하 여 시각화 했다.- 다운로드 및 cogli (https://sourceforge.net/projects/cogli1/)의 최신 버전을 설치.
- OxDNA 시뮬레이션에서 토폴로지 및 구성 파일에서 cogli를 실행 합니다. 사용법은 다음과 같습니다: '. / cogli1-t < 토폴로지 파일 >< 구성 파일 >'.
- B를 눌러 상자를 숨깁니다.
3입니다. 합성 구조
참고: 합성 메서드를 사용 하면 이전 프로토콜15,18에서 적응 시켰다.
- 설계 된 DNA 스테이플 oligonucleotide 공급자 로부터 구입.
- 이러한 DNA 스테이플 nuclease 무료 물을 사용 하 여 100 μ M에의 농도 조정 합니다.
- 1 개의 관으로 '자유 국가' 구조를 구성 하는 각 DNA 가닥을 수영장 고 각 물가 위한 2 μ M에 농도 조정 합니다.
- 수영장 DNA 잠금 튜브 길이와 잠금 사이트 긴 고 각 물가 위한 2 μ M 농도 조정 합니다. 18, 9, 및 4 잠금 사이트 사용 됩니다. 폴 리 A 가닥 같은 농도에서 브리지 지역에 보완 하는 추가 합니다.
- DNA에 보완 리버스는 수영장 가닥 튜브 길이 여 가닥을 잠글 고 각 물가 위한 2 μ M 농도 조정 합니다.
- 300의 MgCl2 솔루션 준비 nuclease 무료 물 70 μ 1 μ M MgCl2 솔루션의 30 μ를 혼합 하 여 nM. 트리 스 EDTA 솔루션 x 5 95 μ nuclease 무료 물 100 x 트리 EDTA 솔루션의 5 μ를 혼합 하 여 준비 합니다.
- 추가 주식 DNA, MgCl2 솔루션의 1.1 μ의 2 μ, 트리 스 EDTA 솔루션, nuclease 무료 물 7.6 μ와 비 계 DNA의 농도 110의 7.3 μ의 2 μ 혼합 주식의 20 μ 있도록 nM. 설정 비 계 DNA의 최종 농도 40 nM, 200 nM, MgCl2 16 m m와 0.5 Tris EDTA 솔루션 DNA 주식 x.
- 빠르게 60 ° C ° C 당 40 분의 속도에서 4 ° C에서에서 80 ° C에 열 cycler에 멋진 혼합된 재고 솔루션 ° C와 멋진 당 4 분의 속도로 60 ° C에 열
4입니다. 구조의 정화
참고: 모든 구조의 샘플 분석 하기 전에 정화 했다. 이 섹션에서 우리는 이전 문학19에서 적응 못 정화의 프로토콜을 설명 합니다. 이전 문학15,18에 설명 된 대로 샘플 또한 젤 전기 이동 법으로 정화 수 있습니다.
- 5 M NaCl 및 100 x 트리 스-EDTA의 준비.
- 던지다 8000, 트리 스 EDTA x 100의 500 μ와 5 M NaCl의 101 μ nuclease 무료 물 249 μ의 150 μ를 혼합 하 여 강 수-버퍼를 준비 합니다.
- 섹션 3.3에서 300 nM MgCl2 솔루션의 5.5 μ, 섹션 3.3에서 5 배 트리 스 EDTA 솔루션의 10 μ, nuclease 무료 물 84.5 μ를 혼합 하 여 대상 버퍼를 준비 합니다.
- 제 3 강 수-버퍼 섹션 4.2에서 20 μ에서 합성된 구조의 믹스 20 μ. 다음 4 ° c.에서 16000 x g에서 혼합된 재고 회전 상쾌한을 제거 하 고 섹션 4.3에서 대상 버퍼에 펠 릿을 디졸브.
5. '잠긴 상태' 에서' 무료' 아코디언 랙의 재구성
- 구성 실험에 대 한 DNA 잠금 없이 구조를 음성 합성.
- 섹션 3에서 DNA 잠금 가닥을 준비 합니다.
- 합성 구조 20 μ로 원하는 길이의 DNA 잠금 물가의 2 μ를 추가 합니다. DNA 잠금 가닥 농도 구조 보다 5 배 높은 수준 이다.
- 0, 10, 25, 50에 대 한 샘플을 품 어 또는 얼마나 빨리 재구성을 보고 100 분 발생 합니다.
- 100 분 보육에 대 한 샘플 50 ° C에서 30 분 동안 품 어 고 천천히 0.33 ° C/분의 속도로 25 ° C까지 냉각 하십시오.
- 50 분 보육에 대 한 샘플 50 ° C에서 15 분 동안 품 어 고 천천히 0.66 ° C/분의 속도로 25 ° C까지 냉각 하십시오.
- 25 분 화 7.5 분에 대 한 50 ° C에서 샘플을 품 어 고 천천히 1.32 ° C/분의 속도로 25 ° C까지 냉각.
- 10 분 부 화에 대 한 샘플 50 ° C에서 3 분 동안 품 어 고 천천히 3.3 ° C/분의 속도로 25 ° C까지 냉각 하십시오.
- 0 분 보육에 대 한 DNA 가닥을 잠근 후 4 ° C에 게 샘플 추가 됩니다.
- 연결 단계 직후 원치 않는 변성을 방지 하기 위해 4 ° C에 샘플을 식혀 급속 하 게.
6. '자유 국가'에 '고정 상태'에서 아코디언 랙의 재구성
- 음성 합성 구성 실험에 대 한 원하는 길이의 DNA 자물쇠 구조.
- 섹션 3에서 역방향 보완 가닥을 준비 합니다.
- 합성 구조 20 μ로 원하는 길이 잠금 가닥에 무료 리버스는 물가의 2 μ를 추가 합니다. DNA 잠금 가닥 농도 구조 보다 5 배 높은 수준 이다.
- 0, 12, 60, 120에 대 한 샘플을 품 어, 240 분을 보고 얼마나 빨리 재구성 발생 합니다.
- 12, 60, 120, 240 분 보육에 대 한 신속 하 게 샘플 40 ° C에가 열 하 고 천천히 각각에 해당 하는 시간에 대 한 20 ° C까지 냉각 하십시오. 바로 파견 단계 후 4 ° c 원치 않는 변성을 방지 하기 위해 샘플을 식혀 급속 하 게.
- 0 분 인큐베이션 역방향 보완 가닥 추가 된 후 4 ° C에서 샘플을 저장 합니다.
7. 편 영상
참고: 가장 이미징 프로토콜 이전 문학18,20에서 적응 했다.
- 1.25 M NaOH 솔루션 nuclease 무료 물 87.5 μ와 10 M NaOH 솔루션의 12.5 μ를 혼합 하 여 준비 합니다.
- 2 %uranyl 편대 솔루션의 50 μ를 1.25 M NaOH 솔루션의 1 μ를 추가 합니다.
- 3 분, 3 분 동안 최대 속도로 원심 분리기에 대 한 솔루션 소용돌이. 3 분 동안 글로우 방전 가장 격자에 순화 된 샘플의 3 μ 보증금과 빠르게 필터 종이 밖으로 씻어.
- 30 초 동안 준비 uranyl 편대 솔루션의 7 μ 보증금과 빠르게 필터 종이 밖으로 씻어.
- 길이 아코디언 구조를 가장 하 여 몇 군데의 각도 측정 합니다.
8. 무서 워 분석
- Atto 550를 Förster 거리는 6.5 거 647N 염료를 사용 하 여 nm. 주식 58와 주식 117 표 1 에 놓여있는지 이라는 가닥 바꿉니다. 다음 섹션 3에서 설명 하는 방법으로 붙일 이라는 물가 구조를 음성 합성.
- 순화 된 샘플의 농도 측정 합니다.
- 384 microplates 10 nM와 부하 50 μ를 샘플 잘 정상화.
- 기증자와 수락자 염료 550 nm에서 측정 샘플을 자극 570에서 형광 스펙트럼 800 nm는 fluorometer nm.
- 같은 방법으로 기증자 전용 샘플의 형광 스펙트럼을 측정 합니다.
- 650에서 샘플의 염료를 흥분 nm 및 형광 스펙트럼 측정 670에서 800 nm nm. 이것은 수락자의 농도 측정 하입니다.
- 3 샘플, 합성 되 고 별도로 정화는 같은 실험을 반복 하 여 표준 편차를 얻을.
- 무서 워 효율성 비율21아래 방정식에 의해 설명 된 대로 메서드를 계산 합니다.
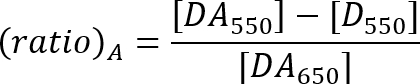
다550: 기증자와 수락자 550 nm 여기에서 샘플의 수락자 피크 형광 강도.
D550: 550 nm 여기에서 기증자 전용 샘플의 수락자 방출 범위에서 형광 강도.
다650: 기증자와 수락자 650 nm 여기에서 샘플의 수락자 피크 형광 강도.
결과
설계 된 6 월 6 일에 의해 DNA 아코디언 랙 oxDNA16,17 에서 시뮬레이션 하 고 결과 그림 6에 표시 됩니다. 시뮬레이션 결과에서 구조 왜곡 없이 원하는 구조 형성은 확인 되었다.
그림 7 에서 가장 이미지는 2, 8, 13, 20 bp 길이의 잠금 구성 된 구?...
토론
이 프로토콜은 디자인, 시뮬레이션, 합성, 그리고 기본적인 2D DNA 아코디언 랙의 분석에서 전체 과정을 소개합니다. 수정 된 설계 및 시뮬레이션 규칙 있다 설명 디자인 규칙의 표준 DNA 종이 접기, 다르기 때문에 DNA 아코디언 랙 추가 뉴클레오티드 유연성14,15크로스 오버에서. 이것에서 우리는 프로토콜 DNA 아코디언 선반을 사용 하 여 다양 한 연구를 가?...
공개
저자 공개할 게 없다
감사의 말
이 연구는 국립 연구 재단의 Korea(NRF) 사역의 과학 및 ICT (MSIT) (2015K1A4A3047345)와 Nano·를 통해 글로벌 연구 개발 센터 프로그램에 의해 부분적으로 지원 되었다 소재 기술 개발 프로그램을 통해 국가 연구 재단의 한국 (NRF) 과학과 ICT (MSIT) (2012M3A7A9671610)에 의해 자금. 서울 대학교에서 공학 연구 연구소가이 작품에 대 한 연구 시설을 제공합니다. 저자 무서 워 분석에 대 한 형광 분광학에 관한 태 영 윤 (생물 과학, 서울 대학교)으로 감사를 인정.
자료
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
참고문헌
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유