JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
종양 세포에서 MicroRNA 수준, 기능 및 관련 표적 유전자를 평가하기 위한 시험관 내 프로토콜
요약
이 프로토콜은 프로브 기반 실시간 중합효소 연쇄 반응(PCR), 설포호다민 B(SRB) 분석, 3' 번역되지 않은 영역(3' UTR) 클로닝 및 루시퍼라제 분석제를 사용하여 관심 있는 miRNA의 표적 유전자를 확인하고 miRNA의 기능을 이해합니다.
초록
MicroRNAs (miRNAs)는 암을 포함하여 몇몇 질병에 있는 수많은 세포내 신호 통로를 조절하기 위하여 인식되는 작은 규정하는 RNA입니다. 이 작은 조절 RNA는 주로 그들의 표적 메신저 RNA (mRNA)의 3' 번역되지 않은 지구 (3' UTR)와 궁극적으로 mRNA의 디코딩 프로세스의 억제 및 표적 mRNA 분해의 확대의 결과로 상호 작용합니다. 발현 수준 및 세포내 기능에 기초하여, miRNA는 종양발생 및 종양 억제 mRNA의 조절 인자로 작용할 수 있다. 수백 또는 수천 개의 계산 예측 표적 중에서 miRNA의 선의의 표적 유전자를 식별하는 것은 관심 있는 miRNA의 역할 및 기본 분자 메커니즘을 분별하는 중요한 단계이다. 다양한 miRNA 표적 예측 프로그램은 가능한 miRNA-mRNA 상호작용을 검색할 수 있다. 그러나, 가장 어려운 질문은 관심의 miRNA의 직접적인 표적 유전자를 확인하는 방법입니다. 이 프로토콜은 miRNA의 기능과 관련된 miRNA 표적을 식별하는 방법에 대한 주요 방법의 재현 가능한 전략을 설명합니다. 이 프로토콜은 miRNA 모방 형질감염에 따른 프로브 기반 실시간 폴리머라제 연쇄 반응(PCR), 설포호다민 B(SRB) 분석사를 사용하여 miRNA 수준, 기능 및 관련 표적 mRNA를 밝히기 위한 단계별 절차에 대한 실용적인 가이드를 제시합니다. , 투여 량 -응답 곡선 생성, 및 루시 퍼래스 분석 유전자의 3'UTR의 복제와 함께, 이는 개별 miRNAs의 역할의 적절한 이해에 필요하다.
서문
MicroRNAs (miRNAs)는 주로 선의의 표적 유전자에서 3'번역되지 않은 영역 (3' UTR)에 반응하여 메신저 RNA (mRNAs)의 번역 및 분해 과정을조절하는 작은 조절 RNA이다 1. miRNAs의 발현은 전사 및 전사 후 메커니즘에 의해 조절될 수 있다. 이러한 규제 메커니즘의 불균형은 암을 포함한 수많은 질병에서 통제되지 않고독특한 miRNAs 발현 수준을 가져온다 2. 단일 miRNA는 다양한 mRNA와 다중 상호작용을 가질 수 있다. 그에 상응하여, 개별 mRNA는 다양한 miRNA에 의해 제어될 수 있다. 따라서 세포내 신호 네트워크는 생리적 장애 및 질병이 시작되고 악화될 수 있는 특유의 발현 된 miRNAs에 의해 복잡하게 영향을 받습니다2,3,4. 5개 , 6. miRNAs의 변경된 표현이 암의 각종 모형에서 관찰되었더라도, miRNAs와 함께 암세포의 매너를 조절하는 분자 기계장치는 아직도 크게 알려지지 않았습니다.
축적 된 증거는 miRNAs의 종양 억제 역할이 암의 유형에 달려 있음을 보여주고있다. 예를 들어, 지게차 상자 o3(FOXO3)를 표적화함으로써, miR-155는 대장암의 세포 증식, 전이 및 화학저항성을 촉진한다7,8. 대조적으로, 신경교종 세포 침입의 제한은 신경발생성 궤적 노치 호몰로그 단백질 2(NOTCH2) 발현9의 조절을 통해 miR-107에 의해 유도된다. miRNA 기능과 관련하여 miRNA 표적 상호 작용의 평가는 miRNAs가 건강하고 병들인 상태 둘 다에 있는 각종 생물학 프로세스를 통제하는 방법을 더 잘 이해하는 필수 불가결한 부분입니다10. 또한, miRNA의 선의의 표적(들)의 발견은 다양한 항암약물을 가진 miRNA 기반 치료에 대한 미세 조정 된 전략을 더욱 제공할 수 있다. 그러나 miRNA 분야의 주요 과제는 miRNA의 직접 표적을 식별하는 것입니다. 여기서, 상세한 방법은 miRNA 표적 유전자 측정을 위한 재현가능한 실험 접근법으로서 제시된다. miRNA 표적 식별을 위한 성공적인 실험 설계는다양한 단계 및 고려사항을 수반한다(도 1). 종양 세포 및 정상 세포에서 성숙한 miRNA 수준의 비교는 관심있는 miRNA를선택하는 일반적인 절차 중 하나일 수 있다(도 1A). 세포 증식에 대한 miRNA의 효과를 검출하기 위해 선택된 miRNA의 기능적 연구는 관심 있는 miRNA의가장 적합한 후보 표적의 목록을 좁히는 것이 중요하다(도 1B). miRNA의 실험적으로 검증된 기능에 기초하여, miRNA 표적 예측 프로그램과 함께 회사내 문헌 및 데이터베이스의 체계적인 검토가 유전자기능에 대한 가장 관련성이 있는 정보를 검색하는 데 요구된다(도 1C). 관심 있는 miRNA의 실제 표적 유전자의 식별은 유전자의 3' UTR의 복제와 함께 루시퍼라아제 분석실험과 같은 실험을 구현함으로써 달성될 수있으며, 실시간 PCR 및 웨스턴 블로팅(도 1D). 현재 프로토콜의 목적은 miRNA 모방 형질감염, 투여량-반응 곡선 생성, 및 프로브 기반 실시간 폴리머라제 연쇄 반응(PCR), 설포호다민 B(SRB) 분석법의 포괄적인 방법을 제공하는 것이다. 유전자의 3' UTR의 복제와 함께 루시퍼라제 분석. 현재 프로토콜은 개별 miRNA의 기능 및 암 치료에서 miRNA의 의미를 더 잘 이해하는 데 유용할 수 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 성숙한 마이크로RNA (miRNA) 발현 분석
-
성숙한 miRNA 상보적인 DNA (cDNA) 종합
- 총 RNA 254 ng와 데옥시리보뉴클리스 I(DNase I) 혼합물 4.5μl을 첨가한 다음, PCR 스트립 튜브에 초순수를 첨가하여 최대 18 μL(그림2A)을만듭니다. 총 반응 수에 기초하여 충분한 양의 DNase I 혼합물을 사용하여 여러 세포주로부터 정제된 각 총 RNA 샘플에 대한 반응을 준비한다.
참고: DNase I 혼합물은 DNase I(1.8 μL), 리보누클레스 억제제(0.3 μL), 및 25 mM MgCl2(2.4 μL)로 구성된다. 재현가능한 전체 RNA를 조달하기 위해 페놀-클로로포름 기반 추출 방법을 사용하는 대신 컬럼 기반 추출 방법을 적용하였다. 페놀-클로로포름 기반 추출방법(11,12)을사용할 경우 일부 miRNA의 추출 수율이 세포 수에 따라 달라질 수 있다고 보고되었다. - 열 사이클러에서 튜브를 배양합니다. 튜브를 37°C에서 10분 동안 실행하고, 90°C에서 5분 배양하여 DNase I을 가열한다. 배양 후 즉시 튜브를 얼음 위에 놓습니다.
- DNase의 7.1 μL을 처리한 총 RNA를 2세트의 새로운 튜브로 처리한 다음, 글리세랄데히드-3-인산탈수소효소(GAPDH) 유전자에대해 1.5 μL의 안티센스 프라이머를 첨가하였다(그림 2B).
참고: cDNA 합성을 위한 총 RNA의 양은 이 단계에서 100 ng가 된다. GAPDH 안티센스 프라이머의 재고 농도는 10 μM이다. GAPDH 안티센스 프라이머를 첨가하는 것은 유전자 특이적 프라이머 방법을 이용한 GAPDH cDNAs의 생성을 위한 것이다. - 열 사이클러를 사용하여 튜브를 배양합니다. 80°C에서 5분 동안 반응한 후 5분 동안 60°C에서 반응합니다.
- 각 반응에 3.4 μL의 역전사(RT) 효소혼합물을 첨가한다(도 2B). RT 효소 혼합물은 100 mM 데옥시리보뉴클레오티드 트리포스페이트(0.15 μL), 10x RT 버퍼(1.5 μL), 리보누클레스 억제제(0.75 μL), 및 역전사 효소(1 μL)로 구성된다. 총 반응 수에 따라 충분한 양의 혼합물을 준비하십시오.
- 각 반응에서 특정 miRNA에 대해 5x RT 프라이머의 3 μL을 첨가합니다(도2B).
참고: 총 부피는 각 반응에 대해 15 μL이다. - 열 사이클러를 사용하여 튜브를 실행합니다. 30분 동안 16°C에서 시작한 다음 30분 동안 42°C에서 반응을 시작하고, 최종적으로 85°C에서 5분동안 5분 동안 유지한다(그림 2B). 단일 가닥 cDNA는 동일한 튜브에서 특정 miRNA 및 GAPDH 유전자 모두에 대해 이 단계에서 생성된다.
- 총 RNA 254 ng와 데옥시리보뉴클리스 I(DNase I) 혼합물 4.5μl을 첨가한 다음, PCR 스트립 튜브에 초순수를 첨가하여 최대 18 μL(그림2A)을만듭니다. 총 반응 수에 기초하여 충분한 양의 DNase I 혼합물을 사용하여 여러 세포주로부터 정제된 각 총 RNA 샘플에 대한 반응을 준비한다.
-
실시간 폴리머라제 연쇄 반응(PCR) 및 데이터 분석
- 1:49 비율로 초순수로 각 cDNA를 희석합니다.
- 특정 miRNA 및 GAPDH에 대한 반응혼합물을 준비한다(표 1). 특정 miRNA 및 GAPDH의 검출을 위해, 각 cDNA 견본을 위한 삼중 반응을 설치하십시오.
- 실시간 PCR 및 데이터 분석을수행합니다(그림 2C). 비교 CT 방법13,14를사용하여 데이터를 분석한다.
2. 마이크로RNA (miRNA) 모방변형
참고: miRNA-107은 1단계에서 선택된다. miRNA-107은 정상 세포와 비교하여 종양 세포에서 하향 조절되기 때문에, miRNA-107은 종양 억제 miRNA라고 추측할 수 있다. 정상 세포와 비교하여 종양 세포에서 상이 조절되는 miRNA의 경우(예를 들어, miRNA-301) miRNA-301에 대한 안티센스 올리고뉴클레오티드는 단계 2, 3 및 4에 적용될 수 있다.
- 계수 챔버 장치로 세포를 계산하고 96 웰 플레이트에서 세포를 플레이트합니다. 세포 밀도는 각 웰에 대해 2 x 103 셀/100 μL입니다. P/S는 형질전환 효율을 감소시킬 수 있기 때문에 페니실린-스트렙토마이신(P/S)을 함유하는 세포 배양 배지를 사용하지 마십시오.
-
miRNA 대조군 모방 및 miRNA-107 모방의 몇몇 최종 농도에서 세포를 트랜스펙트하기 위해 형질전환 혼합물 세트를 준비한다(그림 3).
- miRNA 대조군(25 μM 농도)으로부터 모방 또는 miRNA-107모방, 희석 및 미세원심지 튜브를 이용한 트랜스펙션 시약과 함께 감소된 혈청 매체에서 miRNA-107 모방의 상응하는 양을 첨가한다(도3A). 마이크로파이펫을 사용하여 혼합물을 함유한 올리고를 부드럽게 섞는다. 올리고의 총량(miRNA 모방 대조군 + miRNA-107 모방)은 각각의 웰에서 동일해야 한다. 블랭크 웰은 세포 배양 배지 및 세포 없이 형질전환 시약을 함유하는 환혈성 배지의 100 μL을 포함한다.
- 세포 배양 후드에서 10분 배양 한 후, 혼합물을 함유한 올리고를 다시 부드럽게 혼합한 다음 혼합물의 50 μL을 각 우물에 추가합니다. 형질감염된 세포를 세포 배양 인큐베이터에 보관한다. 6-12시간 배양 후 태아 소 혈청(FBS) 및 P/S를 모두 함유하는 신선한 세포 배양배지로 배지를 함유하는 형질감염 시약을 교체한다. 72 시간 동안 세포를 추가로 배양하십시오. miRNA 모방의 총 치료 기간은 96 시간입니다.
3. 설포호다민 B (SRB) 분석
-
셀 고정
- 플레이트의 각 웰에서 세포 배양 배지를 제거하고 100 μL의 100 μL을 각 웰에 즉시 채웁니다. 조심스럽게 각 우물에서 세포 배양 배지를 흡인하여 바닥에서 어떤 세포 손상 및 분리를 피한다.
참고: 증류수 50 mL에 TCA 분말 20g을 추가하여 40 % TCA를 준비하십시오. 40% TCA에서 1:3 희석 비율로 증류수로 40% TCA를 희석하여 10% TCA를 만듭니다. - 10% TCA가 함유된 플레이트를 냉장고(4°C)에 1시간 동안 보관하십시오.
- 물 욕조에 담그고 여러 번 접시를 씻고 건조시십시오. 우물에 물이 남지 않도록 플레이트를 두드려 우물 내부에서 여분의 물을 제거하십시오. 다음 단계로 가기 전에 접시를 실험실 벤치에 두고 건조시면 됩니다.
- 플레이트의 각 웰에서 세포 배양 배지를 제거하고 100 μL의 100 μL을 각 웰에 즉시 채웁니다. 조심스럽게 각 우물에서 세포 배양 배지를 흡인하여 바닥에서 어떤 세포 손상 및 분리를 피한다.
-
세포 염색
- 빈 웰을 포함하여 각 웰에 0.4 % SRB 용액의 피펫 50 μL. 0.4% SRB 용액이 우물 바닥을 일관되게 덮을 때까지 플레이트를 부드럽게 흔들어 주세요.
참고: 0.4% SRB 용액을 1% 아세트산 100 mL에 0.4 g의 SRB 분말을 첨가하여 준비하고 사용하십시오. 용액을 조심스럽게 흔들어 섞어 줍니다. 0.4% SRB 용액병을 알루미늄 호일과 같은 가벼운 보호 재료로 감쌉니까? 0.4% SRB 솔루션을 냉장고에 보관하십시오. - 40 분 내지 60 분 동안 배양 한 후 1 % 아세트산으로 헹구어 접시를 씻으하십시오. 언바운드 염료가 완전히 씻을 때까지 접시를 씻으소서 (그림3B).
- 다음 단계로 가기 전에 접시를 실험실 벤치에 두고 건조시면 됩니다.
참고: 플레이트는 3.3 단계로 가기 전에 완전히 건조되어야합니다.
- 빈 웰을 포함하여 각 웰에 0.4 % SRB 용액의 피펫 50 μL. 0.4% SRB 용액이 우물 바닥을 일관되게 덮을 때까지 플레이트를 부드럽게 흔들어 주세요.
-
흡광도 측정
- 트리스 베이스 솔루션(10 mM)의 피펫 100 μL을 블랭크 웰을 포함한 해당 웰로 포함한다. 접시를 셰이커에 10 분 동안 보관하십시오. 492 nm에서 흡광도를 측정하십시오.
4. 복용량 응답 곡선의 생성
- 스프레드시트에서 SRB 분석 데이터를 분석합니다. 각 그룹의 흡광도 값에서 빈 흡광도를 빼고 각 그룹의 흡광도 값의 평균(AVE) 및 표준 편차(STD)를 계산합니다.
- 평균 흡광도 비율 계산(AVE%) 및 표준 편차 (STD %) SRB 분석법의 흡광도 값을 사용하여 각 그룹의.
참고: miRNA 대조군의 AVE%는 처리된 군을 모방한 100%이다. 다음 공식을 사용하여 STD%를 계산합니다: STD% = (각 군의 STD/ 대조군 모방 처리군의 AVE 흡광도) x 100. - 처리 농도, AVE%, STD%를 포함한 원시 데이터를 수직으로 정렬하여 소프트웨어로 가져옵니다. 로그 0이 정의되지 않았기 때문에 X축의 첫 번째 농도를 0에가까운 값으로 설정합니다(예: 0.01).
- 그래프 만들기 탭을 클릭하고 단순 분산 오류막대를 선택합니다. 워크시트 열을 기호 값으로 선택하고 다음을 클릭합니다. 데이터 형식 패널에서 XY 쌍을 선택하고 다음을 클릭합니다. 선택 데이터 패널에서 해당 데이터 열을 선택합니다. 마무리 버튼을 클릭하여 플롯을 작성합니다.
참고: X축은 농도를 나타내고, Y축은 각 농도(AVE%)의 평균 흡광도 비율을 나타내고, 오차 막대는 각 농도(STD%)의 표준 편차 비율을 가리킵니다. - X축을 두 번 클릭하여 축의 배율 유형과 배율을 수정합니다. 선형에서 로그로 배율 유형을 변경합니다. 시작 범위와 끝 범위 번호를 각각 0.01 및 200으로 수정합니다.
-
모든 분산형 플롯을 마우스 오른쪽 단추로 클릭하고 곡선 맞춤을 선택하고 사용자가 정의한 하위 범주로 이동합니다. [응답] 곡면을 선택하고 다음 단추를 클릭한 다음 완료 버튼을 클릭합니다. 투여량-응답 곡선은 이제 보고서 탭(그림4A)과함께 생성된다.
- 용량 응답 곡선생성을 위해 소프트웨어에 방정식 1을 입력하려면 분석 탭을 클릭하고 회귀 마법사를선택합니다. 방정식 범주에서 사용자 정의로 이동한 다음 새 단추를 클릭합니다. 방정식 1, 변수, 초기 매개변수 및 구속조건을 해당 빈 상자에 삽입합니다(그림4B,C). 단추로 추가를 클릭하고 방정식이름을 용량 응답곡선으로 설정합니다. 이제 수식 이름이 수식 범주에 정의된 하위 범주 사용자 에서 생성됩니다. f는 수학식 1에서 셀 생존율(% % 셀 생존율)의 백분율을 나타냅니다.
방정식 1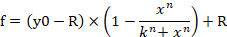
- 보고서 탭으로 이동한 다음 n, k 및 R 값을 확인합니다.
참고 : y0은 miRNA 대조군 모방 처리 그룹의 100 % 세포 생존성을 나타내고, n은 힐 형 계수 (플롯의 기울기)를 나타내고, k는 miRNA-107 모방의 최대 효과의 50 %를 생성하는 miRNA-107 모방의 농도를 나타냅니다 (절반 최대 억제 농도, IC50)및 R은 잔류 영향을 받지 않는 분획(저항 분율)(저항 분율)(15)을 나타낸다. 투여량-응답 곡선을 생성하는 데 사용되는 방정식은 y0에서 R 값(있는 경우)까지의 범위를 100%(도4A)로인식합니다. 따라서 y0에서 0(도4A)의값을 기준으로 계산되는 조정된 k(IC50)값을 획득할 필요가 있다. 조정된 k(IC50)는다른 ICx값(예: IC10 ~ IC90)과함께 수학식 1에서 파생된 수학식 2를 사용하여 얻을 수 있습니다. 수학식 1에서 수학식 2의 파생은 보충 그림1에 표시됩니다.
방정식 2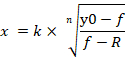
- 방정식 2가 적용되는 셀에서 왼쪽 마우스 버튼을 두 번 클릭합니다. 방정식 2 및 생성된 투여량 응답 곡선의 파라미터를 사용하여 IC10에서 IC 90(그림 4D)에이르는 ICx의 조정된 값을 계산할 수 있습니다.
- 동일한 부호를 입력한 다음 수식이 셀의 대괄호로 시작됩니다. 수식을 입력할 때 해당 열과 행에 달러 기호를 추가하여 n, k 및 R값을 절대 셀 참조로 고정하여 수식을 행으로 자동채울 때 이러한 고정 값이 변경되지 않도록 합니다(그림 4D). 또는 방정식 2를 사용하여 조정된 값을 수동으로 계산할 수 있습니다.
참고: IC90 값은 R 값이 10보다 크므로 결정되지 않습니다. 또한 R 값이 20이상이면 IC80의 값도 결정되지 않습니다(도4D).
5. 관심 있는 마이크로RNA의 직접 표적 유전자 검증
참고: SRB 분석, miRNA-107 과 같은 기능적 실험을 수행한 후 종양 억제 miRNA로 확인되고 miRNA-107이 종양유전자를 직접 표적으로 하는 것이 매우 가능하다. TargetScan(http://www.targetscan.org/vert_71/)과 같은 miRNA 표적 예측 프로그램을 사용하여 모든 예측 대상 유전자목록을 확인한 다음 PubMed및 및 및 진 카드.
- 3' 번역되지 않은 영역(UTR)의 복제를 위한 프라이머 설계
- 유전자의 이름을 GeneCards(https://www.genecards.org/)에 넣고 유전자의 기호를 클릭합니다. 유전자의 Ensembl ID를 클릭하여 Ensembl 게놈 브라우저로 평가한 다음 전사체 표에서 성적증명서 ID를 클릭합니다. 그 후 왼쪽에 있는 성적 증명서 기반 디스플레이 목록에 있는 Exons를 클릭합니다.
- 3' UTR의 뉴클레오티드 서열을 복사하여 프라이머 설계 프로그램에 붙여 넣습니다. 이 프로그램에서 시퀀스를 다시 복사하여 워드 프로세서에 붙여 넣습니다. miRNA 결합 서열의 존재뿐만 아니라 복제에 사용되는 제한 효소 부위의 존재를 확인한다.
참고: 3' UTR 내에 제한 효소 인식 부위가 없는 경우, 복제를 위해 선택된 제한 효소는 다음 단계로 사용될 수 있다. - 프라이머 설계 프로그램에서는 3' UTR 시퀀스를 받아들이고 다음 조건으로 전진 및 역방향 프라이머를 설계하기 시작합니다. 길이: 20-30 뉴클레오티드, Tm: 45-58 °C, GC%: 40-60%. 두 프라이머의 Tm 값 간의 차이는 5°C 미만이어야 한다. 본 연구에 사용된 프라이머 서열은 보충 도2에 제공된다. 설계된 프라이머에 제한 효소 인식 서열과 4개의 무작위 뉴클레오티드를 추가합니다.
- 그라데이션 PCR
- 1회 어닐링 온도당 설계된 프라이머를 포함한 25 μL의 PCR 반응 혼합물을 준비한다(표 2). 총 반응 수에 따라 충분한 양의 혼합물을 준비하십시오. 파이펫팅하여 용액을 혼합하고 각 튜브에 25 μL의 반응 혼합물을 추가합니다. 몇 초 동안 튜브를 원심 분리합니다.
- 변성 단계에서 확장 단계까지 35-40PCR 주기를 수행합니다. PCR 주기를 다음 단계로 설정: 1분 동안 98°C(1주기, 폴리머라제 활성화 단계), 10초용 95°C(변성 단계), 45°C-68°C, 30초(어닐링 단계), 68°C(연장 단계, 1000 bp당 10s-1분), 3분 종료 단계 68°C 그리고 마지막으로 4 °C까지 식힙니다.
- PCR 제품을 실행하고 DNA 사다리가있는 1 % 아가로즈 젤에 밴드를 확인하십시오. 최고의 어닐링 온도를찾습니다(그림 5A). 다음 단계에 가장 좋은 어닐링 온도를 사용하여 유전자의 3' UTR을 다시 증폭시다.
- 이중 소화
- 튜브에서 두 가지 제한 효소인 XhoI(또는 AsiSI) 및 NotI를포함하는 반응 혼합물을 만든다(표 3). 수조 (37 °C)를 사용하여 3-4 시간 동안 혼합물을 배양합니다.
- 이중 소화 제품을 1% 아가로즈 젤에 바르고 자외선 아래에서 밴드를 자른다. 루시퍼라제 벡터의 경우, 겔에서 실행하기 전에, 결찰 단계 동안 재원형화를 방지하기 위해 또 다른 1시간 동안 알칼리성 인산염 10U와 이중 소화 벡터를 반응시킨다.
- 이중 소화 된 PCR 제품 및 절제 된 밴드에서 루시 퍼라제 벡터를 정화합니다.
- 루시퍼라제 벡터에 PCR 제품 결찰
- DNA 리가아제(표 4)를 포함하는 결찰 반응혼합물의 20 μL을 만든다.
참고: 루시퍼라제 벡터에 대한 PCR 생성물(삽입)의 몰 비율은 3:1일 수 있다. 1:1 또는 2:1. - 10-15 s에 대한 튜브를 잠시 원심분리하고 열 사이클러를 사용하여 밤새 16°C에서 배양한다.
참고: 대안적으로, 튜브는 결찰을 위해 2-3일 동안 4°C에서 배양될 수 있다. 이 단계에서, PCR 삽입체는 레닐라 리포터 유전자의 하류에 위치하는영역내로 클로닝될 것이다(도 5B). 유전자의 복제 된 3'UTR에 miRNA의 결합은 레닐라 활성에서 감소 시킬 수 있습니다. 반딧불 루시퍼라제는 레닐라 발현 레벨의 정상화를 위한 것이다.
- DNA 리가아제(표 4)를 포함하는 결찰 반응혼합물의 20 μL을 만든다.
- 변환 및 식민지 PCR
- 유능한 세포를 함유하는 튜브에 결찰 혼합물 (3-5 μL)을 추가합니다. 튜브를 부드럽게 두드리고 얼음에 보관하십시오 (20 분).
참고 : 결찰 혼합물을 추가하기 전에 얼음에 유능한 세포를 동결 해제하십시오. - 튜브를 빠르고 부드럽게 열 블록으로 옮니다. 열 충격 (30 s-1 분 동안 42 °C)에 따라 튜브를 얼음에 20 분 동안 놓습니다.
- 루리아 베르타니 (LB) 한천 접시에 유능한 세포를 확산. 하룻밤 동안 인큐베이터 (37 °C)에서 유능한 세포를 성장시다.
참고: 암피실린 (50-100 μg / mL)은 한천 접시에 포함되어 있습니다. - 개별 식민지를 선택하고 초순수가 함유 된 8 스트립 튜브 중 하나에서 대장균을 다시 중단하십시오. 이 단계를 반복하여 무작위로 선택된 4-8 콜로니(그림5C)에서 대장균을 다시 일시 중단합니다.
- 대장균 현탁액 25 μL을 다른 8 스트립 튜브 세트로 옮김. 지금, 대장균 현탁액의 관의 2 세트가 있습니다.
참고: 한 튜브는 식민지 PCR용이고 다른 하나는 예방 접종용입니다. 대장균 현탁액은 4°C(도 5C)에서일시적으로 보관할 수 있다. - 대장균 현탁액을 사용하여 콜로니 PCR을 수행합니다. 이 단계는 콜로니에 삽입이 포함되어 있는지 확인하는 것입니다. 유전자의 3' UTR을 항양하고 분리하는 최고의 콜로니를 선택한다(도5C).
참고: 선택된 유전자의 각 3' UTR에 대해 5.1-5.5 단계를 반복한다. 유전체 DNA를 대장균 현탁액으로 대체하여 표 2에 나타낸 PCR 반응의 상태를 따르십시오.
- 유능한 세포를 함유하는 튜브에 결찰 혼합물 (3-5 μL)을 추가합니다. 튜브를 부드럽게 두드리고 얼음에 보관하십시오 (20 분).
- 루시퍼라아제 분석
- 24웰 플레이트를 준비합니다. 각 웰에 대해 500 μL 세포 배양 배지에 1-2 x 104 세포를사용한다. P/S를 사용하면 형질전환 효율을 떨어뜨릴 수 있으므로 형질감염에 P/S를 함유하는 세포 배양 배지를 사용하지 마십시오.
- 트랜스펙트 50 ng의 루시퍼라아제 벡터를 대조군 모방 또는 형질전환 시약을사용하여 특정 miRNA 모방을 가진 세포 내로 의한 트랜스펙트(도 5D). 특정 miRNA의 효과를 하나 이상의 농도에서 스크리닝하는 경우, 각 웰에서 올리고의 총량을 동일하게 유지한다(2단계 참조).
- 다음날 인산완식염수(PBS)를 사용하여 우물 내부를 두 번 씻으세요.
- 200 μL의 라시스 시약을 웰에 적용하고 루시퍼라아제 활성을 측정하기 전에 세포 라시스를 충분히 수행하십시오.
참고: 접시를 적어도 15 분 동안 흔들기 접시에 보관하십시오. - 5-10 μL의 세포 용해액을 새로운 튜브로 옮기고 시약 I. 즉시 파이펫팅하여 용액을 혼합하고 발광계를 사용하여 반딧불 루시퍼라아제 활성을 읽습니다.
참고: 10-15 s에 대한 반딧불 루시 퍼라아제 활동을 읽어보십시오. - 동일한 튜브에 시약 II 100 μL을 추가한 다음 두 번 파이펫팅하여 혼합합니다. 루미노미터를 사용하여 10-15s의 레닐라 루시퍼라아제 활동을 읽어보십시오. 각 샘플에 대해 5.6.5 단계와 5.6.6단계를 반복합니다.
- 레닐라와 반딧불의비율을 계산합니다(그림 5E).
참고: 반딧불의 활동은 세포 내로 루시퍼라아제 생성물의 형질감염 효율을 나타낸다.
Access restricted. Please log in or start a trial to view this content.
결과
miRNA 수준의 성공적이고 정확한 확인은 질병의 발달 및 진행에서 miRNA의 예상 역할에 기초하여 miRNA의 분류가 가능한 데이터의 해석에 중요합니다. miRNA-107 및 miRNA-301의 수준은 프로브 기반 정량적 PCR을 사용하여 3개의 췌장 세포주에서 측정되었다. 특정 miRNA 및 동일한 반응에서 기준 유전자 모두의 cdNAs의 합성은 데이터의 재현성을 증가시킬 수 있다. PANC-1 및 CAPAN-1은 인간 췌장 덕트 선암 세포주이?...
Access restricted. Please log in or start a trial to view this content.
토론
관심있는 miRNA의 기능을 가진 선의의 miRNA 표적의 결정을 위한 전략은 miRNA의 다중 역할의 이해를 위해 필수적입니다. miRNA 표적 유전자의 식별은 세포에서 miRNAs에 의해 변조된 세포 신호 이벤트를 해석하기 위한 지침일 수 있다. miRNA의 기능적으로 중요한 표적 유전자의 공개는 암에 있는 miRNA 기지를 둔 치료를 개발하기 위하여 근본적인 지식을 제공할 수 있습니다.
마이크로어...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 것이 없다.
감사의 말
이 연구는 교육부의 지원을 받는 국립연구재단(NRF)을 통해 기초과학 연구 프로그램에 의해 지원되었다(2017R1D1A3B03035662); 및 한림대학교 연구기금, 2017(HRF-201703-003)
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SPL Life Sciences | 50015 | |
| 24-well plate | Thermo Scientific | 142475 | |
| 50 mL conical tube | SPL Life Sciences | 50050 | |
| 6-well plate | Falcon | 353046 | |
| 6x DNA loading dye | Real Biotech Corporation | RD006 | 1 mL |
| 8-cap strip | Applied Biosystems | N8010535 | For cDNA synthesis |
| 8-tube strip | Applied Biosystems | N8010580 | For cDNA synthesis |
| 96-well plate | Falcon | 353072 | |
| Acetic acid | Sigma | A6283-1L | 1 L |
| Agarose A | Bio Basic | D0012 | 500 g |
| Alkaline phosphatase | New England Biolabs | M0290S | 10,000 U/mL |
| Ampicillin | Bio basic Canada Inc | AB0028 | 25 g |
| AriaMx 96 tube strips | Agilent Technologies | 401493 | For real time PCR |
| AriaMx real-time PCR system | Agilent Technologies | G8830A | qPCR amplification, detection, and data analysis |
| AsiSI | New England Biolabs | R0630 | 10,000 units/mL |
| CAPAN-1 cells | ATCC | HTB-79 | |
| Cell culture hood | Labtech | Model: LCB-1203B-A2 | |
| Counting chambers with V-slash | Paul Marienfeld | 650010 | Cells counter |
| CutSmart buffer | New England Biolabs | B7204S | 10X concentration |
| DMEM | Gibco | 11965-092 | 500 mL |
| DNA gel extraction kit | Bionics | DN30200 | 200 prep |
| DNA ladder | NIPPON Genetics EUROPE | MWD1 | 1 Kb ladder |
| DNase I | Invitrogen | 18068015 | 100 units |
| Dual-luciferase reporter assay system | Promega | E1910 | 100 assays |
| Fetal bovine serum | Gibco | 26140-079 | 500 mL |
| HIT competent cells | Real Biotech Corporation(RBC) | RH617 | Competent cells |
| HPNE cells | ATCC | CRL-4023 | |
| LB agar broth | Bio Basic | SD7003 | 250 g |
| Lipofectamine 2000 | Invitrogen | 11668-027 | 0.75 mL |
| Lipofectamine RNAiMax | Invitrogen | 13778-075 | 0.75 mL |
| Luminometer | Promega | Model: E5311 | |
| Microcentrifuge tube | Eppendorf | 22431021 | |
| Microplate reader | TECAN | Infinite F50 | |
| miRNA control mimic | Ambion | 4464058 | 5 nmole |
| miRNA-107 mimic | Ambion | 4464066 | 5 nmole |
| miRNeasy Mini Kit | Qiagen | 217004 | 50 prep |
| Mupid-2plus (electrophoresis system) | TaKaRa | Model: AD110 | |
| NotI | New England Biolabs | R3189 | 20,000 units/mL |
| Oligo explorer program | GeneLink | For primer design | |
| Optical tube strip caps (8x Strip) | Agilent Technologies | 401425 | For real time PCR |
| Opti-MEM | Gibco | 31985-070 | 500 Ml |
| PANC-1 cells | ATCC | CRL-1469 | |
| Penicillin/streptomycin | Gibco | 15140-122 | 100 mL |
| Phosphate buffer saline | Gibco | 14040117 | 1000 mL |
| Plasmid DNA miniprep S& V kit | Bionics | DN10200 | 200 prep |
| PrimeSTAR GXL DNA polymerase | TaKaRa | R050A | 250 units |
| Shaker | TECAN | Shaking platform | |
| Shaking incubator | Labtech | Model: LSI-3016A | |
| Sigmaplot 14 software | Systat Software Inc | For dose-response curve generation | |
| Sulforhodamine B powder | Sigma | S1402-5G | 5 g |
| SYBR green master mix | Smobio | TQ12001805401-3 | Binding fluorescent dye for dsDNA |
| T4 DNA ligase | TaKaRa | 2011A | 25,000 U |
| TaqMan master mix | Applied Biosystems | 4324018 | 200 reactions, no AmpErase UNG |
| TaqMan microRNA assay (hsa-miR-107) | Applied Biosystems | 4427975 | Assay ID: 000443 (50RT, 150 PCR rxns) |
| TaqMan microRNA assay (hsa-miR-301) | Applied Biosystems | 4427975 | Assay ID: 000528 (50RT, 150 PCR rxns) |
| TaqMan miR RT kit | Applied Biosystems | 4366597 | 1,000 reactions |
| Thermo CO2 incubator (BB15) | ThermoFisher Scientific | 37 °C and 5% CO2 incubation | |
| Trichloroacetic acid | Sigma | 91228-100G | 100 g |
| Trizma base | Sigma | T4661-100G | 100 g |
| Ultrapure water | Invitrogen | 10977-015 | 500 mL |
| Veriti 96 well thermal cycler | Applied Biosystems | For amplification of DNA (or cDNA) | |
| XhoI | New England Biolabs | R0146 | 20,000 units/mL |
참고문헌
- He, L., Hannon, G. J. MicroRNAs: small RNAs with a big role in gene regulation. Nature Reviews Genetics. 5 (7), 522-531 (2004).
- Park, J. K., Doseff, A. I., Schmittgen, T. D. MicroRNAs Targeting Caspase-3 and -7 in PANC-1 Cells. International Journal of Molecular Sciences. 19 (4), (2018).
- Park, J. K., et al. MicroRNAs-103/107 coordinately regulate macropinocytosis and autophagy. Journal of Cell Biology. 215 (5), 667-685 (2016).
- Henry, J. C., et al. miR-199a-3p targets CD44 and reduces proliferation of CD44 positive hepatocellular carcinoma cell lines. Biochemical and Biophysical Research Communications. 403 (1), 120-125 (2010).
- Hoefert, J. E., Bjerke, G. A., Wang, D., Yi, R. The microRNA-200 family coordinately regulates cell adhesion and proliferation in hair morphogenesis. Journal of Cell Biology. 217 (6), 2185-2204 (2018).
- Anfossi, S., Fu, X., Nagvekar, R., Calin, G. A. MicroRNAs, Regulatory Messengers Inside and Outside Cancer Cells. Advances in Experimental Medicine and Biology. 1056, 87-108 (2018).
- Khoshinani, H. M., et al. Involvement of miR-155/FOXO3a and miR-222/PTEN in acquired radioresistance of colorectal cancer cell line. Japanese Journal of Radiology. 35 (11), 664-672 (2017).
- Gao, Y., et al. MicroRNA-155 increases colon cancer chemoresistance to cisplatin by targeting forkhead box O3. Oncology Letters. 15 (4), 4781-4788 (2018).
- Catanzaro, G., et al. Loss of miR-107, miR-181c and miR-29a-3p Promote Activation of Notch2 Signaling in Pediatric High-Grade Gliomas (pHGGs). International Journal of Molecular Sciences. 18 (12), (2017).
- Akbari Moqadam, F., Pieters, R., den Boer, M. L. The hunting of targets: challenge in miRNA research. Leukemia. 27 (1), 16-23 (2013).
- Brown, R. A. M., et al. Total RNA extraction from tissues for microRNA and target gene expression analysis: not all kits are created equal. BMC Biotechnology. 18 (1), (2018).
- Kim, Y. K., Yeo, J., Kim, B., Ha, M., Kim, V. N. Short structured RNAs with low GC content are selectively lost during extraction from a small number of cells. Molecular Cell. 46 (6), 893-895 (2012).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Park, J. K., Seo, J. S., Lee, S. K., Chan, K. K., Kuh, H. J. Combinatorial Antitumor Activity of Oxaliplatin with Epigenetic Modifying Agents, 5-Aza-CdR and FK228, in Human Gastric Cancer Cells. Biomolecules & Therapeutics. 26 (6), 591-598 (2018).
- Xia, X., et al. Downregulation of miR-301a-3p sensitizes pancreatic cancer cells to gemcitabine treatment via PTEN. American Journal of Translational Research. 9 (4), 1886-1895 (2017).
- Lee, K. H., et al. Epigenetic silencing of MicroRNA miR-107 regulates cyclin-dependent kinase 6 expression in pancreatic cancer. Pancreatology. 9 (3), 293-301 (2009).
- van Tonder, A., Joubert, A. M., Cromarty, A. D. Limitations of the 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-2H-tetrazolium bromide (MTT) assay when compared to three commonly used cell enumeration assays. BMC Research Notes. 8, 47(2015).
- Wang, P., Henning, S. M., Heber, D. Limitations of MTT and MTS-based assays for measurement of antiproliferative activity of green tea polyphenols. PloS One. 5 (4), e10202(2010).
- Wu, L., Belasco, J. G. Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs. Molecular Cell. 29 (1), 1-7 (2008).
- Jin, Y., Chen, Z., Liu, X., Zhou, X. Evaluating the microRNA targeting sites by luciferase reporter gene assay. Methods in Molecular Biology. , 117-127 (2013).
- Ma, Z., et al. Gamma-synuclein binds to AKT and promotes cancer cell survival and proliferation. Tumour Biology. 37 (11), 14999-15005 (2016).
- Pan, Z. Z., Bruening, W., Giasson, B. I., Lee, V. M., Godwin, A. K. Gamma-synuclein promotes cancer cell survival and inhibits stress- and chemotherapy drug-induced apoptosis by modulating MAPK pathways. Journal of Biological Chemistry. 277 (38), 35050-35060 (2002).
- Martinez-Sanchez, A., Murphy, C. L. MicroRNA Target Identification-Experimental Approaches. Biology (Basel). 2 (1), 189-205 (2013).
- Lee, E. J., et al. Expression profiling identifies microRNA signature in pancreatic cancer. International Journal of Cancer. 120 (5), 1046-1054 (2007).
- Nuovo, G. J., et al. A methodology for the combined in situ analyses of the precursor and mature forms of microRNAs and correlation with their putative targets. Nature Protocols. 4 (1), 107-115 (2009).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44 (1), 31-38 (2008).
- Diederichs, S., Haber, D. A. Dual role for argonautes in microRNA processing and posttranscriptional regulation of microRNA expression. Cell. 131 (6), 1097-1108 (2007).
- Orellana, E. A., Kasinski, A. L. Sulforhodamine B (SRB) Assay in Cell Culture to Investigate Cell Proliferation. Bio Protocol. 6 (21), (2016).
- Lawrie, C. H. MicroRNAs in hematological malignancies. Blood Reviews. 27 (3), 143-154 (2013).
- Quah, B. J., Warren, H. S., Parish, C. R. Monitoring lymphocyte proliferation in vitro and in vivo with the intracellular fluorescent dye carboxyfluorescein diacetate succinimidyl ester. Nature Protocols. 2 (9), 2049-2056 (2007).
- Xing, Z., Li, D., Yang, L., Xi, Y., Su, X. MicroRNAs and anticancer drugs. Acta Biochimica et Biophysica Sinica. 46 (3), 233-239 (2014).
- Moeng, S., et al. MicroRNA-107 Targets IKBKG and Sensitizes A549 Cells to Parthenolide. Anticancer Research. 38 (11), 6309-6316 (2018).
- Chou, T. C. Drug combination studies and their synergy quantification using the Chou-Talalay method. Cancer Research. 70 (2), 440-446 (2010).
- Flamand, M. N., Gan, H. H., Mayya, V. K., Gunsalus, K. C., Duchaine, T. F. A non-canonical site reveals the cooperative mechanisms of microRNA-mediated silencing. Nucleic Acids Research. 45 (12), 7212-7225 (2017).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유