Method Article
RNA 차세대 염기 서 열 분석 및 생물 정보학 파이프라인은 궤적 특이 수준에서 발현 된 라인-1s를 식별 한다
요약
여기에서, 우리는 궤적 특정 수준에서 라인 1 발현을 식별 하기 위해 생물 학적 접근법 및 분석을 제시 한다.
초록
긴 산재 요소-1 (라인/L1s)은 게놈 불안정성 및 돌연변이 유발을 초래 하는 유 전체에 복사 하 고 무작위로 삽입할 수 있는 반복적인 원소입니다. 개별 수준에서 L1 좌 위의 발현 패턴을 이해 하는 것은이 돌연 변이 요소의 생물학에 대 한 이해를 빌려 줄 것입니다. 이 자율 요소는 99%가 잘리고 결함이 있지만 50만 이상의 복사본을 가진 인간 게놈의 상당 부분을 차지 합니다. 그러나, 그들의 풍부 하 고 지배적 인 결함 사본의 수는 다른 유전자의 일부로 서 표현 된 L1 관련 서 열에서 진정으로 표현 된 L1s을 식별 하는 것을 어렵게 만든다. 또한 요소의 반복적인 특성으로 인해 어떤 특정 L1 궤적을 표현 하는지 확인 하는 것도 쉽지 않습니다. 이러한 어려움을 극복 하기 위해, 우리는 궤적 특정 레벨에서 L1 발현을 식별 하는 RNA-서 열 생물 학적 접근법을 제시 한다. 요약 하자면, 우리는 세포질 rna를 모으고, 폴 리아 데 화 전사체를 선택 하 고, 가닥 특이 적 rna-서 열 분석을 활용 하 여 인간 기준 게놈에서 L1 좌 위에 읽기를 고유 하 게 매핑합니다. 우리는 시각적으로 자신의 프로모터에서 전사를 확인 하 고 각 개별 L1 궤적의 mappability에 대 한 계정으로 매핑 된 전사체 읽기를 조정 하는 고유 하 게 매핑된 읽기와 각 L1 궤적을 선별. 이러한 접근법은 소수의 전체 길이 L1 원소 로부터 발현을 검출 하는이 프로토콜이 프로토콜의 능력을 입증 하기 위해 DU145, 전립선 종양 세포 라인에 적용 되었다.
서문
레트로바이러스는 RNA 중간체를 통해 카피 앤 페이스트 메커니즘으로 게놈에서 "점프" 할 수 있는 반복적인 DNA 요소입니다. 레트로바이러스의 한 서브 세트는 긴 산재 요소-1 (라인/L1s)으로 알려져 있으며, 500 개 이상의 복사본1로 인간 게놈의 여섯째를 만든다. 그들의 풍요로 움에도 불구 하 고, 이러한 사본의 대부분은 결함 및 활성 것으로 생각 하는 추정 80-120 L1 요소와 잘립니다2. 전체 길이 L1은 5 ' 및 3 ' 번역 되지 않은 영역, 내부 프로모터 및 관련 안티 센스 프로모터, 2 개의 비중첩 열린 판독 프레임 (orfs) 및 신호와 polya 꼬리를 가진 길이 약 6kb이 고,5 . 인간에서, L1s는 나이가 많은 가족 들에 비해 시간이 지남에 따라 더 독특한 서 열 돌연변이를 축적 한 것으로 진화 시대에 의해 구별 되는 하위 가족 들로 이루어져 있다 L1HS6,7. L1s는 유일 하 게 자율적이 고, 인간 레트로바이러스 이며 그들의 ORFs는 역 역전사, 효소, 및 RNPs를 RNA 결합 및 샤 페론과 함께 해독 하 고 게놈에 삽입 하는 데 필요한 활동을 표적으로 하는 과정에서 해독 한다 역 전사8,9,10,12.
L1s의 재 변이는 이식 돌연변이 유발, 표적 부위 삭제 및 재배열을 포함 하는 다양 한 메카니즘에 의해 인간 생식 계열 질환을 야기 하는 것으로 보고 되었으며,13,14 , 16. 최근 L1s는 다양 한 상피 암에서 관찰 되 고 있는 돌연 변이 원소의 발현 및 삽입 사건 증가에 따라 발암 및/또는 종양 진행에 역할을 할 수 있다는 가설을가지고 있다17,18 . 모든 200 출산19에 하나의 새로운 L1 삽입이 있는 것으로 추정 된다. 따라서, 활발 하 게 발현 하는 L1s의 생물학을 더 잘 이해 하는 것이 필수적 이다. 다른 유전자의 전사체 내에서 발견 되는 반복 되는 본질과 불완전 한 사본의 풍부 함은 이러한 수준의 분석을 어렵게 만들었습니다.
다행히도, 높은 처리량 시퀀싱 기술의 출현으로, 분석을 통해 L1s 특정 수준에서 진정으로 표현 하는 것을 식별 하기 위해 진전을 이루었습니다. RNA 차세대 염기 서 열 분석을 사용 하 여 발현 된 L1s를 가장 잘 식별 하는 방법에 대 한 다른 철학이 있습니다. 궤적 특정 수준에서 L1 전사체를 맵핑하는 데 제안 된 두 가지 합리적인 접근법만이 있었다. 하나는 L1 폴 리아 데 닐 화 신호와 플 랭킹 시퀀스 (20)를 통해 판독 하는 잠재적 인 전사에만 초점을 맞추고 있다. 우리의 접근은 L1 요소 사이의 작은 시퀀스 차이를 활용 하 고 유일 하 게 하나의 궤적21에 매핑되는 RNA-서 열 판독을 매핑합니다. 이러한 방법 모두는 성적 증명서의 정량화 측면에서 한계가 있다. 정량화는 각각의 L1 궤적 (21)의 ' 고유 mappability '에 대 한 보정을 추가 하거나 특정 궤적 (22)에 고유 하 게 맵핑 될 수 없었던 다중 매핑 판독을 재분배 하는 보다 복잡 한 알고리즘을 사용 하 여 잠재적으로 개선 될 수 있다. 여기서, 우리는 궤적 특이 수준에서 발현 된 L1 원소를 동정 하기 위해 단계별로 RNA 추출 및 차세대 염기 서 열 분석 및 생물 정보학 프로토콜을 상세히 설명 한다. 우리의 접근 방식은 기능 L1 요소의 생물학에 대 한 우리의 지식을 최대한 활용 합니다. 이것은 기능 L1 요소가 l1 요소의 시작 부분에서 시작 되는 l1 프로모터 로부터 생성 되어야 한다는 것을 알고 포함 하 고, 세포질에서 번역 되어야 하 고 그의 전사체는 게놈과 공동 선형 이어야 한다. 간단히, 우리는 신선 하 고, 세포질 rna를 수집 하 고, 폴 리아 데 화 전사체를 선택 하 고, 가닥 특이 적 rna-서 열 분석을 활용 하 여 인간 기준 게놈에서 L1 좌 위에 읽기를 고유 하 게 매핑합니다. 이러한 정렬 된 읽기는 여전히 광범위 한 수동 큐 레이 션을 요구 하 여, 진정으로 표현 된 L1으로 궤적을 지정 하기 전에 L1 프로모터 로부터 전사체 읽기가 시작 되는지 확인 합니다. 우리는 DU145 전립선 종양 세포 선 샘플에이 접근을 적용 하 여 비활성 사본의 질량에서 상대적으로 몇 개의 활발 하 게 전사 된 L1 멤버를 식별 하는 방법을 보여줍니다.
프로토콜
1. 세포질 RNA 추출
- 다음 방법을 통해 세포를 얻습니다.
- 100% 콘 유창 함, T-75 플라스 크에서 살아있는 세포를 수집 하십시오.
- 차가운 PBS 5Ml에서 플라스 크를 2 회 세척 하 고 마지막으로 세척 한 후에 긁어 내어 15 mL 원뿔형 튜브로 전달 한다. 1000 x g 및 4°c에서 2 분 동안 원심 분리 하 고 상층 액 (재료 표)을 조심 스럽게 제거 하 고 버립니다.
- 조직 표본에서 세포를 수집 합니다.
- 해 부 되는 것에서 1 시간 이내에 세포질 RNA 추출을 위한 조직을 준비 하 고 항상 얼음에 유지. 장기 저장을 위해, RNA 억제제 해결책은 제조자의 프로토콜 (물자의 테이블)에 따라 해 부 후에 최대 72 시간 동안 조직을 저장 하기 위하여 사용 합니다.
- 주사위 10 µm3 개의 시료를 멸 균 된 증발 기에 5 ml의 차가운 PBS로 균질 화 하 고 15 ml 원뿔형 튜브로 이송 한 후 4°c에서 1000 x g 에서 2 분간 원심 분리 하 고 상층 액을 조심 스럽게 제거 하 고 삭제 합니다 (재질 표 < a0/>). /c8 >).
- 100% 콘 유창 함, T-75 플라스 크에서 살아있는 세포를 수집 하십시오.
- 세포 펠 렛에 용 해 완충 액 2 mL를 넣고 얼음에 5 분간 혼합 하 여 배양 합니다.
- 150 mm 염화 나트륨, 50 mm 7.4 헵 digitonin 및 25 μ g/m l/밀리 리터 (재료 표)로 신선한 용 해 완충 제를 준비 합니다.
- 혈장 막에 침투 하는 데 필요한 용 해 완충 액에서의 digitonin의 최소 농도가 세포 유형에 따라 변할 수 있으므로, 세포 용 해 완충 액으로 처리 된 셀이 혈장 막을 잃고 온전한 핵 막을 유지 한다는 것을 현미경 적으로 확인 한다.
- 사용 직전에 1000 U/m l RNase 억제제 (재료 표)를 추가 하십시오.
- 1000 x g 및 4°c에서 1 분 동안 원심 분리 하 고, 상층 액을 수집 한다.
- 상층 액을 추가 하 여 트리 졸 7.5 mL와 클로로 포 름의 1.5 mL를 미리 냉장 시켰다. 클로로 포 름을 필요로 하는 모든 단계는 깨끗 한 화학 후드 (재료의 테이블) 내부에서 수행 해야 합니다.
- 3220 x g 및 4°c에서 35 분 동안 원심 분리기.
- 수성 부분 (상부 층)을 신선 하 게 냉장 된 15 mL 튜브로 이송 한다.
- 클로로 포 름과 소용돌이 4.5 mL를 추가 합니다.
- 3220 x g 및 4°c에서 10 분 동안 원심 분리기.
- 수성 부분을 신선 하 게 냉각 된 튜브로 이송 하십시오.
- 4.5 mL의이 소 프로 판 올을 넣고 잘 흔들어 밤새-80 ° c (재료 표)에서 배양 합니다.
- 3220 x g 및 4°c에서 45 분간 원심 분리기.
- 이 소 프로 판 올을 제거 하 고, 100% 에탄올 15 mL를 추가 합니다 (재료 표).
- 10 분 동안 3220 x g 에서 원심 분리기.
- 에탄올을 제거 하 고 물기를 제거한 후 약 1 시간 동안 건조 시킵니다.
- 멸 균 된 면봉을 사용 하 여 남아 있는 에탄올 (재료 표)을 모두 닦아 냅니다.
- 펠 릿 크기 (재료 표)에 따라 rnase가 없는 물에 100 μ l의 샘플 200을 다시 일시 중단 합니다.
- 전기 영동 기술을 사용 하 여 샘플을 석은 제조업체의 제품23 (재료 표)에 따라 시료의 품질과 농도를 결정 합니다.
- 시료는 RNA-서 열 분석에 적합 합니다. 린 > 824.
2. 차세대 염기 서 열 분석
- 적어도 5000만 쌍 단 100 bp 판독을 생성 하기 위한 차세대 염기 서 열 분석 플랫폼을 사용 하 여 시퀀싱 될 세포질 RNA 샘플을 제출 한다.
- 폴 리-데 니 화 RNAs 및 스트랜드 특정 시퀀싱을 선택 합니다.
3. 주석 작성 (기존 주석이 있는 경우 선택 사항)
- 전체 길이 L1 어노테이션을 작성 하거나 전체 길이 L1 어노테이션을 다운로드 합니다 (보조 파일 1a).
- 테이블 브라우저 도구 (https://genome.ucsc.edu/cgi-bin/hgTables)와 UCSC 게놈 브라우저에서 라인-1 요소에 대 한 반복 Masker 주석을 다운로드 합니다. 포유동물 clade, 인간 게놈, hg19 어셈블리 (또는 더 업데이트 된 게놈에 대 한 hg38)를 지정 하 고 클래스 이름 아래에 "LINE1"를 필터링 합니다. FL-L1-BLAST로. gtf 파일 및 레이블로 다운로드 하십시오.
- 인간 게놈 내의 프로모터 영역을 포괄 하는 L 1.3 전장 L1의 제 1 300 bp의 로컬 블 라스트 검색을 실행 하 고 6000 bp 다운스트림을 추가 하 여 주석 파일에 L1 좌표의 끝을 생성 한다. Gtf 파일에 저장 하 고 FL-L1-RM로 라벨을 지정 합니다.
- 를 사용 하 여 반복 Masker 주석과 프로모터 기반 L1 주석과 FL-L1-BLAST_RM (소프트웨어 패키지)로 레이블을 교차 시킵니다.
- 리눅스 터미널에서이 명령을 사용 하 여: 베드 도구 교차-FL-L1-BLAST FL-L1-RM > FL-L1-BLAST_RM .
- 교차 된 FL-L1 주석을 위쪽과 아래쪽 가닥으로 분리 합니다.
- FL-L1-BLAST_RM를 스프레드시트 소프트웨어에 복사 하 고 ' 마이너스 '와 ' 더하기 ' 가닥으로 정렬 한 다음, 염색체 위치로 정렬 합니다.
- 두 개의 새 스프레드시트 문서를 작성 하 고, 하나는 마이너스 가닥의 전체 길이 L1s 교차 하는 좌표와 하단 가닥에 하나씩, FL-L1-BLAST_RM_minus와 FL-L1-BLAST_RM_plus로 저장 합니다.
- 두 개의 새 문서를 .txt 파일로 저장 합니다.
- Mac2unix 프로그램을 사용 하 여 .txt 파일을 올바른 주석 파일 (소프트웨어 패키지)으로 변환 합니다.
- 터미널에서이 명령을 사용 하 여: Mac2unix.sh FL-L1-BLAST_RM_minus .
- 터미널에서이 명령을 사용 하 여: Mac2unix.sh FL-L1-BLAST_RM_plus .
- .Gff 확장자를 가진 새 파일을 저장 합니다.
- 또는 AWK를 사용 하 여 + 및 – 가닥과 연관 된 행을 필터링 할 수 있습니다.
- 다음 명령을 사용 하 여 + 가닥: awk '/+/' FL-L1_BLAST_RM > FL-L1_BLAST_RM_plus를 가져옵니다.
- 다음 명령줄을 사용 하 여-가닥: awk '//' FL-L1_BLAST_RM > FL-L1_BLAST_RM_minus를 가져옵니다.
4. 정렬 파이프라인을 판독 하 여 표현 된 L1s 식별
| 옵션 | 설명 |
| – p | 이는 컴퓨터에서 정렬을 실행 하는 데 사용 해야 하는 스레드 수를 자세히 설명 합니다. 컴퓨터 메모리가 클수록 더 많은 스레드가 허용 되 고 경험적으로 d가 되어야 합니다. |
| – m 1 | 이것은 다른 게놈 일치 보다 더 나은 게놈에서 하나의 일치가 있는 읽기를 허용 하는 프로그램을 알려줍니다. |
| -y | 이는 모든 가능한 일치 항목에 대해 매핑 검색을 수행 하 고 고정 된 개수의 일치에 도달한 후에 종료를 허용 하지 않는 tryhard 스위치입니다. |
| – v 3 | 이것은 단지 게놈에 3 개 이하의 불일치를 가진 매핑된 읽기를 위해 메모리를 활용 하는 프로그램을 허용 합니다. |
| – X 600 | 이는 서로 600 베이스 내에 매핑되는 쌍으로 된 읽기만 허용 합니다. 이것은 읽기 쌍이 게놈에 있는 공동 선형이 고 처리 한 RNA 분자를 관련 시키는 s에 대하여 선택 합니다. |
| – 천 kmbs 8184 | 이 명령은 각 L1 관련 읽기에 대해 가능한 많은 양의 정렬을 처리 하기 위한 추가 메모리를 할당 합니다. |
표 1: 보우 타이에 대 한 명령줄 옵션.
- 보 타이를 사용 하 여 관심 있는 RNA-서 열 샘플을 사용 하 여 얼라인 먼 트-말단 시퀀싱 fastq 파일을 실행 합니다.
참고: 고유 정렬에 필요한 매개 변수가 특히이 버전의 bowtie (소프트웨어 패키지) 에서만 발견 되기 때문에 Bowtie1를 사용 하 고 Bowtie2 하지 않아야 합니다. Bowtie는 L1 생물학 및 표현과 관련 된 매 염, 연속 읽기를 평가 하기 위해 스타와 같은 스플라이스 인식 얼 라이너를 통해 사용 됩니다.- 리눅스 터미널에서이 커맨드 라인을 사용 하 여: 보 타이-10m1-s600 y-hg_X_Y_M_index의 8184 hg_sample_1 hg_sample_2. | 삼 툴 스 정렬-hg_sample_sorted를 사용 하는 것이 더 나을 것입니다. 보우 타이의 명령행 옵션에 대 한 설명은 표 1 의 내용을 참조 하십시오.
- 스트랜드는 samtools (소프트웨어 패키지) 및 다음 Linux 명령을 사용 하 여 출력 bam 파일을 분리 합니다. 표준 차세대 시퀀싱 프로토콜을 사용 하지 않는 경우 실제 플래그 값이 다를 수 있습니다.
- 이 명령행을 사용 하 여 맨 위 가닥: 삼 도구 뷰-h hg_sample_sorted를 선택 합니다. | $2 = = 163 $2 83 | 삼 도구 뷰-bS->의 hg_sample_sorted_topstrand입니다. "3. [3]" [3] [1]
- 이 명령행을 사용 하 여 맨 아래 가닥에 대해 선택 합니다. 삼 도구 뷰-h hg_sample_sorted | | $2 = = 147 $2 99 | 삼 도구 뷰-bS-> hg_sample_sorted_bottomstrand를 표시 하는 것입니다. 2.1) "[3]
- Bedtools (소프트웨어 패키지)를 사용 하 여 L1 좌 위의 주석에 대 한 읽기 카운트를 생성 합니다.
- 이 명령행을 사용 하 여 상단 가닥의 감지 방향에서 L1s에 대 한 읽기 카운트를 생성 합니다. bedtools 범위-abam FL-L1-BLAST_RM_plus hg_sample_sorted_topstrand bam > hg_sample_sorted_bowtie_tryhard_plus_top.
- 이 명령행을 사용 하 여 아래쪽 가닥의 감지 방향으로 L1s에 대 한 읽기 카운트를 생성 합니다. bedtools 범위-abam FL-L1-BLAST_RM_minus hg_sample_sorted_bottomstrand bam > hg_sample_sorted_bowtie_tryhard_minus_bottom.
- 5.1.1 단계에서 bam 파일을 인덱싱하면 통합 유전체학 뷰어 (IGV)25 (소프트웨어 패키지)에서 볼 수 있습니다.
- 다음 명령줄을 사용 합니다. 삼 도구 색인 hg_sample_sorted
- 배치 모드를 사용 하 여 한 번에 통과 하는 RNA-서 열 샘플의 수를 증가 시키기 위해, 슈퍼 컴퓨터 스크립트를 사용 하 여 human_bowtie 라는 4.1 단계를 완료 하 고 4.2-4.3 단계를 완료 하는 스크립트를 human_L1_pipeline 라고 하며 스크립트를 작성 하 여 완료 합니다. 4.4 단계는 bam_index 라는 생성 되었습니다. 이러한 스크립트는 추가 파일 2 에서 스크립트를 실행 하기 위한 관련 슈퍼 컴퓨터 명령과 함께 찾을 수 있습니다.
5. 수동 큐 레이 션
- 주석이 추가 된 각 L1 궤적에 매핑된 읽기에 대 한 스프레드시트를 생성 합니다.
- 4.3.2 단계와 라벨 페이지에서 생성 된 hg_sample_sorted_bowtie_tryhard_minus_bottom를 "마이너스-하단"으로 복사 합니다.
- J 열에서 찾은 가장 높은 읽기 수를 기준으로 모든 열을 정렬 합니다.
- 4.3.1 단계에서 만든 hg_sample_sorted_bowtie_tryhard_plus_top를 통해 다른 스프레드 시트에서 "탑 플러스"로 라벨을 복사 합니다.
- J 열에서 찾은 가장 높은 읽기 수를 기준으로 모든 열을 정렬 합니다.
- ' 결합 '으로 표시 된 세 번째 페이지를 만들고 ' 마이너스 하단 ' 및 ' 플러스 톱 ' 페이지에서 10 개 이상의 읽기를 사용 하 여 모든 좌 위를 추가 합니다.
- J 열에서 찾은 가장 높은 읽기 수를 기준으로 모든 열을 정렬 합니다.
- IGV25 (소프트웨어 패키지)에 다음 파일을 로드 합니다. 1) 주석 유전자를 시각화 하기 위해 관심 있는 참조 게놈, 2) FL-L1-BLAST_RM L1 주석을 시각화 하기 위해, 3) hg_sample_sorted에서 매핑된 자막을 시각화 4) hg_genomicDNA_sorted는 게놈 영역의 mappability를 평가 합니다.
- 각 bam 파일과 관련 된 적용 범위 및 연결 행을 제거 합니다.
- Hg_sample_sorted 및 hg_genomicDNA_sorted를 압축 하 여 모든 IGV 트랙이 한 화면에 맞도록 합니다.
- 4.3.2 단계와 라벨 페이지에서 생성 된 hg_sample_sorted_bowtie_tryhard_minus_bottom를 "마이너스-하단"으로 복사 합니다.
- 수동 선별.
- 스프레드시트 "결합" 페이지에 나열 된 좌 위에서 좌표를 사용 하 여 igv25 (소프트웨어 패키지)에서 좌 위 라고 하는 보기.
- 최대 5 킬로바이트의 L1 방향으로 업스트림 읽기가 없는 경우, 진정한 자체를 표현 하는 궤적을 선별 합니다.
- 행에 녹색을 레이블로 지정 하 고 왜 그것이 확실 하 게 표현 된 L1 인지 확인 합니다.
참고: L1의 지역 업스트림이 매핑 불가능 한 경우이 규칙에 대 한 예외가 존재 합니다. 이 경우에는 빨간색을 컬러로 라벨을 지정 하 고 L1 프로모터의 상류 영역의 발현을 평가할 수 없으므로 L1's 표현식은 자신 있게 결정할 수 없습니다.
- 행에 녹색을 레이블로 지정 하 고 왜 그것이 확실 하 게 표현 된 L1 인지 확인 합니다.
- 최대 5kb의 업스트림 읽기가 있는 경우, 궤적을 선별 하는 것은 진정으로 자체 프로모터를 표현 하지 않습니다.
- 행 빨간색 레이블을 컬러로 표시 하 고 왜 확실 하 게 표현 된 L1이 아닙니다.
- 궤적을 읽는 것과 동일한 방향으로 발현 된 유전자의 인 트 론 내로 발현 되는 경우에는, l1의 상류를 읽는 것과 동일한 방향으로 표현 유전자의 하류에, 또는 주석이 없는 발현 패턴의 경우에는 거짓으로 유전자를 선별 한다 L1의 광고 업스트림.
참고:이 규칙에 대 한 예외는 L1 프로모터 시작 사이트와 직접적으로 겹치는 최소 읽기가 있지만 L1의 약간 업스트림에 적용 됩니다. 이 같은 L1 케이스의 다른 읽기 업스트림이 없는 경우,이 L1을 진정으로 표현 하는 것이 좋습니다. 행 녹색 색상에 레이블을 지정 하 고 왜 그것은 확실 하 게 표현 L1.
- 궤적에 대 한 매핑된 판독의 패턴이 mappability의 특정 L1's 영역과 상관 되지 않는 경우에, 거짓 될 가능성이 있는 L1 궤적을 선별.
참고: 예를 들어 l1이 매우 매핑 가능 하지만 l1 내에서 압축 된 영역에서 읽기 더미를가지고 있는 경우 자체 프로모터에서 l1 표현과 관련 될 가능성이 적고 엑손 또는 ltrs와 같은 주석이 없는 소스에서 발생 가능성이 더 높습니다. 이 같은 경우에, 오렌지로 좌 위를 선별 하 고 궤적 의심 되는 이유를 참고. UCSC 게놈 브라우저에서 L1 위치를 확인 하 여 의심 스러운 더미-업의 소스를 확인 합니다. - 유 전체를 산발적으로 표현 하는 게놈 환경 내에 있는 경우에는 궤적을 진정으로 표현 하지 않는다.
참고: 예를 들어, 읽기는 L1의 업스트림 10kb를 나타낼 수 있지만 모든 10kb 또는 그래서 매핑된 읽기 및 L1와 정렬 그 읽기 중 일부. 이러한 L1s는 자체 프로모터에서 발현 될 가능성이 적고, 주석이 없는 게놈 표현의 패턴으로 인해 매핑 읽기가 더 높습니다. 이 같은 경우에, 오렌지로 좌 위를 선별 하 고 궤적 의심 되는 이유를 참고.
6. 참조 게놈에서 mappability를 평가 하기 위한 정렬 전략 읽기 (기존에 정렬 된 게놈 DNA 데이터 셋이 있는 경우 선택 사항)
- 전체 게놈 DNA 시퀀스 파일을 다운로드 하 고. fq 파일로 변환
- 여기에서 찾을 수 있는 NCBI 웹 사이트로 이동: https://www.ncbi.nlm.nih.gov/sra
- 입력 Wgs헬 라 쌍의 끝.
- 군에 의해 결과아래 호모 사피엔스 선택 합니다.
- 쌍을 이루는 샘플을 선택 하 고 다음 샘플과 같이 100 이상의 bp를 사용 하 여 읽습니다. https://www.ncbi.nlm.nih.gov/sra/ERX457838
- 다음과 같이 실행 을 선택한 다음 메타 데이터 를 선택 하 여 읽기 길이를 확인 합니다. https://trace.ncbi.nlm.nih.gov/Traces/sra/?run=ERR492384
- 전체 게놈 DNA 서 열 데이터를 다운로드 하려면 Linux 터미널에서이 명령을 입력 하십시오 . 2.9.2-mac64/빈/프리페치-X 100G ERR492384
참고: SRA 툴킷 프리페치 함수는 NCBI 사이트 (소프트웨어 패키지)에서 발견 된 가입 번호 "ERR492384를 다운로드 합니다. "100G"은 다운로드 한 데이터의 양을 100 기가바이트에 제한 합니다. - Linux 터미널에서이 명령을 입력 하십시오: fastq 덤프--분할 파일 ERR492384
참고:이는 다운로드 한 게놈 DNA 데이터 셋을 두 개의 fastq 파일로 분할 합니다.
- 보우 타이를 사용 하 여 정렬을 실행 합니다.
- 정렬에 대 한 리눅스에서이 명령을 사용 하 여: 보 타이-p 10M1-s600 y-Hg_X_Y_M_index의 8184 hg_genomicDNA_1 hg_genomicDNA_2---| 삼 도구 보기-hbus-| 도구 정렬-hg_genomicDNA_sorted.
- 보우 타이 정렬 (소프트웨어 패키지)에 사용 되는 파라미터를 이해 하려면 4.1 단계를 참조 하십시오.
- Genomically 정렬 된 bam 파일을 다운로드 하 여 작성자 요청 시 사용할 수 있는 mappability를 평가 합니다.
- 정렬에 대 한 리눅스에서이 명령을 사용 하 여: 보 타이-p 10M1-s600 y-Hg_X_Y_M_index의 8184 hg_genomicDNA_1 hg_genomicDNA_2---| 삼 도구 보기-hbus-| 도구 정렬-hg_genomicDNA_sorted.
- 4.2.1 단계에서 bam 파일을 인덱싱하기 위해 samtools을 사용 하 여 IGV25 (소프트웨어 패키지)에서 볼 수 있도록 하 여 수동 큐 레이 션에 대해 알립니다.
- 리눅스에서이 커맨드 라인을 사용 하 여: 삼 도구 색인 hg_genomicDNA_sorted
- 각 L1 좌 위의 mappability 평가
- Bedtools 프로그램, FL-L1 어노테이션 및 정렬 된 게놈 서 열 데이터 (소프트웨어 패키지)를 사용 하 여 L1 좌 위에 고유 하 게 매핑된 읽기 수를 판별 하십시오.
- 리눅스에서이 커맨드 라인을 사용 하 여: 침대 도구 범위-abam FL-L1-BLAST_RM-b hg_genomicDNA_sorted bam ≫ L1_Mappability_hg_genomicDNA.
- 400 고유 읽기가 그것에 정렬 되 면 전체 커버리지 mappability을가지고 L1 궤적을 지정 합니다.
- 각 L1에 대 한 게놈 DNA 정렬 판독 400을 확장 하거나 축소 하는 데 필요한 요인을 결정 합니다.
- 개별적인 L1 궤적 매핑 가능성에 따라 발현의 스케일링 척도를 갖는 것으로, 단계 6.4.3에서 결정 된 인자 들을 곱 하 게 정렬 하는 RNA 전사체의 수는 4 ~ 5 항에서 확실 하 게 발현 L1s 결정 한다.
- Bedtools 프로그램, FL-L1 어노테이션 및 정렬 된 게놈 서 열 데이터 (소프트웨어 패키지)를 사용 하 여 L1 좌 위에 고유 하 게 매핑된 읽기 수를 판별 하십시오.
결과
도 1 에 도식 적으로 설명 된 단계 들이 인간 전립선 종양 세포 DU145에 적용 되었다. RNA 샘플은 사이토 플 라이 크를 준비 하 고, 폴 리-선택 된 가닥 특이 적, 쌍 단 프로토콜에서 시퀀싱 된 다음 세대 였다. 보우 타이를 사용 하 여, 페어-엔드 시퀀싱 파일은 다른 게놈 위치와 비교 하 여 쌍 단 판독을 하나의 게놈 위치와 더 잘 일치 시키는 유일한 일치만 허용 하도록 정렬 되었습니다. DU145 sequence 파일은 작성자 요청 시 사용할 수 있는 bam 파일을 만드는 인간 참조 게놈에 정렬 되었습니다. Bedtools을 사용 하 여 전체 길이 L1s에 매핑된 읽기 수에 DU145 가닥으로 분리 된 bam 파일에서 데이터를 추출 했습니다. 이러한 판독은 스프레드 시트에서 가장 큰 것부터 가장 작은 것까지 정렬 되었고 IGV의 각 L1 궤적 주위의 게놈 환경을 검사 하 여 수동으로 선별 하 여 진위를 확인 했습니다 (보조 표 1). 샘플이 진정으로 표현 되도록 선별 된 경우, 그것은 오른쪽 가장 열에 그것의 수용에 대 한 설명과 함께 색상 코드 녹색 이었다. 상기 방법 섹션에 기재 된 지침에 따라 진정으로 표현 되는 L1 좌 위의 예는 도 2a-b에 나타내 었 다. 샘플이 확실 하 게 표현 되는 것으로 거부 된 경우, 가장 오른쪽 열에 거부 이유가 있는 빨간색으로 색으로 구분 되었습니다. L1 좌 위의 예는 방법 섹션에 기재 된 자신의 다음의 가이드라인 이외의 프로모터 로부터의 발현 때문에 거부 된 것은 도 2의c-e 에 상세히 설명 되어 있다.
여기서, 온전한 프로모터 영역이 있는 전장 L1s 연구 되었다. 이러한 구분이 수행 되지 않으면 잘린 L1s에서 시작 되는 전사 노이즈의 큰 소스가 도입 됩니다. DU145에서의 잘린 L1s의 예는 도 3a-b 에 나타나 있으며,이는 RNA-서 열 판독을 고유 하 게 매핑된 것으로 확인 되었다. 그러나 IGV에서는, 그 전사체가 잘린 L1 으로부터 개시 되지 않았지만, 발현 된 유전자 로부터 또는 하류에서 L1 서 열을 포함 하는 것이 명백 하다.
전반적으로 DU145에서, 수동 큐 레이 션 후에 진정한 표현으로 거부 되는 전체 길이 l1 좌 위 및 읽기의 비율은 약 50%입니다 (보충 표 2) l1 매핑된 전사체 판독의 높은 수준을 시연 그렇지 않으면 수동 큐 레이 션 없이 거짓 긍정으로 기록 됩니다. 구체적으로, DU145에는 총 3152 읽기의 감지 방향으로 고유 하 게 매핑 된 판독을 갖는 전체 전장 L1 좌 위 114이 있었지만, 60 좌 위는 1879 판독으로 수동 큐 레이 션 후에 자신의 프로모터를 발현 하는 것으로 확인 되었다 ( 보조 표 1). 이는 세포질 mRNA를 선택 함으로써 L1 생물학과 무관 한 발현을 감소 시키기 위해 걸음 수를 취한 경우에도 마찬가지 이다. DU145에서 가장 높은 수준의 매핑된 전사체가 있는 궤적은 진정한 L1을 표현 하지 않았기 때문에 거부 되었습니다 (그림 4). 전반적으로 특정 l1 좌 위에 대 한 매핑된 전사체의 수는 수동 큐 레이 션 후에 진정으로 표현 된 대로 허용 되 고 거절 된 l1 좌 위 사이에서 유사 하 게 범위 (도 4).
수동 큐 레이 션 후에 고유 하 게 매핑하는 읽기의 수는 175에서 DU145 범위에서 특정 L1 좌 위를 표현 하 여 10 번의 읽기에서 임의로 선택 된 최소 컷오프를 읽습니다 (그림 5). L1s에 고유 하 게 매핑된 전사체 판독을 식별 하는 이러한 접근법은 발현을 정확 하 게 정량화 하는 능력을 제한 한다. 이를 고려 하기 위해, 각 궤적의 mappability에 따라 수정 계수가 만들어졌습니다. 이 수정 계수를 만들기 위해, 첫 번째 bedtools은 모든 전체 길이 L1 좌 위에 정렬 하 고 가장 낮은 매핑 된 전사체 읽기 (보조 )에서 그 좌 위에 맞춰진 헬 라는 게놈 bam 파일에서 고유 하 게 매핑된 읽기의 수를 추출 하는 데 사용 되었다 그림 1). 그것은 임의로 지정 되었다 400와 L1s 전체 커버리지 mappability 했다. 글 라이 게놈 시퀀싱 샘플에서 l1 궤적에 맵핑 할 수 있는 판독의 수는 400 읽기에 상대적으로 스케일링 되었고, 그 스케일링 된 숫자는 각각에 매핑된 읽기의 수에 곱 게 DU145의 l1 좌 위를 표현 하였다 (보조 표 2) . 예상 대로, mappability에 대 한 더 큰 수정 점수를 가진 L1 요소는 L1PA2 같은 젊은 하위 가족에서 왔다 (보충 표 2). 일단 각 궤적에서의 mappability 점수에 대 한 판독을 조정 하였으며, 대부분의 좌 위에 대 한 발현에 대 한 정량은 증가 하였다 (도 6). 고유 하 게 매핑되는 읽기의 수는 DU145의 mappability 교정으로 특정 L1 좌 위를 표현 했다 612에서 4 읽기 및 가장 낮은 표현 좌 위의 재정렬이 있었다 (그림 6).
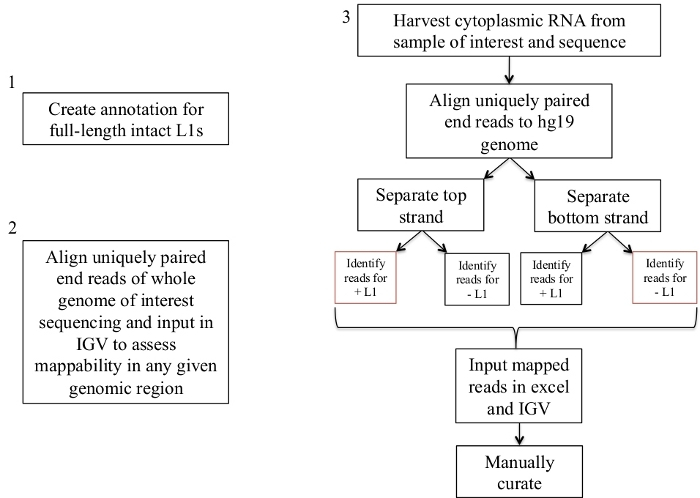
그림 1: 워크플로우 회로도
인간 샘플에서 발현 된 L1s를 식별 하는 단계는 그래픽으로 설명 된다. 적절 한 파일을 이미 사용할 수 있는 경우 1과 2 단계를 반복할 필요가 없습니다. 이러한 적절 한 파일은 보충 파일 1a-b 및 보충 파일 2에서 다운로드할 수 있습니다. 빨간색 상자는 동일한 감지 방향으로 L1s에 매핑하는 읽기 수를 계산 하기 위해 bedtools 커버리지 프로그램을 사용 하는 단계를 나타냅니다. 센스 지향 매핑 읽기와 이러한 좌 위 수동으로 큐레이터 해야 하는 L1s입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
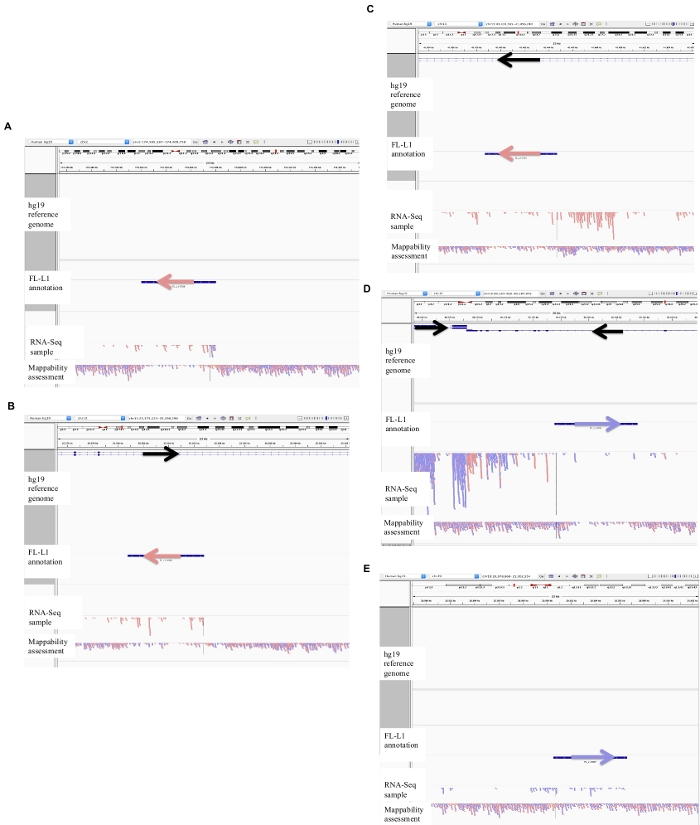
그림 2: DU145에서 큐 레이 팅 된 L1 좌 위의 예
IGV에 로드 된 참조 게놈은 레퍼런스 게놈 버전 (보충 파일 1)과 일치 하는 전체 길이 L1 gff 어노테이션 파일, DU145 bam 파일, 그리고 마지막으로 모든 저자에 게 사용 가능한 mappability를 평가 하는 게놈 라 헬 라 bam 파일입니다. 요청. 주석이 달린 L1의 방향을 시각화 하는 데 도움이 되도록 화살표가 추가 되었습니다. 빨간색 화살표와 읽기는 오른쪽에서 왼쪽으로 순서 대로 방향이 조정 됩니다. 화살표와 파란색의 읽기는 왼쪽에서 오른쪽으로 순서 대로 방향이 조정 됩니다. a) igv에서,이 l1 궤적은 5 kb를 초과 하는 센스 방향으로 l1의 상류가 판독 되지 않기 때문에 자체 프로모터를 발현 하는 것으로 보인다. 이 L1은 낮은 mappability를 가지 며, 유전자에는 없고, 예상 되는 안티 센스 프로모터 활성 (26)의 증거를 가진다. b) igv에서,이 l1 로커 스는 5kb 이상의 센스 방향으로 l1의 상류가 판독 되지 않기 때문에 자체 프로모터를 발현 하는 것으로 보인다. 이 L1은 낮은 mappability과 반대 방향의 유전자 내에 있다. c) igv에서,이 l1 궤적은 5kb 내에서 동일한 방향으로 업스트림 읽기가 존재 함에 따라 표현 된 l1으로 서 거부 되었다. 이 L1은 동일한 방향의 유전자 내에 있기 때문에 전사체의 판독은 발현 된 유전자의 프로모터 로부터 유래 되는 것이 가장 가능성이 있다. d) igv에서,이 l1 궤적은 5kb 내에서 동일한 방향으로 업스트림 읽기가 존재 함에 따라 표현 된 l1으로 서 거부 되었다. 이 L1은 동일한 방향으로 고도로 발현 된 유전자의 하류에 있어서, 전사체 판독은 가장 가능성이 발현 된 유전자의 프로모터 로부터 유래 하 고 정상적인 유전자 종결자를 넘어서 연장 된다. e) igv에서,이 l1 궤적은 5kb 내에서 동일한 방향으로 업스트림 읽기가 존재 함에 따라 표현 된 l1으로 서 거부 되었다. 이 L1은 참조 유전자 내의 주석이 달린 유전자 내에 있거나 그 근처에 있지 않기 때문에 L1 요소의 이러한 전사체 내 및 업스트림의 기원은 주석이 없는 프로모터를 제안 한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
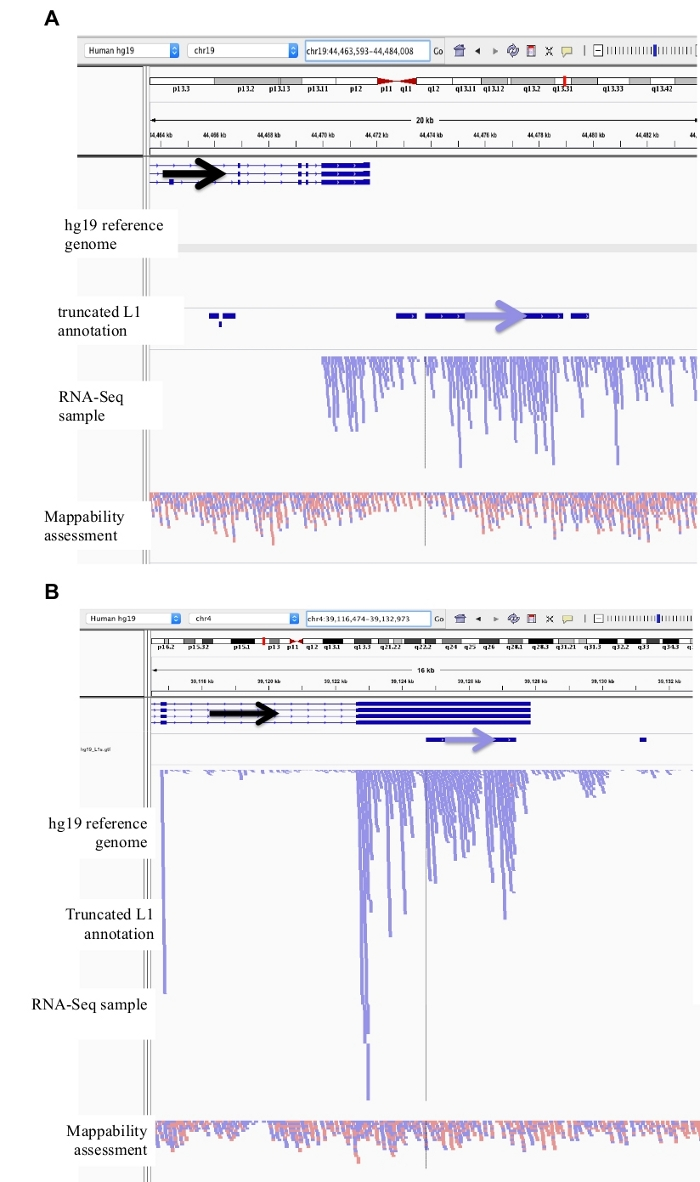
그림 3: 배경 소음은 잘린 L1s 발생 합니다.
L1 주석은 배경 소음의 주요 원인으로 잘린 L1s 포함 되지 않습니다. 주석이 달린 L1의 방향을 시각화 하는 데 도움이 되도록 화살표가 추가 되었습니다. 화살표와 파란색의 읽기는 왼쪽에서 오른쪽으로 순서 대로 방향이 조정 됩니다. a) 입증은 2706 BPS 인 L1MB5 sufamily에서 잘린 L1의 예입니다. IGV에서 읽기는 발현 된 유전자의 하류 확장에서 기인 한다는 것이 명백 하다. b)는 잘린 L1의 또 다른 예를 나타낸다. 이 L1은 4767 bp 길이의 L1PA11입니다. IGV에서는 l1을 고유 하 게 매핑하는 것은 l1이 내에 있는 표현 된 엑손에서 시작 된다는 것이 명백 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
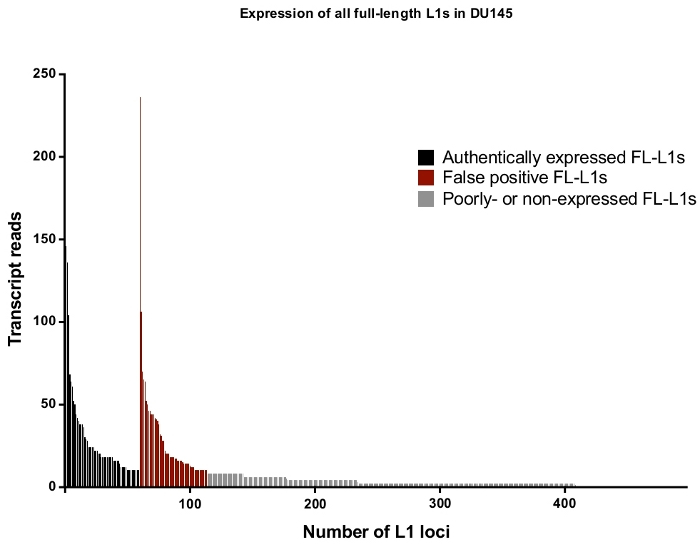
그림 4: DU145 전립선 종양 세포 대사에서 발현 되는 인간 게놈에서 온전한 전체 길이 그대로 L1s에 고유 하 게 매핑되는 전사체 판독.
블랙에서 특정 좌 위는 진정으로 수동 큐 레이 션 후에 표현 되 고 적색으로는 구체적인 좌 위는 수동 큐 레이 션 후에 진정으로 표현 된 판독으로 거부 되는 것으로 확인 되어야 한다. 회색은 각각에 매핑 10 개 미만의 읽기와 좌 위입니다. 이러한 좌 위는 자막 읽기의 작은 부분을 나타내므로 수동으로 선별 되지 않았습니다. X 축 눈금 표시는 모든 100 전체 길이, 손상 되지 않은 L1s 나타냅니다. 약 4500 좌 위는 매핑 읽기가 0 인 것 처럼 그래픽으로 표시 되지 않습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
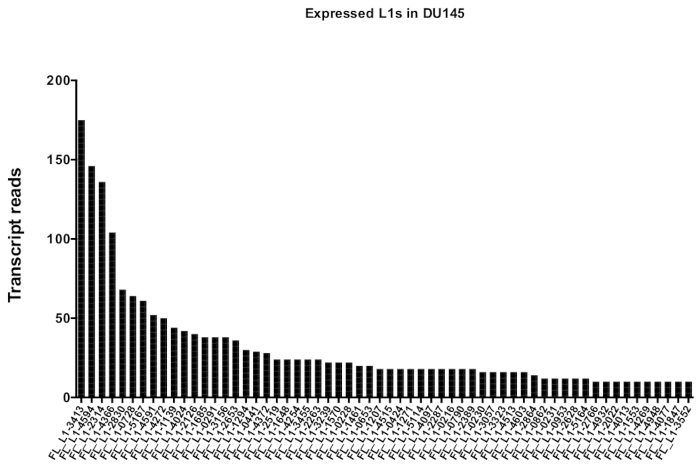
그림 5: DU145 전립선 종양 세포 대사에서 진정으로 전체 길이의 온전한 L1s을 표현 하는 것을 유일 하 게 매핑하는 전사체 읽기.
표시는 수동 큐 레이 션 후 DU145 세포에서 특정 좌 위에 매핑하는 전사체 판독의 숫자입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
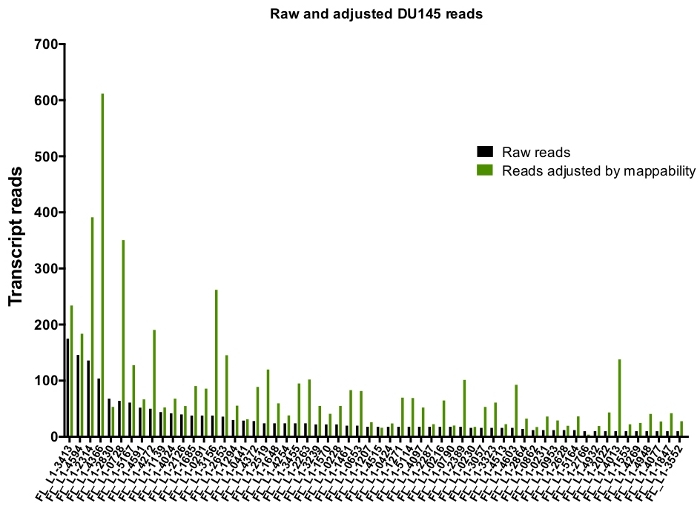
그림 6: 맵 읽기에 의해 조정 될 때, 확실 하 게 L1을 표현 하는 매핑.
DU145 셀에서 L1 좌 위를 수동으로 선별 하는 데 매핑되는 좌 위 별 mappability 점수로 조정 된 성적표 수입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
보조 파일 1: 방향에 따라 온전한 인간 L1s 전체 길이에 대 한 주석. a) FL-L1-BLAST_RM_minus. b) FL-L1-BLAST_RM_plus. 이 파일을 다운로드 하려면 여기를 클릭 하십시오.
보조 파일 2: 섹션 4에 자세히 설명 된 생물 정보학 파이프라인을 자동화 하는 데 사용 되는 슈퍼 컴퓨터 스크립트입니다. 이 파일을 다운로드 하려면 여기를 클릭 하십시오.
보조 그림 1: L1 mappability를 결정 하는 데 사용 되는 게놈 DNA 샘플.
표시 되는 것은 게놈에서 모든 5000 전장 L1 좌 위에 고유 하 게 매핑되는 헬 라 세포 라인 시료 로부터 게놈 전사체 판독의 수 이다. 400는 L1에 지도를 읽을 때 L1은 전체 커버리지 mappability을가지고 지정 되었다. 이 그림을 다운로드 하려면 여기를 클릭 하십시오.
추가 표 1: DU145에서 L1s의 수동 큐 레이 션. 이 표를 다운로드 하려면 여기를 클릭 하십시오.
보조 표 2: mappability 조정을 DU145에서 선별 된 L1s. 이 표를 다운로드 하려면 여기를 클릭 하십시오.
토론
L1 활성은 질병27,28,29에 기여 하는 유전적 손상 및 불안정성을 야기 하는 것으로 나타났다. 약 5000의 전체 길이 L1 사본 중, 수십 개의 진화 하는 젊은 L1s 대부분의 개조 활동2를 차지 합니다. 그러나, 증거가 있다 심지어 일부 오래 된, 소급 적 인 L1s는 여전히 단백질을 손상 DNA를 생산할 수 있는30. 게놈 불안정성 및 질병에서 L1s의 역할을 충분히 인식 하기 위해, 궤적 특이 수준에서 L1 발현이 이해 되어야 한다. 그러나 l1과 관련이 없는 다른 RNAs에 내장 된 L1-관련 서 열의 높은 배경은 진정한 L1 표현을 해석 하는데 중요 한 과제가 된다. 식별 하 고 따라서 개별 L1 좌 위의 표현 패턴을 이해 하는 또 다른 도전은 많은 짧은 읽기 시퀀스가 하나의 고유 한 궤적에 매핑하는 것을 허용 하지 않는 그들의 반복적인 성격 때문에 발생 합니다. 이러한 과제를 극복 하기 위해, 우리는 RNA-서 열 데이터를 사용 하 여 개별적인 L1 좌 위의 발현을 식별 하는 전술한 접근법을 개발 하였다.
우리의 접근법은 높은 레벨을 필터링 합니다 (99% 이상). L1의 서 열에서 생성 된 전사 노이즈의 여러 단계를 취하여 L1-전치와 무관 하다. 첫 번째 단계는 세포질 RNA의 준비를 포함 한다. 세포질 RNA에 대 한 선택 하 여, L1 관련 된 읽기 핵에서 발현 된 인 트로 닉 mRNA 내에서 발견은 크게 고갈. 시퀀싱 라이브러리 준비에서, L1s 관련이 없는 전사 노이즈를 감소 시키기 위해 취한 또 다른 단계는 폴 리아 데 닐 화 전사체의 선택을 포함 한다. 이것은 비 mRNA 종에서 찾아낸 L1 관련 전사체 잡음을 제거 합니다. 다른 단계는 안티 센스 L1 관련 전사체를 동정 하 고 제거 하기 위해 스트랜드 특이 적 시퀀싱을 포함 한다. L1s에 매핑되는 RNA-서 열 전사체의 수를 동정 할 때 기능적인 프로모터 영역과 함께 전장 L1s 대 한 주석의 사용은 또한 잘린 L1s에서 비롯 된 배경 잡음을 제거 한다. 마지막으로, L1 소급 성과 관련이 없는 L1 서 열의 전사 잡음을 제거 하는 마지막 중요 한 단계는 RNA-서 열 전사체가 맵핑된 것으로 확인 된 전장 L1s의 수동 큐 레이 션 이다. 수동 큐 레이 션은 L1 프로모터 로부터 발현이 시작 되는 것을 확인 하기 위해 주변 게놈 환경의 맥락에서 각각의 생물 학적으로 식별 가능 하 게 표현 된 L1 궤적을 가시화 하는 것을 포함 한다. 이러한 접근법은 DU145, 전립선 종양 세포 라인에 적용 되었다. 배경 소음을 줄이기 위해 준비 관련 단계를 모두 수행 하는 경우에도 l1 좌 위의 약 50%가 다른 전사 소스에서 발생 하는 l1 배경 노이즈로 거부 되었습니다 (그림 4). 신뢰성 있는 결과를 생성 하는 데 필요한 엄격 함을 강조 합니다. 수동 큐 레이 션을 사용 하는이 접근법은 노동 집약적 이지만 전체 길이 L1을 둘러싼 게놈 환경을 평가 하 고 이해 하기 위해이 파이프라인을 개발 하는 데 필요 합니다. 다음 단계에는 일부 큐 레이 션 규칙을 자동화 하 여 필요한 수동 큐 레이 션의 양을 줄이는 것이 포함 되지만, 게놈 표현의 아직 완전히 알려지지 않은 성질로 인해, 참조 게놈에서 주석이 없는 소스 발현의 경우, 낮은 영역 mappability, 및 참조 게놈의 구성과 관련 된 복잡 한 요소도이 시점에서 L1 큐 레이 션을 완전히 자동화할 수 없습니다.
시퀀싱으로 개별 l1 좌 위의 발현을 식별 하는 두 번째 과제는 반복적인 L1 전사체의 매핑에 관한 것 이다. 이 정렬 전략에서는, 전사체가 맵핑 되기 위해 참조 게놈에 고유 하 고 동시에 선형적으로 정렬 되어야 합니다. 코드를 매핑하는 쌍을 이루는 끝 시퀀스를 선택 하면 참조 게놈에서 발견 되는 L1 좌 위에 고유 하 게 정렬 되는 전사체의 양이 증가 합니다. 이 독특한 매핑 전략은 특히 하나의 L1 궤적에 대 한 읽기 매핑의 호출에 자신감을 제공 합니다, 그것은 잠재적으로 식별 될 각각의 표현 량을 과소 평가 하지만, 반복적 인 L1. 이 과소 평가에 대해 대략 정확 하 게 하기 위해, 그 mappability에 기초한 각 L1 궤적에 대 한 "mappability" 점수는 고유 하 게 매핑된 전사체 판독의 수를 개발 하 고 적용 하였다 (도 6). 그것은 이상적으로, mappability는 일치 하는 WGS 샘플에 따라 전체 길이 L1에 걸쳐 전체 커버리지 읽기에 득점 해야한다. 여기에서, 우리는 DU145 전립선 종양 세포 주에 l1 좌 위에 매핑 읽기 팽창 또는 수축 하기 위해 각 L1 좌 위의 mapp 능력 점수를 결정 하기 위해 헬 라 셀의 wgs를 사용 합니다. 이 mappability 계산은 조 수정 점수입니다, 하지만 선택 된 ' 완전 한 커버리지 mappability '의 400 읽기는 마음에 종양 세포 주의 동적 특성으로 결정 되었다. 그것은 보충 그림 1에서 관찰 될 수 있다, 매핑된 읽기의 매우 높은 숫자와 함께 몇 L1 좌 위가 있다. 이러한 가능성이 그 좌 위 완전 한 mappability 범위를 대표 하는 것으로 선택 되지 않은 이유입니다, 참조 게놈 내에 있지 않습니다 헬 내에서 중복 된 염색체 서 열에서 온다. 대신에 보충도 1 에 따라 100%의 판독 커버리지의 평균이 400 읽기 전후에 발생 하 고이 평균이 DU145 종양 전립선 세포 라인에도 적용 된다고 가정 하였다.
100-200 bp와의이 정렬 전략은 RNA-서 열 기술 또한 우선적으로 이전 L1s 그들을 더 매핑 할 수 있도록 독특한 돌연변이 시간이 지남에 축적으로 참조 게놈 내에서 진화 한 오래 된 L1s에 대 한 선택. 이 접근법은 L1s의 최 연소 뿐만 아니라 비 참조 다형성 L1s 식별에 관해서 제한 된 감도를가지고 있습니다. L1s의 막내를 확인 하기 위해, 우리는 더 이상 읽기21을 사용 하 게 pacbio 같은 L1 전사체와 시퀀싱 기술의 5 ' 경주 선택을 사용 하는 것이 좋습니다. 이것은 더 독특한 맵핑을 허용 하 고 따라서 표현 된 젊은 L1s의 자신감 있는 식별을 가능 하 게 합니다. RNA-서 열 및 PacBio 접근법을 함께 사용 하면 진정으로 표현 된 L1s의 보다 포괄적인 목록으로 이어질 수 있습니다. 확실 하 게 표현 된 다형성 L1s를 식별 하기 위해 첫 번째 다음 단계는 참조 게놈에 다형성 서 열의 구성 및 삽입을 포함 합니다.
반복 시퀀스를 연구 하는 데 있어서 생물학적 및 기술적 과제는 큰 것 이지만, RNA 시퀀싱 기술을 사용 하 여 전환에 관한 L1 시퀀스의 전사 노이즈를 제거 하는 위의 엄격한 절차를 통해 선별 하기 시작 합니다. 전사 배경 소음의 큰 수준과 자신 있게 그리고 엄격 하 게 개별 궤적 수준에서 L1 식 패턴과 수량을 식별 하는 것입니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는 DU145 전립선 종양 세포에 대 한 박사 얀 동에 게 감사 하 고 싶습니다. 우리는 슈퍼 컴퓨터 스크립트를 만드는 그의 지도와 조언에 네이 선 Ungerleider 감사 드립니다. 이 작품 중 일부는 PD에 GM121812 R01 NIH 교부 금, VPB에 AG057597 R01, 그리고 5TL1TR001418에 대 한 투자를 TK로 지원 했다. 우리는 또한 암 십자군과 Tulane 암 센터 생물 정보학 코어에서 지원을 인정 하 고 싶습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 M HEPES | Affymetrix | AAJ16924AE | |
| 5 M NaCl | Invitrogen | AM9760G | |
| Agilent bioanalyzer 2100 | Agilent technologies | ||
| Agilent RNA 6000 Nano Kit | Agilent technologies | 5067-1511 | |
| bedtools.26.0 | https://bedtools.readthedocs.io/en/latest/content/installation.html | ||
| bowtie-0.12.8 | https://sourceforge.net/projects/bowtie-bio/files/bowtie/0.12.8/ | ||
| Cell scraper | Olympus plastics | 25-270 | |
| Chloroform | Fisher | C298-500 | |
| Digitonin | Research Products International Corp | 50-488-644 | |
| Ethanol | Fisher | A4094 | |
| Gibco (Phosphate Buffered Saline) | Invitrogen | 10-010-049 | |
| Homogenizer | Thomas Scientific | BBI-8541906 | |
| IGV 2.4 | https://software.broadinstitute.org/software/igv/download | ||
| Isopropanol | Fisher | A416-500 | |
| mac2unix | https://sourceforge.net/projects/cs-cmdtools/files/mac2unix/ | ||
| Q-tips | Fisher | 23-400-122 | |
| RNAse later solution | Invitrogen | AM7022 | |
| RNaseZap RNase Decontamination Solution | Invitrogen | AM9780 | |
| samtools-1.3 | https://sourceforge.net/projects/samtools/files/ | ||
| sratoolkit.2.9.2 | https://github.com/ncbi/sra-tools/wiki/Downloads | ||
| SUPERase·In RNase Inhibitor | Invitrogen | AM2694 | |
| Trizol | Invitrogen | 15-596-018 | |
| Water (DNASE, RNASE free) | Fisher | BP2484100 |
참고문헌
- International Human Genome Sequencing. Initial sequencing and analysis of the human genome. Nature. 409, 860 (2001).
- Brouha, B., et al. Hot L1s account for the bulk of retrotransposition in the human population. Proceedings of the National Academy of Sciences of the United States of America. 100 (9), 5280-5285 (2003).
- Dombroski, B. A., Mathias, S. L., Nanthakumar, E., Scott, A. F., Kazazian, H. H. Isolation of an active human transposable element. Science. 254 (5039), 1805 (1991).
- Swergold, G. D. Identification, characterization, and cell specificity of a human LINE-1 promoter. Molecular and Cellular Biology. 10 (12), 6718-6729 (1990).
- Speek, M. Antisense promoter of human L1 retrotransposon drives transcription of adjacent cellular genes. Molecular and Cellular Biology. 21 (6), 1973-1985 (2001).
- Deininger, L., Batzer, M. A., Hutchison, C. A., Edgell, M. H. Master genes in mammalian repetitive DNA amplification. Trends in Genetics. 8 (9), 307-311 (1992).
- Boissinot, S., Chevret, P., Furano, A. L1 (LINE-1) Retrotransposon Evolution and Amplification in Recent Human History. Molecular Biology and Evolution. 17 (6), 915-918 (2000).
- Khazina, E., Weichenrieder, O. Non-LTR retrotransposons encode noncanonical RRM domains in their first open reading frame. Proceedings of the National Academy of Sciences of the United States of America. 106 (3), 731-736 (2009).
- Martin, S. L., Bushman, F. D. Nucleic acid chaperone activity of the ORF1 protein from the mouse LINE-1 retrotransposon. Molecular and Cellular Biology. 21 (2), 467-475 (2001).
- Feng, Q., Moran, M. H., Kazazian, H. H., Boeke, J. D. Human L1 Retrotransposon Encodes a Conserved Endonuclease Required for Retrotransposition. Cell. 87 (5), 905-916 (1996).
- Mathias, S. L., Scott, A. F., Kazazian, H. H., Boeke, J. D., Gabriel, A. Reverse transcriptase encoded by a human transposable element. Science. 254 (5039), 1808 (1991).
- Luan, D. D., Korman, M. H., Jakubczak, J. L., Eickbush, T. H. Reverse transcription of R2Bm RNA is primed by a nick at the chromosomal target site: A mechanism for non-LTR retrotransposition. Cell. 72 (4), 595-605 (1993).
- van den Hurk, J. A. J. M., et al. Novel types of mutation in the choroideremia (CHM) gene: a full-length L1 insertion and an intronic mutation activating a cryptic exon. Human Genetics. 113 (3), 268-275 (2003).
- Miné, M., et al. A large genomic deletion in the PDHX gene caused by the retrotranspositional insertion of a full-length LINE-1 element. Human Mutation. 28 (2), 137-142 (2007).
- Solyom, S., et al. Pathogenic orphan transduction created by a nonreference LINE-1 retrotransposon. Human Mutation. 33 (2), 369-371 (2012).
- Hancks, D. C., Kazazian, H. H. Roles for retrotransposon insertions in human disease. Mobile DNA. Mobile DNA. 7, 9-9 (2016).
- Tubio, J. M. C., et al. Mobile DNA in cancer. Extensive transduction of nonrepetitive DNA mediated by L1 retrotransposition in cancer genomes. Science. 345 (6196), 1251343-1251343 (2014).
- Ewing, A. D., et al. Widespread somatic L1 retrotransposition occurs early during gastrointestinal cancer evolution. Genome Research. 25 (10), 1536-1545 (2015).
- Beck, C. R., Garcia-Perez, J. L., Badge, R. M., Moran, J. V. LINE-1 elements in structural variation and disease. Annual Review of Genomics and Human Genetics. 12, 187-215 (2011).
- Philippe, C., et al. Activation of individual L1 retrotransposon instances is restricted to cell-type dependent permissive loci. eLife. 5, e13926 (2016).
- Deininger, P., et al. A comprehensive approach to expression of L1 loci. Nucleic Acids Research. 45 (5), e31-e31 (2017).
- Jin, Y., Tam, O. H., Paniagua, E., Hammell, M. TEtranscripts: a package for including transposable elements in differential expression analysis of RNA-seq datasets. Bioinformatics. 31 (22), 3593-3599 (2015).
- . . Agilent RNA 6000 Nano Kit Guide. , (2017).
- Mueller, O. L., Schroeder, A. . RNA Integrity Number (RIN) –Standardization of RNA Quality Control. , (2016).
- Robinson, J. T., et al. Integrative genomics viewer. Nature Biotechnology. 29, 24 (2011).
- Speek, M. Antisense promoter of human L1 retrotransposon drives transcription of adjacent cellular genes. Molecular Cellular Biology. 21 (6), 1973-1985 (2001).
- Belancio, V. P., Deininger, L., Roy-Engel, A. M. LINE dancing in the human genome: transposable elements and disease. Genome Medicine. 1 (10), 97-97 (2009).
- Iskow, R. C., et al. Natural Mutagenesis of Human Genomes by Endogenous Retrotransposons. Cell. 141 (7), 1253-1261 (2010).
- Scott, E. C., et al. A hot L1 retrotransposon evades somatic repression and initiates human colorectal cancer. Genome Research. 26 (6), 745-755 (2016).
- Kines, K. J., Sokolowski, M., deHaro, D. L., Christian, C. M., Belancio, V. P. Potential for genomic instability associated with retrotranspositionally-incompetent L1 loci. Nucleic Acids Research. 42 (16), 10488-10502 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유