Method Article
인간 면역결핍 바이러스/원숭이 면역결핍 바이러스 연구에서의 차세대 바이러스 RNA/DNA in situ 혼성화 응용 분야
요약
여기에서는 포르말린 고정 파라핀 포매(FFPE) 조직에서 특정 바이러스 RNA 또는 DNA 염기서열을 식별하기 위한 차세대 현장 혼성화 분석을 제시합니다. 이 접근 방식을 사용하면 매우 높은 민감도와 특이성으로 24시간 이내에 RNA 및 DNA의 낮은 사본을 시각화할 수 있습니다.
초록
In situ hybridization은 조직 절편의 개별 세포 내에서 특정 RNA 또는 DNA 염기서열을 식별하는 강력한 기술로, 생리학적 과정과 질병 발병에 대한 중요한 통찰력을 제공합니다. ISH(In situ hybridization)는 바이러스에 감염된 세포의 위치를 평가하기 위해 수년 동안 사용되어 왔지만, 최근에는 조직 형태를 보존하면서 단일 분자 시각화를 달성하기 위해 동시 신호 증폭 및 배경 억제를 허용하는 고유한 프로브 설계 전략으로 차세대 ISH 접근 방식이 개발되었습니다. 이 차세대 ISH는 분지형 PCR과 같은 접근법을 기반으로 하지만, 현장에서 수행되며 포르말린 고정 파라핀 포매(FFPE) 조직에서 RNA 또는 DNA를 일상적으로 검출하는 데 있어 기존 ISH 방법 또는 현장 PCR 접근 방식보다 더 쉽고 민감하며 재현성이 높습니다. 지난 몇 년 동안 우리 실험실은 다수의 FFPE 조직 내에서 인간 면역결핍(HIV) 및 원숭이 면역결핍(SIV) 바이러스 RNA(vRNA) 및/또는 바이러스 DNA(vDNA) 양성 세포를 검출하기 위해 이 ISH 플랫폼을 적용해 왔습니다. 이 상세한 기술 원고를 통해 우리는 차세대 ISH를 연구에 사용하는 데 관심이 있는 모든 개인과 우리의 지식과 조언을 공유하고자 합니다.
서문
ISH는 세포 또는 조직 섹션 내의 특정 DNA 또는 RNA 염기서열에 대한 상보적 DNA, RNA 또는 변형된 핵산 가닥(즉, 프로브)을 표적으로 하고 시각화하는 데 사용되는 실험적 접근 방식입니다. ISH는 조직 내 특정 핵 염기서열의 특정 국소화 및 시각화를 가능하게 하며, 이는 qPCR과 같은 다른 인기 있는 기술을 사용하여 얻을 수 없는 귀중한 정보인 표적과 세포 환경 간의 발현 수준, 조직, 분포 및 상호 작용을 이해하는 데 중요합니다. 최근까지 ISH는 일반적으로 표지된 상보적 DNA 또는 상보적 RNA(리보프로브)를 사용하여 수행되었습니다. 이러한 프로브는 방사선, 형광 또는 항원 표지 염기(예: 35S, FITC 및 디곡시제닌)와 직접 결합한 다음 각각 자가방사선 촬영, 형광 현미경 또는 면역조직화학 검출 방법을 사용하여 조직에서 국소화 및 정량화되었습니다. 이러한 현장 기술은 계속해서 가치 있는 접근 방식이지만, 노동 집약적이지 않고, 더 간단하고, 더 빠르고, 민감하고, 구체적인 접근 방식을 개발하기 위해서는 개선의 여지가 충분합니다.
숙주 전령 RNA(mRNA) 검출을 위해 2012년에 처음 기술된 대안적인 상용 차세대 ISH 접근법(예: RNAscope 분석)은 분기 PCR을 기반으로 합니다. mRNA 검출은 FFPE 세포 및 조직에서 수행되며, 개별 세포에서 단일 RNA 분자 시각화에 가까운 민감도로수행됩니다 1. 이 접근법의 특이성은 신호 전치증폭기가1을 순차적으로 결합하기 위해 두 개의 이중 Z 표적 프로브가 각각의 상보적 RNA(또는 DNA) 염기서열에 연속적으로 결합하는 고유한 조건에서 달성됩니다. 이를 통해 branched DNA(bDNA)1,2와 유사한 후속 혼성화 단계를 통해 신호 증폭 캐스케이드를 시작할 수 있습니다. 또한 이 접근 방식은 매우 빠르고 쉬우며 단 1일(<8시간) 만에 결과를 얻을 수 있으며, 이는 Radio-ISH 1,2를 포함한 대체 기술을 사용할 경우 최대 4주가 걸리는 것에 비해 상당한 이점입니다. 이 차세대 ISH는 HIV/SIV 연구에 대한 새로운 관점과 기회를 열어주었습니다. HIV 치료의 주요 장애물은 질병의 초기 단계에 형성된 세포 및 조직 저장소입니다 3,4. 이 기술의 전반적인 목표는 바이러스 저장소로 작용하고 감염된 숙주 내에서 지속되는 주요 조직 구획을 식별, 국소화 및 궁극적으로 이해하는 것입니다. 이는 HIV에 대한 효과적인 치료 전략을 개발하는 데 도움이 될 것입니다.
본 원고에서는 당사의 duplex next-generation RNA/DNA multiplex ISH protocol(예: RNAscope/DNAscope)에 대해 자세히 설명하고, 차세대 ISH를 시료 및 특정 target에 최적화하기 위해 기존 RNA ISH protocol을 어떻게 수정했는지 설명합니다. 이 프로토콜을 사용하면 5μm 조직 절편 내에서 HIV/SIV 바이러스 RNA 및 바이러스 DNA의 시각화, 국소화 및 정량화가 가능합니다. vRNA와 vDNA의 동시 시각화는 두 개의 맞춤형 프로브 세트, 즉 vDNA 코딩 가닥을 표적으로 하는 하나의 센스(C1 SIVmac239 Gag-Pol-Sense 프로브 [416141-C1])와 바이러스 게놈의 다른 영역을 다루는 vRNA 전사체(C2 SIVmac239 Vif-Env-Nef-Tar-Anti-Sense 프로브 [416131-C2])를 표적으로 하는 하나의 안티 센스를 결합하여 수행됩니다(표 1)을 사용하여 두 개의 서로 다른 시각화 채널인 C1 및 C2를 사용할 수 있습니다. 이 프로토콜에서 채널 C1 및 C2를 사용하면 신호를 서로 다른 색상(즉, 빨간색의 AP, 갈색의 HRP)으로 시각화하고 다양한 접근 방식으로 프로브를 감지할 수 있습니다. 조직 고정 처리 및 절단을 제외하고 이 분석은 2일이 소요됩니다. 여기에 제시된 것은 세포 펠릿 또는 조직 절편에서 수행할 수 있는 이중 vRNA 및 vDNA in situ 혼성화 프로토콜입니다.
프로토콜
1. 단면 및 슬라이드 준비
- 파라핀 블록을 다듬고 마이크로톰을 사용하여 +/-1 μm 절편 5개를 자릅니다. 절편 또는 세포 펠릿을 40-45°C RNase free 수조의 하전 현미경 슬라이드에 장착합니다. 37 ° C 또는 RT에서 밤새 자연 건조합니다.
참고: 슬라이드는 실온(RT)에서 최대 3개월, 4°C에서 6개월 동안 보관할 수 있습니다. - FFPE 슬라이드를 파라핀화합니다.
- 건조 오븐에서 60°C에서 1시간 동안 슬라이드를 굽습니다.
- 흄 후드에서 두 개의 슬라이드 염색 접시에 ~200mL의 신선한 자일렌을 채우고 두 개의 추가 염색 접시에 ~200mL의 신선한 100% 에탄올을 채웁니다. 용기를 뚜껑으로 덮으십시오.
- 슬라이드를 랙에 놓고 첫 번째 자일렌 함유 접시에 담그십시오. RT에서 5-10분 동안 교반하면서 배양합니다.
- 슬라이드를 두 번째 자일렌 함유 접시에 넣고 교반하면서 RT에서 5-10분 동안 배양합니다.
- 즉시 100% 에탄올이 함유된 접시에 슬라이드를 놓습니다. 교반과 함께 RT에서 5-10분 동안 슬라이드를 배양합니다.
- 즉시 100% 에탄올이 함유된 두 번째 접시에 슬라이드를 놓고 교반하면서 RT에서 5-10분 동안 배양합니다.
- 에탄올에서 랙을 제거하고 랙 측면을 가볍게 두드려 과도한 에탄올을 제거한 다음 RNase free water에서 5-15분 동안 헹굽니다.
2. 오븐 준비
- 하이브리드화 오븐을 켜고 온도를 40°C로 설정합니다.
- 천이나 튼튼한 흡수성 종이 타월을 트레이에 넣고 이중 증류수로 완전히 적셔 습도를 조절합니다.
- 뚜껑이 있는 트레이를 오븐에 넣고 오븐 도어를 닫습니다. 사용하기 전에 트레이를 30°C에서 40분 이상 예열하십시오. 사용하지 않을 때는 트레이를 오븐에 보관하십시오.
3. 열 유도 항원결정기 회수
- 0.5x 구연산염 기반 ISH 혼성화 타겟 회수 버퍼(10nmol/L, pH = 6, 재료 표 참조)를 준비합니다. 가열판의 비커에 넣고 끓입니다.
- 슬라이드를 끓는 타겟 회수 버퍼에 30분 동안 넣어 열 유도 에피토프 회수를 수행합니다.
- 대상 회수 버퍼에서 슬라이드를 제거하고 즉시 이중 증류수로 세척합니다. 공기 건조하기 전에 100% 에탄올에서 5분 동안 탈수하십시오.
- 슬라이드가 자연 건조되면 소수성 장벽 펜을 적용하여 슬라이드의 조직 부분을 둘러쌉니다. 소수성 장벽이 공기 중에서 완전히 건조되도록 하십시오.
4. 프로테아제 전처리
- 건조된 슬라이드를 잠금 슬라이드 랙에 놓고 멸균, 저온 PBS로 1:5 비율로 희석하여 프로테아제 전처리 시약(프로테아제 분해 용액, 2.5μg/mL)을 준비합니다. 잘 섞는다.
참고: 농도가 다른 세 가지 다른 프로테아제 시약이 시판되는 키트에 제공됩니다. 프로테아제 III(표준), 프로테아제 IV(강) 및 프로테아제 플러스(경증). 최적의 조건은 조직 유형, 고정 및 두께에 따라 달라지기 때문에 연구에서 구현하기 전에 프로테아제 분해 시간 및 희석을 경험적으로 테스트합니다(논의 참조). - 조직 부분을 완전히 덮기 위해 희석된 단백질 분해 효소 용액을 슬라이드에 분배합니다. 즉시 오븐에서 40°C에서 20분 동안 슬라이드를 배양하고(1.4단계에서 준비) 슬라이드가 습한 혼성화 트레이에 밀봉되었는지 확인합니다. 프로토콜의 나머지 부분 동안 조직 섹션이 건조되지 않도록 하십시오.
- 즉시 잠금 슬라이드 랙을 이중 증류수가 채워진 세척 트레이에 담그어 3번 헹굽니다.
- 과산화효소 용액을 각 조직 절편에 떨어뜨려 완전히 덮음으로써 내인성 과산화효소 차단을 수행합니다. RT에서 10분 동안 슬라이드를 배양합니다. 완료되면 이중 증류수로 섹션을 3x 헹굽니다.
5. 프로브 혼성화 및 신호 증폭
알림: 증발을 방지하려면 배양 단계에서 조직이 건조되지 않도록 습도 조절 트레이가 제대로 밀봉되었는지 확인하십시오. 세척 단계에서 습도 챔버를 오븐에 다시 넣어 40°C로 유지되도록 합니다.
- 제조업체에서 제안한 대로 C2 프로브 1 부피와 C2 프로브 50 부피를 튜브에 피펫팅하여 C2 프로브와 C1 프로브를 1:50 비율로 혼합합니다. 튜브를 여러 번 뒤집습니다. 사용하기 전에 침전물을 용해시키기 위해 40°C 오븐에서 대상 프로브 혼합물을 ~10분 동안 예열합니다.
참고: 혼합 타겟 프로브는 최대 6개월 동안 4°C에서 보관할 수 있습니다. - 물에서 슬라이드를 제거하십시오. 헹구고 슬라이드를 두드리거나 튕겨 조직 부분에서 과도한 수분을 제거합니다. 즉시 슬라이드에 프로브를 분배하여 각 조직 섹션이 기포 없이 완전히 덮여 있는지 확인합니다. 프로브 혼합물을 40°C의 습도 챔버에서 밤새 배양합니다.
- 다음 날 오븐에서 슬라이드를 꺼내 RT에서 0.5분 동안 5x 세탁 버퍼가 들어 있는 세탁 트레이에 넣습니다. 세탁 단계를 한 번 더 반복합니다.
- 세탁 버퍼에서 슬라이드를 제거합니다. 헹구고 슬라이드를 두드리거나 튕겨 조직 섹션에서 과도한 세척 버퍼를 제거합니다.
- 슬라이드의 혼성화 완충액 B(20% 포름아미드, 5x SSC, 0.3% 리튬도데실 설페이트, 10% 덱스트란 설페이트, 차단 시약)에 즉시 사용 가능한 AMP 1 시약(2 nmol/L)을 시판하여 기포 없이 조직 단면을 완벽하게 커버할 수 있습니다. 습도 챔버에서 40 °C에서 30 분 동안 배양합니다. 1.6.3-1.6.4단계를 반복하여 각각 2분 동안 세탁을 수행합니다.
- 시판되는 AMP 디스펜싱 2. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 15분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세탁을 수행합니다.
- 시판되는 AMP 디스펜싱 3. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 30분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세탁을 수행합니다.
- 시판되는 AMP 디스펜싱 4. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 15분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세탁을 수행합니다.
- 시판되는 AMP 디스펜싱 5. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 30분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
- 시판되는 AMP 6을 디스펜싱합니다. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 15분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
- 감지하기 전에 1x TBS-Tween 20(0.05% v/v)에서 슬라이드를 한 번 헹굽니다. 세척 버퍼에서 슬라이드를 제거하고 헹구고 슬라이드를 두드리거나 튕겨 조직 섹션에서 과도한 세척 버퍼를 제거합니다. 즉시 1x TBS-Tween 버퍼로 채워진 세척 트레이에 넣습니다.
6. 채널 1(C1) 대상 신호 감지
참고: 이는 알칼리성 인산가수분해효소 라벨을 포함하는 2-plex 검출 키트( 재료 표 참조)의 Red Alkaline Phosphatase 및 Fast Red Chromogen Amplification 6 및 발색 검출을 사용하여 수행됩니다. 빠른 빨간색을 기판으로 사용하여 빨간색 신호를 생성합니다.
- Fast RED-B를 Fast RED-A로 1:60 희석하여 Fast Red-A를 사용하여 Fast Red(FR) 작업 용액을 준비합니다. 잘 섞는다. 침전물을 줄이고 더 깨끗한 신호를 얻으려면 주사기를 사용하여 0.45μm MCE 멤브레인을 통해 발색 용액을 여과합니다.
알림: 5분 이내에 Fast RED-B 솔루션을 사용하십시오. 직사광선이나 자외선에 노출시키지 마십시오. - TBS-Tween에서 슬라이드를 제거하고 헹구고 슬라이드를 두드리거나 튕겨 조직 섹션에서 여분의 버퍼를 제거합니다.
- 혼합되고 여과된 FR 용액을 각 조직 섹션에 분배하고 각 섹션이 완전히 덮여 있는지 확인합니다. RT에서 6-8분 동안 배양합니다. 현미경으로 관찰하십시오.
- 슬라이드를 0.5x 세척 버퍼 2x로 헹굽니다. 세척 버퍼에서 슬라이드를 제거하고 헹구고 슬라이드를 두드리거나 튕겨 조직 섹션에서 과도한 세척 버퍼를 제거합니다.
- 시판되는 AMP 7을 디스펜싱합니다. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 10분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
- 시판되는 AMP 8을 디스펜싱합니다. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 15분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
- 시판되는 AMP 디스펜싱 9. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 30분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
- 시판되는 AMP 10을 디스펜싱합니다. 티슈 부분이 기포 없이 완전히 덮여 있는지 확인하십시오. 습도 챔버에서 슬라이드를 40°C에서 30분 동안 배양합니다. 5.3-5.4단계를 반복하여 각각 2분 동안 세척을 수행합니다.
7. 채널 2(C2) 대상 신호 감지
참고: 이 작업은 시중에서 판매되는 Brown HRP 및 DAB Chromogen Kit를 사용하여 수행됩니다( 재료표 참조). 2-plex 검출의 증폭 10에는 양 고추 냉이 과산화 효소 라벨이 포함되어 있으며 발색 검출은 DAB를 사용하여 갈색 신호를 생성함으로써 수행됩니다.
- DAB 신호를 최적으로 감지하려면 시중에서 판매되는 키트를 사용하고 제조업체의 지침을 따르십시오( 재료 표 참조). 현미경으로 관찰하십시오.
주의: DAB는 독성이 있습니다. 이 화학 물질을 취급하고 폐기할 때 적절한 예방 조치와 안전 지침을 따르십시오.
8. counterstaining 및 장착
- 슬라이드를 헤마톡실린으로 대조염색합니다.
- 슬라이드를 교반하는 동안 50% 신선한 여과된 헤마톡실린으로 슬라이드를 30초 동안 대조염색합니다. 슬라이드가 자주색으로 표시됩니다. 물이 맑아질 때까지 슬라이드를 위아래로 흔들면서 즉시 흐르는 물로 헹굽니다. 조직 부분은 보라색으로 유지됩니다.
- 더 나은 대비를 얻으려면 카트라스테인된 슬라이드를 탄산리튬으로 포화된 증류수에 1분 동안 놓습니다. 슬라이드를 교반하는 동안 흐르는 물에 최소 3회 이상 철저히 헹굽니다. 최종 헹굼을 위해 이중 증류수를 사용하십시오.
- FR은 유기 용제에 민감하기 때문에 FR로 염색된 슬라이드는 수성 장착 매체로 덮고 RT에서 밤새 건조해야 합니다.
- 슬라이드를 장착합니다.
- 수성 장착 매체로 덮인 조직 부분이 건조한지 확인하십시오.
- 장착 시약을 사용하여 덮개가 미끄러지기 전에 크실렌에 슬라이드를 담그십시오. 커버슬립과 티슈 섹션 사이에 기포가 생기지 않도록 반드시 제거하고 RT에서 16시간 동안 건조시키십시오.
9. CellProfiler5를 사용한 RNAscope를 위한 정량적 이미지 분석 프로토콜
- 간단히 말해서, 소프트웨어가 헤마톡실린과 FR 염색을 별도의 이미지로 분리하는지 확인하십시오. 관심 대상을 식별하고 측정합니다: 핵, 비리온, 양성 세포 및 응집체 FR 양성 염색. 측정값을 CSV 파일에 저장하고 분석된 이미지를 저장합니다.
- "색상 혼합 해제" 옵션을 선택하면 얼룩이 분리되고 원래 관심 영역(ROI)이 별도의 헤마톡실린 및 FR 이미지로 분할됩니다.
- 헤모시데린, 문신 또는 이와 유사한 특징이 분석을 방해하는 경우 헤마톡실린, FR 및 DAB와 함께 두 번째 "혼합되지 않음" 단계를 추가합니다. 두 번째 FR 이미지를 사용하여 가장 강렬한 FR 픽셀을 찾습니다. 이 두 번째 이미지는 실제 FR 염색을 위한 마스크로 사용됩니다.
- 선택적으로, 임계값을 설정하기 전에 염색된 이미지를 매끄럽게 합니다. 이는 경험적 결정이며 항상 필요한 것은 아닙니다. "IdentifyPrimaryObjects"를 사용하여 개별 염색된 이미지를 임계값으로 설정하여 양수 픽셀을 선택합니다.
- 세 가지 유형의 개체(비리온, 비리온 응집체, 생산적 세포)를 식별합니다.
- 핵이 헤마톡실린으로 염색된 직경 4-100픽셀(px)의 물체인지 확인합니다. 임계값 설정 후 구멍을 줄이고 채웁니다. "IntenseFastRed" 개체는 직경이 4-100px이며 비리온, 양성 세포 및 B 세포 여포(BCF)의 여포 수지상 세포(FDC)에서 볼 수 있는 것과 같은 응집체 양성 염색을 포함합니다. 이 이미지는 거짓 양성(예: 헤모시데린)을 필터링하는 데 사용됩니다.
- FR 작은 긍정이 2-12px 지름의 개체인지 확인합니다. 이 측정에는 비리온 및 vDNA+ 세포가 포함됩니다. 이 범위를 벗어난 모든 개체를 폐기합니다. 임계값 설정 후 구멍을 줄이고 채웁니다.
- FR 큰 긍정(9-100px 지름 개체)을 확인합니다. 이 측정에는 vRNA+ 양성 세포와 응집체 양성 염색이 포함됩니다. 임계값 설정 후 감소. 작은 FR 포지티브 오브젝트와 큰 FR 포지티브 오브젝트의 크기가 겹칠 수 있습니다. 이후 단계에서 분리됩니다.
참고: 소프트웨어(예: Cellprofiler)는 개체 크기에 픽셀을 사용하며 여기에 사용된 픽셀은 0.2510μm/픽셀(40x)로 스캔한 슬라이드에서 파생된 것입니다.
- 결과를 정의하고 추출합니다.
- 개체를 식별한 후 virion, 생산적인 감염 세포 및 핵의 수에 대한 결과를 정의하고 추출합니다.
- 거짓 긍정(예: 헤모시데린)을 제거하도록 설정된 IntenseFastRed 개체와 함께 FastRedSmallPositives를 마스킹하여 비리온을 식별합니다.
- 다음으로, 양성 세포를 식별하고 FR 양성 염색을 집계합니다. IntenseFastRed 및 virion 개체 세트로 마스킹하여 FastRedLargePositives에서 거짓 긍정 및 virions를 제거합니다.
- Nuclei로 다시 마스킹하여 정제된 FastRedLargePositives에서 양성 세포를 추출합니다. 더 이상 접촉하지 않는 개체를 분할하고 개체 영역별로 결과를 필터링하여 작은 개체(≤6픽셀)를 제거합니다. 이렇게 하면 핵 겹침을 마스킹하여 생성된 반점이 제거됩니다. 결과는 양성 세포입니다.
- 마지막으로 집계 FR 긍정을 정의합니다. 이 단계에서는 IdentifyTertiaryObjects를 사용하여 더 큰 부모 개체에 포함된 개체를 찾습니다. 이 경우 구체화된 FastRedLargePositives 개체 집합이 부모이고 양수 셀이 뺍니다.
- virion과 양성 세포의 수를 세십시오.
- 집계 양수 영역을 측정하고 픽셀에서 mm2로 변환합니다. 선택적으로, 분석에서 직접 계수 대신 표준 세포와 비리온 크기를 사용해야 하는 경우 비리온과 양성 세포가 차지하는 면적을 mm2 단위로 기록합니다.
- 원본 이미지에 긍정적 인 개체를 오버레이하고 결과를 저장하십시오.
참고: ISH 데이터는 더 나은 이해와 qPCR 데이터와의 비교를 용이하게 하기 위해 106 핵(세포)당 virion 수와 106 nuclei(세포)당 생산적으로 감염된 vRNA+ 세포 수로 보고되지만 결과는 mm2 단위의 조직 면적으로도 보고될 수 있습니다.
결과
이전 원고 2,6,7,8,9,10,11에서 우리는 vRNA 또는 vDNA를 검출하는 차세대 ISH 플랫폼을 SIV/HIV 게놈의 5' gag-pol 부분을 표적으로 하는 센스 프로브(vDNA)와 게놈의 3' 절반(vif, vpx, vpr, tat, env 및 nef)뿐만 아니라 5' 게놈의 TAR 요소(표 1)를 포함할 수 있다. 이 접근법은 전사 활성 세포(vRNA+, vDNA+)를 전사 비활성 (추정 잠복) 감염된 세포 또는 동일한 조직 섹션2에 전사 무능력 프로바이러스(vRNA-, vDNA+)가 있는 세포와 구별합니다(그림 1).
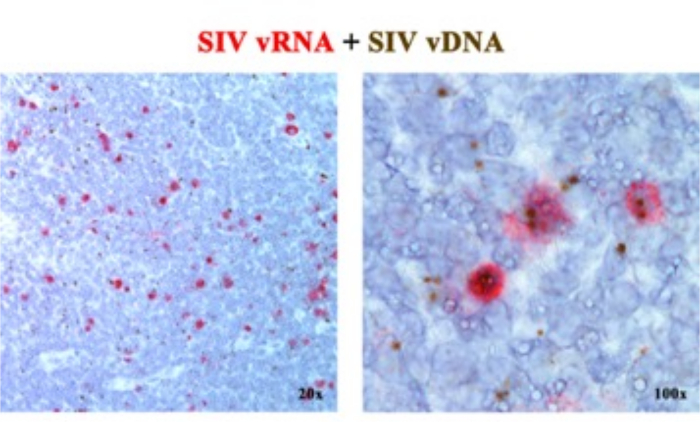
그림 1: 동일한 조직 절편에서 바이러스 RNA 및 vDNA 검출. 급성 SIV에 감염된 RM 림프절에서 RNA 부합화(빨간색) 및 DNA 부합화(갈색) 분석의 조합은 동일한 조직 절편에서 vRNA와 vDNA를 검출할 수 있는 능력을 입증하고 전사 침묵의 vDNA+ vRNA- 세포를 현장에서 식별할 수 있는 강력한 접근 방식을 제공합니다. 이 그림은 Deleage C. et al.2에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| Single Plex RNA/DNAscope 프로브 세트 | |||
| 이름 | ACD 카탈로그 # | ZZ의 수 | 묘사 |
| SIVmac239(안티 센스) | 312811 | 83 | D01065.1의 1251-9420bp 이내를 대상으로 하는 안티센스 프로브(gag, pol, vif, vpx, vpr, tat, env 및 nef) |
| SIVmac239(감지) | 314071 | 83 | D01065.1의 1251-9420bp 이내 역방향 가닥을 대상으로 하는 감지 프로브(gag, pol, vif, vpx, vpr, tat, env 및 nef) |
| V-HIV1-Clade A(안티 센스) | 416101 | 80 | HIV-1 Clade A Consensus의 879-7629bp 이내를 대상으로 하는 안티센스 프로브(gag, pol, vif, vpr, tat, rev, vpu, env 및 nef) |
| V-HIV1-Clade A(감지) | 426341 | 80 | HIV-1 Clade A Consensus의 879-7629bp 이내 역가닥을 대상으로 하는 감지 프로브(gag, pol, vif, vpr, tat, rev, vpu, env 및 nef) |
| V-HIV1-Clade B(안티센스) | 416111 | 78 | AF324493.2의 854-8291bp 이내를 대상으로 하는 안티센스 프로브, HIV-1 Clade B NL4-3(gag, pol, vif, vpr, tat, rev, vpu, env 및 nef) |
| V-HIV1-Clade B(감지) | 425531 | 78 | AF324493.2의 854-8291bp 이내의 역방향 가닥을 대상으로 하는 감지 프로브, HIV-1 Clade B NL4-3(gag, pol, vif, vpr, tat, rev, vpu, env 및 nef) |
| V-HIV1-Clade D(안티센스) | 416121 | 76 | HIV-1 Clade D Consensus의 894-7697bp 이내를 대상으로 하는 안티센스 프로브(gag, pol, vif, vpr, tat, rev, vpu, env, nef) |
| V-HIV1-Clade D(감지) | 426351 | 76 | HIV-1 Clade D Consensus의 894-7697bp 이내에서 역방향 가닥을 표적으로 하는 감지 프로브(gag, pol, vif, vpr, tat, rev, vpu, env, nef) |
| Multi-Plex RNA/DNAscope 프로브 세트 | |||
| 이름 | ACD 카탈로그 # | ZZ의 수 | 묘사 |
| V-SIVmac239-개그-폴-센스-C1 | 416141-C1 | 40 | D01065.1의 1251-4093bp 이내의 역방향 가닥을 대상으로 하는 감지 프로브(gag 및 pol) |
| V-SIVmac239-vif-env-nef-tar-C2(안티센스) | 제416131-C2호 | 47 | D01065.1의 5381-10257bp 이내를 대상으로 하는 안티센스 프로브(vif, vpx, vpr, tat, env, nef 및 TAR 요소) |
| V-HIV1-Clade_B-개그 폴 센스-C1 | 제444051-C1호 | 40 | AF324493.2의 854-3940bp 이내, HIV-1 Clade B NL4-3(gag 및 pol)을 대상으로 하는 감지 프로브 |
| V-HIV1-Clade_B-vif-vpr-tat-rev-vpu-env-nef-tar-C2(안티센스) | 제444061-C2호 | 40 | AF324493.2의 5042-9673bp 이내, HIV-1 Clade B NL4-3(vif, vpr, tat, env, nef 및 TAR 요소)을 대상으로 하는 안티센스 프로브 |
| V-HIV1-Clade_C-개그 폴 센스-C1 | 444021-C1 | 48 | HIV-1 Clade C conensus sequence의 888-5032bp 이내 역방향 가닥을 표적으로 하는 감지 프로브(gag 및 pol) |
| V-HIV1-Clade_C-vif-vpr- rev-vpu-env-nef-tar-C2(안티센스) | 제444041-C2호 | 49 | HIV-1 Clade C conensus sequence의 5078-9698bp 이내를 대상으로 하는 Anti-sense 프로브(vif, vpr, tat, env, nef 및 TAR 요소) |
| V-HIV1-Clade_AE-개그 폴 센스-C1 | 444011-C1 | 55 | AF259954.1의 890-4812bp 이내의 역 가닥을 표적으로 하는 감지 프로브, HIV-1 Clade AE(gag 및 pol) |
| V-HIV1-Clade_AE-vif-vpr-tat-rev-vpu-env-nef-tar-C2(안티센스) | 제444031-C2호 | 57 | AF259954.1, HIV-1 Clade AE(vif, vpr, tat, env, nef 및 TAR 요소)의 5052-9694bp 이내를 표적으로 하는 안티센스 프로브 |
표 1: HIV-1 및 SIV의 vRNA 및 vDNA를 표적으로 하는 프로브 목록.
토론
In situ hybridization은 잘 보존된 환경에서 표적을 국소화하기 위해 각 중요한 단계를 조정할 수 있도록 핵산 화학, 세포 생물학 및 조직학에 대한 엄격함과 기본 지식이 필요한 세심한 분석법입니다. 이 논의에서는 정확하고 해석 가능한 결과를 얻기 위해 문제 해결이 중요한 중요한 단계를 강조하고자 합니다.
조직의 고정 및 처리는 매우 중요하며 분석이 최상의 결과를 얻을 수 있도록 사전에 해결해야 합니다. 중성 완충 PFA(4% 신선하게 준비된) 고정제는 이중 분석에 최적입니다. 그러나 적절한 동결 절제 후 고정 조건으로 동결 조직(OCT)에서 분석을 수행할 수도 있습니다.
조직 절편의 전처리는 중요한 단계입니다. 이 분석에는 두 가지 전처리 단계가 있습니다: 첫 번째는 열 유도 항원결정기 회수(HIER)입니다. 이 단계는 메틸렌 브리지 가교의 역전과 고정 조직에 필요한 단백질 구조의 복원에 중요합니다. 이 처리의 효율성은 시간, 온도, 회수 완충액 유형 및 pH에 따라 다릅니다. 두 번째 전처리는 프로테아제 유도 에피토프 회수(PIER)입니다. 이 단계는 펩타이드를 절단하여 항원 또는 뉴클레오티드를 노출시키고 단백질분해효소 K, 트립신, 펩신을 포함한 효소를 사용합니다. 이는 조직 형태와 관심 대상 모두를 잠재적으로 손상시킬 수 있는 매우 민감한 단계입니다. 효소의 농도와 배양 시간 및 온도는 이 과정에서 매우 중요합니다. 과소화는 핵 경계가 불량하고 정량화 단계에서 어려움을 겪게 합니다. RNA/DNA 표적에 대한 최적의 접근과 관심 조직이나 표적을 손상시키지 않는 전처리 조건 사이의 균형을 찾는 것이 중요합니다. 각 조직 유형은 이러한 전처리 각각에 대한 민감도 수준이 다르며 각 매개변수(효소 농도, 시간, 온도)를 경험적으로 테스트해야 합니다.
세척 버퍼의 엄격성은 온도, 염분 및 세제의 농도, 시간의 세 가지 주요 매개변수를 기반으로 합니다. 세척 버퍼는 식염수 시트르산 나트륨 버퍼(SSC)이며, 버퍼 내의 염 농도는 세척 단계 중 엄격성을 제어합니다. 프로토콜에서 ACD는 0.1x SSC, 0.03% 리튬 도데실 설페이트의 최종 농도에서 세척 버퍼를 사용할 것을 권장합니다. DNAscope 및 멀티플렉스 최적화를 연구하는 동안, 최종 농도 0.05x SSC에서 세척 버퍼를 사용하면 DNA 신호를 시각화하는 데 더 나은 결과를 얻을 수 있으며 감지 프로브의 하룻밤 배양으로 인한 비특이적 표적 혼성화를 줄이는 데 상당한 도움이 된다는 것을 확인했습니다.
검출 접근법, 발색체(적색 또는 갈색) 대 형광의 선택은 분석을 시작하기 전에 조직 유형과 목표에 따라 신중하게 생각해야 합니다. 적색 발색 접근법은 빨간색이 조직에서 자연적으로 발견되지 않기 때문에 좋은 대비를 제공합니다. 갈색 발색체는 적색 발색원과 유사한 결과를 제공합니다. 그러나 조직에 존재하는 일부 혈액 분해 산물은 유사한 색을 가지고 있으며 문신 잉크는 정량화하는 동안 갈색 신호와 분리하기 어렵다는 점을 명심하는 것이 중요합니다. 형광 검출 접근법을 통해 다양한 세포 마커를 명확하게 구별할 수 있으며, 멀티플렉싱은 vRNA 및/또는 vDNA가 있는 세포를 표현형화하기 위한 완벽한 분석을 제공합니다.
프로브의 특이성과 분석의 품질을 보장하기 위해 여러 대조군이 필요합니다. 새로 설계된 각 프로브는 알려진 양성 및 음성 대조군 조직 또는 세포 펠릿에서 테스트해야 합니다. 당사는 종종 표적 염기서열을 포함하는 플라스미드를 생성하고 세포주에 transfection을 수행하여 positive control을 생성합니다. 각 실행에 대해 알려진 음성 조직(HIV 또는 SIV 음성), 프로브 희석제만 포함하는 프로브 없는 대조군 및 RNase 처리된 대조군을 추가하여 분석의 품질과 특이성을 보장합니다.
정량화는 매우 중요한 단계이며 질문에 따라 적절한 도구와 알고리즘을 사용하여 수행해야 합니다. 이 원고에서는 여러 옵션을 평가한 후 선택한 이미지 분석 소프트웨어(예: Cellprofiler)를 제시했습니다. 우리는 이 소프트웨어가 우리의 요구에 가장 적합한 소프트웨어라고 추정했지만 사용할 수 있는 수많은 이미지 분석 소프트웨어 프로그램이 있습니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
이 프로젝트는 계약 번호에 따라 국립 암 연구소(National Cancer Institute), 국립 보건원(National Institutes of Health)의 연방 기금으로 전액 자금을 지원받았습니다. HHSN261200800001E 및 Oregon National Primate Research Center NIH 보조금 상 P51OD011092(J.D.E). 이 간행물의 내용이 반드시 보건복지부의 견해나 정책을 반영하는 것은 아니며, 상표명, 상용 제품 또는 조직에 대한 언급이 미국 정부의 승인을 암시하지도 않습니다. 듀플렉스는 Advanced Cell Diagnostics의 도움으로 개발되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| ACD HybEZII Hybridization system (110V) with ACD EZ-Batch Slides system | ACD | 321710 | Hybridization oven |
| CAT Hematoxylin | Biocare medical | CATHE-GAL | colorstain |
| Clear-Mount | ELECTRON MICROSCOPY SCIENCES | 17985-15 | mounting reagent for red chromogen |
| Immpact DAB Peroxidase Kit | Vector | SK-4105 | Used to reveal HRP - DAB (Brown) to replace the DAB coming in the ACD kit |
| lithium carbonate | Fisher chemical | L119-500 | bluing solution |
| paraformaldehyde | ELECTRON MICROSCOPY SCIENCES | 15714-S | for tissue fixation (4%) |
| PBS | life technology | 14190-136 | |
| Permount Mounting Medium | ThermoFisher Scientific | SP15-100 | mounting regaent for brown chromogen |
| Prolong Gold | ThermoFisher Scientific | P36930 | mounting regaent for fluorescence |
| ribonucleases A | ThermoFisher Scientific | 12091039 | for RNAse treatment in DNAscope protocol |
| ribonucleases T1 | Roche | R1003 | for RNAse treatment in DNAscope protocol |
| RNAscope 2.5, 2-plex detection reagent | ACD | 322430 | Brown and red kit chromogen detection |
| RNAscope Target Retrieval Reagents | ACD | 322000 | retrieval buffer |
| SuperFrost Plus Glass Slides | ThermoFisher Scientific | 12-550-17 | |
| TBS | BOSTON BIOPRODUCTS | BM-301-4L | for washes |
| TSA Plus Fluorescence palette kit (Cy3, Cy5, TMR, Fluorescein) | Perkin elmer | NEL760001KT | HRP Fluorescence detection |
| Tween 20 | SIGMA | P1379-1L | for washes |
| XYLENE 20LT | ThermoFisher Scientific | AC422680200 |
참고문헌
- Wang, F., et al. RNAscope: a novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnosis. 14 (1), 22-29 (2012).
- Deleage, C., et al. Defining HIV and SIV Reservoirs in Lymphoid Tissues. Pathogens and Immunity. 1 (1), 68-106 (2016).
- Sengupta, S., Siliciano, R. F. Targeting the Latent Reservoir for HIV-1. Immunity. 48 (5), 872-895 (2018).
- Churchill, M. J., Deeks, S. G., Margolis, D. M., Siliciano, R. F., Swanstrom, R. HIV reservoirs: what, where and how to target them. Nature Reviews Microbiology. 14 (1), 55-60 (2016).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), e2005970 (2018).
- Deleage, C., Chan, C. N., Busman-Sahay, K., Estes, J. D. Next-generation in situ hybridization approaches to define and quantify HIV and SIV reservoirs in tissue microenvironments. Retrovirology. 15 (1), 4 (2018).
- Deleage, C., Turkbey, B., Estes, J. D. Imaging lymphoid tissues in nonhuman primates to understand SIV pathogenesis and persistence. Current Opinion in Virology. 19, 77-84 (2016).
- Estes, J. D., et al. Defining total-body AIDS-virus burden with implications for curative strategies. Nature Medicine. 23 (11), 1271-1276 (2017).
- Mavigner, M., et al. Simian Immunodeficiency Virus Persistence in Cellular and Anatomic Reservoirs in Antiretroviral Therapy-Suppressed Infant Rhesus Macaques. Journal of Virology. 92 (18), (2018).
- Peterson, C. W., et al. Differential impact of transplantation on peripheral and tissue-associated viral reservoirs: Implications for HIV gene therapy. PLoS Pathogen. 14 (4), e1006956 (2018).
- Deleage, C., et al. Impact of early cART in the gut during acute HIV infection. Journal of Clinical Investigation Insight. 1 (10), e87065 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유