Method Article
위장 유기 체의 유전 공학에 대 한 보편적이 고 효율적인 전기 기광 프로토콜
요약
이 프로토콜은 더 큰 플라스미드를 가진 4개의 다른 위장 오르가노이드 실체의 형질전환에 대한 효율적인 전기 천공 방법을 기술한다(10 kB의 범위까지). 그것은 하루 이내에 수행 할 수 있으며 광범위한 준비 또는 특별한, 비용 집약적 인 전기 천공 버퍼가 필요하지 않습니다.
초록
전기 개출은 플라즈마 멤브레인의 전기 투과에 의해 다양한 종류의 분자로 형질 전환하는 일반적인 방법입니다. 지난 몇 년 동안 1 차 적인 환자 물자에 대 한 배양 방법으로 organoids의 사용 증가, 이 3D 문화 시스템에서 유전 공학에 대 한 구성 요소의 효율적인 전송 방법 필요. 특히 오르가노이드의 경우 유전 적 조작의 효율성은 성공적인 형질 감염에 달려 있습니다. 따라서,이 프로토콜은 오르가노이드의 전기 화를 용이하게하고 다른 엔티티에서 보편적 인 기능을 증명하기 위해 개발되었다. 인간 대장, 췌장, 간 및 위암 오르가노이드는 비교하여 크고 작은 플라스미드로 성공적으로 전기화되었습니다. GFP 인코딩 벡터에 기초하여, 형질감염 효율은 FACS에 의해 결정되었다. 세포또는 특별한, 비용 집약적 인 전기 천공 버퍼의 광범위한 준비는 필요하지 않으며, 프로토콜은 하루 이내에 수행 할 수 있습니다.
서문
최근에는 다양한 정상 및 암 조직을 위해 새로운 3D 세포 배양 시스템인 오르가노이드가 개발되었습니다. 오르가노이드는 기능적으로 그리고 형태학적으로 기원의 조직에 매우 가깝습니다. 그들은 다른 종에서 생성 될 수 있으며, 쉽게 확장 할 수 있으며, 게놈으로 안정적이며 유전적으로 수정 할 수 있어 유전자 조사를위한 이상적인 모델 시스템1,2,3. CRISPR (클러스터된 정기적으로 간격을 두는 짧은 Palindromic 반복)/Cas9 시스템과 같은 유전 공학 기술은 다양한 조작을 가능하게 합니다. 클론의 선택은 정의된 미디어 조건에 의해 실현될 수 있고, 예를 들어, APC(선종성 용종염 대장균) 녹아웃 클론4,5에대한 WNT 리간드 철수에 의해 실현될 수 있다. 대안적으로, 선택 마커는 타겟팅 벡터6,7의상동 지시 수리에 의해 도입되어야 한다. 종종 두 개 이상의 플라스미드를 도입해야 하기 때문에 효율적인 형질 전환이 중요한 매개 변수가 됩니다. 또한, 비특이적 오프 타겟 효과를 줄이기 위해, Cas9 엔도너첼리스의 과도 발현이바람직하다 8.
전기 천공은 DNA, RNA, 단백질 또는 다른 거대 분자와 세포를 트랜스펙트하는 비교적 간단한 방법입니다. 전기 펄스에 의해, 세포막은 더 투과성이 되고 증가한 장악력9를일으키는 원인이 됩니다. 이전에 발표된 대장 오르가노이드의 전기개광 프로토콜에서 돼지박 GFP(녹색 형광 단백질)를 발현하는 GFP(7.4 kB)를 가진 30% 효율을 4일절차에서 도달하였다 10. 다음 프로토콜은 단일 가이드 RNA(sgRNA) 및 Cas9 엔도뉴클리스 서열(예: 9.3 kb를 가진 벡터로서 px458)에 대한 큰 플라스미드 인코딩을 가진 암 또는 건강한 오르가노이드의 효율적인 형질변환을 용이하게 하기 위해 개발되었다. 전체 전기 천공 과정은 특별한 전기 천공 완충제없이 하루 내에 수행 될 수 있으며, 다른 위장 유기 체, 즉 췌장 덕트 선암 (PDAC), 대장암 (CRC), 담관 암 (CCC) 및 위암 (GC) 오르간 사이의 적어도 비교 가능한 효율로 수행 할 수 있습니다.
프로토콜
윤리승인은 TU 드레스덴(#EK451122014)의 윤리위원회로부터 획득되었다.
1. 전기 포공 전 의 오르노이드 문화와 준비
- 앞서 설명한 바와 같이 조직 소화에 의한 오르가노이드를 설정하고 지하 매트릭스에서 해당 엔티티 특이적 배양 배지로 확장한다(개요 는 표 1 및 재료 표참조)11,12,13,14,15,16, 17.
참고 : 인간 조직 샘플의 경우 윤리위원회의 동의 및 승인이 필요합니다. - 전기 화 후 파종을 위해 37 °C에서 48 웰 플레이트를 미리 따뜻하게합니다.
- 기저 배지w/o 항생제뿐만 아니라 10 μM Y-27632 및 3 μM CHIR99021을 포함하는 항생제(표 1참조)를 포함하는 엔티티 특이적 오르가노이드 배양 배지를 준비한다.
참고 : 항생제의 철수는 독성 효과를 줄이기 위해 중요합니다. Y-27632 및 CHIR99021 개량세포 회수10. -
오르가노이드의 준비 (그림 1참조)
- 배양 배지에서 전기 개광 샘플 당 48 웰 플레이트에서 5 개의 오르가노이드 우물을 재배하십시오.
참고 : 증식 오르가노이드를 사용해야합니다 (마지막 분할 후 약 2-3 일). - 잘 당 10 μM Y-27632를 포함하여 해리 시약 230 μL (재료 표참조)을 준비하십시오.
- 각각의 웰로부터 배양배지를 제거하고 제조된 해리 혼합물의 230 μL에서 오르가노이드를 기계적으로 해리한다. 15 mL 튜브에 전기 개화 샘플 당 5 웰을 풀.
- 10-15 세포의 클러스터가 발생할 때까지 37 °C에서 5-15 분 동안 와류와 배양하여 혼합합니다. 따라서 해리를 현미경으로 확인하십시오. 최대 10 mL의 항생제를 사용하여 기저 배지를 추가하여 소화를 중지하십시오.
참고 : 이 단계는 매우 중요합니다! 배양이 너무 짧으면 전기 천공 효율이 감소하지만 소화가 길면 생존력이 감소합니다. - 실온에서 5 분 동안 450 x g의 원심 분리기를 폐기하고 4 mL의 전기 천자 버퍼로 두 번 씻으십시오 (재료 표참조).
- 배양 배지에서 전기 개광 샘플 당 48 웰 플레이트에서 5 개의 오르가노이드 우물을 재배하십시오.
2. 전기 화기
참고: 다음 프로토콜은 사각파 및 분리된 포링 및 전송 펄스 서열을 할 수 있는 전기 포어기를 위해 개발되었습니다(그림 2참조). 선택적으로, 임피던스 값뿐만 아니라 샘플로 전달되는 전압, 전류 및 에너지는 재현 가능한 실험을 위한 제어로 측정될 수 있습니다.
- 플라스미드 DNA 30 μg를 함유한 전기 천공 완충제(재료 표참조)에서 오르가노이드 펠릿을 다시 중단합니다.
참고: 사용되는 플라스미드 DNA의 농도는 전기 천공 공정 중에 최적의 염 농도를 위해 5 μg/μL을 초과해야 합니다. 따라서 벡터의 준비를 위해 내분비 플라스미드 맥시 키트(재료 표참조)가 권장됩니다. 최대 45 μg의 DNA를 세포 독성 효과 없이 사용할 수 있습니다. - 기포를 생성하지 않고 2mm 간격폭의 전기 광자 큐벳에 완전한 DNA-오르가노이드 혼합물을 분배하십시오.
- 후지이 외10에 따라 전기 천자 매개변수를 설정합니다(표 2, 도 2참조).
- 손가락으로 큐벳을 눌러 거품없이 약간 세포를 혼합합니다. 큐벳을 큐벳 챔버에 넣습니다.
- 전기 포더의 Ω 버튼을 누르고 임피던스 값을 기록합니다.
참고: 30-40Ω 사이의 임피던스로 최상의 결과를 나타낸 것입니다. 일반적으로 30-55 Ω 사이의 범위에 있어야합니다. 그렇지 않은 경우, 다음 측면을 제어하십시오 : 사용되는 큐벳의 갭 폭, 전기 포더의 케이블 연결, 가능한 기포, 정확한 부피와 전기 천공 혼합물의 염 농도. - 시작 버튼을 눌러 전기 천공 프로그램을 실행하고 표시된 전류, 전압 및 에너지값을 제어합니다.
참고: 측정된 전압, 전류 및 에너지값은 설정된 전기 천공 파라미터와 일치해야 합니다. 반복되는 실험을 비교하려면 이러한 데이터를 기록하는 것이 유용할 수 있습니다. - 전기 천공 후, 즉시 500 μL의 배양 배지를 항생제로 추가하십시오 (CHIR99021 및 Y-27632; 1.3 단계 참조). 위아래로 파이펫팅하여 섞어서 흰색 폼을 해리합니다.
참고 : 백색 폼은 전기 개화 과정 후에 나타나며 많은 수의 세포가 부착됩니다. 따라서 세포를 잃지 않는 것이 매우 중요합니다. - 큐벳에서 완전히 전기 개출 큐벳에 속하는 파이펫을 사용하여 새로운 15 mL 튜브로 샘플을 옮김을 옮니다(재료 표참조). 큐벳을 기저 배지로 다시 헹구는 것이 좋습니다.
- 세포의 재생을 위해 실온에서 40 분 동안 배양하십시오.
3. 세포의 파종
- 실온에서 5 분 동안 450 x g의 세포를 원심 분리하고 상한체를 버립니다.
- 펠릿을 지하 매트릭스 100 μL에서 다시 중단하고 종자 20 μL을 미리 따뜻하게 한 48 웰 플레이트에 떨어뜨립니다 (단계 1.2 참조). 중합을 위해 37 °C에서 10 분 동안 배양하고 재배 된 오르가노이드의 다음 분할까지 Y-27632 및 CHIR99021로 보충되는 배양 배지250 μL을 추가합니다 (약 5-7 일).
4. 형질감염 효율의 결정
참고: 일반적으로, 추가적인 형질감염 제어로서 형광 마커를 운반하는 벡터를 전기화하는 것이 좋습니다. 선택된 마커 및 그의 발색환 성숙에 따라 형광은 약 24-48 시간 포스트 형질 감염18내에서 볼 수 있을 것이다.
- 형질전환 대조군에서 24-48시간 후에 형광을 현미경으로 확인한다(도 4B참조).
-
형광 활성화 세포 스캐닝 (FACS)
- 1.4.2-1.4.4 단계와 유사하게 세포를 수확하고 단일 세포가 있을 때까지 약 10-20 분 동안 소화합니다. 최대 10 mL의 인산염 버퍼링 식염수(PBS)를 추가합니다.
- 실온에서 5 분 동안 450 x g의 원심 분리기를 폐기하고 상한체를 버립니다.
- 선택적으로 살아있는 세포의 차별을 위해: 인산완충식염수(PBS) 1mL에서 펠릿을 재중단하고 적합한 항체(재료 표참조) 또는 프로피듐 요오드화물(PI)을 추가합니다. 매우 신중하게 만 도청하고 어둠 속에서 실온에서 30 분 동안 배양하여 혼합. 10 mL의 PBS, 원심 분리기로 씻고 상한제를 버립니다.
- PBS의 200 μL에서 세포 펠릿을 다시 중단하고 선택적으로 FACS 튜브로 100 μm 세포 스트레이너를 통해 현탁액을 필터링합니다.
- 적절한 게이팅 전략을 사용하여 FACS 기계에 의해 세포를 분석합니다(그림 3; 도4A)형질전환 효율을 결정한다.
결과
4개의 상이한 암 실체(CRC, CCC, PDAC, GC)의 오르가노이드는 30 μg의 작은 플라스미드(pCMV-EGFP, 4.2 kb) 또는 대형 플라스미드(px458, 9.3 kb)를 사용하여 3회 이상 전기포질하였다. 두 벡터 모두 GFP 카세트를 운반하여 유세포측정에 의한 전기포공 후 형질감염 효율 48h의 측정을 가능하게 한다. 살아있는 세포만을 분석하기 위해, 스캐닝 전에 생명-사멸 항체로 염색을 수행하였다. 게이팅 전략은 그림 3에나와 있습니다.
4개의 오르가노이드 엔티티 에서 4.2 kB 크기의 플라스미드를 더 큰 플라스미드에 비해 더 높은 효율로 형질감염하였다(도 4참조). 작은 플라스미드의 가장 효율적인 형질감염은 92.1±5.2% GFP 양성 세포를 가진 PDAC 오르가노이드에 도달한 반면, 큰 플라스미드는 46.7±3.7%의 효율로 형질감염되었다(평균 ±표준 편차, n=3). 췌장암 오르가노이드에 비해, 더 큰 플라스미드는 53.4±11.7%의 평균 효율을 가진 CRC 오르가노이드로 보다 효율적으로 형질화되었고, 반면 작은 플라스미드는 84.3±5.8%의 평균 효율로 형질감염되었다. 트랜스펙트에 가장 어려운 엔티티는 위암 오르가노이드였다: 크고 작은 플라스미드 둘 다에 대해, 가장 낮은 형질감염 효율은 이 엔티티(각각 32.3±12.7% 및 74.1±5.5%)에 도달하였다. CCC 오르가노이드는 소형 플라스미드에 대해 83.0±13.1%의 평균 형질감염 효율을 나타내고 큰 플라스미드에 대해 39.5±10.4%를 수득하였다.
개념의 증거로서, 인간 정상 위 오르가노이드는 Cas9, GFP 및 TP53을표적으로 하는 2개의 sgRNAs를 위한 px458_Conc2 플라스미드 인코딩으로 전기화되었다. 엑소온 8에 대한 Cas9 유도 이중 가닥 브레이크는 비상동성 최종 결합(NHEJ)에 의해 수리되었고, 그 결과 프레임 시프트 및 결과적으로 유전자의 녹아웃이 발생하였다(보충 도 1참조).
표 1: 기저 매질, 소화 혼합물 및 재배 매체의 조성. 이 파일을 보려면 여기를 클릭하십시오 (다운로드 오른쪽 버튼을 클릭하십시오).

표 2: 후지이 외10에따른 전기 천공 설정 .
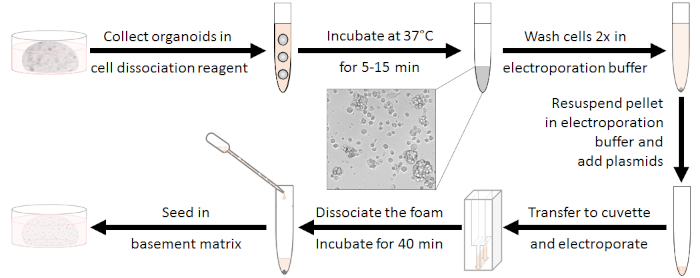
그림 1: 전기 개광 준비 워크플로우. 첫째, 오르가노이드는 10-15 세포의 클러스터에 해리되어야하고 항생제는 씻어 해야합니다. 전기 화 후 흰색 거품을 해리 할 필요가있다. 세포는 실온에서 40 분 동안 재생 한 후 시드 할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
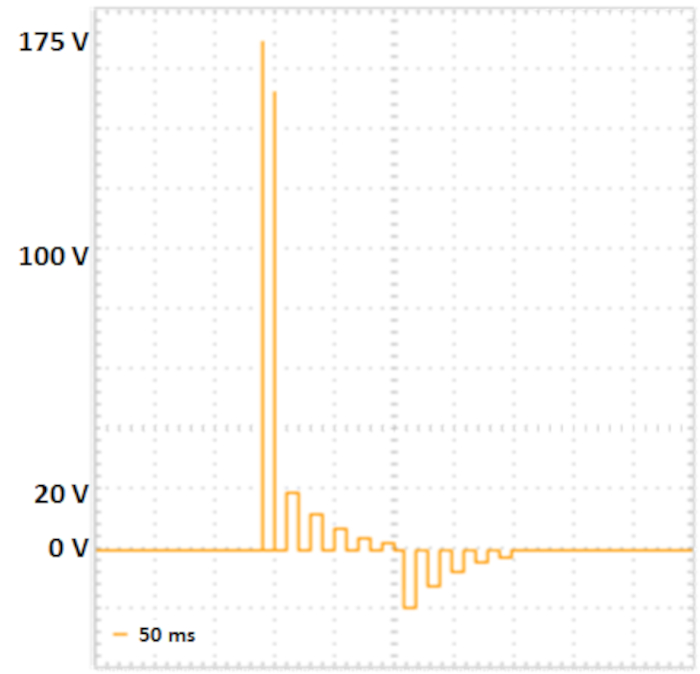
그림 2: 2단계 전기 개공. 전압이 없는 짧은 지속 시간을 가진 두 개의 포링 펄스(각각 5ms, 50ms, 전압 감쇠 10%)에 대해 각각 175V 및 157.5V 세포막의 모공 형성으로 이어질 수 있습니다. 다음 전달 펄스는 세포로 DNA를 전달합니다: 5개의 양성 전달 펄스 (20V, 12V, 7.2V, 4.32 V 및 2,592V, 각각 50ms, 50ms, 전압 감쇠 40%), 5개의 극성 교환 전송 펄스(20V, 12V, 7.2V, 4.32V 및 2,592V, 각각 50ms, 전압 40%)에 대해 일시 정지. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
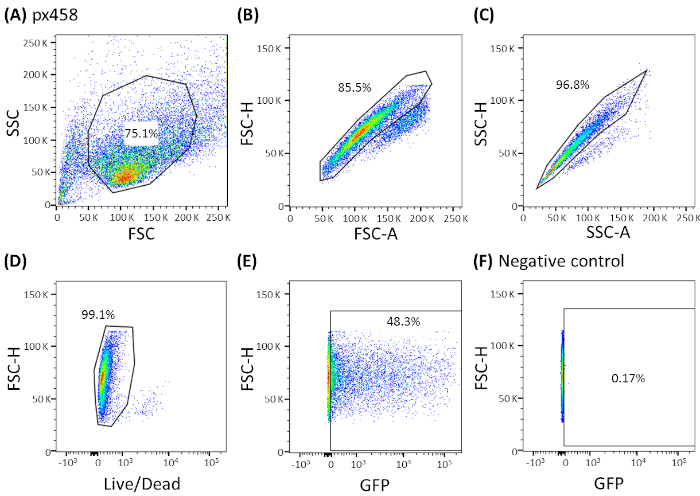
그림 3: CCC 오르가노이드에 의해 도시된 대표적인 게이팅 전략. 모든 전기포상 오르가노이드는 전기천공 후 유세포분석48시간으로 분석되었다. 플라스미드 DNA 없이 전기화된 세포를 음성 대조군으로 사용하였다. 게이트는 세포 형상에 대한 게이팅(A)게이팅,(B,C)단일 세포에 대한 게이팅(doublet discrimination),(D)살아있는 세포에 대한 게이팅(apoptotic 세포에 대한 항체로 염색됨) 및(E,F)마지막으로 eGFP 발현 세포(FITC 채널)에 대한 게이팅. FSC = 정방향 분산; SSC = 측면 분산. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
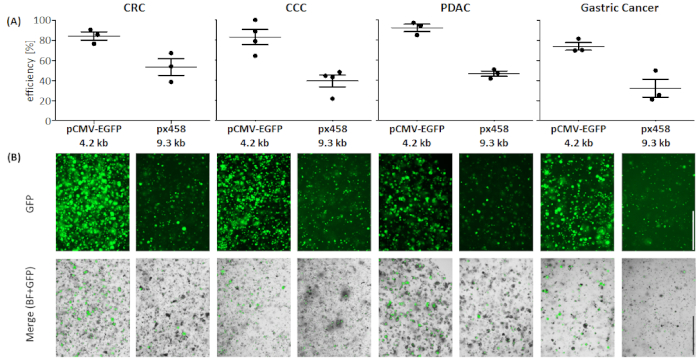
그림 4: 4개의 오르가노이드 엔티티의 전기개공 효율. (A)FACS 분석 (n = 34, 평균 표준 편차 및 각 단일 값이 도시됨) 및(B)형광 현미경에 의한 시각적 비교. 스케일 바 = 1,000 μm. BF = 밝은 필드; CCC = 담관암; CRC = 대장암; GC = 위암; PDAC = 췌장 덕트 선암. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
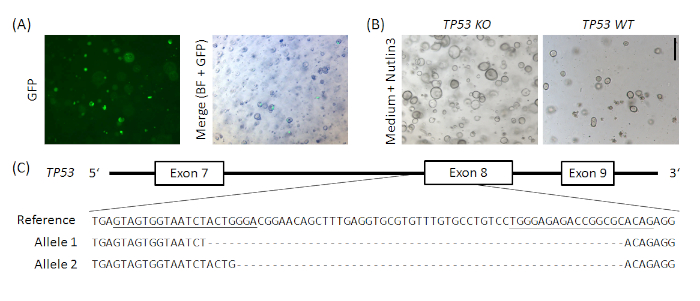
보충 그림 1: 정상적인 인간 위 오르가노이드에서 TP53의 예시CRISPR/Cas9 기반 녹아웃. px458_Conc2 벡터(재료 표참조)는 2gRNA 콘케이트머 벡터, 본경 구19로부터의관대한 선물, px45820을결합하여 복제하였고, 그 결과 2sGRNAs, Cas9 및 GFP에 대한 플라스미드 인코딩이 생성되었다. TP53을 표적으로 하는 2개의 sgRNAs는 황금 문 복제에 의해 px458_Conc2 벡터에서 도입되었다(Andersson-Rolf 등19). 플라스미드 DNA의 10 μg는 인간 정상 위 오르가노이드(A)에서 전기로 하였다. 클론은 Nutlin3 투여(B)에의해 선택되었고 TP53 녹아웃은 TOPO TA 클로닝 및 대두의 시퀀싱에 의해 확인되었으며, 여기서 하나의 클론(C)에 대해 예시하였다. sgRNAs는 참조에 밑줄이 그어져 있습니다. 배율 막대 = 200 μm. BF = 밝은 필드. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
이 프로토콜은 다양한 오르가노이드 엔티티의 전기 천공을 효율적이고 빠르고 쉽게 수행할 수 있는 방법에 대한 자세한 지침을 제공합니다. PDAC, CRC, CCC 및 GC에서 제시된 종양 오르가노이드에 추가, 그것은 뿐만 아니라 건강한 조직에서 파생된 오르가노이드를 위해 성공적으로 작동합니다. 프로토콜은 하루 이내에 수행 할 수 있습니다. 발표된 오르가노이드 형질감염 프로토콜에서 전체 절차는 재배매체10,21의상이한 유형을 가진 제제의 이틀을 포함하여 4일간 지속되었다. 우리의 프로토콜에는 특별한 전처리가 필요하지 않습니다. 전기 개공 전에 전기 개화 완충액으로 세척함으로써 매체의 항생제 성분을 세척하고 최적의 임피던스 값을 위한 식염수 농도의 조정을 달성했습니다. 그럼에도 불구하고 성공적인 전기 천공에 대한 몇 가지 중요한 측면을 고려해야합니다.
셀
Fujii et al.10에 의한 전기 천공 프로토콜에서 단일 세포로 오르가노이드를 해리시키고 20 μm 세포 스트레이너를 통해 필터링하는 것이 좋습니다. 우리의 손에서 단일 세포로의 소화는 세포의 생존력을 강하게 감소시입니다. Merenda등. 21에서제안한 바와 같이, 우리는 또한 10-15 세포의 클러스터에 오르가노이드를 해리하고 단일 세포 해리에 비해 감소 된 효율을 결정할 수 없었다. 전기 천공 후, 부착 된 세포가 손실되지 않도록 흰색 거품을 해리하는 것은 매우 중요한 단계입니다.
2D 세포 배양의 경우, 10분 이상 최대 40분의 전기천공 후 재생 시간이 특히 대형플라스미드(22)의생존성 및 형질감염 효율을 증가시키는 것으로 나타났다. 시험 실험에서, 동일은 이 프로토콜에 있는 전기 천공 후에 40 분의 배양 단계로 이끌어 내는 organoids를 위해 문서화될 수 있었습니다. 전기천공으로부터의 회복을 증가시키기 위해, 우리는 5~7일 동안 Rho 관련 단백질 키나아제(ROCK) 억제제 Y-27632로배양하였다. 유사하게, 글리코겐 신타제 키나제 3(GSK3) 억제제 CHIR99021의 추가 적인 보충은 단일 세포가10을회복하는 데 도움을 주기 위한 것이다.
설정
사용되는 전기 포더의 장점 중 하나는 임피던스가 최적의 조건을 위해 전기 개공 전에 측정 될 수 있다는 것입니다. 제조업체에 따르면 임피던스 값은 30-55Ω이어야 합니다. 30-40Ω의 임피던스 값은 최적의 효율성을 보여주었습니다. 예비 실험에서, 포링 펄스의 상이한 전압 및 펄스 길이 값은 생존율에 대한 효율의 최적 비율을 찾기 위해 변화되었다. 요약하면, 우리는 여기에 설명된 상이한 엔티티에서 Fujii 등10의 기재된 값을 확인할 수 있었다.
Dna
상이한 DNA 양의 효과는 샘플당 최대 45 μg의 DNA를 예비 실험에서 시험하였다. 세포 독성 효과를 감지 할 수 없습니다. 형질감염 효율은 포화 및 30 μg를 가진 용량 의존적인 방법으로 증가하였다. 따라서 최종 프로토콜에서 샘플당 30 μg를 사용했지만 물론 증가될 수 있습니다(예를 들어, 병렬로 더 많은 플라스미드의 전기 포추화). 추가적으로, DNA의 순도 그리고 사격량은 아주 중요한 것처럼 보입니다. 5 μg/μL을 초과하는 농도는 최적의 형광 효율을 보여주었습니다.
예상대로, 9.3 kB 플라스미드는 더 작은 4.2 kB 플라스미드보다 낮은 효율로 형질감염될 수 있다(도 4참조). 10kB보다 더 큰 플라스미드의 사용은 효율성을 더욱 감소시킬 것으로 예상됩니다. 미래 응용을 위해, 이 유전자 운반대는 그(것)들을 더 작게 만드는 플라스미드의 세균성백본이 부족하기 때문에, 벡터로 minicircle DNA를 시험하는 것이 흥미로웠습니다. 이렇게 하면 형질 전환 효율이 향상됩니다. 더욱이, 오르가노이드의 CRISPR 기반 조작의 경우 리보뉴클레오프로테인(RNP) 복합체로서 Cas9에 결합된 sgRNAs의 직접 전기화는 대안 또는 추가25일수 있다.
공개
저자는 공개 할 것이 없다.
감사의 말
줄리안 포그럽, 앤-크리스틴 메이네케, 맥스 헤이덕에게 뛰어난 기술 지원을 부탁드립니다. 자금은 도이치 크렙실페(111350 및 70112925), 산더 스티퉁(2014.104.1), 헥터 스티퉁(No M65.2) 및 유럽 연합(ERC No 639050)이 제공했다.
자료
| Name | Company | Catalog Number | Comments |
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. |  |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. |  |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. |  |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 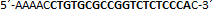 |
참고문헌
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유