Method Article
예방 접종 된 마우스에서 항원 특정 CD8 T 세포의 세포 주기 분석을위한 DNA / Ki67 기반 유동 세포 분석
* 이 저자들은 동등하게 기여했습니다
Erratum Notice
요약
클로날 팽창은 항원 특이적 T 세포 반응의 주요 특징이다. 그러나, 항원 반응 T 세포의 세포 주기는 기술적인 한계 때문에 부분적으로, 제대로 조사되었습니다. 우리는 예방 접종 마우스의 비장 및 림프절에서 복제 확장 항원 특정 CD8 T 세포를 분석하는 유동 세포 측정 방법을 설명합니다.
초록
생체 내항원 특이적 T 세포의 세포 주기는 몇 가지 방법을 사용하여 검사되었으며, 모두 몇 가지 한계를 가지고 있습니다. Bromodeoxyuridine (BrdU)는 S-phase에 있거나 최근에 완료된 세포를 표시하고, 카박스피플루오레세인 succinimidyl ester (CFSE)는 분할 후 딸 세포를 검출합니다. 그러나, 이러한 염료는 분석 시 세포 주기 단계의 식별을 허용하지 않는다. 대안적 접근법은 정지상G0을제외한 세포 주기의 모든 단계에서 세포에 의해 높게 발현되는 마커인 Ki67을 악용하는 것입니다. 불행하게도, Ki67은 이 단계에서 남아 있거나, 사이클링으로진행하거나, G0으로이동할 수 있는 G1의 세포로부터 미토시스에 전념하는 S-phase에서 세포를 분리하지 않기 때문에 추가 분화를 허용하지 않는다.
여기서는 마우스 이차 림프구 기관의 상이한 세포 주기 단계에서 T 세포의 "스냅샷"을 캡처하기 위한 유동 세포 측정 방법을 설명합니다. 이 방법은 Ki67 및 DNA 염색을 주요 조직 적합성 복합체(MHC)-펩타이드 멀티머 염색 및 혁신적인 게이팅 전략과 결합하고,G0,G1, S-G/M 상에서 항원 특이적 CD8 T 세포를 성공적으로 분화할 수 있도록 하는 것은 인간 면역결핍 바이러스(HIV)-1의 모델 항원 개그를 운반하는 바이러스 벡터를 이용한 백신 접종 후 마우스의 비장 및 배수 림프절의 S-G2/M상에서.1.
이 방법의 중요한 단계는 분석 민감도를 높이고 현재 분석 기준에 의해 누락되었을 고도로 활성화/증식하는 항원 특이적 T 세포를 포함하는 DNA 염료 및 게이팅 전략의 선택이었다. DNA 염료, Hoechst 33342는 막및 세포 내 염색을 보존하면서 G0/G1 및 G2/M DNA 피크의 고품질 차별을 얻을 수 있었습니다. 이 방법은 생체 내에서 T 세포 반응에 대한 지식을 증가시키고 면역 모니터링 분석을 개선할 수 있는 큰 잠재력을 가지고 있습니다.
서문
Naïve T 세포는 항원 프라이밍시 클로날 팽창 및 분화를 겪습니다. 분화 된 T 세포는 항원 클리어런스와 항원 특이적 기억유지에 필수적인 이펙터 기능을 표시하며, 이는 오래 지속되는 보호의 핵심입니다. 1차 반응의 첫 번째 단계 동안, 림프구내 의 전문 틈새 시장 내의 항원 제시 세포(APC)와의 순진한 T 세포 상호 작용은 클론 팽창 단계1,2,3을특징으로 하는 거대한 T 세포 증식을 유도하는 데 중요하다. T 세포-APC 상호작용은 T 세포 클론 자손4,5,6,7의양과 질에 영향을 미치는 항원, 공동 자극 신호 및 수용성 인자(사이토카인 및 케모키네)의 농도 및 지속성에 의해 미세하게 조절된다.
T 세포 클론 확장의 집중적인 연구에도 불구하고 항원 프라이밍 T 세포가 항원 인식 부위에서 전체 세포 주기를 완료했는지 또는 세포 주기 진행 중에 다른 장기로 마이그레이션하는지 여부는 아직 알려지지 않았습니다. 지식의 이 부족은 세포 주기 분석에 사용할 수 있는 공구의 속성 때문입니다. 여기에는 핵 마커, Ki67 및 세포 염료에 특이적 단일 클론 항체(mAbs)가 포함되며, 세포 주기의 S-phase를 겪은 세포를 식별하거나 딸 세포와 그 조상들 사이에서 차별(예: 카박스시플루오레신 succinimidyl(ESTERSE).
그러나, CFSE 및 BrdU와 같은 세포 라벨링 염료는, 특정 기관에서 발견된 세포가 로컬로 증식되었는지 또는 오히려 분할후이 사이트로 마이그레이션되었는지 여부에 대한 결정을 허용하지 않는다8,9. 더욱이, 핵 내 단백질인 Ki67은 G 0(Ki67-음성 세포)의 세포와 다른 세포 주기 단계(Ki67 양성 세포)의 세포만 구별할 수 있다. 따라서, Ki67 분석은 활성 증식(즉, S, G2또는 M)에서 세포를G1의세포와 구별하지 않으며, 이는 G1에서 장기간 분할 또는 체류또는10,11로되돌아갈 수 있다.
여기서, 예방접종된 마우스의 비장 및 림프절(LNs)으로부터 항원 특이적 CD8 T 세포의 세포 주기 분석을 위한 새로운 유동 세포측정 방법을 설명한다(도1). 상기 방법은 이전에 마우스골수(BM) 조혈세포의 세포주기를 분석하기 위해 사용하였던 Ki67 및 DNA 염색의 조합을이도(13,14)이다. 여기서는 최근 발표된 혁신적인 게이팅전략(12)과함께 KI67 플러스 DNA 염색을 CD8 T 세포 클론 확장 의 분석에 성공적으로 적용했습니다. G0,G1,S-G2/M상에서 백신 접종 된 마우스의 비장 및 배수 LN에서 항원 특이적 CD8 T 세포를 명확하게 구별 할 수 있었습니다.
프로토콜
마우스는 Plaisant 동물 시설에 보관되었고, 이 작업은 이탈리아 보건부 승인 번호 1065/2015-PR에 따라 수행되었습니다. 이 프로토콜은 국내 및 국제 법률 및 정책에 따라 동물 관리 지침을 따랐습니다(UE 지침 2010/63/UE; 이탈리아 입법 법령 26/2014).
1. 중간 및 염색 용액의 준비
- 완벽한 매체 준비: 로스웰 파크 메모리얼 인스티튜트(RPMI) 배지 2m글루타민, 100 U/mL 페니실린/스트렙토마이신, 50 μM 베타-메카토에탄올, 태아 소 세럼(FBS)의 10% 부피/부피(v/v)
- 염색 버퍼 준비: Ca2+/Mg2 + (PBS) 없이 인산염 완충 식염수 1% 무게/볼륨 (w/v) 소 혈 청 알 부 안 부 안 부 (BSA) 및 2 mM 에틸렌디아민테트라 아제성 산 디나트륨 소금 (EDTA)
2. 마우스 치료
- 프라임 7-8주 된 여성 Balb/c 마우스는 인간 면역결핍 바이러스.m(HIV)-1-개그 발현-침팬지 아데노바이러스 벡터(ChAd3-gag)의 사두근에서107개의 바이러스 성 입자를 투여한다.
- 프라이밍 후 1-4 개월, i.m. HIV-1 개그 표현 수정 된 vaccinia 앙카라 바이러스의 주사에 의해 마우스 를 한 번 밀어 106 플라크 형성 단위의 용량.
- 3일째에 부스트 후, 자궁 경부 탈구에 의해 증폭된 마우스를 희생하고, 치료되지 않은 마우스와 병행하여 분석하십시오.
- 사두근 (iliac, popliteal 및 inguinal)과 비장을 증폭 및 치료되지 않은 마우스에서 배출하는 LN을 수확하십시오. 더욱이, 치료되지 않은 마우스로부터 두 개의 뒷다리로부터 BM을 수집하고, 이 BM을 유동 세포계 설정에 사용하고 세포 주기 분석을 위한 양수 대조군(도2)을사용한다.
참고: 이전에 설명된 바와 같이 ChAd3-gag 및 MVA 개그 벡터를생성합니다.
3. LN, 비장 및 BM 세포를 배출하는 격리
- 비장 및 LN 세포의 격리
- 15mL 튜브 2개 각각에 5mL의 완전한 배지를 놓고 얼음 위에 보관하여 장기를 수집할 준비를 합니다.
- 자궁 경부 탈구로 성인 마우스를 희생하십시오.
- 마우스를 등에 놓고 70% v/v 에탄올로 피부 표면을 살균합니다.
- inguinal LN을 수집하려면 가위로 복부에 ~ 1cm 세로 절개를 하고 집게로 절개를 늘게 하십시오.
- 피부의 내부 표면에 잉구인 LN을 시각화하고 집게로 수확합니다. 3.1.1 단계에서 제조된 2개의 15mL 튜브 중 하나에 잉구이널 LN을 놓습니다.
- 비장을 수집하려면 가위로 간절을 하고 비장을 제거하십시오. 주변 결합 조직을 절단한 후 비장을 제2 15mL 튜브에 넣고 3.1.1 단계에서 제조하였다.
- iliac LN을 수집하려면 창자를 옆으로 옮기고 열등한 베나 카바 에 가까운 iliac LN을 시각화한 다음 집게를 사용하여 수집합니다. 잉구이날 LN을 포함하는 동일한 튜브에 iliac LN을 놓습니다.
참고: 염색을 위한 충분한 LN 셀(섹션 4 참조)을 얻으려면 한 마우스에서 포라이트, 잉게날 및 iliac LN을 풀링하는 경우가 많습니다. 이 LNs는 모두 사두근 (i.m. 예방 접종 부위)를 배출하고 있습니다. 이 프로토콜은 풀이 된 LN의 15mL 튜브 를 하나만 사용합니다. - 포라이트 LN을 수집하려면 뒷다리의 피부를 잡고 근육을 밝히기 위해 부드럽게 아래쪽으로 당깁니다. 그런 다음, 무릎 관절 아래 근육 사이에 집게를 삽입하고, 포라이트 LN을 수집합니다.
참고: 3.1.7 이후의 참고 를 참조하십시오. - 비장을 완전한 배지 5mL로 채워진 60mm 배양 접시 내에 70 μm 셀 스트레이너에 넣습니다. 5mL 주사기 플런저를 사용하여 전체 분리될 때까지 장기를 부드럽게 으깬다.
- 스트레이너를 제거하고 셀 서스펜션을 깨끗한 15mL 튜브로 옮기십시오.
- 문화 접시에 완전한 배지 5mL을 추가하고 모든 세포가 회복되었는지 확인하기 위해 접시와 여과기를 조심스럽게 씻으세요. 15 mL 튜브에 비장 세포 서스펜션의 나머지 와 수영장.
- 풀링된 잉구이날, iliac 및 포플라이트 LN의 경우 비장에 대해 3.1.9 ~ 3.11 단계에서 사용되는 유사한 절차에 따라 단일 셀 서스펜션을 준비합니다.
- 4°C에서 10분 동안 400 × g의 원심분리기 세포. 상체를 버리고 PBS에서 세포 펠릿을 다시 중단합니다.
- 적혈구 용해 버퍼와 PBS에서 0.04 % v / v 트라이팬 블루를 사용하여 Neubauer 챔버로 세포를 계산합니다.
- BM 셀의 격리
- 15mL 튜브에 5mL 완성 매체를 배치하고 얼음 위에 보관하여 뒷다리 컬렉션을 준비합니다.
- 자궁 경부 탈구로 성인 마우스를 희생하십시오.
- 70% v/v 에탄올로 피부 표면을 살균합니다.
- 가위로 복부 피부에 ~1cm의 횡방향 절개를 하고, 절단의 양쪽에 있는 피부를 단단히 잡고, 천천히 아래쪽으로 당겨 뒷다리의 근육을 발견합니다.
- 뒷다리 의 뒤쪽에서 피부를 제거하려면 마우스를 척추 위치에 두고 무릎 아래에 클램프를 놓고 위쪽으로 당겨 근육을 노출시하십시오.
- 골반/엉덩이 관절과 발목 : 한 뒷다리의 두 사지에서 뼈를 잘라.
- 3.2.1 단계에서 준비된 15mL 튜브로 뒷다리를 옮기십시오. 튜브를 얼음 위에 보관하십시오.
- 15mL 튜브에서 뒷다리를 가져 와서 티슈 페이퍼로 옮기십시오. 경골을 제거하기 위해 무릎 관절 바로 아래에 뒷다리를 잘라. 주위 근육에서 대퇴골과 경골을 해부하고, 가위를 사용하여 과도한 조직을 제거하고, 티슈 페이퍼를 적십합니다.
- 내부 골수 샤프트를 노출하는 가위로 뼈 끝을 잘라. BM 추출 튜브에 경골과 대퇴골을 삽입하십시오(3.2.9.1-3.2.9.218의준비 참조), 하단에 가장 넓은 끝이 있습니다.
- 팁 끝 부분과 100 μL 선에서 200 μL 파이펫 팁을 잘라냅니다.
- 중간 부분을 팁의 상부, 더 큰 섹션에 넣고 1.5 mL 마이크로 퍼지 튜브에 놓습니다.
- BM 추출 튜브를 800 × g에서 1 분 동안 회전합니다.
- 뼈를 버리고, 모든 클러스터를 제거하기 위해 완전한 매체의 1 mL에서 펠릿을 적극적으로 다시 분리합니다. 15mL 튜브 의 상단에 배치 된 70 μm 필터를 통해 셀 서스펜션을 필터링합니다.
- BM 추출 튜브를 매번 1mL의 완전한 배지로 두 번 세척합니다. 70 μm 필터를 통해 필터를 필터링하고, 3.2.11 단계에서 얻은 셀 서스펜션의 나머지 부분과 함께 부피를 풀.
참고: 단일 15 mL 튜브는 마우스의 양쪽 뒷다리에서 세포를 포함합니다. - 4°C에서 10분 동안 400 × g의 원심분리기 세포. 상체를 버리고 PBS에서 세포 펠릿을 다시 중단합니다.
- 적혈구 용해 버퍼와 PBS에서 0.04 % v / v 트라이팬 블루를 사용하여 Neubauer 챔버로 세포를 계산합니다.
4. 비장, LN 및 BM 세포의 염색
- 3개의 하위 그룹으로 염색될 세포 견본을 분할합니다: 처리되지 않은 마우스에서 BM 세포를 포함하여 보상을 위한 세포 견본은Hoechst 33342 (Hoechst로 불린) 및 처리되지 않은 마우스에서 비장 세포가 죽은 세포 염료 보상을 위한 죽은/살아있는 세포 혼합을 준비하는 데 이용되는 것을 처리되지 않은 마우스에서 만 염색하는 것을 위한; 세포 주기 분석을 위한 양성 제어,치료되지 않은 마우스로부터의 BM 샘플로 구성된; 치료되지 않은 쥐의 비장 및 LN 샘플을 포함하는 실험 샘플.
참고: 개그 별 CD8 T 셀의 충분한 수를 분석하기 위한 충분한 비장 및 LN 셀이 있는지 확인합니다. 3개의 예방 접종된 마우스에서 풀링된 비장 세포와 풀링된 LN 세포를 사용해야 하며, 각각 3개의 ×106세포를 함유하고 있는 풀링된 세포의 2개 이상의 동일한 샘플을 염색할 필요가 있다. Hoechst 염색 단계에서 동일한 샘플을 병합합니다. 유사하게, 3개의 처리되지 않은 마우스에서 얼룩 풀린 비장 세포 및 LN 세포, 끝에 동일한 견본을 병합합니다. 계측기 및 보상 설정에 사용할 처리되지 않은 마우스에서 비장 세포의 얼룩이 없는 샘플을 따로 놓습니다. - 죽은 세포 염료 보상을 위해 죽은 / 살아있는 세포 믹스를 준비하십시오 (세포의 이 혼합은 죽은 세포 염료로만 염색됩니다).
- 65°C에서 수유를 가열합니다.
- 비장 세포의 알리쿼트(~3~×106)를섭취하십시오.
- 세포 현탁액을 마이크로퍼지 튜브로 옮기고 65°C에서 5분 동안 수조에 넣고 즉시 얼음 위에 10분 간 놓습니다.
- 열사세포를 1:1의 비율로 라이브 비장 세포(~3×106)와혼합하고, 혼합물의 절반을 96개의 잘 둥근 바닥 플레이트로 옮기십시오(~3×106세포/죽은 세포 염색 제어를 위한 우물).
- 실험 샘플의 데드 셀 염색, 세포 주기 분석을 위한 양성 제어 및 죽은/라이브 셀 믹스
- 전비, LN, BM 세포(3× 106세포/웰), 및 데드/라이브 셀 믹스(section 4.2)를 96웰 원형 바닥 플레이트로 옮기고, 스테인링 방식(단계 4.1)에 따라 원심분리기는 4°C에서 3분 동안 ×.
- PBS에서 희석된 죽은 세포 염료의 50 μL에서 각 세포 펠릿을 재연하고, 즉시 3회 위아래로 피펫팅하여 재연한다.
- 4°C에서 30분 동안 배양하여 빛으로부터 보호합니다.
- 염색 버퍼로 셀을 2번 세척; 200 μL을 가진 처음으로 250 μL로 두 번째. 각 세척 원심분리기당 400g에서 4°C에서 3분 동안 플레이트를 ×.
- 상체를 버리고 PBS의 20 μL에서 셀 펠릿을 다시 중단합니다.
- 주요 조직적합성 복합체(MHC)-펩타이드 멀티머 및 mAbs를 사용하여 멤브레인 셀 염색.
- 스테인링 방식(흐름 사이토미터 설정, 표 1)에따라 필요한 볼륨을 고려하여 다음 시약을 준비합니다.
- 적절한 희석에 따른 염색 버퍼에서 mAb 2.4G2를 희석(재료표참조); 염색할 각 샘플에 대해 이 희석의 10 μL을 사용하십시오.
참고: 2.4G2 mAb블록 비항원 특이적 면역글로불린을 FcγIII 및 FcγII 수용체에 차단한다. - 적절한 희석을 얻기 위해 염색 버퍼에서 H-2k (d) AMQMLKETI 알로피코시아니 (APC)라벨 테트라머 (Tetr-gag)를 희석하십시오 (재료의 표참조); 염색할 각 샘플에 대해 이 희석의 20 μL을 사용하십시오.
- 이전에 적정 실험에서 결정된 적절한 희석(재료 표참조)에 따라 염색 버퍼에서 mAbs를 희석시킴으로써 항체 혼합물을 준비; 염색될 각 시료에 대해, 이 항체 혼합물의 20 μL을 사용하십시오.
참고: 여기서, 안티 CD3e 페리딘 엽록소 단백질 (PerCP-Cy5.5) (클론 145-2C11), 안티 CD8a 화려한 자외선 (BUV805) (클론 53-6.7), 및 안티 CD62L phycoerythrin cyanine7 (PECy7) (MELone 14) 사용하였다.
- 적절한 희석에 따른 염색 버퍼에서 mAb 2.4G2를 희석(재료표참조); 염색할 각 샘플에 대해 이 희석의 10 μL을 사용하십시오.
- 이전에 희석된 2.4G2 mAb(단계 4.4.1.1)의 10μL을 추가하고 빛으로부터 보호된 4°C에서 10분 동안 배양합니다.
- 이전에 희석된 테트르 개그 APC(4.4.1.2단계)와 H-2k(d) AMQMLKETI 피코레테린(PE) 펜타머(펜트 개그)의 10μL을 추가합니다. 4°C에서 15분 동안 배양하여 빛으로부터 보호합니다.
- 이전에 준비된 항체 믹스(Step 4.4.1.3)의 20μL을 추가하고, 빛으로부터 보호되는 4°C에서 15분 인큐베이션을 한다.
참고: 따라서 최종 부피는 우물당 80 μL입니다(단계 4.3.5, 단계 4.4.2 ~ 4.4.4). - 염색 버퍼의 200 μL로 셀을 세척합니다. 4°C에서 5분 동안 400 × g의 원심분리기.
- 염색 버퍼의 250 μL에서 세포 펠릿을 다시 중단하고 셀 서스펜션을 5mL 튜브로 옮기습니다. 튜브에 염색 버퍼 1mL을 추가하고 원심분리기를 4°C에서 5분 동안 400 × g에서 추가합니다.
- BM 세포의 알리쿼트(3×106세포)를 사용하여 Hoechst 채널(Hoechst 33342)을 보정하고 자외선 레이저(유량 사이토미터 설정(표2)에의해 흥분되고, 세포 현탁액을 5mL 튜브로 전송한다. 튜브에 염색 버퍼 1mL을 추가하고 원심분리기 400 × g를 4°C에서 5분 동안 추가합니다.
- 스테인링 방식(흐름 사이토미터 설정, 표 1)에따라 필요한 볼륨을 고려하여 다음 시약을 준비합니다.
5. 고정/투과성
- 제조업체의 지침에 따라 고정/투과성 희석 의 3 부분으로 고정/투과성 농축물의 1부분을 희석하여 신선한 고정/투과화 버퍼를 준비하십시오.
- 상체및 펄스 소용돌이를 폐기하여 샘플을 완전히 분해합니다.
- 스테인처리되지 않은 비장 세포(3 x 106,셀 샘플 목록 참조, 섹션 4.1) 및 소용돌이가 있는 튜브를 포함하여 각 튜브에 갓 준비된 고정/투과화 버퍼 1mL을 추가합니다.
- 4 °C에서 16 시간 동안 배양하십시오.
참고: 프로토콜은 여기에서 일시 중지할 수 있습니다.
6. 세포 내 염색
- Ki67 염색
- 제조업체의 지시에 따라 충류 버퍼 10배를 증류수로 희석하여 신선한 투과화 버퍼 1x를 준비하십시오. 사용하기 전에 투과화 버퍼 1x는 0.45 μm 필터를 통해 필터링하여 응집체를 제거해야 합니다.
- 희석 mAb Ki67 형광 이소티오네이트(FITC) (clone SolA15)에서 투과완 버퍼 1배(재료표참조), 적정 실험에서 이전에 결정된 바와 같이(샘플당 100 μL의 최종 부피).
- 각 튜브에 3mL의 투과화 버퍼1x를 추가하고, 원심분리기는 실온(RT)에서 5분 동안 400 × g로 추가합니다.
- 상신을 버리고 6.1.3 단계를 반복하십시오.
- 상체를 버리고, 이전에 희석된 mAb Ki67 FITC(6.1.2단계)의 100 μL에서 세포 펠릿을 다시 분리한다.
- 빛으로부터 보호된 RT에서 30분 동안 배양합니다.
- 4mL의 충류 버퍼 1x로 셀을 2회 세척합니다. 각 세척 원심분리기는 400 g에서 RT에서 5 분 동안 ×.
- PBS에서 세포 펠릿을 재연하여 다음과 같은 부피를 고려한다: 350 μL의 PBS 샘플은 유동 세포계에서 직접 획득되는; 시료에 대한 PBS의 250 μL은 혈류 세포측정(섹션 6.2)이 되기 직전에 Hoechst와 함께 배양된다.
- DNA 염색
- PBS에 4 μg/mL Hoechst의 250 μL을 각 샘플에 추가합니다(Hoechst의 최종 농도는 2 μg/mL).
참고: PBS에서 250 μL의 2개 이상의 동일한 시료가 제조된 경우, 이 단계에서 병합하고 PBS에서 4 μg/mL Hoechst 용액의 동일한 부피를 추가합니다(Hoechst의 최종 농도는 2 μg/mL). 세포의 수는 DNA 염색 단계에 크게 영향을 미칩니다. 각 샘플에서 동일한 셀 번호를 사용합니다. 약간 감소된 세포 수(예를 들어, 이전 세척 단계의 세포 손실로 인해)조차도 DNA에 더 높은 Hoechst 결합을 초래하고 Hoechst 강도가 높다는 점에 유의하십시오. - 빛으로부터 보호된 RT에서 15분 동안 배양합니다.
- RT에서 5 분 동안 400 × g에서 샘플을 원심 분리합니다.
- PBS의 350 μL에서 세포 펠릿을 다시 중단합니다.
- PBS에 4 μg/mL Hoechst의 250 μL을 각 샘플에 추가합니다(Hoechst의 최종 농도는 2 μg/mL).
7. 보상 비드 샘플 준비
- 염색 버퍼에서 mAb를 적절히 희석하여 항체의 5 μL을 준비합니다.
참고: 실험에 사용되는 각 불소-컨쥬게이트 mAb에 대해 해당 보상 비드 샘플을 준비합니다. - 소용돌이 부정적인 제어 및 안티 쥐 / 햄스터 Ig, θ 사용 전에 컴프 비드.
- 각 샘플에 대해 음수 제어 컴비드 1방울(~20 μL)과 안티래트/햄스터 이그, k 컴비드 한 방울을 소개합니다.
- 미리 희석된 항체(7.1단계)의 5μL을 튜브에 넣고 위아래로 파이프를 넣습니다.
- 4°C에서 15분 동안 배양하여 빛으로부터 보호합니다.
- 염색 버퍼 2mL로 샘플을 세척합니다. 4°C에서 5분 동안 400 × g의 원심분리기.
- 상체를 버리고 각 튜브및 소용돌이에 PBS 500 μL을 추가하여 펠릿을 다시 중단하십시오.
8. 유동 세포계에서 의기및 보상 설정 및 실험 샘플 수집
참고: 사이토미터 구성의 흐름 사이토미터설정(표 2)을참조하십시오.
- 일반 기기 및 보상 설정
- 샘플 수집을 위한 소프트웨어를 열고(재료 표참조) 작업 영역 리본 섹션에서 새 실험을 클릭하고 새 빈 실험을 선택하여 새 실험을만듭니다.
- 생성된 실험을 두 번 클릭하여 엽니다.
- 사이토미터 설정 창에서 매개 변수를 클릭하고 전방 분산(FSC) 및 사이드 스캐터(SSC) 매개 변수를 포함한 스테닝 패널에 사용되는 모든 채널(예: PE, APC 등)을 선택합니다.
- 로그 스케일의 선택을 해제하여 선형 배율을 Hoechst 매개 변수로 선택하고 FCS, SSC 및 Hoechst에 대한 전압 펄스의 너비(W)를 확인합니다.
참고: 모든 매개 변수는 선형 축척인 FSC 및 SSC를 제외한 logarithmic(로그) 축척으로 기본적으로 표시됩니다. 모든 파라미터는 전압 펄스의 영역(A) 및 높이(H)에 의해 분석됩니다. - 전역 워크시트에서x축에 FSC-A가 있는 점 플롯과 y축의 SSC-A를 만듭니다.
- 수집 대시보드에서 데이터 수집을 클릭하여 스테인드비린지 비장 샘플을 실행합니다.
- 매개 변수 섹션의 전압 값을 수정하여 셀을 시각화하도록 적절한 FSC 및 SSC 설정을 설정하고 전역 워크시트의작업 공간 도구 모음의 다각형 게이트를 클릭하여 FSC-A/SSC-A 도트 플롯에 표시되는 모든 셀을 선택하는 게이트를 만듭니다.
- x 축에 각 형광 매개 변수와 함께 히스토그램에 게이트 된 세포를 표시합니다.
- 각 형광 파라미터에 대한 스테인드 셀의 음수 및 양성 신호 사이의 명확한 분리를 갖도록 형광 검출기(PMT)를 조정하기 위해 얼룩지지 않고 완전히 염색된 비장 샘플을 실행합니다.
- 보상 설정을 수행하려면 작업 공간 리본 및 보상 설정 섹션에서 실험을 클릭하고 보상 컨트롤 만들기를 선택합니다. 확인되지 않은 컨트롤 튜브/웰 포함 및 확인을 클릭합니다.
참고: 이 작업을 수행하면 보상 컨트롤이라는 표본과 선택한 각 매개 변수에 해당하는 여러 시트가 포함된 일반 워크시트가 생성됩니다. - 보상 구슬 샘플을 실행합니다(섹션 7 참조) 적절한 FSC 및 SSC 설정을 설정하여 Cytometer 창의 FSC 매개 변수에서 전압 값과 5,000의 획득 임계값을 수정하여 구슬을 시각화합니다.
- 비드 모집단의 P1 게이트를 조정하고 양수 및 음수 피크가 모두 x축에 표시되는지 확인합니다. 각 보상 비드 샘플에 대해 이 작업을 반복하고 마지막으로 수집 대시보드의 레코드 데이터를 클릭하여 각 샘플 파일을 기록합니다(각 샘플에 대해 최소 5,000개의 이벤트를 기록).
- 기록된 각 비드 샘플에 대해 P2 및 P3 게이트를 각각 양수 및 음수 피크에 설정합니다.
- 보상을 위해 셀 샘플을 실행합니다(4.2 단계 및 4.4.7 단계 및 섹션 5 및 6)를 참조하십시오. FSC 및 SSC 전압과 임계값값을 수정하여 셀을 시각화하고, P1 게이트를 조정하고, 마지막으로 각 샘플 파일을 기록합니다(최소 10,000개의 이벤트를 기록). P2 및 P3 게이트를 각각 양수 및 음수 피크에 설정합니다.
참고: Hoechst 채널의 보정을 위해 G0/G1을 음수 피크(P3)와 G2/M을양수(P2)로 사용한다. - 작업 공간 리본 섹션과 보상 설정 섹션에서 실험을 클릭하여 보상 계산을선택합니다.
- 생성된 보정 설정의 이름을 지정하고 현재 실험에 저장합니다.
- 실험 샘플 수집
- 브라우저 도구 모음에서 새 표본을 클릭하여 표본을 열고 전역 워크시트에서게이팅 전략을 만듭니다.
참고: 샘플 수집의 게이팅 전략은 그림 3 및 섹션 9에 설명된 샘플 분석과 유사합니다. - x축에 CD3-A가 있는 히스토그램에 모든 이벤트 모집단을 표시합니다. 간격 게이트를 만들어 CD3+ 셀만 선택합니다.
- 수집 대시보드에서저장소 게이트를 LN 샘플의 모든 이벤트및 비장 샘플의 모든 이벤트 또는 CD3+ 셀로 선택합니다.
- 실험 용 샘플을 저속으로 실행하고 마지막으로 예방 접종 된 마우스의 각 샘플에 대해 적어도 100-200 항원 별 CD8 T 세포를 수집하도록 모든 파일을 기록합니다.
참고: 실험 용 샘플의 파일 크기는 일반적으로 큰 (30-120 MB), 특히 항원 특정 CD8 T 세포의 주파수가 낮은 경우. 따라서, 이벤트의 높은 숫자 (> 1 × 106)적어도 100-200 항원 특이 CD8 T 세포를 기록하기 위해 수집되어야한다. 큰 파일은 후속 데이터 분석 프로세스를 느리게 할 수 있습니다. 비장 샘플에서 CD3+ 셀만 획득하면 파일 크기를 더 작게 유지하는 데 도움이 됩니다(위의 단계 8.2.2 참조). - 세포 주기 분석에 대한 양성 제어를 실행하고 기록, 즉, 치료되지 않은 마우스에서 BM 샘플.
- 브라우저 도구 모음에서 새 표본을 클릭하여 표본을 열고 전역 워크시트에서게이팅 전략을 만듭니다.
9. 데이터 분석
- 소프트웨어를 열고(재료테이블참조) 작업 공간 리본 섹션에서 그룹 만들기 섹션을 클릭하여 분석할 다른 장기에 해당하는 다른 그룹을 만듭니다(즉, 그룹 "a-LNs"를 만듭니다.) "b-비장"; "c-BM").
참고: 새로 만든 그룹이 그룹 목록에 표시되고 "보상" 그룹은 소프트웨어에 의해 자동으로 생성됩니다. - 그룹 이름을 두 번 클릭하여 그룹 수정 창을 열고 새로 만든 그룹이 동기화되어 있는지 확인합니다. 그렇지 않은 경우 동기화된 함수에 체크표시를 삽입합니다.
- 해당 그룹에서 각 .fcs 파일을 드래그합니다.
- "a-LNs" 그룹으로 시작하는 게이팅 전략을 만듭니다.
- 그래프 창을 열기 위해 그룹의 완전히 스테인드 된 샘플을 두 번 클릭합니다. x 축은 fcs 파일과 같이 레이블이 지정됩니다(흐름 사이토미터 설정, 표 2참조).
- y축의 x 축에 DNA-A및 DNA-W를 사용하여 이 샘플을 위해 획득한 총 이벤트를 도트 플롯에 표시합니다.
- 그래프 창의 게이팅 도구 섹션에서 사각형을 클릭하여 단일 셀 채우기만 선택합니다.
참고: 단일 세포는 다음과 같이 DNA-A 값을 가지고 있습니다: 2N (낮음): 2N과 4N (중간), 또는 4N (높음)과 동일합니다), DNA-W 값은 그들 모두에 대해 동일합니다 (그림 3의단계 1). - 직사각형 게이트의 중앙을 두 번 클릭하여 y축의 x축 및 죽은 셀 염료에 FSC-A 매개 변수가 있는 도트 플롯에 단일 셀을 표시합니다.
- 그래프 창의 게이팅 도구 섹션에서 다각형을 클릭하여 라이브 셀 채우기만 선택합니다. 살아있는 세포는 죽은 세포 염료에 대한 음수 (도 3의단계 2).
- 다각형 게이트의 중앙을 두 번 클릭하여 y축의 x축 및 SSC-A 매개 변수에 FSC-A 매개 변수가 있는 점 플롯에 셀을 표시합니다.
- 사각형을클릭하고 해당 그래프12(그림 3의3단계)에 모든 단일 라이브 셀을 포함하는 "편안한" 게이트를 만듭니다.
- "편안한" 게이트 의 중앙을 두 번 클릭하여 x 축에 CD3가 있는 점 플롯에 셀을 표시하고 y축에 CD8을 표시합니다.
- 다각형 (그림 3의4 단계)을 클릭하여 CD3+CD8+ 셀을 선택합니다.
- CD3+CD8+ 게이트의 중앙을 두 번 클릭하여 x 축에 Tetr-gag와 y 축의 펜트 개그와 함께 점 플롯에 셀을 표시합니다.
- 다각형(도 3의5단계)을 클릭하여 항원 특이적 CD8 T 세포(테트르 개그와 펜트 개그 모두에 대해 양성)를 선택한다.
- 개그 별 게이트의 중앙을 두 번 클릭하여 x축에 DNA-A가 있는 점 플롯에 세포를 표시하고y축(그림 4)에Ki67을 표시한다.
- 그래프 창의 게이팅 도구 섹션에서 Quad를 클릭하여 서로 다른 세포 주기 단계에서 셀을 선택합니다.
참고: G0 상에 있는 세포는 Ki67neg DNA 저세포 (왼쪽 사분면 하부); G1의 세포는 Ki67pos-DNA 저(왼쪽 위 사분면)입니다. S-G2/M의세포는 Ki67pos-DNA 중간/높음(오른쪽 상단 사분면)(그림4)이다. - 한 샘플에서 만든 게이팅 전략을 해당 그룹에 복사하여 그룹의 모든 샘플에 게이트를 적용합니다.
- "A-LN 그룹"에 대해 9.5~ 9.18단계를 반복합니다.
- 모든 게이트가 "b-비장" 그룹의 각 샘플에 적합한지 확인합니다. BM 세포(positive control)의 세포 주기를 분석하려면"이완된" 게이트의 중앙을 클릭하여 y축의 x축 및 Ki67에 DNA-A를 사용하여 점도플롯에 세포를 표시한다.
- 모든 게이트가 3군의 각 샘플(즉, 비장, LN 및 BM의 셀)에 적합한지 확인합니다.
참고: 세포 주기(step 9.17)를 위한 단하나 세포 집단 게이트(step 9.7) 및 쿼드 게이트는 주로 샘플(섹션 6.2)의 Hoechst 염료 강도의 가능한 약간의 차이로 인해 다른 샘플에서 서로 다른 게이트 좌표를 가질 수 있다. 이러한 이유로 각 샘플에서 세포 주기에 대한 단일 셀 모집단 게이트및 쿼드 게이트를 수정해야 할 수도 있습니다. 그룹 이름을 두 번 클릭하고 그룹 속성에서 동기화를 제거합니다. 이 작업을 사용하면 그룹의 다른 모든 샘플에서 동일한 게이트를 수정하지 않고 하나의 샘플에서 게이트를 수정할 수 있습니다. 동기화 제거 후 필요한 경우 게이트를 수정합니다. - 이 분석에서 얻은 결과를 시각화하려면 작업 공간 리본 섹션의 레이아웃 편집기를 클릭하여 엽니다. 샘플 창의 게이팅 전략의 각 게이트를 레이아웃 편집기로 드래그하고 게이팅 전략의 순서에 따라 플롯을 배치합니다. 필요한 경우 레이아웃의 해당 플롯을 두 번 클릭하고 그래프 정의 창에서 적절한 형식을 선택하여 그래프 유형을 변경합니다.
- 그룹을 클릭하고 레이아웃 리본의 함수에 의해 반복하여 각 기관에서 얻은 결과를 시각화하고 다른 샘플을 비교합니다.
결과
도 1에서요약된 프로토콜에 따라 Balb/c 마우스의 비장, LNs 및 BM으로부터세포의 세포 주기 단계는 형광 DNA 염료, Hoechst 및 항 Ki67 mAb를 사용하여 분석하였다. 이러한 염색은 세포 주기의 다음 단계에서 세포의 분화를 허용했다: G0 (Ki67neg, DNAlow로 정의 된 DNA의 2N), G1 (Ki67pos, DNAlow), 및 S-G2/M (Ki67pos, 2N과 4N 사이에 구성 된 DNA 함량, 또는 DN/DN로 정의 된 DNA의 4N).
우리는 먼저 이전에 발표 된 결과13,14를 재현하기 위해 BM 세포의 세포 주기 분석을 수행한 다음 관심있는 세포, 즉 CD8 T 세포를 분석했습니다. 도 2는 BM 세포의 세포 주기 분석의 전형적인 예를나타낸다(도 2A). 이 프로토콜은 G0/G1 및 G2/MDNA 피크의 낮은 계수(CV)를 산출하여 DNA 염색의 우수한 품질을 나타내며(도2B,CV < 2.5를 가진 예를 나타낸다; CV는 항상 모든 실험에서 5<).
그런 다음 예방 접종된 마우스로부터 항원 특이적 CD8 T 세포에 동일한 프로토콜을 적용했습니다. BALB/c 마우스는 HIV-1 개그를 운반하도록 설계된 프라이밍용 채드3 개그와 MVA-gag를 사용하여 HIV-1의 항원 개그에 대한 예방 접종을 받았습니다. 날 (d) 3 포스트 부스트에서, 우리는 비장과 배수 LN에서 개그 특정 CD8 T 세포의 주파수를 분석했다. 우리는 기존의 전략과 는 달리, 고도로 활성화 된 항원 반응 CD8 T 세포(12)를검출하는 데 적합한 면역 반응의 초기 단계에서 T 세포에 대한 새로 정의 된 게이팅 전략을 활용하였다. 우리는 다섯 단계의 새로운 전략을 실행했습니다. 1단계에서는 DNA-A/-W 게이트에 의한 이중 또는 골재를 배제하고, 2단계에서는 죽은 세포 마커 배제에 의해 살아있는 세포를 확인했습니다. 3단계에서는 표준좁은 림프구게이트(12)대신 비통상적 "완화" FSC-A/SSC-A게이트(그림 3A)를사용하여 관심인구를 규명하였다. CD3+CD8+ 셀 (그림 3A의4 단계)에 게이팅 후, 우리는 두 개의 서로 다른 MHC 멀티머, 즉 펜트 개그와 테트개그 (그림 3A의단계 5)를 사용하여 개그 특정 CD8 T 세포를 식별했습니다. 치료되지 않은마우스(도 3B 및 C,단계 5)의 염색 배경을 증가하지 않고, 예방 접종 된 마우스에서 개그 특정 CD8 T 세포 검출의 감도를 향상시키기 위해 하나 대신 두 개의 멀티머를 사용했다. 따라서, 우리는 성공적으로 예방 접종 마우스에서 치료되지 않은 마우스 (0.00 % 및 0.00 % 항원 특이 CD8 T 세포, LNs및 비장에서 각각) 예방 접종 마우스에서 (0.46 % 및 0.29% LNs 및 비장의 항원 특이적 CD8 T 세포, 각각, 도 3B 및 C)를구별하였다.
특히, 이 프로토콜을 통해 우리는 LNs의 항원 특이적 CD8 T 세포 게이트와 치료되지 않은 마우스의 비장(보통 0.00% 및 최대 0.02%)에서 매우 낮은 배경을 가질 수 있었습니다. 개그 특이적 및 개그별 FSC-A/SSC-A 플롯의 비교는 개그 특이적 세포가 높은 SSC-A 및 FSC-A(도3D)를가지고 있음을 보여주었으며, 이러한 세포를 포착하기 위해 "편안한" FSC-A/SSC-A 게이트를 사용할 필요성을 확인하였다. 그런 다음 상이한 세포 주기 상에서 개그 특이적 CD8 T 세포의 백분율을 평가하였다(그림4A). 우리는 비장에서 개그 특정 CD8 T 세포가 배수 LN에서 더 많은 3 사후 부스트 (18.60 %와 33.52 %, 각각)에서 S-G2/ M 단계에서 세포의 높은 비율을 포함하는 것으로 나타났습니다.
더욱이, 우리는 S-G2/M상에서 개그 특이적 CD8 T 세포가 동일한 장기(도4B)로부터총 CD8 T 세포에 오버레이될 때 높은 FSC-A 및 SSC-A를 가졌다는 것을 발견했습니다. 개그 특이적 CD8 T 세포에 의한 CD62L 발현은 LN(도4C)에서G0내의 몇 세포를 제외하고 활성화된 T 세포에 대해 예상대로 낮았다. 모두, 이들 결과는 "이완된" 게이트(도 3A, B 및 C의3단계)가 증식하는 항원 특이적 CD8 T세포(12)를모두 포함하도록 요구되었다는 것을 확인하였다. 프로토콜은 상이한 세포 주기 상에서 세포에 의한 분석 및 CD62L 발현의 분석 시 항원 특이적 CD8 T 세포의 세포 주기 단계의 "스냅샷" 평가를 위해 매우 유용했다.

도 1: 항원 특이적 CD8 T 세포의 세포 주기 분석을 위한 프로토콜의 구성. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
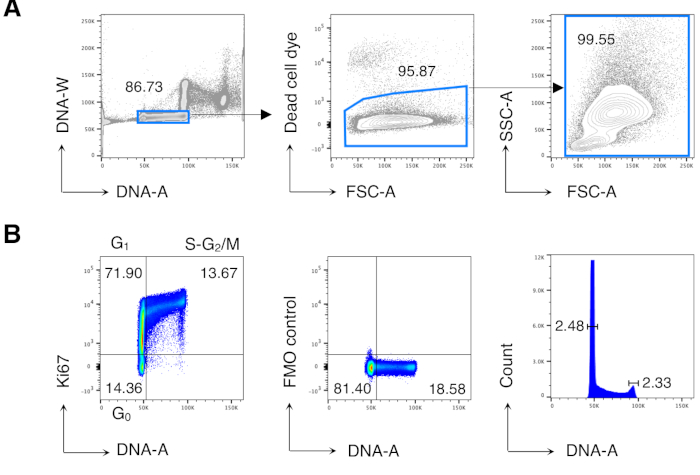
그림 2: BM 세포의 세포 주기 분석. 치료되지 않은 Balb/c 마우스에서 BM 세포는 혈류 세포측정에 의해 염색되고 분석되었다. (A) 게이팅 전략의 예. 우리는 DNA-A/-W 플롯(왼쪽)의 단일 세포에 게이트를 두었고, 그 후 죽은 세포 염료 배제(가운데)에 의해 살아있는 세포에 문이 있었습니다. 그런 다음 모든 BM 셀(오른쪽)에 대해 "편안한" FSC-A/SSC-A 게이트가 사용되었습니다. (B) BM 세포의 세포 주기 분석의 예 (왼쪽). 우리는 세포 주기의 다음 단계에서 세포를 식별하기 위해 Ki67및 DNA 염색의 조합을 사용: G0 (아래 왼쪽 사분면, Ki67neg-DNAlow 세포), G1 (상단 왼쪽 사분면, Ki67pos-DNAlow), S-G2/M (오른쪽 상단 사분면, Ki67pos-DNA 중간/높은). 키67 mAb(가운데)와 DNA 히스토그램(오른쪽)의 형광 마이너스 원(FMO) 제어가 도시된다. DNA 히스토그램 플롯에서, 좌우 게이트는 G0/G1 및 G2/MDNA 피크에 각각 대응하며, 숫자는 각 피크의 변이계(CV)를 나타낸다. 다른 모든 플롯에서 숫자는 표시된 게이트의 셀 백분율을 나타냅니다. 그림은 5개 중 1개의 대표적인 실험을 보여줍니다. 각 실험에서, 우리는 3 마우스에서 풀린 BM 세포를 분석했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
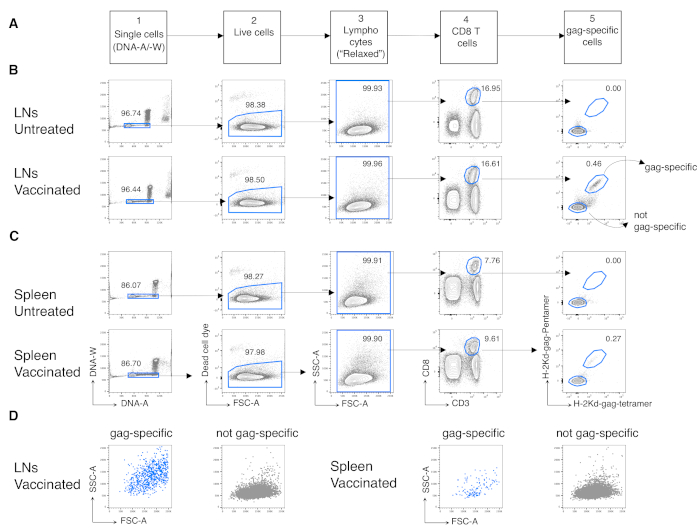
그림 3: LNs 및 비장으로부터 항원 특이적 CD8 T 세포의 분석. Balb/c 마우스는 Chad3-gag와 근육질(i.m.) 프라이밍되었고 MVA-gag로 i.m를 높였습니다. 3일째에, 예방 접종 및 치료되지 않은 대조군 마우스로부터 LN 및 비장 세포를 배출하는 후 혈류 세포측정에 의해 염색되고 분석하였다. (A) 단일 세포를 식별하는 5단계로 게이팅 전략의 계획(1단계); 라이브 셀 (2 단계); 림프구 (3 단계); CD8 T 셀(4단계); 개그 별 세포(5단계). (B-C) 플롯의 예: (B) LNs 및 (C) 비장의 세포 분석 (상단) 및 예방 접종 (하단) 마우스. 우리는 1 단계에서 DNA-A / -W 플롯에 단 하나 세포를 확인했습니다. 그런 다음, 2단계에서, 우리는 죽은 세포 염료 배제에 의해 살아있는 세포를 선택했습니다. 3단계에서는 림프구에 대해 비정식 "편안한" 게이트를 사용했습니다. 4단계에서는 CD3 및 CD8의 이중 발현으로 CD8 T 셀을 확인했습니다. 그런 다음 5단계에서 개그 별 세포를 식별했으며, 불소크롬 라벨이 부착된 H-2kd-개그 펜타머(펜트 개그)와 H-2kd-개그-테트라머(Tetr-gag)를 각각 묶는 능력에 따라 개그 특이성 세포를 확인했습니다. (D) 상기와 같이 게이팅 후 개그 특이적(파란색)과 개그 특이적(grey) 세포의 FSC-A/SSC-A 프로파일. 숫자는 표시된 게이트의 셀 백분율을 나타냅니다. 그림은 5개 중 1개의 대표적인 실험을 보여줍니다. 각 실험에서, 우리는 3명의 예방 접종마우스및 3개의 처리되지 않은 마우스에게서 풀로 풀린 비장을 분석하고 풀로 한 LN 세포를 분석했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
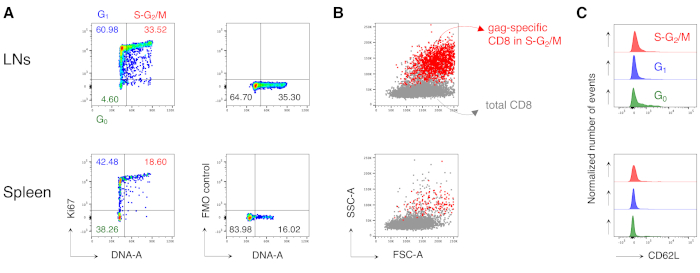
도 4: 항원 특이적 CD8 T 세포의 세포 주기 분석. 마우스는 도 3에서와 같이 예방 접종을 받았고, 개그 특이적 세포의 세포 주기 분석은 도3에서와 같이 5단계로 게이팅 후 3일째 에 수행되었다. (a)예방 접종된 마우스의 LNs(위) 및 비장(bottom)으로부터 개그 특이적 CD8 T 세포의 세포 주기 분석의 예. 세포 주기 단계는 도 2B에서와같이 확인되었다. 패널은 G0,G1,S-G 2/M(왼쪽) 및 형광 마이너스 원(FMO) 제어에서 Ki67 mAb(오른쪽)의 세포를 나타낸다. 숫자는 표시된 게이트의 셀 백분율을 나타냅니다. (B) FSC-A/SSC-A 도트 플롯은 S-G2/M상(빨간색)에서 개그 별 CD8 T 세포를 총 CD3+CD8+ T 세포(회색)에 오버레이하여 백신 접종된 마우스의 LNs(위쪽)와 비장(아래쪽)에서 겹쳐낸다. (C) G 0(녹색)에서 개그 특이적 CD8 T 세포에 의해 CD62L 발현을 나타내는 히스토그램을 상쇄하고, G1(파란색), S-G 2/M(빨간색)에서 LNs(위) 및 백신 접종 된 마우스의 비장(bottom)에서 분리된다. y축은 정규화된 이벤트 수를 나타냅니다. 이 수치는 총 15개의 마우스를 가진 5개의 독립적인 실험 중 1개의 대표적인 예를 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 재료 : 흐름 세포계 설정. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
T 세포 복제 확장집중적으로 연구되었지만, 일부 측면은 알 수없는 상태로 남아 있습니다. 이러한 관점에서, 우리는 마우스 모형에 있는 백신 주사 후에 초기에 항원 특정 CD8 T 세포의 세포 주기를 분석하기 위하여 고과민성 유동 세포 측정 방법을 설치했습니다. 상기 프로토콜은 이전에 마우스13,14에서BM 조혈 세포의 세포 주기를 분석하기 위해 사용된 Ki67 및 DNA 염색의 조합을 기반으로 한다. 항원 특이적 CD8 T 세포에 프로토콜을 적용하기 위해 DNA 염료 의 선택, 다른 샘플에 걸쳐 유사한 DNA 염색을 얻기 위한 적절한 조건, 데이터 분석을 위한 게이팅 전략을 포함하여 몇 가지 중요한 문제를 고려해야 했습니다.
많은 염료는 프로피듐 이오디드와 7-아미노락티노마이신 D를 포함하여 DNA 염색에 사용할 수 있습니다. 우리는 Ki67 염색에 필요한 멤브레인 염색 및 가벼운 고정 / 투과화 프로토콜과 호환되었기 때문에 Hoechst를 선택했습니다. 동시에, Hoechst를 염색하면 우수한 품질의 DNA 히스토그램을 얻을 수 있었으며, 즉 G0/G1 및 G2/MDNA 피크는 일반적으로 다른 DNA 염료로 얻은 DNA 피크보다 훨씬 낮은 계수(CV)를 가졌다( DRAQ519). 실제로, Hoechst는 살아있는 세포20에서도DNA를 얼룩지게 할 수 있습니다.
일부 전략은 동일한 실험의 다른 샘플에서 Hoechst 강도의 변동을 피하기 위해 사용되었다. Hoechst 염색은 시간 동안 염료 강도의 감소를 최소화하기 위해 유동 세포계에서 시료 수집 직전에 수행되었다. 수많은 샘플과 큰 실험에서 프로토콜을 재현하는 데 관심이있는 사람들을 위해 한 번에 몇 가지 샘플에 Hoechst 염색을 수행하는 것이 좋습니다. 또 다른 단점은 Hoechst 강도가 염료를 가진 잠복기 도중 세포 수에 의해 무겁게 영향을 받을 수 있다는 것입니다. 이러한 이유로, 우리는 강력하게 DNA 염색을위한 샘플 당 세포의 동일한 수와 동일한 볼륨을 사용하는 것이 좋습니다. 유동 세포계에서 획득하기 위해 많은 수의 세포가 필요한 경우, 두 개 이상의 동일한 샘플을 준비하고 Hoechst 염색 단계 직전에 병합하는 것이 좋습니다.
프로토콜의 핵심은 데이터 분석을 위한 게이팅 전략입니다. 최근에는 면역 반응 초기에 T 세포 분석을 위한 새로운 전략을 발표하여 항원 특이적 T세포(12)의검출 민감도를 높일 수 있게 되었다. 이 전략을 다음과 같이 여기에 표시된 데이터에 적용했습니다. 첫째, 우리는 DNA-A/W 플롯에서 세포 응집체를 제외했습니다. 둘째, 죽은 세포를 게이팅 한 후, 우리는 FSC / SSC 플롯 ("편안한 게이트")에서 상당히 큰 림프구 게이트를 사용했습니다. 이 전략에 의해, 우리는 이러한 세포가 높은 FSC-A 및 SSC-A를 가지고 있기 때문에, 일반적으로 현재 게이팅 전략에 의해 놓친 S-G2/M에 고도로 활성화 된 항원 특이CD8 T 세포를 포함 할 수 있었다. 요약하자면, 데이터 분석은 항원 특이적 T 세포의 활성/증식의 민감한 검출을 얻는 데 필수적인 방법의 중요한 부분을 나타낸다.
이 방법은 면역 반응의 초기 단계에서 중요한 T 세포 데이터를 누락할 가능성을 방지하고 T 세포 면역 모니터링을 위한 새로운 관점을 엽니다. 향후 개선은 G2와 M21사이의 분화를 허용하는 인히스톤 3에 대한 염색을 포함할 수 있다. 현재 한계는 세포가 핵 마커, Ki67에 대한 얼룩을 위해 고정되고 투과화되어야 한다는 것입니다. 따라서 셀은 정렬 및 후속 기능 분석과 같은 다른 유형의 분석에 사용할 수 없습니다. 더욱이, Hoechst를 포함하여 DNA 염료는 일반적으로 게놈 DNA 분석을 방해하고 평가의 이 모형을 위해 적합하지 않습니다. 다른 세포 주기 단계와 상관 관계가 있고 살아있는 세포에 얼룩질 수 있는 막 마커의 확인은 이 한계를 극복할 수 있었습니다. 결론적으로, 이 방법은 예방 접종, 감염, 면역 매개 질환 및 면역 요법과 같은 여러 맥락에서 T 세포를 활성화/증식하는 평가를 위한 큰 잠재력을 가지고 있습니다.
공개
A. Folgori와 S. Capone은 Reithera Srl. A. 니코시아의 직원으로 특허 출원 WO 2005071093 (A3) "침팬지 아데노바이러스 백신 캐리어"에 발명가로 임명되었습니다. 다른 저자는 공개 할 것이 없습니다.
감사의 말
이 작품은 MiUR 프로젝트 2017K55HLC_006 의해, 로미아에 의해 지원되었다, 5 × 에 의해 1000 아소시아지오네 이탈리아어 리스크카 술 칸크로에서 보조금 (AIRC). 다음 테트라머는 NIH 테트라머 시설을 통해 획득되었다: APC-컨쥬게이드 H-2K (d) HIV 개그 197-205 AMQMLKETI.
자료
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
참고문헌
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- . Mouse phenotype Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020)
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Erratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021. Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:

Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유