JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
세포외 플럭스 분석을 이용한 단일 3D 미세조직 구상체의 미토콘드리아 에너지 대사 탐구
Erratum Notice
요약
이 프로토콜은 사용자가 Seahorse 세포외 플럭스 분석을 사용하여 3D 암 세포주 유래 구상체에서 미토콘드리아 에너지 대사를 조사하는 데 도움이됩니다.
초록
구상체라고 불리는 3차원 (3D) 세포 응집체는 최근 몇 년 동안 시험관 내 세포 배양의 최전선이되었습니다. 세포를 2차원, 단세포 단층(2D 배양)으로 배양하는 것과는 달리, 스페로이드 세포 배양은 세포외 매트릭스 단백질의 발현, 세포 신호전달, 유전자 발현, 단백질 생산, 분화 및 증식을 포함하여 생체 내에 존재하는 생리적 세포 구조 및 특성을 촉진, 조절 및 지원한다. 3D 배양의 중요성은 종양학, 당뇨병, 줄기 세포 생물학 및 조직 공학을 포함한 많은 연구 분야에서 인정 받고 있습니다. 지난 십 년 동안 구상체를 생산하고 신진 대사 기능과 운명을 평가하기 위해 개선 된 방법이 개발되었습니다.
세포외 플럭스(XF) 분석기는 XF24 섬 포획 플레이트 또는 XFe96 스페로이드 마이크로플레이트를 사용하여 구상체와 같은 3D 미세조직에서 미토콘드리아 기능을 탐색하는 데 사용되었습니다. 그러나, XF 기술을 이용한 구상체에서 미토콘드리아 에너지 대사를 조사하는 뚜렷한 프로토콜과 최적화는 상세히 설명되지 않았다. 이 백서에서는 XFe96 XF 분석기가 있는 스페로이드 마이크로플레이트를 사용하여 단일 3D 구상체에서 미토콘드리아 에너지 대사를 조사하기 위한 상세한 프로토콜을 제공합니다. XF 기술은 서로 다른 암 세포주를 사용하여 크기가 다른 것뿐만 아니라 다른 부피, 세포 번호, DNA 함량 및 유형의 3D 구상체에서 세포 호흡을 구별 할 수있는 것으로 입증되었습니다.
올리고마이신, BAM15, 로테논 및 안티마이신 A의 최적의 미토콘드리아 이펙터 화합물 농도는 3D 구상체에서 미토콘드리아 에너지 대사의 특정 파라미터를 조사하는 데 사용됩니다. 이 백서에서는 또한 구상체에서 얻은 데이터를 정상화하는 방법에 대해 논의하고 XF 기술을 사용하여 스페로이드 대사를 탐구 할 때 고려해야 할 많은 고려 사항을 다룹니다. 이 프로토콜은 고급 시험관 내 스페로이드 모델에 대한 연구를 추진하는 데 도움이됩니다.
서문
생물학적 연구에서 시험관 내 모델의 발전은 지난 20 년 동안 빠르게 진행되었습니다. 이러한 모델에는 이제 오르간-온-칩 양식, 오가노이드 및 3D 미세조직 구상체가 포함되며, 이들 모두는 시험관내 및 생체내 연구 사이의 번역을 개선하기 위한 공통적인 초점이 되었다. 고급 시험관 내 모델, 특히 구상체의 사용은 조직 공학, 줄기 세포 연구, 암 및 질병 생물학 1,2,3,4,5,6,7 및 유전 독성 학 8,9,10, 나노 물질 독성학 11을 포함한 안전성 테스트를 포함한 여러 연구 분야에 걸쳐 있습니다. 12,13,14, 및 약물 안전성 및 효능 시험8,15,16,17,18,19.
정상 세포 형태학은 생물학적 표현형과 활성에 매우 중요하다. 세포를 3D 미세조직 구상체로 배양하면 세포가 형태학, 표현형 기능 및 아키텍처를 채택할 수 있으며, 이는 생체 내에서 관찰되는 것과 비슷하지만 고전적인 단층 세포 배양 기술로 포획하기는 어렵습니다. 생체내 및 시험관내 둘 다에서 , 세포 기능은 세포 미세환경에 의해 직접적으로 영향을 받는데, 이는 세포 통신 및 프로그래밍에 한정되지 않는다(예를 들어, 세포-세포 접합 형성, 세포 틈새를 형성할 기회); 즉각적인 환경에서 호르몬 및 성장 인자에 대한 세포 노출 (예를 들어, 염증 반응의 일부로서 세포 사이토카인 노출); 물리적 및 화학적 매트릭스의 조성 (예를 들어, 세포가 뻣뻣한 조직 배양 플라스틱 또는 탄성 조직 환경에서 성장하는지 여부); 그리고 가장 중요한 것은 세포 신진 대사가 영양과 산소에 대한 접근뿐만 아니라 젖산과 같은 대사 폐기물의 처리에 의해 어떻게 영향을 받는지입니다.
대사 플럭스 분석은 정의된 시험관내 시스템 내에서 세포 대사를 검사하는 강력한 방법입니다. 특히, XF 기술은 무손상 세포 및 조직의 세포 생물 에너지학의 실시간 변화를 실시간으로 분석 할 수있게합니다. 많은 세포 내 대사 사건이 수초에서 분 정도 내에 발생한다는 것을 감안할 때, 실시간 기능적 접근법은 시험관 내에서 손상되지 않은 세포 및 조직에서 세포 대사 흐름의 실시간 변화를 이해하는 데 가장 중요합니다.
이 논문은 강제 응집 접근법을 사용하여 시험관내 3D 스페로이드 모델로서 암 유래 세포주 A549 (폐 선암종), HepG2/C3A (간세포 암종), MCF-7 (유방 선암종) 및 SK-OV-3 (난소 선암종)을 배양하기 위한 프로토콜을 제공한다 (그림 1). 또한 (i) 애질런트 XFe96 XF 분석기를 사용하여 단일 3D 구상체의 미토콘드리아 에너지 대사를 조사하는 방법을 자세히 설명하고, (ii) 단일 3D 구상체를 사용하여 XF 분석을 최적화하는 방법을 강조하고, (iii) 이 접근법을 사용하여 3D 스페로이드 대사를 조사하는 데 있어 중요한 고려 사항 및 한계에 대해 논의합니다. 가장 중요한 것은,이 논문은 산소 소모율 (OCR)의 계산이 산화 인산화 및 세포 구상체에서 미토콘드리아 기능을 결정할 수있게 해주는 데이터 세트가 수집되는 방법을 설명합니다. 이 프로토콜에 대해 분석되지는 않았지만 세포외 산성화 속도 (ECAR)는 XF 실험에서 OCR 데이터와 함께 측정되는 또 다른 매개 변수입니다. 그러나 ECAR은 종종 XF 데이터 세트에서 잘못 해석되거나 잘못 해석됩니다. 우리는 기술 제조업체의 기본 접근 방식에 따라 ECAR 계산의 한계에 대한 논평을 제공합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
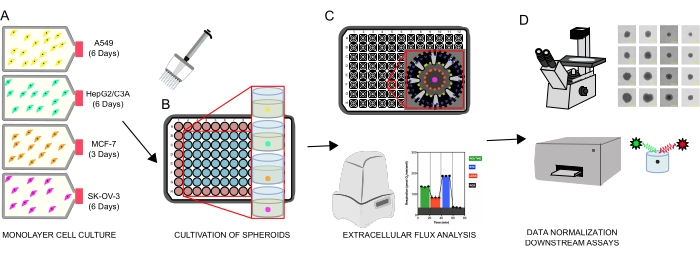
그림 1: 세포 구상체 생성을 위한 그래픽 워크플로우, 세포외 플럭스 분석 및 다운스트림 분석. 네 개의 암 세포주를 단층(A)으로 선택적으로 배양하고, 조직 배양 플라스크로부터 분리하고, 초저 부착 96-웰 마이크로플레이트에 시딩하여 구상체(B)를 형성하였다. A549 폐 암종, HepG2/C3A 간 암종, SK-OV-3 난소 선암종 및 MCF-7 유방 암종 세포를 1 × 10 3-8 × 103 세포/웰에서 시딩하고 7일까지 성장시켜 단일 구상체를 형성하고 지속적인 관찰 및 평면 측정을 통해 스페로이드 시딩 밀도 및 배양 시간을 최적화하였다. 일단 형성되면, 단일 구상체를 무혈청 XF 배지로 세척하고, 폴리-D-리신 (C)으로 예비코팅된 스페로이드 분석 마이크로플레이트에 조심스럽게 시딩하였다. 스페로이드는 해결하기 위해 몇 가지 프로토콜을 사용하여 XFe96 분석기를 사용하여 세포외 플럭스 분석을 실시하였다: (1) 기저 미토콘드리아 호흡 반응을 위한 최적의 스페로이드 크기; (2) 미토콘드리아 호흡기 억제제의 최적화된 적정; (3) 마이크로플레이트 웰 내의 스페로이드 배치의 최적화. (d) 사후 XF 분석, 위상차 현미경 및 스페로이드 DNA 정량화를 데이터 정규화 및 기타 하류 시험관내 분석에 사용하였다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 암세포주를 3D 체외구 상체로 육성
| 세포주 | 묘사 | 배양 배지 | 근원 |
| A549 | 폐 암종 세포주 | RPMI 1640 | 인증된 세포 배양의 유럽 컬렉션 (ECACC) |
| 피루베이트 나트륨 (1 mM) | |||
| 페니실린 - 스트렙토 마이신 - (100 U / mL - 100 mg / mL) | |||
| 10 % (v / v) FBS | |||
| 헵G2/C3A | 간암종 세포주, 모체 HepG2 세포주의 클론 유도체 | 증권 시세 표시기 | 미국 조직 문화 컬렉션 (ATCC) |
| 페니실린 - 스트렙토 마이신 - (100 U / mL - 100 mg / mL) | |||
| 10 % (v / v) FBS | |||
| MCF7 | 유방 선암종 세포주 | RPMI 1640 | 인증된 세포 배양의 유럽 컬렉션 (ECACC) |
| 피루베이트 나트륨 (1 mM) | |||
| 페니실린 - 스트렙토 마이신 - (100 U / mL - 100 mg / mL) | |||
| 10 % (v / v) FBS | |||
| SK-OV-3 | 난소 선암종 세포주 | RPMI 1640 | 인증된 세포 배양의 유럽 컬렉션 (ECACC) |
| 피루베이트 나트륨 (1 mM) | |||
| 페니실린 - 스트렙토 마이신 - (100 U / mL - 100 mg / mL) | |||
| 10 % (v / v) FBS | |||
| 구성 요소 | RPMI 분석 배지 (최종 부피 50 mL) | ||
| 기본 매체 | 애질런트 해마 XF RPMI, pH 7.4 | ||
| 포도당 (1 M 멸균 스톡) | 11 mM (0.55 mL 원액) | ||
| L-글루타민 (200 mM 멸균 스톡) | 2 mM (0.5 mL의 원액) | ||
| 소듐 피루베이트 (100 mM 멸균 스톡) | 1 mM (0.5 mL의 원액) |
표 1: 암 세포주 배지 및 XF 배지 조성물.
- 표준 무균 조직 배양 기술을 사용하여 모든 세포주를 배양하고 적합한 분석 키트를 사용하여 마이코플라즈마가 없는지 확인합니다.
- 세포주를 T75 조직 배양 플라스크 또는 이와 동등한 권장 배지를 사용하여 배양한다(표 1). 세포주를 65-80% 컨플루언시로 배양하고 최대 25 계대까지 정기적으로 계대시킨다.
- Dulbecco의 변형된 인산염 완충 식염수(DBPS)에서 세포 배양 플라스크를 두 번 헹구십시오.
- 3 mL의 세포 해리 시약( 표 참조)으로 플라스크로부터 세포를 분리하고 37°C에서 5분 동안 현미경으로 분리를 확인하였다.
- 분리된 세포 현탁액을 부드럽게 흡인하여 단일 세포 현탁액을 보장하고, 7 mL의 완전한 조직 배양 배지로 세포 해리 시약을 비활성화시킨다.
- 세포를 300 × g 에서 5분 동안 원심분리하여 수집하고, 상층액을 버리고, 세포를 완전 배지에 재현탁시킨다.
- 혈구세포계 또는 자동화된 세포 계수기를 사용하여 세포를 계수하고 시딩에 필요한 원하는 세포 밀도로 적정한다.
참고: 전체 96웰 플레이트를 100 μL/웰에서 4 × 103 세포/웰에서 시드하려면 세포를 12mL의 권장 부피로 4 × 104 세포/mL로 적정해야 합니다. - 세포 현탁액을 멸균 저장소로 데칸트하고, 100 μL의 세포 현탁액을 다채널 피펫터를 사용하여 세포 기피제 마이크로플레이트의 각 웰에 분배한다.
참고: 마이크로플레이트의 내부 60개 웰만 시드하고 나머지는 DPBS로 채워야 합니다. 이것은 증발 장벽을 형성하고, 플레이트 전체에 걸쳐 스페로이드 균질성을 보장하며, 플레이트 에지 효과를 최소화합니다. - 스페로이드 마이크로플레이트를 300 × g 에서 15분 동안 원심분리하여 세포를 느슨한 응집체 내로 강제한다.
- 플레이트를 스페로이드 형성을 보장하기 위해 최소 3일 동안 37°C, 5%CO2 에서 인큐베이션한다.
- 표준화 된 실험실 관행을 사용하여 위상차 현미경을 수행하여 구상체의 성장을 모니터링하십시오. 세포 배양 배지를 3일마다 또는 매주 두 번 반부피 배지 교환을 수행하여 보충한다.
2. 세포외 플럭스 (XF) 기술을 사용하여 단일 구상체의 미토콘드리아 에너지 대사 조사
- 분석 준비 (하루 전)
- 4x 배율에서 위상 대비가 있는 반전된 광학 현미경을 사용하여 스페로이드 생존력을 확인하여 시료 간의 온전한 스페로이드 구조, 형태학 및 전반적인 균일성을 보장합니다.
- 센서 카트리지에 수분을 공급합니다.
- 교정제 ~20 mL를 원뿔형 튜브에 넣는다.
- 교정제를 함유하는 원뿔형 튜브를CO2 가 아닌 37°C 인큐베이터에 하룻밤 동안 놓는다.
- 분석 키트에서 내용물을 제거합니다.
- 유틸리티 플레이트에서 센서 카트리지를 분리하고 유틸리티 플레이트 옆의 작업대에 거꾸로 놓습니다.
- 200 μL의 멸균ddH2O를 다채널 P300 피펫을 사용하여 센서 카트리지 유틸리티 플레이트의 각 웰에 피펫한다.
- 센서 카트리지를 유틸리티 플레이트 위에 놓습니다.
- 각 웰의 수위가 센서 프로브를 잠수할 만큼 충분히 높은지 확인하십시오.
- 조립된 센서 카트리지를 CO2가 아닌 37°C 인큐베이터로 옮기고 밤새 그대로 둡니다.
참고: 이 단계는 분석 개시 12-72시간 전에 수행될 수 있다.
- 코트 스페로이드 분석 마이크로플레이트
- 무균 기술을 사용하여 30 μL/웰의 멸균 Poly-D-Lysine (0.1 mg/mL) 용액을 스페로이드 마이크로플레이트에 첨가하고 실온에서 30분 동안 배양한다.
- 스페로이드 마이크로플레이트의 각 웰에서 용액을 흡인하고, 플레이트를 반전시킨 다음, 티슈 페이퍼 위에 단단히 두드려 잔류 용액을 제거합니다.
- 200 μL/웰의 멸균 ddH2O로 플레이트를 2회 세척하십시오.
- 최종 세척 후, 마이크로플레이트를 뒤집어 티슈 페이퍼 위에 단단히 두드려 잔류 물을 제거한다.
- 플레이트를 사용하기 전에 30분 동안 공기 건조시키거나 향후 사용을 위해 4°C에서 보관하십시오.
참고: 스페로이드 분석 마이크로플레이트는 스페로이드가 마이크로플레이트의 바닥에 고정되도록 분자 접착제로 코팅되어야 합니다. 분자 접착제가 없으면 구상체가 빠져 나와 분석 결과를 방해 할 수 있습니다. 다른 분자 접착제는 또한 예비코팅 플레이트를 위한 Poly-D-리신에 대한 대안으로서 사용될 수 있다. 예비코팅된 플레이트는 4°C에서 저장될 수 있지만, 분석 개시 전에 실온으로 평형화되도록 방치되어야 한다.
- XF 분석 배지 준비
- 표 1에 상술된 바와 같이 XF RPMI 배지를 준비하고, 0.22 μm 시린지 필터로 멸균 여과한다.
- 분석 준비 (분석 전 1 시간)
- 보충된 XF RPMI 분석 배지를 37°C로 예열한다.
- 코팅된 스페로이드 분석 마이크로플레이트를 비-CO2 37°C 인큐베이터 또는 건식 욕조에서 예열한다.
- 센서 카트리지를 준비합니다.
- 교정제와 센서 카트리지가 들어있는 원뿔형 튜브를 공기 인큐베이터에서 꺼냅니다.
- 유틸리티 플레이트에서 센서 카트리지를 분리하고 작업 표면에 거꾸로 놓습니다.
- P300 다채널 피펫을 사용하여 유틸리티 플레이트에서 물을 흡인하고 버립니다.
- 교정 용액을 멸균 시약 저장소에 붓고 P300 다중 채널 피펫을 사용하여 미리 예열된 교정제 200μL/웰을 유틸리티 플레이트에 추가합니다.
- 센서 카트리지를 집어 들고 유틸리티 플레이트 위에 다시 올려 놓아 센서가 교정기에 잘 잠겨 있는지 확인합니다.
- 조립된 센서 카트리지를 포트 주입 용액을 로드할 준비가 될 때까지 다시 비CO2 37°C 인큐베이터 내로 옮깁니다.
- 구상체를 분석 배지로 세척한다.
- 스페로이드 배양 플레이트를 37°C, 5%CO2 인큐베이터에서 제거하고 스페로이드 전달 단계 전에 스페로이드를 현미경으로 관찰하여 이들의 완전성을 보장한다.
- 스페로이드 플레이트의 모든 웰을 배경 보정 웰을 포함하여 180μL/웰의 예열 분석 매질로 로드합니다.
- 7 cm 페트리 접시를 3 mL의 분석 배지로 부분적으로 채운다.
- 넓은 오리피스 피펫 팁이 로딩된 다채널 피펫을 사용하여, 피펫터를 10-50 μL의 흡인 부피로 설정하여 96-웰 배양 플레이트의 구상체를 7cm 페트리 접시로 옮깁니다.
- 시드 구상체를 예비코팅된 스페로이드 분석 마이크로플레이트에 넣는다.
- 해부 현미경 및 라이트박스 장치를 사용하여, 아래에 상술된 바와 같이 페트리 디쉬로부터 스페로이드 분석 마이크로플레이트로 구상체를 옮긴다.
- 넓은 오리피스 피펫 팁이 장착된 단일 채널 피펫터의 부피를 20μL로 설정하고 단일 구상체를 조심스럽게 흡인합니다. 팁을 스페로이드 분석 마이크로플레이트의 각 웰의 중앙에 직접 배치하고 중력이 단일 스페로이드를 각 웰의 중앙으로 용출시킬 수 있도록 합니다. 즉, 피펫 팁에서 어떤 배지도 배출하지 말고 모세관 작용이 피펫 팁에서 스페로이드를 인출하도록 합니다. 용출을 확인하기 위해, 피펫터의 내용물을 현미경으로 7cm 페트리 접시에 다시 피펫팅할 수 있다.
참고: 단일 스페로이드의 중력 용출은 일반적으로 스페로이드 크기/밀도에 따라 15-30초가 걸립니다. 이 시간 동안 피펫터는 제거해서는 안됩니다. 모든 배경 보정 웰에는 구상체가 없어야하며 분석 배지 만 포함되어야합니다. 현미경으로 각 구상체의 위치를 확인하십시오. 각 스페로이드는 이상적으로 각 웰의 중심 내에 위치해야합니다. - 일단 모든 구상체가 스페로이드 분석 마이크로플레이트로 옮겨지면, 플레이트를 분석 전에 최소 1시간 동안 37°C에서 비CO2 인큐베이터로 옮긴다.
- 넓은 오리피스 피펫 팁이 장착된 단일 채널 피펫터의 부피를 20μL로 설정하고 단일 구상체를 조심스럽게 흡인합니다. 팁을 스페로이드 분석 마이크로플레이트의 각 웰의 중앙에 직접 배치하고 중력이 단일 스페로이드를 각 웰의 중앙으로 용출시킬 수 있도록 합니다. 즉, 피펫 팁에서 어떤 배지도 배출하지 말고 모세관 작용이 피펫 팁에서 스페로이드를 인출하도록 합니다. 용출을 확인하기 위해, 피펫터의 내용물을 현미경으로 7cm 페트리 접시에 다시 피펫팅할 수 있다.
- 해부 현미경 및 라이트박스 장치를 사용하여, 아래에 상술된 바와 같이 페트리 디쉬로부터 스페로이드 분석 마이크로플레이트로 구상체를 옮긴다.
3. XF 분석을위한 센서 카트리지에 화합물 제조 및 로딩
| 주입 전략 | 화합물(포트) | XFe96 마이크로웰 개시 부피(μL) | 원하는 최종 우물 농도 | 포트 부피(μL) | 최종 XFe96 마이크로웰 주입 후 부피(μL) | 작업 재고 집중 |
| 1 | 올리고마이신 (A) | 180 | 3 ug/mL | 20 | 200 | 30 μg/mL |
| 로테논 (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 항 마이신 A (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 2 | BAM15 (A) | 180 | 5 μM | 20 | 200 | 50 μM |
| 로테논 (B) | 200 | 2 μM | 20 | 220 | 22 μM | |
| 항 마이신 A (B) | 200 | 2 μM | 20 | 220 | 22 μM |
표 2: XFe96 분석기를 사용하여 단일 3D 구상체의 미토콘드리아 에너지 대사를 프로빙하기 위한 미토콘드리아 화합물 농도.
- 완전히 보충된, 예열된 XF RPMI 분석 배지를 사용하여 표 2 에 언급된 바와 같이 각 화합물의 작업 스톡 농도를 준비한다.
- 카트리지 플레이트(유틸리티 플레이트에 연결됨)를 열 방향으로, 1-12 방향을 왼쪽에서 오른쪽으로 맞춥니다.
- 로딩 가이드를 사용하는 경우, 포트 A가 먼저 로딩되는 경우와 같이 잘 로딩된 절차에 따라 카트리지 플레이트 위에 놓으십시오(예: 포트 A가 먼저 로드되는 경우) 가이드의 왼쪽 위 모서리에 A 가 표시되는지 확인하십시오.
- 각 화합물의 작업 용액을 적절한 저장소로 옮기고 보정된 P100 다채널 피펫을 사용하여 20μL를 모든 해당 포트에 분배합니다. 각 컴파운드에 대해 나머지 포트에 대해 반복합니다.
참고: 센서 카트리지 플레이트에 포트를 사용하지 않는 경우 이러한 포트는 비워 두거나 분석 매체로 채울 수 있습니다. 특정 포트 문자의 선택만이 사용되는 경우, 그 문자에 대응하는 다른 포트가 분석 매체로 로딩되도록 보장하십시오; 그렇지 않으면 공기가 우물에 주입되어 우물의 결과를 손상시킵니다. - 포트 로딩 후, 플레이트 로딩 가이드(사용되는 경우)를 제거하고 센서 카트리지를 로딩하기 위한 분석기를 준비합니다.
참고: 포트를로드 한 직후에 분석이 실행되지 않는 경우 센서 카트리지에 뚜껑을 다시 놓고 기계에 적재 할 준비가 될 때까지 플레이트를 37 °C 공기 인큐베이터에 다시 넣으십시오.
4. 분석 설계, 주입 전략 및 데이터 수집
- 분석 실행
- 분석기의 전원을 켜고 컨트롤러(컴퓨터)에 연결합니다.
참고: 이는 웨이브 컨트롤러 소프트웨어의 위젯 패널에 있는 계측기 연결 상태를 통해 확인할 수 있습니다. - WAVE 소프트웨어에서 템플릿 페이지로 이동하여 실험에 대한 분석 템플릿 파일을 찾은 다음 두 번 클릭하여 엽니다.
참고: 분석 템플릿이 템플릿 보기에 나타나지 않으면 템플릿 파일을 공유 네트워크 드라이브 또는 USB 플래시 드라이브에서 템플릿 폴더로 가져옵니다. - 분석을 시작하려면 분석 실행 탭을 클릭합니다.
참고: 그룹 정의가 플레이트 맵 내에 올바르게 할당되면 페이지 오른쪽의 녹색 눈금으로 표시된 대로 분석을 실행할 준비가 됩니다. 이 단계에서, 임의의 추가 정보는 분석 요약 페이지 또는 비워 둔 페이지에 입력될 수 있다; 다음 단계로 진행합니다. 3D 미세조직 구상체에서 미토콘드리아 조절제의 지연된 침투로 인해(그림 2), 표 3에 설명된 측정 프로토콜 정보를 사용하십시오.
- 분석기의 전원을 켜고 컨트롤러(컴퓨터)에 연결합니다.
| 측정 기간 | 주입 수 및 포트 | 측정 세부 정보 | 기간 기간(h:min:s) |
| 교정 | 해당 사항 없음 | XF 분석기는 측정이 정확한지 확인하기 위해 항상 이 보정을 수행합니다. | 00:20:00 (평균이며 기계마다 다를 수 있음) |
| 평형 | 해당 사항 없음 | 평형화는 교정 후에 발생하며 권장됩니다. | 00:10:00 |
| 기저 | 해당 사항 없음 | 사이클 = 5 | 00:30:00 |
| 믹스 = 3:00 | |||
| 대기 = 0:00 | |||
| 측정 = 3:00 | |||
| 올리고마이신/BAM15 | 주입 1(포트 A) | 사이클 = 10 | 01:00:00 |
| 믹스 = 3:00 | |||
| 대기 = 0:00 | |||
| 측정 = 3:00 | |||
| 로테논 + 안티마이신 A | 주입 2(포트 B) | 사이클 = 10 | 01:00:00 |
| 믹스 = 3:00 | |||
| 대기 = 0:00 | |||
| 측정 = 3:00 | |||
| 총 시간: | 03:00:00 | ||
표 3: XFe96 분석기를 사용하여 단일 3D 구상체의 미토콘드리아 에너지 대사를 프로빙하기 위한 프로토콜 설정.
- 실행 시작을 클릭하여 저장 위치 대화 상자를 표시합니다.
- 결과 파일의 저장 위치를 입력하고 조립된 센서 카트리지를 분석기 측면의 도어에서 나타나는 열 트레이에 놓습니다. 열 트레이가 자동으로 열리고 화면에 교정 유틸리티 플레이트 로드 메시지가 표시될 때까지 기다립니다. 화면의 지시를 따르기 전에 i) 유틸리티 플레이트에 센서 카트리지가 제대로 맞는지 확인하고, ii) 센서 카트리지에서 뚜껑을 분리하고, iii) 유틸리티 플레이트에서 센서 카트리지의 올바른 방향을 확인하십시오.
- 화면의 명령에 따라 센서 카트리지 교정을 시작합니다.
참고: 교정을 완료하는 데 걸리는 시간은 약 10-20분입니다(37°C에서의 분석의 경우). - 센서 카트리지 교정 후 웨이브 컨트롤러의 화면 지침에 따라 스페로이드 마이크로플레이트를 분석기에 로드하여 12분 평형 단계를 시작합니다.
참고 : 흰색 눈금이있는 녹색 상자는 그 우물에 대한 '양호한'교정을 나타냅니다. 어떤 우물이 '좋은'교정을 제공하지 못하면 빨간색 상자와 흰색 십자가로 표시됩니다. 이러한 웰은 수정 분석 탭을 사용하여 분석이 완료된 후 임의의 분석으로부터 주목되고 배제되어야 한다 . - 기계가 평형 단계를 완료한 후(계측기 프로토콜에 설명된 대로) 분석기가 기준 측정값을 자동으로 획득하기 시작할 때까지 기다립니다.
- 실험을 완료하려면 WAVE 컨트롤러의 화면에 표시되는 명령을 따릅니다.
참고: 스페로이드 마이크로플레이트가 분석기에서 제거되면 센서 카트리지를 폐기하고 필요한 경우 추가 분석을 위해 스페로이드 플레이트를 따로 두십시오(예: 이중 가닥(ds) DNA 정량화). 마이크로플레이트가 추가 분석을 위해 필요하지 않은 경우, 센서 카트리지와 함께 폐기될 수 있다. - 분석 대화 상자가 나타날 때까지 기다렸다가 결과를 보거나 템플릿 보기로 돌아갑니다.
5. 데이터 정규화 및 분석 전략 - 사후 분석 정규화 및 다운스트림 분석 (선택적 단계)
- 데이터 정규화
- 스페로이드 데이터를 정규화하려면 스페로이드 크기 및 부피를 계산하고 스페로이드 분석에서 dsDNA를 정량화하기 위한 데이터 정규화 전략과 관련된 일련의 프로토콜을 참조하십시오. 이들은 보충 파일로 포함되었습니다. 보충 파일 1 및 보충 파일 2를 참조하십시오.
- 데이터 분석
- 자동화된 분석 생성기 중 하나로 데이터를 내보내려면 WAVE 컨트롤러의 데이터 내보내기 명령에 따라 분석 유형과 일치하는 내보내기 생성기를 선택합니다. 또는 데이터 파일을 내보내고 Seahorse 분석에 업로드합니다.
참고: 보고서 생성기 및 Seahorse 분석의 단점은 데이터 분석이 XF 분석이 설계된 방식으로 제한되며 측정 주기 전반에 걸쳐 평균을 취할 수 없다는 것입니다. 계측기 소프트웨어에서 데이터 세트를 수동으로 내보내면 이와 관련하여 사용자가 선호할 수 있습니다. 3D 구상체의 미토콘드리아 호흡을 평가하기위한 주사 전략이 일반적인 'MitoStress'테스트의 주입 전략과 다를 수 있다는 점을 감안할 때, 일련의 스프레드 시트 템플릿이 개발되어 3D 세포 배양에 특화된 이러한 데이터 세트를 분석하는 데 도움이되며 요청시 제공 될 것입니다. 이들 데이터 템플릿 파일들은 도 2에서 상세하고 설명된 주요 미토콘드리아 호흡 파라미터들에 대한 데이터를 제공할 것이다. - 데이터를 분석하려면 데이터를 WAVE 컨트롤러 소프트웨어에서 스프레드시트 보고서로 내보내고 분석을 위해 독립적인 스프레드시트 템플릿을 사용합니다.
- 자동화된 분석 생성기 중 하나로 데이터를 내보내려면 WAVE 컨트롤러의 데이터 내보내기 명령에 따라 분석 유형과 일치하는 내보내기 생성기를 선택합니다. 또는 데이터 파일을 내보내고 Seahorse 분석에 업로드합니다.
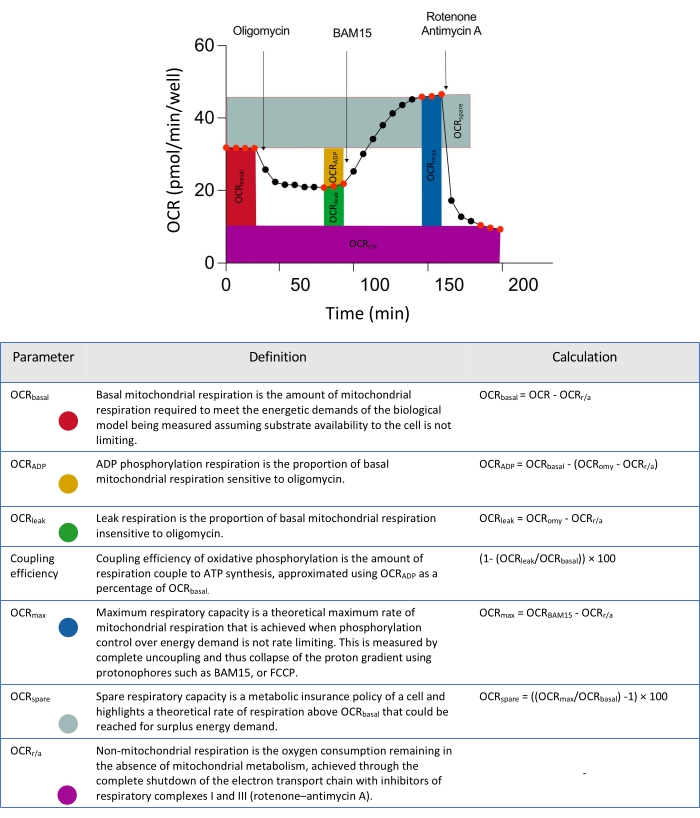
그림 2: 세포외 플럭스 데이터 분석에서 파생된 파라미터에 대한 개략적인 설명자. 약어 : OCR = 산소 소비율. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
결과
잘 형성되고 컴팩트한 구상체를 얻기 위해, 각 세포주는 시딩 밀도 및 배양 기간 동안 개별적으로 최적화되었다(도 3). A549, HepG2/C3A 및 SK-OV-3 세포주는 초기에 느슨한 응집체를 형성하였고, 이는 배양 7일 후까지 명확하게 정의된 둘레를 갖는 둥근 구상체로 진행되지 않았다. 반대로, MCF-7 세포는 3일 이내에 구상체를 형성할 수 있었다. 모든 스페로이드 모델에 대한 배양 기간 ?...
Access restricted. Please log in or start a trial to view this content.
토론
주요 결과 및 결과
이 논문은 XFe96 XF 분석기와 함께 일련의 암 유래 세포주를 사용하여 단일 3D 구상체의 미토콘드리아 에너지 대사를 조사하는 상세한 프로토콜을 제공한다. 강제 응집을 위한 세포 기피제 기술을 사용하여 A549, HepG2/C3A, MCF7 및 SK-OV-3 세포 구상체의 신속한 배양을 위한 방법이 개발되고 기술된다. 이 프로토콜은 (1) 스페로이드 배양 프로토콜의 최적화 및 기술 제조업?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 선언 할 이해 상충이 없습니다.
감사의 말
N.J.C는 Sygnature Discovery Ltd (BB/M01116X/1, 1940003)와 함께 BBSRC MIBTP CASE Award의 지원을 받았습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
참고문헌
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
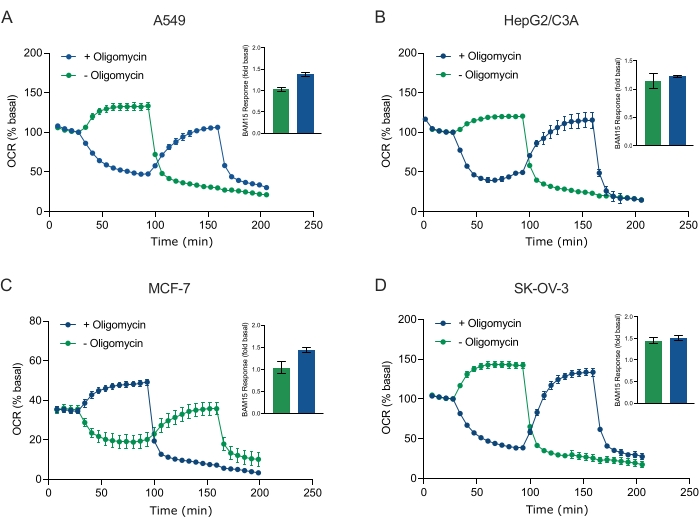
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
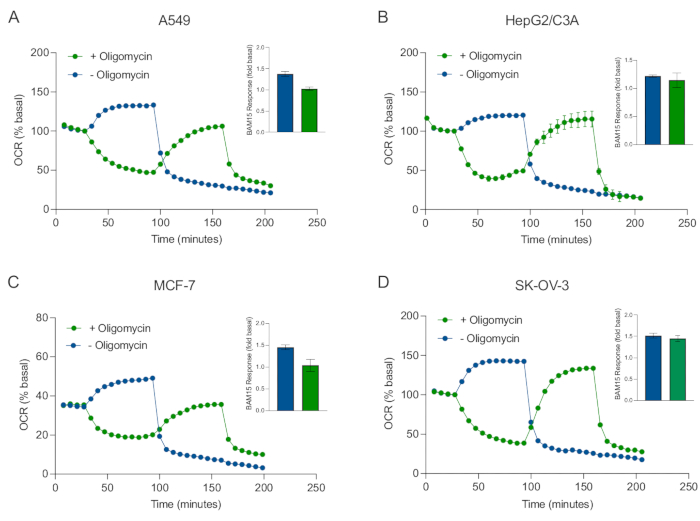
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유