JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
렌티바이러스 매개 CRISPR/Cas9 유전자 편집을 이용한 녹아웃 근육 세포주 개발
요약
이 프로토콜은 가이드 RNA의 설계부터 녹아웃 클론의 세포 클로닝 및 특성화에 이르기까지 CRISPR/Cas9를 사용하여 녹아웃 근세포를 생성하는 방법을 설명합니다.
초록
클러스터형 조절 간격 짧은 고진구 반복(CRISPR)/Cas 9의 한 가지 중요한 적용은 특히 유전자 진단 중에 확인된 질병과 관련된 새로운 유전자/단백질의 기능을 연구하기 위한 녹아웃 세포주의 개발입니다. 이러한 세포주의 개발을 위해서는 두 가지 주요 쟁점이 얽혀 있어야합니다 : 선택된 세포에 고효율로 CRISPR 도구 (Cas9 및 가이드 RNA)를 삽입하고 선택한 유전자의 특정 결실에 대한 Cas9 활성의 제한. 여기에 기술된 프로토콜은 근육 세포와 같은 형질감염되기 어려운 세포에서 CRISPR 도구의 삽입에 전념한다. 이 프로토콜은 공개적으로 이용가능한 플라스미드로 생산된 렌티바이러스의 사용에 기초하며, 이를 위해 모든 클로닝 단계가 관심있는 유전자를 표적으로 하기 위해 기술된다. Cas9 활성의 조절은 KamiCas9라고 불리는 이전에 기술된 시스템의 적응을 사용하여 수행되었으며, 여기서 Cas9를 표적화하는 가이드 RNA를 코딩하는 렌티바이러스를 사용한 세포의 형질도입은 Cas9 발현의 점진적 폐지를 허용한다. 이 프로토콜은 RYR1-녹아웃 인간 근육 세포주의 개발에 적용되었으며, 이는 근육 세포 내 칼슘 방출 및 여기 수축 결합에 관여하는이 중요한 칼슘 채널의 녹아웃을 확인하기 위해 단백질 및 기능적 수준에서 더욱 특성화되었습니다. 여기에 설명 된 절차는 근육 세포 또는 다른 트랜스펙션하기 어려운 다른 유전자에 쉽게 적용될 수 있으며 인간 세포에서 이러한 유전자를 연구하는 데 유용한 도구를 생산할 수 있습니다.
서문
유전자 시퀀싱의 진행과 특정 조직에서 알려지지 않은 기능의 유전자에서 돌연변이의 확인과 함께, 새로운 표적 유전자의 기능을 이해하고 관련 병리 생리학 적 메커니즘에 대한 개입을 확인하는 관련 세포 모델의 개발은 필수적인 도구를 구성합니다. 또한, 이들 모델은 미래의 치료 개발1,2에 매우 중요하며, 실험에서 동물의 사용을 줄이기 위한 국제 권고와 직선으로 녹아웃 동물 모델의 개발에 대한 흥미로운 대안을 구성한다. CRISPR/Cas9를 사용한 유전자 편집은 현재 사용 가능한 가장 강력한 도구 중 하나로, 많은 녹아웃/녹인 모델을 개발할 수 있었으며, CRISPR/Cas9를 사용한 표적 유전자 검증은 CRISPR/Cas93의 가장 널리 사용되는 응용 프로그램 중 하나입니다. 유전자 편집의 성공은 표적 세포 모델에 CRISPR 도구 (가이드 RNAs 및 뉴클레아제 Cas9)를 도입하는 능력에 달려 있으며, 이는 근육 세포4와 같은 많은 형질감염이 어려운 세포에서 도전이 될 수 있습니다. 이 도전은 바이러스, 일반적으로 렌티 바이러스의 사용으로 극복 할 수 있으며, 이는 많은 세포 유형을 효율적으로 형질 도입하고 전이유전자를 전달할 수있는 큰 이점을 가지고 있습니다. 그러나 그것의 주요한 단점은 숙주 세포 게놈에서 이식유전자의 통합이며, 통합 부위에 국소화된 유전자의 잠재적 변경과 이식유전자의 영구적인 발현으로 이어지며, 이는 뉴클레아제 Cas9의 경우 해로운 결과를 초래할 것이다5. Merienne과 동료6에 의해 스마트 솔루션이 제안되었는데, 이는 Cas9 유전자 자체를 표적으로 하는 가이드 RNA의 세포로의 도입으로 구성되어 Cas9 불활성화로 이어진다. 이 전략의 적응은 사용자 친화적이고 다재다능한 프로토콜로 제시되어 형질감염이 어려운 세포에서 거의 모든 유전자를 녹아웃 할 수 있습니다.
여기에 제시된 프로토콜의 목표는 불멸화된 근육 세포에서 관심있는 유전자의 불활성화를 유도하는 것이다. 그것은 불멸화 된 세포의 다른 유형에서 관심있는 모든 유전자를 녹아웃하는 데 사용할 수 있습니다. 여기에 기재된 프로토콜은 가이드 RNAs 및 이들의 클로닝을 렌티바이러스 플라스미드로 설계하고, 렌티바이러스 벡터에서 CRISPR 도구를 생산하고, 세포를 상이한 렌티바이러스로 형질도입하고, 세포를 복제하여 균질한 편집된 세포주를 생산하는 단계를 포함한다.
이 프로토콜을 사용하여, 불멸화된 인간 골격근 세포는 세포내 칼슘 방출 및 근육 수축에 관여하는 필수 칼슘 채널인 제1형 리아노딘 수용체(RyR1)의 결실과 함께 개발되었다7. 유전자의 녹아웃(KO)은 웨스턴 블롯을 사용하여 단백질 수준에서, 그리고 칼슘 이미징을 이용한 기능적 수준에서 확인되었다.
프로토콜
근육 생검은 유럽 권장 사항 및 프랑스 법률에 따라 연구 용 Tissues 은행 (Myobank, EU 네트워크 EuroBioBank, 파리, 프랑스의 파트너)으로부터 입수되었습니다. 서면 정보에 입각 한 동의는 모든 개인으로부터 얻어졌습니다. 불멸의 근모세포는 V. Mouly 박사 (프랑스 파리의 Myology Institute)에 의해 친절하게 생산되었으며, 프로토콜은 Myology Institute 윤리위원회 (MESRI, n AC-2019-3502)의 승인을 받았습니다.
1. 크리스퍼 가이드 디자인
- 결실될 유전자의 영역을 확인한다. ensembl.org 또는 genome.ucsc.edu 와 같은 게놈 브라우저 도구를 사용하여 게놈 서열을 검색하고 두 영역의 염색체 좌표를 결정하여 삭제할 영역의 양쪽에서 가이드 RNA (gRNA)를 찾습니다.
- 여기에 사용된 유전자의 경우, RYR1 유전자의 FASTA 서열과 엑손 101의 서열을 다음과 같이 구한다. 앙상블에서 인간 게놈의 최신 버전에서 RYR1 을 검색하고 첫 번째 항목을 선택한 다음 단백질 코딩 서열의 전사체를 클릭하십시오. 그런 다음 Exons를 클릭하여 유전자의 엑손 목록으로 리디렉션하십시오.
- 다운로드 시퀀스를 클릭하고 게놈 시퀀스 만 선택하여 전체 유전자의 전체 합의 서열을 다운로드하십시오. 유전자의 엑손과 인트론 목록을 아래로 스크롤하고 표적화 된 것을 선택하십시오.
- 유전자에서 상응하는 뉴클레오티드 서열을 찾는다. 결실될 엑손의 바로 상류 및 하류의 인트론의 뉴클레오티드 서열을 선택하고, 이는 gRNAs를 검색하는데 사용될 것이다.
- 여기에서 가이드 1 및 가이드 2라고 하는 두 개의 gRNA를 단계 1.1에서 식별된 영역(삭제될 영역의 상류 및 하류의 인트론)에서 Crispor.tefor.net8과 같은 온라인 도구를 사용하여 설계한다. 수백 염기쌍(bp)으로 분리된 두 개의 gRNA를 선택하고, 각 gRNA의 서열은 프로토스페이서 인접 모티프(PAM) 없이 정확히 20개 뉴클레오티드 길이이다. 오프 타겟을 제한하기 위해 사용할 수있는 최상의 가이드를 선택하십시오. 가이드 설계의 예는 그림 1 을 참조하십시오.
- 두 가이드 사이의 서열은 결실될 것으로 예상되고, 관심있는 유전자의 녹아웃을 초래할 것이며, 따라서 관심 유전자에서 필수 서열 또는 필수 엑손이 삭제되도록 두 가이드의 위치를 선택한다. 삭제 된 서열 / 엑손이 유전자의 대체 전사체에만 독점적으로 존재하지 않거나 단백질의 중요한 부분을 인코딩하므로 그 결실은 기능적 녹아웃을 초래합니다.
참고: Cas9 절단 부위가 프로토스페이서 인접 모티프(PAM)의 3 bp 상류에서 발생할 것으로 예측되지만, 더 큰 거리에서의 절단도 발생할 수 있으며, 따라서 좋은 해결책은 인트론에서의 절단과 같은 절단 부위의 정확한 국소화에 의존하지 않는다.
- 두 가이드 사이의 서열은 결실될 것으로 예상되고, 관심있는 유전자의 녹아웃을 초래할 것이며, 따라서 관심 유전자에서 필수 서열 또는 필수 엑손이 삭제되도록 두 가이드의 위치를 선택한다. 삭제 된 서열 / 엑손이 유전자의 대체 전사체에만 독점적으로 존재하지 않거나 단백질의 중요한 부분을 인코딩하므로 그 결실은 기능적 녹아웃을 초래합니다.
- 다음 서열을 가지기 위해 PAM이 없는 각 gRNA에 대한 역보체(RC) 서열을 결정한다: 가이드 1 및 가이드 1-RC, 가이드 2, 및 가이드 2-RC.
- 표 1에 제시된 프라이머를 주문하여 플라스미드의 클로닝을 수행한다. 프로토콜 전반에 걸쳐, 멸균H2O에서 10 nM의 농도로 프라이머를 사용한다.
참고: 이들 프라이머에서 gRNAs에 첨가된 서열(굵게 및 밑줄이 그어진 것)은 각각 가이드, 프로모터 및 트랜스-활성화-Crispr RNA(tracrRNA) 전후의 플라스미드의 서열에 상응하며, 프라이머와 플라스미드 사이의 양호한 중첩을 보장하기 위해 변형되어서는 안 된다.
2. 플라스미드 클로닝
참고: 이 단계에서, gRNAs는 렌티바이러스 생산을 위해 플라스미드 백본에 삽입될 것이다. 두 gRNA를 코딩하는 카세트는 먼저 중첩된 프라이머를 사용하여 연속적인 중합효소 연쇄 반응(PCR)에 의해 생산된다. 그런 다음 새로운 카세트를 렌티 바이러스 백본 플라스미드 #87919에 삽입합니다.
- 다음 플라스미드를 얻는다: 렌티바이러스 벡터에서 CRISPR 가이드 RNA를 암호화하는 플라스미드 #87919 및 렌티바이러스 벡터에서 SpCas9 서열을 암호화하는 플라스미드 #87904.
- 카세트 구성
참고: 복제 프로토콜은 다음과 같이 요약되어 있습니다. 그림 2.- 2 μL의 플라스미드 #87919, 2 μL의 primer_XmaIF, 2 μL의 primer_Guide1R, 25 μL의 중합효소 혼합물, 및 19 μL의H2O.로 PCR(A) 반응을 실행한다: 98°C에서 5분의 초기 변성, 이어서 98°C에서 30초의 30 사이클, 60°C에서 30초, 72°C에서 1분 45초, 72°C에서 7분의 최종 연신율. 단계 1.4에서 설명된 프라이머의 Tm은 60°C이다.
참고 : 프로그램 1의 신장 시간이 상당히 길어 보이지만이 신장 시간은 올바른 크기의 충분한 재료를 생산할 수 있도록 선택되었습니다. 실제로, 플라스미드에서 반복되는 서열 (RNA 가이드 후 tracrRNA의 서열은 플라스미드 #87919에서 세 번 반복됨)으로 인해 예상되는 DNA의 PCR 증폭이 어렵고 연속적인 PCR에서 더 작은 밴드가 추가로 생성됩니다. 따라서, 상이한 PCR 산물들 사이의 경쟁으로 인해, 신장 시간이 증가되거나(가장 긴 것을 선호하고 마지막에 충분한 정제된 물질을 갖기 위해), 또는 터치 다운 PCR(프로그램 2)이 긴 단편(예를 들어 단계 2.2.6에서 PCR(최종)에 대해 기술됨)에 대해 사용되었다. - PCR 산물을 트리스-보레이트-EDTA 완충액(TBE)의 1% 아가로스 겔 상에서 분리하고, 300 bp 단편을 절제하고 정제한다. 제조자의 지시에 따라 전용 키트를 사용하여 정제를 수행하고 최종 부피 20 μL에서 용출하십시오. 정제된 단편을 직접 사용하거나 단계 2.2.5를 위해 -20°C에서 보관하십시오.
- PCR 프로그램 1을 사용하여 2 μL의 플라스미드 #87919, 2 μL의 primer_Guide1F, 2 μL의 primer_Guide2R, 25 μL의 중합효소 혼합물 및 19 μL의H2O로 PCR (B) 반응을 실행한다. PCR 산물을 TBE의 1% 아가로스 겔 상에서 분리하고, 용출 완충액 20 μL에서 400 bp 단편을 절제하고 정제한다. 정제된 단편을 직접 사용하거나 단계 2.2.5를 위해 -20°C에서 저장한다.
- PCR 프로그램 1을 사용하여 2 μL의 플라스미드 #87919, 2 μL의 primer_Guide2F, 2 μL의 primer_BlpIR, 25 μL의 중합효소 혼합물 및 19 μL의H2O로 PCR (C) 반응을 실행한다. PCR 산물을 TBE 완충액의 1% 아가로스 겔 상에서 분리한다. 600 bp 단편을 20 μL의 용출 완충액에서 절제하고 정제한다. 정제된 단편을 직접 사용하거나 단계 2.2.6을 위해 -20°C에서 저장한다.
참고: 900 bp에서의 또 다른 단편은 단계 2.2.1의 참고에 설명된 바와 같이, 플라스미드 내의 반복된 영역 상의 프라이머의 혼성화로 인해 보일 수 있다. 있는 경우 이 밴드는 폐기해야 합니다. - PCR 프로그램 1을 사용하여 2 μL의 용출 PCR A (단계 2.2.2로부터), 2 μL의 용출 PCR B (단계 2.2.3부터), 2 μL의 primer_XmaIF, 2 μL의 primer_Guide2R, 25 μL의 중합효소 믹스 및 19 μL의H2O로 PCR (D) 반응을 실행한다. PCR 산물을 TBE 완충액 중의 1% 아가로스 겔 상에서 분리하는 단계; 700 bp 단편을 20 μL의 용출 완충액에서 절제하고 정제한다. 정제된 단편을 직접 사용하거나 단계 2.2.6을 위해 -20°C에서 저장한다.
참고: 다른 밴드는 반복된 영역 상의 프라이머의 혼성화로 인해 >1,000 bp, 400 bp 및 300 bp에서 보일 수 있으며 폐기되어야 한다. - 4 μL의 용출 PCR C (단계 2.2.4로부터), 4 μL의 용출 PCR D (단계 2.2.5로부터), 4 μL의 primer_XmaIF, 4 μL의 primer_BlpIR, 50 μL의 중합효소 혼합, 및 34 μL의H2O로 PCR(최종) 반응을 실행한다. 사용된 프로그램은 다음(PCR 프로그램 2)이다: 98°C에서 5분 동안 초기 변성; 6 사이클: 98°C에서 30 s, 66°C에서 30 s (사이클 당 1 °C의 혼성화 온도의 감소), 72°C에서 1 분 45; 35 사이클: 98°C에서 30초, 60°C에서 30초, 72°C에서 1분 45초, 및 72°C에서 5분의 최종 신장.
참고: 이 최종 증폭을 위한 PCR 프로그램은 각 가이드 바로 뒤에 두 개의 반복된 tracrRNA 서열을 포함하는 최종 증폭의 크기가 크기 때문에 이전 PCR 프로그램과 다른 터치 다운 PCR입니다. - PCR 산물을 TBE 완충액 중의 1% 아가로스 겔 상에서 분리하는 단계; 최종 카세트를 소비하고 정제하고, 이는 용출 완충액 20 μL에서 약 1,300 bp로 이동한다. 용출된 생성물을 정량화한다. 정제된 단편을 직접 사용하거나 단계 2.3.2.1을 위해 -20°C에서 저장한다. 이것은 렌티바이러스 플라스미드에 삽입될 최종 생성물이다.
참고: 다른 단편은 폐기되어야 하는 불완전한 PCR 단편에 상응하는 >1000 bp, 400 bp 및 300 bp에서 보일 수 있다.
- 2 μL의 플라스미드 #87919, 2 μL의 primer_XmaIF, 2 μL의 primer_Guide1R, 25 μL의 중합효소 혼합물, 및 19 μL의H2O.로 PCR(A) 반응을 실행한다: 98°C에서 5분의 초기 변성, 이어서 98°C에서 30초의 30 사이클, 60°C에서 30초, 72°C에서 1분 45초, 72°C에서 7분의 최종 연신율. 단계 1.4에서 설명된 프라이머의 Tm은 60°C이다.
- 렌티바이러스 플라스미드 골격에 gRNAs 카세트를 삽입한다.
- 제한 효소 XmaI 및 BlpI로 플라스미드 #87919의 이중 소화에 의해 플라스미드를 선형화한다.
- 15 μL의 권장 완충액, 15 μL의 플라스미드 (1μg/μL), 7.5 μL의 BlpI 효소 (10 U/μL 중), 7.5 μL의 XmaI 효소 (10 U/μL 중) 및 112.5 μL의 H2O.H2O. 37°C에서 1시간 동안 인큐베이션한 다음, 65°C에서 20분 동안 인큐베이션한다. 총량을 1% 아가로스 겔에 로딩하고, 절단하고∼10 kb 플라스미드를 적절한 키트로 정제한다. 용출은 20 μL의 완충액에서 수행되고, 용출된 생성물은 광학 밀도 측정을 사용하여 정량화된다.
참고: Sun 및 coll9에 의해 기술된 프로토콜과 같은 큰 DNA 단편에 대해 적절한 정제 프로토콜을 사용하십시오.
- 15 μL의 권장 완충액, 15 μL의 플라스미드 (1μg/μL), 7.5 μL의 BlpI 효소 (10 U/μL 중), 7.5 μL의 XmaI 효소 (10 U/μL 중) 및 112.5 μL의 H2O.H2O. 37°C에서 1시간 동안 인큐베이션한 다음, 65°C에서 20분 동안 인큐베이션한다. 총량을 1% 아가로스 겔에 로딩하고, 절단하고∼10 kb 플라스미드를 적절한 키트로 정제한다. 용출은 20 μL의 완충액에서 수행되고, 용출된 생성물은 광학 밀도 측정을 사용하여 정량화된다.
- gRNA 카세트와 플라스미드를 리게이트한다. 단계 2.2.7로부터 gRNA 카세트로 반응 믹스를 제조하고, 단계 2.3.1.1로부터 선형화된 플라스미드를 준비하고, 2 μL의 효소 및H2O를 첨가하여 최종 부피가 10 μL를 갖는다. 50°C에서 15분 동안 인큐베이션하여 p_guides이라 불리는 최종 플라스미드를 생성한다.
참고 : DNA 카세트 양은 50-100 ng 사이이어야하며 플라스미드 양은 100-200 ng 사이이어야하며 2 : 1 몰비가되어야합니다.
- 제한 효소 XmaI 및 BlpI로 플라스미드 #87919의 이중 소화에 의해 플라스미드를 선형화한다.
- 새로 준비된 플라스미드 2μL를 사용하여 Stbl3(50μL) 또는 XL10-Gold와 같은 화학적으로 유능한 E.Coli 를 변형시키고 항생제 없이 37°C에서 1시간 성장시킨 후 100μg/mL 암피실린으로 LB 한천 플레이트에 퍼뜨립니다. 37°C에서 하룻밤 동안 인큐베이션한다. 몇 개의 식민지를 선택하고 제조업체의 지침에 따라 상업용 키트를 사용하여 미니 준비를하십시오.
- Primer_XmaI을 가진 미니프렙 DNA에 대해 PCR 증폭을 수행하고 PCR 프로그램 1을 사용하여 Primer_BlpR다( 도 2 참조). PCR 산물을 TBE 완충액의 1% 아가로스 겔 상에서 분리한다. 약 1300 bp의 예상 크기로 밴드를 가진 몇 개의 콜로니 (~ 5)를 선택하십시오.
- Primer_XmaI 또는 Primer_BlpR를 사용하여 선택된 콜로니의 DNA 시퀀싱을 수행하여 gRNA 카세트의 올바른 삽입을 확인합니다.
참고: 서열 검증 콜로니 중 하나가 연구에 추가로 사용되고, 플라스미드를 p_guides이라고 한다. - 단계 2.2(카세트 구성)부터 프라이머-킬러 F 및 R, 프라이머-mCherry F 및 R을 사용하여 반복한다. 플라스미드는 p_Killer라고합니다.
3. 렌티바이러스 생산
- 제조업체의 지침에 따라 내독소가 없는 maxi-prep 키트를 사용하여 필요한 모든 플라스미드를 다량 생산하고 정제합니다. 분취량을 2 μg/μL에서 준비합니다. -20°C에서 보관하십시오.
- 세포의 제조 (제1일)
- 10% 소 태아 혈청(FBS)과 1% 페니실린/스트렙토마이신으로 보충된 둘베코 변형 이글 배지(DMEM) 고글루코스 피루베이트로 구성된 배지 16 mL에 플레이트당 1 x 106 HEK293 세포로 시딩된 145 cm의 18개의 플레이트를 준비한다. 세포를 37°C에서, 5%CO2 인큐베이터에서 3일 동안 증폭시킨다.
참고: 렌티바이러스의 생산은 일회용 보호복, 보호 캡 및 장갑을 포함한 적응된 보호 장비를 사용하여 생물안전 레벨 2 실험실에서 조심스럽게 수행되어야 합니다. 모든 실험은 필터 팁이있는 층류 후드 (BSLII 안전 캐비닛)에서 수행되어야합니다. 렌티바이러스 및 사용된 모든 플라스틱/유리 폐기물을 포함하는 모든 용액은 에탄올 70% 또는 기타 바이러스 불활성화제로 비활성화해야 합니다.
- 10% 소 태아 혈청(FBS)과 1% 페니실린/스트렙토마이신으로 보충된 둘베코 변형 이글 배지(DMEM) 고글루코스 피루베이트로 구성된 배지 16 mL에 플레이트당 1 x 106 HEK293 세포로 시딩된 145 cm의 18개의 플레이트를 준비한다. 세포를 37°C에서, 5%CO2 인큐베이터에서 3일 동안 증폭시킨다.
- 세포의 형질감염 (4일째)
- 세포의 합류를 확인하고 60 % -65 % 합류에 도달했는지 확인하십시오.
- 각 플레이트에 대해 함유하는 형질감염 용액을 준비한다: 20.8 μg의 목적하는 플라스미드 (p-guides, 또는 p-Killer, 또는 pCas9 #87904), 엔벨로프를 코딩하는 플라스미드 4.8 μg (VSV-G, #8454), 렌티바이러스 포장을 위한 플라스미드 psPAX2 (#12260) 20.8 μg, 136 μL의 인산칼슘, 136 μL의H2O로 조정하고, 1,000 μL의 최종 부피로 조정한다. 이 용액을 2x HEPES-완충 식염수 (HBS) 1 mL에 적가하고 교반 하에 첨가한다.
참고 : 시약의 최적 준비를 보장하기 위해 모든 플레이트에 대한 혼합을 동시에 준비하지 마십시오. 동시에 6 개의 플레이트에 대한 믹스를 준비하십시오 (예 : 18 개의 플레이트에 대해, 6 개의 플레이트에 대해 세 번 믹스를 준비하십시오. - 실온(RT)에서 적어도 10분 동안 인큐베이션하고, 용액 2 mL를 세포에 적가한다. 형질감염 시약을 플레이트를 뒤로, 앞으로, 위, 아래로 부드럽게 교반하면서 균질화하고, 37°C, 5%CO2 에서 적어도 5시간 동안 인큐베이션한다.
참고: 이 시점부터 렌티바이러스 생산이 끝날 때까지 두 번째 장갑, 보호 슬리브 및 일회용 플래스트론을 포함한 추가 보호 장비를 착용하십시오. - 형질감염 후 다섯 시간 후, 플레이트로부터 배지를 제거하고 PBS로 헹구어 형질감염 시약을 제거한다. 신선한 배지 12 mL를 첨가하고, 37°C, 5%CO2에서 48시간 인큐베이션한다.
- 바이러스 입자의 수집 (Day 6)
- 모든 접시에서 배지를 수집하고 풀링하십시오. 4°C에서 5분 동안 800 x g 에서 원심분리하여, 세포 파편을 펠릿화하였다. 0.45 μm 필터를 사용하여 상청액을 여과한다(다중 필터 필요).
- 스윙 버킷 로터에서 4°C에서 2시간 동안 68,300 x g 에서 원심분리한다. 상층액을 제거하고 튜브를 5-10 분 동안 종이의 안전 캐비닛 아래에서 거꾸로 뒤집어 가능한 한 많은 액체를 제거한 다음 펠렛 당 100 μL의 HEK 증식 배지를 첨가하십시오. 4°C에서 적어도 2시간 후, 상하로 피펫팅하여 펠렛을 재현탁시킨다. 재현탁 된 모든 펠렛을 풀링하십시오.
참고: 펠릿은 분취 전에 4°C에서 하룻밤 동안 배지에 방치할 수 있다. - 10 μL 또는 25 μL 샘플 크기의 분취량 렌티 바이러스 (사용에 따라 다름) 및 액체 질소로 스냅 동결. -80°C에서 보관한다. 해동된 분취량을 얼리지 마십시오.
- LV-가이드, LV-킬러 및 LV-Cas9를 생산하기 위해 관심있는 다른 플라스미드와 함께 3.2 내지 3.4단계를 반복한다.
참고: 대안으로 렌티바이러스는 회사 또는 바이러스 시설에서 구입할 수 있습니다.
4. 렌티바이러스 적정
참고: 바이러스 적정은 HEK293 세포에서 수행됩니다. 적정은 관심있는 세포에 대해 세포 당 정확한 수의 렌티바이러스 (렌티바이러스의 배치가 무엇이든간에)를 후속 단계에서 통합하는 것이 중요합니다. 세포를 효율적으로 형질도입하는 바이러스 입자의 수를 감염의 다중성(MOI)이라고 한다: MOI 10은 따라서 세포당 10개의 바이러스 입자의 도입에 상응한다. 동결 / 해동 사이클이 렌티 바이러스의 생존 가능성에 영향을 미치기 때문에 적정은 냉동 렌티 바이러스 분취량으로 수행되며 각 후속 실험은 동일한 풀의 새로운 분취량으로 수행됩니다. 하나의 적정 방법이 여기에 기술되어 있지만, 다른 방법이 사용될 수 있다.
- 1일째에, 바닥에 유리 커버슬립이 있는5개의 35mm 플레이트와 커버슬립이 없는 두 개의 35mm 플레이트에 플레이트당 1 x 10 5개의 세포를 시드하십시오.
- 2일째에, 커버슬립이 없는 두 플레이트로부터, 트립신화 후 세포의 수를 수집 및 계수하고, 플레이트 당 평균 세포량(N)을 결정한다.
- 증식 배지에서 1/10으로 희석된 렌티바이러스 100 μL를 준비한다. 5개의 배양된 플레이트를 1 내지 50 μL의 희석된 바이러스의 상이한 부피의 희석된 바이러스로 형질감염시킨다. 이 프로토콜을 위해, 다음 부피를 사용하여 5개의 플레이트를 형질도입한다: 1 μL, 5 μL, 10 μL, 20 μL, 50 μL. 37°C에서, 5%CO2에서 48시간 동안 인큐베이션한다.
- 4일째에, 커버슬립을 4% 파라포름알데히드에서 20 내지 30분 동안 RT에서 인큐베이션함으로써 세포를 고정시킨다. LV-Cas9의 경우, 세포를 RT에서 10분 동안 포스페이트 버퍼 염수 (PBS) 중 0.1% 트리톤 X100으로 투과시키고, PBS-0.1% 트리톤 X100-2% 염소 혈청-0.5% 소 혈청 알부민 (BSA)에 포화시켜 RT에서 20분 동안 고정시키고, RT에서 45분 동안 1차 항체 항-V5 (희석 1/400)로 라벨링하고, 이어서 형광 2차 항체와 함께 RT에서 30분 동안 인큐베이션하였다. RT에서 10분 동안 Hoescht(PBS에서 10μg/mL)로 핵을 라벨링합니다.
- 커버슬립을 슬라이드에 장착하고 20x 대물렌즈가 장착된 형광현미경을 사용하여 관찰합니다. LV-가이드 및 LV-Killer의 경우, 고정 후 핵 라벨링으로 직접 진행하고 커버슬립을 장착합니다. 각 커버슬립에 대해, 시야에서 핵의 총 수(총 세포 수)와 라벨링된 세포의 수(V5 또는 mCherry로 표시)를 계수하고, 각 커버슬립에 대해 표지된 세포의 비율을 결정한다.
- 형질도입된 세포의 비율이 10% 이상이고 50%를 초과하지 않는 커버슬립을 선택하십시오. 이러한 커버슬립에 대한 형질도입 효율(X)을 결정하고, 이러한 형질도입 효율을 얻기 위해 사용된 희석된 바이러스의 부피(V, μL)를 주목한다.
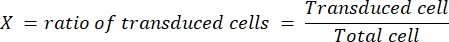
- 다음 공식에 따라 바이러스의 역가 (감염성 입자, ip/mL로 표시됨)를 확인합니다.

희석 인자는 단계 4.3에서 수행된 렌티바이러스의 희석이다. 우리는 HEK 세포에서 결정된 LV-Cas9에 대해 1 x 109 ip/mL의 최종 역가를, LV-가이드에 대해 1 x10 10 ip/mL의 최종 역가를 일상적으로 얻었다.
5. 근원세포 형질도입
참고: 불멸화된 근아세포는 이전에 생산된 세 개의 렌티바이러스로 연속적으로 형질도입된다. 이들은 20% FBS, 2% 페니실린/스트렙토마이신, 2% 울트로서 G로 보충된 Ham's F10으로 구성된 증식 배지에서 50% 미만의 밀도로 유지되고, 37°C, 5%CO2에서 배양되었다.
- 다음 공식에 따라 LV-Cas9에 대한 MOI 10 및 LV-킬러에 대한 LV 가이드 및 MOI 20으로 선택한 세포 수를 치료하는 데 필요한 렌티바이러스의 부피를 결정하십시오.
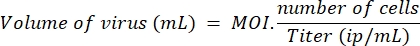
참고: 병렬 실험에서, 형질도입 효율은 대조군 렌티바이러스(lenti-GFP)를 사용하여 근모세포와 HEK에서 비교되었으며, 우리는 HEK 세포에 비해 근원세포를 효율적으로 형질도입하기 위해 다섯 배 더 많은 렌티바이러스가 필요하다는 것을 결정하였다. 따라서, HEK에서 측정된 MOI 10은 근모세포 당 두 개의 바이러스 입자에 상응한다. 여기에 사용된 MOI는 HEK 세포에 대해 계산된다. - 1일째에, 96-웰 플레이트를 웰 당 100 μL의 증식 배지 중 10,000개의 세포로 시드한다. 2일째에, 단계 5.1에서 계산된 LV-가이드 및 LV-Cas9의 적절한 부피를 추가하여 안전 캐비닛 아래에 세포를 형질도입한다. 세포를 7일째까지 인큐베이터로 복귀시킨다.
참고: 특히 LV-Cas9의 경우 25보다 높은 MOI를 사용하면 고농도의 렌티바이러스에서 높은 세포 사멸로 인해 편집된 세포의 수가 개선되지 않을 수 있습니다. - 7 일째에 트립신화를 수행하고 세포를 계산하십시오. 세포를 새로운 플레이트에서 40% 내지 50%의 컨플루언시로 시드하고 세포를 인큐베이터로 복귀시킨다. 5시간 후, 세포를 20의 MOI에서 LV-킬러로 형질도입한다(단계 5.1에서 계산된 부피). 트랜스펙션 후 5 내지 10일 동안, 적어도 두 개의 계대 동안 세포를 증폭시키고, 항상 <50%의 낮은 컨플루언시로 유지한다. 세포는 다음 단계인 세포 클로닝을 위해 준비되며, 그들의 성장이 정상으로 복귀될 때(분열 시간에 의해 추정됨).
참고: 형질도입 후 증식이 약간 느려질 수 있습니다. 정상 세포 성장은 세포의 분열 시간의 추정에 의해 이러한 실험 절차 전에 결정될 수 있다.
6. 세포 복제
참고: 근원세포 형질도입이 어렵고 100% 효율에 도달하지 못하기 때문에, 렌티바이러스를 사용하는 경우에도 완전히 교정된 세포주를 얻기 위해서는 세포 복제가 필요합니다. 이것은 불멸화 된 세포 또는 몇 주 / 개월 동안 배양되고 증폭 될 수있는 세포에서만 가능합니다.
- 트립신화하고 세포를 계산하십시오. 세포를 10 cells/mL의 증식 배지에서 희석하고 100 μL/웰의 배지가 들어있는 96-웰 플레이트에 1 세포/웰로 세포를 시드한다.
참고 : 시드 될 플레이트의 수는 예상되는 유전자 편집의 확률에 따라 다르며 2 ~ 10 개의 플레이트가 일상적으로 사용됩니다. - 세포의 성장을 모니터링하고, 적어도 35 mm 플레이트에 도달할 때까지 더 큰 플레이트에서 각 웰을 점진적으로 증폭시키고, 근아세포의 합류도를 50% 이하로 유지한다. 이 단계는 사용 된 세포와 우물에서 한 번 분리되면 성장할 수있는 능력에 따라 2-6 주간 지속될 수 있습니다.
7. 복제 선택
참고: 이 단계는 성장하는 클론 중 어느 것이 적절하게 수정되었는지 확인하기 위해 수행됩니다.
- 추정적으로 변형된 서열을 둘러싸는 영역을 증폭시키기 위해 첫 번째 가이드(Primer_BeforeGuide1F) 앞에 위치한 프라이머와 두 번째 가이드(Primer_AfterGuide2R) 뒤에 위치한 또 다른 프라이머로 구성된 프라이머 세트를 설계한다. 여기에 사용된 프라이머에 대해서는 표 1 을 참조한다.
- 각 클론으로부터 세포를 수집하고, 향후 증폭을 위해 적어도 300,000개의 세포를 아끼고, 나머지 세포에 대한 임의의 표준 프로토콜을 사용하여 게놈 DNA를 추출한다.
- 많은 수의 클론이 성장하고, 편집되지 않은 클론을 버리기 위해, 동일한 튜브에 있는 다섯 개의 클론으로부터 세포를 풀링하여 이 풀에서 DNA를 추출하고 PCR로 테스트함으로써 신속한 테스트를 수행한다. 필요한 만큼 복제본을 반복합니다. 그런 다음, 개별 분석을 수행하기 위해 편집된 셀을 포함하는 풀을 추가로 분리한다.
- 다음과 같이 PCR에 의한 편집을 제어한다. 제조사의 지시 및 프라이머 파라미터에 따라 1 μL의 Primer_BeforeGuide1F, 1 μL의 Primer_AfterGuide2R, 12.5 μL의 중합효소 혼합물, 3 μL의 게놈 DNA 및 7.5 μL의H2O. 증폭으로 PCR 반응을 준비한다. 1% 아가로스 겔에서 실행하여 편집된 클론을 확인합니다.
- 다음과 같이 시퀀싱을 통해 편집을 제어합니다. 선택한 클론의 생어 시퀀싱을 수행하여 결실을 확인하고 각 클론에서 편집이 어떻게 수행되었는지 확인합니다. 하나 이상의 편집된 클론을 유지하여 표적화된 유전자만이 변형되었는지, 관찰된 생리학적 효과를 담당하는지 확인하고, 후속 실험에서 대조군 클론(CTRL)으로 사용될 편집되지 않은 클론을 유지한다.
- 선택한 클론을 확장합니다. 일단 각 클론의 컨플루언시가 약 50%에 도달하면, 세포를 트립신화하고 세포를 더 큰 접시에 플레이트화하고, 생화학적 및 기능적 특성화를 수행하기에 충분한 세포가 생성될 때까지(보통 클론 당 1 x 106개 이상), 장래의 사용을 위해 각 클론의 냉동 분취량을 저장한다.
8. 편집된 클론의 특성화
참고: 일단 몇 개의 클론이 DNA 시퀀싱에 의해 선택되고 확인되면, 표적 유전자의 결실은 웨스턴 블롯을 사용하여 단백질 수준에서, 그리고 기능적 세포 분석이 가능한 경우 기능적 수준에서 확인할 수 있습니다. RYR1-KO의 경우, RyR1이 칼슘 채널이기 때문에, 배양된 세포 상에서 칼슘 이미징을 사용하여 기능적 특성화가 수행되었다.
- 편집된 클론에서의 단백질 발현
참고: RyR1은 분화된 근튜브(10)에서만 발현된다. 이의 발현은 웨스턴 블롯을 사용하여 근관에서 평가되었으며, RyR1의 단백질 수준에서의 결실 및 Cas9 단백질의 결실을 확인했다.- 플레이트 200,000 세포를 라미닌으로 코팅된 35 mm 플레이트 내의 약 1.76cm2 의 표면 상의 증식 배지 (상기 기술, 단계 5) (칼슘이 있는 PBS 중 10 mg/mL에서의 라미닌 200 μL 방울에 상응하는 표면). 일단 세포가 37°C, 5% CO2에서 2-3시간 동안 배양된 후 플레이트에 달라붙으면, 배양 배지를 DMEM 저글루코스 + 10% 말 혈청 + 1% 페니실린/스트렙토마이신으로 구성된 분화 배지로 옮기고, 세포를 6일 동안 배양기로 복귀시킨다.
- 분화 6일 후, 세포를 수집하고 프로테아제 억제제로 보충된 200 μL의 RIPA로 용해시킨다. Folin Lowry 방법11을 사용하여 단백질 농도를 결정한다.
- 15 μg의 단백질을 로딩하고, Laemmli 변성 완충액 중의 RT에서 30분 동안 변성시킨 후, 5%-15% 구배 아크릴아미드 겔 상에서 처리하였다. 전기영동 분리 후, 단백질을 임모빌론 P 상에서 4시간 동안 0.8 V에서11시간 동안 전달한다.
- 멤브레인을 0.1% 트윈 20 및 5% 탈지 분유를 함유하는 PBS 중의 RT에서 30분 동안 포화시킨 후, RT에서 2시간 동안 또는 4°C에서 하룻밤 동안 동일한 완충액에 희석된 1차 항체로 멤브레인을 인큐베이션하고, 멤브레인을 PBS-0.1% 트윈 20으로 5분 동안 5x 세척하고, RT에서 1시간 동안 이차 항체와 함께 멤브레인을 인큐베이션한다. 사용된 1차 항체는 하기: Cas9를 검출하기 위한 V5-태그에 대한 항체 (희석: 1/5000), 로딩 대조군으로서 항-GAPDH (희석: 1/1000), 항-RyR1 항체 12,13 (희석: 1/10.000), DHPR의 알파 1 서브유닛에 대한 항체 (희석: 1/1000) 및 미오신 중쇄 MF20에 대한 항체 (희석: 1/1000).
- 멤브레인 5x를 PBS-0.1% 트윈 20으로 5분 동안 세척하고, 과량의 액체를 건조시키고, 화학발광 기질을 첨가한다. 기질 제공자가 권장하는 대로 진행하여 화학발광 신호를 검출한다.
- 편집된 클론의 기능적 특성화
참고: RyR1의 기능은 CTRL 또는 KO 클론14로부터 생성된 분화된 근관에서 칼슘 이미징을 사용하여 평가되었다.- 플레이트 50,000 세포를 35 mm 디쉬의 중앙에0.2 cm2 표면 상에 라미닌으로 코팅하고(표면은 50 μL 라미닌 방울로 덮고, 칼슘이 있는 PBS 중 10 mg/mL에서) 단계 8.1.1에 기재된 바와 같이 6일 동안 분화를 유도하였다. 생물학적 삼중항을 갖기 위해 각 자극에 대해 세 개의 플레이트를 준비하십시오.
- 50 μL의 myotube를 플루오4-다이렉트로 로딩하고, 분화 배지에서 1:1로 희석하고, 37°C에서 30분 동안 인큐베이션하였다. 1mg/mL의 포도당이 보충된 KREBS 완충액으로 세포를 두 번 헹구십시오.
- 10x 목표를 사용하여 반전 형광 현미경 또는 공초점 현미경으로 형광 변화를 측정합니다. 현미경 무대에 플레이트를 설치하고 90 초 동안 초당 1 프레임에서 수집을 시작하십시오.
- 남아있는 KREBS를 제거하고, RyR1 직접 자극을 위해 막 탈분극 (140 mM 최종 농도) 또는 4 CmC (500 μM 최종 농도) 2 mL의 KCl을 첨가하여 프레임 25에서 세포를 자극한다. 기록된 현장에 최소 10개의 근관이 존재하는지 확인하십시오.
- 전용 소프트웨어를 사용하여 각 myotube의 형광 변화를 정량화하십시오. 접시 당 적어도 10 개의 myotubes (이상적으로는 접시 당 20-30 개의 myotubes)를 분석하기 위해 선택하고, 각 myotube의 장축에 선 (또는 관심 영역 (ROI))을 그리고 모든 프레임에 대해이 선을 따라 형광 F를 수집합니다.
- 프레임 1 내지 24에 대응하는 초기 형광 값, F0을 결정한다. 형광 변이(F-F0)/F0을 0~90초의 시간 함수로 플로팅합니다. 실험을 세 번 반복하여 세 개의 서로 다른 배양물로부터 적어도 90개의 미오튜브로부터 형광 변이를 얻는다. 90개의 근관에 대한 모든 결과를 풀링하고 각 시간 프레임에서 (F-F0)/F0의 평균 ± SEM을 계산합니다. 각 자극 및 각 클론에 대한 칼슘 방출의 피크 진폭을 정량화한다.
결과
이 프로토콜은 건강한 피험자15 (소위 HM 세포, 인간 근모세포의 경우)로부터의 불멸화 근모세포에 적용되었으며, 여기서 RyR1은 RyR1 단백질을 코딩하는 RYR1 유전자를 녹아웃시키기 위해 이전에 특성화16이었다. 가이드 RNA의 설계는 유전자의 엑손 101 및 인트론 101의 일부를 포괄하는 서열을 삭제하도록 만들어졌다. 엑손 101의 일부의 결실은 판독 프레임의 ?...
토론
병리학에 관여하는 알려지지 않은 기능의 유전자를 특성화하는 길의 주요 단계는 이러한 유전자의 기능을 연구하기위한 관련 세포 모델의 개발입니다. CRISPR / Cas9를 사용한 유전자 편집의 사용은 기하 급수적으로 성장하는 연구 분야이며, 여기에 제시된 녹아웃 모델의 개발은 가장 널리 사용되는 응용 분야 중 하나입니다. 이러한 맥락에서, 우리는 여기서 관심있는 임의의 유전자에서 인간 세포?...
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
이 작품은 협회 Française contre les myopathies (AFM-Téléthon)와 Auvergne-Rhône Alpes Région (AURA)의 보조금으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Anti-CACNA1S antibody | Sigma-Aldrich | HPA048892 | Primary antibody |
| Blp I | NE BioLabs | R0585S | Restriction enzyme |
| CalPhos Mammalian Transfection Kit | Takara | 631312 | Transfection kit |
| Easy blot anti Mouse IgG | GeneTex | GTX221667-01 | HRP secondary antibody |
| Easy blot anti Rabbit IgG | GeneTex | GTX221666 | HRP secondary antibody |
| Fluo-4 direct | Molecular Probes | F10472 | Calcium imaging |
| GAPDH(14C10) Rabbit mAb | Cell Signaling Technology | #2118 | Primary antibody |
| HindIII | Fermentas | ER0501 | Restriction enzyme |
| InFusion HD Precision Plus | Takara | 638920 | Ligation kit |
| MasterMix Phusion High Fidelity with GC | ThermoFisher Scientific | F532L | Mix for PCR reaction with High fidelity Taq polymerase and dNTPs |
| Myosin Heavy Chain antibody | DHSB | MF20 | Primary antibody |
| NucleoBond Xtra Maxi EF | Macherey-Nagel | REF 740424 | Maxipreparation kit for purification of plasmids |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609 | DNA purification |
| NucleoSpin Tissue | Macherey-Nagel | 740952 | Kit for DNA extraction from cell |
| One Shot Stbl3 Chemically Competent E. coli | ThermoFisher Scientific | C737303 | Chemically competent cells |
| Plasmid #87904 | Addgene | 87904 | Lentiviral plasmid encoding the SpCas9 (for LV-Cas9) |
| Plasmid #87919 | Addgene | 87919 | Lentiviral backbone for insertion of cassette with guides (for LV-guide-target) |
| Plasmid #12260 | Addgene | 12260 | Lentiviral plasmid encoding lentiviral packaging GAG POL |
| Plasmid #8454 | Addgene | 8454 | Lentiviral plasmid encoding envelope protein for producing lentiviral and MuLV retroviral particles |
| V5 Tag Monoclonal Antibody | Invitrogene | R96025 | Primary antibody |
| XL10-Gold Ultracompetent Cells | Agilent | 200317 | Chemically competent cells |
| Xma I | NE BioLabs | R0180S | Restriction enzyme |
참고문헌
- Claussnitzer, M., Susztak, K. Gaining insight into metabolic diseases from human genetic discoveries. Trends in Genetics. 37 (12), 1081-1094 (2021).
- Fuster-García, C., García-Bohórquez, B., Rodríguez-Muñoz, A., Millán, J. M., García-García, G. Application of CRISPR tools for variant interpretation and disease modeling in inherited retinal dystrophies. Genes. 11 (5), 473 (2020).
- Modell, A. E., Lim, D., Nguyen, T. M., Sreekanth, V., Choudhary, A. CRISPR-based therapeutics: current challenges and future applications. Trends in Pharmacological Sciences. 43 (2), 151-161 (2022).
- Olson, E. N. Toward the correction of muscular dystrophy by gene editing. Proceedings of the National Academy of Sciences of the United States of America. 118 (22), (2021).
- Wu, X., Kriz, A. J., Sharp, P. A. Target specificity of the CRISPR-Cas9 system. Quantitative Biology. 2 (2), 59-70 (2014).
- Merienne, N., et al. The self-inactivating KamiCas9 system for the editing of CNS disease genes. Cell Reports. 20 (12), 2980-2991 (2017).
- Marty, I., Fauré, J. Excitation-contraction coupling alterations in myopathies. Journal of Neuromuscular Diseases. 3 (4), 443-453 (2016).
- Concordet, J. P., Haeussler, M. CRISPOR: intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Research. 46, 242-245 (2018).
- Sun, Y., Sriramajayam, K., Luo, D., Liao, D. J. A quick, cost-free method of purification of DNA fragments from agarose gel. Journal of Cancer. 3, 93-95 (2012).
- Flucher, B. E., Conti, A., Takeshima, H., Sorrentino, V. Type 3 and type 1 ryanodine receptors are localized in triads of the same mammalian skeletal muscle fibers. The Journal of Cell Biology. 146 (3), 621-630 (1999).
- Hess, H. H., Lees, M. B., Derr, J. E. A linear Lowry--Folin assay for both water-soluble and sodium dodecyl sulfate-solubilized proteins. Analytical Biochemistry. 85 (1), 295-300 (1978).
- Garibaldi, M., et al. Dusty core disease' (DuCD): expanding morphological spectrum of RYR1 recessive myopathies. Acta Neuropathologica Communications. 7 (1), 3 (2019).
- Marty, I., et al. Biochemical evidence for a complex involving Dihydropyridine receptor and Ryanodine receptor in triad junctions of skeletal muscle. Proceedings of the National Academy of Sciences of the United States of America. 91 (6), 2270-2274 (1994).
- Oddoux, S., et al. Triadin deletion induces impaired skeletal muscle function. Journal of Biological Chemistry. 284 (50), 34918-34929 (2009).
- Mamchaoui, K., et al. Immortalized pathological human myoblasts: towards a universal tool for the study of neuromuscular disorders. Skeletal Muscle. 1, 34 (2011).
- Cacheux, M., et al. Functional characterization of a central core disease RyR1 mutation (p.Y4864H) associated with quantitative defect in RyR1 protein. Journal of Neuromuscular Diseases. 2 (4), 421-432 (2015).
- Luis, A. The old and the new: Prospects for non-integrating lentiviral vector technology. Viruses. 12 (10), 1103 (2020).
- Leenay, R. T., Beisel, C. L. Deciphering, communicating, and engineering the CRISPR PAM. Journal of Molecular Biology. 429 (2), 177-191 (2017).
- Salmon, P., Trono, D. Production and titration of lentiviral vectors. Current Protocols in Neurosciences. , (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유