Method Article
노인의 연료 활용 및 일주기 리듬을 평가하기 위한 최소 침습 방법 사용
요약
말초 혈액 단핵 세포를 활용하여 노화 환자에 대한 연료 활용 및 일주기 리듬의 시너지 효과를 평가하기 위해 새롭고 최소 침습적인 프로토콜이 제시됩니다.
초록
노화는 신체 장애 및 만성 질환의 위험에 시너지 효과적이고 독립적으로 기여하는 여러 생리학적 변화와 관련이 있습니다. 노화와 관련된 신체 장애의 원인은 복잡하고 복합적이지만, 미토콘드리아 기능의 저하는 많은 노인의 기능 저하의 진행과 일치하는 것으로 보입니다. 노화에 따라 미토콘드리아 기능이 감소하는 이유는 여전히 파악하기 어렵지만, 새로운 과학에 따르면 연료 대사와 일주기 리듬이 모두 미토콘드리아 기능에 영향을 미칠 수 있습니다.
최근 연구에 따르면 일주기 리듬은 노화와 함께 방해를 받게 되며, 일주기 리듬이 흐트러지면 미토콘드리아 기능에 영향을 미치고 많은 노화 관련 만성 질환과 겹치는 병리학적 결과를 초래합니다. 미토콘드리아 기능을 직접 평가하기 위한 현재의 정량적 방법은 침습적이며 일반적으로 근육 생검이 필요하기 때문에 잠재적인 통증과 위험의 인지된 수준을 감안할 때 참가자 모집 및 연구 순수에 어려움을 초래할 수 있습니다. 따라서 세포 수준에서 미토콘드리아 기능의 변화와 노인의 일주기 패턴을 평가하기 위한 혁신적이고 상대적으로 비침습적인 프로토콜이 조정되었습니다. 특히, 실시간 대사 플럭스 분석기는 차등 기질 가용성 하에서 백혈구의 미토콘드리아 생체 에너지 기능을 평가하는 데 사용됩니다.
미토콘드리아 생체 에너지학 및 일주기 리듬 결과와 교차 상관관계를 맺기 위해 백혈구에서 일주기 시계 유전자의 발현도 분석됩니다. 이러한 혁신적인 방법론적 접근 방식은 노인의 미토콘드리아 기질 선호도와 일주기 리듬을 연구하기 위한 최소 침습적 방법을 제공함으로써 향후 임상 시험에 도움이 될 것으로 믿어집니다.
서문
지난 세기의 발전으로 인해 기대 수명이 증가하고 고령화 인구가 증가했습니다. 미래를 내다보면 미국에서 65세 이상 성인의 비율은 2020년에서 2050년까지 5% 증가할 것으로 예상됩니다1. 이러한 기대 수명의 증가는 건강 수명(독립적인 기능과 관련된 수명)의 증가를 의미하지는 않습니다. 현실은 노화가 세포 대사와 생리에 영향을 미치는 수많은 생물학적 변화를 동반하여 인지 및 신체 기능의 점진적인 저하를 초래한다는 것입니다 2,3. 인간의 기대 수명이 계속 증가함에 따라4세가 되어도 기능적 능력과 독립성을 보존해야 할 필요성이 커지고 있습니다.
나이가 들면서 신체 기능과 독립성이 저하되는 것은 여러 요인에 의해 발생한다는 것은 오래 전부터 알려져 왔지만, 만성 질환 및 급성 자극 사건의 발병과 관련이 있는 경우가 많다5. 반대로, 이러한 신체 기능 및 근육 특성의 감소는 단일 질병과 명확한 연관성이 없는 연령에 따른 장애 발병과 관련이 있는 것으로 나타났습니다6. 만성 질환 및 신체 장애의 정확한 원인을 알기 어렵기 때문에 미토콘드리아 기능의 손상은 만성 질환의 발병 및 진행, 고령 성인의 신체 기능 상실과 일치하는 것으로 생각되어 왔습니다 7,8.
미토콘드리아는 많은 세포 과정에 필요한 대부분의 아데노신 삼인산(ATP)을 제공합니다9. 고도의 산화성 조직은 적절한 에너지 생산을 위해 미토콘드리아에 의존합니다. 노화, 산화 능력 및 미토콘드리아 ATP 합성이 감소합니다. 이러한 감소는 부분적으로 미토콘드리아 DNA(mtDNA)의 산화적 손상에 기인하며, 이로 인해 mtDNA 돌연변이 및 결실이 점진적으로 축적됩니다10. mtDNA 돌연변이 및 결실의 축적은 기능적 전자 전달 사슬 단백질의 형성을 감소시켜 세포의 ATP 생성 능력을 감소시킵니다. 노화와 관련된 미토콘드리아 기능의 저하는 심장 및 골격근과 같은 고도의 산화 조직에서 가장 두드러진다11. 연구에 따르면 나이가 많은 쥐 샘플의 비복근 미토콘드리아는 젊은 샘플에 비해 ATP 생성 및 함량이 약 50% 감소하는 것으로 나타났습니다12. 또한, 인간 골격근에서 미토콘드리아 ATP 생산 능력은 수명이 10년마다 약 8%씩 감소하는 것으로 나타났습니다13. 이러한 발견은 노화와 관련된 미토콘드리아 기능의 감소가 유기체의 에너지 생산 감소에 기여할 수 있음을 시사합니다.
미토콘드리아 활성의 핵심 조절인자는 과산화소체 증식제 활성화 수용체 γ(PPARγ) 공동 활성제-1(PGC-1α)인 것으로 생각됩니다14. PGC-1α 활성의 저하 또는 풍부도의 감소는 미토콘드리아 산화 활성의 감소로 이어지며, 결과적으로 에너지 생산의 저하로 이어집니다. 또한, 미토콘드리아 질의 저하가 골격근의 질에 영향을 미칠 수 있으며, 이로 인해 근감소증, 다이나페니아의 발병 또는 악화가 발생할 수 있으며, 기능적 능력 저하가 발생할 수 있다15,16. 노화와 관련된 미토콘드리아 기능과 골격근의 질적 저하에 대한 증거는 미토콘드리아 손상과 기능 저하의 발병 기전 사이의 연관성을 시사한다1, 7. 최근에는 기능적 지역사회 거주 노인에서 이러한 현상이 확인되었으며, 이는 골격근 미토콘드리아 대사의 감소가 이 인구 집단의 이동성 감소를 예측한다는 것을 보여줍니다18. 나이가 들면서 미토콘드리아 기능이 감소하는 정확한 메커니즘은 불분명하지만, 최근의 증거에 따르면 생체 시계와 미토콘드리아 기능 사이의 상호 작용이 강조되어 미토콘드리아 연료 이용과 생물 발생에 영향을 미친다19.
연료 활용
미토콘드리아 기능은 연료 대사와 골격근 조직의 세포 수준에서 사용되는 연료 유형에 의해 영향을 받는 것으로 보인다11. 연료 고갈 기간, 특히 인간의 탄수화물 고갈 기간 동안 (미토콘드리아) 에너지 생산에 대한 연료 선호도가 바뀝니다. 포도당 수치가 낮으면 연료 선호도가 포도당에서 지방산 및 산 유래 케톤체로 이동합니다. 이 대사 전환은 지방 세포에서 지질 대사의 상향 조절에 의해 표시되며, 이어서 혈액으로의 케톤 방출이 증가합니다4. 케토제닉 다이어트를 통해 연료 활용도를 포도당에서 케톤으로 전환하면 미토콘드리아 활성산소종 생성, 항산화 방어, ATP 합성, 생물발생에 유익한 효과가 있는 것으로 보인다20.
탄수화물에서 지질 대사로의 대사 전환은 환경적 영양소 가용성이 낮은 기간과 글리코겐 저장량이 고갈된 시기에 발생합니다. 이 스위치가 시작되면 저장된 트리글리세리드는 글루코신생식을 위한 기질인 글리세롤과 유리 지방산으로 분해되어 간으로 운반되어 β산화를 통해 아세틸 코엔자임 A(아세틸 CoA)로 산화됩니다. 케톤체는 주로 간에서 3개의 아세틸 CoA 분자를 β-하이드록시-β-메틸글루타릴-CoA로 2단계 응축하여 합성된 다음 아세토아세테이트 및 3-β에타 하이드록시부티레이트를 포함한 케톤체로 추가 처리됩니다21. 이러한 케톤체는 몸 전체의 조직에 분포되어 있으며, 심장, 뇌, 골격근에서 가장 많이 소비된다21. 노화와 함께 미토콘드리아 지방산 산화가 손상되어 대사 스위치에 영향을 미칩니다22. 미토콘드리아 연료 활용의 장애는 미토콘드리아 기능 장애를 더욱 악화시켜 노화 관련 질병 및 기능 저하에 기여하는 것으로 제안되었습니다23.
말초혈액 단핵세포(PBMC)의 미토콘드리아 산소 소비량 변화는 기능 장애 및 혈관화와 관련된 패턴을 평가하기 위해 연구되었습니다. Hartman 등 은 산소 소비와 다양하게 매개된 확장 사이의 상관관계를 결정하기 위한 연구를 수행했으며, 그 결과 미토콘드리아 기능 장애와 혈관 평활근 세포 기능 장애 사이의 연관성을 시사하는 것으로 밝혀졌다24. 다른 장기와 관련하여, PBMC는 호흡량 측정에 의해 결정된 바와 같이 더 높은 인지 및 뇌 기능과 상관관계가 있었다25. 따라서 PBMC 생체 에너지학 및 호흡 능력은 신체 전반의 장기 또는 조직의 기능적 능력을 평가하기 위한 잠재적인 바이오마커 역할을 할 수 있습니다.
일주기 리듬
미토콘드리아 건강에 영향을 미치는 또 다른 중요한 요인은 일주기 리듬입니다. 일주기 리듬은 환경 신호가 없을 때 발생하는 행동 및 생리학의 ~24시간 진동입니다26. 이러한 리듬은 시스템 및 조직의 항상성을 지원하기 위해 예측적인 방식으로 기능합니다. 일주기 리듬의 기초가 되는 메커니즘은 일주기 시계 27(circadian clock27)이라고 하는 전사-번역 피드백 루프입니다. 지난 15년 동안 생체 시계 메커니즘이 몸의 거의 모든 세포에 존재한다는 것이 입증되었습니다28. 시간을 유지하는 것 외에도, 분자 시계 메커니즘은 일주기 시계 출력29이라고 하는 유전자 발현의 일일 프로그램에도 기여합니다. clock output 유전자는 각 조직 유형에 고유하며 세포 대사, 자가포식, 복구 및 항상성에 중요한 경로와 기능적으로 연관되어 있습니다. 최근의 증거에 따르면 미토콘드리아 건강은 생체 시계 기능에 의존하며 미토콘드리아 생물 발생, 연료 활용 및 미토파지를 포함한 미토콘드리아 기능에 영향을 미친다30.
전임상 및 임상 연구에서 새롭게 드러난 증거는 노화 전반에 걸쳐 일주기 리듬에 장애가 있음을 입증했다31. 여기에는 정상적인 수면 및 각성 주기의 중단, 심부 체온 리듬의 진폭 감소,31단계의 변화에 적응하는 지연된 능력 등이 포함됩니다. 예를 들어, 한 연구에서는 성체 마우스와 늙은(20+ 개월) 마우스의 일주기 시스템에 조명 일정을 6시간 변경함으로써 도전했습니다. 늙은 쥐는 활동 패턴을 새로운 빛 시간표에 다시 동반하는 데 더 오래 걸리는 것으로 나타났습니다32. 일주기 행동의 변화와 일치하게, 조직 시계를 분석한 결과, 노화 코호트에서 중추 조직 시계와 말초 조직 시계가 모두 손상된 것으로 나타났습니다.
보다 최근에는 여러 그룹이33세의 서로 다른 조직에 걸쳐 일주기 시계와 시계 출력에 대한 전사체 분석을 수행했습니다. 이러한 연구의 결과는 나이가 들면서 생체 시계 출력의 대규모 재프로그래밍이 있음을 강조합니다. 이는 core clock가 timing function을 유지하더라도 매일 발현되는 유전자가 크게 다르다는 것을 의미합니다. 예를 들어, 2 개의 연구는 24 시간 동안 4 시간마다 인간 피험자로부터 근육 생검을 수집했으며, 그 결과는 야행성 설치류와 주간 인간 사이에서 시계 유전자 발현의 최고점과 최저점이 역전된다는 것을 확인했습니다34 , 35 , 36 . 이는 clock gene 발현을 활성 단계 대 휴지 단계(light 대 dark가 아님)에만 기준으로 비교할 때 근육의 clock gene 발현 패턴이 종 간에 거의 동일하다는 것을 나타냅니다. 이러한 연령과 관련된 시계 출력의 변화는 미토콘드리아 기능, DNA 손상 및 복구, 자가포식과 같은 노화의 알려진 특징을 포함하는 경로 조절의 장애를 초래하는 것으로 제안됩니다37.
연구 근거
미토콘드리아 기능과 신체 기능 저하 사이의 연관성은 잘 확립되어 있습니다. 그러나 미토콘드리아 기능 장애의 근본적인 원인은 여전히 논쟁의 주제로 남아 있습니다. 최근 연구에 따르면 세포 연료 활용과 일주기 리듬이 이 과정에서 중요한 역할을 할 수 있습니다. 근육 생검 샘플에서 미토콘드리아 산소 소비량을 측정하는 것과 같은 미토콘드리아 기능을 평가하는 전통적인 방법은 종종 고통스럽고 침습적인 것으로 인식되며, 이는 특히 허약하고 근감소증이 있는 성인과 같이 근육량이 적은 인구 집단에서 참여를 방해할 수 있습니다38.
이러한 한계를 감안할 때, 노인의 세포 연료 이용률과 일주기 리듬의 변화를 평가하기 위한 덜 침습적인 방법이 필요합니다. 이 연구는 이 집단의 연료 대사와 일주기 리듬을 평가하는 데 사용할 수 있는 새로운 최소 침습 프로토콜을 평가하는 것을 목표로 합니다. 이 연구의 결과는 노화 관련 변화와 의학적 또는 행동적 중재에 대한 반응에 대한 더 나은 이해에 기여할 것이며, 이 분야의 향후 연구를 위한 모델 역할을 할 것입니다.
프로토콜
인간 참가자와 관련된 절차는 연구 윤리 위원회(Florida Ethics Policy 1.0104)와 플로리다 대학의 기관 검토 위원회(Institutional Review Board)의 승인을 받았습니다.
1. 미토콘드리아 기능
- 말초 혈액 단핵 세포(PBMC)의 분리
- 특수 8mL 채혈 튜브(16mm x 125mm, 0.1M sodium citrate anticoagulant 또는 sodium heparin anticoagulant 포함)를 사용하여 PBMC를 수집합니다( 재료 표 참조).
알림: 채혈 튜브에는 요변성 폴리에스터 젤과 구배 매체( 재료 표 참조)로 구성된 혈액 분리 매체가 포함되어 있어 백혈구 분리를 향상시킵니다. - 실행 가능한 결과를 얻기 위해 채취 후 2시간 이내에 8mL 세포 준비 튜브를 처리합니다.
- 채혈관에 혈액을 채운 후 혼합하고(부드러운 반전 또는 믹서로) 최대 2시간 동안 실온(RT)에 보관합니다.
- 피팅 스윙 아웃 로터의 채혈 튜브를 2,000 × g 의 속도로 상온에서 15분(또는 30,000G-min에 해당, 2,000×g을 초과하지 않음) 동안 원심분리합니다.
참고: 이 튜브는 일반 15mL 원심분리 튜브보다 높습니다. 따라서 올바른 로터를 사용하는 데 각별한 주의를 기울여야 합니다. - 플라즈마층의 ~80%를 흡인하여 생물학적 유해 폐기물 용기에 버리고 15mL 원심분리기 튜브에 채혈 튜브(이송 피펫 사용)에서 세포층을 수집합니다.
참고: 15mL 튜브를 사용하는 경우 7mL의 세포층을 추가하고 14mL에 멸균 인산염 완충 식염수(PBS)를 채웁니다. PBS에 대한 세포의 비율은 1:1이어야 합니다. 필요한 경우 여러 개의 튜브를 사용하십시오. 뒤집어서 부드럽게 섞습니다. - 상온에서 900 × g 에서 5분(또는 4,500 G-min에 해당) 동안 스윙 아웃 로터의 원심분리기.
- 세포 펠릿을 방해하지 않고 PBS 용액을 흡인하여 생물학적 유해 폐기물 용기에 폐기합니다.
- 부드럽게 triturating하여 1mL의 기저 배지(BM, Table of Materials(재료표 참조)에 세포 펠릿을 재현탁합니다. BM을 총 10mL에 넣고 반전시켜 부드럽게 섞습니다. 셀 계수를 수행합니다.
- 900 × g 의 스윙 아웃 로터에서 세포 현탁액을 RT에서 5분 동안 원심분리합니다.
참고: 생체 에너지 평가를 위해 당일에 세포를 사용하는 경우 1.1.10단계로 진행합니다. 셀을 저장하기 위해 고정된 경우 1.1.13단계로 진행합니다. - 원하는 세포 농도를 달성하기 위해 다음 세포 펠릿의 재현탁을 위한 분석 배지의 부피(AM, 재료 표 참조)를 계산합니다.
참고: 예를 들어, 50μL에서 150,000 cells/well의 시딩 밀도의 경우 세포 현탁액의 원하는 농도는 300만 cells/mL입니다. - 세포 펠릿을 방해하지 않고 상층액을 흡인하고 생물학적 유해 폐기물 용기에 폐기합니다.
- 세포 펠릿을 1mL 이하의 AM으로 부드럽게 재현탁시킵니다. 원하는 세포 농도에 대해 계산된 총 부피에 AM을 추가합니다. 이제 세포를 코팅된 세포 배양 플레이트에 파종할 준비가 되었습니다( 재료 표 참조).
참고: 다음 단계는 저장을 위해 셀을 고정하기 위한 단계(1.1.13-1.1.16)이며 당일 절차에는 적용되지 않습니다. - 세포를 동결하는 경우, 세포 펠릿을 방해하지 않고 상층액을 흡인하고 생물학적 유해 폐기물 용기에 폐기합니다.
- 원하는 농도(5-1,000만 cells/mL)의 동결 매체( 재료 표 참조)에서 세포를 부드럽게 재현탁합니다. 표지된 cryovials에서 원하는 부피의 분취량.
- 냉동 용기( 재료 표 참조)를 사용하여 세포 현탁액을 최소 4시간 동안 또는 -80°C에서 밤새 천천히 동결합니다.
- 4 시간 후 또는 다음날 아침, cryovials를 액체 질소 저장소로 옮겨 증기 상에 상주해야합니다.
- 측정 전날, 세포 배양 마이크로플레이트( 재료 표 참조)의 웰 바닥을 세포 접착제(재료 표 참조)로 코팅합니다. 제조업체의 지침에 따라 권장 농도 22.4μg/μL로 접착제를 0.1M 중탄산나트륨(pH 8.0)에 적절한 부피의 접착제 원액을 희석하여 접착제를 준비합니다. 세포 배양 마이크로플레이트의 각 웰에 25μL의 접착제를 도포합니다.
- 20분 후 각 웰에서 남은 액체를 빨아들이고 200μL의 멸균 정제수로 웰을 두 번 세척합니다. 플레이트를 생물 안전 캐비닛에서 건조시킵니다 (~ 2 시간 동안).
- 플레이트를 알루미늄 호일로 싸거나 밀폐된 보조 용기에 넣고 4°C의 냉장고에 보관합니다.
- 특수 8mL 채혈 튜브(16mm x 125mm, 0.1M sodium citrate anticoagulant 또는 sodium heparin anticoagulant 포함)를 사용하여 PBMC를 수집합니다( 재료 표 참조).
- thereal-time metabolic flux analyzer를 사용한 PBMC의 생체 에너지 평가
참고: 플럭스 분석기 및 다중 모드 리더 사용( 재료 표), 웰당 150,000 PBMC의 최적 파종밀도, 2μM의 최종 언커플러 시안화물-4(트리플루오로메톡시) 페닐히드라존(FCCP) 농도 및 4μM의 최종 Hoechst 33342 농도(주입 후 최소 20분의 배양 시간)를 측정했습니다.- 하이드로 부스터로 센서 카트리지에 수분을 공급하려면 상자에서 유틸리티 플레이트가 있는 센서 카트리지를 제거하고 카트리지 "센서 업"을 벤치에 놓습니다. Pipet 200 μL의 보정제(재료 표 참조)를 유틸리티 플레이트의 각 웰에 삽입하고 하이드로부스터를 유틸리티 플레이트에 단단히 놓은 다음 센서 카트리지에 놓습니다. 갇힌 기포를 검사하고 제거하고 37°C non-CO2 인큐베이터에 밤새 넣습니다.
- 플럭스 분석기를 켠 다음 분석 분석 소프트웨어를 엽니다( 재료 표 참조). 온도를 37°C로 설정합니다. 설정 온도의 안정화를 위해 기기를 밤새 켜 두십시오.
알림: 다음 프로그래밍 단계(1.2.4-1.2.10)는 실험 전날 또는 당일에 수행할 수 있습니다 - 분석 소프트웨어(재료 표 참조) 파일을 프로그래밍하려면 기판 산화 응력 테스트용 템플릿을 엽니다. Group Definitions(그룹 정의)로 이동하여 Injection Strategies(주입 전략) 탭을 열고 Inhibitor + Sub Ox Stress Test(억제제 + Sub Ox Stress Test)를 Etomoxir + Sub Ox Stress Test(에토목서 + Sub Ox Stress Test)로 편집한 다음 두 가지 주입 전략(UK5099 + Sub Ox Stress Test 및 BTPES + Sub Ox Stress Test)을 추가합니다. 각 주입 전략 내의 주입 조건 창에서 포트 A에 대한 A를 클릭하고 각 억제제의 최종 농도(에토목시르: 4μM; UK5099: 2 μM; BTPES: 3 μM)입니다.
- 포트 B, C 및 D를 클릭하고 각각의 최종 농도를 입력합니다(포트 B: 올리고마이신 농도[1.50μM], 포트 C: FCCP[2.0μM]; 포트 D: 로테논 + 안티마이신 A[0.5μM]). 포트 D의 화합물 창에 Hoechst 33342, 4μM를 추가합니다.
- Pretreatments(전처리) 탭을 열고 해당하는 경우 전처리를 선택합니다. 예: Control(제어), Experimental(실험) 또는 customary(예: Baseline 및 Follow up). 필요한 경우 아래 창에 설명을 추가합니다.
- Assay Media(분석 미디어) 탭을 열고 Media(미디어) 풀다운 메뉴에서 RPMI Medium(RPMI 배지), pH 7.4를 선택하고, 로트 번호, Media(Media) 준비 인력, 준비 시간 및 Media에 추가된 보충제(10mM 포도당, 2mM 글루타민, 1mM 피루브산, Table of Materials(재료표 참조))를 입력합니다.
- Cell Type(셀 유형) 탭을 열고 Name(이름)으로 PBMC-Subject ID, Cell Type(세포 유형)으로 PBMC, Seeding density(시딩 밀도)로 150000, Source(소스)로 Subject ID, 세포를 준비한 인력, 준비 날짜 및 세포 해동일(해당되는 경우)을 입력합니다.
- Generate Groups(그룹 생성)를 클릭하고 Plate Map(플레이트 맵)으로 이동하여 96웰 플레이트의 웰을 각 그룹에 할당합니다. 웰 A1, A12, H1 및 H12를 배경 웰로 지정된 상태로 유지합니다.
- Protocol( 프로토콜)로 이동하여 Equilibrate(평형) 상자(기본 설정)를 선택하고 다음 정보를 입력합니다. 기준선: 5가지 측정 주기: 3분 혼합, 0분 대기 및 3분 측정; 매체 또는 억제제(포트 A): 6가지 측정 주기: 3분 혼합, 0분 대기 및 3분 측정; 올리고마이신(포트 B): 3가지 측정 주기: 3분 혼합, 0분 대기 및 3분 측정; FCCP(포트 C): 3가지 측정 주기: 3분 혼합, 0분 대기 및 3분 측정; Rotenone + Antimycin A + Hoechst 33342 (Port D) : 3 가지 측정주기: 3 분 혼합, 0 분 대기 및 3 분 측정.
- 생체 에너지 평가 당일, 냉장고에서 코팅된 세포 배양 마이크로플레이트를 꺼내 생물 안전 캐비닛에서 RT까지 예열합니다.
- 97mL의 예열된 BM과 보충제를 혼합하여 분석 배지(AM)를 준비합니다: 피루브산 1mL(최종 농도: 1mM), 포도당 1mL(최종 농도 10mM) 및 글루타민 1mL(최종 농도: 2mM). 사용할 때까지 37°C의 AM을 비 CO2 인큐베이터에 두십시오.
- 세포를 시딩하려면 A1, H1, A12 및 H12(백그라운드 웰로 할당됨)를 제외한 각 웰에 150,000개의 PBMC가 포함된 50μL의 세포 현탁액(1.1.12부터)을 추가합니다.
위험: 최적의 시딩 밀도는 이전에 결정되었습니다. - 세포판을 뚜껑으로 덮고 세포의 균일한 분포를 촉진하기 위해 생물 안전 캐비닛의 RT에서 60분 동안 세포를 휴지시킵니다.
중요: 현미경으로 세포를 관찰하여 웰 전체에 걸쳐 원하는 균질한 세포 분포를 확인합니다. - 세포 이미징의 경우, 첫 번째 세포 배양 몇 시간 전에 다중 모드 리더( 재료 표 참조)를 켭니다. 이미징 소프트웨어를 열기 전에 리더의 시작 프로토콜이 완료될 때까지 기다립니다( 재료 표 참조). 이미징 소프트웨어를 열고 예열 온도를 37°C로 설정합니다.
- 분석 시약을 준비하려면 분석 키트의 지침에 따라 표 1에 설명된 대로 약간의 수정을 가하여 시약을 재구성합니다.
중요: 최적의 최종 FCCP 및 Hoechst33342 농도가 이전에 결정되었습니다. - 60분 동안 세포를 안정시킨 후 세포 배양 현미경으로 웰을 검사하고 세포 응집과 같은 시각적 이상을 기록하십시오.
- 60 × g 의 마이크로플레이트용 스윙아웃 로터가 있는 원심분리기에서 RT의 세포 배양 플레이트를 최소 가속 및 최소 차단으로 1분 동안 원심분리합니다. 플레이트를 돌리고 40×g에서 1 분 동안 다시 원심분리합니다. 가속과 브레이크를 최소로 설정합니다. non-CO2 incubator에서 37 ° C에서 25-30 분 동안 세포를 배양합니다.
참고: 세포 이미징이 수행되면 대신 예열된 다중 모드 리더에서 세포 플레이트를 배양하고( Table of Materials(재료 표 참조) 이 배양 기간(첫 번째 세포 배양) 동안 각 웰의 명시야 이미지를 촬영합니다. - 바코드 스캐너로 세포 배양 플레이트의 바코드를 스캔하고(재료 표 참조), 명시야 스캔 시작을 선택하고, 세포 배양 플레이트를 플레이트 트레이에 올려 놓고, 트레이 후퇴를 시작합니다. 스캔할 세포가 포함된 웰을 선택하고 스캔을 시작합니다.
- 첫 번째 세포 배양 동안, 표 1에 표시된 대로 센서 카트리지의 포트에 시약을 로드합니다 : 포트 (A): 20 μL of AM (대조군), 에토목시르(미토콘드리아로의 장쇄 지방산 수송 억제제), UK 5099 (2-Cyano-3-(1-phenyl-1H-indol-3-yl)-2-propenoic acid; 미토콘드리아 피루브산 운반체 억제제) 또는 BPTES (Bis-2-(5-phenylacetamido-1, 3,4-thiadiazol-2-yl)에틸 설파이드; 글루타민이 글루타메이트로 전환되는 억제제); 포트 (B) : 22 μL의 올리고마이신 (ATP 합성효소 억제제); 포트 (C) : 25 μL의 FCCP (uncoupler carbonyl cyanide-4 (trifluoromethoxy) phenylhydrazone); 포트 (D) : 27 μL의 로테논 / 항 마이신 A / Hoechst 33342 (각각 미토콘드리아 복합체 I 및 III의 억제제 및 핵 염료). 센서 카트리지를 37°C non-CO2 인큐베이터에 5분 더 넣습니다.
참고: 포트 로드에 대한 제조업체의 지침을 따르십시오. - 첫 번째 세포 배양 후 및 세포 이미징이 완료된 후(해당되는 경우) 인큐베이터 또는 플레이트 리더에서 세포 배양 플레이트를 꺼내고 각 웰에 따뜻한 AM을 웰당 180μL의 최종 부피로 추가합니다. cell plate를 non-CO2 incubator에 37°C로 15-25분 더 놓습니다(두 번째 세포 배양).
- 두 번째 세포 배양 중에 센서 보정을 시작하여 대사 분석을 시작합니다. 센서 카트리지의 보정을 시작할 준비가 되면 Run Assay(분석 실행 )를 클릭합니다. 메시지가 표시되면 센서 카트리지를 37°C non-CO2 인큐베이터에서 플럭스 분석기 트레이로 옮기고 보정을 시작합니다. 분석 분석 소프트웨어의 지시에 따라 보정이 완료된 후 셀 플레이트의 유틸리티 플레이트를 교체합니다.
참고: 보정에는 약 20분이 소요됩니다. - 대사 분석 후 소프트웨어 지시에 따라 기기에서 센서 카트리지와 셀 플레이트를 제거합니다. 셀 플레이트에서 센서 카트리지를 제거하고 셀 플레이트를 뚜껑으로 덮습니다.
- 예비 실험에서 Hoechst 염료 배양 시간을 결정한 후 플레이트의 바코드를 스캔하고 소프트웨어 지시에 따라 다중 모드 리더로 각 세포 함유 웰의 형광 이미지를 획득합니다.
참고: 이미징 및 분석 소프트웨어가 상호 연결되어 있어 세포 계수에 대한 데이터 정규화를 위해 세포 계수 데이터를 분석 데이터 파일로 가져올 수 있습니다.
2. 일주기 시계 유전자 발현
참고: 참가자의 PBMC에서 시계 유전자 발현은 RNA 혈액 키트를 사용하여 RNA를 분리하여 검토됩니다( 재료 표 참조).
- 참가자의 혈액 3mL를 안정화 시약 6mL와 함께 RNA 튜브( 재료 표 참조)에 직접 추출합니다. 완전히 섞일 때까지 10초 동안 소용돌이.
참고: 안정화 시약이 참가자의 혈액 샘플과 완전히 혼합되지 않으면 연구 오류의 위험이 높아집니다. - 안정화된 혈액 샘플 1.0mL를 적혈구 용해 완충액 5mL와 함께 15mL 튜브로 옮깁니다. 얼음 위에서 10-15분 동안 배양합니다. 배양 중에 튜브를 2번 소용돌이칩니다. 튜브를 4°C에서 400× g 으로 10분 동안 원심분리합니다.
- 펠릿을 확인하고 용해된 적혈구가 포함된 상층액을 조심스럽게 붓습니다. 적혈구 용해 완충액 2mL를 추가하고( 재료 표 참조), 세포를 재현탁하고 잠시 와류를 일으킨 다음 400 × g 에서 4°C에서 10분 동안 원심분리합니다.
- 상층액을 붓고 튜브를 흡수성 종이에 1-2분 동안 거꾸로 둡니다. 다음 단계를 수행하기 전에 깨끗한 종이로 튜브 가장자리 주변의 액체를 닦아내십시오.
- 600μL의 RNA 용해 완충액을 조심스럽게 추가합니다( 재료 표 참조). 제조업체의 프로토콜에 따라 β-메르캅토에탄올을 추가하고 펠릿을 재현탁합니다.
- 용해물을 일회용 세포 용해물 균질화기( 재료 표 참조) 스핀 컬럼에 직접 피펫하고 2mL 수집 튜브에 넣고 최대 속도로 2분 동안 원심분리하여 균질화합니다. 일회용 cell-lysate homogenizer 스핀 컬럼을 버리고 homogenized lysate를 저장합니다.
- 균질화된 용해물에 1부피(600μL)의 70% 에탄올을 첨가하고 피펫팅으로 혼합합니다. 형성되었을 수 있는 침전물을 포함하여 샘플을 2mL 수집 튜브의 새 스핀 컬럼에 조심스럽게 피펫팅하되 가장자리를 적시지 마십시오. >8,000 × g에서 15초 동안 원심분리기.
- 스핀 컬럼( 재료 표 참조)을 새 2mL 수집 튜브로 옮깁니다. 700μL의 엄격한 세척 버퍼( 재료 표 참조)를 스핀 컬럼에 적용하고 >8,00× 0g 에서 15초 동안 원심분리기를 사용하여 세척합니다. 플로우스루를 폐기합니다.
- 스핀 컬럼을 조심스럽게 열고 500μL의 순한 세척 버퍼를 추가합니다( 재료 표 참조). 캡을 닫고 3분 동안 최대 속도(20,000× g)로 원심분리기를 닫습니다.
- 스핀 컬럼을 1.5mL 마이크로 원심분리 튜브로 옮기고 30-50μL의 RNase-free 물을 실리카 멤브레인에 직접 피펫으로 주입합니다. >8,000 × g 에서 1분 동안 원심분리기를 사용하여 RNA를 용리합니다. 한 번 더 반복합니다.
참고: RNA는 -80°C에서 보관할 수 있습니다. - 제조업체의 프로토콜에 따라 RNase-free DNase 세트( 재료 표 참조)를 사용하여 컬럼에서 DNase 처리를 수행합니다.
- 제조업체의 프로토콜에 따라 총 RNA 500ng 및 실시간 중합효소연쇄반응(PCR) 시스템( 재료 표 참조)을 사용하여 cDNA를 생성합니다. 모든 cDNA 샘플을 RNase가 없는 물에 1:25로 희석하고 4mL를 사용하여 정량적 역전사 PCR(qRT-PCR)을 수행합니다.
- 프라이머 첨가 방법( 재료 표 참조)을 사용하여 표 2에 표시된 각 프라이머를 10mM씩 사용하여 qRT-PCR을 완료합니다. Real-Time 시스템을 사용하여 qRT-PCR을 완료합니다( 재료 표 참조).
- Rpl26 mRNA 수치를 사용하여 선택한 유전자의 mRNA 수치를 정규화합니다. 2-DDCt 방법을 사용하여 상대 정량화를 계산합니다.
- 주어진 mRNA의 발현이 일주기 진동을 나타내는지 확인하기 위해 하나의 실험 조건40에서 일주기 리듬을 검출하기 위해 LR_rhythmicity39 가능성 기반 테스트(p≤ 0.01 사용)를 활용하십시오.
3. 데이터 분석 계획
참고: 약물 사용에 따라 참가자를 분류하기 위해 의료 목록이 사용됩니다43.
- 연령, 성별, 체중, 키, 혈압, 심박수 및 기타 위험 요인을 공변량으로 포함해야 하는 혼합 효과 선형 모형을 사용합니다.
- 개체 내 데이터 상관 관계를 설명하기 위해 무작위 효과 항을 포함해야 합니다.
- 모델 피팅의 경우 계층적 원칙에 따라 역방향 변수 선택을 구현해야 합니다.
- 적합된 모형에서 적합모형의 모든 공변량이 주어지면 시점 1부터 시점 2까지의 변화를 측정합니다.
결과
제안된 프로토콜에는 방법론에 대한 검증 역할을 하는 예비 데이터가 포함되어 있습니다. 이 프로토콜은 미토콘드리아 기능 및 세포 연료 활용을 검사하기 위한 실시간 대사 플럭스 분석기와 일주기 리듬 유전자(예: BMAL1, CLOCK, Nfil2, Nr1d1, Dbp, Cry1, Per2)를 분석하기 위한 RNA 추출 및 qRT-PCR을 통합합니다.
초기 분석 후 10일 후에 5명의 대조군 참가자로부터 분리된 인간 PBMC의 산소 소비율(OCR)이 그림 1에 제시되어 있습니다. 이 데이터는 사전값과 사후값을 비교하는 데 사용되며 대조군, 에토목시르, UK5099 및 BPTES 주입 후 기저 호흡, 급성 반응, 최대 호흡 및 예비 용량의 평균값을 보여줍니다. 특히, 그림 1C 는 에토목시르 주입 후 유의한 음성 급성 반응을 보여주지만, 기저 호흡, 최대 호흡 또는 예비 용량에 대해서는 유의한 효과가 관찰되지 않았습니다.
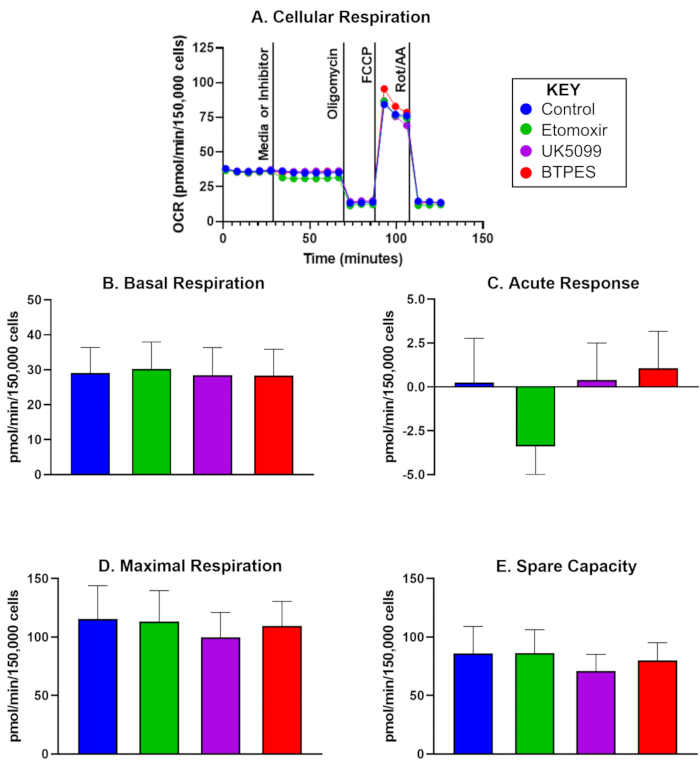
그림 1: 분리된 인간 말초 혈액 단핵 세포(PBMC)의 산소 소비량.(A) 대조군에서 분리한 PBMC의 실시간 산소 소비율(OCR, pmol/(min∙150,000 cells)을 플럭스 분석기로 측정하고 기질 산화 분석으로 평가. 세포는 150,000 cells/well의 밀도로 파종되었습니다. 첫 번째 주사는 배지(대조군) 또는 억제제(에토목시르, UK5099 또는 BPTES, 자세한 내용은 텍스트 참조)였으며 기초 세포 호흡수를 측정한 후 수행되었습니다. 미토콘드리아 기질 한계에 대한 급성 반응은 억제제 주입 전과 후의 기저 OCR의 차이로 결정되었습니다. ATP 합성효소 억제제인 올리고마이신(Oligomycin)은 ATP 생산 결합 호흡을 억제하고 양성자 누출 호흡을 생성합니다. FCCP는 언커플러(uncoupler)로서 최대한의 비결합 호흡을 유도합니다. 로테논 및 항마이신 A(각각 복합체 I 및 III의 억제제)는 비미토콘드리아 호흡을 제외한 모든 것을 억제합니다(자세한 내용은 텍스트 참조). (B-E) 세포 호흡의 정량화(n = 5, 데이터는 평균 ± SD로 표시됨). (B) 억제제 주입 전 기초 OCR, (C) 억제제에 대한 급성 반응(억제제 주입 전 기초 비율에 대한 OCR 변화), (D) 최대 OCR, (E) 예비 용량(첫 번째 주입 후 최대 OCR과 기초 OCR의 차이). 에토목시르 주입에 대한 급성 반응(C)은 높은 에너지 수요(D) 동안 OCR에 눈에 띄는 영향을 미치지 않고 다른 기질 그룹에 비해 기초 조건에서 에너지 기질로서 지방산에 대한 OCR의 의존성이 더 높다는 것을 시사할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 화합물 | 화합물에 AM (μL) 첨가 | 재고(μM) | 작업 재고에 대한 재고 (μL) | 작업 재고의 경우 AM(μL) | 작업 재고(μM) | 작업 재고(μL) [포트] | 최종 농도 (μM) |
| 에토목시르 | 700 | 160 | 500 | 1500 | 40 | 20 [A] | 4 |
| 영국5099 | 700 | 80 | 500 | 1500 | 20 | 20 [A] | 2 |
| 비피테스 | 700 | 120 | 500 | 1500 | 30 | 20 [A] | 3 |
| 올리고 | 420 | 150 | 300 | 2700 | 15 | 22 [나] | 1.5 |
| FCCP (영어) | 720 | 100 | 600 | 2400 | 20 | 25 [기음] | 2 |
| 썩음/AA/H | 540 | 50 | 300 | 2700 | 5 | 27 [디] | 0.5 |
표 1: 기질 산화 테스트를 위한 시약 준비 및 스톡, 작업 및 최종 용액의 농도. 모든 시약은 세포 mito stress test 또는 substrate oxidation stress test kit의 일부입니다. 약어: oligo = oligomycin; FCCP = 언커플러 카르보닐 시안화물-4(트리플루오로메톡시) 페닐히드라존; Rot/AA/H = 로테논/항마이신 A/Hoechst 33342. Etomoxir, UK5099, BTPES: 각각 지방산, 포도당 및 글루타민 산화 억제제.
| 바흐1 | 앞으로 – GCACGACGTTCTTTCTTCTGT |
| 리버스 – GCAGAAGCTTTTTCGATCTGCTTTT | |
| 시계 | 앞으로 – CGTCTCCACAACCCTTCCTCAAC |
| 리버스 – GTAAATGCTGCCTGGGTGGA | |
| 크라이1 | 앞으로 – ACTGCTATTGCCCTGTTGGT |
| 리버스 – GACAGGCAAATAACGCCTGA | |
| 당1 | 앞으로 – ATTCGGGTTACGAAGCTCCC |
| 리버스 – GGCAGCCCTTTCATCCACAT | |
| 퍼2 | 앞으로 – CATGTGCAGTGGAGCAGATTC |
| 리버스 – GGGGTGGTAGCGGATTTCAT | |
| Rev-erb α | 포워드 – ACAGATGTCAGCAATGTCGC |
| 리버스 – CGACCAAACCGAACAGCATC |
표 2: 일주기 시계 유전자 프라이머.
토론
나이가 들면서 미토콘드리아 기능의 저하와 일주기 리듬 조절이 노화 관련 질병의 원인으로 점점 더 많이 여겨지고 있습니다. 식단 및 신체 활동과 같은 생활 방식 수정을 통해 생체 리듬을 변경하는 것은 건강한 노화를 촉진하고 노화와 관련된 이동성 저하를 줄일 수 있는 잠재적인 전략입니다. 그러나 미토콘드리아 기능을 직접 평가하는 현재의 방법은 침습적이며 종종 근육 생검이 필요하기 때문에 인지된 통증과 위험으로 인해 참가자 모집 및 유지에 어려움을 겪을 수 있습니다.
혈액 채취와 같은 덜 침습적인 방법을 통해 일주기 및 대사 건강의 마커를 평가하면 향후 연구에서 치료 대상을 탐색하고 테스트하는 데 유용한 결과를 제공할 수 있습니다. 이러한 최소 침습적 방법은 일주기 리듬과 신진대사 건강 사이의 복잡한 상호 작용과 기능에 미치는 영향에 대한 새로운 통찰력을 제공함으로써 이 분야를 크게 발전시킬 수 있는 잠재력을 가지고 있습니다. 이 연구의 목표는 세포 에너지 대사와 일주기 리듬 사이의 관계를 평가하는 것입니다. 특히, 생체 에너지 플럭스 분석은 참가자의 백혈구에서 일주기 유전자 그룹의 유전자 발현 모니터링과 함께 다양한 기질 가용성 조건에서 미토콘드리아 기능을 평가하는 데 사용됩니다. 분석의 두 부문, 즉 생체 에너지와 유전자 발현을 사용함으로써 이 두 가지 기본 과정 사이의 관계에 대한 포괄적인 이해를 달성할 수 있습니다.
일주기 관점에서 이 시계열 데이터의 통계적 분석은 일주기 리듬의 강도, 범위 및 타이밍에 대한 통찰력을 제공합니다. 결론적으로, 유기체 수준에서 유전자 발현 분석, 세포 생물 에너지학 및 대사 측정의 통합은 인간의 에너지 대사와 일주기 리듬 간의 상호 작용을 밝힐 새롭고 혁신적인 접근 방식을 구성합니다.
파일럿 연구에서 PBMC의 OCR에서 지방산 활용 제한에 대한 급성 반응을 감지했습니다(카르니틴 팔미토일 전이효소 1a의 억제제인 에토목시르 주사 후). 이 발견은 이 특정 참가자 그룹의 PBMC에서 기저 호흡 중 에너지 기질로 지방산에 의존할 수 있음을 시사합니다. 그러나 최대 호흡에는 영향을 미치지 않았으며, 이는 포도당 및 글루타민과 같은 대체 에너지원이 높은 에너지 수요 동안 지방산의 감소된 활용을 보상할 수 있음을 시사합니다. 향후 연구에서는 a) PBMC의 생체 에너지학이 전신 에너지를 반영하는지, b) 시간 제한 식사와 같은 중재가 에너지 기질 선호도에 영향을 미칠 수 있는지 여부를 조사해야 합니다.
PBMC의 플럭스 분석과 관련하여 몇 가지 중요한 단계가 있습니다. 먼저, 실험 샘플을 평가하기 전에 각 웰 내부와 각 플레이트에 걸쳐 세포가 연속적으로 균일하게 분포되어 있는지 확인하여 세포 파종 밀도(웰당 세포)를 최적화해야 하며, 최종 FCCP 농도는 농도 0, 0.125, 0.25, 0.5, 1.0 및 2.0μM을 사용하여 농도 테스트 실행을 실행하여 최적화해야 합니다. 또한 해당되는 경우 Hoechst 33342 염색은 제조업체의 지침에 따라 최적화해야 합니다. 둘째, 세포 매개변수에 대한 대사 데이터의 정규화는 실험 간 데이터의 비교 가능성에 매우 중요합니다. 본 프로토콜에서는 Hoechst 33342-염색 세포 및 세포 이미징 장치를 사용하여 플럭스 분석기 분석 종료 후의 세포 계수를 설명합니다. 적절한 장치를 사용할 수 없는 경우 총 세포 단백질 또는 웰당 핵 DNA 함량과 같은 대체 정규화 방법을 적용할 수 있습니다. 제안된 것과 비교하여 프로토콜 내에서 사용할 수 있는 주목할만한 수정 사항이 있습니다. 구체적으로, 프로토콜은 여기에서 제안된 두 개의 키트와 비교하여 세 가지 억제제 각각에 대한 개별 분석 키트를 사용하여 완료할 수 있습니다( 재료 표 참조).
노인의 에너지 대사와 일주기 리듬 사이의 상호 작용을 연구하기 위한 대리모로 PBMC를 사용하는 것은 치료에 대한 반응이 다른 조직과 기관의 반응을 정확하게 반영할 수 있다는 가정에 의해 제한됩니다. 이 접근법은 새롭고 최소 침습적이지만, 뇌, 간, 골격근과 같은 다양한 조직과 기관이 다양한 조건에서 다르게 반응할 수 있다는 점을 인정하는 것이 중요합니다. 전임상 연구는 먹이를 먹은 마우스와 공복 마우스에서 시계 유전자 발현이 변경되어 간과 근육 조직에서 BMAL1 표적 유전자의 부분적인 상향 조절로 이어졌지만 다른 마우스의 하향 조절로 이어졌다는 것을 입증했습니다41. 이러한 말초 조직과 기관은 대사 과정을 매우 잘 나타내며 시계 유전자 발현 메커니즘에 영향을 미치는 환경적 단서의 영향을 받을 수 있습니다42. 말초 조직, 장기 및 중앙 생체 시계 사이의 관계를 완전히 이해하기 위해서는 추가 연구가 필요합니다.
또 다른 제한 사항은 참가자가 처방전을 복용해도 실격되지 않는다는 것인데, 이는 통계 분석에 제한을 가할 수 있습니다. 이러한 제한에 대응하기 위해, 향후 연구에서는 약물을 복용하는 노인 집단에서 검증된 의료 목록이 사용될 것이다43. 참가자는 프로토콜의 데이터 검토 섹션에 기록된 권장 약물에 따라 분류됩니다. 약물 치료제는 1) 기능 저하를 가속화하고, 2) 기능 저하를 늦추고, 3) 골격근 기능에 영향을 미치는 것으로 나타났습니다.
마지막으로, 인간의 골격근 미토콘드리아 산화 능력은 낮과 밤의 리듬을 나타내며, 06:00 pm에서 11:00 pm 사이에 최고조에 달하고 08:00 am에서 11:00 am44 사이에 감소합니다. 이것이 PBMC의 미토콘드리아 산화 능력에 적용되는지는 아직 명확하지 않습니다. 그러나 예비 데이터에 따르면 PBMC와 미토콘드리아 대사는 관련이 있는 것으로 나타났습니다45. 근육 생검 및 PBMC의 변화에 대한 정보가 명확하지 않다는 점을 감안할 때 결과를 분석할 때 주의를 기울여야 합니다. 이러한 한계를 감안할 때, 프로토콜의 유효성과 효율성을 보장하는 데 도움이 될 수 있는 귀중한 컨텍스트와 통찰력을 제공할 수 있으므로 프로토콜을 평가하고 개발할 때 이 정보를 염두에 두는 것이 중요합니다.
우리가 아는 한, 이 프로젝트에서 제안된 방법을 통해 연료 사용 패턴이나 생체 리듬을 평가한 이전 연구는 없습니다. 우리의 목표는 미토콘드리아 연료 활용 및 일주기 건강 지표의 변화에 대한 반응성을 조사하는 것입니다. 이 연구는 고감도 바이오마커를 측정하기 위한 최소 침습적 방법을 제시하며, 이는 근육 생검이 불가능한 향후 중재 연구에서 대안으로 사용될 수 있습니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
이 연구는 임상 및 중개 과학 연구소(NIH/NCRR UL1TR000064)의 지원을 받아 NIH/NIA P30AG028740(Older American's Independence Center)의 자금 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Agilent Technologies Cell Imaging | Agilent Technologies | Cell image software | |
| Agilent/Seahorse Wave desktop program | Agilent Technologies | 5994-0039EN | Software used to analyse data from the celluar analyser and stress test assay |
| Agilent/Seahorse XFe96 Flux Analyzer | Agilent Technologies | S7800B | Real-time cellular flux analyzer; flux analyser |
| Bar Code Reader | Agilent Technologies | G2615-90007 | |
| Seahorse Wave Desktop Software | Agilent technologies | Data acquisition software; assay analysis; wave program | |
| Seahorse XF 1.0 M Glucose solution | Agilent Technologies | 103577-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 100 mM Pyruvate solution | Agilent Technologies | 103578-100 | Supplement to basal medium to make assay medium |
| Seahorse XF 200 mM Glutamine solution | Agilent Technologies | 103579-100 | Supplement to basal medium to make assay medium |
| Seahorse XF Cell Mito Stress Test | Agilent Technologies | 103015-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF Mito Fuel Flex Test | Agilent Technologies | 103260-100 | Mitochondrial bioenergetic function assay |
| Seahorse XF RPMI Medium | Agilent Technologies | 103576-100 | Basal medium for PBMCs |
| Seahorse XFe96 FluxPak mini | Agilent Technologies | 102601-100 | Sensor cartridges and cell culture microplates |
| Cytation 1 Cell Imaging Multi-Mode Reader | Agilent/BioTek | Multimode reader to image cells | |
| CPT Sodium Heparin Tube, 16 x 125 mm x 8.0 mL | Becton Dickinson | 362753 | Blood collection tubes for isolation of peripheral blood mononuclear cells |
| CellTak Cell and Tissue Adhesive | Corning | 354240 | Cell adherent to coat cell culture microplate |
| Phosphate Buffered Saline | Corning | 21-040-CV | Buffer to wash blood cells |
| Ficoll Paque Plus | Cytiva | GE17-1440-02 | Gradient medium |
| Lunar Prodigy DXA scanner | General Electric | EN 60601-2-7 5.1 | Whole body lean mass and fat/lean tissue mass ratio |
| Freezing container, Nalgene Mr. Frosty | MilliporeSigma | C1562 | Freezing container used to slow-freeze cell suspension |
| Buffer EL. | Qiagen | 79217 | Erythrocyte lysis buffer |
| Buffer RLT | Qiagen | 79216 | RNA lysis buffer |
| Buffer RPE | Qiagen | 1018013 | Mild washing buffer |
| Buffer RW1 | Qiagen | 1053394 | Stringent washing buffer |
| QIAamp DNA Micro Kit | Qiagen | 56304 | DNA preps: QIAamp MinElute Columns, Proteinase K, Carrier RNA, Buffers, Collection Tubes (2 ml) |
| QIAamp RNA Blood Mini Kit | Qiagen | 52304 | RNA blood kit; Used to isolate RNA |
| QIAshredder | Qiagen | 79656 | disposable cell-lysate homogenizers |
| RNase-Free DNase Set | Qiagen | 79254 | Used to perform DNA digest |
| 2-Mercaptoethanol (Reagent) | Thermo Fisher Scientific | MFCD00004890 | |
| 2-mL collection tubes, 100 count | Thermo Fisher Scientific | AM12480 | |
| Fast SYBR Green Master Mix | Thermo Fisher Scientific | 4385612 | Primers are added to this and used to carry out qRT-PCR |
| Microcentrifuge Tubes, 1.5 mL | Thermo Fisher Scientific | 69715 | Used to hold RNA purification filter during RNA purification |
| Narrow p1000 pipette tips | Thermo Fisher Scientific | 02-707-402 | |
| QuantStudio 3 Real-Time PCR System, MiniAmp Plus Thermal Cycler, and 96-Well Plates Package | Thermo Fisher Scientific | A40393 | |
| Tempus Blood RNA Tube | Thermo Fisher Scientific | 4342792 | RNA Tube |
| Tempus Spin RNA Isolation kit | Thermo Fisher Scientific | 4380204 | RNA extraction and isolation |
참고문헌
- UN Department of Economic and Social Affairs. Population Division 2019, World Population Prospects. UN Department of Economic and Social Affairs. , (2019).
- Anton, S., Leeuwenburgh, C. Fasting or caloric restriction for healthy aging. Experimental Gerontology. 48 (10), 1003-1005 (2013).
- Dziechciaż, M., Filip, R. Biological psychological and social determinants of old age: Bio-psycho-social aspects of human aging. Annals of Agricultural and Environmental Medicine. 21 (4), 835-838 (2014).
- Anton, S. D., et al. Flipping the metabolic switch: understanding and applying the health benefits of fasting. Obesity. 26 (2), 254-268 (2018).
- Fried, L. P., Guralnik, J. M. Disability in older adults: evidence regarding significance, etiology, and risk. Journal of the American Geriatrics Society. 45 (1), 92-100 (1997).
- Manini, T. Development of physical disability in older adults. Current Aging Science. 4 (3), 184-191 (2011).
- Chung, H. Y., et al. Molecular inflammation: underpinnings of aging and age-related diseases. Ageing Research Reviews. 8 (1), 18-30 (2009).
- Sun, N., Youle, R. J., Finkel, T. The mitochondrial basis of aging. Molecular Cell. 61 (5), 654-666 (2016).
- Tarasov, A. I., Griffiths, E. J., Rutter, G. A. Regulation of ATP production by mitochondrial Ca2. Cell Calcium. 52 (1), 28-35 (2012).
- Chistiakov, D. A., Sobenin, I. A., Revin, V. V., Orekhov, A. N., Bobryshev, Y. V. Mitochondrial aging and age-related dysfunction of mitochondria. Biomed Research International. 2014, 238463 (2014).
- Boengler, K., Kosiol, M., Mayr, M., Schulz, R., Rohrbach, S. Mitochondria and ageing: role in heart, skeletal muscle and adipose tissue. Journal of Cachexia, Sarcopenia, and Muscle. 8 (3), 349-369 (2017).
- Drew, B., et al. Effects of aging and caloric restriction on mitochondrial energy production in gastrocnemius muscle and heart. American Journal of Physiology Regulatory, Integrative and Comparative Physiology. 284 (2), R474-R480 (2003).
- Short, K. R., et al. Decline in skeletal muscle mitochondrial function with aging in humans. Proceedings of the National Academy of Sciences. 102 (15), 5618-5623 (2005).
- Musci, R. V., Hamilton, K. L., Miller, B. F. Targeting mitochondrial function and proteostasis to mitigate dynapenia. European Journal of Applied Physiology. 118 (1), 1-9 (2018).
- Picca, A., et al. Targeting mitochondrial quality control for treating sarcopenia: lessons from physical exercise. Expert Opinion on Therapeutic Targets. 23 (2), 153-160 (2019).
- Fernandez-Marcos, P. J., Auwerx, J. Regulation of PGC-1α, a nodal regulator of mitochondrial biogenesis. The American Journal of Clinical Nutrition. 93 (4), 884-890 (2011).
- Kim, Y., Triolo, M., Hood, D. A. Impact of aging and exercise on mitochondrial quality control in skeletal muscle. Oxidative Medicine and Cellular Longevity. 2017, 3165396 (2017).
- Wang, H., Hiatt, W. R., Barstow, T. J., Brass, E. P. Relationships between muscle mitochondrial DNA content, mitochondrial enzyme activity and oxidative capacity in man: alterations with disease. European Journal of Applied Physiology and Occupational Physiology. 80 (1), 22-27 (1999).
- Tian, Q., et al. Muscle mitochondrial energetics predicts mobility decline in well-functioning older adults: The baltimore longitudinal study of aging. Aging Cell. 21 (2), e13552 (2022).
- Sardon Puig, L., Valera-Alberni, M., Cantó, C., Pillon, N. J. Circadian rhythms and mitochondria: connecting the dots. Frontiers in Genetics. 9, 452 (2018).
- Gano, L. B., Patel, M., Rho, J. M. Ketogenic diets, mitochondria, and neurological diseases. Journal of Lipid Research. 55 (11), 2211-2228 (2014).
- Liesa, M., Shirihai, O. S. Mitochondrial dynamics in the regulation of nutrient utilization and energy expenditure. Cell Metabolism. 17 (4), 491-506 (2013).
- Lesnefsky, E. J., Chen, Q., Hoppel, C. L. Mitochondrial metabolism in aging heart. Circulation Research. 118 (10), 1593-1611 (2016).
- Hartman, M. L., et al. Relation of mitochondrial oxygen consumption in peripheral blood mononuclear cells to vascular function in type 2 diabetes mellitus. Vascular Medicine. 19 (1), 67-74 (2014).
- Mahapatra, G., et al. Blood-based bioenergetic profiling is related to differences in brain morphology in African Americans with Type 2 diabetes. Clinical Science. 132 (23), 2509-2518 (2018).
- Moore-Ede, M. C. Physiology of the circadian timing system: predictive versus reactive homeostasis. The American Journal of Physiology. 250 (5), R737-R752 (1986).
- Young, M. W. Life's 24-hour clock: molecular control of circadian rhythms in animal cells. Trends in Biochemical Sciences. 25 (12), 601-606 (2000).
- Yoo, S. H., et al. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proceedings of the National Academy of Sciences. 101 (15), 5339-5346 (2004).
- Zhang, R., Lahens, N. F., Ballance, H. I., Hughes, M. E., Hogenesch, J. B. A circadian gene expression atlas in mammals: implications for biology and medicine. Proceedings of the National Academy of Sciences. 111 (45), 16219-16224 (2014).
- de Goede, P., Wefers, J., Brombacher, E. C., Schrauwen, P., Kalsbeek, A. Circadian rhythms in mitochondrial respiration. Journal of Molecular Endocrinology. 60 (3), R115-R130 (2018).
- Hood, S., Amir, S. The aging clock: circadian rhythms and later life. The Journal of Clinical Investigation. 127 (2), 437-446 (2017).
- Sellix, M. T., et al. Aging differentially affects the re-entrainment response of central and peripheral circadian oscillators. The Journal of Neuroscience. 32 (46), 16193-16202 (2012).
- Sato, S., et al. Circadian reprogramming in the liver identifies metabolic pathways of aging. Cell. 170 (4), 664-677 (2017).
- Lundell, L. S., et al. et al. feeding alters lipid and amino acid metabolite rhythmicity without perturbing clock gene expression. Nature Communications. 11 (1), 4643 (2020).
- Perrin, L., et al. Transcriptomic analyses reveal rhythmic and CLOCK-driven pathways in human skeletal muscle. eLife. 7, e34114 (2018).
- Gutierrez-Monreal, M. A., Harmsen, J. -. F., Schrauwen, P., Esser, K. A. Ticking for metabolic health: the skeletal-muscle clocks. Obesity. 28, S46-S54 (2020).
- Wolff, C. A., et al. Defining the age-dependent and tissue-specific circadian transcriptome in male mice. bioRxiv. 42 (1), 111982 (2023).
- Wilson, D., Breen, L., Lord, J. M., Sapey, E. The challenges of muscle biopsy in a community based geriatric population. BMC Research Notes. 11 (1), 830 (2018).
- Ding, H., et al. Likelihood-based tests for detecting circadian rhythmicity and differential circadian patterns in transcriptomic applications. Briefings in Bioinformatics. 22 (6), 224 (2021).
- Ding, Z., Lamb, T. M., Boukhris, A., Porter, R., Bell-Pedersen, D. Circadian clock control of translation initiation factor eIF2α activity requires eIF2γ-dependent recruitment of rhythmic PPP-1 phosphatase in Neurospora crassa. mBio. 12 (3), e00871 (2021).
- Di Francesco, A., Di Germanio, C., Bernier, M., de Cabo, R. A time to fast. Science. 362 (6416), 770-775 (2018).
- Kalfalah, F., et al. Crosstalk of clock gene expression and autophagy in aging. Aging. 8 (9), 1876-1895 (2016).
- Psaty, B. M., et al. Assessing the use of medications in the elderly: methods and initial experience in the cardiovascular health study. Journal of Clinical Epidemiology. 45 (6), 683-692 (1992).
- van Moorsel, D., et al. Demonstration of a day-night rhythm in human skeletal muscle oxidative capacity. Molecular Metabolism. 5 (8), 635-645 (2016).
- Janssen, J. J. E., et al. Extracellular flux analyses reveal differences in mitochondrial PBMC metabolism between high-fit and low-fit females. American Journal of Physiology. Endocrinology and Metabolism. 322 (2), E141-E153 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유