JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
글루칸 포스파타제의 기질 결합을 측정하기 위한 Concanavalin A 기반 침강 분석
요약
이 방법은 글루칸 포스파타제와 아밀로펙틴의 결합 친화도를 정량화하기 위한 렉틴 기반 시험관 내 침강 분석을 설명합니다. 이 공동 침강 분석은 글루칸 포스파타제 기질 결합을 측정하는 데 신뢰할 수 있으며 다양한 가용화된 글루칸 기질에 적용할 수 있습니다.
초록
글루칸 포스파타제는 동물의 글리코겐 및 식물의 전분과 같은 글루칸 기질을 탈인산화하는 이중 특이성 포스파타제(DSP)의 더 큰 계열에 속합니다. 모델 글루칸 기질을 가진 글루칸 포스파타제의 결정 구조는 DSP와 탄수화물 결합 도메인으로 만들어진 뚜렷한 글루칸 결합 계면을 나타냅니다. 그러나 생리학적으로 관련된 기질과의 글루칸-글루칸 포스파타제 상호작용의 정량적 측정은 글루칸 포스파타제 효소 계열의 생물학적 이해와 에너지 대사 조절의 기본입니다. 이 원고는 다른 글루칸 기질에 대한 글루칸 포스파타제의 기질 결합 친화도를 검출하도록 설계된 Concanavalin A(ConA) 기반 시험관 내 침강 분석을 보고합니다. 개념 증명으로서, 글루칸 포스파타제 Arabidopsis thaliana Starch Excess4 (SEX4) 및 아밀로펙틴의 해리 상수(KD)를 측정하였다. SEX4 돌연변이 및 글루칸 포스파타제 효소 계열의 다른 구성원의 특성화는 단백질-탄수화물 상호 작용의 차등 결합을 평가하기 위한 이 분석의 유용성을 추가로 입증합니다. 이러한 데이터는 광범위한 전분 및 글리코겐 상호작용 단백질을 특성화하기 위한 이 분석의 적합성을 입증합니다.
서문
글루칸 포스파타제는 단백질 티로신 포스파타제(PTP) 슈퍼패밀리1 내에서 이중 특이성 포스파타제(DSP)의 기능적으로 다양한 서브패밀리의 구성원입니다. 그들은 광범위하게 발산하는 광합성 유기체, 인간, 척추 동물, 일부 무척추 동물 및 원생 생물을 포함한 대부분의 생명체에서 발견되었습니다 2,3,4. 식물에는 전분 과잉4 (SEX4), 성 4 (LSF1) 및 성 4 (LSF2) 5,6,7과 같은 세 가지 알려진 글루칸 포스파타제가 포함되어 있습니다. 글루칸 포스파타제가 결핍된 식물은 일시적인 전분 분해 및 잎의 전분 축적 비율이 감소합니다 8,9. Laforin은 척추 동물과 인간의 글리코겐을 탈인산화하는 글루칸 포스파타제 계열의 창립 멤버입니다 3,10. 라포린의 돌연변이는 신경퇴행성 라포라병을 유발하는데, 이는 치명적인 상염색체 열성 형태의 간질이다11. 글루칸 포스파타제는 글리코겐과 전분 대사에 필요하며 식물의 전분 함량을 조절하고 신경퇴행성 라포라병을 치료하는 중요한 효소로 부상했습니다12,13. 모델 글루칸 기질을 가진 글루칸 포스파타제에 대한 최근의 X선 결정학 연구는 기질 결합 및 글루칸 탈인산화의 촉매 메커니즘에 대해 밝혔습니다14,15,16,17. 그러나 글루칸 포스파타제가 생리학적 기질에 어떻게 결합하는지에 대한 현재의 이해는 불완전합니다.
전분은 80%-90% 아밀로펙틴과 10%-20% 아밀로오스로 만들어진 포도당의 불용성 중합체입니다18. 식물 글루칸 포스파타제의 기질은 글리코겐 및 전분 과립과 같은 인산화된 탄수화물 분자입니다. 인산화된 글루코실 잔기는 1:600 포스페이트:글루코실 잔기 비율로 존재한다. 흥미롭게도, 인산염은 아밀로펙틴 분자에만 존재한다19. 주요 식물 글루칸 포스파타제 SEX4는 전분 과립에 작용하여 아밀로펙틴 분자를 탈인산화합니다. 구조 유도 돌연변이 유발 연구와 결합된 SEX4의 X선 결정 구조는 글루칸 구조 내의 다양한 위치에 대한 SEX4의 고유한 기질 특이성을 입증했습니다15. 우리는 최근에 SEX4의 생물학적으로 관련된 활성이 가용화된 아밀로펙틴 기질에 작용할 때만 관찰될 수 있음을 보여주었습니다20. 그러나 글루칸-SEX4 상호작용을 이해하는 것은 기질의 구조적 복잡성, 더 넓은 결합 특이성, 단백질과 그 기질 사이의 낮은 결합 친화도로 인해 어려운 것으로 입증되었습니다. 이러한 문제는 등온 적정 열량측정법(ITC), 핵자기 공명(NMR) 분광법 및 효소 결합 면역흡착 분석법(ELISA) 기반 분석과 같은 단백질-리간드 상호작용에 일반적으로 사용되는 방법을 활용하는 능력을 방해했습니다.
흥미롭게도 탄수화물-단백질 상호 작용에 대한 우리의 이해의 대부분은 렉틴을 연구하는 데서 비롯되었습니다. 콘카나발린 A(ConA)는 원래 잭빈에서 추출한 콩과 식물 렉틴 계열의 단백질입니다. ConA는 높은 특이성으로 탄수화물을 결합하므로 약물 표적화 및 전달 응용 분야에 사용하기에 유리합니다. 비환원성 α-D-만노실 및 α-D-글루코실을 함유하는 다양한 기질에 대한 ConA의 결합이 광범위하게 연구되었다19,20. 상업적으로 이용 가능한 ConA-결합 세파로스 비드는 일반적으로 당단백질과 당지질을 정제하는 데 사용된다21. ConA는 포도당 잔기의 C3, C4 및 C6 하이드록실 그룹을 통해 이러한 글루칸에 결합합니다. ConA-Sepharose 비드는 또한 글리코겐-단백질 및 전분-단백질 상호작용의 결합을 측정하는 데 성공적으로 사용되었습니다22,23. 이 연구에서 우리는 ConA-Sepharose 비드를 사용하여 글루칸 포스파타제-아밀로펙틴 상호작용의 결합 특이성을 측정하기 위한 결합 분석을 개발했습니다.
이전에는 글루칸 포스파타제 기질 결합 능력 14,20,24를 평가하기 위해 ConA 기반 침강 분석이 사용되었습니다. 이 연구에서는 글루칸-글루칸 포스파타제와 탄수화물 상호작용의 결합 친화도를 결정하는 새로운 방법을 개발하는 데 동일한 전략이 사용되었습니다. 이 방법은 또한 다양한 가용화 탄수화물-단백질 상호 작용을 조사하는 데 이점이 있습니다.
프로토콜
1. ConA-Sepharose 비드의 제조
- 67mM HEPES(pH 7.5), 10mM MgCl2 및 0.2mMCaCl2를 포함하는 결합 완충액 250mL를 만듭니다. 1 M NaOH 용액을 사용하여 pH를 조절한다.
- ConA-Sepharose 비드 현탁액 250μL를 1.5mL 미세원심분리기 튜브에 피펫팅합니다. 내용물을 10,000 x g 에서 4°C에서 30초 동안 원심분리합니다. 상청액을 버리십시오.
참고: 분석에 사용된 각 아밀로펙틴 농도에 대해 250mL 미세 원심분리기 튜브에 1.5μL의 ConA-Sepharose 비드가 필요합니다. - 750 μL의 결합 완충액을 250 μL의 ConA-Sepharose 비드가 들어 있는 각 튜브에 추가합니다. 튜브를 10,000 x g 에서 4°C에서 1분 동안 원심분리합니다. 상청액을 제거합니다. 이 단계를 2x 반복하여 비드가 적절하게 세척되고 결합 완충액과 평형을 이루도록 합니다.
2. 아밀로펙틴 용액의 제조
- 10mg/mL 감자 아밀로펙틴의 원액을 만드십시오. 아밀로펙틴은 수 불용성이며 열에 의해 용해 될 수 있습니다. 가용화하려면 감자 아밀로펙틴 0.1g을 증류수 10mL에 첨가한다. 현탁액을 80°C의 수조에서 1시간 동안 또는 용액이 더 이상 탁해지지 않을 때까지 가열합니다.
- 용액이 뭉치는 것을 방지하기 위해 반복적인 볼텍싱과 함께 실온(RT)으로 돌아가도록 합니다.
- 알코올-알칼리성 처리는 아밀로펙틴 기질을 가용화하는 대체 방법입니다. 이 방법을 사용하여 가용화하려면 아래 단계를 따르십시오.
- 0.5g의 아밀로펙틴 기질을 5mL의 20% 에탄올과 5mL의 2M NaOH에 현탁합니다. RT에서 15-20 분 동안 내용물을 세게 저어줍니다.
- 다음으로, 물 10 mL를 첨가하고, 2 M HCl을 첨가하여 용액의 pH를 6.5로 조정한다. 증류수로 결과 용액의 부피를 50mL로 올려 10mg/mL 아밀로펙틴 용액을 만든다.
- 10mg/mL 가용화된 아밀로펙틴 용액을 희석하여 일련의 2mL의 희석된 아밀로펙틴 용액을 만듭니다. 예를 들어, 10mg/mL의 반감액을 수행하여 일련의 아밀로펙틴 농도(5mg/mL, 2.5mg/mL, 1.25mg/mL, 0.625mg/mL, 0.3125mg/mL, 0.156mg/mL, 0.078mg/mL, 0.039mg/mL, 0.019mg/mL 및 0mg/mL)를 준비합니다.
3. ConA 세파로스의 제조 : 아밀로펙틴 비드
- 각 희석된 아밀로펙틴 용액 250μL를 결합 완충액에 미리 평형화된 ConA-Sepharose 비드 250μL가 들어 있는 1.5mL 미세원심분리기 튜브에 추가합니다. 내용물을 잘 섞는다. 해당 아밀로펙틴 농도로 튜브에 라벨을 붙입니다.
- 4°C에서 30분 동안 회전하는 휠에서 내용물을 배양합니다.
참고: ConA-Sepharose:amylopectin 결합 복합체는 20분 후 시간이 지남에 따라 변화가 없습니다. 평형에 도달했는지 확인하기 위해 배양 시간을 10분에서 1시간까지 다양하게 하여 30분 배양 시간을 선택했습니다. - 튜브를 10,000 x g 에서 1분 동안 원심분리합니다. 새로 라벨이 붙은 1.5mL 미세 원심분리기 튜브에 상층액을 수집합니다. 이들 상청액 분획을 저장하여 D-글루코스 분석12 (아밀로펙틴의 산 가수분해에 이어 효소 분석을 통한 글루코스의 UV 측정)를 수행합니다. 이 단계는 모든 아밀로펙틴이 비드에 결합되도록 하는 데 필요합니다.
- 750 μL의 결합 완충액을 ConA-Sepharose:amylopectin beads에 첨가합니다. 튜브를 10,000 x g 에서 1분 동안 원심분리합니다. 결합되지 않은 아밀로펙틴 분자를 제거하기 위해 상청액을 버립니다.
- 충분한 세척을 위해 3.4단계를 반복합니다. 각 튜브에는 이제 다양한 양의 아밀로펙틴 기질에 결합된 ConA-Sepharose 비드가 포함되어 있습니다.
4. ConA-Sepharose와 SEX4 배양 : 아밀로펙틴 비드
- 250μL의 ConA-Sepharose:amylopectin 비드를 10μg의 SEX4 단백질, 10mM 디티오트레이톨(DTT) 및 10μM 프로테아제 억제제 칵테일(PIC)을 포함하는 결합 완충액 100μL와 혼합합니다. 각 튜브의 총 부피는 350μL입니다.
참고: 프로테아제 억제제 칵테일은 불필요한 SEX4 분해를 방지하기 위한 예방 단계로 추가됩니다. 이 단계는 선택 사항입니다. 이 분석에서는 재조합 단백질인 Arabidopsis thaliana SEX4(AtSEX4)를 사용합니다. 정제된 단백질은 화학발광을 통해 단백질을 검출하는 데 필요한 N-말단 히스티딘 태그를 함유하고 있습니다. 글루칸 포스파타제 정제에 대한 상세한 정보는 이전 간행물 14,20,24에 기재되어 있다. - 단백질과 ConA-Sepharose:amylopectin 비드 현탁액을 4°C에서 45분 동안 부드럽게 회전시키면서 인큐베이션합니다.
참고: 복합체의 평형에 도달하기 위해 45분의 배양 시간이 선택됩니다. - 튜브를 10,000 x g 에서 1분 동안 원심분리합니다. 겔 로딩 팁을 사용하여 상층액 50μL를 새로운 1.5mL 미세원심분리 튜브에 조심스럽게 피펫팅합니다. 수집된 상층액 분획 50μL가 들어 있는 각 튜브에 4x SDS-PAGE 염료 20μL와 물 10μL를 추가합니다. 샘플을 95°C에서 10분 동안 가열합니다. SDS-PAGE 젤을 실행하기 위해 이러한 샘플을 저장합니다. "상층액(S)"이라고 표시된 10개의 새 튜브에 해당 기질 농도가 있는지 확인합니다.
- 750μL의 결합 완충액을 ConA-Sepharose:amylopectin:SEX4 비드에 추가하여 비드에서 결합되지 않은 단백질을 제거합니다. 튜브를 10,000 x g 에서 1분 동안 원심분리합니다. 적절한 세척을 위해 이 단계를 한 번 더 반복합니다. 상청액을 버리십시오.
- 세척된 ConA-Sepharose:amylopectin:SEX4 비드가 들어 있는 튜브에 4x SDS-PAGE 염료 20μL와 증류수 80μL를 추가합니다. 샘플을 95°C에서 10분 동안 가열하고 10,000 x g 에서 1분 동안 원심분리합니다.
- 펠릿을 버리고 SDS-PAGE 젤을 실행하기 위해 상청액을 저장합니다. 상층액 80μL를 새 튜브에 피펫으로 넣고 "펠릿(P)"으로 표시합니다.
5. SDS-PAGE 젤 실행
- 결합되지 않은 단백질 샘플 40μL(2.3단계에서 제조, 표지된 S)를 가장 낮은 기질 농도에서 가장 높은 농도까지 4%-12% 프리캐스트 폴리아크릴아미드 겔 웰에 로드하되, 단백질 분자량 마커를 로드하기 위해 첫 번째 레인을 자유롭게 유지합니다. 두 번째 젤을 사용하여 2.5단계에서 만든 10개의 결합된 단백질 샘플(P로 표시됨)을 로드합니다.
- 새로 준비된 1x SDS-PAGE 실행 버퍼를 장치의 두 챔버에 추가합니다. 150V에서 35분 동안 또는 염료 전면이 젤 바닥에 도달할 때까지 젤을 실행합니다.
- 장치에서 런 젤을 제거하고 스페이서와 유리판을 제거합니다. 분리된 겔을 사용하여 웨스턴 블롯 분석을 실행합니다.
6. 화학발광 검출을 위한 웨스턴 블로팅14,15
참고: 이 방법은 사용자가 실험실에 가지고 있는 웨스턴 블로팅 장비에 따라 쉽게 수정/조정할 수 있습니다.
- 트리스 염기 5.8g, 글리신 2.9g, SDS 0.37g, 메탄올 200mL를 포함하는 전사 완충액 1L를 만듭니다.
- 크기가 분리된 단백질을 폴리아크릴아미드 겔에서 니트로셀룰로오스 막으로 옮깁니다. 스폰지, 여과지, 겔 및 니트로셀룰로오스 멤브레인을 웨스턴 전달 프로토콜14,15에 따라 간략하게 조립합니다. 70V에서 1시간 동안 작동합니다.
- 비특이적 단백질 결합을 방지하기 위해 1%-5% 소 혈청 알부민(BSA) 또는 우유 단백질의 단백질 용액이 포함된 니트로셀룰로오스 막을 50mL의 TBST 완충액(20mM Tris[pH 7.5], 150mM NaCl, 0.1% Tween 20)에 1시간 동안 배양합니다. TBST 버퍼를 사용하여 멤브레인을 3배 세척하여 결합되지 않은 차단 용액을 제거합니다.
- 양고추냉이 과산화효소(HRP) 결합 항체와 함께 1시간 동안 His-tagged 단백질을 특이적으로 배양합니다. 멤브레인을 TBST 완충액으로 3x 세척하여 결합되지 않은 항체를 제거합니다. 최적의 재현성과 감도를 위해 항체를 TBST에 1:2,000 희석하여 사용합니다.
- HRP 효소 결합 항체는 SEX4 단백질의 히스티딘 태그에 특이적으로 결합하여 화학발광 시약이 있는 상태에서 밴드를 생성합니다. 디지털 이미징의 경우 1.5mL 튜브에 화학발광 기질 용액(각각 750μL)을 동일한 비율로 혼합한 용액을 만듭니다. 용액에서 적어도 5분 동안 멤브레인을 배양합니다.
- 멤브레인 단백질 면이 아래로 향하도록 블롯 스캐너에 놓고 수집 소프트웨어를 실행하여 펠릿 및 상청액 분획 모두에서 단백질을 정량화합니다.
7. 데이터 분석
- 블롯 스캐너와 함께 수집 소프트웨어를 사용하여 정량적 신호 측정을 수행합니다. 상층액 및 펠릿 분획의 모든 정량적 측정을 로드된 총 단백질로 정규화합니다.
참고: 이 소프트웨어를 사용하면 상청액 및 펠릿 분획에서 각 단백질 밴드의 강도를 정량화할 수 있습니다. - 포화 결합 실험에서 단백질 결합 대 아밀로펙틴 농도의 백분율을 표시합니다. 데이터 분석 소프트웨어를사용하여 데이터를 Y = Bmax x X/(KD + X)에 피팅하고 KD를 계산합니다.
참고: Bmax는 최대 특이 결합, Y축은 백분율입니다.tage 결합된 단백질, X축은 아밀로펙틴 농도입니다.
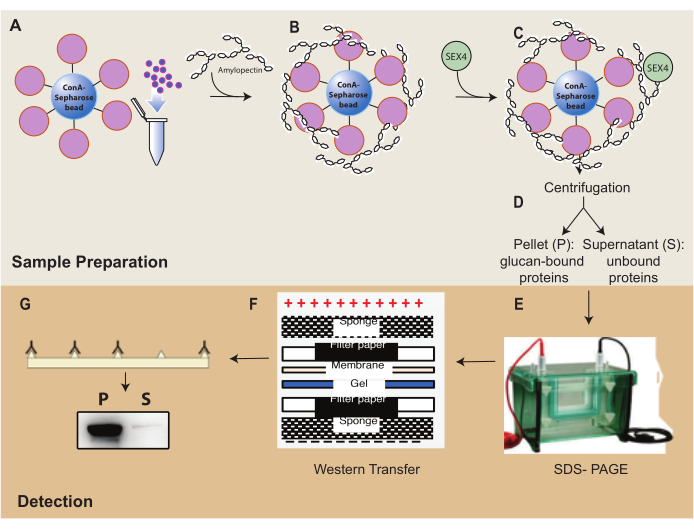
그림 1: ConA-Sepharose 침강 분석 워크플로우 개요. (a) ConA-Sepharose 비드의 제조. (B) 아밀로펙틴 기질을 사용한 배양. (C) SEX4 단백질로 배양. (D) 원심분리를 통한 결합 및 결합되지 않은 단백질 분획의 분리. (e) SDS-PAGE를 통한 단백질 분리. (F) 웨스턴 블롯 분석. (g) His-tagged SEX4 단백질의 화학발광 검출. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
글루칸 포스파타제 단백질 계열의 주요 특징 중 하나는 글루칸 기질에 결합하는 능력입니다. 먼저, ConA-Sepharose:amylopectin beads에 대한 SEX4의 결합능을 SDS-PAGE를 이용하여 분석하였다(도 2A). 소 혈청 알부민(BSA)은 ConA-Sepharose:amylopectin 비드에 대한 단백질의 비특이적 결합을 검출하기 위한 음성 대조군 역할을 했습니다. 단백질의 SDS-PAGE 분석은 펠렛 분획에서 SEX4 단백질의 존재와 ...
토론
이 연구는 글루칸-글루칸 포스파타제 상호작용의 결합 친화도를 측정할 수 있는 새로운 시험관 내 침강 분석의 성공적인 개발을 보여줍니다. 분석 설계는 포도당의 하이드록실 잔기를 통해 글루칸에 대한 렉틴 ConA의 특이적 결합을 활용하여 가용화된 탄수화물 기질을 세파로스 비드에 간접적으로 포획합니다. 이를 통해 원심분리를 통해 결합된 단백질 분획과 결합되지 않은 ?...
공개
저자는 이해 상충을 선언하지 않습니다.
감사의 말
이 연구는 국립 과학 재단 상 MCB-2012074의 지원을 받았습니다. 저자는 귀중한 토론과 지원을 해준 플로리다 대학교 생화학 및 분자 생물학과의 Craig W. Vander Kooi 박사에게 감사드립니다. 저자는 또한 플로리다 대학교 생화학 및 분자 생물학과의 Matthew S. Gentry 박사에게 그의 지원에 감사드립니다. 웨스턴 블롯 이미징을 위해 LICOR C-digit 블롯 스캐너를 사용할 수 있도록 허락해 주신 Skidmore College Neuroscience 프로그램의 의장인 Sara Lagalwar 박사에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
참고문헌
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유