JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
RPA-CRISPR/Cas12a-SPM 및 딥러닝 기반의 DNA 바이러스 검출 시스템
요약
DNA 바이러스의 미량 검출을 위한 CRISPR/Cas12a 시스템과 재조합 중합효소 증폭을 결합한 프로토콜을 제시하고, 현장 진단 DNA 바이러스 검출을 위한 인공 지능 지원 분류를 통해 휴대용 스마트폰 현미경을 구축합니다.
초록
당사는 DNA 바이러스의 미량 검출을 위해 재조합 중합효소 증폭(RPA)과 CRISPR/Cas12a 시스템을 결합한 빠르고 구현하기 쉬우며 감도가 높은 염기서열 특이적 POC(point-of-care) DNA 바이러스 검출 시스템을 보고합니다. 표적 DNA는 RPA와 CRISPR/Cas12a에 의해 별도로 증폭 및 인식되며, 이는 형광단 소광체 표지된 DNA 리포터를 절단하고 형광을 일반화하는 Cas12a의 부수적 절단 활성을 유발합니다. POC 검출을 위해 휴대용 스마트폰 현미경은 형광 이미지를 촬영하도록 제작되었습니다. 또한, 높은 정확도를 달성하는 양성 또는 음성 샘플의 이진 분류를 위한 딥 러닝 모델이 시스템 내에 배포됩니다. 이 DNA 바이러스 POC 검출 시스템의 예로 개구리 바이러스 3(FV3, Ranavirus 속, Iridoviridae과)를 테스트하였으며, 검출 한계(LoD)는 40분 이내에 10aM에 도달할 수 있습니다. 숙련된 작업자와 부피가 큰 기기가 필요 없는 휴대용 소형 RPA-CRISPR/Cas12a-SPM은 인공 지능(AI) 지원 분류 기능을 통해 POC DNA 바이러스 검출에 큰 잠재력을 보여주며 이러한 바이러스의 확산을 방지하는 데 도움이 될 수 있습니다.
서문
최근 몇 년 동안 2014년 에볼라 바이러스 질환(EVD) 유행1 및 20182, 2015년 중동호흡기증후군(MERS)3, 2015년 지카 바이러스 질병 유행4, 중증 급성 호흡기 증후군 코로나바이러스 2(SARS-CoV-2)5로 인한 코로나바이러스 감염증 2019(COVID-19), 2022년 원숭이두창 바이러스(MKPV)에 의한 원숭이두창 6 등 다양한 바이러스에 의한 전염병의 유행이 빈번하게 발생했습니다을 클릭합니다. 이러한 전염병의 갑작스런 발생은 많은 사망자를 발생시키고 막대한 경제적 손실과 사회적 불안을 초래합니다. 감염을 신속하게 진단하고 바이러스의 추가 확산을 방지하기 위해 신속하고 정확한 탐지 시스템이 시급히 필요합니다.
최근에는 CRISPR(clustered regular interspaced short palindromic repeats) 및 CRISPR-associated (Cas) 단백질이 전 세계적으로 주목을 받고 있으며 핵산 검출에서 유망한 결과를 보여주었습니다 7,8,9,10,11,12,13,14,15 . CRISPR RNA(crRNA)에 의해 유도되는 CRISPR/Cas12a 단백질은 표적 DNA에 결합하여 절단합니다. 이 활성은 trans-cleavage로 알려진 비특이적 단일 가닥 DNA(ssDNA)의 방출로 이어지며 핵산 검출을 위한 검출 신호를 향상시키는 데 사용할 수 있습니다. 중합효소연쇄반응(PCR), 정량적 Real-Time PCR(qPCR) 및 효소 결합 면역흡착 분석법(ELISA)과 같은 일부 기존 검출 방법은 현장 진료(POC) 검출에 복잡하고 시간이 많이 걸리며 비용이 많이 듭니다. 이전 연구에서는 CRISPR/Cas12a 기술을 기반으로 아프리카돼지열병(ASFV)에 대한 자동화되고 통합되며 비용 효율적인 검출 시스템을 성공적으로 개발했습니다. 이 시스템에서는 증폭 없이 2시간 내에 1pM의 검출 한계를 달성했습니다. CRISPR/Cas12a 시스템과 재조합 효소 중합효소 증폭(RPA)이 결합되어 미량 DNA 검출의 감도와 특이성을 향상시킵니다. 다른 등온 증폭 기술과 비교할 때 RPA는 정교한 온도 제어 장비 없이 반응 시간이 짧기 때문에 설계가 간단하고 작동이 편리합니다.
병원체의 POC 검출을 위해 스마트폰 현미경(SPM), 휴대용 형광측정기 또는 측면 플로우 스트립과 같은 기기가 결과 판독을 위해 개발되었습니다 16,17,18. SPM은 카메라를 통해 이미지를 캡처하고 빠른 데이터 분석을 위해 일부 모바일 애플리케이션에 업로드합니다. 이러한 현미경은 고감도의 휴대용, 저렴 및 소형 신호 획득 시스템을 만들고 H5N1, 지카 바이러스 및 SARS-CoV-219,20와 같은 병원체를 감지하는 데 이점을 보여주었습니다. 따라서 표적 DNA 바이러스의 RPA-CRISPR/Cas12a 검출에 의해 트리거되는 형광 신호를 포착할 수 있는 휴대용 SPM을 구축했습니다. 형광단과 소광체를 연결하는 ssDNA 리포터 프로브는 CRISPR/Cas12a가 타겟 DNA 바이러스를 인식하면 절단되며, 형광단에서 방출되는 형광은 SPM으로 캡처할 수 있습니다.
SPM21의 형광 이미지로부터 결과 정보를 얻는 데 일반적으로 사용되는 전문 소프트웨어와 비교하여, 일부 전문가들은 형광 이미지(22)를 얻은 후 기계 학습 및 딥 러닝을 사용하여 바이러스 DNA의 농도를 정량화하는데, 이는 더 많은 시간이 소요됩니다. 의료 이미지를 분류할 때, 종래의 신경망(CNN)은 종단 간 방식으로 원시 픽셀화된 이미지로부터 특징을 학습하는 데 자주 사용된다 23,24,25,26. AlexNet, DenseNet-121 및 EfficientNet-B7과 같은 인기 있는 CNN 기반 딥 러닝 모델이 이 분야에 성공적으로 적용되었습니다27,28. 그러나 특정 영역에서 대규모 데이터 세트를 얻는 것은 어려울 수 있으므로 전이 학습이 필요합니다29,30. 이 접근 방식은 대규모 데이터 세트로 딥 러닝 모델을 사전 학습시키며, 사전 학습된 모델은 작은 데이터 세트로 새 작업의 시작점으로 사용됩니다. 이 기법은 대규모 데이터 세트의 필요성을 줄이고, 과적합을 방지하며, 훈련 시간을 단축할 수 있다31. 여기에서는 positive 및 negative 샘플의 형광 이미지의 이진 분류를 위해 전이 학습과 함께 딥 러닝 모델을 사용합니다.
이 방법에서는 DNA 바이러스의 미량 검출을 위해 RPA와 CRISPR/Cas12a 시스템을 결합합니다. 표적 DNA는 RPA와 CRISPR/Cas12a에 의해 별도로 증폭 및 인식되며, 이는 형광단 소광체 표지된 DNA 리포터를 절단하고 형광을 일반화하는 Cas12a의 부수적 절단 활성을 유발합니다. POC 검출을 위한 형광 이미지를 촬영하고 이진 분류를 위한 딥 러닝 모델을 개발하기 위해 휴대용 SPM을 구축합니다. 구축된 POC 탐지 시스템의 회로도는 그림 1에 나와 있습니다. 숙련된 작업자와 부피가 큰 기기 없이도 인공 지능(AI) 지원 분류 기능을 갖춘 RPA-CRISPR/Cas12a-SPM은 POC DNA 바이러스 검출에 대한 큰 잠재력을 보여줍니다.
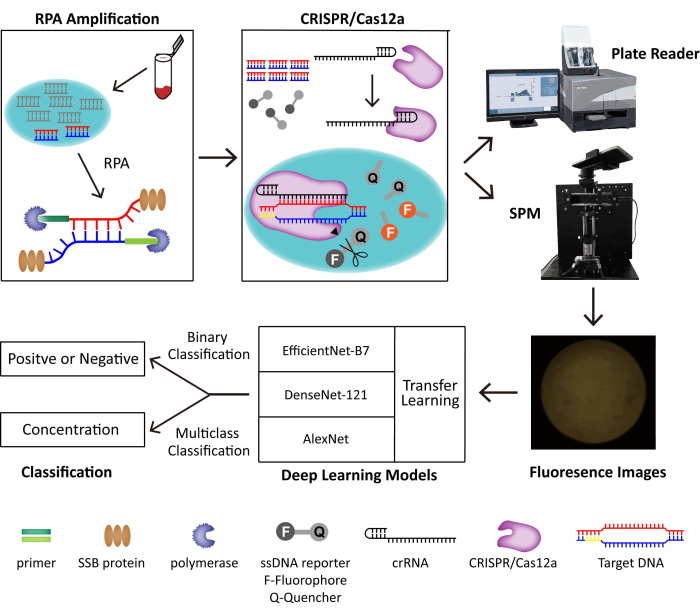
그림 1: 수집된 이미지에 대한 AI 분류와 함께 RPA-CRISPR/Cas12-SPM 검출 시스템의 회로도. 동물 유래 샘플의 핵산은 PINDBK에 의해 방출됩니다. 바이러스의 표적 DNA는 RPA-CRISPR/Cas12a 시스템에 의해 증폭되고 특이적으로 인식됩니다. CRISPR/Cas12a는 crRNA와 결합하고 Cas12a-crRNA 복합체는 표적 DNA와 결합하여 ssDNA 리포터 프로브에서 CRISPR/Cas12a의 부수적 절단을 유발합니다. 리포터의 형광단이 방출되고 형광은 상용화된 플레이트 리더 또는 당사가 제작한 SPM에 의해 감지됩니다. 전이 학습을 통한 AlexNet, DenseNet-121 및 EfficientNet-B7을 포함한 세 가지 딥 러닝 모델을 사용하여 형광 이미지를 분류합니다. 이 그림은 Lei et al.35의 허가를 받아 재사용되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 시료 처리
- 이중 가닥 DNA 바이러스인 개구리 바이러스 3(FV3, Ranavirus 속, Iridoviridae과)를 예로 들어 보겠습니다. FV3 검출을 위한 타겟으로 major capsid (mcp) 유전자를 선택하십시오. 이는 이 유전자가 보존성이 뛰어나고 일반적으로 라나바이러스 검출 타겟으로 간주되기 때문입니다. 선택된 타겟 시퀀스는 표 1에 나와 있습니다.
참고: Frog Virus 3은 이 프로토콜의 예로 사용됩니다. - 표적 DNA 절편 준비를 위해 FV3 및 감염성 비장 및 신장 괴사 바이러스(ISKNV, 또 다른 Ranavirus)의 mcp 유전자 DNA 절편을 사용합니다.
참고: 이 연구에서 표적 DNA 단편은 재료 표에 나열된 회사에서 상업적으로 획득했습니다. 그들은 후속 탐지의 대상 및 제어로 간주됩니다.
| 이름 | 순서 | ||
| FV3 엠피 | NTS: 5' ... gtaacccggctttcGGGCAGTTTCGGTCGGCGTtcccaggtcg... 3' (240 bp) | ||
| TS: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 bp) | |||
| 이스큰브 MCP | NTS: 5' ... ggccatgccaatttTGGGCAGGATTTAGTGTGACGgtggcgaggg... 3' (231 bp) | ||
| TS: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 bp) | |||
표 1: 이 방법에서 선택된 타겟 염기서열.
2. RPA 반응
- 타겟 염기서열에 대한 RPA 프라이머 쌍을 설계하고 합성합니다. RPA 프라이머 쌍의 시퀀스는 표 2에 설명되어 있습니다.
- 5x RPA 반응 완충액(표 3) 및 MgCl2(100mM)를 준비합니다.
- 표 4에 자세히 설명된 대로 4가지 주요 RPA 효소(UvsX, UvsY, GP32, Bsu 단백질)를 1x RPA 반응 완충액과 미리 설계된 프라이머를 혼합합니다.
- 혼합물을 철저히 소용돌이치십시오.
- 각 RPA 반응에 대해 1단계에서 얻은 타겟 1μL를 첨가하고 다시 와류로 철저히 혼합합니다.
- 7μL의 MgCl2 (100mM)를 추가하여 반응을 시작합니다. 각 RPA 반응의 최종 부피는 50μL입니다.
- 37°C에서 30분 동안 분석을 수행합니다.
- RPA 제품은 4°C에서 며칠 동안 보관할 수 있습니다. RPA 제품의 성능이 저하됨에 따라 더 나은 진단 결과를 얻기 위해 가능한 한 빨리 추가 탐지에 사용하십시오.
- [선택 사항] DNA 겔 전기영동을 수행합니다.
- 5 μL의 RPA 제품을 꺼내고, 적절한 부피의 6x DNA 로딩 버퍼를 추가하고, 트리스 아세테이트 EDTA(TAE) 버퍼에서 DNA 겔 전기영동을 수행합니다.
- 로딩 버퍼 밴드가 겔 바닥에 도달할 때까지 약 120V 아래에서 전기 영동을 약 20분 동안 실행합니다.
- 표적 염기서열이 샘플에서 성공적으로 증폭되었는지 여부를 확인하려면 샘플의 밴드 크기와 marker의 밴드 크기를 비교합니다.
| 이름 | 순서 |
| RPA 입문서 F | ATGTCTTCTGTaactggttcaggtatcaca |
| RPA 입문서 R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
표 2: 이 방법에 사용된 RPA 프라이머.
| 구성 요소 | 원래 농도 | 덧셈 |
| 나무못 20,000 | - | 114의 mg의 |
| ATP (영문) | 100 밀리미터 | 125 마이크로리터 |
| dNTPs (영문) | 25의 mM | 48 마이크로리터 |
| 트리스-HCl | 1개 M | 125 마이크로리터 |
| 디에이티(DTT) | 1개 M | 125 마이크로리터 |
| 포스포크레아틴 | 1개 M | 250 마이크로리터 |
| 크레아틴 키나아제 | 10 μg/μL | 50 μL |
| ddH2O | - | 277 마이크로리터 |
| 총 볼륨 | - | 1mL |
표 3: 5x RPA 반응 완충액(pH 7.5)의 조성.
| 구성 요소 | 원래 농도 | 덧셈 |
| 5x RPA 반응 버퍼 | - | 10 μL |
| UvsX 단백질 | 5mg/mL | 2.6 마이크로리터 |
| UvsY 단백질 | 5mg/mL | 0.9 μL |
| GP32 단백질 | 5mg/mL | 2.54 마이크로리터 |
| Bsu 단백질 | 5mg/mL | 0.88 마이크로리터 |
| 포워드 프라이머 | 100 마이크로미터 | 0.25 마이크로리터 |
| 반전된 뇌관 | 100 마이크로미터 | 0.25 마이크로리터 |
| ddH2O | - | 24.58 마이크로리터 |
| 과녁 | - | 1 μL |
| *마그네슘Cl2 | 100 밀리미터 | 7 μL |
| 총 볼륨 | 50 μL | |
| *RPA 반응을 시작하려면 MgCl2 를 마지막으로 추가해야 합니다. | ||
표 4: RPA 반응의 구성.
3. SPM 없이 CRISPR/Cas12a 검출
- CRISPR/Cas12a에 대한 형광단과 소광체를 연결하는 표적 염기서열의 crRNA(Table of Materials)와 ssDNA 리포터 프로브를 사용합니다. 여기서 카르복시 테트라메틸로다민(TAMRA)은 ssDNA 리포터 프로브의 5' 말단에서 형광단으로 연결되고 블랙홀 퀜처-2(BHQ2)는 3' 말단에서 소광체로 연결됩니다. crRNA 및 ssDNA reporter의 상세한 염기서열은 표 5에 기재되어 있다.
- 10x CRISPR/Cas12a 반응 완충액으로 Lachnospiraceae 박테리아 Cas12a(LbCas12a) 단백질을 준비합니다.
- 섹션 2의 RPA 반응 산물 1μL를 LbCas12a-crRNA 복합체 및 500nM ssDNA 리포터 프로브가 있는 1x CRISPR/Cas12a 반응 버퍼에 100μL 반응 부피에 용해시킵니다.
- LbCas12a와 crRNA를 혼합한 후 혼합물을 5분 이상 그대로 두어 기능성 복합체를 형성합니다. 배양 후 반응 혼합물에 다른 성분을 추가하고 37°C에서 전체 반응을 수행합니다. 각 CRISPR/Cas12a 반응의 최종 부피는 100μL입니다. 각 CRISPR/Cas12a 반응에서 각 성분의 자세한 농도는 표 6에 설명되어 있습니다.
- 37°C에서 30분 동안 100μL CRISPR/Cas12a 검출 반응을 수행합니다.
- 535 nm의 여기 파장과 595 nm의 방출 파장(60의 이득)에서 마이크로플레이트 리더를 통해 형광 신호를 지속적으로 검사합니다.
참고: 다양한 파장의 excitation 및 emission light 검출은 이전에 설계된 ssDNA 리포터 프로브에서 fluorophore 및 quencher를 선택하느냐에 따라 달라집니다. - 이렇게 수집된 데이터에 대해 제어 값을 양성 샘플의 측정값으로 나누어 모든 데이터를 정규화한 다음 2표본 t-검정 분석을 위해 적분합니다.
| 이름 | 순서 | |
| FV3에 대한 LbCas12a crRNA | 우아우우쿠아쿠아구과가우GGGCAGCAGTTTTCGGTCGGCGT | |
| ssDNA 리포터 | /5탐라/타트/3BHQ2 | |
표 5: 이 방법에 사용된 CRISPR/Cas12a crRNA 및 ssDNA 리포터의 염기서열.
| 구성 요소 | 원래 농도 | 덧셈 |
| 네버퍼 r2.1 | - | 10 μL |
| LBA Cas12a(CPF1) | 10 마이크로미터 | 0.5 μL |
| crRNA (영문) | 10 마이크로미터 | 0.625 마이크로리터 |
| LbCas12a/crRNA 복합체가 결합할 때까지 최소 5분 동안 기다립니다. | ||
| DNA 리포터 | 100 마이크로미터 | 0.5 μL |
| ddH2O | - | 87.375 마이크로리터 |
| 과녁 | - | 1 μL |
| 총 볼륨 | - | 100 μL |
표 6: CRISPR/Cas12a 반응의 조성.
4. SPM 설정
- excitation path의 경우, 레이저 강도를 감쇠시키기 위해 ND(Neutral Density) 필터를 통과하도록 레이저 빔을 설정합니다.
- aspherical lens(이하 L1)에서 collimated beam을 생성하고 dichroic mirror(DM)에 의해 반사합니다.
- 대물렌즈(20x)를 통해 샘플이 배치되는 유리 슬라이드에 빛을 비추어 샘플의 형광을 비추고 여기시킵니다. 샘플 스테이지는 정밀한 초점면 조정을 가능하게 하여 빔을 대물렌즈의 뒤쪽 초점면으로 향하게 합니다. 위에서 언급한 단계는 SPM 기기의 여기 경로를 형성합니다.
- 방출 경로의 경우 외부 렌즈(이하 L2라고 함)를 배치하여 대물렌즈의 반대쪽에 중간 이미지를 형성합니다. 대물렌즈는 샘플을 비추는 동시에 방출 신호를 수집합니다.
- 방출 경로 끝에 위치한 스마트폰을 사용하여 샘플의 형광 신호를 기록합니다. 흔들리지 않도록 안정된 브래킷을 사용하십시오.
- L1과 스마트폰 카메라 사이에 대역 통과 필터를 설정하여 여기된 빛을 필터링하고 샘플에서 방출된 빛만 카메라에 도달하도록 하여 감지를 최적화할 수 있습니다.
- 이동식 배포를 위해 브레드보드에 SPM 설정을 고정합니다.
참고: RPA-CRISPR/Cas12a 반응을 기반으로 한 형광 검출을 위한 SPM 장치의 회로도 및 물리적 외관은 그림 2에 나와 있습니다. CRISPR/Cas12a 검출은 다음 단계에서 설명하는 전처리된 유리 슬라이드에서 이루어집니다.
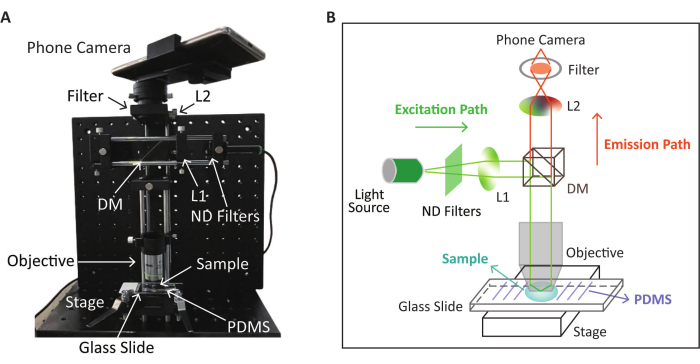
그림 2: 형광 검출에 사용되는 SPM 장치의 개략도 및 물리적 외관. (A) RPA-CRISPR/Cas12a 반응 후 형광 이미지 수집을 위한 SPM 장치의 물리적 외관. (B) RPA-CRISPR/Cas12a 반응을 기반으로 한 형광 검출을 위한 SPM 장치의 개략도. 이 그림은 Lei et al.35의 허가를 받아 수정(조정된 이미지 위치 및 색상)되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. SPM를 가진 탐지를 위한 유리제 활주의 처리
- 베이스와 경화제를 10:1의 비율로 혼합하여 폴리디메틸실록산(PDMS)을 준비한 다음 80°C의 핫플레이트에서 2시간 동안 굽습니다.
- PDMS와 유리 슬라이드를 모두 처리하십시오(길이: 75mm; 높이: 50mm)를 산소 플라즈마 처리로 120초 동안 누른 다음 함께 누릅니다.
- 유리/PDMS를 95°C에서 2시간 동안 굽습니다. 그것은 Si-O-Si 결합에 의해 영구적으로 밀봉됩니다. PDMS는 투명도가 높고 자동 형광이 없어 SPM 검출에 도움이 됩니다. 유리 슬라이드 및 PMDS의 처리는 He et al.32에 따라 수행됩니다.
6. SPM을 이용한 CRISPR/Cas12a 검출
- CRISPR/Cas12a에 대한 형광단과 소광체를 연결하는 표적 염기서열의 crRNA(Table of Materials)와 ssDNA 리포터 프로브를 사용합니다. 이 방법에서 카르복시 테트라메틸로다민(TAMRA)은 ssDNA 리포터 프로브의 5' 말단에서 형광단으로 연결되고 3' 말단에서 블랙홀 퀜처-2(BHQ2)로 연결됩니다. crRNA 및 ssDNA reporter의 상세한 염기서열은 표 5에 기재되어 있다.
- 10x CRISPR/Cas12a 반응 완충액으로 Lachnospiraceae 박테리아 Cas12a(LbCas12a) 단백질을 준비합니다.
- 섹션 2의 RPA 반응 산물 1μL를 LbCas12a-crRNA 복합체 및 500nM ssDNA 리포터 프로브가 있는 1x CRISPR/Cas12a 반응 버퍼에 100μL 반응 부피에 용해시킵니다.
- LbCas12a와 crRNA를 혼합한 후 혼합물을 5분 이상 그대로 두어 기능성 복합체를 형성합니다. 배양 후 반응 혼합물에 다른 성분을 추가하고 37°C에서 반응을 완료합니다. 각 CRISPR/Cas12a 반응의 최종 부피는 100μL입니다. 각 CRISPR/Cas12a 반응에서 각 성분의 자세한 농도는 표 6에 설명되어 있습니다.
- 전처리된 유리 슬라이드에서 100μL CRISPR/Cas12a 검출 반응을 수행하고 커버슬립으로 덮습니다. 유리 슬라이드를 실온에서 반응하여 10분 동안 배양합니다.
- SPM으로 형광 신호를 측정합니다. 검출 반응이 있는 유리 슬라이드를 SPM의 스테이지에 놓고 적절한 거리를 유지하고 초점 거리와 선명도를 조정한 다음 반응의 시야를 찾아 초점을 맞춰 이미지를 캡처합니다.
알림: 샘플의 데이터를 대략적인 농도 범위로 조정할 수 있도록 먼저 표준 곡선을 얻어야 합니다. 10 nM, 1 nM, 100 pM, 10 pM, 또 다른 바이러스인 10 pM을 포함하여 다양한 농도의 정제된 표적이 사용됩니다(RPA 이전).
7. 데이터 세트 및 데이터 증강
- 섹션 6의 검출 분석에서 형광 이미지를 데이터 세트로 수집합니다. 데이터 병렬 처리를 보장하기 위해 각 샘플에 대해 최소 3개의 병렬 감지를 반복합니다.
- 더 높은 병렬 처리를 달성하기 위한 몇 가지 적절한 방법을 승인할 수 있습니다. 예를 들어, 각 샘플에서 형광 이미지를 수집할 때 수동으로 초점을 맞추고 상대적으로 더 밝은 필드를 찾아 이미지를 촬영할 수 있습니다. 동시에 각 샘플을 촬영하여 5개 이상의 다른 위치에서 형광 신호를 얻을 수 있습니다.
- 각 이미지의 평균 회색 값과 ImageJ로 농도 그룹의 평균 회색 값의 표준 편차를 측정합니다.
- 데이터 정리를 위한 강도 범위[중앙값 - 표준 편차, 중앙값 + 표준 편차]를 설정합니다.
참고: 초기 단계에서 얻은 이미지에는 큰 차이가 있는 이미지가 있을 수 있으므로 이미지를 선별해야 합니다. 이미지의 농도가 설정된 임계값을 벗어나면 이상값으로 간주되어 제외되어야 합니다. - 농도가 증가하는 정제된 대상에 대한 이미지를 각각 오름차순으로 0-6으로 레이블을 지정합니다.
- 시스템의 견고성을 향상시키고 과잉 맞춤을 방지하려면 Python에서 transform 함수를 통해 수평 뒤집기, 수직 뒤집기, 랜덤 노이즈와 같은 이미지 증강 기술을 구현하십시오. 이는 데이터 세트에 변형을 도입하는 데 도움이 됩니다.
8. 전이 학습
- 백본 네트워크로 분류를 위해 딥러닝 모델 AlexNet33 을 채택합니다.
- 섹션 7에서 사전 훈련된 모델의 제약 조건을 충족하려면 Python의 변환 함수를 통해 입력 이미지를 224픽셀 x 224픽셀 x 3채널(높이와 너비 224픽셀, 빨강, 녹색 및 파랑 채널 깊이 3채널)로 모양을 변경합니다.
참고: 이 단계는 transform을 포함하여 전이 학습에서 이기종 데이터에 대한 일반적인 전처리입니다. - ImageNet 데이터 세트와 함께 사전 훈련된 백본 네트워크를 사용하여 학습된 중간 숨겨진 계층의 가중치를 활용하면서 기능을 추출합니다.
- 형광 분류 작업 컨텍스트에서 원래 ImageNet 작업을 위해 1,000개의 뉴런을 포함했던 신경망의 최종 완전 연결 계층을 2개 또는 7개의 뉴런이 있는 완전 연결 계층으로 대체합니다.
- 혼동 행렬, 정확도, 정밀도, 재현율 및 Lawton 및 Viriri34를 기반으로 하는 F1 점수를 포함한 일련의 메트릭을 사용하여 설정 학습 모델의 성능을 평가합니다.
결과
이 방법은 DNA 바이러스에 대한 빠르고 구현하기 쉬우며 감도가 높은 POC(point-of-care) 검출 시스템에 중점을 둡니다. RPA 반응을 위한 프라이머 쌍 설계와 CRISPR/Cas12a 반응을 위한 crRNA 설계는 RPA-CRISPR/Cas12a 반응의 효율성에 영향을 미치고 후속 검출 및 분류에 영향을 미치기 때문에 필수적인 두 가지 부분입니다.
이 방법에서 FV3는 DNA 바이러스 검출의 한 예로 간주?...
토론
이 방법에서는 AI 지원을 받아 빠르고 구현하기 쉬우며 감도가 높은 염기서열 특이적 POC DNA 바이러스 검출 시스템을 개발합니다. 검체를 채취한 후 RPA를 적용하여 표적 염기서열을 증폭하면 CRISPR/Cas12a가 표적 DNA를 인식하고 형광을 방출하여 검출 신호를 확대할 수 있습니다. 휴대용 스마트폰 현미경은 형광 이미지를 촬영할 수 있도록 제작되었으며, 전이 학습을 통한 딥 러닝 모델은 양성 및 음성 ...
공개
저자는 밝힐 것이 없습니다.
감사의 말
이 작업은 중국 국립 자연 과학 재단 31970752, 과학, 기술, 선전시 혁신 위원회(JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001)의 지원을 받습니다. 심천 베이 연구소 공개 자금, SZBL2020090501004; 중국 박사후 연구원 과학 재단 2020M680023; 및 중화인민공화국 해관 총국 2021HK007.
자료
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
참고문헌
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유