JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
TACI: 3D 칼슘 이미징 분석을 위한 ImageJ 플러그인
요약
TrackMate Analysis of Calcium Imaging(TACI)은 z축의 움직임을 검사하고 각 z-스택의 최대값을 식별하여 해당 시점에서 세포의 강도를 나타내는 3D 칼슘 이미징 분석을 위한 오픈 소스 ImageJ 플러그인입니다. 측면(x/y) 방향으로 겹치지만 다른 z-평면에서 겹치는 뉴런을 분리할 수 있습니다.
초록
신경과학 연구는 복잡한 이미징 및 계산 도구를 사용하여 데이터 세트에서 포괄적인 정보를 추출하도록 발전했습니다. 칼슘 이미징은 신뢰할 수 있는 결과를 얻기 위해 정교한 소프트웨어가 필요한 널리 사용되는 기술이지만 많은 실험실에서 최신 표준을 충족하도록 프로토콜을 업데이트할 때 계산 방법을 채택하는 데 어려움을 겪고 있습니다. 프로그래밍 지식과 소프트웨어에 대한 페이월의 부족으로 인해 어려움이 발생합니다. 또한 관심 세포는 칼슘 이미징 중에 모든 방향으로 움직임을 나타냅니다. 측면(x/y) 방향의 모션을 수정하기 위해 많은 접근 방식이 개발되었습니다.
이 백서에서는 새로운 ImageJ 플러그인인 TACI(TrackMate Analysis of Calcium Imaging)를 사용하여 3D 칼슘 이미징에서 z축의 모션을 검사하는 워크플로우에 대해 설명합니다. 이 소프트웨어는 뉴런이 나타나는 모든 z-위치에서 최대 형광 값을 식별하고 이를 사용하여 해당 t-위치에서 뉴런의 강도를 나타냅니다. 따라서 이 도구는 측면(x/y) 방향으로 겹치지만 별개의 z-평면에 나타나는 뉴런을 분리할 수 있습니다. ImageJ 플러그인인 TACI는 3D 칼슘 이미징 분석을 위한 사용자 친화적인 오픈 소스 계산 도구입니다. 우리는 온도 변동 동안 모든 방향으로의 움직임을 표시하는 파리 유충 온도에 민감한 뉴런과 파리 뇌에서 얻은 3D 칼슘 이미징 데이터 세트를 사용하여 이 워크플로를 검증했습니다.
서문
세포 내 칼슘 수치는 신경 흥분성의 정확한 지표입니다. 칼슘 이미징은 세포 내 칼슘의 변화를 측정하여 신경 활동을 이해합니다1. 신경과학 연구에서는 유전적 접근을 통해 특정 뉴런 세트에서 비침습적으로 발현될 수 있는 GCaMP 2,3과 같은 유전적으로 암호화된 칼슘 지표(GECI)를 포함하여 세포 내 칼슘 농도를 측정하는 기술의 개발로 인해 이 방법을 점점 더 많이 사용하고 있습니다. 레이저와 현미경 부품의 비용 절감은 칼슘 이미징의 사용도 증가시켰다4. 중요한 것은 칼슘 이미징을 통해 자유롭게 움직이는 동물에서 단일 뉴런과 큰 뉴런 집단을 동시에 기록하고 연구할 수 있다는것입니다 5.
그럼에도 불구하고, 칼슘 이미징 데이터의 분석은 (1) 시간 경과에 따른 개별 세포의 형광 변화를 추적하는 것을 포함하고, (2) 형광 신호가 간헐적으로 사라지거나 뉴런 반응과 함께 다시 나타나고, (3) 뉴런이 모든 방향, 특히 초점면 안팎으로 이동하거나 여러 평면에 나타날 수 있기 때문에 어렵습니다4. 6입니다. 수동 분석은 시간이 많이 걸리고 기록 길이와 뉴런 수가 증가함에 따라 실용적이지 않습니다. 칼슘 이미징 분석 프로세스를 가속화하기 위해 다양한 소프트웨어 프로그램이 개발되었습니다. 이전에는 소프트웨어가 제한된 실험 맥락에서 설계되어 다른 실험실에서 소프트웨어를 채택하기가 어려웠습니다. 소프트웨어 공유에 대한 현대적 표준을 충족시키려는 최근의 노력은 상이한 그룹들에 걸쳐 칼슘 이미징 데이터를 일관되게 분석할 수 있는 몇몇 도구들의 개발로 이어졌다 7,8,9,10,11,12,13,14,15,16,17,18,19 . 그러나 이러한 도구의 대부분은 프로그래밍 지식이 필요하거나 상용 소프트웨어에 의존합니다. 프로그래밍 지식과 소프트웨어 페이월의 부족으로 인해 연구원들은 이러한 방법을 채택하지 못합니다. 또한, 이러한 도구들 중 다수는 x/y 모션을 교정하는 데 초점을 맞추지만, z축의 모션도 명시적으로 진단하고 교정해야 한다6. z-드리프트를 나타내고 여러 z-평면에 나타나는 뉴런에 초점을 맞춘 3D 칼슘 이미징을 분석하기 위한 계산 도구가 필요합니다. 이상적으로 이 도구는 오픈 소스 소프트웨어를 사용해야 하며 다른 실험실에서 쉽게 채택할 수 있도록 프로그래밍 지식이 필요하지 않아야 합니다.
여기에서 우리는 3D 칼슘 이미징 데이터를 분석하기 위해 새로운 ImageJ 플러그인인 TACI를 개발했습니다. 먼저, 필요한 경우 소프트웨어의 이름을 바꾸고 3D 칼슘 이미징 데이터를 z 위치별로 구성합니다. 관심 있는 세포는 각 z 위치에서 추적되고 형광 강도는 TrackMate 또는 기타 계산 도구에 의해 추출됩니다. 그런 다음 TACI를 적용하여 z축의 모션을 검사합니다. z-스택의 최대값을 식별하고 이를 사용하여 해당 시점에서 셀의 강도를 나타냅니다. 이 워크플로우는 모든 방향의 움직임 및/또는 측면(x/y) 방향에서 겹치지만 다른 z 위치에 나타나는 뉴런이 있는 3D 칼슘 이미징을 분석하는 데 적합합니다. 이 워크플로우를 검증하기 위해 뇌의 파리 유충 온도에 민감한 뉴런과 버섯 뉴런의 3D 칼슘 이미징 데이터 세트가 사용되었습니다. 참고로 TACI는 오픈 소스 ImageJ 플러그인이며 프로그래밍 지식이 필요하지 않습니다.
프로토콜
1. 칼슘 이미징
- 파리 애벌레 준비
참고: 파리와 유충은 25시간 12시간 명암 주기에서 12°C로 유지됩니다.- CO2로 파리를 마취하십시오. 20-45마리의 수컷과 20-45마리의 암컷을 각각의 플라이 바이알에 분류하고,CO2 노출로부터 회복하기 위해 적어도 24시간 내지 48시간을 준다.
알림: CO2 에 대한 비행 노출은 가능한 한 최단 시간 동안 지속되어야 합니다. - 유충의 나이를 맞추려면 효모 과립이 들어있는 새 바이알에 파리를 두드리고 4-8 시간 동안 알을 낳습니다. 파리를 새 바이알로 뒤집어 제거합니다.
- 20% w/v 자당 용액 10mL를 사용하여 72시간에 유충을 수집합니다.
- CO2로 파리를 마취하십시오. 20-45마리의 수컷과 20-45마리의 암컷을 각각의 플라이 바이알에 분류하고,CO2 노출로부터 회복하기 위해 적어도 24시간 내지 48시간을 준다.
- 현미경 및 온도 제어 설정
- 컨포칼 현미경( 재료 표 참조)과 z축 피에조 스테이지에서 이미징을 수행합니다.tage 다음 설정을 사용하여 스테이지 인서트가 있습니다. 레이저, 아르곤; 스캔 모드, 프레임; 프레임 크기, 512 x 512; 속도, 최대; 채널/비트 심도, 1/8비트; 줌, 1.5; z-스택, 슬라이스 = 15, 유지 = 슬라이스; 초점 장치 및 전략, 명확한 초점; 초점, 에 대한 명확한 초점.
- 열전달 화합물에 의해 Peltier 냉각 모듈을 방열판에 부착하여 열전 냉각기를 구축합니다. Peltier는 2A 전원 공급 장치로 구동됩니다.
- 열전대 마이크로프로브를 데이터 수집 디바이스에 부착하여 온도를 기록합니다.
- 칼슘 이미징
- 유충을 1x 인산염 완충 식염수(PBS)로 3배 헹굽니다.
- 1x PBS 75μL를 유리 슬라이드 중앙에 피펫팅합니다.
- 1x PBS에 하나 또는 두 개의 유충을 넣고 유충 근처에 열전대 마이크로 프로브를 놓습니다.
알림: 유충과 마이크로프로브 사이의 거리는 ~5mm여야 합니다. 유충은 마이크로 프로브가 너무 가까이 위치하면 움직일 수 있습니다. 거리가 멀면 온도 판독값이 정확하지 않을 수 있습니다. - 유충과 열전대 마이크로 프로브를 유리 커버 슬립으로 덮습니다. 커버슬립을 매니큐어로 밀봉합니다.
- 슬라이드를 현미경 스테이지에 놓고 25x 대물렌즈를 사용하여 초점을 찾은 다음 열전 냉각기를 슬라이드에 놓습니다.
알림: Peltier는 유충이 온도 자극을 전달하는 슬라이드에 직접 배치됩니다. - 컨포칼 소프트웨어에서는 관심 있는 형광 셀에 초점을 맞춥니다. 과포화를 피하기 위해 레이저 출력을 조정하십시오. z-스택 설정에서 첫 번째 및 마지막 슬라이스 위치를 설정합니다.
알림: 광표백을 방지하기 위해 녹음하기 전에 가능한 가장 낮은 레이저 출력을 사용하십시오. - z-스택 스캐닝과 온도 기록을 동시에 시작합니다.
- Peltier에 전원을 공급하는 전원 공급 장치를 제어하여 Peltier 표면 온도를 변경합니다. 전원을 켜면 온도가 낮아지고 전원을 끄면 온도가 높아집니다.
- z-stack 스캐닝 및 온도 기록을 중지합니다.
2. 3D 칼슘 이미징 데이터 분석
- 칼슘 이미징 데이터를 TIFF 파일로 내보내고 내부 TIFF 파일의 기본 이름과 동일한 이름의 폴더에 저장합니다.
참고: 파일 이름에는 쉼표가 포함되어서는 안 됩니다.- 다음 매개변수를 사용하여 ZEN(블랙 에디션)에서 칼슘 이미징 데이터를 내보냅니다. 파일 유형, TIFF; 압축, LZW; 채널, 2; Z-위치, 모두; 시간, 모두; 단계, 1; 지역, 전체.
- 설치
- Github(https://github.com/niflylab/TACI_CalciumImagingPlugin/releases)에서 TACI-Calcium_Imaging.jar를 다운로드합니다.
- 메뉴 바에서 플러그인을 클릭한 다음 드롭다운 메뉴에서 설치를 클릭하여 FIJI에 플러그인을 설치합니다(플러그인 | 설치). 그런 다음 FIJI를 다시 시작합니다.
참고: 플러그인을 사용하지 마십시오 | 플러그인을 설치합니다. - Plugins( 플러그인) 를 클릭한 다음 TACI-Calcium Imaging (Plugins | TACI-칼슘 이미징).
- RENAME 함수를 사용하여 TIFF 파일 이름을 필요한 구조로 변환합니다.
참고: 이 도구는 filename_h#t#z#c#.tif 기본 구조로 사용합니다(h# 및 c#은 선택 사항입니다. #: 양의 정수). 이미지 파일 이름이 기본 구조가 아닌 경우 RENAME 함수를 실행해야 합니다.- 폴더 찾아보기를 클릭하여 TIFF 이미지의 이름을 바꿔야 하는 폴더를 선택합니다.
- 매개 변수 정보를 입력합니다. 파일 이름, 위상, 최대 T-위치, 최대 Z-위치 및 채널을 포함한 5개의 매개변수가 나열됩니다. 각 매개 변수에는 Preceding Text, Parameter Value 및 Order의 세 가지 값이 있습니다.
참고: 이 정보는 대/소문자를 구분합니다. 이미지 파일 이름에 매개 변수 앞에 구가 포함되어 있는 경우 해당 구를 해당 매개 변수의 선행 텍스트 로 입력합니다. 이미지 파일 이름에 모든 매개 변수 뒤에 문구가 포함되어 있는 경우 문구를 Post Text로 입력합니다.- 파일 이름, 최대 T-위치 및 최대 Z-위치를 입력하고 최대 T-위치 및 최대 Z-위치의 매개변수 값에 모든 숫자가 포함되도록 해야 합니다.
- 폴더 이름을 사용하여 파일 이름이 자동으로 채워질 때까지 기다립니다.
- TIFF 파일 이름에 위상 및 채널이 포함되어 있지 않으면 해당 매개 변수 값을 비워 두고 [순서]에서 [Na]를 선택합니다.
- 이름 바꾸기를 클릭하여 이름과 _r 같은 폴더를 만듭니다. 폴더에서 TIFF 파일 이름이 ORGANIZE 함수와 호환되도록 재구성되었는지 확인합니다.
참고: TIFF 이미지의 파일 이름에 _r 추가되었습니다.
- ORGANIZE 함수를 사용하여 동일한 z 위치의 TIFF 이미지를 한 폴더에 저장합니다.
참고: 폴더 이름은 내부에 있는 TIFF 파일의 기본 이름과 같아야 하며 쉼표가 없어야 합니다.- 폴더 찾아보기를 클릭하여 TIFF 이미지를 구성해야 하는 폴더를 선택합니다.
- 매개 변수 CSV 파일(param.csv)이 있는 경우 매개 변수 값이 자동으로 채워질 때까지 기다립니다.
참고: param.csv 파일에는 필수 형식이 있습니다. 파일 이름, 위상, position_t, position_z, 채널 및 is_gray를 포함한 매개 변수는 열 A에서 시작하여 왼쪽에서 오른쪽으로 1행에 채워야 합니다. 각 매개 변수에 해당하는 값은 2행에 채워야 합니다. - 매개변수 CSV 파일(param.csv)이 없는 경우 매개변수 값을 수동으로 입력합니다. 위상 및 채널의 매개변수 값에는 문자가 포함되고 T 위치 및 Z 위치의 매개변수 값은 t 및 z 위치의 가장 큰 수 여야 합니다. 이미지 파일 이름에 위상 또는 채널이 포함되어 있지 않으면 Na를 입력합니다.
- 필요한 경우 회색조 TIFF 이미지를 만듭니다. Are images gray ?(이미지가 회색입니까?) 상자를 선택 취소하여 이미지를 회색 음영으로 만듭니다.
- 구성을 클릭하여 이름과 _gray_stacks 같은 폴더를 만들고 폴더에 같은 이름과 _#(#: z-positions)의 폴더를 생성합니다. TIFF 파일이 z 위치에 따라 해당 폴더로 정렬되고 매개 변수와 해당 값을 찾을 수 있는 param.csv이라는 파일이 생성되는지 확인합니다.
- FIJI의 TrackMate를 사용하여 각 z 위치에서 관심 세포의 형광 강도를 추출합니다.
참고: 이 단계는 다른 이미징 소프트웨어로도 수행할 수 있습니다.- FIJI의 z 위치 폴더에서 TIFF 이미지를 엽니다.
- 플러그인을 클릭하여 TrackMate 실행 | 추적 | TrackMate를 선택하고 필요한 경우 다음 매개변수를 조정합니다.
- DoG 또는 LoG 감지기를 사용하십시오.
- Blob 지름, 임계값 및 중앙값 필터를 변경합니다. 블롭 지름을 셀의 지름과 비슷하게 조정합니다. 셀이 타원형이면 Blob 지름을 보조 축과 비슷하게 조정합니다.
참고: 임계값 을 높이면 신호 강도에 영향을 주지 않고 배경 소음이 신호로 포착되는 것을 방지할 수 있습니다(보충 그림 1). 그러나 임계값 이 증가하면 실제 신호를 놓칠 수 있습니다(보충 그림 1). 신호가 강하면 중앙값 필터를 사용하여 소금과 후추 노이즈를 줄입니다. - 관련 없는 신호의 전부는 아니더라도 일부를 제거하도록 필터를 설정합니다. 필터 X와 Y는 공간 필터입니다. 필터 X와 Y를 사용하여 실제 신호에서 멀리 떨어져 있는 관련 없는 신호를 제거합니다.
알림: 한 이미지에 필터가 설정되어 있을 때 실제 신호가 제거되지 않았는지 확인하기 위해 다른 모든 이미지를 확인하는 것이 중요합니다. - 연결 최대 거리, 갭 닫기 최대 거리 및 갭 닫기 최대 프레임 간격을 설정합니다. 특히 샘플이 시간이 지남에 따라 크게 이동할 때 연결 최대 거리 및 간격 폐쇄 최대 거리를 Blob 직경의 3x-5배로 설정하면 트랙 수를 줄이는 데 도움이 됩니다. 간격 닫기 최대 프레임 간격을 스택의 이미지 수로 설정합니다.
- 관심 영역(ROI)의 형광 강도를 CSV 파일로 내보냅니다. 이전 TrackMate 버전을 사용하는 경우 작업 선택 창에서 모든 스팟 통계 내보내기를 선택합니다. TrackMate 버전이 7.6.1 이상인 경우 디스플레이 옵션 창에서 스팟을 선택합니다. 대화형 파일을 CSV 파일로 내보내거나 저장합니다.
참고: 두 파일 모두 이미지 창과 대화합니다. ROI를 강조 표시하면 이미지 창에 해당 ROI가 표시됩니다. 이러한 파일에는 해당 시점(POSITION_T)에서 관심 있는 셀의 평균 강도(MEAN_INTENSITY 또는 MEAN_INTENSITY_CH1)가 포함됩니다. 동일한 TRACK_IDs는 서로 다른 시점에서 동일한 ROI를 나타내야 합니다. 그러나 항상 그런 것은 아니며 필요한 경우 수동으로 수정해야 할 수도 있습니다. TrackMate가 특정 시점에서 ROI를 인식하지 못하면 해당 시점이 표시되지 않습니다.
- 배경 형광 강도를 추출하고 Background_list.csv 파일을 만듭니다.
참고: 이 연구에서, 각 z-위치에 대한 배경 강도는 형광 신호를 포함하지 않고 다른 시점에서 온 3 내지 5개의 인접한 동일한 크기 얼룩의 평균값을 사용하여 추정되었습니다.- Background_list.csv 파일에는 필수 형식이 있습니다. 각 열에는 Neuron 0부터 시작하는 하나의 뉴런 정보가 포함됩니다. 1행의 뉴런 번호(예: Neuron 0)를 입력합니다. 그런 다음 분석된 각 z-위치에 대한 배경 강도를 제공합니다(Neuron 0에 대해 5개의 z-위치가 분석된 경우) Neuron 0 아래에 5개의 배경 강도 값을 입력합니다.
참고: Background_list.csv 파일은 TACI EXTRACT 함수에 필요합니다. 배경이 무시할 수 있는 경우 모든 뉴런의 배경 강도가 0인 Background_list.csv 제공해야 합니다.
- Background_list.csv 파일에는 필수 형식이 있습니다. 각 열에는 Neuron 0부터 시작하는 하나의 뉴런 정보가 포함됩니다. 1행의 뉴런 번호(예: Neuron 0)를 입력합니다. 그런 다음 분석된 각 z-위치에 대한 배경 강도를 제공합니다(Neuron 0에 대해 5개의 z-위치가 분석된 경우) Neuron 0 아래에 5개의 배경 강도 값을 입력합니다.
- EXTRACT 함수를 사용하여 각 t 위치에서 최대 형광 강도를 식별하고 방정식 (1)과 같이 ΔF/F0을 계산합니다.
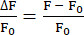 (1개)
(1개)- 폴더를 만들고 Neuron 0부터 시작하는 뉴런 번호를 사용하여 이름을 지정합니다. 해당 뉴런의 형광 정보가 포함된 CSV 파일을 폴더에 저장합니다. 각 CSV 파일에는 하나의 z 위치 정보가 포함되어 있으므로 CSV 파일 수는 분석된 z 위치 수와 같습니다. CSV 파일의 이름을 Mean_Intensity#.csv(#은 z 위치를 나타냄)로 지정하고 각 CSV 파일에는 POSITION_T 및 MEAN_INTENSITY 또는 MEAN_INTENSITY_CH1의 두 개 이상의 열이 포함됩니다.
참고: 관심 있는 모든 셀에 대한 폴더를 만듭니다. - 2.7.1단계에서 만든 Background_list.csv 파일과 폴더를 하나의 폴더에 저장합니다. 파일 찾아보기를 클릭하여 이 폴더를 선택합니다.
참고: Background_list.csv 파일의 배경 값 수는 각 뉴런에 대한 Mean_Intensity#.csv 파일 수와 일치해야 합니다. - 배경 파일이 자동으로 채워집니다. T 위치 수에 대해 가장 큰 수의 t-위치를 채웁니다.
- 추출(Extract)을 클릭하여 각 뉴런에 대한 CSV 파일 및 플롯을 포함하는 결과 폴더를 생성합니다. CSV 파일에는 각 t 위치에서 최대 형광 강도 및 ΔF/F0에 대한 정보가 포함되어 있습니다. 플롯은 t-위치에 대한 ΔF/F0의 꺾은선형 차트입니다.
참고: 이 연구에서 ΔF/F0 는 방정식 1을 사용하여 계산되었습니다. 각 z-위치의 제1 값은F0를 사용하였다. 이 F0 이 적절하지 않은경우 20 플러그인은 python_files 폴더의 각 뉴런에 대한 원시 데이터를 포함하는 파일을 제공합니다.
- 폴더를 만들고 Neuron 0부터 시작하는 뉴런 번호를 사용하여 이름을 지정합니다. 해당 뉴런의 형광 정보가 포함된 CSV 파일을 폴더에 저장합니다. 각 CSV 파일에는 하나의 z 위치 정보가 포함되어 있으므로 CSV 파일 수는 분석된 z 위치 수와 같습니다. CSV 파일의 이름을 Mean_Intensity#.csv(#은 z 위치를 나타냄)로 지정하고 각 CSV 파일에는 POSITION_T 및 MEAN_INTENSITY 또는 MEAN_INTENSITY_CH1의 두 개 이상의 열이 포함됩니다.
- MERGE 함수를 사용하여 각 뉴런의 ΔF/F 0 평균을 구하고, SEM을 계산하고, t-위치에 대한 평균 ΔF/F0 를 플로팅합니다.
- 파일 찾아보기를 클릭하여 EXTRACT 함수로 만든 결과 폴더를 선택합니다.
- 가장 많은 수의 t-포지션으로 T 포지션 수를 채웁니다.
- 결합(Merge)을 클릭하여 merged_data.csv 파일과 Average_dF_F0.png 플롯을 포함하는 merged_data 폴더를 생성합니다. CSV 파일에는 각 t 위치의 평균 및 SEM ΔF/F0에 대한 정보가 포함되어 있습니다. 그림은 t-위치에 대한 평균 ΔF/F0의 꺾은선형 차트입니다.
결과
3D 칼슘 이미징 분석의 워크플로우
이 연구에서는 새로운 ImageJ 플러그인인 TACI를 개발하고, z-드리프트를 추적하고 여러 z-위치에 나타나는 개별 세포의 반응을 정확히 찾아내는 3D 칼슘 이미징을 분석하는 워크플로우를 설명했습니다(그림 1). 이 도구에는 RENAME, ORGANIZE, EXTRACT 및 MERGE의 네 가지 기능이 있습니다...
토론
이 연구에서는 새로운 ImageJ 플러그인인 TACI를 개발하고 3D 칼슘 이미징을 분석하는 워크플로를 설명했습니다. 현재 사용 가능한 많은 도구는 x/y 모션을 수정하는 데 중점을 두고 있지만 z축의 모션도 명시적으로 진단하거나 수정해야 합니다6. 살아있는 유기체에서 이미지를 획득하는 동안 유기체가 고정되어 있어도 z축의 움직임은 피할 수 없으며 온도 변화와 같은 일부 자극은 ?...
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
Fralin Imaging Center의 Zeiss LSM 880을 사용하여 칼슘 이미징 데이터를 수집했습니다. IMARIS 소프트웨어에 도움을 주신 Michelle L Olsen 박사님과 Yuhang Pan 박사님께 감사드립니다. 원고에 대한 건설적인 의견을 주신 Lenwood S. Heath 박사님과 GitHub README 파일에 대한 의견을 주신 Steven Giavasis에게 감사드립니다. 이 작업은 NIH R21MH122987(https://www.nimh.nih.gov/index.shtml) 및 NIH R01GM140130(https://www.nigms.nih.gov/)에 의해 L.N.에 의해 지원되었습니다. 자금 제공자는 연구 설계, 데이터 수집 및 분석, 출판 결정 또는 원고 준비에 아무런 역할도 하지 않았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Blunt Fill Needel | BD | 303129 | |
| Calcium chloride dihydrate | Fisher Scientific | 10035-04-8 | Fly food ingredient |
| Carbon dioxide | Airgas | UN1013 | Size 200 High Pressure Steel Cylinder |
| CO2 bubbler kit | Genesee | 59-180 | |
| Confocal microscope LSM880 | Zeiss | 4109002107876000 | An inverted Axio Observer Z1, equipped with 5 lasers, 2 standard PMT detectors, 32-channel GaAsP dectectors, an Airyscan detector, and Definite Focus.2. |
| DAQami software | Measurement Computing | ||
| Dextrose | Genesee | 62-113 | Fly food ingredient |
| Drosophila Agar | Genesee | 66-111 | Fly food ingredient |
| Ethanol | Decon Labs, Inc. | 64-17-5 | Fly food ingredient |
| Fly line: Ir21a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: Ir21a-Gal80 | Dr. Lina Ni lab | ||
| Fly line: Ir68a-Gal4 | Dr. Aravinthan DT Samuel lab | A kind gift | |
| Fly line: Ir93a-Gal4 | Dr. Paul Garrity lab | A kind gift | |
| Fly line: UAS-GCaMP6 | Bloomington Drosophila Stock Center | 42750 | |
| Flypad | Genesee | 59-114 | |
| General purpose forged brass regulator | Gentec | G152 | |
| Gibco PBS pH 7.4 (1x) | Thermo Fisher Scientific | 10010-031 | |
| Green Drosophila tubing | Genesee | 59-124 | |
| Heat transfer compound | MG Chemicals | 860-60G | |
| Heatsink | Digi-Key Electronics | ATS2193-ND | Resize to 12.9 x 5.5 cm |
| Illuminator | AmScope | LED-6W | |
| Inactive Dry Yeast | Genesee | 62-108 | Fly food ingredient |
| Incubator | Pervical | DR-41VL | Light: dark cycle: 12h:12h; temperature: 25 °C; humidity: 40-50% RH. |
| Methyl-4-hydroxybenzoate | Thermo Scientific | 126965000 | Fly food ingrediete |
| Micro cover glass | VWR | 48382-126 | 22 x 40 mm |
| Microscope slides | Fisher Scientific | 12-544-2 | 25 x 75 x 1.0 mm |
| Nail polish | Kleancolor | ||
| Narrow Drosophila vials | Genesee | 32-113RL | |
| Objective | Zeiss | 420852-9871-000 | LD LCI Plan-Apochromat 25x/0.8 Imm Corr DIC M27 |
| Peltier cooling module | TE Technology | TE-127-1.0-0.8 | 30 x 30 mm |
| Plugs | Genesee | 49-102 | |
| Power Supply | Circuit Specialists | CSI1802X | 10 volt DC 2.0 amp linear bench power supply |
| Princeton Artist Brush Nepture | Princeton Artist Brush Co. | Series 4750, size 2 | |
| Sodium potassium L-tartrate tetrahydrate | Thermo Scientific | 033241-36 | Fly food ingredient |
| Stage insert | Wienecke and Sinske | 432339-9030-000 | |
| Stereo Microscope | Olympus | SZ61 | Any stereo microscope works |
| T-Fitting | Genesee | 59-123 | |
| Thermocouple data acquisition device | Measurement Computing | USB-2001-TC | Single channel |
| Thermocouple microprobe | Physitemp | IT-24P | |
| Yellow Cornmeal | Genesee | 62-101 | Fly food ingredient |
| Z-axis piezo stage | Wienecke and Sinske | 432339-9000-000 |
참고문헌
- Grienberger, C., Konnerth, A. Imaging calcium in neurons. Neuron. 73 (5), 862-885 (2012).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nature Biotechnology. 19 (2), 137-141 (2001).
- Zhang, Y., et al. jGCaMP8 fast genetically encoded calcium indicators. Janelia Research Campus. , (2020).
- Robbins, M., Christensen, C. N., Kaminski, C. F., Zlatic, M. Calcium imaging analysis - How far have we come. F1000Research. 10, 258 (2021).
- Oh, J., Lee, C., Kaang, B. K. Imaging and analysis of genetically encoded calcium indicators linking neural circuits and behaviors. The Korean Journal of Physiology & Pharmacology. 23 (4), 237-249 (2019).
- Stringer, C., Pachitariu, M. Computational processing of neural recordings from calcium imaging data. Current Opinion in Neurobiology. 55, 22-31 (2019).
- Pnevmatikakis, E. A., Giovannucci, A. NoRMCorre: An online algorithm for piecewise rigid motion correction of calcium imaging data. Journal of Neuroscience Methods. 291, 83-94 (2017).
- Nguyen, J. P., Linder, A. N., Plummer, G. S., Shaevitz, J. W., Leifer, A. M. Automatically tracking neurons in a moving and deforming brain. PLoS Computational Biology. 13 (5), 1005517 (2017).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. EMC2: A versatile algorithm for robust tracking of calcium dynamics from individual neurons in behaving animals. bioRxiv. , (2021).
- Giovannucci, A., et al. CaImAn an open source tool for scalable calcium imaging data analysis. Elife. 8, 38173 (2019).
- Delestro, F., et al. In vivo large-scale analysis of Drosophila neuronal calcium traces by automated tracking of single somata. Scientific Reports. 10, 7153 (2020).
- Cantu, D. A., et al. EZcalcium: Open-source toolbox for analysis of calcium imaging data. Frontiers in Neural Circuits. 14, 25 (2020).
- Eglen, S. J., et al. Toward standard practices for sharing computer code and programs in neuroscience. Nature Neuroscience. 20 (6), 770-773 (2017).
- Pachitariu, M., et al. Suite2p: Beyond 10,000 neurons with standard two-photon microscopy. bioRxiv. , (2017).
- Corder, G., et al. An amygdalar neural ensemble that encodes the unpleasantness of pain. Science. 363 (6424), 276-281 (2019).
- Lagache, T., Hanson, A., Pérez-Ortega, J. E., Fairhall, A., Yuste, R. Tracking calcium dynamics from individual neurons in behaving animals. PLoS Computational Biology. 17 (10), 1009432 (2021).
- Kolar, K., Dondorp, D., Zwiggelaar, J. C., Høyer, J., Chatzigeorgiou, M. Mesmerize is a dynamically adaptable user-friendly analysis platform for 2D and 3D calcium imaging data. Nature Communications. 12, 6569 (2021).
- Moein, M., et al. CaSiAn: A Calcium Signaling Analyzer tool. Bioinformatics. 34 (17), 3052-3054 (2018).
- Zhou, P., et al. Efficient and accurate extraction of in vivo calcium signals from microendoscopic video data. Elife. 7, 28728 (2018).
- Neugornet, A., O'Donovan, B., Ortinski, P. I. Comparative effects of event detection methods on the analysis and interpretation of Ca(2+) imaging data. Frontiers in Neuroscience. 15, 620869 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Fazeli, E., et al. Automated cell tracking using StarDist and TrackMate. F1000Research. 9, 1279 (2020).
- Chen, T. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Ni, L., et al. The ionotropic receptors IR21a and IR25a mediate cool sensing in Drosophila. Elife. 5, 13254 (2016).
- Omelchenko, A. A., et al. Cool and warm ionotropic receptors control multiple thermotaxes in Drosophila larvae. Frontiers in Molecular Neuroscience. , (2022).
- Sanchez-Alcaniz, J. A., et al. An expression atlas of variant ionotropic glutamate receptors identifies a molecular basis of carbonation sensing. Nature Communications. 9 (1), 4252 (2018).
- Hernandez-Nunez, L., et al. Synchronous and opponent thermosensors use flexible cross-inhibition to orchestrate thermal homeostasis. Science Advances. 7 (35), (2021).
- Klein, M., et al. Sensory determinants of behavioral dynamics in Drosophila thermotaxis. Proceedings of the National Academy of Sciences of the United States of America. 112 (2), 220-229 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유