JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
역전사 루프 매개 등온 증폭을 통한 SARS-CoV-2 바이러스 검출
요약
여기에서는 RT-LAMP(reverse transcription loop-mediated isothermal amplification)를 통해 인간 샘플에서 SARS-CoV-2 바이러스를 검출하는 방법을 표준화하고 구현하기 위한 완전한 프로토콜을 제공합니다. 60분 이내에 완료되는 이 방법은 저렴한 비용과 저렴한 장비를 사용하여 모든 실험실이나 현장 진료에 적용할 수 있습니다.
초록
중증 급성 호흡기 증후군 코로나바이러스 2(SARS-CoV-2) 바이러스는 인간의 건강에 큰 영향을 미쳤습니다. 많은 사람들이 감염으로 사망하기 때문에 현대 사회에 계속 위협이 되고 있습니다. 이 질병은 황금 표준 실시간 중합효소 연쇄 반응(RT-PCR)과 같은 혈청학적 및 분자 검사를 사용하여 진단됩니다. 마지막은 전문 인프라, 값비싼 장비 및 숙련된 인력이 필요하기 때문에 몇 가지 단점이 있습니다. 여기에서는 인간 샘플에서 RT-LAMP(reverse transcription-loop-mediated isothermal amplification)를 사용하여 SARS-CoV-2 바이러스를 검출하는 데 필요한 단계를 설명하는 프로토콜을 제시합니다. 이 프로토콜에는 silico에서 프라이머를 설계하고, 시약을 준비하고, 증폭 및 시각화하기 위한 지침이 포함되어 있습니다. 표준화되면 이 방법은 저렴한 비용과 저렴한 장비를 사용하여 60분 이내에 모든 실험실 또는 현장 진료에 쉽게 구현하고 적용할 수 있습니다. 다양한 병원체를 감지하는 데 적응할 수 있습니다. 따라서 현장 및 보건소에서 적시에 역학 감시를 수행하는 데 잠재적으로 사용될 수 있습니다.
서문
중증 급성 호흡기 증후군 코로나 바이러스 2 (SARS-CoV-2)는 코로나 바이러스 질병 2019 (COVID-19)를 유발합니다. 세계보건기구(WHO)는 2020년 1월 30일 국제적 공중보건 비상사태를, 2020년 3월 11일 팬데믹을 선포했습니다. 팬데믹으로 인해 이 기사가 작성된 시점을 기준으로 7억 6천만 명 이상의 확진자와 687만 명의 사망자가 발생했습니다1.
이 바이러스의 영향은 전염병 탐지 및 통제를 개선하기 위해 더 우수하고, 더 정확하고, 더 빠르고, 더 널리 사용 가능한 감시 도구의 필요성을 강조했습니다 2,3. 팬데믹 기간 동안 SARS-CoV-2 진단 검사는 핵산, 항체 및 단백질 검출을 기반으로 했지만 핵산의 RT-PCR 검출이 황금 표준입니다4. 그러나 RT-PCR에는 몇 가지 제한 사항이 있습니다. 분자 생물학 교육을 받은 특수 장비, 인프라 및 인력이 필요하므로 전문 실험실로 적용이 제한됩니다. 또한 표본을 실험실로 운반하는 데 걸리는 시간을 제외하고 시간이 많이 소요되며(4-6시간) 며칠이 걸릴 수 있습니다5. 이러한 제약으로 인해 효율적인 검체 처리와 비상 계획 및 역학 관리에 필요한 정보를 얻을 수 없습니다.
RT-LAMP(Reverse Transcription-Loop-Mediated Isothermal Amplification)는 RT-PCR에 비해 몇 가지 장점이 있어 특히 자원이 제한된 환경에서 미래의 POCT(point-of-care diagnostic test)를 설계하는 데 매력적인 전략이 된다6. 첫째, 표적 서열에서 6-8개의 영역, 즉 DNA또는 RNA 7,8을 인식하는 4-6개의 프라이머를 사용하기 때문에 매우 특이적입니다. 둘째, 일정한 온도에서 작동하기 때문에 증폭을 생성하기 위해 실시간 열 순환기와 같은 정교한 장비가 필요하지 않으며 이를 작동하기 위해 고도로 훈련된 인력이 필요하지 않습니다. 셋째, 반응 시간이 매우 짧고(~60분) 매우 전문화되지 않은 시약이 사용되어 비용 효율적인 도구입니다6. 앞서 언급한 바와 COVID-19 팬데믹으로 인한 보건 비상 사태를 감안할 때, 이 기법은 모든 연구 실험실에서 빠르고 저렴하며 간단하게 구현할 수 있는 대체 진단 방법으로 볼 수 있다9.
이 기사에서는 열순환기와 수조를 사용하여 비색 방법으로 SARS-CoV-2를 검출하기 위해 RT-LAMP를 표준화하고 구현하기 위한 프로토콜에 대해 설명합니다(그림 1). 임계점, 한계 및 이를 발전시키기 위한 대안에 대해 논의합니다.
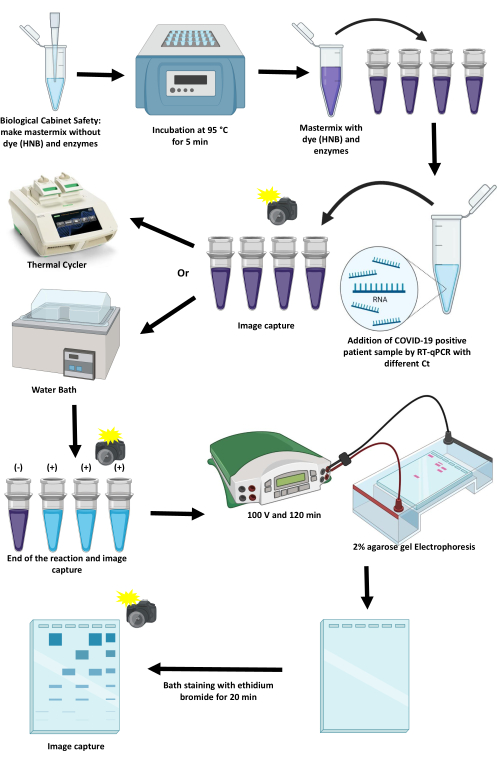
그림 1: RT-LAMP 기법을 사용하여 SARS-CoV-2를 증폭하기 위한 프로토콜 체계. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
사용된 샘플은 Fundación Valle del Lili University 병원의 임상 실험실에서 제공되었으며 RT-qPCR 기술을 사용하여 COVID-19 양성 반응을 보인 환자의 정제된 RNA와 일치했습니다. 모든 환자는 연구에 대한 사전 동의를 제공했으며, 이 연구는 Fundación Valle del Lili University 병원의 인간 연구를 위한 생명윤리 위원회의 승인을 받았습니다.
1. RT-LAMP 프라이머 설계 및 준비
참고: LAMP 프라이머는 NEB(New England BioLabs) LAMP, Primer Explorer 및 LAMP assay versatile analysis(LAVA)를 포함한 다양한 플랫폼과 함께 사용할 수 있습니다. 그러나 이 프로토콜의 경우 NEB LAMP 도구가 사용되었습니다. 프라이머 설계는 NextStrain 데이터베이스10에서 얻은 SARS-CoV-2 게놈을 사용하여 수행할 수 있습니다. 표 1 은 이 프로토콜에 사용된 프라이머 세트를 보여줍니다.
- LAMP용 프라이머 설계
- 바이러스 게놈 염기서열을 얻습니다.
- 시퀀스 정렬을 수행하여 합의 시퀀스를 얻습니다.
- NEB LAMP 프라이머 설계 도구 플랫폼11로 이동하여 퀵 가이드의 지침을 따릅니다. 이 도구는 프라이머 탐색기 V5와 동일한 결과를 생성하지만 출력이 훨씬 사용자 친화적입니다. Primer Explorer 사용자 설명서를 Primer Design의 가이드로 사용하십시오.
- 프라이머 세트의 열역학적 평가
- Primer-Dimer12 도구를 사용하여 얻은 프라이머에 대한 열역학 분석을 수행합니다.
- 프라이머 시퀀스를 도구에 넣습니다. 그런 다음 Multiplex Analysis and Dimer Structure Report 옵션을 선택합니다.
- ΔG가 -5 이상인 프라이머 세트를 선택합니다.
- 설계된 프라이머의 특이성 평가
- BLAST13 의 뉴클레오티드 수집(nt/nt) 데이터베이스를 사용하여 각 프라이머를 분석합니다.
- 첫 번째 BLAST 분석을 수행하려면 Refseq_rna 데이터베이스를 선택하고 Orthocoronavirinae 아과에 속하는 속 그룹으로 검색을 필터링합니다. 알파코로나바이러스(taxid:693996), 감마코로나바이러스(taxid:694013), 델타코로나바이러스(taxid:1159901)입니다. 또한 H1N1 아형(taxid:114727), 인플루엔자 A 바이러스(taxid:11320) 및 인플루엔자 B 바이러스(taxid:11520)로 동시 순환하는 다른 바이러스에 대해 염기서열을 평가합니다.
- 두 번째 BLAST 분석을 수행하려면 Betacoronavirus GenBank를 선택하고 Coronaviridae(taxid:11118) 및 SARS(taxid:694009)로 검색을 필터링합니다. 이 그룹에는 박쥐에서 발견되는 게놈, Betacoronavirus(taxid:694002)를 포함하여 확인된 모든 SARS 코로나바이러스 게놈의 염기서열이 포함되어 있습니다.
- 이 프로토콜의 경우 프라이머가 표적 게놈인 SARS-CoV-2 이외의 게놈과 일치하지 않도록 합니다.
- 프라이머 준비
- 이요건조된 프라이머가 들어 있는 바이알을 마이크로 원심분리기(10,000 x g, 실온[RT]에서 1분)로 회전시켜 튜브 개방 중 손실을 방지합니다.
- 이요건조된 분말을 0.1% 디에틸 피로카보네이트(DEPC) 물 또는 뉴클레아제가 없는 물에 최종 농도 100μM(표 2)로 재수화하고 위아래 피펫팅으로 완전히 용해시킵니다. 그런 다음 마이크로 원심분리기에서 최대 속도(10,000 x g, RT에서 1분)로 회전하여 튜브 바닥에 있는 모든 프라이머 용액을 수집합니다.
- 표 2에 보고된 바와 같이 전방 내부 프라이머(FIP), 후방 내부 프라이머(BIP), 전방 외부 프라이머(F3), 후방 외부 프라이머(B3), 루프 백워드(LB) 및 루프 포워드(LF) 프라이머를 사용하여 생물안전 캐비닛 아래에 2x 프라이머 혼합물을 준비합니다. 손실을 방지하려면 마이크로 원심분리기로 빠른 회전(10,000 x g, RT에서 1분)을 수행하기 전에 프라이머 용액을 피펫팅하거나 부드럽게 소용돌이치십시오.
- 장기 보관을 위해 10x 프라이머 혼합물을 -20°C에서 보관하십시오. 그러나 너무 많은 동결-해동 주기를 피하기 위해 여러 샘플에 관계없이 최대 5개의 실험에 대해 충분히 준비하십시오.
주의사항 더 작은 부피의 프라이머 혼합물이 필요한 경우 새 부피를 계산하여 값을 조정하십시오(표 2). 또한 RT-LAMP 반응에는 루프 프라이머가 필요하지 않기 때문에 RdRp 및 RdRp/Hel 세트에는 LF 프라이머가 포함되어 있지 않습니다. 결과적으로 LF 프라이머의 부피를 뉴클레아제가 없는 물 또는 0.1% DEPC 물로 교체합니다.
2. RT-LAMP 반응
- 제조업체의 지침에 따라 층류 캐비닛을 켜고 공기 흐름이 안정화될 때까지 최소 3분 동안 기다립니다.
- 공기 흐름이 안정되면 무균 기술을 사용하여 캐비닛의 내부 표면을 청소하고 살균합니다. 이를 위해 1000ppm 4차 암모늄(염화벤잘코늄), 2% 차아염소산염, 3% 과산화수소 및 70% 에탄올 순서로 소독제를 사용하십시오.
알림: 이 경우 무균 기술은 소독제를 도포하고 이전에 청소한 표면을 거치지 않고 냅킨으로 객실 내부에서 외부로 제거하는 것을 수반합니다. - 2.2 단계의 소독제를 사용하여 동일한 순서로 객실에 들어갈 재료를 청소하십시오.
참고: 마이크로피펫, 필터 팁 박스, 1.5mL 및 0.6mL 튜브가 있는 플라스크, 0.2mL PCR 튜브, 랙 및 400mL 비커를 캐비닛에 가져와야 합니다. - 냅킨과 니트릴 장갑을 기내에 반입하십시오. 그런 다음 캐비닛을 끄고 15분 동안 자외선(UV)에 노출시킵니다.
주의: 장기간 방사선 노출로 인한 조직 및 DNA 손상을 방지하려면 2.4단계에서 설정한 시간이 만료될 때까지 자외선을 피하십시오.
주의사항 프로토콜을 시작하기 전에 그림 2 에 표시된 조립을 수행하고 2.4단계를 완료한 후 수조를 시작하십시오. 금속 용기에 식수를 거의 가득 채우고 철제 실험실 가열판의 온도를 90 ° C로 설정하는 것이 중요한데, 이는 시스템의 온도가 ~ 66.3 ° C가 될 수 있으므로 수은 온도계로 모니터링됩니다. - 조사 기간이 끝나면 캐비닛을 다시 시작하고 1.1단계의 권장 사항을 따르십시오.
- 시약(표 3, 표 4 및 표 5)을 얼음이 채워진 냉각기 또는 작은 폴리스티렌 냉장고에 넣습니다. 70% 에탄올로 세척한 후 용기를 캐비닛에 넣습니다.
- 0.6mL 미세 원심분리기 튜브에서 증폭할 유전자의 LAMP 혼합물(RdRp, N-A 및 RdRp/Hel)을 준비하고 다음 구성 요소만 추가합니다: 10x 완충액, MgSO4, dNTPs, 1x 프라이머 믹스 및 뉴클레아제가 없는 물 또는 0.1% DEPC 물; 균질화하기 위해 피펫팅으로 잘 섞습니다.
주의: 캐비닛 내부의 부적절한 취급 및 동작으로 인해 시약 오염의 위험이 높습니다. 이 문제를 완화하기 위해 다음 규칙을 따라야 합니다: (i) 멸균 및 필터 팁을 사용하십시오. (ii) 각 시약에 대해 하나의 팁을 사용하십시오. (iii) 층류를 방해하지 않도록 천천히 조심스럽게 움직입니다. (iv) 질서를 유지하고 최소한의 재료를 사용합니다. (v) 다른 장갑을 사용하여 혼합물을 준비하고 유전 물질을 첨가하십시오.
알림: 모든 시약, 특히 효소는 온도 변화로 인해 변성되고 중합효소 활성이 변경될 수 있으므로 얼음에 보관하십시오. - 캡을 닫은 상태로 0.6mL 튜브를 가열 블록에 넣고 95°C에서 5분 동안 배양합니다.
알림: l을 시작하기 전에 최소 30분 동안 캐비닛 외부에 있는 1.5-2.0mL 튜브의 가열 블록을 켜십시오.amp 혼합물 준비를 하고 수은 또는 알코올 온도계로 온도(95°C)를 모니터링합니다. - 배양이 완료되면 튜브를 얼음이 채워진 폴리스티렌 쿨러에 5분 동안 넣습니다.
- 튜브를 층류 캐비닛으로 되돌리고 효소 DNA 중합효소(Bst 3.0), 역전사 효소 및 고충실도 DNA 중합효소(표 3, 표 4 및 표 5)를 추가하여 LAMP 혼합물 준비를 완료합니다. 비색 검출을 사용하는 경우 염료 하이드록시나프톨 블루(HNB)를 추가합니다.
- 이 시약을 첨가한 후 LAMP 시약을 피펫팅하여 효소와 염료를 용해시켜 잘 혼합합니다.
- 기포가 생기지 않도록 주의하면서 각 PCR 튜브에 22.0μL의 혼합물을 채웁니다. 그런 다음 3.0μL의 0.1% DEPC 물 또는 뉴클레아제가 없는 물을 음성 대조군 또는 템플릿 없는 튜브(NTC)에 추가하고 나머지 튜브는 추가(유전 물질)를 위해 따로 보관합니다.
참고 : Bst 3.0 효소가 활성화되어 반응이 조기에 시작되지 않도록 샘플이 추가될 때까지 PCR 튜브를 얼음이 채워진 냉각기에 보관하십시오. - 캐비닛에서 모든 재료를 제거하고 70% 에탄올을 사용하여 표면을 청소합니다. 그런 다음 제조업체의 지침에 따라 전원을 끕니다.
- 별도의 영역에서 각 PCR 튜브에 3μL의 샘플을 추가하고 완전히 균질화합니다. 이를 위해 20μL 마이크로피펫과 필터 팁을 사용하십시오.
주의: 유전 물질을 첨가하는 데 사용되는 마이크로피펫은 이 목적으로만 사용해야 하며 혼합물을 준비하는 데 사용할 수 없습니다. 이러한 방식으로 시약의 오염을 방지할 수 있습니다. 또한 RNA 분해 가능성을 줄이기 위해 RNA 샘플을 항상 얼음 위에 보관하십시오. 샘플 추가를 위해 일회용 가운, 모자, N95 마스크, 레깅스, 실험실 고글 및 니트릴 장갑과 같은 개인 보호 장비(PPE)를 사용하십시오. - 비색 반응을 수행하기 전에 고품질 카메라로 PCR 튜브의 사진을 찍습니다. HNB의 시작 색상은 보라색입니다.
- 다음 시스템 또는 장비에서 반응을 수행하십시오: (i) 열 순환기 및 (ii) 수조.
- 열 순환기: 튜브를 반응 블록에 증착하고 thermopro를 설정합니다.file ( 표 6 참조) 장비에.
- 수조: 튜브를 원형 용기에 넣고 튜브가 나오지 않도록 잘 조정하십시오. 그 후, 표 2에 나열된 온도에서 수조(그림 6A, B)에 용기를 넣습니다.
- 수조의 경우 튜브가 시스템 내부에 들어가면 타이머를 60분 동안 시작합니다(표 6).
- 반응 시간 후 열 순환기 또는 수조에서 튜브를 제거하고 전기 영동 실행을 위해 4 °C에서 또는 사용할 때까지 -20 °C에서 보관하십시오.
- 비색 반응이 수행된 경우 고품질 카메라를 사용하여 PCR 튜브의 사진을 촬영합니다. HNB의 최종 색상은 하늘색입니다.
3. 아가로스 겔의 증폭 산물 분석
참고: 이러한 단계는 비색 반응을 위한 추가 검사 또는 표준화 단계 중 성능 제어로 제안됩니다. 이는 이 기술이 이러한 테스트를 수행하는 실험실에 큰 오염 위험을 초래할 수 있기 때문입니다.
- 가장자리 고무가 벽에 닿도록 베드를 전기영동 챔버 내부에 배치하여 아가로스(내부 챔버)를 추가할 수 있는 밀폐된 공간을 만듭니다(그림 3A, B).
- 3.1단계를 완료한 후 500mL 비커에 필요한 양의 아가로스를 칭량하여 2% 겔을 얻습니다. 그 후, 필요한 부피의 0.5x 트리스 아세테이트 EDTA(TAE) 완충액을 추가하고 1-2분 동안 전자레인지에 넣습니다.
알림: 아가로스는 오븐에서 꺼낼 때 덩어리가 없고 반투명하면 완전히 녹습니다. 이것이 확인되지 않으면 제대로 겔화되지 않은 영역이 남아 증폭 산물의 전기영동 실행 및 시각화가 변경될 수 있습니다. - 오븐에서 비커를 꺼내고 3.1단계에서 만든 내부 챔버에 아가로스를 붓습니다(그림 3C). 그런 다음 기포가 없는지 확인하고 기포가 있으면 마이크로피펫 팁을 사용하여 제거합니다.
- 빗을 배열하여 웰을 형성하고 아가로스를 실온(RT)에서 약 30분 동안 겔에 둡니다.
- 이 시간이 끝나면 5mL의 0.5x TAE 완충액을 추가하여 빗과 겔이 들어있는 베드를 쉽게 제거할 수 있습니다. 그런 다음 웰이 양극에 오도록 겔을 배치합니다(그림 3D).
- 제조업체가 지정한 용량까지 0.5x TAE 버퍼로 전기영동 챔버를 채우고 전극이 버퍼와 접촉하도록 합니다.
- 겔의 첫 번째 웰에 3μL의 분자량 마커를 추가하고 9μL의 NTC와 각 샘플을 다음 웰에 추가합니다. 7 μL의 증폭 산물과 3 μL의 로딩 버퍼를 결합하여 만듭니다. 그런 다음 이 혼합물 9μL를 겔의 웰에 로드합니다.
- 전기영동 챔버를 뚜껑으로 덮고 케이블을 색상 패턴으로 전원 공급 장치 포트에 연결합니다. 전원을 다음 매개변수로 설정하십시오: 100V 및 일정한 암페어 120분 동안.
- 전기영동 실행이 완료된 후 염색 용액(에티듐 브로마이드)이 있는 용기에 겔을 넣고 30분 동안 배양합니다.
- 배양 후 염색 용액에서 젤을 제거하고 지퍼 잠금 백에 넣습니다. 이렇게 하면 앰플리콘을 시각화하는 데 사용될 장비의 오염을 방지할 수 있습니다.
- Amersham Imager 600과 같은 트랜스일루미터 또는 이미저에서 겔을 시각화합니다.
결과
프로토콜의 구현은 위에서 설명한 프로토콜에 따라 각 타겟 유전자에 대한 프라이머 세트를 설계하는 것으로 시작됩니다. 2020년 6월, NextStrain 데이터베이스에서 5,000개의 SARS-CoV-2 게놈을 얻었으며 콜롬비아 게놈의 10%를 대표했습니다. 이러한 염기서열은 프라이머 설계 과정에서 사용된 합의 염기서열을 얻기 위해 정렬되었습니다. 표 1 은 프라이머 RdRp/Hel 및 RdRp에 대해 선택된 프라이?...
토론
RT-LAMP는 분자 진단을 수행하기 위한 보완적인 방법론으로 간주되지만, 프로토콜을 표준화하고 구현할 때 고려해야 하는 몇 가지 제한 사항과 중요한 단계도 있습니다.
SARS-CoV-2 검출을 위한 LAMP 표준화는 마스터 믹스에서 다음 매개변수와 구성 요소를 평가했습니다: (a) 프라이머의 농도 및 정렬 온도; (b) 효소의 농도; (c) 마그네슘 농도; (d) 반응 시간; (e) BSA, DMSO, 구아니디늄 클?...
공개
나탈리아 캄필로-페드로자(Natalia Campillo-Pedroza)는 BioDx: Diagnóstico y Soluciones Biotecnológicas S.A.S.의 CEO입니다. 나머지 저자는 이해 상충이 없다고 선언합니다.
감사의 말
이 연구는 콜롬비아의 Sistema General de Regalías(보조금 번호 BPIN 2020000100092)와 Universidad Icesi - Convocatoria Interna(보조금 번호 CA0413119)의 지원을 받았습니다. MFVT는 또한 Universidad de los Andes의 조교수 기금으로 자금을 지원받았습니다. 자금 지원 기관은 설계, 활동 실행, 데이터 수집, 데이터 분석 및 원고 준비에 참여하지 않았습니다. Sars -CoV-2 샘플의 바이러스 RNA를 제공해 준 Fundación Valle del Lili 대학병원과 원고에 대한 의견을 제시해 준 Dr. Alvaro Barrera-Ocampo에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 kb DNA Ladder | SOLIS BIODYNE | 07-12-00050 | Store at -20 °C |
| 50x TAE Electrophoresis Buffer | ThermoScientific | B49 | Store at roome temperature |
| Accuris High Fidelity Polymerase | ACCURIS LIFE SCIENCE REAGENTS | PR1000-HF-200 | It can be used in case Q5 High-Fidelity DNA polymerase cannot be purchased. For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Agarose | PanReacAppliChem | A8963,0100 | N/A |
| Bst 3.0 DNA Polymerase 8000 IU/mL | New England BioLabs | M0374S/M0374L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | Store at -20 °C |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220-25G | Handle it with caution under an extraction cabinet |
| GeneRuler 100 bp Plus DNA Ladder, ready-to-use | ThermoScientific | SM0322 | Store at -20 °C |
| Hydroxy naphthol blue disodium salt | Santa Cruz Biotechnology | sc-215156B | N/A |
| Q5 High-Fidelity DNA polymerase 2000 IU/mL | New England BioLabs | M0491S/M0491L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
| WarmStart RTx Reverse Transcriptase 15000 IU/mL | New England BioLabs | M0380S/M0380L | For the enzyme, make aliquots of an appropriate volume and store at -20 °C until use. |
참고문헌
- World Health Organization. . Who coronavirus (COVID-19) dashboard (no date). , (2023).
- Ibrahim, N. K. Epidemiologic surveillance for controlling Covid-19 pandemic: types, challenges and implications. Journal of Infection and Public Health. 13 (11), 1630-1638 (2020).
- Rojas-Gallardo, D. M., et al. COVID-19 in Latin America: Contrasting phylodynamic inference with epidemiological surveillance. (Molecular epidemiology of COVID-19 in Latin America). medRxiv. , (2020).
- Liu, R., et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clinica Chimica Acta. 505, 172-175 (2020).
- Kevadiya, B. D., et al. Diagnostics for SARS-CoV-2 infections. Nature Materials. 20 (5), 593-605 (2021).
- Tomita, N., Mori, Y., Kanda, H., Notomi, T. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nature Protocols. 3 (5), 877-882 (2008).
- Li, Y., Fan, P., Zhou, S., Zhang, L. Loop-mediated isothermal amplification (LAMP): A novel rapid detection platform for pathogens. Microbial Pathogenesis. 107, 54-61 (2017).
- Notomi, T., Mori, Y., Tomita, N., Kanda, H. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. Journal of Microbiology. 53 (1), 1-5 (2015).
- Augustine, R., et al. Loop-mediated isothermal amplification (LAMP): A rapid, sensitive, specific, and cost-effective point-of-care test for coronaviruses in the context of COVID-19 pandemic. Biology (Basel). 9 (8), 182 (2020).
- . Nextstrain Available from: https://nextstrain.org/ (2023)
- . Neb Lamp, NEB LAMP Available from: https://lamp.neb.com/ (2023)
- . Blast: Basic local alignment search tool (no date) Available from: https://blast.ncbi.nlm.nih.gov/ (2023)
- Zhang, Y., et al. Rapid molecular detection of SARS-CoV-2 (COVID-19) virus RNA using colorimetric LAMP. medRxiv. , (2020).
- Lu, R., et al. Development of a novel reverse transcription loop-mediated isothermal amplification method for rapid detection of SARS-CoV-2. Virologica Sinica. 35 (3), 344-347 (2020).
- Najafov, A., Hoxhaj, G. . PCR Guru. , (2017).
- Zhang, Y., et al. Enhancing colorimetric loop-mediated isothermal amplification speed and sensitivity with guanidine chloride. Biotechniques. 69 (3), 178-185 (2020).
- Ramírez-Chavarría, R. G., et al. Automatic analysis of isothermal amplification via impedance time-constant-domain spectroscopy: A SARS-CoV-2 case study. Chemosensors. 11 (4), 230 (2023).
- Haque, M. F. U., et al. A novel RdRp-based colorimetric RT-LAMP assay for rapid and sensitive detection of SARS-CoV-2 in clinical and sewage samples from Pakistan. Virus Research. 302, 198484 (2021).
- Donia, A., et al. Integration of RT-LAMP and microfluidic technology for detection of SARS-CoV-2 in wastewater as an advanced point-of-care platform. Food and Environmental Virology. 14, 364-373 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유