JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
EEG 및 심혈관 신호를 공동으로 분석하기 위한 오픈 소스 EEGLAB 플러그인인 BrainBeats
요약
BrainBeats 도구 상자는 EEG 및 심혈관(ECG/PPG) 신호를 공동으로 분석하도록 설계된 오픈 소스 EEGLAB 플러그인입니다. 여기에는 HEP(Heartbeat-evoked Potentials) 평가, 기능 기반 분석 및 EEG 신호에서 심장 아티팩트 추출이 포함됩니다. 이 프로토콜은 두 개의 렌즈(HEP 및 기능)를 통해 뇌-심장 상호 작용을 연구하는 데 도움이 되어 재현성과 접근성을 향상시킵니다.
초록
뇌와 심혈관계 간의 상호 작용은 인체 생리학에 대한 이해를 높이고 건강 결과를 개선할 수 있는 잠재력으로 인해 점점 더 많은 주목을 받고 있습니다. 그러나 이러한 신호의 멀티모달 분석은 지침, 표준화된 신호 처리 및 통계 도구, 그래픽 사용자 인터페이스(GUI), 대규모 데이터 세트를 처리하거나 재현성을 높이기 위한 자동화의 부재로 인해 까다롭습니다. 표준화된 EEG 및 심박변이도(HRV) 특징 추출 방법에는 또 다른 공백이 존재하여 임상 진단 또는 기계 학습(ML) 모델의 견고성을 훼손합니다. 이러한 제한 사항에 대응하여 BrainBeats 도구 상자를 소개합니다. 오픈 소스 EEGLAB 플러그인으로 구현된 BrainBeats는 세 가지 주요 프로토콜을 통합합니다: 1) 밀리초 정확도로 시간 고정 뇌-심장 상호 작용을 평가하기 위한 심장 박동 유발 전위(HEP) 및 진동(HEO); 2) 다양한 뇌 및 심장 지표 간의 연관성/차이를 조사하거나 강력한 특징 기반 ML 모델을 구축하기 위한 EEG 및 HRV 특징 추출; 3) EEG 분석을 수행하는 동안 잠재적인 심혈관 오염을 제거하기 위해 EEG 신호에서 심장 아티팩트를 자동으로 추출합니다. 이 세 가지 방법을 동시 64채널 EEG, ECG 및 PPG 신호를 포함하는 오픈 소스 데이터 세트에 적용하기 위한 단계별 자습서를 제공합니다. 사용자는 그래픽 사용자 인터페이스(GUI) 또는 명령줄을 사용하여 고유한 연구 요구 사항에 맞게 매개변수를 쉽게 미세 조정할 수 있습니다. BrainBeats는 뇌와 심장의 상호 작용 연구에 더 쉽게 접근하고 재현할 수 있도록 해야 합니다.
서문
오랫동안 환원주의적 접근은 인간의 생리학과 인지에 대한 과학적 탐구를 지배해 왔다. 이 접근 방식에는 복잡한 신체 및 정신 과정을 더 작고 관리하기 쉬운 구성 요소로 해부하여 연구원들이 개별 시스템에 독립적으로 집중할 수 있도록 하는 것이 포함되었습니다. 이 전략은 인간의 몸과 마음의 복잡하고 상호 연결된 특성을 연구하는 데 어려움이 있었기 때문에 생겨났습니다 1. 환원주의는 신경2 또는 심장3 통신에 대한 이온 채널과 활동 전위의 역할을 설명하는 것과 같이 개별 하위 시스템을 고립되어 이해하는 데 중요한 역할을 했습니다. 그러나 이러한 고립된 시스템이 더 큰 공간적, 시간적 규모에서 어떻게 상호 작용하는지에 대한 우리의 이해에는 상당한 격차가 남아 있습니다. 다중(통합적 또는 생태적) 틀은 인체를 복잡한 다차원적 시스템으로 간주하며, 여기서 마음은 뇌의 산물이 아니라 생명체의 활동, 즉 인체의 일상 기능 내에서 뇌를 통합하는 활동으로 간주한다4. 멀티모달(multimodal)과 환원주의(reductionist) 접근법은 배타적이지 않은데, 이는 우리가 전체 뇌 없이 하나의 뉴런을 연구할 수 없고, 개별 뉴런의 특성을 이해하지 않고는 전체 뇌를 연구할 수 없는 것과 같다. 그들은 함께 인간의 건강, 병리학, 인지, 심리학 및 의식에 대한 보다 포괄적이고 시너지 효과를 내는 이해를 위한 길을 닦습니다. 본 방법은 뇌파검사(EEG)와 심혈관 신호, 즉 심전도(ECG) 및 광혈류측정법(PPG)의 공동 분석을 제공함으로써 뇌와 심장 사이의 상호 작용에 대한 다중 모드 연구를 용이하게 하는 것을 목표로 합니다. MATLAB에서 EEGLAB 플러그인으로 구현된 이 툴박스는 기존의 방법론적 한계를 해결하고 과학 분야의 접근성과 재현성을 용이하게 하기 위해 오픈 소스로 만들어졌습니다. 최신 지침과 권장 사항을 설계 및 기본 매개 변수에 구현하여 사용자가 알려진 모범 사례를 따르도록 권장합니다. 제안된 도구 상자는 1) 심장 박동 유발 전위 연구, 2) EEG 및 ECG/PPG 신호에서 특징 추출, 3) EEG 신호에서 심장 아티팩트 제거에 관심이 있는 연구자 및 임상의에게 귀중한 리소스가 될 것입니다.
심장-뇌 연구
심장과 뇌의 관계는 기능적 자기 공명 영상(fMRI) 및 양전자 방출 단층 촬영(PET)과 같은 신경 영상 방법을 통해 역사적으로 연구되어 왔습니다. 연구진은 이러한 도구를 사용하여 심혈관 조절과 관련된 일부 뇌 영역(예: 심박수및 혈압 조작5)을 강조하거나, 심박수가 BOLD 신호6에 미치는 영향을 보여주거나, 관상동맥 심장 질환(즉, 스트레스 유발 혈압7)에 기여할 수 있는 뇌-신체 경로를 식별했다). 이러한 연구는 중추신경계(CNS)와 심혈관 기능 간의 복잡한 상호 작용에 대한 이해를 크게 향상시켰지만, 이러한 신경 영상 기법은 비용이 많이 들고 가용성이 제한적이며 통제된 실험실 환경에 국한되어 있어 실제 및 대규모 응용 분야에 대한 실용성이 제한됩니다.
이와는 대조적으로, EEG 및 ECG/PPG는 더 다양한 환경과 인구 집단 또는 장기간에 걸쳐 뇌-심장 상호 작용을 연구할 수 있는 잠재력을 제공하여 새로운 기회를 제공하는 더 저렴하고 휴대 가능한 도구입니다. ECG는 심장이 수축하고 이완할 때 피부(일반적으로 가슴 또는 팔)에 배치된 전극을 통해 각 심장 박동에서 생성되는 전기 신호를 측정합니다8. PPG는 광원(예: LED)과 광검출기(일반적으로 손가락 끝, 손목 또는 이마에 배치)를 사용하여 미세혈관 조직의 혈액량 변화(즉, 혈류 및 맥박수)를 측정하며, 혈액이 주변 조직보다 더 많은 빛을 흡수하는 방식에 의존합니다9. 두 방법 모두 심혈관 기능에 대한 중요한 정보를 제공하지만 용도가 다르고 고유한 데이터 유형을 제공합니다. ECG와 마찬가지로 EEG는 세포외 기질, 조직, 두개골 및 두피를 통해 전파되는 수천 개의 대뇌 피질 뉴런의 동기화 된 활동에 의해 생성 된 전기장을 기록하여 두피 표면(10)에 배치 된 전극에 도달합니다. 따라서 EEG 및 ECG/PPG의 사용은 뇌-심장 상호 작용의 기저에 있는 생리적, 인지적, 정서적 과정과 인간의 건강과 웰빙에 미치는 영향에 대한 이해를 증진시키는 데 큰 가능성을 가지고 있습니다. 따라서 BrainBeats 도구 상자를 사용하여 EEG, ECG/PPG 신호에서 심장-뇌 상호 작용을 캡처하는 것은 임상 진단 및 예측, 빅 데이터 머신 러닝(ML), 실제 자가 모니터링11 및 모바일 뇌/신체 영상(MoBI)12,13과 같은 과학 분야에 특히 유용할 수 있습니다.
EEG 및 ECG 신호를 공동으로 분석하기 위한 두 가지 접근 방식
EEG와 심혈관 신호 간의 상호 작용을 연구하는 두 가지 주요 접근 방식이 있습니다.
시간 도메인: 이벤트 관련 전위(ERP)의 심장 박동 유발 전위(HEP) 및 시간-주파수 영역의 심장 박동 유발 발진(HEO): 이벤트 관련 스펙트럼 섭동(ERSP) 및 시험 간 일관성(ITC). 이 접근법은 뇌가 각 심장 박동을 어떻게 처리하는지 조사합니다. 밀리초(ms) 정확도로 이 방법을 사용하려면 두 시계열이 완벽하게 동기화되고 심장 박동이 EEG 신호에 표시되어야 합니다. 이 접근법은 최근 몇 년 동안 관심을 끌었습니다 14,15,16,17,18,19.
특징 기반 접근법: 이 접근법은 연속 신호에서 EEG 및 심박 변이도(HRV) 특징을 추출하고 이들 간의 연관성을 검사합니다. 이것은 EEG (종종 정량적 EEG 또는 qEEG20라고 불림), ECG 21,22,23 및 PPG 24,25,26에 대해 독립적으로 수행되었습니다. 이 접근 방식은 상태 및 특성 관련 변수를 모두 캡처하여 유망한 응용 프로그램을 제시합니다. EEG 및 심혈관 신호 모두에 대해 기록이 길수록 특성 변수 27,28,29가 더 우세합니다. 따라서 응용 프로그램은 기록 매개변수에 따라 달라집니다. 특징 기반 분석은 정신 및 신경 장애, 치료 반응 또는 재발을 예측하기 위한 새로운 정량적 지표를 제공하여 점점 더 많은 관심을 받고 있습니다 30,31,32,33,34,35. 이 접근법은 대규모 실제 데이터 세트(예: 클리닉, 원격 모니터링)에서 특히 매력적이며, 이는 웨어러블 신경 기술11의 최근 혁신 덕분에 더 쉽게 얻을 수 있습니다. 덜 탐구된 응용 분야는 특정 뇌와 심장 기능 간의 연관성을 식별하여 중추 신경계 역학의 기저에 있는 잠재력을 강조하는 것입니다. 심박 변이도(HRV)는 ECG 및 PPG 신호 모두에서 계산할 수 있습니다. 자율 신경계(ANS)에 대한 정보를 제공하여 심장 박동 사이의 시간 간격(즉, 정상에서 정상까지의 간격)의 변화를 측정합니다27. 교감신경(SNS) 활동이 증가하면(예: 스트레스 또는 운동 중) 일반적으로 HRV가 감소하는 반면, 부교감신경(PNS) 활동(예: 이완 중)은 HRV를 증가시킵니다. 느린 호흡 속도는 일반적으로 PNS 활동이 증가하여 HRV를 증가시키며, 특히 짧은 녹음(<10분)의 경우 더욱 그렇습니다27. HRV 점수가 높을수록 일반적으로 더 탄력적이고 적응력이 뛰어난 ANS를 나타내는 반면, HRV가 낮으면 스트레스, 피로 또는 근본적인 건강 문제를 나타낼 수 있습니다. 장시간 HRV 기록(즉, 최소 24시간)은 심혈관 질환, 스트레스, 불안 및 일부 신경학적 상태를 포함한 다양한 건강 상태에 대한 예측 예후를 제공한다27. 혈압, 심박수 또는 콜레스테롤 수치와 같은 측정은 심혈관계의 상태에 대한 정보를 제공합니다. 이와는 대조적으로, HRV는 심장이 스트레스에 어떻게 반응하고 스트레스로부터 회복되는지를 보여주는 역동적인 측면을 추가합니다.
기존 방법에 비해 BrainBeats의 장점
아래에서 검토한 바와 같이 심혈관 및 EEG 신호를 서로 독립적으로 처리할 수 있는 도구가 존재하지만 공동으로 분석할 수는 없습니다. 더욱이, 심혈관 신호를 처리하는 데 사용 가능한 대부분의 수단은 비용이 많이 드는 라이선스를 필요로 하거나, 자동화된 처리를 허용하지 않거나(특히 대규모 데이터 세트에 유용함), 투명성과 재현성을 방해하는 독점 알고리즘을 가지고 있거나, 그래픽 사용자 인터페이스(GUI)를 제공하지 않음으로써 고급 프로그래밍 기술을 필요로 합니다36. 우리가 아는 한, GUI를 통한 HEP/HEO 분석을 지원하는 4개의 오픈 소스 MATLAB 툴박스, 즉 ecg-kit 툴박스(37), BeMoBIL 파이프라인(38), HEPLAB EEGLAB 플러그인(39), CARE-rCortex 툴박스(40)가 있습니다. HEPLAB, BeMoBI 및 ecg-kit는 심장 박동을 감지하고 EEG 신호에 표시하여 HEP 분석을 용이하게 하지만 통계 분석을 제공하지 않거나 시간 영역(즉, HEP)으로 제한됩니다. CARE-rCortex 플러그인은 ECG 및 호흡 신호, 시간-주파수 영역 분석, 통계, HEP/HEO 분석에 적합한 고급 기준선 정규화 및 보정 방법을 지원하여 이러한 문제를 해결했습니다. 그러나 유형 1 오류(즉, 거짓 양성)의 통계적 보정을 위해 Bonferroni 방법을 사용하며, 이는 EEG 응용 프로그램에 대해 너무 보수적이고 생리학적으로 건전하지 않아 유형 II 오류(즉, 거짓 음성)의 증가로 이어집니다41. 또한 이 툴박스는 자동화를 위한 명령줄 액세스를 제공하지 않습니다. 마지막으로, 최근 연구에서는 기준선 보정 방법 42,43,44를 권장하는데, 이는 신호 대 잡음비(SNR)를 줄이고 통계적으로 불필요하고 바람직하지 않기 때문입니다.
이러한 제한 사항을 해결하기 위해 현재 MATLAB 환경에서 오픈 소스 EEGLAB 플러그인으로 구현된 BrainBeats 툴박스를 소개합니다. 이전 방법에 비해 다음과 같은 장점이 있습니다.
1) 사용하기 쉬운 GUI 및 명령줄 기능(자동화된 처리를 수행하려는 프로그래머용). 2) R 피크 감지, RR 아티팩트 보간, HRV 메트릭 계산과 같은 심혈관 신호 처리를 위한 검증된 알고리즘, 매개변수 및 지침(예: 윈도우잉, 재샘플링, 정규화 등에 대한 임플란트 지침)27,45,46). 이는 Vest 등이 이러한 처리 단계에서 미미한 차이가 어떻게 다른 결과로 이어질 수 있는지를 보여주었기 때문에 HRV 지표의 재현성 및 임상적 적용 가능성의 부족에 기여하기 때문이다46. 3) 필터링 및 윈도잉 44,47, 재참조 48,49, 비정상 채널 및 아티팩트 제거(50,51,52), 최적화된 ICA 분해 및 독립 구성 요소 분류 53,54,55,56을 포함한 EEG 신호 처리를 위한 검증된 알고리즘, 기본 매개변수 및 지침. 사용자는 툴박스를 사용하여 필요에 맞게 툴박스를 사용하기 전에 모든 전처리 매개변수를 미세 조정하거나 선호하는 방법으로 EEG 데이터를 전처리할 수도 있습니다(예: EEGLAB clean_rawdata 플러그인50,52, BeMoBIL 파이프라인(38), PREP 파이프라인(57 등) ). 4) ECG 신호의 심장 박동 유발 전위(HEP, 즉 시간 영역) 및 진동(HEO, 웨이블릿 또는 FFT 방법을 사용한 이벤트 관련 스펙트럼 섭동, 시험 간 일관성은 표준 EEGLAB 소프트웨어를 통해 사용 가능). 유형 1 오류에 대한 수정 사항이 있는 파라메트릭 및 비파라메트릭 통계는 EEGLAB의 표준 소프트웨어를 통해 사용할 수 있습니다. 비모수 통계에는 다중 비교를 위한 순열 통계 및 시공간 보정(예: 시공간 군집화 또는 임계값 없는 군집 향상)이 포함됩니다(예: 시공간 군집화 또는 임계값 없는 군집 향상)58,59. 사용자는 LIMO-EEG 플러그인을 사용하여 계층적 선형 모델링을 구현할 수 있으며, 이는 내부 및 피험자 간 분산을 잘 설명하고 유형 I 및 II 오류60,61에 대한 강력한 제어를 통해 가정없는 질량 단변량 접근 방식을 구현합니다. HEP/HEO 데이터 통계 분석은 채널 및 독립 구성 요소 도메인에서 수행할 수 있습니다. 5) PPG 신호에서 HEP/HEO 및 HRV 분석(HEP/HEO에 대한 최초). 6) 최초로 EEG 및 HRV 특징의 공동 추출을 지원합니다. 7) 이 툴박스는 필요한 다양한 처리 단계에서 신호를 검사하고 주제 수준에서 출력을 검사할 수 있는 다양한 데이터 시각화를 제공합니다.
| 메서드 | ECG에서 R-피크 검출 | PPG에서 R파를 감지합니다 | HEP/HEO (헅/HEO) | EEG & HRV 특징 | EEG에서 심장 아티팩트 제거 | GUI (그래픽 인터페이스) | 명령줄 |
| ECG 키트 | X | X | X | X | |||
| 비모빌 | X | X | X | ||||
| 헵랩 | X | X | X | X | |||
| 케어-r코텍스 | X | X | X | X | |||
| 브레인비트 | X | X | X | X | X | X | X |
표 1: 기존의 유사한 방법과 관련하여 BrainBeats가 가져온 참신함.
독자가 이 방법이 자신에게 적합한지 판단하는 데 도움이 되는 정보
이 툴박스는 EEG 및 ECG/PPG 데이터가 있는 모든 연구자 또는 임상의에게 적합합니다. 플러그인은 아직 별도의 파일에서 EEG 및 ECG/PPG 신호 가져오기를 지원하지 않습니다(이 기능은 곧 사용할 수 있음). 이 툴박스는 HEP/HEO 분석을 수행하거나, 표준화된 방법으로 EEG 및/또는 HRV 특징을 추출하거나, 단순히 EEG 신호에서 심장 인공물을 제거하려는 모든 사람에게 적합합니다. BrainBeats의 전반적인 흐름과 방법을 요약한 블록 다이어그램은 그림 1 을 참조하십시오.
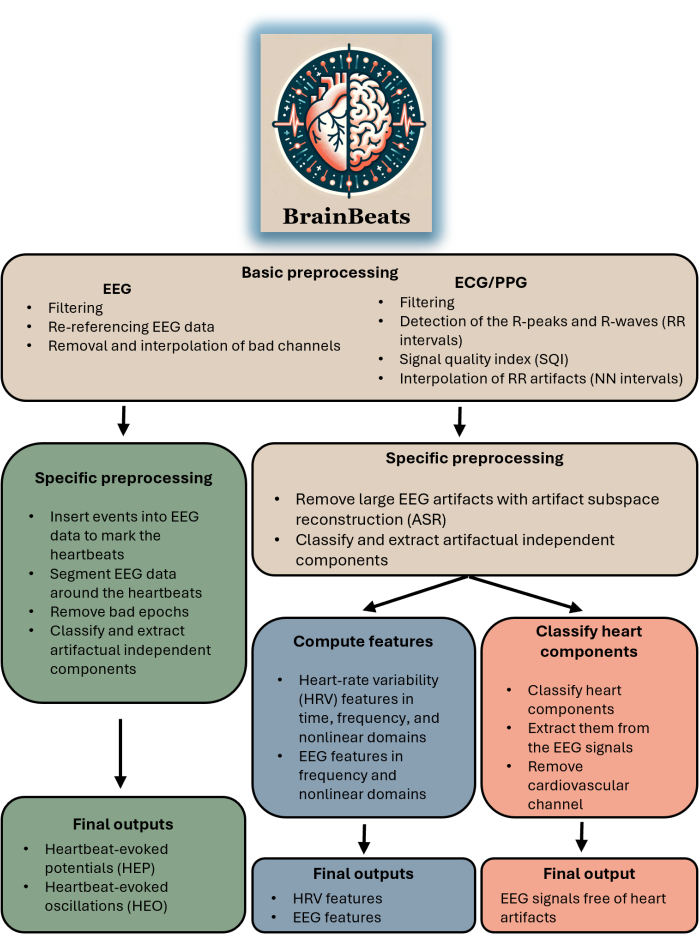
그림 1. BrainBeats의 전체 아키텍처와 흐름을 요약한 블록 다이어그램. 세 가지 방법에서 공통적인 작업은 갈색입니다. HEP(Heartbeat-evoked Potentials) 및 HEO(Oscillations)와 관련된 작동은 녹색입니다. EEG 및 HRV 특징 추출과 관련된 작업은 파란색입니다. EEG 신호에서 심장 인공물을 제거하는 것과 관련된 작업은 빨간색입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
각 참가자로부터 정보에 입각한 동의를 얻었고, 우랄 연방 대학 윤리 위원회는 실험 프로토콜을 승인했습니다.
1. BrainBeats 요구 사항
- 컴퓨터에 MATLAB과 EEGLAB을 설치합니다. EEGLAB은 https://github.com/sccn/eeglab 에서 다운로드할 수 있으며 컴퓨터의 어느 곳에서나 압축을 풀거나 Git 사용자를 위해 복제할 수 있습니다. 설치에 대한 자세한 내용은 GitHub 페이지를 참조하십시오.
- 경로 설정 버튼을 클릭하여 MATLAB의 홈 패널에 EEGLAB 폴더의 경로를 추가합니다. 압축을 푼 파일이 있는 eeglab 폴더를 선택하고 저장 > 닫기를 클릭합니다.
- MATLAB의 명령 창에 eeglab 을 입력하여 EEGLAB을 시작합니다.
- 파일 > EEGLAB 확장 관리를 클릭하여 BrainBeats 플러그인을 설치합니다. 검색 창에 brainbeats를 입력하고 목록에서 BrainBeats 플러그인을 선택한 다음 설치/업데이트를 클릭합니다.
- 샘플 데이터 세트를 EEGLAB에 로드합니다. File(파일) > Load existing data(기존 데이터 로드)를 클릭합니다. EEGLAB 폴더로 이동하고, 플러그인 폴더로 이동하고, BrainBeats 폴더로 이동하여 sample_data 폴더를 엽니다. dataset.set 파일을 선택합니다.
참고: 이 데이터셋은 오픈 소스 다중 주제 데이터셋62,63의 sub-032(눈을 뜨고 쉬고 있는 상태)에 해당합니다. 1000Hz 샘플링 속도에서 동시 EEG(64개 채널), ECG(1개 채널) 및 PPG(1개 채널)가 포함되어 있기 때문에 선택되었습니다. ECG 및 PPG 신호는 actiCHamp 시스템의 보조 입력을 사용하여 수집되었습니다. EEG 데이터는 10-20 시스템에 따라 배치된 활성 전극으로 기록되었으며, FCz는 온라인 기준으로, Fpz는 접지 전극으로, 임피던스는 25kOm 미만으로 유지되었습니다. ECG는 오른쪽 손목에 배치된 하나의 활성 전극, 왼쪽 손목에 있는 기준 전극, 팔꿈치에서 원위부 3cm에 있는 왼쪽 안쪽 팔뚝의 지면을 사용하여 기록되었습니다. PPG는 왼쪽 검지에서 기록되었습니다. EEG, ECG 및 PPG 데이터는 동일한 증폭기로 동시에 기록되었기 때문에 시간 동기화됩니다. 자세한 내용은 참조62,63을 참조하십시오. - Save Outputs(출력 저장) 확인란을 선택하여 EEGLAB에 로드된 원본 파일과 동일한 위치에 해당 .set 파일의 모든 항목을 저장합니다.
참고: 이 자습서에서는 EEG, ECG 및 PPG 데이터를 하나의 EEGLAB 데이터 세트로 병합하고, 3D BEM(경계 요소 방법) 전극 좌표를 로드하고, 신호를 250Hz로 다운샘플링하여 파일 용량(사용자 다운로드용)을 줄이고 컴퓨팅 시간을 단축했습니다. 이 데이터 세트에는 비정상적인 EEG 채널이 없었기 때문에 BrainBeats의 불량 채널 감지 및 제거 알고리즘을 설명하기 위해 채널 TP9를 인위적으로 수정했습니다. 마찬가지로, BrainBeats의 아티팩트 제거 기능을 설명하기 위해 파일 시작 부분의 큰 EEG 아티팩트와 3-6초의 시간 채널에서 고주파 근육 같은 아티팩트를 인위적으로 시뮬레이션합니다.
2. 심장 박동 유발 전위(HEP) 및 진동(HEO)
- BrainBeats의 첫 번째 일반 사용자 인터페이스(GUI)를 열어 주요 매개변수를 선택합니다. EEGLAB에서 도구 > BrainBeats >1st level (subject level)을 클릭합니다. 실행할 분석으로 Heartbeat-evoked potential HEP 를 선택하고, 심장 데이터 유형으로 ECG 를 선택하고, 버튼을 클릭하여 ECG라고 표시된 ECG 채널을 선택할 채널 목록을 표시합니다(또는 텍스트 상자에 직접 입력). Visualize outputs (recommended)(출력 시각화(권장) ) 및 Save outputs(출력 저장 ) 옵션을 선택된 상태로 유지하고 Ok(확인)를 클릭합니다.
- 이전 선택 사항(예: ECG 데이터를 사용한 HEP 분석)에 따라 두 번째 GUI 창이 나타납니다. EEG 및 ECG 신호 모두에 대한 전처리 파라미터를 선택합니다. ECG 전처리 및 EEG 전처리 상자의 선택을 취소하여 전처리를 해제하여 BrainBeats를 시작하기 전에 데이터가 이미 전처리되었는지 확인합니다. 이 EEG 데이터는 러시아에서 기록되었으므로 전처리 EEG 섹션에서 전력선 소음을 50Hz로 변경합니다. 딸깍 하는 소리 OK 시작합니다.
- 감지된 PPG 채널을 제거할지 확인하는 경고 메시지가 나타납니다. 툴박스는 ECG와 PPG를 동시에 분석(또는 다른 보조 채널)하도록 설계되지 않았고 데이터 세트에 보관하면 심각한 오류(예: 아티팩트 제거, ICA 분해 불량 등)가 발생하기 때문입니다. Yes(예)를 클릭합니다. BrainBeats는 몇 가지 검사를 수행하고, 몇 가지 기본 매개 변수를 설정하고, ECG 신호를 전처리하고 RR 간격을 계산하기 위해 EEG 데이터에서 ECG를 분리합니다.
- ECG 및 RR 시계열은 BrainBeats의 데이터 형식, 명확성 향상, 매개변수 튜닝 및 컴퓨팅 시간에 맞게 조정된 Physionet Cardiovascular Signal 도구 상자46,64의 검증된 알고리즘을 사용하여 전처리됩니다(알고리즘 검증을 위한 참조 참조). 플러그인은 RR 간격, 타임스탬프, 필터링된 ECG 신호, R-피크 지수 및 심박수(HR)를 출력합니다. GUI 또는 명령줄을 통해 이러한 매개변수를 조정합니다.
참고: ECG 신호는 검증된 맞춤형 필터(1-30Hz)를 사용하여 대역 통과 필터링되고 신호를 스캔하여 Pan-Tompkins(P&T) 방법65를 사용하여 QRS 복합파 및 R-피크를 식별하고, 최상의 성능을 위해 미분, 제곱, 적분 및 평활화를 포함한 일부 신호 처리 작업을 구현합니다. P&T 에너지 임계값은 큰 충돌로 인한 중단을 방지하기 위해 샘플 속도와 평활화된 ECG 값을 기반으로 추정됩니다. RR 구간 변동성이 중앙값의 1.5배를 초과하면 누락된 피크를 검색합니다. 평균 R-피크 기호는 30초 세그먼트에 걸쳐 계산되며, 피크 포인트는 내화 기간 검사를 통해 정제되어 플랫라인 조건을 관리하고 일관된 감지를 보장합니다. - 다음으로, BrainBeats는 앞뒤로 검색 및 생리학적 임계값을 사용하여 비정상적인 RR 간격 또는 RR 간격 내의 급증을 식별합니다. 신호 품질 지수(SQI)는46으로 계산됩니다. RR 시계열의 20% 이상에 RR 아티팩트(생리학적 한계를 벗어나거나 SQI가 0.9 미만)가 포함된 경고를 표시하는 경우 이를 확인합니다. 플롯에는 필터링된 ECG 신호, 식별된 R-피크, NN 간격 및 보간된 아티팩트가 표시됩니다( 그림 2 참조).
참고: RR 아티팩트는 기본적으로 NN(Normal-to-Normal) 간격을 얻기 위해 모양 보존 조각별 3차 방법을 사용하여 보간되지만 제거하거나(권장하지 않음) 필요한 경우 다른 보간 방법(선형, 3차, 최근접 이웃, 이전/다음 이웃, 스플라인, 3차 컨볼루션 또는 수정된 Akima 3차)을 사용합니다. 여러 ECG 채널이 있는 경우 각각에 대해 RR 간격이 추정되고 다음 단계에 대해 RR 아티팩트 수가 가장 적은 채널이 선택됩니다. - 오른쪽/왼쪽 화살표를 눌러 자세히 관찰하려면 R-피크의 확대된 30초 창을 스크롤합니다. 데이터에 여러 ECG/PPG 채널이 포함된 경우 RR 간격에 대해 신호 품질 지수가 가장 좋은 채널을 사용합니다. BrainBeats는 현재 ECG와 PPG 신호를 동시에 지원하지 않습니다. 자습서를 위해 두 데이터 형식을 모두 포함하는 샘플 데이터 세트를 선택했습니다.
- ECG 신호가 완료되면 BrainBeats 대역 통과는 기본적으로 비선형 인과 최소 위상 FIR 필터를 사용하여 1-40Hz에서 EEG 데이터를 필터링하여 심장 박동 전후 기간 사이의 번짐 활동을 줄이고 인과 관계를 보존하며 원치 않는 그룹 지연을 방지합니다44. 이는 하트비트 전 기간을 검사하는 사용자에게 특히 중요합니다. 저역 통과 필터가 전력선 주파수보다 높은 값으로 설정된 경우(예: 전력선 주파수가 50Hz인 80Hz 저역 통과) 날카로운 노치 필터를 사용하여 선 노이즈 아티팩트를 제거합니다. 그런 다음 EEG 데이터는 30개 미만의 채널이 감지되지 않는 한 REST 알고리즘(HEP 분석(49)에 가장 적합)을 사용하여 무한대로 재참조됩니다(이 경우 신뢰할 수 있게 재참조될 수 없으며 사용자에게 알리기 위해 경고가 생성됨).
- 그런 다음 BrainBeats는 비정상적인 EEG 채널을 감지, 제거 및 보간합니다(그림 3). 기본 매개변수가 5초보다 큰 평평한 선(clean_flatlines 알고리즘), 최대 고주파 노이즈 표준 편차 10, 창 길이 5초(저속 주파수 아티팩트52를 더 잘 캡처하기 위해), 인접 채널 간의 최소 상관 관계 .65 및 최대 허용 부분 33%(clean_channels 알고리즘)인지 확인합니다.
참고: RANSAC 샘플 수는 기본적으로 500개로 설정되어 불량 채널 제거의 수렴 및 재현성을 높입니다(계산 시간이 증가하더라도). - 다음으로, R-피크를 이벤트 마커로 EEG 데이터에 삽입하여 각 하트비트를 표시하고 기준선 제거 없이 이러한 마커 주변의 데이터를 분할합니다(지침43,66에 따라; 그림 4). NN 간격은 길이가 다르고 EEG는 일정한 길이로 분할되어야 하므로 IBI(Interbeat-Interval) 데이터의 5번째 백분위수(즉, 가장 짧은 IBI의 5%가 떨어지는 값, 히스토그램에서 빨간색 점선으로 표시됨, 그림 5 참조)를 사용하여 R-peak 이벤트 후 최소 epoch 크기 컷오프를 추정합니다.
참고: 이 5번째 백분위수 값은 HEP/HEO 분석에 대한 관심 기간이 하트비트49,67 후 200-600ms이므로 너무 짧지 않도록 하면서 가능한 한 많은 epoch를 보존하기 위한 좋은 절충안입니다. - R-피크 -300ms 전에서 R-피크 이후 5번째 백분위수 값까지 EEG 데이터를 세그먼트화하고 시간 0의 R-피크를 사용합니다. 지침49,67에 따라 550ms보다 짧거나 둘 이상의 R-피크(ERP/ERSP를 바이어스할 수 있음)를 포함하는 epoch를 거부합니다. RMS(Root-Mean-Square) 및 SNR(Signal-to-Noise Ratio) 메트릭과 MATLAB의 isoutlier 함수를 사용하여 큰 EEG 아티팩트를 포함하는 Epoch를 감지합니다(그림 6). 아티팩트 시대가 제거됩니다.
- 기본 Infomax 알고리즘을 사용하여 블라인드 소스 분리를 수행하고 최상의 성능을 위한 데이터 순위를 고려합니다53. 원하는 경우, Picard(빠른) 옵션을 선택하여 빠른 계산54,54를 위한 사전 조건화된 독립 구성 요소 분석(PICARD) 알고리즘을 선택하거나 필드 ICA 방법에 대해 Modified Infomax(길지만 복제 가능) 옵션을 선택하여 수정된 Infomax 알고리즘을 선택합니다(이 작업은 훨씬 더 긴 계산 시간을 포함함). ICLabel 플러그인56을 사용하여 IC를 자동으로 분류하여 비뇌 인공물을 추출합니다(안구 구성 요소는 90% 신뢰도로 제거되는 반면, 근육, 선 잡음 및 채널 잡음은 99% 신뢰도로 제거됨; 그림 7).
- 첫 번째 GUI 창에서 Visualize outputs 옵션을 선택한 상태로 유지하여 최종 EEG 시계열, 그랜드 평균 HEP(그림 8 상단), 각 하트비트에 대한 HEP(그림 8 하단) 및 그랜드 평균 하트비트 유발 진동(HEO; 그림 9). 이벤트 관련 스펙트럼 섭동(ERSP, 즉, 심장 박동에 걸친 EEG 전력의 변화) 측면에서 HEO를 조사합니다. 그림 9 상단) 및 시험 간 위상 일관성(ITC, 즉, 심장 박동에 걸친 위상 각도의 일관성; 그림 9 하단).
참고: ERSP는 기본 3-사이클 웨이블릿(Hanning-tapered 윈도우 적용, 패드 비율 2)을 사용하여 계산되며, 더 높은 주파수에 사용되는 웨이블릿의 사이클 수가 가장 높은 주파수(1 - 0.8)에서 등가 FFT 윈도우의 사이클 수의 최대 20%까지 천천히 확장됩니다. 이는 함수에 의해 측정된 개별 창의 모양과 결과 시간/빈도 창에서 해당 모양을 제어합니다. 설명을 위해 임의의 기준선이 제거되고, ERSP는 문헌에 기술된 전형적인 HEO 효과, 즉 전두중심 전극(17,67)에 걸친 알파 대역(8-13Hz)에서 300-450ms 후 심장 박동을 포착하기 위해 주파수 7-25Hz에 대해 계산됩니다. 더 낮은 주파수는 interbeat intervals에 의해 정의되는 짧은 epoch 크기 때문에 추정할 수 없습니다. 비모수(순열) 통계는 p-값 0.05에 대한 HEO를 시각화하기 위해 적용되며, 이는 거짓 발견률(FDR, 즉, 유형 1 오류 또는 패밀리별 오류)에 대해 수정됩니다. 이러한 플롯은 튜토리얼 목적 또는 단일 시행 분석을 위해 생성됩니다. - 전처리 플롯은 여러 단계를 시각화하기 위해 생성됩니다. 이 기능을 끄려면 Visualize preprocessings 상자를 선택 취소합니다. 최종 EEG 데이터(R-피크 주위로 정리 및 분할)에는 ERP/ERSP 분석이 편향될 수 있으므로 ECG 데이터가 포함되지 않습니다. 최종 출력에서 심장 채널을 보존하려면 심장 채널 유지 상자를 선택합니다.
참고: 필요한 경우 다음 파일(다음 조건 또는 참가자)을 처리하기 전에 여기에서 일시 중지합니다. - BrainBeats는 EEGLAB의 기록 기능을 지원합니다. 모든 작업이 끝나면 MATLAB의 명령 창에 eegh 를 입력하여 GUI에서 수동으로 선택한 파라미터를 사용하여 단일 명령줄을 통해 위의 모든 단계를 반복하는 명령줄을 인쇄하므로 쉽게 자동화할 수 있습니다. EEGLAB 구조에서 전처리 출력(예: 심혈관 시계열의 신호 품질 지수, NN 간격, 제거된 EEG 채널, 세그먼트 및 구성 요소 등)을 찾습니다(EEG.brainbeats.preprocessings). 모든 매개변수는 EEG.brainbeats.parameters에서도 내보내집니다.
- 고급 사용자의 경우 다음 명령줄을 사용하여 기본 매개 변수를 사용하여 위의 모든 단계를 수행합니다(자세한 옵션은 BrainBeats 리포지토리의 자습서 스크립트 참조).
이지랩; 닫다; % GUI 없이 EEGLAB 시작
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('파일 이름','dataset.set',
'파일 경로', 전체 파일 (main_path, 'sample_data')); %샘플 데이터셋 로드
EEG = brainbeats_process(EEG,'분석',
'hep','heart_signal','ECG', 'heart_channels',
{'ECG'},'clean_eeg',참); % BrainBeats1st 레벨을 시작하여 기본 매개변수로 HEP 분석용 파일을 처리합니다. - 위의 단계는 ECG 신호에서 HEP/HEO를 수행했습니다. PPG 신호에 대해 다음 단계를 사용합니다.
- 다음 단계에서는 PPG 신호를 사용하여 동일한 작업을 수행합니다. 이전 작업에서 덮어썼으므로 동일한 데이터 세트를 다시 로드하고(1.5단계 참조) BrainBeats의 첫 번째 GUI를 다시 열어 기본 매개변수를 선택합니다. 도구 > BrainBeats >1st level (subject level)을 클릭합니다. 실행할 분석으로 HEP(Heartbeat-evoked potentials) 를 선택하고, 심장 데이터 유형으로 PPG 를 선택하고, PPG 채널을 선택할 채널 목록을 표시하는 버튼을 클릭합니다. Visualize outputs (recommended)(출력 시각화(권장) ) 및 Save outputs(출력 저장 ) 옵션을 선택된 상태로 유지하고 OK(확인)를 클릭합니다.
- 두 번째 GUI 창은 2.2단계와 유사한 방식으로 팝업됩니다. 유일한 차이점은 이제 ECG 신호 대신 PPG 신호를 처리하도록 매개변수를 미세 조정할 수 있다는 것입니다. OK(확인 )를 클릭하여 기본 매개변수로 실행합니다.
- 감지된 추가 ECG 채널을 제거할지 확인하는 경고 메시지가 나타납니다. 다시 말하지만, 이것은 예상된 것입니다. Yes(예)를 클릭합니다. 기본적으로 이 툴박스는 PPG 신호를 전처리하고, 펄스파를 감지하여 RR 구간을 구하고, RR 아티팩트(있는 경우)를 식별하고, 이를 보간합니다(그림 10). 단계 2.7. 을 2.12로 변경합니다. 가 수행되고 동일한 플롯과 출력이 생성되지만 PPG 신호에서 감지된 R 파를 기반으로 합니다( 그림 11 및 그림 12 참조).
참고: R 파는 지정된 창 내에서 신호의 기울기를 사용하여 감지됩니다. 그런 다음 기울기가 동적 임계값을 초과할 때 전위 펄스에 플래그가 지정되며, 이는 감지 이력 및 신호 특성에 따라 조정됩니다. 그런 다음 알고리즘은 눈을 감는 기간 내에 검색하여 최대 기울기를 정확히 찾아낸 후 임계값을 통해 맥파의 시작을 결정합니다. R파 피크는 시작 근처의 계곡으로 식별되고 해당 위치가 기록됩니다. 이 알고리즘은 전체 신호를 반복하여 감지 임계값을 지속적으로 조정하고 R-wave 피크를 식별한 다음 RR 간격을 계산하는 데 사용합니다. - 고급 사용자의 경우 다음 명령줄을 사용하여 기본 매개 변수를 사용하여 위의 모든 단계를 수행합니다(자세한 옵션은 BrainBeats 리포지토리의 자습서 스크립트 참조).
이지랩; 닫다; % GUI 없이 EEGLAB 시작
main_path = fileparts(which('eegplugin_
BrainBeats.m')); cd(main_path);
EEG = pop_loadset('파일 이름','dataset.set',
'파일 경로', 전체 파일 (main_path, 'sample_data')); %샘플 데이터셋 로드
EEG = brainbeats_process(EEG,'분석',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',참); % BrainBeats1st 레벨을 시작하여 기본 매개변수로 HEP 분석용 파일을 처리합니다.
3. EEG 및 HRV 특징 추출
- 동일한 데이터 세트를 다시 로드합니다(1.5단계 참조; 이전 작업에서 덮어썼으므로 파일 > 기존 데이터 세트 로드>dataset.set 선택)를 클릭하고 기본 GUI를 다시 열어 기본 매개 변수를 선택합니다(2.1단계; 도구 > BrainBeats > 1 수준)를 클릭합니다. 분석 유형으로 EEG 및 HRV 특징 추출을 선택하고, 심장 신호 유형으로 ECG를 선택하고, 전극 레이블 목록에서 ECG를 선택합니다. 확인을 클릭합니다.
- 두 번째 GUI 창은 2.2단계와 같이 나타나지만 EEG 전처리 및 HRV 및 EEG 기능 추출에 대한 매개변수가 다릅니다. 아래 설명된 대로 이러한 매개 변수를 설정합니다. OK(확인 )를 클릭하여 기본 매개변수로 시작합니다.
- HRV 섹션에서 freq. options 버튼을 클릭하여 HRV 전력(기본값은 normalized Lomb-Scargle periodogram으로 설정), 창 겹침(기본값은 25%로 설정) 및 두 번째 수준 정규화(기본적으로 설정되지 않음, 자세한 내용은 아래 참고 참조)를 수행하는 방법을 선택합니다.
- EEG features 섹션에서 freq. 옵션을 클릭하여 파워 스펙트럼 밀도(PSD, 기본값 = 1-40Hz), 단위(데시벨, μV2/Hz 또는 총 전력으로 정규화), 윈도우 유형(기본값 = 해밍), 윈도우 겹침(기본값 = 50%), 윈도우 길이(기본값 = 2초), 그리고 각 대역에 대한 주파수 경계 유형.
참고: HRV 전력은 기본적으로 정규화된 Lomb-Scargle 주기도를 사용하여 계산되며, 이는 리샘플링이 필요하지 않고(따라서 원본 정보를 더 잘 보존) 불균일하게 샘플링된 데이터, 누락된 데이터 및 노이즈(NN 간격에서 일반적)를 가장 잘 처리합니다68. 정규화된 버전은 신호의 분산에 따라 전력을 조정하여 다양한 노이즈 수준에 덜 민감하고, 주기적 구성 요소의 상대적 강도에 더 집중하며, 다른 녹음 또는 주제에서 더 비교할 수 있는 결과를 제공합니다. 사용할 수 있는 다른 방법으로는 비정규화된 Lomb-Scargle 주기도, Welch 방법 및 FFT(Fast Fourier Transform)가 있습니다. Welch 및 FFT 방법에 대해 리샘플링이 자동으로 수행되어 필요한 정기적으로 샘플링된 시계열을 생성합니다. 두 번째 수준 정규화는 각 주파수 대역의 전력을 총 전력으로 나누어 적용할 수 있으며, 이를 통해 전체 전력에 대한 각 주파수 구성 요소의 상대적 기여도를 보다 직관적으로 측정할 수 있습니다. 4개의 대역을 모두 사용할 수 있고 최소 24시간의 신호가 필요한 경우에만 의미가 있기 때문에 기본적으로 비활성화되어 있습니다. 이러한 알고리즘은 Physionet Cardiovascular signal processing toolbox46에서 조정된 것입니다.
- 감지된 추가 PPG 채널을 제거할지 확인하는 동일한 경고 메시지가 나타납니다. 다시 말하지만, 이것은 예상된 것입니다. Yes(예)를 클릭합니다. BrainBeats는 2.4단계와 같이 ECG 데이터를 전처리하고 NN 간격을 동일하게 추출하기 시작합니다. 그런 다음 시간(SDNN, RMSSD, pNN50), 주파수(ULF, VLF, LF, HF, LF:HF 비율, 총 전력) 및 비선형(푸앵카레, 위상 정류 신호 평균, 퍼지 엔트로피 및 프랙탈 차원) 영역의 NN 간격에서 심박 변이도(HRV) 기능을 추출합니다.
참고: BrainBeats는 파일 길이를 자동으로 확인하여 최소 요구 사항(예: ULF-HRV 전원에는 24시간의 데이터 필요)이 충족되었는지 확인하고, 그렇지 않은 경우 경고 메시지를 보내고, 신뢰할 수 없는 추정을 방지하기 위해 이러한 기능을 내보내지 않습니다. BrainBeats는 HRV 지표 추정을 위한 지침 및 권장 사항을 따릅니다 27,45. - BrainBeats는 2.7단계와 같이 EEG 데이터를 전처리합니다. (그림 3 참조). 큰 아티팩트는 ASR(아티팩트 부분 공간 재구성) 알고리즘50,52(기본 SD 기준은 30으로 설정되고 사용 가능한 RAM의 80%를 사용하여 속도 향상)를 사용하여 연속 데이터에서 자동으로 감지됩니다. 이러한 큰 아티팩트는 EEG 데이터에서 제거됩니다(그림 13 참조). GUI에서 이러한 매개변수를 조정하려면 ASR이 있는 불량 세그먼트를 거부할 임계값 및 ASR에 사용할 사용 가능한 RAM 필드를 선택합니다.
참고: EEG 및 심혈관 시계열은 각 신호에서 개별적으로 추정되기 때문에 기능 모드에 대해 시간을 동기화할 필요가 없습니다. 따라서 EEG 아티팩트는 EEG 데이터에서 직접 제거할 수 있으며(빨간색, 그림 13), 아티팩트를 포함하는 epoch가 두 시계열 모두에 대해 거부된 HEP 모드와 달리 ms 정확도의 시간 동기화가 해당 방법에 필수적입니다. - HEP와 동일한 알고리즘 및 매개변수를 사용하여 ICA를 수행하되(2.11단계 참조), 이번에는 99% 신뢰도로 감지되면 심장 구성 요소가 제거됩니다(관련 심장 관련 신호를 제거하고 싶지 않기 때문에 HEP/HEO에 대해 보존됨).
- 3.2단계에서 주파수 도메인 상자가 선택되므로 BrainBeats는 델타(1-3Hz), 세타(3-7Hz), 알파(8-13Hz), 베타(13-30Hz) 및 감마(30+Hz) 주파수 대역에 대한 평균 전력 스펙트럼 밀도(PSD), 개별 알파 주파수(IAF) 및 사용 가능한 모든 (대칭) 전극 쌍의 알파 비대칭과 같은 주파수 도메인 기능을 추출합니다. 이러한 기능을 확인하십시오.
참고: PSD를 데시벨(dB)로 변환하면 녹음물과 피사체 간의 결과를 쉽게 비교할 수 있습니다. 주파수 바운드는 종래의 바운드(예를 들어, 알파 대역에 대해 미리 정의된 8-13Hz) 또는 개별화된 경계로 설정될 수 있으며, 이들은 개별적인 차이(69 )를 설명하기 위해 전력 스펙트럼 밀도의 분포로부터 검출된다(예를 들어, 알파 대역의 경우 7.3-12.6Hz). 이 알고리즘은 알파 대역을 위해 설계되었으며, 다른 대역에 대해서는 특히 파워 스펙트럼 분포에 피크가 없는 경우 성능이 좋지 않습니다. 개별 알파 주파수(IAF)는 분할 피크 또는 모호한 피크(69)를 더 잘 처리하기 위해 알파 무게 중심을 사용하여 추정된다. 알파 비대칭은 지침에 따라 계산됩니다(50% 겹치는 2-s 해밍 창, 왼쪽 채널의 알파 파워 로그에서 오른쪽 채널의 알파 파워 로그를 뺀 값)47. 따라서 양수 값은 왼쪽보다 오른쪽 알파 파워가 더 크다는 것을 나타내며 그 반대의 경우도 마찬가지입니다. 알파 비대칭은 각 전극으로부터의 알파 파워를 모든 전극(47)에 걸쳐서 합산된 알파 파워로 나눔으로써 정규화될 수 있다. 대칭 쌍은 쎄타 거리를 사용하여 얻어지며, EEG 데이터에는 전극 좌표가 포함되어야 합니다. - 3.2단계에서 비선형 도메인 상자를 선택하면 BrainBeats는 각 EEG 채널에 대한 퍼지 엔트로피와 프랙탈 차원을 추출합니다.
참고: 비선형 영역 특징은 스펙트럼 측정에 의해 놓쳐지는 뇌의 비선형적이고 복잡한 역학을 포착하는 것으로 생각되며, 다양한 신체 시스템 간의 상호 작용을 조사하는 데 특별한 가능성을 보여줍니다 70,71,72. 퍼지 엔트로피는 대안(샘플 및 근사 엔트로피)보다 더 안정적이고 강력하지만 계산 시간이 더 오래 걸립니다(특히 샘플링 속도가 높은 긴 EEG 시계열의 경우). 이 문제를 해결하기 위해 EEG 신호의 길이가 2분보다 길고 샘플링 속도가 100Hz보다 큰 경우 자동으로 다운샘플링(또는 팩터가 정수가 아닌 경우 데시메이션)하여 90Hz(즉, 기본 저역 통과 필터와 일치하고 라인 노이즈 아티팩트를 최대한 피하기 위해 45Hz의 나이퀴스트 주파수에 해당)로 감소합니다. 또한 EEG 기능을 추정할 때 병렬 컴퓨팅이 기본적으로 활성화되어 특히 많은 EEG 채널을 사용할 수 있는 경우 컴퓨팅 시간이 단축됩니다. - 첫 번째 GUI에서 출력 시각화 를 선택하여(3.1단계 참조) HRV 및 EEG 데이터(그림 14)에 대한 전력 스펙트럼 밀도(PSD)를 표시하는 플롯과 일부 EEG 특징을 표시하는 두피 지형도(그림 15)를 생성합니다.
참고: EEG.brainbeats.preprocessing에서 일부 전처리 출력을 찾을 수 있으며 EEG.brainbeats.parameters에서 사용되는 모든 매개 변수를 찾을 수도 있습니다. 우리는 사용자가 연구 결과의 재현성을 높이기 위해 이러한 결과를 과학 간행물에 보고할 것을 권장합니다. - 첫 번째 GUI 창에서 출력 저장 상자를 선택한 상태로 유지하여 EEG.brainbeats.features의 EEGLAB .set 파일로 내보낸 모든 기능을 저장하고 데이터셋이 로드된 동일한 폴더의 .mat 파일에 저장합니다.
- BrainBeats는 EEGLAB의 기록 기능을 지원합니다. 모든 작업이 끝나면 MATLAB의 명령 창에 eegh 를 입력하여 GUI에서 수동으로 선택한 매개 변수를 사용하여 단일 명령 줄을 통해 위의 모든 단계를 반복할 수 있는 명령 줄을 인쇄하여 작업을 쉽게 자동화하고 복제할 수 있습니다.
- 고급 사용자의 경우 다음 명령을 사용하여 위의 모든 단계를 수행합니다.
이지랩; 닫다; % GUI 없이 EEGLAB 시작
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('파일 이름','dataset.set',
'파일 경로', 전체 파일 (main_path, 'sample_data')); % 샘플 데이터 세트를 로드합니다.
EEG = brainbeats_process(EEG, '분석',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - 이전 단계에서는 ECG 신호에서 HRV 특징을 추출했습니다. PPG 신호에서 HRV 특징을 추출하려면 다음 단계를 사용하십시오(EEG 특징은 동일함).
- 작업에 의해 덮어쓰여졌으므로 동일한 데이터 세트를 다시 로드하고(1.5단계) 기본 GUI를 다시 엽니다(2.5단계). 분석을 위해 EEG 및 HRV 특징 추출 을 선택하고 심장 신호 유형으로 PPG 를 선택하고 채널 이름으로 PPG 를 선택합니다. 확인을 클릭합니다.
- 이제 2번째 GUI 창에 PPG 전처리와 HRV 및 EEG 기능을 추출하기 위한 매개변수가 표시됩니다. OK(확인 )를 클릭하여 기본 매개변수로 실행합니다. 매개변수는 2.17단계에 설명되어 있습니다.
- 감지된 ECG 채널을 제거할지 확인하는 경고 메시지가 나타납니다. 툴박스는 ECG와 PPG를 동시에(또는 다른 보조 채널) 분석하도록 설계되지 않았고 데이터 세트에 보관하면 심각한 오류(예: 아티팩트 제거, ICA 분해 불량 등)가 발생할 수 있기 때문에 이는 예상된 결과입니다. Yes(예)를 클릭합니다.
- BrainBeats는 PPG 신호를 전처리하고 2.5단계와 같이 NN 간격을 추정합니다. 3.2단계와 동일하게 NN 간격에서 추출된 HRV 특징을 확인합니다. 단, NN 구간은 이제 PPG 신호에서 얻어졌습니다. EEG 신호는 3.2단계와 같이 전처리됩니다. BrainBeats는 PSD를 플로팅합니다(그림 16). 여기서 유일한 차이점은 ECG가 아닌 PPG에서 얻은 NN 간격에서 추정된 PSD입니다.
- 고급 사용자의 경우 다음 명령을 사용하여 위의 모든 단계를 수행합니다.
이지랩; 닫다; % GUI 없이 EEGLAB 시작
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('파일 이름','dataset.set','파일 경로',
fullfile(main_path, 'sample_data')); %샘플 데이터셋 로드
EEG = brainbeats_process(EEG,'분석',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. EEG 신호에서 심장 인공물을 추출합니다.
- 샘플 데이터 세트를 로드합니다(1.5단계 참조).
- 도구 > BrainBeats > 1st level (주제 수준)을 클릭하여 기본 GUI 창을 열고 분석 유형으로 EEG 신호에서 심장 아티팩트 추출을 선택하고 심장 신호 유형으로 ECG를 선택한 다음 전극 레이블 목록에서 ECG를 선택합니다. 확인을 클릭합니다.
- 2번째 GUI 창에는 전처리 매개변수가 표시됩니다. 전력선 노이즈 를 50Hz(유럽)로 설정하고, 필요한 경우 신뢰 수준을 편집하고, 부스트 모드(베타) 상자를 선택하고, 샘플 데이터 세트의 EEG 신호가 사전 처리되지 않으므로 확인을 클릭하여 기본 매개변수로 실행합니다.
참고: 심장 구성 요소를 감지하기 위한 신뢰도 수준은 기본적으로 80%로 설정되며, 일부 데이터 세트의 경우 너무 낮거나 너무 높을 수 있습니다. 이 값을 높이면 심장 구성 요소를 감지할 가능성은 높아지지만 해당 감지의 신뢰성은 떨어집니다. 부스트 모드(베타)는 선택 사항이며 심장 신호를 EEG 신호에 주입하여 분류 성능을 향상시키는 것을 목표로 합니다. - 감지된 추가 PPG 채널을 제거할지 확인하는 경고 메시지가 나타납니다. 툴박스는 ECG와 PPG를 동시에(또는 다른 보조 채널) 분석하도록 설계되지 않았고 데이터 세트에 보관하면 심각한 오류(예: 아티팩트 제거, ICA 분해 불량 등)가 발생할 수 있기 때문에 이는 예상된 결과입니다. Yes(예)를 클릭합니다.
- ECG 신호는 대역통과 필터링되어 1Hz 미만의 느린 주파수 드리프트와 20Hz 이상의 고주파 잡음을 제거합니다(비인과 제로 위상 FIR 필터 사용). 3.4단계와 같이 EEG 신호를 전처리합니다.
- 선조건화된 실제 데이터에 대한 ICA 알고리즘(PICARD)을 사용하여 독립 성분 분석(ICA)을 수행합니다. 이 옵션을 변경하려면 4.3단계의 GUI에서 복제를 위해 표준 Infomax 알고리즘 또는 수정된 Infomax 알고리즘을 선택합니다. 그런 다음 ICLabel을 사용하여 독립 성분을 자동으로 분류합니다. 성분이 80% 신뢰도로 심장 성분으로 분류되면 기본적으로 EEG 데이터에서 자동으로 감지되고 추출됩니다.
- 첫 번째 기본 GUI(4.2단계)에서 Visualize outputs 상자를 유지하여 제거된 구성 요소(그림 17 왼쪽)의 두피 지형과 심장 구성 요소(빨간색, 그림 17 오른쪽)를 추출한 후 최종 EEG 시계열(파란색, 그림 17 오른쪽)을 시각화합니다.
참고: ECG 채널은 ECG 관련 구성 요소의 추출을 확인하기 위해 시각화를 위해 유지되지만 더 이상 관련 정보가 포함되지 않으므로 이 단계 후에 제거됩니다. - 고급 사용자의 경우 다음 명령줄을 사용하여 다음 단계를 수행합니다.
이지랩; 닫다; % GUI 없이 EEGLAB 시작
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('파일 이름','dataset.set','파일 경로',
fullfile(main_path, 'sample_data'));
뇌파 = brainbeats_process(EEG,'분석','rm_heart',
'heart_signal','ECG', ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'부스트',true);
결과
먼저 BrainBeats 플러그인을 사용하여 EEG 및 ECG 데이터를 사전 처리하고, 아티팩트를 식별 및 제거하고, 심장 박동 유발 전위(HEP) 및 진동(HEO)을 분석했습니다. BrainBeats는 ECG 신호와 일부 RR 아티팩트에서 RR 간격을 성공적으로 감지했습니다(그림 2). BrainBeats는 또한 명령 창에서 하트비트의 11/305(3.61%)가 아티팩트로 플래그가 지정되고 보간되었다고 보고했습니다. RR 간격(보간 전)?...
토론
프로토콜의 중요한 단계
중요한 단계는 1.1-1.4단계에 설명되어 있습니다. 경고 및 오류 메시지는 사용자가 문제가 발생할 수 있는 이유(예: EEG 데이터에 로드되지 않은 전극 위치, 초저주파 HRV의 신뢰할 수 있는 측정값을 계산하기에 너무 짧은 파일 길이, 신뢰할 수 있는 분석을 위한 신호 품질이 너무 낮음 등)를 이해하는 데 도움이 되도록 툴박스의 다양한 위치에 구현됩니다. 각 기...
공개
저자는 밝힐 것이 없습니다.
감사의 말
노에틱 과학 연구소(Institute of Noetic Sciences)가 이 연구를 지원했습니다. BrainBeats의 알고리즘 중 일부를 개발하기 위해 조정된 원본 오픈 소스 알고리즘의 개발자에게 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
참고문헌
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유