JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
힘줄의 유전자 발현 수준에 대한 공간 고해상도 분석
요약
이 문서에서는 힘줄에 대해 최적화된 in situ 프로토콜을 수행하는 방법을 설명합니다. 이 방법은 조직 준비, 단면 투과화, 프로브 설계 및 신호 증폭 방법에 대해 설명합니다.
초록
최근 몇 년 동안 다양한 의료 및 생물학 분야에서 고분해능 전사체학을 위한 많은 프로토콜이 개발되었습니다. 그러나 기질이 풍부한 조직, 특히 힘줄은 세포 수가 적고 세포당 RNA 양이 적으며 기질 함량이 높기 때문에 분석이 복잡하여 남겨졌습니다. 최근의 가장 중요한 단세포 도구 중 하나는 힘줄의 유전자 발현 수준에 대한 공간 분석입니다. 이러한 RNA 공간 도구는 새로운 집단과 알려지지 않은 집단의 특정 세포를 찾고, 단일 세포 RNA-seq 결과를 검증하고, 단일 세포 RNA-seq 데이터에 조직학적 컨텍스트를 추가하기 위해 힘줄에서 특히 매우 중요합니다. 이러한 새로운 방법은 탁월한 감도로 세포의 RNA를 분석하고 단일 세포 수준에서 단일 분자 RNA 표적을 검출할 수 있게 하여 힘줄을 분자적으로 특성화하고 힘줄 연구를 촉진하는 데 도움이 될 것입니다.
이 방법 논문에서는 단일 세포 수준에서 온전한 세포 내 표적 RNA를 검출하기 위해 새로운 in situ hybridization assay를 사용하여 조직학적 섹션에서 공간 유전자 발현 수준을 분석하는 데 사용할 수 있는 방법에 중점을 둘 것입니다. 먼저, 사용 가능한 다양한 분석을 위해 힘줄 조직을 준비하는 방법과 배경 잡음 없이 높은 감도와 높은 특이도로 표적 특이적 신호를 증폭하는 방법에 중점을 둘 것입니다. 그런 다음 이 백서에서는 특정 투과화 방법, 다양한 프로브 설계 및 현재 사용 가능한 신호 증폭 전략에 대해 설명합니다. 단일 세포 해상도에서 서로 다른 유전자의 전사 수준을 분석하는 이러한 고유한 방법을 통해 다양한 동물 모델 및 인간 힘줄 조직의 젊고 나이가 많은 집단에서 힘줄 조직 세포를 식별하고 특성화할 수 있습니다. 이 방법은 또한 뼈, 연골 및 인대와 같은 다른 기질이 풍부한 조직에서 유전자 발현 수준을 분석하는 데 도움이 됩니다.
서문
힘줄은 근육과 뼈 1 사이에 힘을 전달할 수 있는 결합 조직입니다. 발달적으로, 축 방향 건세포(axial tenocytes)는 소미트(somites)2의 경피(sclerotome) 내의 중간엽 세포(mesenchymal cell)에서 유래한다. 사지 힘줄은 측판 중배엽에서 파생됩니다. 그리고 두개골 힘줄은 두개골 신경능선계통 3,4에서 발생합니다. 힘줄은 경피성 전사 인자5의 발현으로 특징지을 수 있지만, 테노모듈린(tenomodulin), 모호크(mohawk) 및 조기 성장 반응 1/2 6,7,8,9를 포함한 여러 마커도 힘줄 발달에 중요한 역할을 합니다.
힘줄에 대한 몇 가지 알려진 마커에도 불구하고, 일반적으로 힘줄에는 생체 역학적 특성의 구배에 걸쳐 있는 세포가 포함되어 있기 때문에 보다 심층적인 특성화는 여전히 어렵습니다. 근긴장 접합부(myotendinous junction), 힘줄 중간 몸체(tendon mid-body) 및 더 석회화된 부착부(enthesis)에서 힘줄 세포는 인장 특성이 다양한 세포 외 기질(extracellular matrices)에 존재합니다. 힘줄은 연조직과 경조직 사이의 기계적 강도 차이로 인한 인장 응력을 견뎌야 하기 때문에 힘줄에 있는 세포의 공간적 조직은 힘줄의 기능에 특히 중요합니다. 그러나 이러한 힘줄 하위 집단에 대해서는 알려진 바가 거의 없습니다.
많은 고분해능 공간 전사체 도구를 사용하여 단일 세포 RNA 염기서열 분석 또는 현장 혼성화를 포함하되 이에 국한되지 않는 세포 하위 집단을 규명하기 시작할 수 있습니다. 그러나 이러한 공간 프로파일링 분석은 현저해부 또는 절편 후 조직 전반에서 RNA 발현을 밝히는 데 도움이 되지만 힘줄 조직에서 수행할 경우 어려울 수 있습니다. 힘줄은 건조 질량10 기준 콜라겐의 거의 86%로 구성된 매트릭스가 풍부한 조직으로, 염기서열분석을 위해 세포를 추출하는 것이 어렵습니다. 기질에서 세포를 분리하는 것의 복잡성,힘줄 11의 저세포 특성, 상대적으로 낮은 RNA 수로 인해 힘줄은 분석하기 어려운 조직입니다.
이 논문에서는 조직 준비, 투과화 및 프로브 설계 방법을 제공하여 힘줄에 활용하기 위해 새로운 in situ hybridization assay를 최적화하는 방법을 제시합니다. 기존 염기서열분석 기술과 결합하면 연구자들이 개발 중인 힘줄, 성인 힘줄 또는 손상된 힘줄에 걸쳐 분석 민감도와 특이도를 높여 힘줄 하위 집단을 공간적으로 특성화하는 데 도움이 될 수 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
모든 동물 실험은 IACUC(Institutional Animal Care and Use Committee) 및 AAALAC 지침에 따라 수행되었습니다. 실험은 매사추세츠 종합 병원에서 승인된 프로토콜 #2013N000062 따라 수행되었습니다. 이 연구에서는 C57BL/J6 마우스(생후 5주 및 P0)를 사용했습니다. 이 프로토콜에 사용되는 모든 재료, 시약 및 기기와 관련된 자세한 내용은 재료 표를 참조하십시오.
1. 샘플 준비 및 정착
- CO2 챔버에서 마우스를 안락사시킨 후 안락사의 2차 방법으로 자궁경부 탈구를 수행합니다. 그런 다음 가위를 사용하여 고관절12를 따라 쥐의 뒷다리를 자릅니다. 섬광 바이알에 쥐 뒷다리를 4% 파라포름알데히드(PFA) 용액에 충분히 담그고 샘플을 완전히 덮고 조직을 24시간 동안 고정합니다.
참고: 4% PFA 대신 10% 포르말린을 고정에 사용할 수도 있습니다. 고정 후, 샘플은 추가 처리까지 인산염 완충 식염수(PBS)에 보관될 수 있습니다. 뒷다리 전체를 고정함으로써 힘줄의 장력을 유지할 수 있고 부착부, 힘줄 중간 부위 또는 근긴장 접합부를 검사할 수 있는 기회를 가질 수 있습니다. - 섬광 바이알에서 PFA를 제거한 후 샘플을 덮을 만큼 충분한 PBS를 추가하여 1x PBS로 3 x 10분 동안 샘플을 세척합니다. 그런 다음 뒷다리를 4°C에서 0.5M EDTA에 1-2주 동안 교반하면서 담그고 2-3일마다 새로운 EDTA로 교체합니다.
- EDTA를 제거한 후 1x PBS로 샘플을 3 x 10분 동안 세척하고 샘플을 70% EtOH로 탈수한 다음 원하는 방향으로 파라핀에 삽입합니다. 기존의 FFPE 절편 기술을 사용하여 3μm 두께의 조직을 절단하고 절편을 처리된 하전 현미경 슬라이드로 옮깁니다( 재료 표 참조)13.
참고: 다음 단계를 진행하기 전에 섹션을 4°C에 보관할 수 있습니다. - 탈파라핀화
- 60°C의 핫 플레이트에서 슬라이드를 1시간 동안 구운 다음 슬라이드를 실온(RT)에 놓으십시오. 슬라이드에서 파라핀 왁스를 제거하려면 슬라이드를 슬라이드 랙에 놓고 자일렌으로 채워진 염색 접시에 넣습니다. 신선한 자일렌을 3 x 5분 동안 반복합니다.
주의 : 크실렌은 위험합니다. 조심히 다루십시오. - 섹션을 재수화하려면 RT에서 100% EtOH로 2회, dH2O로 희석된 75% EtOH에서 1회, PBS에서 희석된 50% EtOH에서 1회, PBS에서 희석된 25% EtOH로 1회, PBS에서 2회 세척합니다.
참고: 모든 재수화 단계는 슬라이드 랙의 슬라이드와 염색 접시의 다양한 EtOH 용액으로 수행됩니다.
- 60°C의 핫 플레이트에서 슬라이드를 1시간 동안 구운 다음 슬라이드를 실온(RT)에 놓으십시오. 슬라이드에서 파라핀 왁스를 제거하려면 슬라이드를 슬라이드 랙에 놓고 자일렌으로 채워진 염색 접시에 넣습니다. 신선한 자일렌을 3 x 5분 동안 반복합니다.
2. RNAscope(상용화된 ISH) 프로토콜14 적응
- 솔루션 및 프로브 준비
- pH 8에서 25mM의 Tris-HCl, 10mM의 EDTA 및 50mM의 포도당을 결합하여 절편화된 샘플의 전처리를 위한 TEG 용액을 준비합니다. 모든 샘플이 처리되거나 과도하게 준비될 수 있도록 충분한 부피를 만드십시오. RT에서 필터 살균 및 보관; 사용하기 전에 용액을 적절하게 혼합하십시오.
- 모든 세탁을 위해 충분한 1x 세척 버퍼( 재료 표 참조)를 준비하고 RT에 보관하십시오.
- 40°C 수조 또는 인큐베이터에서 관심 프로브를 10분 동안 예열합니다. 그런 다음 ISH 프로토콜14에 따라 프로브를 준비합니다.
- 시료의 전처리
- 권장되는 전처리 용액을 사용하는 대신 TEG 완충액이 들어있는 염색 접시에 슬라이드를 60°C에서 4시간 동안 담그십시오.
참고: TEG 완충액 배양은 조직이 특히 조밀하거나 관심 RNA 결합 모티프의 항원 검색이 어려운 경우 최대 6시간까지 연장할 수 있습니다. 이 경우 여기에 정지 지점을 추가하고 슬라이드를 건조시켜 4°C 냉장고에서 밤새 보관할 수 있습니다. - 버퍼에서 샘플을 제거하고 건조시킵니다. 그런 다음 소수성 장벽 펜으로 샘플 주위를 그리고, 프로테아제 IV가 들어있는 다른 염색 접시에 담그고, 40 °C에서 45 분 동안 오븐에서 배양합니다.
참고: 성체 마우스에는 프로테아제 IV를 권장합니다. P0와 같은 어린 마우스를 사용하는 경우 프로테아제 III와 같은 더 가벼운 효소를 30분 동안 사용하는 것이 좋습니다. 다른 연령에 대한 문제를 해결할 때 힘줄의 무결성을 관찰하고 성능 저하 여부를 확인하십시오. 조직이 해리되거나 분해되는 경우 배양 시간을 줄이거나 사용하는 프로테아제를 변경하십시오. - 샘플을 TEG 버퍼에서 30분 동안 다시 세척한 다음 ISH 프로토콜14에 따라 프로브를 하이브리드화합니다.
- 샘플 상단에 50-100 μL의 장착 시약을 추가합니다. 현미경 검사를 위해 샘플 위에 커버슬립을 놓습니다. 이미징할 때 고배율(40x 대물렌즈 이상)을 사용하여 신호를 봅니다.
- 권장되는 전처리 용액을 사용하는 대신 TEG 완충액이 들어있는 염색 접시에 슬라이드를 60°C에서 4시간 동안 담그십시오.
3. HCR ISH 프로토콜15 적응
- 후접사
- 4% PFA(PBS 중), PBT 용액(PBS 중 0.1% Tween-20) 및 PBS 내 Proteinase K(스톡 농도 10mg/mL, 최종 농도 5μg/mL)를 준비합니다.
- 첫 번째 접미사 단계를 시작하려면 4% PFA가 함유된 염색 접시에 슬라이드를 5분 동안 담그십시오. 그런 다음 4% PFA 용액을 제거하고 이후 단계에서 사용하기 위해 보관하십시오. PBT 용액으로 슬라이드를 2 x 5분 동안 헹구고 PBT 용액을 제거합니다. Proteinase K 용액(5μg/mL)이 들어 있는 염색 접시에 슬라이드를 5분 동안 놓고 PBT 용액에서 2 x 5분으로 헹굽니다.
- 두 번째 접미사 단계를 시작하고 슬라이드를 4% PFA에 5분 동안 담급니다.
참고: 이것은 3.1.2단계에서 저장된 것과 동일한 4% PFA입니다. - PBT 용액에서 슬라이드를 3 x 5분 동안 헹굽니다.
알림: 모든 후고정 단계는 슬라이드 랙의 슬라이드와 각 용액이 포함된 염색 접시에서 수행됩니다.
- 아세틸화(Acetylation)
- 무수아세트산 625μL, 1M 트리에탄올아민(TEA) 완충액 3.3mL 및 ddH2O246mL를 함유하는 아세틸화 용액을 준비합니다. 반드시 잘 섞어 준비 후 즉시 사용하십시오.
- 슬라이드를 아세틸화 용액에 10분 동안 넣은 다음 PBT 용액에서 3 x 5분으로 헹굽니다. ddH2O에서 슬라이드를 헹구고 30분 동안 자연 건조시킵니다. 그런 다음 소수성 장벽 펜으로 샘플 주위를 그립니다.
알림: 모든 아세틸화 단계는 슬라이드 랙의 슬라이드와 각 용액이 포함된 염색 접시에서 수행됩니다
- 교 잡
- 혼성화 버퍼를 37°C로 예열하고 가습 챔버를 37°C로 예열합니다.
주의: 버퍼에는 위험 물질인 포름아미드가 포함되어 있습니다. 슬라이드가 충분히 건조되지 않은 경우 실험실용 물티슈로 가장자리를 닦아서 과도한 dH2O를 제거합니다. - 100μL의 혼성화 완충액에 각 프로브 혼합물의 0.4pmol을 첨가하여 프로브 용액을 준비합니다.
알림: dHCR 이미징을 사용하는 경우 더 높은 농도의 프로브를 사용하여 프로브 혼성화 효율을 개선하십시오. - 3 M NaCl (4 L의 최종 부피에서 701.1 g의 NaCl)과 0.3 M의 Na3CH6H5O7 • 2H2O (352.8 g)를 결합한 다음 HCl 또는 10 N NaOH를 첨가하여 최종 pH를 7로 조정하여 4 L의 염화나트륨 구연산염 완충액 (20x SSC)을 준비합니다. 4 L에 도달하도록 충분한 H2O를 추가합니다. 20x SSC를 5x로 희석하고 10% Tween 20을 충분히 추가하여 총 부피의 0.1%가 되도록 하여 5x SSCT를 준비합니다.
- 샘플에 200μL의 혼성화 완충액을 추가하고, 슬라이드를 가습 챔버에 넣고, 슬라이드를 10분 동안 배양합니다. 혼성화 용액을 제거하고 실험실용 물티슈로 가장자리를 닦아서 슬라이드의 초과 버퍼를 배출합니다.
- 준비된 프로브 용액 100μL를 샘플 위에 추가하고 샘플에 커버슬립을 놓습니다. 오븐에서 하룻밤 동안 또는 5x SSC와 50% 포름아미드를 함유한 용액으로 가습된 챔버에서 약 12-16시간 동안 배양합니다. 온도를 37°C로 설정합니다.
참고: 배양은 하룻밤 동안 이루어지기 때문에 커버슬립을 추가하면 증발을 최소화할 수 있습니다. - 37°C 수조에 5x SSCT 용액의 부분 표본을 남겨두고 RT에 두 번째 부분 표본을 남겨둡니다. 다음 단계에서 샘플 수와 세척 횟수를 기준으로 필요한 이러한 부분 표본의 부피를 예상합니다.
- 혼성화 버퍼를 37°C로 예열하고 가습 챔버를 37°C로 예열합니다.
- 세탁
- 이전에 예열된 5x SSCT 용액을 사용하여 75% 세척 버퍼/25% 5x SSCT, 50% 세척 버퍼/50% 5x SSCT 및 25% 세척 버퍼/75% 5x SSCT 용액을 준비합니다.
- 여분의 프로브를 제거하려면 75% 세척 버퍼/25% 5x SSCT, 50% 세척 버퍼/50% 5x SSCT 및 25% 세척 버퍼/75% 5x SSCT에서 37°C에서 세척당 15분 동안 슬라이드를 직렬로 담그십시오. 커버 슬립이 샘플에서 떠오르도록 합니다.
- 슬라이드를 100% 5x SSCT로 2 x 15분 동안 배양한 다음 RT에서 5분 동안 5x SSCT에 담급니다.
알림: 세척 단계는 슬라이드 랙의 슬라이드와 각 용액이 포함된 염색 접시 또는 용액을 물방울로 첨가하여 수행할 수 있습니다.
- 증폭
- 3μM 스톡의 2μL를 스냅 쿨링하여 헤어핀 h1 6 pmol과 헤어핀 h2 6 pmol을 준비합니다(95°C에서 90초 동안 가열하고 RT로 냉각). 이 과정에서 머리핀을 빛으로부터 보호하십시오.
참고: HCR 헤어핀 h1 및 h2는 헤어핀 보관 버퍼에 제공되며 스냅 냉각 준비가 되어 있습니다. 시원한 h1 및 h2 헤어핀을 별도의 튜브에 끼웁니다. - 실험실용 물티슈로 가장자리를 닦아내어 슬라이드를 건조시킵니다. 그런 다음 샘플 위에 200μL의 증폭 버퍼를 추가하고 RT에서 30분 동안 가습된 챔버에 놓습니다. RT에서 100μL의 증폭 버퍼에 snapcooled h1 헤어핀과 snapcooled h2 헤어핀을 추가하여 헤어핀 혼합물을 준비합니다.
- 증폭 버퍼를 제거하고 실험실용 물티슈로 가장자리를 닦아 슬라이드의 과도한 액체를 배출합니다. 이전에 준비된 헤어핀 혼합물 100μL를 추가하고 샘플 위에 파라필름을 놓습니다. 슬라이드를 최소 4시간 동안 또는 RT의 어두운 가습 챔버에서 하룻밤 동안 배양합니다.
- 여분의 헤어핀을 제거하려면 염색 접시에 슬라이드를 5x SSCT로 담그고 RT에서 30분 동안 배양합니다. 신선한 5x SSCT로 30분 동안 세척을 반복하고 5분 동안 세 번째로 세탁을 반복합니다.
- 실험실용 물티슈로 가장자리를 닦아내어 슬라이드를 건조시키고 샘플 위에 50-100 μL의 장착 시약을 추가합니다. 현미경 검사를 위해 샘플 위에 커버슬립을 놓습니다. 이미징할 때 고배율(40x 대물렌즈 이상)을 사용하여 신호를 봅니다.
- 3μM 스톡의 2μL를 스냅 쿨링하여 헤어핀 h1 6 pmol과 헤어핀 h2 6 pmol을 준비합니다(95°C에서 90초 동안 가열하고 RT로 냉각). 이 과정에서 머리핀을 빛으로부터 보호하십시오.
Access restricted. Please log in or start a trial to view this content.
결과
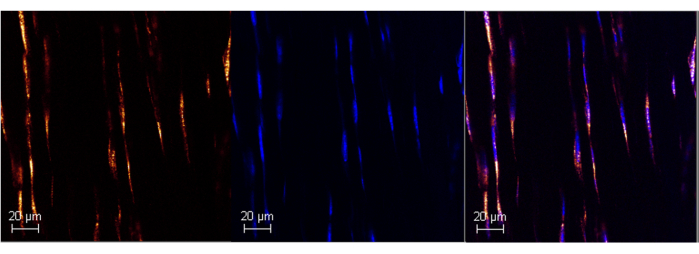
그림 1: RNAScope를 사용한 성체 마우스 아킬레스건에서 Poly A RNA 발현. 상용화된 ISH assay를 사용한 마우스 아킬레스건(왼쪽 패널)의 성공적인 Poly A 라벨링의 대표 이미지. DAPI와의 공동 국소화는 프로브의 특이성(중간 및 오른쪽 패널)을 확인하...
Access restricted. Please log in or start a trial to view this content.
토론
이 논문에서는 기존 ISH 도구를 활용하여 높은 수준의 특이성과 민감도로 힘줄 조직에 사용할 수 있도록 수정 사항을 설명합니다. 힘줄은 매트릭스 밀도가 높은 조직이기 때문에 유사한 수준의 프로브 침투 및 특이성을 달성하기 위해 프로토콜 조정이 자주 이루어져야 합니다. 힘줄 조직의 이러한 특정 투과화 방법과 신호 증폭 전략은 논의된 ISH 프로토콜의 효능을 개선?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
저자는 이러한 프로토콜의 개발 및 문제 해결에 대한 지원과 격려에 대해 Jenna Galloway와 Galloway Lab 구성원에게 감사드립니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
참고문헌
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689(2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853(2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288(2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753(2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968(2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806(2020).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유