Method Article
PyDesigner v1.0: 확산 자기 공명 이미징을 위한 DESIGNER 파이프라인의 Python 구현
* 이 저자들은 동등하게 기여했습니다
요약
여기에서는 일반적인 dMRI 아티팩트를 수정하고 확산 텐서 이미징(DTI), 확산 첨도 이미징(DKI), 파이버 볼 이미징(FBI), 미세 구조 모델링(백질 무결성[WMTI] 및 파이버 볼 백질[FBWM]) 및 트랙토그래피 출력을 생성할 수 있는 Python 기반 확산 자기 공명 이미징(dMRI) 처리 파이프라인인 PyDesigner를 소개합니다.
초록
PyDesigner는 dMRI 전처리 및 텐서 추정을 위한 Gibbs 및 NoisE 제거(DESIGNER) 파이프라인(Dv1)을 사용하는 원래 확산 매개변수 EStImation을 기반으로 하는 Python 기반 소프트웨어 패키지입니다. 이 소프트웨어는 비상업적 연구를 위해 공개적으로 제공되며 임상 치료에 사용할 수 없습니다. PyDesigner는 FSL 및 MRtrix3의 도구를 결합하여 노이즈 제거, Gibbs 링잉 보정, 와전류 모션 보정, 브레인 마스킹, 이미지 스무딩 및 라이시안 바이어스 보정을 수행하여 다중 확산 측정의 추정을 최적화합니다. Windows, Mac 및 Linux의 여러 플랫폼에서 사용할 수 있으며, DKI, DTI, WMTI, FBI 및 FBWM 데이터 세트와 tractography ODF 및 .fib 파일에서 일반적으로 사용되는 메트릭을 정확하게 파생할 수 있습니다. 또한 파일 형식에 구애받지 않으며 .nii, .nii.gz, .mif 및 dicom 형식 형식의 입력을 허용합니다. 사용자 친화적이고 설치가 쉬운 이 소프트웨어는 데이터 무결성을 평가하기 위해 신호 대 잡음비 그래프, 이상치 복셀 및 헤드 모션을 보여주는 품질 관리 메트릭도 출력합니다. 또한 이 dMRI 처리 파이프라인은 다중 에코 시간 데이터 세트 처리 및 기능 파이프라인 사용자 지정을 지원하므로 사용자는 다양한 사용자 요구 사항을 충족하기 위해 사용할 프로세스와 생성되는 출력을 지정할 수 있습니다.
서문
확산 MRI(dMRI)는 뇌의 미세 구조 특성에 대한 비침습적 연구에 널리 적용됩니다. 많은 dMRI 방법이 제안되었지만 일반적으로 사용되는 두 가지는 확산 텐서 이미징(DTI)과 확산 첨도 이미징(DKI)입니다. 이러한 기술은 밀접한 관련이 있으며, DKI는 확산 비가우시안성1의 정량화를 포함하는 DTI의 확장입니다. 둘 다 다양한 스칼라 확산 측정을 제공하고 백질 섬유 tractography의 구축을 가능하게 합니다. DTI와 DKI의 중요한 장점은 확산 물리학에 대한 견고한 기반을 가지고 있어 그 타당성이 조직 미세구조 2,3에 대한 자세한 가정에 의존하지 않는다는 것입니다. 이를 통해 DTI와 DKI는 모든 연령과 질병 상태의 참가자를 위해 뇌와 신체 전체에 적용될 수 있습니다.
원시 확산 가중 이미지(DWI)는 신호 잡음, 동작, 깁스 링잉 및 와전류 왜곡을 포함한 여러 요인에 의해 저하되기 때문에 확산량4을 계산하기 전에 전처리를 사용해야 합니다. DWI의 전처리는 현재 고도로 발전된 활발한 연구 분야입니다. 그러나 필요한 처리 단계를 일관된 결과를 제공하는 단일 파이프라인으로 결합하는 것은 dMRI 획득의 세부 사항에 따라 여러 사용자 정의 설정을 조정해야 하고 전처리 단계가 수행되는 순서가 결과에 영향을 미치기 때문에 어렵습니다. 이러한 이유로 DWI5에 대한 전처리를 최적화, 표준화 및 간소화하기 위해 2016년에 Gibbs 및 NoisE Removal을 사용한 확산 매개변수 EStImation(DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) 파이프라인이 처음 제안되었습니다. DESIGNER Dv1은 FSL, MRtrix3, MATLAB 및 Python에 내장된 소프트웨어 도구를 사용하여 전처리 및 확산/첨도 텐서 추정5을 통한 이미지 보정을 포함하는 원활하고 완전한 DWI 프로세스를 생성합니다. 전처리 단계를 켜거나 끄는 제어 플래그를 사용하면 DWI 수정을 선택적으로 수행할 수 있습니다. DESIGNER는 (i) Marchenko-Pastur 주성분 분석(MP-PCA) 잡음 제거6, (ii) Gibbs 울림 보정7, (iii) EPI(echo-planar imaging) 왜곡 보정8, 와전류 보정9, 모션 보정10 및 이상치 치환11, (iv) B1 바이어스 필드 보정, (v) 브레인 마스크 생성, (vi) 평활화, (vii) 라이시안 노이즈 바이어스 보정 및 (viii) b0 정규화. 이 특정 순서로 전처리하면 정확도와 효과적인 신호 대 잡음비(SNR)5가 모두 향상됩니다. PyDesigner의 각 단계는 선택 사항이며 사용자 기본 설정에 따라 사용하거나 건너뛸 수 있습니다. 예를 들어, 평활화는 일부 데이터 세트에 필요한 전처리 단계가 아닐 수 있습니다. 필터링 오류를 완화하는 데 도움이 되지만 초고품질 데이터 세트에는 필요하지 않을 수 있습니다. 따라서 사용자는 데이터에 필요한 단계만 사용하도록 선택할 수 있습니다.
여러 플랫폼에서 DESIGNER를 구현하는 것은 운영 체제 및 환경 설정의 차이로 인해 어렵습니다. 특히, DESIGNER Dv1이 주로 MATLAB으로 작성된다는 사실은 Python-MATLAB 인터페이스를 가능하게 하는 데 필요한 복잡한 구성 요구 사항으로 인해 발생하는 상당한 이식성 문제를 야기합니다. 또한 MATLAB, Python 및 종속성 버전의 다양한 조합은 이 파이프라인의 재현성을 손상시킵니다. 이러한 이유로 우리는 완전히 Python 기반인 PyDesigner를 개발했습니다. 이 dMRI 처리 파이프라인을 사용하면 원활한 전처리가 가능할 뿐만 아니라 파이프라인을 Docker 컨테이너에 통합할 수 있으므로 이식성과 재현성이 크게 향상됩니다. 또한 PyDesigner는 MATLAB 코드를 대체하여 모든 라이선스 비용을 절감하고 접근성을 개선합니다.
이 dMRI 처리 파이프라인은 DESIGNER에서 도입한 핸즈프리 접근 방식을 보강하고, 몇 가지 새로운 기능을 추가하고, FSL 및 MRtrix3의 도구를 통합하여 전처리를 수행합니다. Numpy12, SciPy13 및 CVXPY14,15와 같은 표준 수학 Python 라이브러리는 DESIGNER의 MATLAB 부분을 Python 코드로 대체하는 데 사용되었습니다. 이 소프트웨어는 Github16에서 공개적으로 제공되고 사용할 수 있습니다. 이 백서의 목표는 당사 소프트웨어를 검증하거나 유사한 소프트웨어와 비교하는 것이 아니라 사용자가 원하는 경우 PyDesigner를 사용하여 데이터를 처리하기 위한 단계별 가이드를 제공하는 것입니다.
프로토콜
소프트웨어를 개발하고 테스트하는 데 사용된 모든 데이터는 기관 검토 위원회가 승인한 연구 프로토콜에 따라 수집되었습니다.
참고: PyDesigner는 MacOS 10.14를 기반으로 구축되었으며 Windows에서 실행하려면 WSL(Linux용 Windows 하위 시스템)이 필요합니다. Linux/Mac 시스템에서도 동일한 명령을 사용할 수 있습니다.
1. 운영 체제 호환성
- Windows 시스템의 경우 링크를 따라 WSL17 을 설치한 다음, 2.6단계로 진행합니다.
- Unix 기반 시스템(예: Linux 및 Mac)의 경우 섹션 2로 진행합니다.
참고: Unix 기반 시스템은 기본적으로 PyDesigner 및 해당 종속성을 설치할 수 있습니다.
2. FSL 설치
참고: 모든 PyDesigner 테스트는 FSL 버전 6.0.2로 수행되었으며 v6.0.2 이상을 사용하는 것이 좋습니다.
- FSL wiki18 로 이동하여 다운로드/설치를 선택합니다. Windows 사용자의 경우 2.6단계로 이동합니다.
- Installing FSL(FSL 설치) 제목 아래에서 Download FSL(FSL 다운로드 )을 선택합니다. 그런 다음, FSL 설치 소프트웨어 라이선스를 선택하고, 계약을 읽고, 동의를 선택합니다.
- 필수 필드를 입력하고 등록을 선택합니다.
- Download FSL(FSL 다운로드) 링크(페이지의 첫 번째 링크)를 선택합니다. 그런 다음 설치 지침 링크(페이지의 두 번째 링크)를 선택합니다. 다음 지침에 따라 각 운영 체제에 대한 FSL을 다운로드합니다.
참고: FSL은 설치하는 데 약 10분이 걸립니다. - 성공적인 설치를 확인하려면 새 터미널 창에 다음을 입력합니다.
$ flirt -version. 그러면 버전 번호가 반환됩니다. 3단계로 진행합니다. - Windows 사용자의 경우 FSL19에 대한 WSL 설치 가이드를 따릅니다.
3. Conda 환경 만들기
- Python 환경을 관리하기 위해 conda 패키지를 설치합니다. 사용자는 Miniconda 또는 Anaconda를 선택할 수 있습니다.
- Miniconda/Anaconda20을 다운로드합니다. Mac 및 Linux 사용자는 3.2단계로 진행합니다.
- Windows의 경우 WSL21 내에서 conda를 설정합니다.
참고: 다음 지침은 Miniconda에 대한 것이지만 Anaconda에서도 동일한 명령이 작동합니다. conda는 더 이상 Python 3.7을 지원하지 않지만 Python 3.7로 conda 환경을 만들 수 있으므로 이 프로토콜을 설명된 대로 정확하게 구현할 수 있습니다.
- 터미널에 다음을 입력하여 conda 환경을
$ conda create -n mri python=3.7만듭니다. 모든 PyDesigner 관련 작업에 이 conda 환경을 사용합니다.- Enter 키를
y입력하라는Proceed ([y]/n)?메시지가 표시되면 .
참고: -n 플래그: 이것을 사용하여 conda 환경의 이름을 나타냅니다. 비디오에서 conda 환경의 이름은 mri입니다.
참고:python=PyDesigner는 이 버전에서 빌드되고 테스트되었으므로 버전 3.7을 사용하는 것이 좋습니다.
- Enter 키를
4. MRtrix3 설치
- 다음 명령을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - MRtrix322 의 다운로드 페이지를 열고 해당 운영 체제에 대한 다운로드 지침을 따릅니다. 설치 코드는 특정 종속성에 대한 확인을 요청합니다. 다운로드 프로세스를 계속하려면 입력하십시오
y.- MRtrix3 설명서를 인쇄하여 터미널에 다음을 입력하여 설치를 확인합니다.
$ mrinfo -h
- MRtrix3 설명서를 인쇄하여 터미널에 다음을 입력하여 설치를 확인합니다.
5. PyDesigner 설치
- 새 터미널 창을 열고 터미널에 다음을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - 다음 명령을 실행합니다.
$ pip install PyDesigner-DWI - 성공적인 설치를 확인하려면 터미널에 다음을 입력하여 버전 번호를 인쇄합니다.
$ pydesigner -v- "module not found" 오류가 나타나면 프로토콜 끝에 있는 문제 해결 섹션(섹션 10)을 참조하십시오.
참고: $ pydesigner -h 는 소프트웨어의 명령과 구문을 설명하는 도움말 문서를 인쇄합니다. PyDesigner는 Github를 통해서도 다운로드할 수 있습니다. 웹 사이트23의 설치 지침을 참조하십시오.
- "module not found" 오류가 나타나면 프로토콜 끝에 있는 문제 해결 섹션(섹션 10)을 참조하십시오.
6. 데이터 준비
참고: PyDesigner는 .nii, .nii.gz, .mif 및 DICOM과 같은 여러 파일 형식에서 작동합니다. 비디오에서 DICOM은 이 dMRI 처리 파이프라인을 사용하기 전에 정렬되어 NIfTI로 변환됩니다. NIfTI 파일은 비식별화되어 있고 시퀀스당 하나의 파일이 있기 때문에 작업하기가 더 쉽습니다.
- DICOM을 정렬하려면 DicomSort 도구24를 사용합니다.
참고: 동일한 작업을 수행하는 사용 가능한 다른 모든 DICOM 정렬 도구를 사용할 수 있습니다. - DICOM을 정렬한 후 dcm2niix25를 사용하여 NIfTI 파일로 변환합니다.
- 터미널에 다음 명령을 입력하여 dcm2niix를 설치합니다.
$ sudo apt installdcm2niix - 다운로드가 완료되면 터미널에 다음을 입력하고 dcm2nii에 대한 설명서를 인쇄하여 설치를 확인하십시오.
$ dcm2niix -h
- 터미널에 다음 명령을 입력하여 dcm2niix를 설치합니다.
- dcm2niix를 실행하려면 터미널에 다음 명령을 입력하십시오.
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
참고: -f: 출력 파일의 파일 이름 구조를 지정하려면 원하는 파일 이름 구조와 함께 이 태그를 따릅니다. 비디오는 -f %s_%p_%d 를 사용합니다. %s, %p 및 %d는 각각 계열, 프로토콜 번호 및 설명에 대한 표기법입니다. 모든 파일 구조가 작동합니다. dcm2niix의 예상 출력은 .bval, .bvec, .json 및 .nii 파일입니다. TOPUP 데이터를 획득한 경우 .json 및 .nii TOPUP 파일이 있습니다.
7. PyDesigner 기본 사용법
참고: PyDesigner는 처리 매개 변수 및 컴퓨터 기능에 따라 단일 데이터 세트에 대해 최대 1.5시간이 걸릴 수 있습니다.
- 표준 처리를 실행하려면 플래그
-s또는-standard를 사용하십시오. 이 명령은 노이즈 제거6, Gibbs-ringing 보정7, EPI 왜곡 보정8, 와전류 모션 보정9, 뇌 및 뇌척수액(CSF) 마스크 생성, 스무딩, 라이시안 바이어스 보정, 이상치 감지11, 텐서 추정 및 파라메트릭 맵 생성 순서로 권장 전처리 파이프라인을 실행합니다. - 기본 PyDesigner 명령을 실행하려면 먼저 터미널에 다음 명령을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - 다음 명령을
$ pydesigner -h입력하여 소프트웨어에 대한 도움말 문서를 인쇄합니다. 이 문서에서는 PyDesigner에 사용할 수 있는 모든 명령과 구문에 대해 설명합니다. - 다음 예제를 사용하여 명령을 구조화합니다.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - 다음과 같은 중요한 플래그를 유의하십시오.
--verbose: 이를 사용하여 데이터를 처리하는 PyDesigner 콘솔을 볼 수 있습니다(필수 아님).--rpe pairs #: TOPUP 시퀀스를 사용하는 사용자가 TOUP을 사용하여 EPI 왜곡 보정8 프로세스의 속도를 높일 수 있도록 합니다. #은 TOPUP 시퀀스에서 사용할 B0 볼륨의 수를 나타냅니다. #=1을 사용하면 왜곡 필드의 과대 추정을 방지하고 이 필드를 만드는 데 필요한 시간을 줄일 수 있습니다. 사용자는 신뢰할 수 있는 결과를 얻기 위해 일상적으로 #=1을 사용했습니다. #=1을 사용할 때 PyDesigner는 기본적으로 첫 번째 볼륨을 사용합니다.
8. 고급 PyDesigner 사용: 사용자 개인의 요구에 맞게 소프트웨어 조정
참고: PyDesigner를 조정할 때, 플래그를 생략하십시오 -s ; 이 플래그는 자동 전처리를 위한 명령입니다.
- 터미널에 다음 명령을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - 소프트웨어의 웹 사이트23 플래그 목록을 참조하십시오. 각 플래그를 사용하면 사용자가 PyDesigner의 각 부분을 개별적으로 실행할 수 있습니다.
- 명령을 구조화합니다. 사용자가 수행하려는 각 시퀀스에 대한 플래그로 시작합니다. 그런 다음 PyDesigner를 입력 및 출력 파일로 보냅니다. 예를 들어:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - 예 1: 텐서 피팅을 단독으로 실행합니다(추가 전처리 없음).
- 터미널에 다음 명령을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - 플래그 목록은 PyDesigner 웹 사이트23 을 참조하십시오. 이 예에서는 를 사용합니다
-m. -m: 이 플래그를 추가하여 텐서 피팅을 뇌 복셀 내에서만 제한합니다. 이렇게 하면 텐서 피팅 + 이상값 감지 속도가 빨라집니다.- 다음 예제를 사용하여 명령을 구조화합니다.
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
참고: 텐서 피팅에 대한 입력은 전처리된 .nii 파일입니다.
- 터미널에 다음 명령을 입력하여 conda 환경을 활성화합니다.
- 예제 2: 노이즈 제거만 실행합니다(추가 전처리 없음).
- 터미널에 다음을 입력하여 conda 환경을 활성화합니다.
$ conda activate mri - 플래그 목록은 PyDesigner 웹 사이트23 을 참조하십시오. 이 예에서는 를 사용합니다
-n. - -n: 이 플래그를 추가하여 노이즈 제거 단계를 완료합니다. 각 주체의 출력 폴더에 있는 DWI_preproccessed.nii라는 레이블이 지정된 이전에 전처리된 DWI 파일을 입력합니다.
- 다음 예제를 사용하여 명령을 구조화합니다.
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- 터미널에 다음을 입력하여 conda 환경을 활성화합니다.
9. 향후 PyDesigner 업데이트
참고: 업데이트는 웹사이트23에서 찾을 수 있습니다.
- PyDesigner를 업데이트하려면 새 터미널 창에 다음을 입력합니다.
$ pip install --upgrade PyDesigner-DWI
참고: PyDesigner가 Github16을 통해 설치된 경우 git을 사용하여 업데이트해야 합니다.
10. 설치 문제 해결
- PyDesigner를 다운로드할 때 누락된 Python 모듈이 있는 경우 "module not found" 오류 코드가 나타납니다.
- 터미널에 다음을 입력하여 누락된 모듈을 수정합니다.
$ pip install nameofmodule - 그런 다음 터미널에 다음을 입력하여 첫 번째 설치 명령을 반복합니다.
$ pip install PyDesigner-DWI - 이제 PyDesigner가 완전히 설치되어야 합니다. 5.3단계로 돌아가십시오. 동일한 오류가 발생하면 누락된 Python 모듈이 모두 설치될 때까지 이 프로세스를 반복합니다.
- 터미널에 다음을 입력하여 누락된 모듈을 수정합니다.
11. 출력 오류 문제 해결
- 문제가 발생하면 log_command.json 파일을 검토하여 전처리 오류를 식별합니다. 중간 파일을 검토하여 특정 오류를 확인합니다.
- 문제 해결에 도움이 필요하면 GitHub페이지 16에서 요청을 제출하세요.
12. 예제 데이터셋으로 PyDesigner 실행
- OpenNeuro에서 예제 데이터 세트를 다운로드하십시오26.
참고: 이 예제 데이터 세트에는 원시 데이터와 전처리된 데이터가 모두 포함되어 있습니다. 원시 데이터는 PyDesigner 예제 처리를 위한 입력으로 사용됩니다. 전처리된 데이터는 iMac OS v12.4에서 dMRI 처리 파이프라인을 사용하여 원시 데이터를 처리한 후 생성된 출력으로 구성됩니다. 사용자는 원시 데이터를 다운로드하고, PyDesigner를 사용하여 이러한 데이터를 처리하고, 출력을 제공된 예제 출력과 비교하여 PyDesigner가 올바르게 실행되고 있는지 확인할 수 있습니다. 원시 데이터는 기본 폴더(/PyDesigner Example Dataset/sub-01)에 포함되고 전처리된 데이터는 derivatives 폴더(/PyDesigner Example Dataset/derivatives/sub-01)에 포함됩니다. 달리 지정하지 않는 한 OpenNeuro 데이터 세트는 기본 사용자 폴더에 다운로드됩니다. - Mac/Linux에서 새 터미널 또는 Windows에서 WSL을 엽니다.
참고: 표준 PyDesigner 처리 파이프라인을 실행하려면 아래 예제 명령 구조를 따르십시오. 아래 명령을 터미널/WSL에 입력합니다. 이 예제에서 " ./user_download_folder/"는 예제 데이터 폴더의 경로를 나타냅니다. 이 경로는 개별 사용자 컴퓨터에서 예제 데이터가 저장되는 위치에 따라 다릅니다. - 출력을 위한 새 폴더를 만들려면 터미널에 다음을 입력합니다.
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - conda 환경을 활성화하려면 터미널에 다음을 입력합니다.
$ conda activate mri - 예제 데이터 세트에서 PyDesigner를 실행하려면 터미널에 다음을 입력합니다.
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
참고: 이 예에서 ./user_download_folder/는 예제 데이터 폴더의 경로를 나타냅니다. 이 경로는 각 사용자의 컴퓨터에 따라 다릅니다. 12.3-12.5단계의 명령은 8.3단계의 구조를 따릅니다. 이 명령은 PyDesigner의 표준 처리 플래그인 -s 플래그를 사용하며, 이 플래그는 노이즈 제거6, Gibbs 링잉 보정7, 왜곡해제 8,9, 브레인 마스킹, 스무딩 및 라이시안 바이어스 보정 순서로 처리 단계를 실행합니다. - 소프트웨어가 올바르게 설치되면 모든 출력이 새로 생성된 PyDesigner_Outputs 폴더에 채워집니다. PyDesigner에서 생성된 전처리 파일을 OpenNeuro에서 다운로드한 "derivatives" 폴더에 있는 예제 데이터셋 전처리된 폴더와 비교합니다.
- 대표 결과 섹션을 참조하여 해당 메트릭 맵을 예제 데이터 세트 결과의 메트릭 맵과 비교합니다.
결과
PyDesigner 소프트웨어는 원시 확산 데이터에 여러 이미지 보정 단계를 적용하고 분석을 수행할 때 원시 파일의 정확도를 개선하는 데 사용되는 출력을 생성합니다. 파이프라인에서 사용할 수 있는 각 단계는 소개에서 논의한 바와 같이 동료 검토 간행물 5,6,7,8,9,10,11 을 통해 이전에 검증되었습니다. 소프트웨어의 출력은 tractography 프로파일, 연결성 매트릭스, 복셀 단위 분석, ROI 분석, fODF 분석, TBSS 및 fixel 기반 분석과 같은 분석에 사용할 수 있습니다.
소프트웨어 웹사이트(23 )는 전처리 파이프라인 동안 생성된 모든 출력 파일을 나열합니다. 각 프롬프트가 실행된 후 콘솔은 완료된 모든 프로세스에 대한 설명을 출력합니다. 출력 파일에는 처리 파일, 메트릭 및 품질 관리의 3가지 유형이 있습니다. 출력 디렉토리 구조는 그림 1에 나와 있습니다. 이러한 파일은 표준 전처리를 사용할 때 사용할 수 있습니다(프로토콜의 섹션 7 참조). 사용자가 고급 사용을 필요로 하는 경우(프로토콜의 섹션 8 참조) 사용 가능한 출력 파일은 완료된 프로세스에 따라 달라집니다.
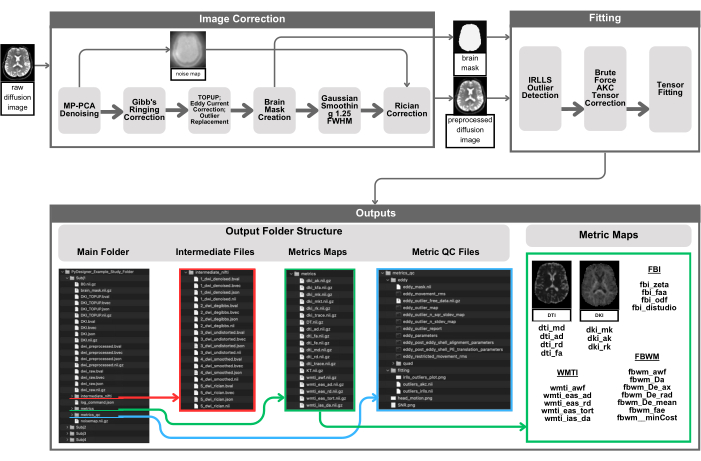
그림 1: PyDesigner 파이프라인의 시각적 표현. 전처리는 PyDesigner(왼쪽 상단)에 입력 4D DWI를 제공하는 것으로 시작하며, 그런 다음 MP-PCA 잡음 제거를 거쳐 잡음이 제거된 4D DWI 및 3D 노이즈 맵을 생성합니다. 그런 다음 잡음이 제거된 4D DWI는 Gibbs ringing correction, Rician bias correction, TOPUP, 와전류 보정 및 이상치 보정을 거칩니다. 그런 다음 브레인 마스크는 후속 단계, 이상치 감지 및 텐서 피팅을 위해 계산되어 브레인 마스크 내에서만 수행하여 계산 속도를 높입니다. 출력은 main subject processing 폴더의 하위 디렉토리(intermediate_nifti, metrics 및 metrics_qc)에서 찾을 수 있습니다. PyDesigner 출력의 스크린샷은 가능한 모든 출력의 전체 목록이 아니라 사용자가 기대할 수 있는 것에 대한 도식적인 시각적 예제를 제공하기 위한 것입니다. 최종 출력과 중간 파일 모두의 출력은 사용된 사용자 입력 데이터 및 처리 플래그에 따라 달라집니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
가공
처리 파일은 PyDesigner 파이프라인 중에 사용되며 루트 출력 디렉터리에 저장됩니다. 각 전처리 단계에서 중간 DWI 파일은 그림 1과 같이 출력 폴더 "intermediate_nifti"에 저장됩니다. 파이프라인의 각 단계를 개별적으로 평가하기 위해 처리 또는 출력과 관련된 문제를 해결할 때 이러한 파일을 참조해야 합니다.
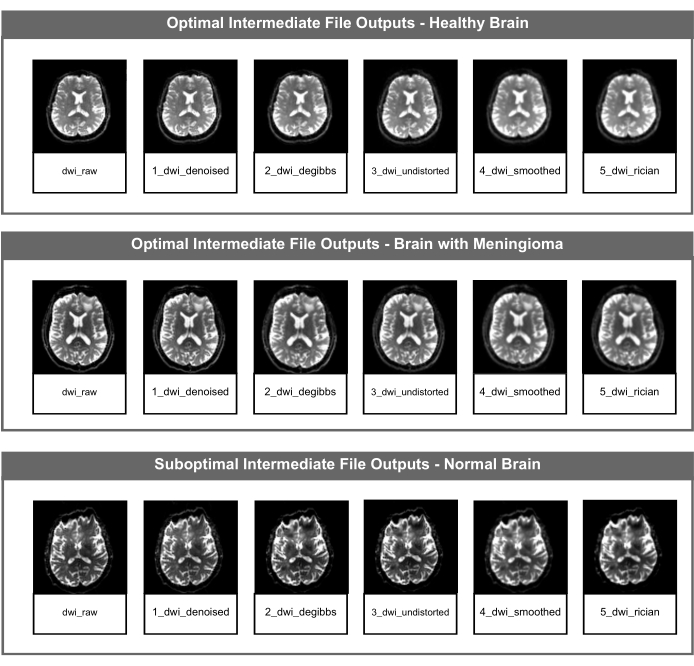
그림 2: 최적 및 차선의 중간 DWI NifTI 파일. 그림은 PyDesigner 파이프라인의 각 이미지 보정 단계에 대한 중간 NIfTI 파일을 보여줍니다. 맨 위 행은 건강한 성인 뇌의 데이터를 사용한 최적의 중간 파일 출력의 예이고, 중간 행은 병리학적 뇌(수막종)의 데이터를 사용한 최적의 중간 파일 출력의 예이며, 맨 아래 행은 뇌 구조나 건강과 관련이 없는 감수성 아티팩트를 가진 건강한 성인 뇌의 데이터를 사용하여 차선의 중간 파일 출력을 표시합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
운율학
이 폴더에는 PyDesigner가 계산하는 모든 파라메트릭 맵이 포함되어 있습니다( 그림 1 참조). 여기에는 DTI/DKI, FBI(fiber ball imaging)/FBWM(fiber ball white matter) 및 WMTI(white matter tract integrity) 메트릭에 대한 파라메트릭 맵이 포함됩니다(표 1)16.

표 1: DTI/DKI 및 FBI/FBWM 메트릭의 예상 값 범위. 이 표에는 PyDesigner에서 생성된 강력한 DTI, DKI, FBI 및 FBWM 메트릭 목록과 예상 값 범위가 포함되어 있습니다. 각 메트릭을 파생하는 데 필요한 시퀀스와 b-값(s/mm2)도 나열됩니다. 나열된 DTI 메트릭은 FA, MD, AD 및 RD입니다. 나열된 DKI 메트릭은 MK, AK, RK 및 KFA입니다. 나열된 FBI 메트릭은 FAA입니다. 나열된 FBWM 메트릭은 AWF, DA, DE_AX, DE_RAD 및 FAE입니다.
사용자는 평균 확산도(MD), 분수 이방성(FA) 및 평균 첨도(MK) 메트릭의 시각적 및 값 품질 관리(QC)를 수행하여 차선의 결과를 식별할 수 있습니다. 이러한 메트릭이 아래 설명된 표준에 의해 최적이 아닌 경우 사용자는 그림 2 에 설명된 각 중간 파일을 보고 어떤 전처리 단계가 실패했는지 확인해야 합니다.
시각적 QC는 차선의 결과(예: 텐서 피팅 문제 및 아티팩트)를 식별하는 데 사용됩니다. 소프트웨어 기본값을 통해 이미지를 조작하지 않도록 시각적 QC에 ImageJ를 사용하는 것이 좋습니다. 그림 3 의 맨 윗줄은 각각 0-3 μm2/ms, 0-1 μm2/ms 및 0-2 μm2/ms의 생물학적으로 그럴듯한 임계값을 사용하는 일반적인 MD, FA 및 MK 메트릭 맵을 보여줍니다(그림 3 [윗줄]). MD 맵은 심실에서 가장 높은 값을 가져야 하고 피질 회백질에서 높은 값을 가져야 합니다(그림 3A [맨 윗줄]). FA 지도에는 뇌 전체에 걸쳐 명확한 명백한 백질 관이 있어야 합니다(그림 3B [맨 윗줄]). MK 맵은 WM에서 높은 값을 가져야 하고 회백질 및 CSF에서 더 낮은 값을 가져야 합니다(그림 3C [맨 윗줄]). 그림 3D [맨 위 행]은 텐서 피팅 문제가 있는 메트릭 맵의 예로, 값이 0인 복셀의 클러스터가 생성됩니다. 문제가 발생하면 log_command.json 파일을 검토하여 전처리 오류를 찾습니다. 중간 파일을 검토하여 특정 오류를 확인합니다. 문제 해결에 도움이 필요하면 PyDesigner GitHub 페이지에서 요청을 제출하세요.
값 품질 관리는 주어진 메트릭 맵의 복셀이 각 데이터 세트의 주제 간에 상대적으로 균일한지 식별하는 데 사용됩니다. 각 맵 및 데이터 세트에 대한 예상 값 범위는 데이터 및 PyDesigner 매개 변수에 따라 달라집니다. 테스트 데이터 세트에서는 FSLeyes 기본 히스토그램 비닝을 사용하여 MD, FA 및 MK에 대해 각각 8000-10,000, 2500-4,000, 5,000-13,000 범위에서 일관된 피크를 보였습니다. 그림 3 의 맨 아래 행은 히스토그램 변동성의 예를 제공합니다. 표 1 에는 이러한 메트릭에 대한 예상 x축 값이 포함되어 있습니다. 더 높거나 낮은 복셀은 데이터 세트의 특징이거나 아티팩트 또는 전처리 문제를 나타냅니다(그림 3D [하단 행]).
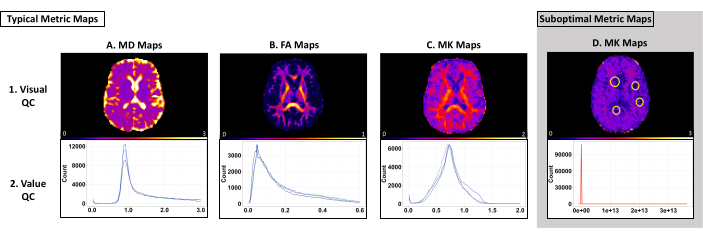
그림 3: 최적 및 최적이 아닌 결과를 제공하는 PyDesigner의 메트릭 맵 및 히스토그램의 예. 맨 위 행은 시각적 QC에 사용되는 단일 주제 MD, FA 및 MK 맵의 예를 보여줍니다. 맨 아래 행에는 값 QC에 사용되는 다중 개체 히스토그램이 표시됩니다. (에이씨) 시각적 및 가치 품질 관리를 통과하는 메트릭 맵 및 히스토그램의 일반적인 예입니다. 각 측정항목 유형에 대한 히스토그램의 각 파란색 선은 개별 데이터셋을 나타냅니다. 각 데이터 세트는 유사한 곡선을 따르며 유사한 범위 내에 있습니다. (D) 시각적 또는 값 품질 관리를 통과하지 못하는 메트릭 맵의 예. 히스토그램의 빨간색 선이 A-C와 다른 곡선을 어떻게 나타내는지 주목하십시오. 이 메트릭 맵에서 원으로 표시된 0 복셀은 전처리 중 텐서 피팅 문제 때문입니다(패널 D, 맨 위 행). 이 히스토그램은 일반적으로 데이터 세트에서 예상보다 높거나 낮은 복셀의 예입니다(패널 D, 맨 아래 행). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
품질 관리
PyDesigner가 데이터를 처리한 후에는 metrics_qc 폴더( 그림 1 참조)를 사용하여 최적화되지 않은 데이터 세트를 식별해야 합니다. 각 데이터 세트에 대해 PyDesigner는 품질 관리에 사용되는 세 개의 플롯을 출력합니다.
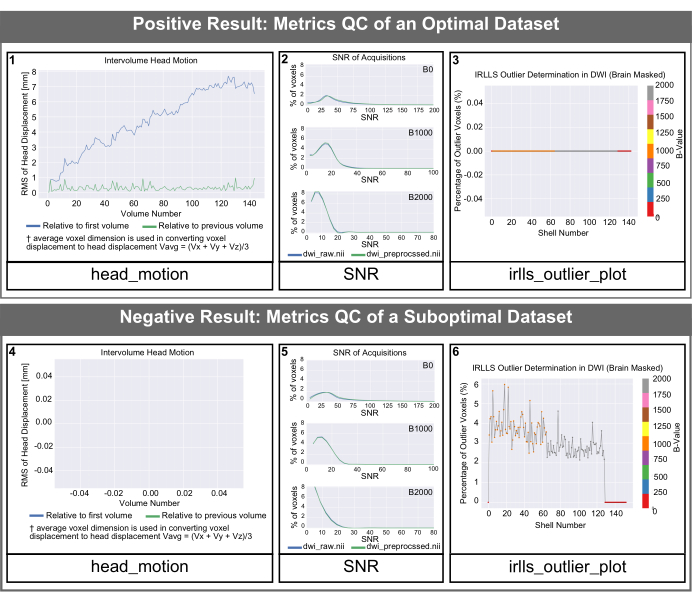
그림 4: 최적 및 최적이 아닌 데이터 세트에 대한 PyDesigner 생성 QC 히스토그램. SNR, 볼륨 간 헤드 모션 및 PyDesigner에 의해 생성된 이상값 히스토그램. 두 행 모두 건강한 성인 뇌의 데이터를 나타냅니다. 맨 위 행은 최적의 데이터 세트에 대한 품질 관리 히스토그램의 예입니다. 맨 아래 행은 뇌 구조 또는 건강과 관련이 없는 감수성 아티팩트가 있는 최적이 아닌 데이터 세트의 품질 관리 결과를 보여줍니다. PyDesigner의 기본 출력에 있는 레이블의 텍스트 크기는 이 그림에 표시되는 것보다 작습니다. 가독성을 위해 이 그림의 텍스트 크기를 늘렸습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 4의 head_motion 그래프는 첫 번째 볼륨과 이전 볼륨에 대한 상대적인 헤드 변위를 보여줍니다. 그림 4(패널 1)에서 볼 수 있듯이 헤드 변위는 일반적으로 작으며 PyDesigner는 모션 및 와전류 모션 보정9를 위한 TOUP과 함께 FSL 프로그램 Eddy를 사용하여 표준 처리 파이프라인에서 이러한 모션 아티팩트를 조정합니다. 최적이 아닌 데이터 세트의 경우 head_motion 그래프가 그림 4(패널 4)와 같이 비어 있는 것처럼 보일 수 있습니다. 이는 와전류 모션 보정이 실패하여 PyDesigner가 그래프를 출력할 수 없음을 나타냅니다. 와전류 정정 로그 파일은 metrics_qc 폴더 내의 Eddy 하위 폴더에서 찾을 수 있습니다(그림 1 참조). 신호 대 잡음비(SNR) 그래프에는 3개의 플롯이 표시됩니다. 각 플롯은 서로 다른 b-값에 대한 것이며 전처리된 데이터와 원시 데이터를 모두 표시합니다. 최적의 데이터 세트를 위해서는 원시 데이터 SNR 피크가 ≥5여야 합니다(그림 4[패널 2]). 최적화되지 않은 데이터 세트의 원시 데이터 SNR 피크는 ≤3입니다(그림 4 [패널 5]). 이상적으로 사용자는 모든 b-값에 대한 SNR 피크가 약간 증가하지만 극적으로 증가하지는 않는 것을 볼 수 있어야 합니다. 이상치 플롯은 metrics_qc 내의 피팅 폴더에 있으며 데이터 세트에서 이상치의 백분율을 보여줍니다(그림 4[패널 3 및 6]). 좋은 데이터 세트는 이상값의 비율이 낮아야 하며, 일반적으로 5% 미만이어야 합니다(그림 4 [패널 3]). 최적화되지 않은 데이터 세트는 그림 4(패널 6)에서 볼 수 있듯이 이상값의 비율이 높습니다.
예제 데이터셋 결과
PyDesigner가 예제 데이터 세트 처리를 완료하면 모든 출력이 "PyDesigner_Outputs" 폴더 내에 포함되어야 합니다. 이러한 출력은 OpenNeuro에서 다운로드한 예제 데이터 세트와 함께 패키지된 "derivatives" 폴더에 있는 출력과 비교할 수 있습니다(MacOS 12.4에서 처리됨). 소프트웨어가 올바르게 실행되면 "PyDesigner_Outputs"와 "derivatives"의 파일 구조가 정확히 동일합니다. 마찬가지로, 하위 폴더 "metrics_qc"에 있는 SNR, 머리 움직임 및 이상치 플롯은 그림 5A의 것과 일치해야 합니다. 메트릭 맵(하위 폴더 "metrics"에 있음)은 FSLeyes, MRIcron, ImageJ 등과 같은 이미징 소프트웨어를 통해 비교할 수 있습니다. 전처리된/메트릭스의 FA, MD 및 MK 값의 히스토그램은 그림 5B에서 볼 수 있습니다. 그림 5B 에 표시된 모든 메트릭 히스토그램은 표 1에 제안된 메트릭 값 스케일에 따라 조정됩니다.
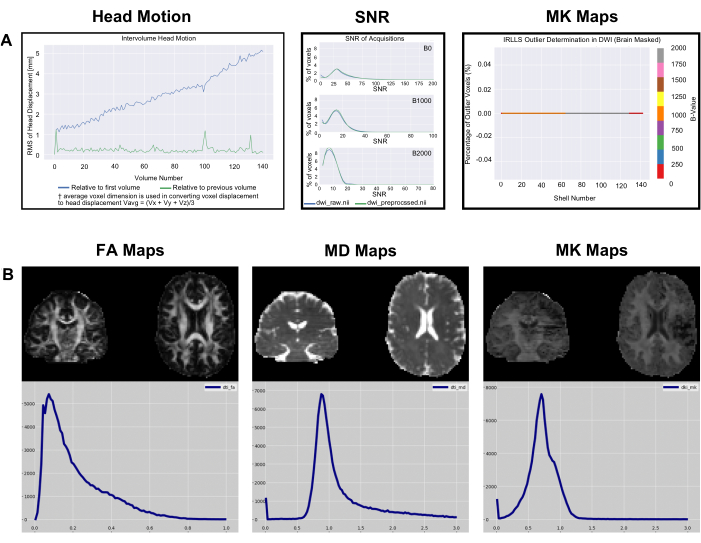
그림 5: 예제 데이터 메트릭 및 메트릭 QC 히스토그램. (A) OpenNeuro에서 다운로드한 예제 데이터에 대해 PyDesigner에서 생성한 SNR, 볼륨 간 헤드 모션 및 이상치 히스토그램. 가독성을 위해 이 그림의 플롯에서 텍스트 크기가 증가되었습니다. (B) 단일 주제 메트릭은 FSLeyes v6.0을 통해 시각화된 동일한 데이터 세트에 대한 FA, MD 및 MK에 대한 복셀 값 수의 히스토그램을 매핑합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
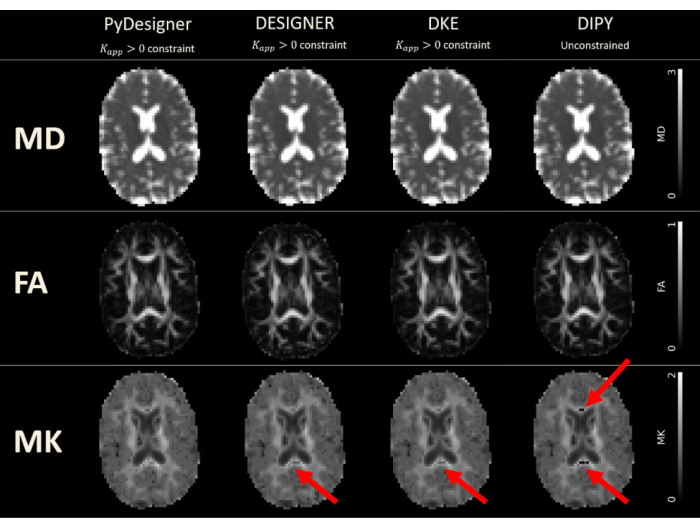
그림 6: PyDesigner, DESIGNER, DKE 및 DIPY에서 파생된 DTI 및 DKI 맵. 텐서 피팅은 PyDesigner, DESIGNER5 및 DKE(Diffusional Kurtosis Estimator)27에서 Kapp > 0 제약 조건으로 수행된 반면, 소프트웨어 제한으로 인해 DIPY(Diffusion Imaging in Python)28 에서 제약 조건이 없는 피팅이 사용되었습니다. MD의 단위는 밀리초당 제곱 마이크로미터(μm2/ms)이고 다른 메트릭은 차원이 없습니다. Gibbs 벨소리 보정의 일반적인 문제로 인한 드롭아웃은 DESIGNER, DKE 및 DIPY에서 생성한 MK 맵에서 볼 수 있습니다. 이 그림은 Dhiman et al.29의 허락을 받아 복제되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
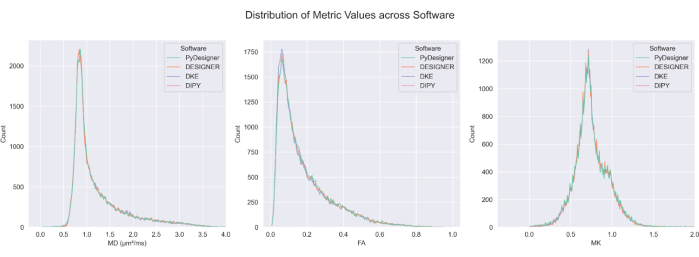
그림 7: 파이프라인 전반의 FA, MD 및 MK 비교. CSF 제외 브레인에서 PyDesigner, DESIGNER5, DKE27 및 DIPY28 의 FA, MD 및 MK에 대한 계산된 값의 분포는 대부분의 복셀에서 유사합니다. 여러 가지 방법 간의 메트릭 맵 계산은 비슷합니다. 이 그림은 Dhiman et al.29의 허락을 받아 복제되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
PyDesigner를 개발하게 된 주된 동기는 DESIGNER의 핵심 요소를 구현하는 동시에 모든 MATLAB 코드를 Python으로 대체하여 이식성과 접근성을 높이는 것이었습니다. PyDesigner와 DESIGNER Dv1은 거의 동일한 출력을 생성합니다29. 그럼에도 불구하고 PyDesigner에는 몇 가지 추가 옵션, 기본 설정 및 사소한 버그 수정이 포함되어 있습니다. 온라인 PyDesigner 문서16 에 이에 대해 자세히 설명되어 있습니다.
PyDesigner는 또한 일반적으로 사용되는 Diffusional Kurtosis Estimator (DKE)27 및 Diffusion Imaging in Python (DIPY)28 DKI 분석 도구29 (그림 6 및 그림 7 참조)와 유사한 결과를 산출하지만, DESIGNER-preprocessed dMRI를 기반으로 생성된 맵은 Ades-Aron et al.5에 의해 입증된 바와 같이 피팅 알고리즘으로 인해 더 정확한 것으로 간주됩니다. PyDesigner, DESIGNER5, DKE27 및 DIPY28의 MD, FA 및 MK 메트릭 맵을 비교하려면 그림 6을 참조하십시오. 그림 7은 각 파이프라인의 MD, FA 및 MK 히스토그램을 비교한 것입니다. 제약된 텐서 피팅, 이상치 감지 및 명백한 첨도 계수 보정을 결합하면 PyDesigner 및 DESIGNER5에서 볼 수 있듯이 보다 강력하고 정확한 텐서 피팅을 얻을 수 있습니다.
DESIGNER Dv1에 비해 PyDesigner의 장점은 NeuroDock Docker 컨테이너30을 통해 사용할 수 있어 이식성이 크게 향상되고 설치가 간단하다는 것입니다. 이 컨테이너는 Windows, Mac OS 및 다양한 Linux 배포판을 포함하여 Docker와 호환되는 모든 주요 OS 플랫폼에서 실행됩니다. 사용자 피드백에 따라 PyDesigner v2.0에는 PyDesigner Docker Container에 대한 업데이트가 포함됩니다. 새 버전의 PyDesigner와 다단계 Dockerfile의 도입으로 도입된 개선 사항은 사용자가 직면하고 있는 모든 기존 문제를 해결할 것입니다. 사용자가 설치에 문제가 있는 경우 PyDesigner 토론 페이지16에 질문을 제출하는 것이 좋습니다. 또한 Docker의 컨테이너 기술을 사용하면 Docker 호환 로컬 클러스터에서 DWI를 신속하게 일괄 처리하기 위해 고성능 클러스터(HPC)에 간단하게 배포할 수 있습니다.
PyDesigner에는 WMTI3, FBI 및 FBWM을 포함하여 DKI를 능가하는 미세 구조 모델링 계산도 포함되어 있습니다. WMTI의 경우 표준 DKI 데이터 세트가 적합하며 관련 미세 구조 매개 변수가 기본적으로 계산됩니다. 그러나 WMTI의 유효성은 FA가 높은 백질 영역(즉, FA ≥ 0.4)으로 제한된다는 점을 강조해야 합니다. 일부 WMTI 메트릭은 주어진 복셀31에서 축삭의 병렬 정렬을 가정하기 때문에 정확도가 제한됩니다. FBI 32,33,34는 대뇌 백질 전체에 적용할 수 있는 고유한 dMRI 방법으로, 높은 b-값(즉, b ≥ 4000 s/mm 2)과 최소 64개의 확산 인코딩 방향(b = 0에 대한 데이터와 함께)으로 샘플링된 dMRI 데이터가 필요합니다. FBI의 주요 출력은 각 백질 복셀에 대한 섬유 배향 밀도 함수(fODF)이며, 이는 백질 견인 조영술에 사용할 수 있고 FBWM의 입력 역할을 할 수 있을 뿐만 아니라 축삭 내 분수 이방성(FAA)입니다. FBWM은 DKI와 FBI의 dMRI 데이터를 활용하여 WMTI와 동일한 매개변수를 추정하지만 정확도가 향상되었으며 FA 값에 관계없이 백질 전체에 적용할 수 있습니다. 따라서 이 추가 데이터를 사용할 수 있는 경우 FBWM 추정치가 WMTI35의 추정치보다 선호됩니다. FBI와 마찬가지로 FBWM은 성인 대뇌 백질에서만 검증되었습니다.
PyDesigner에서 제공하는 회전 불변량(RAS 방향) 외에도 DSIstudio(LPS 방향)와 관련된 .fib 파일은 DTI, DKI 및 FBI용으로 별도로 생성됩니다. .fib 파일에는 각 방법에 대한 tractography 프로파일을 생성하기 위한 ODF 방향 정보가 포함되어 있습니다. 결과 tractography 프로파일은 주제 및 그룹 수준에서 연결 매트릭스를 생성하는 데 사용할 수 있습니다. DKI 및 FBI .fib 파일에는 DTI tractography에서 사용할 수 없는 다방향 정보 릴레이 교차 광섬유 정보가 포함되어 있습니다. 또한 각 .fib 파일 내에는 각 방법의 다양한 회전 불변량이 포함되어 있으며, 이는 tractography의 시딩, 수행 및 중지를 위한 기준으로 다양한 조합으로 사용할 수 있습니다. DSIstudio에 대한 자세한 내용은 웹사이트36에서 확인할 수 있습니다.
PyDesigner의 또 다른 주목할만한 기능은 NifTi(.nii), 압축된 NifTi(.nii.gz), DICOM(.dcm) 및 MRtrix 파일 형식(.mif)과 같은 다양한 파일 입력을 처리할 수 있는 다중 파일 입력입니다. PyDesigner는 입력 형식에 관계없이 헤더 메타데이터에서 수집 정보를 자동으로 식별하고 그에 따라 수정을 수행할 수 있으므로 핸드오프 접근 방식을 지원합니다. 프로토콜의 차이에 관계없이 동일한 명령(위 참조)을 사용하여 다양한 DWI를 처리할 수 있습니다. 따라서 이 소프트웨어는 수동 전처리 단계 및 명령을 최소화하여 시간과 노력을 절약합니다. 최근에 릴리스된 업데이트(v1.0-RC10)에서는 다중 TE(에코 시간) 데이터 세트에 대한 호환성을 도입하여 이 기능이 향상되었습니다. 이를 통해 PyDesigner는 다중 TE DWI에서 TE와 크게 독립적인 이미지 전처리 단계를 실행하여 노이즈와 아티팩트가 최소화된 이미지를 생성할 수 있습니다. 그런 다음 TE 종속 텐서 계산은 각 TE에서 개별적으로 수행되어 확산 또는 첨도 메트릭을 생성합니다.
PyDesigner v1.0은 이미지 전처리 파이프라인 설계에서 엔드포인트를 나타내지 않습니다. 이미지 처리 도구의 개발 및 검증은 활발한 연구 분야입니다. 특히, 이 원고를 준비하는 동안, 이미지 노이즈 제거 및 Gibbs 링잉 제거에 대한 새로운 개발이 제안되었으며, 그 결과 부분 푸리에 획득35로 획득한 dMRI에 대한 Gibbs 아티팩트의 향상된 노이즈 제거 및 보정을 포함하는 수정된 Designer 파이프라인 Designer Dv237이 출시되었으며 DESIGNER Github38에서 사용할 수 있습니다. 마찬가지로, BIDS39 조직 규정 준수는 모든 MRI 분석 파이프라인의 중요한 측면입니다. BIDS 형식을 사용하면 모든 MRI 데이터 유형에 대한 특정 세트 구조를 설정하여 보다 효율적인 데이터 공유 및 파이프라인 구현이 가능합니다. PyDesigner는 현재 BIDS 규격이 아니지만 PyDesigner의 향후 버전에는 BIDS 규격 출력이 포함될 예정입니다. 또한 PyDesigner는 원래 Python 버전 3.7을 사용하여 작성되었습니다. 이 글이 게시될 당시에는 Python 3.7이 구식으로 간주되었습니다. 그러나 이 원고에 포함된 정보는 Python 버전과 독립적입니다. 즉, 본 명세서에 기술된 중요한 전처리 기능(예를 들어, 노이즈 제거, 깁스 링잉 보정 등)은 설치 절차의 변경에 관계없이 동일한 작동 및 개념적 워크플로우를 따를 것이다. 중요한 것은 모든 Python 기반 소프트웨어와 마찬가지로 향후 PyDesigner 버전이 새 버전의 Python으로 업데이트된다는 것입니다. 우리는 시간이 지남에 따라 이러한 개발을 더욱 검증하고 통합하기 위해 노력합니다.
Python 버전 업데이트와 관련된 새로운 문서를 포함하여 PyDesigner의 최신 버전을 보려면 독자는 새로운 분석을 시작하기 전에 웹 사이트를 참조하고 PyDesigner에 관한 질문을 제출할 수 있는 토론 포럼에 참여하는 것이 좋습니다16. 이식성을 위한 Docker 구현은 NeuroDock30이라고 하며, 여기에는 PyDesigner와 해당 종속성이 포함되어 있어 다양한 플랫폼에서 처리할 수 있습니다.
공개
없음.
감사의 말
올리비아 혼(Olivia Horn), 대니얼 렌치(Daniel Lench), 그레이엄 워너(Graham Warner)와의 유익한 토론에 감사드립니다.
이 간행물에 보고된 연구는 미국 국립보건원(National Institutes of Health)의 보조금 R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716, T32DC014435의 지원을 부분적으로 받았습니다. 이 내용은 전적으로 저자의 책임이며 반드시 미국 국립보건원(National Institutes of Health)의 공식 견해를 나타내는 것은 아닙니다. 추가 자금은 Litwin Foundation에서 제공했습니다.
자료
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
참고문헌
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유