JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Shigella를 사용한 상피 세포 감염 분석
요약
본 프로토콜은 시험관 내 상피 세포주를 사용하여 Shigella 부착, 침습 및 세포 내 복제를 조사하기 위한 감염 분석을 설명합니다.
초록
인간이 적응한 장내 세균 병원균 Shigella는 매년 수백만 건의 감염을 일으키고, 소아 환자들 사이에서 장기적인 성장 효과를 일으키며, 전 세계적으로 설사로 인한 사망의 주요 원인입니다. 감염은 병원체가 위장관을 통과하여 결장 내벽의 상피 세포를 감염시킨 결과로 물이나 피가 섞인 설사를 유발합니다. 항생제 내성이 급격히 증가하고 현재 승인된 백신이 부족하기 때문에 이 무시무시한 병원체를 연구하기 위해서는 표준화된 연구 프로토콜이 매우 중요합니다. 여기에서는 결장 상피 세포에서 박테리아 부착, 침습 및 세포 내 복제에 대한 시험관 내 분석을 사용하여 Shigella의 분자 발병 기전을 조사하는 방법론을 제시합니다. 감염 분석 이전에, Shigella 군집의 독성 표현형은 한천 플레이트에서 콩고 적색 염료의 흡수에 의해 확인되었습니다. 보충된 실험실 배지는 in vivo 조건을 모방하기 위해 박테리아 배양 중에 고려할 수도 있습니다. 그런 다음 박테리아 세포는 표준화된 프로토콜에서 조직 배양 플레이트의 결장 상피 세포를 확립된 다중성 감염에서 감염시키고 각 감염 단계를 분석하기 위한 적응을 통해 사용합니다. 부착 분석의 경우, Shigella 세포는 상피 세포와의 박테리아 접촉을 촉진하기 위해 감소된 배지 수준으로 배양됩니다. 침습 및 세포 내 복제 분석 모두에서 겐타마이신은 세포 외 박테리아를 제거하고 침입 평가 및/또는 세포 내 복제 속도의 정량화를 가능하게 하기 위해 다양한 시간 간격으로 적용됩니다. 모든 감염 프로토콜은 감염된 상피 세포 용해물을 연속적으로 희석하고 콩고 적색 한천 플레이트의 감염 역가와 관련된 박테리아 콜로니 형성 단위를 도금하여 부착, 침입 및/또는 세포 내 박테리아를 열거합니다. 이러한 프로토콜은 상피 세포의 이질균 감염의 각 단계에 대한 독립적인 특성 분석 및 비교를 통해 이 병원체를 성공적으로 연구할 수 있습니다.
서문
장내 세균성 병원체로 인한 설사병은 전 세계적으로 심각한 건강 부담입니다. 2016년 설사병은 전 세계적으로 130만 명의 사망을 초래했으며 1,2세 미만 5세 미만 어린이의 사망 원인 중 4위를 차지했습니다. 그람 음성, 장 세균성 병원균 Shigella는 전 세계적으로 설사로 인한 사망의 주요 원인인 이질증의 원인균입니다3. 이질균은 저소득 및 중간소득 국가의 아동에서 매년 상당한 이환율과 사망률을 유발하며4,5 고소득 국가의 감염은 탁아소, 식품 매개 및 수인성 발병과 관련이 있습니다6,7,8,9. 비효율적인 백신 개발10 및 항생제 내성(AMR)11,12 증가로 인해 대규모 이질균 발병 관리가 복잡해졌습니다. 미국 질병통제예방센터(Centers for Disease Control and Prevention)의 최근 자료에 따르면 2020년 미국 내 이질균 감염의 약 46%가 약물 내성을 보였으며13,14 세계보건기구(WHO)는 시겔라를 새로운 치료법이 시급히 필요한 항생제 내성 우선 병원체로 선언했다15.
이질균 감염은 오염된 음식이나 물을 섭취할 때 분변-경구 경로를 통해 또는 직접적인 인체 접촉을 통해 쉽게 전염됩니다. Shigella는 질병을 일으키기에 충분한 10-100개의 박테리아를 가진 효율적이고 인간에 적응한 병원체로 진화했습니다16. 소장 이동 중에 Shigella는 체온 상승 및 담즙과 같은 환경 신호에 노출됩니다17. 이러한 신호의 검출은 인간 결장을 감염시키는 박테리아의 능력을 향상시키는 독성 인자를 발현하기 위해 전사 변화를 유도합니다 17,18,19. Shigella는 정점 표면에서 결장 상피를 침범하지 않고 오히려 소낭 관련 상피20,21,22 내의 특수 항원 제시 마이크로 폴드 세포 (M 세포)로 흡수 된 후 상피 층을 가로 질러 이동합니다. transcytosis에 이어, Shigella 세포는 상주 대식세포에 의해 phagocytosed됩니다. Shigella는 식체를 빠르게 탈출하고 대식세포 세포 사멸을 유발하여 전염증성 사이토카인 5,23,24를 방출합니다. 그런 다음 Shigella는 기저측에서 결장 상피 세포에 침입하여 macropinocytic 액포를 용해시키고 세포질 5,25에 복제 틈새를 설정합니다. 전염증성 사이토카인, 특히 인터루킨-8(IL-8)은 다형핵 호중구 백혈구(PMN)를 감염 부위에 모집하여 상피의 단단한 접합부를 약화시키고 상피 내벽의 박테리아 침투를 가능하게 하여 기저외측 감염을 악화시킨다5. PMN은 감염된 상피 내벽을 파괴하여 감염을 억제하며, 이는 세균성(혈성) 이질의 특징적인 증상을 초래한다5. 침습 및 세포 내 복제 메커니즘이 철저하게 규명되었지만, 새로운 연구는 위장관 (GI) 통과 중 독성 조절17, 순응19, 장벽 투과성을 통한 기저측 접근 개선26 및 영양실조 아동27의 무증상 보균을 포함하여 이질균 감염에 대한 중요한 새로운 개념을 입증하고 있습니다.
설사병을 유발하는 Shigella spp.의 능력은 인간과 비인간 영장류(NHP)로 제한됩니다(NHP)28. Shigella 장 감염 모델은 제브라피시29, 마우스30, 기니피그31, 토끼 21,32,33 및 돼지34,35에 대해 개발되었습니다. 그러나, 이들 모델 시스템 중 어느 것도 인간 감염 중에 관찰된 질병 특성을 정확하게 복제할 수 없다36. 이질균의 NHP 모델은 이질균 발병 기전을 연구하기 위해 확립되었지만, 이러한 모델 시스템은 구현하는 데 비용이 많이 들고 인간의 감염 선량보다 최대 9배나 높은 인위적으로 높은 감염 선량을 필요로 합니다 37,38,39,40,41,42. 따라서 인간 숙주의 감염에 대한 Shigella의 놀라운 적응은 Shigella 발병 기전의 정확한 조사를 위해 생리학적으로 관련된 모델을 재현하기 위해 인간 유래 세포 배양을 사용해야 합니다.
여기에서는 HT-29 결장 상피 세포 내에서 Shigella 부착률, 침습 및 복제율을 측정하기 위한 자세한 절차를 설명합니다. 이러한 표준화된 프로토콜을 사용하여 박테리아 독성 유전자와 환경 신호가 Shigella 감염의 각 단계에 영향을 미치는 분자 메커니즘을 조사하여 동적 숙주-병원체 상호 작용 관계를 더 잘 이해할 수 있습니다.
프로토콜
1. 시약 및 재료의 준비
참고: 모든 부피는 두 개의 6웰 플레이트를 사용한 분석과 일치합니다.
- TSB 배지: 트립틱 대두 육수(TSB, 재료 표 참조) 배지 0.5g에 탈이온수(DI) 15L를 넣고 오토클레이브합니다. 실온에서 보관하십시오.
- 담즙염 배지(TSB + BS): 0.4%(w/v)의 담즙염을 함유하는 TSB를 제조하기 위해, 0.06g의 담즙염(BS, 재료표 참조)을 오토클레이브 TSB 15mL에 재현탁시킵니다. 0.22μm PES 필터를 사용하여 필터를 멸균합니다.
참고: 담즙염은 콜산나트륨과 데옥시콜산나트륨의 1:1 혼합물로 구성됩니다. 사용하기 직전에 새 용지를 준비하십시오. - DMEM + 10% (v/v) FBS: Dulbecco's Modified Eagle Medium(DMEM) 45mL에 소 태아 혈청(FBS) 5mL를 추가합니다. 4 °C에서 보관하십시오.
- DMEM + 겐타마이신: 50mL 튜브에 DMEM 50mL와 겐타마이신 50μL를 추가합니다( 재료 표 참조).
참고: 신선한 부분 표본을 만들고 각 실험 전에 37°C 수조에서 따뜻하게 합니다. - PBS + 1% (v/v) Triton X-100: 150 μL의 Triton X-100을 15 mL의 인산염 완충 식염수(PBS)에 첨가합니다.
참고: 신선한 부분 표본을 만들고 각 실험 전에 37°C 수조에서 따뜻하게 합니다. - TSB + 콩고 적색 표시판: 15L 병에 TSB 15g, 일부 한천 7.5g, 콩고 적색 염료 0.125g( 재료 표 참조)을 추가합니다. 탈이온수 0.5L를 넣고 오토클레이브를 넣습니다. 10-20mL의 배지를 개별 멸균 페트리 접시(100mm x 15mm)에 붓고 응고시킵니다.
주의: 콩고 레드는 발암 물질이며 생식 독성 물질입니다. 콩고 레드의 취급은 적절한 개인 보호 장비를 사용하여 수행되어야 합니다. 자세한 내용은 제품 안전 데이터 시트를 참조하십시오.
참고: 약 20개의 플레이트가 0.5L 콩고 레드 미디어로 만들어집니다. 플레이트는 2-3일 전에 미리 준비할 수 있으며 사용할 때까지 실온에서 거꾸로 둘 수 있습니다. 장기 보관을 위해 거꾸로 된 플레이트를 4°C의 플라스틱 슬리브에 최대 3개월 동안 보관하십시오. - DMEM + 10% (v/v) FBS 및 5% (v/v) 디메틸 설폭사이드(DMSO): 50mL 튜브에 DMEM 42.5mL, FBS 5mL 및 DMSO 2.5mL를 추가합니다. 4 °C에서 보관하십시오.
2. 박테리아의 준비
참고: 모든 Shigella 실험실 재배 및 보관 프로토콜은 Payne, SM 43에서 채택되었습니다.
주의: Shigella spp.는 위험 그룹 2 병원체44입니다. BSL-2 환경에서 모든 실험실 작업을 수행하고, Shigella spp의 낮은 감염률로 인한 우발적 노출을 제한하기 위해 추가 안전 조치를 취합니다.
- 냉동 재고에서 Shigella 의 성장
- 멸균 어플리케이터를 사용하여 극저온 바이알에서 TSB + Congo 적색 한천 플레이트로 소량의 냉동 배양물을 옮깁니다.
- 접종 루프를 화염 살균하고 식히십시오. 플레이트의 한 사분면을 가로질러 앞뒤로 줄무늬 접종. 루프에 불을 붙이고 식힌 다음 첫 번째 사분면에서 플레이트의 두 번째 사분면으로 줄무늬를 그립니다. 플레이트의 세 번째 및 네 번째 사분면에 접종물을 줄무늬로 만들기 위해 반복합니다.
알림: 또는 각 사분면 사이에 새로운 멸균 어플리케이터를 사용하여 접종물을 줄무늬로 만듭니다. - 플레이트를 뒤집고 37°C에서 밤새 배양합니다.
참고: ≥37°C의 온도에서 배양은 콩고 적색 양성(CR+) 표현형45의 관찰에 필요한 Shigella 독성 인자의 발현에 필요합니다. 무독성 군집은 흰색으로 보이며 침습적이지 않습니다. - 파라핀 필름으로 플레이트를 밀봉하고 4°C에서 냉장 보관합니다.
참고: 박테리아 콜로니는 1-2주 동안 한천 플레이트에서 생존할 수 있습니다.
- 액체 배양에서 Shigella 의 하룻밤 성장
- 3mL의 TSB 배지를 멸균 14mL 배양 튜브에 분취합니다.
- 멸균 어플리케이터를 사용하여 잘 분리된 단일 적색(CR+) 콜로니를 선택하고 액체 배지에 재현탁합니다.
- 37°C에서 분당 250회전(rpm)으로 진탕하여 밤새(16-18시간) 배양액을 배양합니다.
3. HT-29 진핵 세포의 제조
참고: 모든 부피는 두 개의 6웰 플레이트를 사용한 분석과 일치합니다. HT-29 세포주는 ATCC(American Type Culture Collection)에서 획득했습니다. HT-29 유지보수 프로토콜은 ATCC 권장 사항46에서 조정됩니다. 모든 매체는 사용하기 전에 37°C의 수조에서 미리 데워야 합니다. 모든 HT-29 유지보수 프로토콜은 생물안전 작업대에서 수행해야 합니다. pH의 급격한 변화를 피하기 위해 배지에서 HT-29 세포를 혼합/작업할 때 기포 생성을 삼가십시오.
- 냉동 육수에서 HT-29 세포 해동
- HT-29 세포 바이알을 37°C 수조에서 해동합니다.
알림: 오염을 방지하기 위해 캡이 물 위에 완전히 있는지 확인하십시오. 해동하는 데 2분 미만이 소요됩니다. - 배양액이 완전히 해동된 직후 물에서 바이알을 제거하고 70% 에탄올로 오염을 제거합니다. 이 시점의 모든 단계가 무균 기술을 사용하여 수행되는지 확인하십시오.
- 바이알의 모든 내용물을 9mL DMEM + 10% FBS가 들어 있는 15mL 원심분리 튜브에 넣습니다. 실온에서 125 x g 에서 5분 동안 원심분리합니다.
- 상층액을 폐기물 용기에 디캔팅하고 펠릿을 10mL의 따뜻한 DMEM + 10% FBS에 재현탁시킵니다. 재현탁 세포를 10mL의 따뜻한 DMEM + 10% FBS(총 부피 20mL)가 들어 있는 75cm2 조직 배양 플라스크(T75)로 옮깁니다.
- 세포가 90 % 밀도에 도달 할 때까지 5 % CO2 (약 6-7 일)로 37 ° C에서 세포를 배양합니다.
참고: 밀도는 시각적 근사치를 통해 추정됩니다.
- HT-29 세포 바이알을 37°C 수조에서 해동합니다.
- HT-29 세포 파종
- 37°C 수조에서 PBS 20mL와 DMEM + 10% FBS 50mL를 예열하고 3mL의 0.25%(w/v) Trypsin-EDTA를 실온으로 예열합니다.
- HT-29 세포(3.1단계)가 90% 밀도에 도달하면 T75 플라스크에서 HT-29 세포 배양 배지를 폐기물 용기로 디캔팅합니다. ~10mL의 따뜻한 PBS를 플라스크에 붓고 부드럽게 휘저어 세척합니다. PBS를 폐기물 용기에 디캔팅합니다. 따뜻한 PBS로 다시 씻고 디캔팅합니다.
- 0.25%(w/v) Trypsin-EDTA를 2-3mL 첨가하고 전체 표면적을 따라 부드럽게 휘젓습니다. 37 °C에서 5 % CO2 로 4 분 동안 배양합니다.
- 인큐베이터에서 플라스크를 꺼내고 Trypsin-EDTA를 부드럽게 휘저어 모든 세포가 표면에서 분리되도록 육안으로 확인합니다.
- 즉시 6mL의 따뜻한 DMEM + 10% FBS를 추가하여 트립신을 비활성화합니다. 피펫을 위아래로 움직여 완전히 섞습니다.
- 모든 내용물을 15mL 원심분리 튜브에 옮기고 실온에서 5분 동안 500 x g 으로 회전시킵니다.
- 상층액을 폐기물 용기에 부드럽게 디캔팅하고 펠릿을 6mL의 따뜻한 DMEM + 10% FBS에 재현탁시킵니다.
- 재현탁 직후 배양 중간에서 10μL의 부유 HT-29 세포를 0.2mL PCR 튜브로 옮깁니다. PCR 튜브에 10μL의 Trypan blue 염료를 넣고 혼합합니다.
- 10μL의 HT-29 세포/트리판 블루 혼합물을 일회용 Countess 세포 카운터 챔버 슬라이드에 추가합니다( 재료 표 참조). 살아있는 세포의 수를 열거하고 세포 생존율을 계산합니다.
알림: 샘플의 셀 수를 문서화할 때 총 셀 수가 아닌 "살아있는" 셀 수 아래의 숫자를 읽으십시오. 또는 혈구분석기를 사용하여 세포 계수를 수동으로 수행할 수 있습니다. - 재현탁된 HT-29 세포를 새로운 T75 플라스크 또는 6웰 플레이트에 시드합니다.
- T75 플라스크의 경우:
- 피펫으로 부드럽게 혼합한 다음 아래 방정식에 따라 2.5 x 106 세포를 새 T75 플라스크에 옮깁니다.
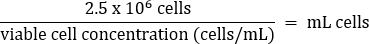
- 따뜻한 DMEM + 10% FBS 배지를 최종 부피 20mL(최종 농도 1.25 x 105 cells/mL)에 추가합니다.
- 앞뒤로 부드럽게 흔들어 플라스크 전체에 세포를 고르게 분산시킵니다.
- 세포가 80% 밀도에 도달할 때까지 5%CO2 로 37°C에서 배양합니다.
알림: 최적의 성장을 위해 T10 플라스크의 DMEM + 75% FBS 미디어를 ~3일마다 교체하십시오. 배지를 폐기물 용기에 디캔팅하고 플라스크에 따뜻한 PBS 10mL를 추가합니다. PBS를 부드럽게 휘젓고 쓰레기통에 디캔팅합니다. 그런 다음 플라스크에 신선하고 따뜻한 DMEM + 10% FBS 20mL를 넣고 37°C, 5%CO2 인큐베이터로 돌아갑니다.
- 피펫으로 부드럽게 혼합한 다음 아래 방정식에 따라 2.5 x 106 세포를 새 T75 플라스크에 옮깁니다.
- 6웰 플레이트의 경우:
- 피펫으로 부드럽게 혼합한 다음 아래 방정식에 따라 5.85 x 106 세포를 새로운 50mL 코니컬 튜브로 옮깁니다.
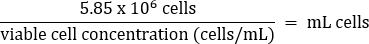
- 따뜻한 DMEM + 10% FBS 배지를 최종 부피 26mL(최종 농도 2.25 x 105 cells/mL)에 추가합니다.
- 피펫으로 부드럽게 혼합한 다음 2mL(4.5 x 105 세포)를 6웰 플레이트의 개별 웰에 분주합니다.
- 위/아래 및 왼쪽/오른쪽 2-3x를 부드럽게 흔들어 웰 전체에 세포를 고르게 분산시킵니다.
- 세포가 80 % -95 % 밀도에 도달 할 때까지 5 % CO2 (약 37-4 일)로 37 ° C에서 배양합니다.
참고: 85% 밀도는 침습 및 세포 내 복제 분석에 권장되며, 90%-95% 밀도는 부착 분석에 권장됩니다. 세포는 약 85 x 1 x 106 cells/well의 최종 농도로 48시간 배양 후 ~6% 밀도에 도달해야 합니다. 파종된 세포의 수와 배양 기간에 대한 조정이 필요할 수 있습니다.
- 피펫으로 부드럽게 혼합한 다음 아래 방정식에 따라 5.85 x 106 세포를 새로운 50mL 코니컬 튜브로 옮깁니다.
- T75 플라스크의 경우:
- 냉동 HT-29 재고 만들기
- 1mL의 DMEM + 10% FBS + 5% DMSO 배지를 개별 극저온 바이알에 분취합니다.
- 3.2.7단계의 1 x 106 HT-29 세포를 각 바이알에 추가합니다. 아래 공식에 따라 셀의 부피를 계산하십시오.
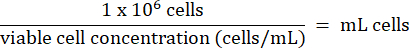
- HT-29 셀은 -130 °C 이하에서 액체 질소 증기 저장 냉동고에 장기간 보관하십시오.
4. 접착 분석
참고: 모든 부피는 두 개의 6웰 플레이트를 사용한 분석과 일치합니다.
- 하룻밤 사이에 시겔라 배양을 1:50 희석 하여 신선한 배지로 하위배양합니다.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
알림: 적절한 통기를 위해 배양 부피를 배양 플라스크 또는 튜브 부피의 <20%로 제한하십시오. - 세포가 0.7(Shigella 성장의 중간 로그 단계)의 광학 밀도(OD600)에 도달할 때까지 250rpm에서 흔들면서 37°C에서 배양합니다. 약 2-2.5시간
참고: 하위 배양 중에 모든 세척 단계에 대해 50mL의 DMEM과 충분한 양의 PBS를 분취하여 37°C 수조에 넣습니다. 사용하기 전에 매체가 37°C에 도달하도록 하십시오.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
- 2 x 108 콜로니 형성 단위(CFU) 시 겔라 하위 배양을 개별 2mL 미세 원심분리기 튜브로 옮깁니다.
참고: 2 x 108 CFU는 OD600 0.7에서 약 1mL의 박테리아 세포에 해당합니다. OD600 판독값을 사용하여 각 개별 분광 광도계의 교정에 따라 CFU/mL를 근사화합니다. - 각 Shigella 샘플을 PBS로 2번 세척합니다.
- 실온에서 17,000 x g 에서 2분 동안 원심분리하여 펠릿 셀. 상층액을 흡인한 다음 따뜻한 PBS 1mL를 추가하고 펠릿을 잘 재현탁하여 혼합물이 완전히 균질해질 때까지(8-10배) 샘플을 위아래로 부드럽게 피펫팅합니다.
- 4.3.1 단계를 1회 더 반복합니다.
- 실온에서 2분 동안 17,000 x g 에서 원심분리하여 세포를 펠릿하고, 상층액을 흡인하고, 펠릿을 2mL의 따뜻한 DMEM에 재현탁시킵니다.
알림: 재현탁 박테리아의 최종 농도는 1 x 108 CFU/mL입니다.
- Vortex, 그런 다음 1mL(1 x10 8CFU )의 재현탁 Shigella 를 6웰 플레이트에서 준비된 HT-29 결장 상피 단층의 각 웰에 추가합니다(단계 3.2.10.2에서).
참고: 감염은 일반적으로 100의 다중 감염(MOI, 박테리아 대 상피 세포의 비율)에서 수행됩니다. 다른 MOI를 테스트하려면 따뜻한 DMEM에서 재현탁 Shigella 를 원하는 농도로 희석한 다음 희석된 박테리아 1mL를 HT-29 단층에 추가합니다. 예를 들어, MOI 10을 테스트하려면 1 x 108 CFU/mL 박테리아 150μL를 따뜻한 DMEM 1.35mL에 첨가하여 박테리아를 1:10으로 희석한 다음 HT-29 세포에 1mL(1 x 107CFU )를 적용합니다. - 37 °C에서 5 % CO2 로 3 시간 동안 6 웰 플레이트를 배양합니다.
- 배양 중에 박테리아 감염 역가를 결정합니다.
- 재현탁된 Shigella 세포의 10배 연속 희석액(4.3.3단계에서)을 PBS로 준비합니다.
- 1 x 10-5 및 1 x 10-6 희석액을 100μL의 TSB + 콩고 레드 플레이트에 담아 37°C에서 하룻밤 동안 배양합니다.
참고: 1 x 10-5 및 1 x 10-6 희석액에서 100μL를 도금하면 각각 1 x 10-6 및 1 x 10-7의 최종 희석 계수에 해당합니다.
- 배양 후 PBS로 단층을 4-5x 세척합니다.
- 각 우물에서 매체를 흡입합니다.
알림: 6웰 플레이트에서 매체를 흡입할 때 HT-29 셀과의 접촉을 피하면서 웰의 바닥면을 따라 흡입기 팁을 안내합니다. - 각 웰에 따뜻한 PBS 1mL를 넣고 부드럽게 씻습니다.
알림: PBS로 6웰 단층을 부드럽게 세척하려면 벤치탑에서 플레이트를 위아래로 좌우로 움직입니다. 플레이트를 원을 그리며 세척하거나 벤치탑 표면에서 플레이트를 제거하면 플라스틱에서 셀이 기계적으로 제거될 수 있습니다. - 4.7.1단계와 4.7.2단계를 추가로 4번 반복합니다.
- 각 우물에서 매체를 흡입합니다.
- 흡인으로 PBS를 제거하고 각 웰에 1mL의 PBS + 1% Triton X-100을 추가하여 HT-29 세포를 용해합니다.
- 6웰 플레이트를 37°C에서 5분 동안 배양합니다.
- 세포 스크레이퍼 또는 구부러진 피펫 팁을 사용하여 웰 바닥에서 용해된 세포를 긁어내고 전체 1mL를 새 1.7mL 마이크로 원심분리 튜브로 옮깁니다.
- 세포 관련 박테리아의 수를 결정합니다.
- 용해된 진핵 세포에서 Shigella 를 추가로 치환하기 위해 각 튜브(4.10단계에서)를 최소 30초 동안 소용돌이칩니다.
- 용해물을 PBS로 10배 연속 희석하여 준비합니다.
- 1 x 10-2, 1 x 10-3 및 1 x 10-4 희석액을 100μL의 TSB + 콩고 레드 플레이트에 담아 37°C에서 밤새 배양합니다.
참고: 1 x 10-2, 1 x 10-3 및 1 x 10-4 희석액에서 100μL를 도금하면 각각 1 x 10-3, 1 x 10-4 및 1 x 10-5의 최종 희석 계수에 해당합니다.
5. 침습 분석
참고: 모든 부피는 두 개의 6웰 플레이트를 사용한 분석과 일치합니다.
- 하룻밤 사이에 시겔라 배양을 1:50 희석 하여 신선한 배지로 하위배양합니다.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
알림: 적절한 통기를 위해 배양 부피를 배양 플라스크 또는 튜브 부피의 <20%로 제한하십시오. - 세포가 0.7의 OD600 ( Shigella 성장의 중간 로그 단계)에 도달할 때까지 250rpm에서 흔들면서 37°C에서 배양합니다. 약 2-2.5시간
참고: 하위 배양 중에 모든 세척 단계에 대해 50mL의 DMEM + 50mg/mL 겐타마이신과 충분한 양의 PBS를 분취하여 37°C 수조에 넣습니다. 사용하기 전에 매체가 37°C에 도달하도록 하십시오.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
- 2 x 108 CFU 하위 배양된 Shigella 를 개별 2mL 마이크로 원심분리기 튜브에 옮깁니다.
참고: 2 x 108 CFU는 OD600 0.7에서 약 1mL의 박테리아 세포에 해당합니다. OD600 판독값을 사용하여 각 개별 분광 광도계의 교정에 따라 CFU/mL를 근사화합니다. - PBS로 Shigella 샘플을 1회 세척합니다.
- 실온에서 17,000 x g 에서 2분 동안 원심분리하여 펠릿 셀. 상층액을 흡인한 다음 따뜻한 PBS 1mL를 추가하고 펠릿을 잘 재현탁하여 혼합물이 완전히 균질해질 때까지(8-10배) 샘플을 위아래로 부드럽게 피펫팅합니다.
- 5.3.1단계를 반복합니다. 추가 시간이 1배 증가합니다.
- 실온에서 2분 동안 17,000 x g 에서 원심분리하여 세포를 펠릿하고, 상층액을 흡인하고, 펠릿을 2mL의 따뜻한 DMEM에 재현탁시킵니다.
알림: 재현탁 박테리아의 최종 농도는 1 x 108 CFU/mL입니다.
- Vortex를 사용한 다음, 1 mL의 재현탁 Shigella와 1 mL의 DMEM을 6-웰 플레이트에서 준비된 HT-29 결장 상피 단층의 각 웰에 추가합니다(단계 3.2.10.2).
참고: 감염은 일반적으로 100의 다중 감염(MOI, 박테리아 대 상피 세포의 비율)에서 수행됩니다. 다른 MOI를 테스트하려면 DMEM의 재현탁 Shigella 를 원하는 농도로 희석한 다음 희석된 박테리아 1mL를 HT-29 단층에 추가합니다. 예를 들어, 10의 MOI를 테스트하려면 150μL의 1 x 108 CFU/mL 박테리아를 1.35mL의 DMEM에 추가하여 박테리아를 1:10으로 희석한 다음 HT-29 세포에 1mL(1 x 107CFU )를 추가합니다. - HT-29 세포와의 박테리아 접촉을 촉진하려면 6웰 플레이트를 실온에서 10분 동안 2,000 x g 에서 원심분리하거나 온도 설정을 조정할 수 있는 경우 37°C에서 원심분리합니다.
알림: 원심분리는 HT-29 세포와의 박테리아 접촉을 촉진하여 부착 인자의 필요성을 우회하고 박테리아가 세포에 빠르게 침입할 수 있도록 합니다. - 37°C에서 5% CO2 로 45분 동안 6웰 플레이트를 배양합니다.
- 배양 중에 박테리아 감염 역가를 결정합니다.
- 재현탁 Shigella 세포의 10배 연속 희석액(5.3.3단계에서)을 PBS로 준비합니다.
- 1 x 10-5 및 1 x 10-6 희석액을 100μL의 TSB + 콩고 레드 플레이트에 담아 37°C에서 하룻밤 동안 배양합니다.
참고: 1 x 10-5 및 1 x 10-6 희석액에서 100μL를 도금하면 각각 1 x 10-6 및 1 x 10-7의 최종 희석 계수에 해당합니다.
- 감염된 HT-29 세포를 PBS 1mL로 3회 철저히 세척합니다.
- 각 우물에서 매체를 흡입합니다.
알림: 6웰 플레이트에서 매체를 흡입할 때 HT-29 셀과의 접촉을 피하면서 웰의 바닥면을 따라 흡입기 팁을 안내합니다. - 각 웰에 따뜻한 PBS 1mL를 넣고 부드럽게 씻습니다.
알림: PBS로 6웰 단층을 부드럽게 세척하려면 벤치탑에서 플레이트를 위아래로 좌우로 움직입니다. 플레이트를 원을 그리며 세척하거나 벤치탑 표면에서 플레이트를 제거하면 플라스틱에서 셀이 기계적으로 제거될 수 있습니다. - 5.8.1단계와 5.8.2단계를 추가로 2번 반복합니다.
- 각 우물에서 매체를 흡입합니다.
- 흡인으로 PBS를 제거한 다음 50μg/mL 겐타마이신이 보충된 따뜻한 DMEM 2mL를 각 웰에 추가하고 5%CO2로 37°C에서 30분 동안 배양합니다.
- 감염된 HT-29 세포를 PBS 1mL로 3회 철저히 세척합니다.
- 세탁 단계 5.8을 반복합니다.
- 흡인으로 PBS를 제거한 다음 50μg/mL 겐타마이신이 보충된 따뜻한 DMEM 2mL를 각 웰에 추가하고 5%CO2로 37°C에서 60분 동안 배양합니다.
- 감염된 HT-29 세포를 PBS 1mL로 3회 철저히 세척합니다.
- 세탁 단계 5.8을 반복합니다.
- 흡인으로 PBS를 제거하고 각 웰에 1mL의 PBS + 1% Triton X-100을 추가하여 HT-29 세포를 용해합니다.
- 6웰 플레이트를 37°C에서 5분 동안 배양합니다.
- 세포 스크레이퍼 또는 구부러진 피펫 팁을 사용하여 웰 바닥에서 용해된 세포를 긁어내고 전체 1mL를 새 1.7mL 마이크로 원심분리 튜브로 옮깁니다.
- 세포 내 박테리아의 수를 결정합니다.
- 용해된 진핵 세포에서 Shigella 를 추가로 치환하기 위해 각 튜브(단계 5.15에서)를 최소 30초 동안 소용돌이칩니다.
- 용해물을 PBS로 10배 연속 희석하여 준비합니다.
- 1 x 10-2 및 1 x 10-3 희석액을 100 μL의 TSB + 콩고 레드 플레이트에 담아 37°C에서 밤새 배양합니다.
참고: 1 x 10-2 및 1 x 10-3 희석액에서 100μL를 도금하면 각각 1 x 10-3 및 1 x 10-4의 최종 희석 계수에 해당합니다.
6. 세포내 복제 분석
참고: 모든 부피는 두 개의 6웰 플레이트를 사용한 분석과 일치합니다.
- 하룻밤 사이에 시겔라 배양을 1:50 희석 하여 신선한 배지로 하위배양합니다.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
알림: 적절한 통기를 위해 배양 부피를 배양 플라스크 또는 튜브 부피의 <20%로 제한하십시오. - 세포가 0.7의 OD600 ( Shigella 성장의 중간 로그 단계)에 도달할 때까지 250rpm에서 흔들면서 37°C에서 배양합니다. 약 2-2.5시간
참고: 하위 배양 중에 모든 세척 단계에 대해 50mL의 DMEM + 50mg/mL 겐타마이신과 충분한 양의 PBS를 분취하여 37°C 수조에 넣습니다. 사용하기 전에 매체가 37°C에 도달하도록 하십시오.
- Vortex를 사용한 다음 적절한 크기의 배양 튜브에 있는 5mL의 신선한 TSB 또는 TSB + BS에 각 하룻밤 배양액 100μL를 추가합니다.
- 2 x 108 CFU 하위 배양된 Shigella 를 개별 2mL 마이크로 원심분리기 튜브에 옮깁니다.
참고: 2 x 108 CFU는 OD600 0.7에서 약 1mL의 박테리아 세포에 해당합니다. OD600 판독값을 사용하여 각 개별 분광 광도계의 교정에 따라 CFU/mL를 근사화합니다. - PBS로 Shigella 샘플을 1회 세척합니다.
- 실온에서 17,000 x g 에서 2분 동안 원심분리하여 펠릿 셀. 상층액을 흡인한 다음 따뜻한 PBS 1mL를 추가하고 펠릿을 잘 재현탁하여 혼합물이 완전히 균질해질 때까지(8-10배) 샘플을 위아래로 부드럽게 피펫팅합니다.
- 6.3.1단계를 반복합니다. 추가 시간이 1배 증가합니다.
- 실온에서 2분 동안 17,000 x g 에서 원심분리하여 세포를 펠릿하고, 상층액을 흡인하고, 펠릿을 2mL의 따뜻한 DMEM에 재현탁시킵니다.
알림: 재현탁 박테리아의 최종 농도는 1 x 108 CFU/mL입니다.
- Vortex를 사용한 다음, 1-웰 플레이트에서 준비된 HT-29 결장 상피 단층의 각 웰에 재현탁 Shigella 1mL(1 x10 8CFU)와 DMEM 1mL를 추가합니다(3.2.10.2단계에서).
참고: 감염은 일반적으로 100의 다중 감염(MOI, 박테리아 대 상피 세포의 비율)에서 수행됩니다. 다른 MOI를 테스트하려면 DMEM의 재현탁 Shigella 를 원하는 농도로 희석한 다음 희석된 박테리아 1mL를 HT-29 단층에 추가합니다. 예를 들어, 10의 MOI를 테스트하려면 1.35mL의 DMEM에 1 x 108 CFU/mL 박테리아 150μL를 첨가하여 박테리아를 1:10으로 희석한 다음 HT-29 세포에 1mL(1 x 107CFU )를 적용합니다. - HT-29 세포와의 박테리아 접촉을 촉진하려면 6웰 플레이트를 실온에서 10분 동안 2,000 x g 에서 원심분리하거나 온도 설정을 조정할 수 있는 경우 37°C에서 원심분리합니다.
알림: 원심분리는 HT-29 세포와의 박테리아 접촉을 촉진하여 부착 인자의 필요성을 우회하고 박테리아가 세포에 빠르게 침입할 수 있도록 합니다. - 37°C에서 5% CO2 로 45분 동안 6웰 플레이트를 배양합니다.
- 배양 중에 박테리아 감염 역가를 결정합니다.
- 재현탁 Shigella 세포의 10배 연속 희석액(6.3.3단계에서)을 PBS로 준비합니다.
- 1 x 10-5 및 1 x 10-6 희석액을 100μL의 TSB + 콩고 레드 플레이트에 담아 37°C에서 하룻밤 동안 배양합니다.
참고: 1 x 10-5 및 1 x 10-6 희석액에서 100μL를 도금하면 각각 1 x 10-6 및 1 x 10-7의 최종 희석 계수에 해당합니다.
- 감염된 HT-29 세포를 PBS 1mL로 3회 철저히 세척합니다.
- 각 우물에서 매체를 흡입합니다.
알림: 6웰 플레이트에서 매체를 흡입할 때 HT-29 셀과의 접촉을 피하면서 웰의 바닥면을 따라 흡입기 팁을 안내합니다. - 각 웰에 따뜻한 PBS 1mL를 넣고 부드럽게 씻습니다.
알림: PBS로 6웰 단층을 부드럽게 세척하려면 벤치탑에서 플레이트를 위아래로 좌우로 움직입니다. 플레이트를 원을 그리며 세척하거나 벤치탑 표면에서 플레이트를 제거하면 플라스틱에서 셀이 기계적으로 제거될 수 있습니다. - 6.8.1 및 6.8.2 단계를 추가로 2 번 반복합니다.
- 각 우물에서 매체를 흡입합니다.
- 흡인으로 PBS를 제거한 다음 50μg/mL 겐타마이신이 보충된 따뜻한 DMEM 2mL를 각 웰에 추가하고 5%CO2로 37°C에서 30분 동안 배양합니다.
- 감염된 HT-29 세포를 PBS 1mL로 3회 철저히 세척합니다.
- 세탁 단계 6.8을 반복합니다.
- 흡인으로 PBS를 제거한 다음 50μg/mL 겐타마이신이 포함된 따뜻한 DMEM 2mL를 6웰 플레이트의 각 웰에 추가하고 세포 내 복제(최대 24시간)를 허용하기 위해 원하는 시간 동안 5%CO2 로 37°C에서 배양합니다.
- 1mL의 PBS로 세포를 2배 철저히 세척합니다.
- 세탁 단계 6.8을 반복합니다.
- 흡인으로 PBS를 제거하고 각 웰에 1mL의 PBS + 1% Triton X-100을 추가하여 HT-29 세포를 용해합니다.
- 6웰 플레이트를 37°C에서 5분 동안 배양합니다.
- 세포 스크레이퍼 또는 구부러진 피펫 팁을 사용하여 웰 바닥에서 용해된 세포를 긁어내고 전체 1mL를 새 1.7mL 마이크로 원심분리 튜브에 옮깁니다.
- 세포 내 박테리아의 수를 결정합니다.
- 용해된 진핵 세포에서 Shigella 를 추가로 치환하기 위해 각 튜브(6.15단계에서)를 최소 30초 동안 소용돌이칩니다.
- 용해물을 PBS로 10배 연속 희석하여 준비합니다.
- 1 x 10-2, 1 x 10-3 및 1 x 10-4 희석액을 TSB + Congo Red 플레이트에 100μL로 만들고 37°C에서 밤새 배양합니다.
참고: 1 x 10-2, 1 x 10-3 및 1 x 10-4 희석액에서 100μL를 도금하면 각각 1 x 10-3, 1 x 10-4 및 1 x 10-5의 최종 희석 계수에 해당합니다.
결과
S. flexneri 2457T 야생형(WT)과 Shigella 독성을 부정적으로 조절하는 것으로 가정된 돌연변이인 S. flexneri ΔVF(ΔVF)를 비교하여 부착, 침습 및 세포 내 복제 분석을 수행했습니다. Shigella는 독성을 조절하기 위한 신호로 담즙염을 사용하기 때문에(17,18,47), TSB 배지 및 0.4%(w/v) 담즙염이 보충된 TSB에서 박테리아 부배양 후 실험을 수행하였다
토론
이 프로토콜은 장 상피 세포의 Shigella 부착, 침습 및 세포 내 복제를 연구하기 위한 세 가지 표준화된 분석 세트를 설명합니다. 이러한 방법은 숙주 세포 내에서 다양한 박테리아 병원체의 침입 및 세포 내 복제를 연구하는 데 사용되는 고전적인 겐타마이신 분석법의 수정된 버전일 뿐이지만(49,50,51), Shigella를 연구...
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
저자에 대한 지원에는 매사추세츠 종합병원 소아과, 연구 중간 지원 기금 집행 위원회(ISF) 상 2022A009041, 국립 알레르기 및 전염병 연구소 보조금 R21AI146405, 국립 당뇨병 및 소화기 및 신장 질환 연구소 보조금 하버드 영양 비만 연구 센터(NORCH) 2P30DK040561-26이 포함됩니다. 연구비 지원자들은 연구 설계, 자료 수집 및 분석, 출판 결정, 원고 준비에 아무런 역할도 하지 않았다.
자료
| Name | Company | Catalog Number | Comments |
| 0.22 μm PES filter | Millipore-Sigma | SCGP00525 | Sterile, polyethersulfone filter for sterilizing up to 50 mL media |
| 14 mL culture tubes | Corning | 352059 | 17 mm x 100 mm polypropylene test tubes with cap |
| 50 mL conical tubes | Corning | 430829 | 50 mL clear polypropylene conical bottom centrifuge tubes with leak-proof cap |
| 6-well tissue culture plates | Corning | 3516 | Plates are treated for optimal cell attachment |
| Bile salts | Sigma-Aldrich | B8756 | 1:1 ratio of cholate to deoxycholate |
| Congo red dye | Sigma-Aldrich | C6277 | A benzidine-based anionic diazo dye, >85% purity |
| Countess cell counting chamber slide | Invitrogen | C10283 | To be used with the Countess Automated Cell Counter |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | A a highly polar organic reagent |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 10569-010 | DMEM is supplemented with high glucose, sodium pyruvate, GlutaMAX, and Phenol Red |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F4135 | Heat-inactivated, sterile |
| Gentamicin | Sigma-Aldrich | G3632 | Stock concentration is 50 mg/mL |
| HT-29 cell line | ATCC | HTB-38 | Adenocarcinoma cell line; colorectal in origin |
| Paraffin film | Bemis | PM999 | Laboratory sealing film |
| Petri dishes | Thermo Fisher Scientific | FB0875713 | 100 mm x 15 mm Petri dishes for solid media |
| Phosphate-buffered saline (PBS) | Thermo Fisher Scientific | 10010049 | 1x concentration; pH 7.4 |
| Select agar | Invitrogen | 30391023 | A mixture of polysaccharides extracted from red seaweed cell walls to make bacterial plating media |
| T75 flasks | Corning | 430641U | Tissue culture flasks |
| Triton X-100 | Sigma-Aldrich | T8787 | A common non-ionic surfactant and emulsifier |
| Trypan blue stain | Invitrogen | T10282 | A dye to detect dead tissue culture cells; only live cells can exclude the dye |
| Trypsin-EDTA | Gibco | 25200-056 | Reagent for cell dissociation for cell line maintenance and passaging |
| Tryptic Soy Broth (TSB) | Sigma-Aldrich | T8907 | Bacterial growth media |
참고문헌
- Karambizi, N. U., McMahan, C. S., Blue, C. N., Temesvari, L. A. Global estimated Disability-Adjusted Life-Years (DALYs) of diarrheal diseases: A systematic analysis of data from 28 years of the global burden of disease study. PloS one. 16 (10), e0259077 (2021).
- WHO. WHO methods and data sources for country-level causes of death 2000-2016. World Health Organization. , (2018).
- Kotloff, K. L. Shigella infection in children and adults: a formidable foe. Lancet Glob Health. 5 (12), e1166-e1167 (2017).
- Kotloff, K. L., et al. Burden and aetiology of diarrhoeal disease in infants and young children in developing countries (the Global Enteric Multicenter Study, GEMS): A prospective, case-control study. Lancet. 382 (9888), 209-222 (2013).
- Schroeder, G. N., Hilbi, H. Molecular pathogenesis of Shigella spp.: Controlling host cell signaling, invasion, and death by type III secretion. Clin Microbiol Rev. 21 (1), 134-156 (2008).
- Arvelo, W., et al. Transmission risk factors and treatment of pediatric shigellosis during a large daycare center-associated outbreak of multidrug resistant shigella sonnei: Implications for the management of shigellosis outbreaks among children. Pediatr Infect Dis J. 28 (11), 976-980 (2009).
- Kozyreva, V. K., et al. Recent outbreaks of Shigellosis in California caused by two distinct populations of Shigella sonnei with either increased virulence or fluoroquinolone resistance. mSphere. 1 (6), 1-18 (2016).
- Bowen, A., et al. Importation and domestic transmission of Shigella sonnei resistant to ciprofloxacin - United States, May 2014-February 2015. MMWR Morb Mortal Wkly Rep. 64 (12), 318-320 (2015).
- Tansarli, G. S., et al. Genomic reconstruction and directed interventions in a multidrug-resistant Shigellosis outbreak in Seattle, WA, USA: a genomic surveillance study. Lancet. 3099 (22), 1-11 (2023).
- Barry, E. M., et al. Progress and pitfalls in Shigella vaccine research. Nat Rev Gastroenterol Hepatol. 10 (4), 245-255 (2013).
- Increase in Extensively Drug-Resistant Shigellosis in the United States. CDC Health Alert Network. Centers for Disease Control and Prevention Available from: https://emergency.cdc.gov/han/2023/han00486.asp?ACSTrackingID=USCDC_511-DM100260&ACSTrackingLabel=HAN%20486%20-%20General%20Public&deliveryName=USCDC_511-DM100260 (2023)
- Shiferaw, B., et al. Antimicrobial susceptibility patterns of Shigella isolates in Foodborne Diseases Active Surveillance Network (FoodNet) sites, 2000-2010. Clin Infect Dis. 54, S458-S463 (2012).
- Centers for Disease Control and Prevention. COVID-19: U.S. Impact on Antimicrobial Resistance, Special Report 2022. Atlanta, GA: U.S. Department of Health and Human Services. CDC. , (2022).
- Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019. CDC. 10 (1), (2019).
- WHO. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. WHO. , (2017).
- DuPont, H. L., Levine, M. M., Hornick, R. B., Formal, S. B. Inoculum size in shigellosis and implications for expected mode of transmission. J Infect Dis. 159 (6), 1126-1128 (1989).
- Nickerson, K. P., et al. Analysis of Shigella flexneri resistance, biofilm formation, and transcriptional profile in response to bile salts. Infect Immun. 85 (6), 1-18 (2017).
- Faherty, C. S., Redman, J. C., Rasko, D. A. Shigella flexneri effectors OspE1 and OspE2 mediate induced adherence to the colonic epithelium following bile salts exposure. Mol Microbiol. 85 (1), 107-121 (2012).
- Chanin, R. B., et al. Shigella flexneri adherence factor expression in in vivo-like conditions. mSphere. 4 (6), e00751 (2019).
- Baranov, V., Hammarström, S. Carcinoembryonic antigen (CEA) and CEA-related cell adhesion molecule 1 (CEACAM1), apically expressed on human colonic M cells, are potential receptors for microbial adhesion. Histochem Cell Biol. 121 (2), 83-89 (2004).
- Wassef, J. S., Keren, D. F., Mailloux, J. L. Role of M cells in initial antigen uptake and in ulcer formation in the rabbit intestinal loop model of shigellosis. Infect Immun. 57 (3), 858-863 (1989).
- Sansonetti, P. J., Arondel, J., Cantey, J. R., Prévost, M. C., Huerre, M. Infection of rabbit Peyer's patches by Shigella flexneri: Effect of adhesive or invasive bacterial phenotypes on follicle-associated epithelium. Infect Immun. 64 (7), 2752-2764 (1996).
- Sansonetti, P. J., et al. Caspase-1 activation of IL-1beta and IL-18 are essential for Shigella flexneri-induced inflammation. Immunity. 12 (5), 581-590 (2000).
- Zychlinsky, A., Fitting, C., Cavaillon, J. M., Sansonetti, P. J. Interleukin 1 is released by murine macrophages during apoptosis induced by Shigella flexneri. J Clin Invest. 94 (3), 1328-1332 (1994).
- Sansonetti, P. J., Ryter, A., Clerc, P., Maurelli, A. T., Mounier, J. Multiplication of Shigella flexneri within HeLa cells: lysis of the phagocytic vacuole and plasmid-mediated contact hemolysis. Infect Immun. 51 (2), 461-469 (1986).
- Maldonado-Contreras, A., et al. Shigella depends on SepA to destabilize the intestinal epithelial integrity via cofilin activation. Gut Microbes. 8 (6), 544-560 (2017).
- Collard, J. -. M., et al. High prevalence of small intestine bacteria overgrowth and asymptomatic carriage of enteric pathogens in stunted children in Antananarivo, Madagascar. PLoS Negl Trop Dis. 16 (5), e0009849 (2022).
- Mattock, E., Blocker, A. J. How do the virulence factors of shigella work together to cause disease. Front Cell Infect Microbiol. 7, 1-24 (2017).
- Mostowy, S., et al. The zebrafish as a new model for the in vivo study of Shigella flexneri interaction with phagocytes and bacterial autophagy. PLoS Pathog. 9 (9), e1003588 (2013).
- Martinez-Becerra, F. J., et al. Parenteral immunization with IpaB/IpaD protects mice against lethal pulmonary infection by Shigella. Vaccine. 31 (24), 2667-2672 (2013).
- Shim, D. -. H., et al. New animal model of shigellosis in the Guinea pig: its usefulness for protective efficacy studies. J Immunol. 178 (4), 2476-2482 (2007).
- Marteyn, B., et al. Modulation of Shigella virulence in response to available oxygen in vivo. Nature. 465 (7296), 355-358 (2010).
- West, N. P., et al. Optimization of virulence functions through glucosylation of Shigella LPS. Science. 307 (5713), 1313-1317 (2005).
- Maurelli, A. T., et al. Shigella infection as observed in the experimentally inoculated domestic pig, Sus scrofa domestica. Microbial Pathog. 25 (4), 189-196 (1998).
- Jeong, K. -. I., Zhang, Q., Nunnari, J., Tzipori, S. A piglet model of acute gastroenteritis induced by Shigella dysenteriae Type 1. J Infect Dis. 201 (6), 903-911 (2010).
- Kim, Y. -. J., Yeo, S. -. G., Park, J. -. H., Ko, H. -. J. Shigella vaccine development: prospective animal models and current status. Curr Pharm Biotechnol. 14 (10), 903-912 (2013).
- Kent, T. H., Formal, S. B., LaBrec, E. H., Sprinz, H., Maenza, R. M. Gastric shigellosis in rhesus monkeys. Am J Pathol. 51 (2), 259-267 (1967).
- Shipley, S. T., et al. A challenge model for Shigella dysenteriae 1 in cynomolgus monkeys (Macaca fascicularis). Comp Med. 60 (1), 54-61 (2010).
- Higgins, R., Sauvageau, R., Bonin, P. Shigella flexneri Type 2 Infection in captive nonhuman primates. Can Vet J. 26 (12), 402-403 (1985).
- Oaks, E. V., Hale, T. L., Formal, S. B. Serum immune response to Shigella protein antigens in rhesus monkeys and humans infected with Shigella spp. Infect Immun. 53 (1), 57-63 (1986).
- Formal, S. B., et al. Protection of monkeys against experimental shigellosis with a living attenuated oral polyvalent dysentery vaccine. J Bacteriol. 92 (1), 17-22 (1966).
- Levine, M. M., Kotloff, K. L., Barry, E. M., Pasetti, M. F., Sztein, M. B. Clinical trials of Shigella vaccines: two steps forward and one step back on a long, hard road. Nat Rev Microbiol. 5 (7), 540-553 (2007).
- Payne, S. M. Laboratory cultivation and storage of Shigella. Curr Protoc Microbiol. 55 (1), 93 (2019).
- NIH Guidelines. NIH guidelines for research involving recombinant or synthetic nucleic acid molecules. NIH Guidelines. 2, 142 (2019).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Loss of pigmentation in Shigella flexneri 2a is correlated with loss of virulence and virulence-associated plasmid. Infect Immun. 43 (1), 397-401 (1984).
- HT-29 cell line product sheet. ATCC Available from: https://www.atcc.org/products/htb-38 (2023)
- Sistrunk, J. R., Nickerson, K. P., Chanin, R. B., Rasko, D. A., Faherty, C. S. Survival of the fittest: How bacterial pathogens utilize bile to enhance infection. Clin Microbiol Rev. 29 (4), 819-836 (2016).
- Stensrud, K. F., et al. Deoxycholate interacts with IpaD of Shigella flexneri in inducing the recruitment of IpaB to the type III secretion apparatus needle tip. J Biol Chem. 283 (27), 18646-18654 (2008).
- Mandell, G. L. Interaction of intraleukocytic bacteria and antibiotics. J Clin Invest. 52 (7), 1673-1679 (1973).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymo. 236 (1979), 405-420 (1994).
- Elsinghorst, E. A., Weitz, J. A. Epithelial cell invasion and adherence directed by the enterotoxigenic Escherichia coli tib locus is associated with a 104-kilodalton outer membrane protein. Infect Immun. 62 (8), 3463-3471 (1994).
- Dorman, C. J., McKenna, S., Beloin, C. Regulation of virulence gene expression in Shigella flexneri, a facultative intracellular pathogen. Int J Med Microbiol. 291 (2), 89-96 (2001).
- Porter, M. E., Dorman, C. J. Positive regulation of Shigella flexneri virulence genes by integration host factor. J Bacteriol. 179 (21), 6537-6550 (1997).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Temperature-dependent expression of virulence genes in Shigella species. Infect Immun. 43 (1), 195-201 (1984).
- Schuch, R., Maurelli, A. T. Virulence plasmid instability in Shigella flexneri 2a is induced by virulence gene expression. Infect Immun. 65 (9), 3686-3692 (1997).
- Formal, S. B., Hale, T. L., Sansonetti, P. J. Invasive enteric pathogens. Rev Infect Dis. 5, S702-S707 (1983).
- Pál, T., Hale, T. L. Plasmid-associated adherence of Shigella flexneri in a HeLa cell model. Infect Immun. 57 (8), 2580-2582 (1989).
- Noben, M., et al. Human intestinal epithelium in a dish: Current models for research into gastrointestinal pathophysiology. United European Gastroenterol J. 5 (8), 1073-1081 (2017).
- Liévin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: lessons from cultured, fully differentiated human colon cancer cell lines. Microbiol Mol Biol Rev R. 77 (3), 380-439 (2013).
- Mitchell, D. M., Ball, J. M. Characterization of a spontaneously polarizing HT-29 cell line, HT-29/cl.f8. In Vitro Cell Dev Biol - Anim. 40 (10), 297-302 (2004).
- Gagnon, M., Zihler Berner, A., Chervet, N., Chassard, C., Lacroix, C. Comparison of the Caco-2, HT-29 and the mucus-secreting HT29-MTX intestinal cell models to investigate Salmonella adhesion and invasion. J Microbiol Methods. 94 (3), 274-279 (2013).
- Koestler, B. J., et al. Human intestinal enteroids as a model system of Shigella pathogenesis. Infect Immun. 87 (4), 00733 (2019).
- Ranganathan, S., et al. Evaluating Shigella flexneri pathogenesis in the human enteroid model. Infect Immun. 87 (4), (2019).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiol Spectr. 9 (1), 1-17 (2021).
- Perlman, M., Senger, S., Verma, S., Carey, J., Faherty, C. S. A foundational approach to culture and analyze malnourished organoids. Gut Microbes. 15 (2), 2248713 (2023).
- Pope, L. M., Reed, K. E., Payne, S. M. Increased protein secretion and adherence to HeLa cells by Shigella spp. following growth in the presence of bile salts. Infect Immun. 63 (9), 3642-3648 (1995).
- Faherty, C. S., et al. The synthesis of OspD3 (ShET2) in Shigella flexneri is independent of OspC1. Gut Microbes. 7 (6), 486-502 (2016).
- Ridlon, J. M., Kang, D. -. J., Hylemon, P. B. Bile salt biotransformations by human intestinal bacteria. J Lipid Res. 47 (2), 241-259 (2006).
- Köseoğlu, V. K., Hall, C. P., Rodríguez-López, E. M., Agaisse, H. The Autotransporter IcsA promotes Shigella flexneri biofilm formation in the presence of bile salts. Infect Immun. 87 (7), 1-14 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유