JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
완보동물 Hypsibius exemplaris에서 qRT-PCR을 통한 상대 전사체 수준 비교를 위한 단일 동물, 단일 튜브 RNA 추출
요약
이 연구는 완보동물 Hypsibius exemplaris 에서 유전자 발현을 분석하기 위한 신속한 RNA 추출 및 전사체 수준 비교 방법을 제시합니다.물리적 용해를 사용하는 이 고처리량 방법은 출발 물질로 단일 완보동물을 필요로 하며 그 결과 정량적 역전사 중합효소 연쇄 반응(qRT-PCR)을 위한 cDNA를 강력하게 생산할 수 있습니다.
초록
완보동물인 Hypsibius exemplaris 는 극한의 환경에서도 살아남을 수 있는 능력으로 유명한 새로운 모델 유기체입니다. 이러한 극한내성의 분자 메커니즘과 유전적 기초를 탐구하기 위해 많은 연구에서 대규모 코호트에서 개별 동물에 이르는 집단에서 수행할 수 있는 RNA 염기서열 분석(RNA-seq)에 의존하고 있습니다. 역전사 중합효소연쇄반응(RT-PCR) 및 RNA 간섭(RNAi)은 이후 RNA-seq 결과를 확인하고 후보 유전자에 대한 유전적 요구 사항을 각각 평가하는 데 사용됩니다. 이러한 연구에는 정량적 RT-PCR(qRT-PCR)에 의한 RNA 추출 및 상대 전사체 수준 측정을 위한 효율적이고 정확하며 경제적인 방법이 필요합니다. 이 연구는 개별 완보동물에서 RNA를 안정적으로 분리할 뿐만 아니라 각 추출에 필요한 시간과 비용을 줄이는 효율적인 단일 완보동물, 단일 튜브 RNA 추출 방법(STST)을 제시합니다. 이 RNA 추출 방법은 정량적 PCR(qRT-PCR)에 의해 여러 전사체를 증폭하고 검출하는 데 사용할 수 있는 cDNA의 양을 생성합니다. 이 방법은 열 충격 조절 단백질인 Heat-Shock Protein 70 β2(HSP70 β2)와 Heat-Shock Protein 90α(HSP90α)의 두 가지 단백질을 암호화하는 유전자의 발현의 동적 변화를 분석하여 검증되었으며, qRT-PCR을 사용하여 열에 노출된 개인의 상대적 발현 수준을 평가할 수 있습니다. STST는 기존의 벌크 및 단일 완보동물 RNA 추출 방법을 효과적으로 보완하여 qRT-PCR에 의한 개별 완보동물 전사 수준을 빠르고 저렴하게 검사할 수 있습니다.
서문
완보동물은 대부분의 다른 형태의 생명체에게 치명적인 극한 조건에서 살아남는 능력으로 유명한 작은 다세포 동물입니다1. 예를 들어,이 동물들은 인간에게 치명적인 전리 방사선 선량의 거의 1000 배에서 살아남을 수 있습니다 2,3,4,5,6,7,8,9,10, 거의 완전한 건조 11,12,13,14,15, 첨가되지 않은 경우 동결 동결 보호제 16,17,18, 그리고 건조 상태에서는 공간의 진공 19,20도 있습니다. 극한 환경에서 생존할 수 있는 독특한 능력으로 인해 이 동물들은 복잡한 다세포 유기체의 극한 내성을 이해하기 위한 기본 모델이 되었습니다 1,21,22,23.
형질전환 및 생식세포 유전자 변형을 포함하여 이 놀라운 동물의 안정적인 유전자 조작은 최근까지 파악하기 어려웠습니다24,25. 따라서 극한내성의 분자 메커니즘을 밝히기 위한 대부분의 실험은 RNA 염기서열분석을 통한 전사 프로파일링을 통해 수행됩니다. 방사선 8,9,26,27,28, 열 스트레스 29, 동결 스트레스12 및 건조 27,30,31,32,33에 이르기까지 다양한 극한 조건에서 완보동물에 대한 많은 가치 있고 유익한 RNA 염기서열분석 데이터 세트가 존재합니다. 이러한 연구 중 일부는 극한내성에 대한 분자적 이해를 조명하기 위해 벌크 RNA 추출 및 정제 방법을 활용했습니다. 그러나 많은 동물에서 RNA 전사체를 대량으로 추출하면 개체 간의 유전자 발현 변이를 분석할 수 없으므로 보다 정제된 데이터 세트의 잠재적인 풍부함을 놓칠 수 있습니다. 중요한 것은 이러한 연구가 종종 환경 스트레스 요인에서 살아남은 동물과 그렇지 않은 동물을 모두 포함하는 이질적인 동물 개체군을 분석한다는 것입니다. 따라서 이러한 연구는 잠재적으로 극적으로 다른 여러 반응 상태의 발현 데이터를 평균화함으로써 혼동을 일으킵니다. 이 문제를 해결하기 위해 Arakawa et al., 201634는 RNA 추출 키트를 적용한 다음 단일34,35,36 또는 여러30,37,38 동물을 입력으로 사용하여 선형 PCR 증폭 단계를 적용하는 우아한 저입력 RNA-seq 파이프라인을 개발했습니다. 이러한 연구는 완보동물의 극한내성22에 대한 이해의 기초가 되었다. 흥미롭게도, 이 프로토콜은 7마리의 동물을 출발 물질로 사용하여 qRT-PCR에도 적용되었습니다24.
대부분의 모델 유기체에서 RNA-seq를 통해 잠재적 표적을 식별한 후 qRT-PCR을 수행하여 RNA-seq에 의해 식별된 전사 변화를 확인하고 고분해능 방식으로 후보 유전자의 발현 시간 경과를 평가합니다. 확인된 유전자의 기능을 테스트하기 위해 이러한 연구 후에는 종종 분자 표적39,40의 RNAi 매개 녹다운과 극한 내성 용량12,41 분석이 뒤따릅니다. 각 RNAi knockdown의 효능은 일반적으로 transcript 풍부도의 감소를 직접 모니터링하여 qRT-PCR에 의해 확인됩니다. 그러나 RNAi는 각 dsRNA가 개체의 수동 미세주입을 통해 전달되어야 하기 때문에 완보동물에서 노동 집약적인 과정입니다 39,40. 이 전략의 낮은 처리량 특성으로 인해 단일 동물에서 qRT-PCR에 맞게 조정된 빠르고 저렴한 RNA 추출 방법은 완보동물 연구에 매우 유용할 것입니다. 이전 방법은 단일 완보동물에서 RNA를 추출하기 위해 개발되었지만, 이러한 프로토콜은 추출을 qRT-PCR과 결합하지 않고 대신 광학 밀도 기반 방법 12,40,41에 의존했습니다. 이러한 과제에 동기를 부여받은 당사는 단일 H. exemplaris에서 qRT-PCR에 사용할 수 있는 RNA를 양과 질로 안정적으로 생성하는 프로토콜을 개발하고자 했습니다.
Caenorhabditis elegans42를 위해 개발된 단일 동물 RNA 추출 프로토콜에서 채택된 STST는 H. exemplaris에 최적화되어 있습니다. 추출 방법은 6개의 빠른 동결-해동 단계로 구성되며, 큐티클을 물리적으로 파괴하여 RNA 추출 및 후속 cDNA 합성을 가능하게 합니다. STST 방법은 Boothby, 201843에 설명된 바와 같이 벌크 RNA 추출 방법에 비해 추출 시간을 24배 이상 단축하고, Arakawa et al., 201634에 설명된 바와 같이 단일 완보동물 RNA 추출 키트에 비해 추출 시간을 30% 단축합니다. 또한 RNA 추출 키트 제제에 비해 샘플-실험자 상호 작용 횟수가 5회에서 1회로 감소하여 외인성 리보뉴클레아제에 의한 오염 위험이 줄어듭니다. 고도로 발현된 유전자를 쿼리할 때 STST 방법은 단일 완보동물당 25개의 정량적 RT-PCR 반응에 충분한 cDNA를 생성하며, 반응당 총 25μL cDNA 부피 중 1μL만 필요합니다. 그러나 주형 농도는 더 낮은 존재비 전사체에 대해 경험적으로 결정되어야 합니다.
35°C에서 20분간 단기간 열충격에 대응하여 열충격단백질-90α(HSP90α)와 열충격단백질70β2(HSP70β2)를 암호화하는 유전자의 차등발현을 조사하여 유전자 발현의 동적 변화를 분석하는 STST 방법의 효능을 평가하였다. 대부분의 진핵생물에서 HSP70β2와 HSP90α는 모두 단기간의 열충격에 노출(20분)된 후 빠르게 상향 조절됩니다42. H. exemplaris 에 대한 분석은 단일 열처리된 완보동물에서 추출한 HSP70β2 및 HSP90α 인코딩 RNA가 모두 단기간의 열 노출 후 통계적으로 유의한 발현 증가를 보였다는 것을 밝혔습니다. 이러한 결과는 STST 프로토콜이 시간이 지남에 따라 개별 동물의 유전자 발현의 동적 변화를 분석하는 데 사용될 수 있음을 보여줍니다.
STST 추출 방법은 빠르고 저렴한 RNA 추출과 qRT-PCR에 의한 전사체 수준의 후속 비교를 용이하게 함으로써 RNA-seq와 같은 기존 실험 방법을 보완해야 합니다. 이 방법은 또한 광학 밀도만 사용하는 것보다 수동으로 주입한 개체에서 RNAi의 효율성과 침투도를 정량적으로 평가하는 데 유용할 것입니다. 마지막으로, 이들의 유사한 표피 구조와 신체적 특성으로 인해, 이 방법은 다른 완보동물 종의 유전자 발현을 분석하는 데에도 효과적일 것으로 보인다44.
프로토콜
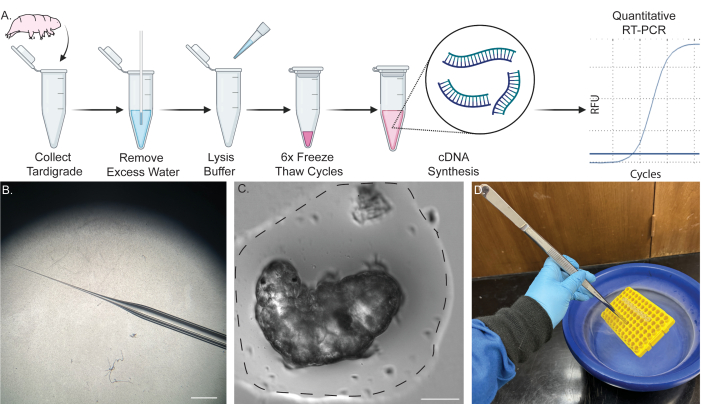
그림 1: 단일 완보동물에서 RNA 추출을 위한 단일 튜브 파이프라인. (A) 6번의 동결-해동 주기 및 후속 cDNA 합성을 포함하여 단일 완보동물에서 RNA를 추출하기 위한 프로토콜을 보여주는 계획. 샘플은 이후에 RT-PCR 및 qRT-PCR에 사용될 수 있습니다. (B) 수분 제거에 사용되는 마이크로피펫 테이퍼의 이미지. 눈금 막대 : 2mm. (C) 소량의 물에서 완보동물의 명시야 이미지 (점선). 성공적인 추출을 위해 표시된 정도까지 대부분의 수분을 제거해야 하며 용해 완충액의 희석을 방지할 수 있습니다. 눈금 막대 : 50 μm. (D) 샘플을 안전하게 빠르게 동결-해동하기 위해 긴 집게를 사용하여 샘플을 액체 질소에 담그는 모습을 보여주는 이미지. 일부 콘텐츠는 BioRender에서 제작되었습니다. Kirk, M. (2022) BioRender.com/d93s511 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고: 그림 1A는 절차의 개략도를 보여줍니다. 완보동물 및 조류 배양 절차에 대한 자세한 내용은 이전에 발표된 보고서 45,46,47을 참조하십시오.
1. 용천수 살균
- 2갤런 물통에서 5L의 샘물을 붓습니다(자세한 내용은 재료 표 참조).
- 오토클레이브 안전 병에 캡을 놓고 소량의 오토클레이브 테이프로 밀봉합니다. 병을 조이지 마십시오. 캡을 위에 놓습니다.
- 건조 단계 없이 습식 사이클에서 50분 동안 샘물을 오토클레이브합니다.
- 물이 실온(RT)에 도달하도록 하고 RT에 보관하기 전에 캡을 단단히 밀봉하십시오.
2. 유리 마이크로 피펫 당기기(피펫 풀러 사용)
- 유리 마이크로피펫(O.D.: 1mm, I.D.: 0.58mm, 길이: 10cm)을 마이크로피펫 풀러에 고정합니다. 가열 필라멘트와의 접촉은 피펫 모양을 변경하고 필라멘트를 손상시킬 수 있으므로 피펫 접촉을 피하십시오.
- 각 필라멘트와 피펫 풀러에 대해 피펫의 당김을 경험적으로 결정합니다. 그러나 최적화를 위한 시작점으로 사용하려면 78°C와 182.2g의 인장 중량의 단일 당김 단계를 사용하십시오.
- 필라멘트가 가열되고 중력이 유리 마이크로피펫을 날카로운 점이 있는 두 개의 유리 마이크로피펫으로 분리하도록 합니다(그림 1B).
- 이 뽑힌 유리 마이크로피펫을 왁스 또는 점토와 함께 밀폐된 100mm 페트리 접시에 보관하여 제자리에 고정하고 날카로운 끝이 깨지는 것을 방지하십시오.
3. 유리 마이크로피펫 당기기(피펫 풀러 없이)
- Bunsen 버너 또는 기타 제어된 화염원을 낮은 설정으로 켜십시오.
- 양손에 한쪽 끝이 있는 유리 마이크로피펫을 가져옵니다.
- 유리가 녹기 시작할 때까지 유리 마이크로피펫의 중앙을 화염 위로 잡습니다. 그런 다음 두 끝을 빠르게 잡아당깁니다. 이것은 두 개의 매우 섬세하고 날카로운 팁을 만듭니다.
- 한 쌍의 멸균 된 미세한 집게로 팁을 가볍게 부러 뜨립니다.
- 이 뽑힌 유리 마이크로피펫을 왁스 또는 점토와 함께 밀폐된 100mm 페트리 접시에 보관하여 제자리에 고정하고 날카로운 끝이 깨지는 것을 방지하십시오.
4. RNA 적출
- 극저온 안전 용기에 0.5L의 액체 질소를 넣습니다.
주의 : 액체 질소는 극저온이며 피부나 눈에 노출되면 화상을 입을 수 있습니다. 취급 시에는 보호복, 스플래시 고글, 니트릴 장갑, 냉동 장갑, 실험실 가운 및 발가락이 막힌 신발을 사용하십시오. 액체를 운송하기 전에 용기가 액체 질소에 안전한지 확인하십시오. 이 단계를 위해 에탄올 드라이 아이스 수조를 사용하는 것도 가능할 수 있습니다. - cDNA 합성 마스터 믹스 만들기: 1 μL의 무작위 헥사머 프라이머, 2 μL의 DNase, 4 μL의 5x RT 버퍼, 1 μL 효소 믹스, 1 μL의 H2O 및 1 μL의 10 mM dNTP를 포함하는 10 μL 용액. 이 용액을 얼음에 보관하십시오.
- 완보분해 완충액(5mM Tris(pH = 8), 0.5%(v/v) 세제 1, 0.5%(v/v) 세제 2, 무균 뉴클레아제가 없는 물에서 0.25mM EDTA)를 준비합니다.
참고: 이 솔루션은 벤치 탑에 6개월 동안 보관할 수 있습니다. 그러나 무균 상태를 유지하고 잠재적인 RNA 오염 소스를 피하십시오. - 추출을 위한 충분한 용해 완충액을 분취합니다(2 μL/완보).
- 완보성 용해 완충액에 RNAse 억제제를 최종 농도 4 U/μL까지 첨가합니다.
- 용액을 얼음 위에 보관하기 전에 2000 x g의 속도로 2000 x g 의 속도로 RT에서 용액을 소용돌이치고 스핀다운합니다.
- 멸균 필터 팁 P1000 피펫을 사용하여 배양에서 실험에 필요한 만큼의 완보동물을 제거하고 멸균 35mm 페트리 접시에 넣습니다.
참고: 완보동물은 이러한 방식으로 처리할 수 있습니다. 일반적으로 조건당 3개의 완보동물이 추출을 위해 처리됩니다. - 오토클레이브 멸균 스프링물 1mL와 멸균 필터 팁 P1000 피펫을 사용하여 완보동물을 세 번 세척합니다. 천천히 위아래로 피펫팅하면 조류 오염 물질을 제거하는 데 도움이 됩니다.
- 25x - 50x 배율의 해부 현미경을 사용하여 이 세척된 배양물에서 단일 완보동물을 멸균 필터 팁 P10 피펫을 사용하여 새로운 멸균 35mm 페트리 접시로 옮깁니다.
- 멸균 필터 팁 P200 피펫을 사용하여 100μL의 무멸균 뉴클레아제에서 단일 완보동물을 세척합니다.
참고: 이 세척 단계는 리보뉴클레아제를 포함한 오염 물질을 추가로 제거하는 데 사용됩니다. - 세척된 완보동물을 멸균 필터 팁 P10 피펫을 사용하여 1-2μL의 멸균 뉴클레아제가 없는 물에 담긴 깨끗한 멸균 PCR 튜브의 바닥으로 옮기고, 완보동물이 팁 측면에 달라붙지 않도록 주의합니다.
- 완보동물을 해부 현미경으로 25배 배율로 시각화합니다.
- 물을 쉽게 제거할 수 있도록 당겨진 유리 마이크로피펫의 끝을 튜브 바깥쪽에서 가볍게 부수십시오. 구멍이 물을 끌어올릴 수 있을 만큼 충분히 큰지 확인하지만 완보동물은 그렇지 않은지 확인하십시오.
- 당겨진 유리 마이크로피펫의 모세관 작용을 사용하여 동물이 직경 약 2완보동물 길이의 작은 물방울로 둘러쌀 때까지 물을 제거합니다.
- 해부 내시경을 통해 수분 제거 과정을 모니터링하여 수위가 적절하고 완보동물이 수분을 유지하는지 확인합니다.
참고: 그림 1C 는 예를 제공합니다.amp제거할 물의 양. 이것은 중요한 단계입니다. 완보동물이 마르는 것을 방지하기 위해 작은 물방울이 완보동물을 둘러싸지만, 용해 완충액이 희석되는 것을 방지하기 위해 가능한 한 많은 과도한 물을 제거해야 합니다. 예를 들어amp잔여 수위의 경우 그림 1C를 참조하십시오. - 물을 제거한 직후 튜브 바닥에 완보 용해 완충액 2μL를 추가하고 잠시 와류를 일으킨 다음 탁상용 원심분리기에서 2000 x g 에서 5초 동안 상온에서 튜브를 원심분리합니다.
- 완보동물이 포함된 샘플을 즉시 PCR 튜브 랙에 넣고 랙으로 단단히 고정되었는지 확인합니다.
- 한 쌍의 길고 거친 집게를 사용하여 랙을 잡고 샘플이 들어 있는 랙을 부드럽게 담그십시오.amp완전히 얼 때까지 액체 질소에 넣습니다(그림 1D).
- 액체 질소에서 랙을 제거하고 즉시 얼음 위에 올려 놓으십시오. 샘플을 해동시킵니다(~45초에서 1분 소요). 15초마다 얼음에서 샘플을 제거하고 육안으로 검사하여 샘플을 모니터링합니다. 샘플이 눈에 띄게 투명해지면 다음 단계로 이동합니다.
- 4.18-4.19단계를 5회 더 반복합니다. 최대 용해 및 추출을 위해 총 6번의 동결-해동 주기가 필요합니다(그림 2A, B).
- 동결-해동이 완료되면 샘플을 얼음 위에 놓고 즉시 다음 단계로 진행합니다. cDNA 준비에 사용할 수 있는 RNA를 감소시키므로 이 시점에서 보관을 위해 샘플을 동결하지 마십시오.
5. cDNA 종합
- 완보성 용해물이 포함된 PCR 튜브에 2μL의 cDNA 합성 마스터 믹스를 추가합니다. 튜브를 짧게 튕기고 탁상용 원심분리기로 2000 x g 의 RT에서 5초 동안 회전시킨 후 얼음 위에서 샘플을 교체합니다.
- 샘플을 thermocycler에 넣고 25°C에서 10분 동안 프라이머를 어닐링하고, 55°C에서 30분 동안 배양하여 역전사를 수행하고, 마지막으로 85°C에서 5분 동안 효소를 열 비활성화합니다.
- 배양 후 즉시 튜브를 얼음 위에 놓고 21μL의 무균 뉴클레아제 물을 첨가하여 샘플을 총 부피 25μL로 희석합니다. 저복제 수 전사체의 경우, 경험적으로 결정된 대로 이 희석 단계를 변경합니다.
6. qPCR
- 더 많은 양의 완보동물로부터 제조된 총 RNA를 사용하여 프라이머 세트의 어닐링 온도를 결정하고, 예를 들어, Boothby, 201843에 제시된 벌크 추출 방법을 결정합니다.
- qRT-PCR을 실행하기 전에 PCR 온도 구배를 실행하여 최적의 어닐링 온도를 결정합니다(이 프로토콜에 사용된 모든 PCR 설정은 표 1 및 표 2 참조).
- 지표 염료 슈퍼 믹스 튜브 하나를 얼음에 넣고 빛으로부터 분리하십시오. 96웰 qPCR 플레이트를 얼음 위에 놓고 5μL의 슈퍼 믹스, 2μL의 물, 각 프라이머(10μM) 1μL 및 cDNA 생성물 1μL를 원하는 웰 수에 놓습니다.
- PCR 플레이트를 플레이트 씰로 밀봉하고 프라이머 세트에 적합한 어닐링 온도를 사용하여 qRT-PCR을 실행합니다(이 백서에 사용된 모든 qRT-PCR 설정은 표 3 참조).
7. 정량화 및 결과 해석
- 결과를 하나 이상의 control housekeeping 유전자와 정량적으로 비교하며, 그 발현은 부과된 조건에 대해 일정할 것으로 예상됩니다. 이 연구를 위해 액틴 유전자를 사용했습니다.
- 각 웰에 대한 Ct-값 또는 사이클 임계값을 구하고 대조군 하우스키핑 유전자 반응의 Ct 값과 비교합니다. 다음 방정식을 사용하여 유전자 발현의 접힘 변화를 계산합니다.
참고: fold gene expression은 각 transcript 및 완보동물에 대해 2-(ΔΔCt)48로 플롯됩니다. - Ct-값에서 전사 번호의 대략적인 추정치를 얻으려면 다음 방정식을 사용하십시오.
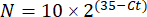
여기서 N은 전사체의 수이고 2는 가정된 PCR 효율 또는 PCR48의 사이클당 형광 배증가입니다.
결과
single-tardigrade RNA 추출의 개발 및 최적화
완보동물에서 RNA 추출을 위해 Ly et al., 201542 의 프로토콜을 채택한 STST 시스템은 제제의 양과 품질을 극대화하도록 최적화되었습니다(그림 1A). RT-PCR은 액틴 전사체에 대해 수행되었으며, 엑손 1 및 2에 걸쳐 527bp 영역을 증폭하여 전사체 수율을 정량화했습니다(이러한 프라이머?...
토론
이 연구는 단일 완보 qRT-PCR에 대한 RNA를 추출하는 효율적인 방법을 제시합니다. STST 방법론을 기존 단일 완보동물 RNA 추출 키트와 직접 비교한 결과, STST RNA 추출은 >200배 더 많은 양의 액틴 RNA 전사체를 산출하고, 시료당 비용을 1달러 미만으로 줄이며, 추출에 필요한 시간을 30% 단축하는 것으로 나타났습니다. 관련 생물학적 질문에 STST를 적용하기 위해 단기 열-충격 반응 ...
공개
저자는 공개할 이해 상충이 없음을 선언합니다.
감사의 말
우리는 NIH Ruth Kirschstein Fellowship # 5F32AG081056-02와 Molly J. Kirk 박사를 지원한 Errett Fisher Post-Doctoral Fellowship, Chaoming Xu를 지원한 Crowe Family Fellowship, 이러한 연구 노력을 지원한 University of California, Santa Barbara Academic Senate Grant 및 NIH 보조금 #R01GM143771 및 #2R01HD081266에 감사드립니다. 저자는 또한 캘리포니아 대학, 산타 바바라 및 캘리포니아 대학, 대통령 사무실의 지원을 받는 캘리포니아 NanoSystems 학회 내의 생물학 Nanostructures 실험실의 사용을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
참고문헌
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유