Method Article
효모 표면 디스플레이에 의한 단백질 공학
요약
이 프로토콜은 관심 항원에 결합하는 단백질 변이체를 풍부하게 하기 위해 효모 표면 디스플레이 선택 캠페인을 수행하기 위한 필수 단계를 설명합니다.
초록
단백질 공학은 주어진 단백질의 기존 기능을 개선하거나 새로운 기능을 생성할 수 있도록 합니다. 단백질 공학 분야에서 가장 널리 사용되는 다재다능한 도구 중 하나는 효모 표면 디스플레이(yeast surface display)로, 무작위 단백질 풀이 효모 표면에서 발현됩니다. 표현형(예: 효모에 표시된 단백질과 관심 항원의 결합)과 유전자형(단백질 변이체에 대한 플라스미드 암호화)의 연결을 통해 원하는 특성과 농축된 변이체의 후속 염기서열분석을 위해 이 라이브러리를 선택할 수 있습니다. magnetic bead selection과 flow cytometric sorting을 결합하면 표적 항원에 대한 결합이 강화된 단백질 변이체를 선택하고 농축할 수 있습니다. 특히, 친화성 성숙(affinity maturation)에 더하여, 표적에 대한 결합은 어떠한 초기 결합 친화성(initial binding affinity)도 없이 달성될 수 있다. 여기에서는 효모 표면 디스플레이 선택 캠페인의 모든 필수 부분을 다루고 일반적인 효모 표면 디스플레이 결과의 예를 제공하는 단계별 프로토콜을 제공합니다. 당사는 효모 표면 디스플레이가 유세포 분석에 액세스할 수 있는 모든 분자 생물학 실험실에서 확립할 수 있는 광범위하게 적용 가능하고 강력한 방법임을 보여줍니다.
서문
효모 표면 디스플레이는 단백질 공학 분야의 핵심 기술 중 하나입니다. 이를 통해 향상된 친화력 또는 안정성과 같은 원하는 특성을 가진 단백질 변형체를 선택할 수 있습니다. 19971 년에 처음 도입 된이 기술은 파지 디스플레이 2,3, 리보솜 디스플레이4 및 포유류 세포 디스플레이 5,6,7 외에 가장 일반적으로 사용되는 디스플레이 기술 중 하나입니다. 관심 단백질(POI)은 단백질을 고정하기 위해 융합하여 효모 세포 표면에 표시됩니다. 다양한 앵커 단백질을 사용할 수 있으며, 가장 일반적으로 POI는 효모 응집소 짝짓기 단백질 Aga2p 1,8의 C-말단에 융합됩니다. 또한 POI는 일반적으로 헤마글루티닌 태그(HA-tag) 및 c-myc 태그와 같은 두 개의 태그로 둘러싸여 있으며, 이를 통해 형광 표지된 항체 및 유세포 분석을 사용하여 디스플레이 수준을 검출할 수 있습니다(그림 1A). 일반적인 효모 선별 캠페인에는 magnetic bead selection과 flow cytometric sorting의 조합이 포함됩니다. 비드 선택은 큰 세포 수를 처리하고 표적 항원에 결합하는 단백질 변이체의 농축을 가능하게 하는데, 이는 항원이 로드된 비드와의 다가 상호 작용이 열성 효과를 유발하여 친화성이 낮은 변이체의 손실을 방지하기 때문입니다(그림 1B). 유세포 분석 및 선택은 표시된 POI 변이체와 표지된 항원의 결합을 시각화할 수 있는 이점을 제공합니다. 결과적으로, 결합 집단을 분류하고 배양할 수 있으며, 이는 여러 선별 라운드를 통해 원하는 특성을 가진 단백질 변이체를 농축할 수 있습니다. 또한, 무작위 돌연변이 유발의 추가 라운드를 수행하여 다양성을 더욱 증가시킬 수 있으며, 따라서 단백질의 친화성 및/또는 안정성에 기여하는 추가 돌연변이를 찾을 가능성을 높일 수 있습니다.
효모 표면 디스플레이는 (a) 진핵생물 발현 기계, 산화 단백질 접힘 및 진핵생물 번역 후 변형(예: N-글리코실화), (b) 단백질 옆에 있는 두 개의 펩타이드 태그 검출로 인한 발현 정규화, (c) 유세포 분석을 통한 선택 진행 상황의 육안 검사(예: 결합 세포의 비율 및 결합 강도) 및 (d) 개별 단백질 돌연변이를 분석할 수 있는 가능성과 같은 특정 이점을 제공합니다. 효모(예: 열안정성 및 친화도 분석)는 힘든 단백질 발현 및 정제에 대한 시간 절약형 대안을 제시합니다9. 실제로, 효모 표면에 표시된 단백질의 친화도(KD값)와 안정성(T50 값)은 모두 생물물리학적 방법 및 용해성 단백질을 사용하여 얻은 데이터와 좋은 상관관계를 보여주었습니다 9,10,11,12. 효모 표면 표시는 다양한 단백질, 예를 들어 항체 단편 13,14,15,16, 10번째 유형 III 피브로넥틴 도메인 17,18, rcSso7d19,20 또는 매듭21의 엔지니어링에 사용되었습니다. 유사하게, 아미노산 코돈 사용뿐만 아니라 무작위 위치를 변경하여 효모 라이브러리 설계를 최적화하기 위한 광범위한 연구가 수행되었습니다 17,22,23. 효모 표면 디스플레이는 안정성 14,15,24,25, 친화도18,26,27, 효소 활성 28,29,30,31 및 단백질 발현32의 엔지니어링에 대해 성공적인 것으로 입증되었습니다. 또한, 작은 분자의 존재 또는 부재 시 조건부 결합과 같은 보다 정교한 응용은 효모 표면 디스플레이20을 사용하여 수행되었습니다.
이 프로토콜에서는 소분자 A112020의 존재 하에서 항원 인간 레티놀 결합 단백질 4(hRBP4)에 대해 선택된 G4 라이브러리(10번째 유형 III 피브로넥틴 도메인, Fn3 기반)의 예와 함께 효모 표면 표시를 사용한 선택 캠페인의 모든 필수 단계를 설명합니다. 이 선택은 분자 스위치로 사용될 수 있는 작은 분자에 의존하는 단백질-단백질 상호 작용을 생성하기 위해 수행되었습니다. 참고로, 효모 표면 표시를 통한 대체 접근법이 가능하지만, 일반적인 효모 선택은 일반적으로 이전의 결합 친화성 없이 표적 항원에 결합하는 것을 목표로 합니다. 당사는 효모 라이브러리 배양, 비드 선택, 유세포 분석 분류 및 오류가 발생하기 쉬운 PCR(epPCR)에 의한 친화성 성숙을 포함하는 효모 선별 캠페인의 모든 단계를 다룹니다. 따라서 이 프로토콜은 이전 효모 표면 표시 프로토콜33,34를 보완하며 주어진 효모 라이브러리 및 선택한 표적 항원과 함께 효모 표면 표시 선택(그림 1)의 기초로 사용할 수 있습니다.
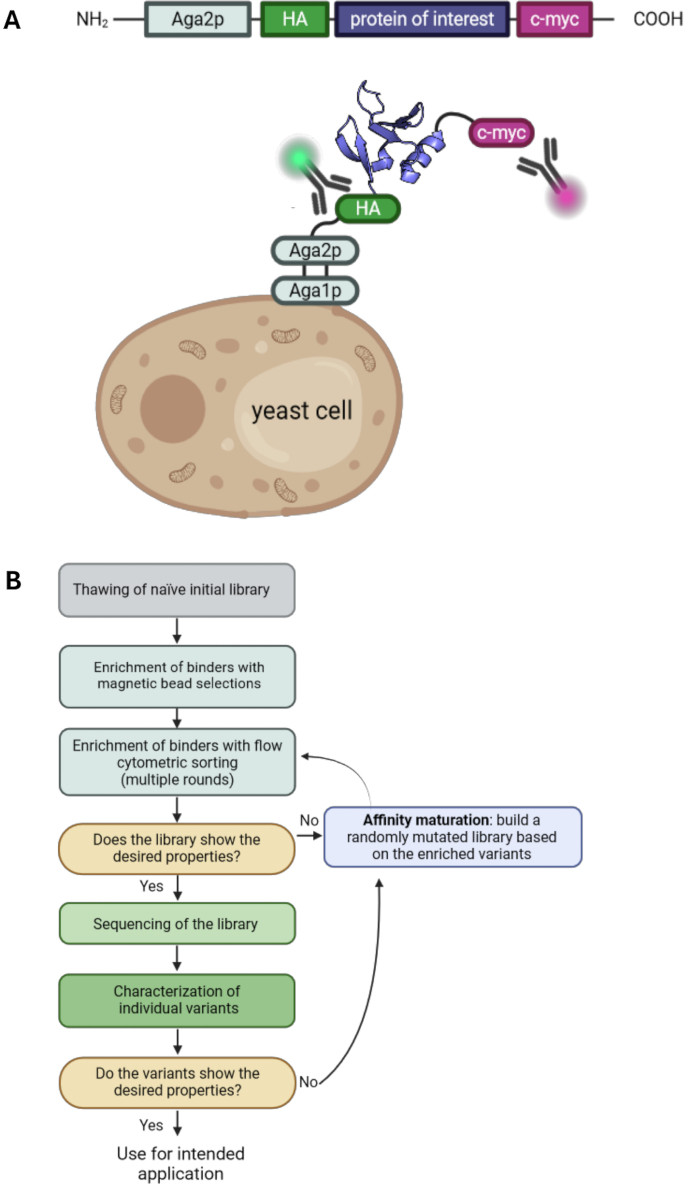
그림 1: 효모 표면 표시의 원리 및 효모 표면 표시 선택을 위한 일반적인 작업 흐름. (A) POI는 효모 표면 디스플레이 벡터로 복제되며 일반적으로 N-말단 HA- 및 C-말단 c-myc-tag가 측면에 있습니다. 이 구조체는 효모 짝짓기 단백질 Aga2p에 융합되어 표면에 표시됩니다. 묘사된 단백질은 PDB ID: 6QBA20의 엔지니어링된 바인더 "RS3"입니다. (B) 효모 표면 디스플레이 선택 캠페인의 일반적인 워크플로우를 보여주는 순서도로, 비드 선택 및 유세포 분석 분류를 통해 원하는 특성을 가진 단백질 변이체의 농축과 친화성 성숙을 위한 epPCR을 결합합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
프로토콜
1. 효모 라이브러리의 해동 및 배양
- RT에서 냉동 효모 라이브러리의 충분한 부분 표본을 해동하고 SD-CAA( 표 1 참조)에서 즉시 약 1의 OD600 (1 x 107 cells/mL에 해당)으로 희석합니다.
참고: 라이브러리의 다양성을 최소 10배 커버해야 합니다. 따라서 라이브러리 크기가 2.5 x 108 클론을 차지하는 경우(이 예에서 사용된 G4 라이브러리의 다양성임) 250mL의 SD-CAA에 최소 2.5 x 109 세포를 접종하여 OD600 of 1에 도달합니다. 이 프로토콜은 액체 배양(라이브러리로 갓 형질전환된 효모 세포)에서도 시작할 수 있습니다. - 생존 가능한 세포의 수를 평가합니다.
- 이 효모 현탁액(OD600 of 1)에서 100μL 분취액을 취하고 마이크로 원심분리 튜브에서 900μL의 SD-CAA 배지를 사용하여 1:10 희석 시리즈를 수행합니다.
- SD-CAA 플레이트에서 최대 10-5 의 희석액과 100μL의 희석액 10-3, 10-4 및 10-5 플레이트를 준비합니다(표 1). 플레이트를 30 ° C에서 2-3 일 동안 배양합니다. 해동 후 생존 가능한 세포의 수가 라이브러리 다양성을 10배 이상 초과하는지 확인합니다.
- 한편, 잔여 세포 현탁액을 계속 진행하고 유리플라스크에 G4 라이브러리를 함유한 효모 배양액을 30°C에서 진탕(180rpm)하면서 하룻밤 동안 배양합니다. 하룻밤 배양 후 섹션 2(효모 표면에서 단백질 발현 유도)를 계속합니다.
- 2-3일의 배양 후 육안 검사로 SD-CAA 플레이트의 콜로니를 계산합니다. 해동 후 생존 가능한 세포의 최종 수를 계산할 때 희석 계수와 도금된 세포(즉, 100μL)와 원래 배양 부피(즉, 250mL)의 비율을 고려하십시오.
Viable cells/mL = 플레이트의 콜로니 수 × 희석 계수
생존 세포(총) = (생존 세포/mL) × 배양 부피
| 중간/버퍼 | 구성 요소 | 농도 [g/L] | 코멘트/설명 | |||
| SD-CAA | D-포도당 | 20 | 모든 매체 성분을 1000mL ddH2O에 용해시키고 일회용 0.22μm 멸균 필터로 멸균 여과액을 용해합니다. | |||
| 효모 질소 염기 | 6.7 | |||||
| 카스미노산 | 5 | |||||
| 구연산 일수화물 | 7.4 | |||||
| Tri-sodium citrate 이수화물 | 10.83 | |||||
| SG-CAA | D-갈락토오스 | 20 | 모든 매체 성분을 1000mL ddH2O에 용해시키고 일회용 0.22μm 멸균 필터로 멸균 여과액을 용해합니다. | |||
| D-포도당 | 2 | |||||
| 효모 질소 염기 | 6.7 | |||||
| 카사미노산 | 5 | |||||
| 디소듐 수소 인산염 헵타하이드레이트 | 10.2 | |||||
| 나트륨 이수소 인산염 일 수화물 | 8.56 | |||||
| SD-CAA 플레이트 | 소르비톨 | 182 | 소르비톨, 인산이수소나트륨 헵타하이드레이트, 인산이수소나트륨 일수화물 및 한천을 900mL ddH2O 및 오토클레이브에 용해시킵니다. 나머지 성분을 100mLddH2O에 용해 및 멸균 여과하고 오토클레이브 매체가 미지근할 때 첨가합니다. | |||
| 디소듐 수소 인산염 헵타하이드레이트 | 10.2 | |||||
| 나트륨 이수소 인산염 일 수화물 | 7.44 | |||||
| 한천 | 15 | |||||
| D-포도당 | 20 | |||||
| 효모 질소 염기 | 6.7 | |||||
| 카사미노산 | 5 | |||||
| YPD | 펩톤 | 20 | 일회용 0.22μm 멸균 필터로 10x D-포도당 스톡(200g/L)과 멸균 여과액을 준비합니다. 펩톤과 효모 추출물을 900mL ddH2O에 용해시키고 오토클레이브를 사용합니다. 미지근하면 100mL 10x D-포도당을 첨가합니다. | |||
| 효모 추출물 | 10 | |||||
| D-포도당 | 20 | |||||
| YPD 플레이트 | 펩톤 | 20 | 일회용 0.22μm 멸균 필터로 10x D-포도당 스톡(200g/L)과 멸균 여과액을 준비합니다. 펩톤, 효모 추출물 및 한천을 900mL ddH2O 및 오토클레이브에 용해시킵니다. 미지근하면 100mL 10x D-포도당을 첨가합니다. | |||
| 효모 추출물 | 10 | |||||
| D-포도당 | 20 | |||||
| 한천 | 15 | |||||
| PBSA (증권 시세 표시기) | 증권 시세 표시기 | 1 | PBS에 BSA를 용해시키고 일회용 0.22μm 멸균 필터로 멸균 여과액을 제거합니다. | |||
표 1: 매체 및 버퍼 조성.
2. 효모 표면에서 단백질 발현 유도
- 하룻밤 배양 후 효모 배양은 일반적으로 2-20 사이의 OD600 에 도달합니다. 광도계로 OD600 을 측정하여 효모 배양의 밀도를 측정합니다.
참고: OD 측정은 0.1-1 범위에서만 선형입니다. 따라서 SD-CAA에서 희석(일반적으로 1:20 및 1:50)을 수행하여 OD600을 측정합니다.- SD-CAA의 세포를 OD600 of 1로 희석하고 다양성이 10배로 덮이도록 합니다(즉, G4 라이브러리의 희석을 위해 2.5 x 109 세포가 사용됨). 세포가 기하급수적 성장 단계에 도달할 수 있도록 흔들면서 30°C에서 배양을 계속합니다.
- 하룻밤 배양에서 효모 라이브러리(15% 글리세롤을 함유한 SD-CAA)의 글리세롤 스톡(다양성 50x 포함)을 준비합니다. 이를 위해 필요한 수의 효모 세포(2000 x g, 3분, 4°C)를 원심분리하고, 상등액을 버리고, 15% 글리세롤이 보충된 1mL의 SD-CAA 배지에 세포 펠렛을 재현탁합니다. 글리세롤 스톡을 -80 °C에서 즉시 동결하십시오.
참고: 효모 배양액이 동결 전에 고정상으로 성장하면 효모 세포의 생존력이 증가합니다. 이후 선별 라운드에서 라이브러리의 다양성이 더 낮을 경우 일반적으로 700μL의 효모 배양액을 사용하고 50% 글리세롤이 보충된 300μL의 SD-CAA와 혼합하는 것으로 충분합니다. 여기서, 효모 배양은 일반적으로 고정상에서 OD600 값 15를 나타내지만 실제 값은 사용 중인 광도계에 따라 달라질 수 있습니다. 따라서 고정상은 반복된 OD600 측정에 의해 결정될 수 있습니다. OD600 값이 2시간의 배양 기간 동안 증가하지 않으면 고정상에 도달합니다.
- 효모 배양 희석 후 5분, 4-6시간 동안 2000 x g 에서 다양성을 커버할 수 있는 적절한 부피의 세포를 원심분리하고(일반적인 OD600 값은 4-6 사이임) 상등액을 폐기합니다.
- 세포 펠릿을 SG-CAA( 표 1 참조)에 재현탁시켜 OD600 of 1을 생성하고 새 유리 플라스크로 옮깁니다.
참고: 갈락토오스 함유 배지에서의 재현탁은 갈락토오스 유도 프로모터에 의해 표면에서 단백질의 발현을 유도합니다. - 180rpm으로 진탕하면서 20°C에서 밤새 효모 배양을 배양하여 POI의 표면 발현을 유도합니다. 하룻밤 유도 후 효모 배양은 일반적으로 2-5 사이의 OD600 값에 도달합니다. 유도 온도를 37°C로 올리면 OD600 값이 4-10일 것으로 예상됩니다.
참고: 온도를 20°C로 낮추면 세포 성장이 느려지고 일반적으로 더 나은 디스플레이 수준을 얻을 수 있습니다. 그러나, 37 °C에서의 유도는 안정화된 돌연변이체35에 대한 선택 압력을 증가시키기 위해 유용할 수 있다.
3. 효모 라이브러리의 첫 번째 bead selection round (positive selection)
참고: 표준 비드 선택 절차에는 6단계가 포함됩니다(표 2).
| 하루 | 걸음 | |
| 0 | 하룻밤 문화 | |
| 1 | 효모 세포 표면의 단백질 발현 유도 | |
| 2 | 1개의 positive selection이 있는 첫 번째 bead selection | |
| 3 | beads의 제거, passaging, 효모 세포 표면의 단백질 발현 유도 및 라이브러리의 동결 | |
| 4 | 3개의 음성 선택과 1개의 양성 선택을 가진 두 번째 비드 선택 | |
| 5 | 구슬 제거 및 라이브러리 동결 | |
표 2: 효모 라이브러리의 bead selection 전도를 위한 일반적인 타임라인.
- 구슬의 준비
- 첫 번째 비드 선택을 위한 비드를 준비합니다( 재료 표 참조): 각 튜브에 대해 10μL의 비오틴 바인더 마그네틱 비드(4 × 105 beads/μL)를 990μL의 PBSA(표 1)에 재현탁탁하고 세척을 위해 튜브를 마그네틱 랙에 2분 동안 놓고 뚜껑을 연 상태에서 튜브를 마그네틱 랙에 2분 동안 놓습니다. 상층액을 조심스럽게 제거하십시오. 1mL PBSA로 세척 단계를 반복합니다.
참고: 동일한 비오틴화 항원을 가진 여러 개의 비드 튜브가 필요한 경우, 이 단계에서 하나의 튜브에서 준비하여 균일한 항원 비드 용액을 생성할 수 있습니다. 이 경우 다음 단계에서 그에 따라 항원의 양을 늘려야 합니다. - 1.5mL 마이크로 원심분리 튜브에 6.7-33pmoles의 비오틴화 항원(여기서: 0.125mg/mL 농도의 hRBP4 6.86μL)과 함께 총 1mL의 PBSA에 비드를 재현탁합니다.
참고: 6.7pmoles의 항원으로 충분하지만 항원이 제한되지 않는 경우 33pmoles를 사용해야 합니다. 이 경우와 같이 저분자 약물 또는 리간드에 결합된 항원에 대해 선택을 수행하는 경우, 이 약물/리간드는 선택 중에 존재해야 하며 이 단계에서 추가해야 합니다(여기서는 5μM A1120). - 4 °C에서 2 시간 동안 부드럽게 흔들거나 회전하여 배양하십시오.
참고: 이 단계에서 셀을 준비할 수 있습니다. - 배양 후 세포에 추가하기 전에 뚜껑을 연 상태에서 튜브를 마그네틱 랙에 2분 동안 놓습니다. 상층액을 제거하고 위에서 설명한 대로 항원이 로드된 비드를 1mL의 PBSA로 세척합니다. 세탁 단계를 반복합니다.
참고: 약물 또는 리간드에 결합된 항원을 선택한 경우 세척 버퍼에는 이 약물 또는 리간드가 과도하게 포함되어야 합니다(예: 5μM A1120). - 항원이 로드된 비드를 50μL의 PBSA에 재현탁시킵니다(해당되는 경우 약물 또는 리간드가 있는 경우).
참고: 여러 튜브에 대해 동일한 항원 로딩 비드를 준비한 경우 그에 따라 부피를 조정하십시오(G4 라이브러리의 경우 여기서 최종 부피는 두 튜브에 대한 100μL 수율 비드입니다).
- 첫 번째 비드 선택을 위한 비드를 준비합니다( 재료 표 참조): 각 튜브에 대해 10μL의 비오틴 바인더 마그네틱 비드(4 × 105 beads/μL)를 990μL의 PBSA(표 1)에 재현탁탁하고 세척을 위해 튜브를 마그네틱 랙에 2분 동안 놓고 뚜껑을 연 상태에서 튜브를 마그네틱 랙에 2분 동안 놓습니다. 상층액을 조심스럽게 제거하십시오. 1mL PBSA로 세척 단계를 반복합니다.
- 효모 세포의 준비
- 상기와 같이 유도 효모 배양액의 OD600 을 측정합니다.
- 라이브러리의 다양성의 10배(2000 x g, 10분, 4°C)를 커버하기에 충분한 셀을 펠릿화합니다. G4 라이브러리의 경우 원심분리를 위해 2.5 x 109 셀을 사용합니다. 상층액을 제거하고 PBSA 10mL로 세포를 세척합니다.
- 다시 원심분리기(2000 x g, 10분, 4°C)를 하고 적절한 부피의 PBSA(여기서는 2mL)에서 1.25 x 109 cells/mL의 농도로 세포를 재현탁하고 마이크로 원심분리 튜브로 옮깁니다.
참고: PBSA의 부피는 세포 수에 따라 다릅니다. - 원심분리기(2000 x g, 3분, 4°C)를 측정하고 950μL의 PBSA에 재현탁합니다.
- 항원이 로드된 비드를 사용한 효모 세포의 배양
- 950μL의 세포 현탁액에 50μL의 항원 로드 비드를 추가하고 진탕 또는 회전하면서 4°C에서 2시간 동안 배양합니다.
- 배양 후, 뚜껑을 연 상태로 세포-항원-비드 용액을 마그네틱 랙에 놓습니다. 뚜껑에서 튜브까지 액체를 피펫팅하여 전체 효모 배양물을 자기 선택에 적용합니다. 바인딩되지 않은 셀을 조심스럽게 버리기 전에 2분 동안 기다립니다. 즉시 PBSA 1mL로 씻어내십시오.
참고: 약물/리간드(이 예에서는 A1120)가 있는 상태에서 선별을 수행하는 경우 PBSA에 존재해야 합니다. - PBSA를 사용한 세척 단계 후 SD-CAA 1mL에 세포를 빠르게 재현탁시켜 건조를 방지합니다.
- SD-CAA 200mL를 추가하고 위에서 설명한 대로 연속 희석을 수행합니다. SD-CAA 플레이트에 100μL를 플레이트화하고 30°C에서 2-3일 동안 배양하여 새로운 최대 다이버시티를 결정합니다. 여기서, 첫 번째 비드 선택 후 G4 라이브러리의 다양성은 1.25 x106 셀입니다.
참고: 원본 라이브러리는 이 선택 라운드에서 10배 오버 샘플링되었기 때문에 선택 후 대부분의 클론이 농축된 풀에 여러 번 존재할 것이라고 가정하는 것이 합리적입니다. 결과적으로 실제 다양성은 더 낮아지며 이 숫자는 최대값으로 간주되어야 합니다. 일반적으로 이 첫 번째 선택 라운드에서 다양성이 최소 100배 감소합니다. 남은 배양물(200mL)을 30°C에서 진탕(180rpm)과 함께 16시간 이상 배양합니다. 프로토콜은 세포를 4°C에 배치하여 야간 배양 후 일시 중지할 수 있습니다.
4. 구슬 제거 및 다음 구슬 선택 라운드 전에 배양
- 위에서 설명한 대로 OD600 을 측정합니다. 다이버시티를 최소 10배(2000 x g, 3분, RT)까지 커버할 수 있을 만큼 충분한 셀을 펠렛화합니다. 상층액을 버리고 SD-CAA 1mL에 세포를 재현탁합니다.
- 마이크로 원심분리기 튜브로 옮기고 뚜껑을 연 상태에서 마그네틱 랙에 2분 동안 놓습니다. 결합되지 않은 세포를 새로운 마이크로 원심분리기 튜브로 옮기고 분리 과정을 반복합니다.
- 결합되지 않은 세포를 100mL의 SD-CAA에 희석하고 3-4의 OD600 에 도달할 때까지 180rpm에서 진탕하면서 30°C에서 배양합니다.
참고: 비드 제거 전에 OD600 이 4 미만이면 배양 없이 바로 세포를 유도할 수 있습니다. - OD600 of 3-4에 도달하면 다양성을 10x (2000 x g, 3 min, RT) 이상 커버할 수 있는 충분한 수의 세포를 원심분리하고 단백질 발현 유도를 위해 SG-CAA에서 OD600 of 1로 재현탁하고 180rpm으로 밤새 진탕하면서 20°C에서 배양합니다.
- 나머지 SD-CAA 효모 배양액이 고정 성장 단계(OD600 이상 15)에 도달하면 다양성을 50배(2000 x g, 3분, RT)를 커버하기에 충분한 세포를 펠릿화합니다. 원심분리(2000 x g, 3분)로 세포를 동결한 다음 15% 글리세롤을 함유한 SD-CAA에 세포 펠렛을 재현탁하고 -80°C에서 보관합니다.
5. 3개의 부정 및 1개의 긍정 선택이 있는 두 번째 비드 선택 라운드
- 상기와 같이 세포와 항원 비드를 준비합니다. negative selection에 대해 (항원을 추가하지 않고) barare beads 용액을 준비합니다.
참고: 3개의 negative selection이 수행되기 때문에 3개의 라운드 모두에 대해 beads를 함께 준비할 수 있습니다. - 세척 후 항원 비드를 50μL의 PBSA에 재현탁하고 베어 비드를 150μL의 PBSA에 재현탁합니다(3가지 음성 선택 모두에 대해 준비된 경우).
- 첫 번째 음성 선택의 경우, PBSA에서 세척된 세포 950μL에 세척된 베어 비드 50μL를 추가하고 4°C에서 1.5시간 동안 배양합니다.
- 배양 후, 노출된 비드 셀 현탁액이 들어 있는 튜브를 뚜껑을 연 상태로 마그네틱 랙에 놓습니다. 뚜껑에 있는 액체를 튜브에 피펫으로 넣고 2분 동안 기다립니다. 결합되지 않은 세포를 새로운 마이크로 원심분리 튜브로 옮기고 세척된 베어 비드 50μL를 추가합니다.
- 이 절차를 반복하여 부정 선택을 두 번 더 수행합니다. 한편, positive selection을 위해 antigen loaded beads를 준비합니다.
- 3번의 음성 선택 라운드 후 50μL의 항원 로드 비드 용액을 세포에 추가하고 4°C에서 2시간 동안 배양합니다.
- 항원이 로드된 비드가 들어 있는 세포를 뚜껑이 열린 마그네틱 랙에 놓고 뚜껑에 있는 액체를 튜브에 피펫으로 넣습니다. 바인딩되지 않은 셀을 버리기 전에 2분 동안 기다립니다.
- 첫 번째 antigen-bead 선택에 대해 설명한 대로 나머지 모든 단계를 수행합니다. 첫 번째 positive selection(단계 3.3.2)과의 유일한 차이점은 세척 단계의 수입니다: PBSA로 한 번 세척하는 대신 PBSA로 bead-cell suspension을 두 번 세척하여 선택의 엄격성을 높입니다.
참고: 일반적으로 다양성은 다시 10배-100배씩 감소합니다. 이 두 번째 bead selection 후 G4 라이브러리의 최대 다양성은 3.45 x 105 cells였습니다. 위에서 언급했듯이 실제 다양성은 도서관의 과다 표본추출로 인해 더 낮을 것으로 예상됩니다.
6. 유세포 분류를 통한 라이브러리 선택
- SG-CAA에서 표면 발현을 밤새 유도한 후 위에서 설명한 대로 OD600 을 측정합니다. 10배의 다양성(2000 x g, 5분, RT)을 커버하기에 충분한 셀을 펠렛화합니다. 상등액을 버리십시오.
- 펠릿을 PBSA에 재현탁시키고 마이크로 원심분리기 튜브로 옮깁니다. 각 튜브의 염색을 위해 3 x 107 셀을 사용합니다. 다양성에 따라 필요한 만큼 튜브를 준비하십시오. 항원 없이 염색할 수 있도록 대조관 1개를 준비합니다.
참고: 예를 들어, 30 x 106 셀을 정렬해야 하는 경우 300 x 106 셀을 펠릿화하여 눈에 보이는 세포 펠릿을 얻고 후속 단계에서 세포 손실을 방지합니다. PBSA 5mL에 재현탁하고 각각 500μL의 부분 표본 3-4개를 마이크로 원심분리 튜브로 옮깁니다. 염색 과정에서 세포의 일부가 손실되기 때문에 분류해야 하는 수보다 약 3배 더 많은 세포를 준비하십시오. 약물/리간드가 있는 상태에서 선택을 수행하는 경우, 약물/리간드 없이 항원만 사용하여 추가 대조군을 준비합니다. - 다시 원심분리기(2000 x g, 5분, RT)를 하고 항원이 포함된 PBSA 200μL(hRBP4와 5μM A1120)에 펠릿을 재현탁하고 4°C에서 1시간 동안 배양합니다.
알림: 이 시점부터 모든 단계는 얼음과 차가운 시약으로 수행해야 합니다. 항체 또는 항원 고갈을 방지하기 위해 모든 시약은 튜브에서 효모에 표시된 단백질의 총 수와 비교하여 화학량론적 초과량으로 첨가되어야 합니다. 그렇지 않으면 항원 및/또는 항체의 제한된 가용성으로 인해 효모 표시 변이체의 결합을 제한할 위험이 있을 수 있습니다. 일반적으로 효모 세포당 5 x 104 분자가 계산에 사용되며 항원 및 항체를 최소 10배 이상 추가하는 것이 좋습니다. 항원이 매우 제한적이면 초과분을 5배로 줄일 수 있습니다. 리간드 고갈을 피하기 위한 계산의 예를 포함한 보다 자세한 설명은 이전 프로토콜9에 제공되어 있습니다. - 배양 후 세포를 원심분리하고(2000 x g, 5분, 4°C) PBSA 1mL로 세척합니다(선택 중에 약물/리간드가 존재하는 경우(이 경우 A1120과 같이 세척 버퍼에 있어야 함).
- 결합된 항원의 염색 및 검출을 위한 항체가 포함된 100μL의 저온 PBSA에 세포를 재현탁하고 4°C에서 30분 동안 배양합니다.
- anti-c-myc 항체(최종 희석 1:100)를 사용하여 표시된 전체 길이 POI를 검출합니다. anti-HA antibody(최종 희석 1:50)를 사용하여 표시된 단백질의 총량을 검출합니다. Penta-His 항체(최종 희석 1:20) 또는 스트렙타비딘(최종 희석 1:200)을 사용하여 각각 6x HIS 태그 또는 비오틴을 포함하는 용해 항원에 결합하는 효모 세포를 선택합니다.
- 검출 시약을 교대로 선별 라운드와 2차 시약에 결합하는 단백질 변이체의 농축을 방지합니다.
참고: 다양한 형광단 조합을 사용할 수 있지만 Alexa Fluor 488 및 Alexa Fluor 647은 유세포 분석에서 보상이 필요하지 않기 때문에 좋은 옵션입니다. 항체 고갈을 방지하기 위해, 튜브에 있는 효모에 표시된 단백질의 총 수와 비교하여 화학량론적 초과량으로 추가해야 합니다. 일반적으로 계산은 효모 세포당 5 x 104 분자를 기반으로 합니다. 보다 상세한 설명은 이전 프로토콜9에 제공되어 있다.
- 배양 후 세포를 원심분리하고(2000 x g, 5분, 4°C) PBSA 1mL(hRBP4 선택 G4 라이브러리의 경우 A1120 5μM 약물/리간드 포함)를 펠릿 및 원심분리에 다시 첨가합니다. 대부분의 상등액을 제거하고 펠릿이 건조되는 것을 방지하기 위해 20-30 μL만 유지하십시오.
- 원하는 특성(즉, A1120이 있는 상태에서 hRBP4에 결합)을 가진 Fn3 분자를 표시하는 효모 세포를 분류 및 분류하기 직전에만 펠릿을 차가운 PBSA에 재현탁합니다.
참고: 게이팅 전략은 그림 2A에 나와 있습니다. - 세포를 SD-CAA 배지로 직접 분류하고, 분류 후 SD-CAA 배지를 더 첨가하고, 180rpm에서 진탕하면서 30°C에서 배양합니다.
참고: 분류 후 부피가 크게 증가하는 경우 원심분리 단계(2000 x g, 5분, RT)를 수행하여 과도한 sheath fluid를 제거하고 세포를 새로운 SD-CAA 배지에 재현탁해야 합니다. 새 튜브에 분류된 세포의 수를 기록하면 다음 분류 라운드를 위한 새로운 최대 다양성이 됩니다. 위에서 언급했듯이 도서관의 실제 다양성은 도서관의 과다 샘플링으로 인해 더 낮아질 것입니다. - 유세포 분석을 여러 차례 반복하여 효모 라이브러리에서 결합 단백질 변이체가 풍부해지는 것을 확인합니다(그림 2B).
참고: 분석 유세포 분석 실험에서 라이브러리를 나란히 비교하는 것이 유용할 수 있습니다. 이후 선별 라운드에서 항원의 농도를 낮추는 것은 낮은 친화성 결합제와 높은 친화성 결합체를 구별하고 낮은 농도에서 표적에 더 높은 결합 신호를 보이는 단백질 변이체를 특별히 분류하는 데 도움이 될 수 있습니다(그림 2C). 이후 선택 라운드에서는 향상된 열 안정성을 위해 분류할 수도 있습니다. 이를 위해 섹션 7에 설명된 대로 epPCR에 의한 라이브러리의 무작위 돌연변이 유발을 수행했습니다. 다른 유세포 분석 분류 라운드와 유사한 세포를 유도, OD600 측정 및 세포 세척을 통해 준비합니다. PBSA의 효모 세포를 얼음 위에 10분 동안 놓은 다음 고온에서 열 충격을 위해 열 사이클러에서 10분 동안 배양합니다. 그 후 다시 얼음 위에 10분 동안 놓고 염색 프로토콜을 진행합니다. 안정화된 변형체는 열 변성에 저항하므로 열 배양 후에도 유지된 결합을 보여줍니다. 야생형 단백질의 초기 열 안정성에 따라 열 충격에 대해 다양한 온도를 테스트할 수 있습니다. 일반적으로 40-80 °C 범위의 온도가 적합합니다. 그러나 48°C 이상에서는 효모 세포의 생존력이 급격히 감소합니다. 따라서 7.1단계에서 논의한 바와 같이 열 충격을 받은 효모 라이브러리를 분류한 후 플라스미드를 추출해야 합니다. 이 경우, EBY100 세포는 다음 분류36라운드로 진행하기 전에 농축 변이체로 새롭게 형질전환되어야 합니다. 열 안정성을 높이기 위한 보다 자세한 프로토콜은 다른 곳에서 찾을 수 있습니다37. 표시된 단백질의 변성이 가역적인 경우 이 방법은 작동하지 않습니다. 또는 분류 전날 20°C가 아닌 37°C에서 단백질 발현을 유도하고 여전히 효율적인 전체 길이 표시를 보이는 단백질 변형체를 분류합니다.
7. 무작위 돌연변이를 도입하기 위한 epPCR을 사용한 친화력 성숙
참고: epPCR을 사용한 친화도 성숙은 첫 번째 유세포 분석 분류 라운드 전에 또는 유세포 분석 분류 라운드 사이에 수행할 수 있습니다. A1120이 있는 상태에서 hRBP4가 있는 G4 라이브러리를 선택하기 위해 1차 유세포 분류 전에 친화성 성숙을 수행했습니다. 이는 또한 bead selection 후 library 크기와 유세포 분석으로 검출할 수 있는 결합 신호에 따라 달라집니다. 특히, bead selection 후의 친화도가 유세포 분석 실험에서 신호를 얻기에 충분하지 않은 경우(세척 단계에서 항원이 빠르게 해리되기 때문에) epPCR은 유세포 분석을 통해 나중에 검출 및 선택할 수 있는 개선된 변형체를 생성할 수 있습니다.
- 다음과 같은 수정 사항과 함께 상용 plasmid miniprep II 키트( 재료 표 참조)를 사용하여 농축 효모 라이브러리에서 효모 miniprep DNA 추출을 수행합니다.
- 하룻밤 배양의 세포 밀도를 측정하고 아침에 0.2의 OD600 으로 희석한 후 30°C에서 진탕하여 배양합니다.
- OD600 이 0.6에 도달하면 600 x g 에서 2분 동안 세포를 원심분리하여 3mL의 배양물을 수확합니다.
- 용액 1 200μL를 펠릿에 추가합니다. 6μL의 자이몰리아제를 추가합니다. 플릭 또는 가벼운 볼텍싱으로 펠릿을 재현탁시키고 37°C에서 3시간 동안 배양합니다.
- 용액 2 200μL를 넣고 부드럽게 섞습니다. 용액 3 400μL를 넣고 부드럽게 섞습니다.
- 3분 동안 최대 속도로 원심분리기를 사용합니다.
- 상층액을 spin-I 컬럼으로 옮기고 30초 동안 최대 속도로 원심분리한 다음 플로우스루를 폐기합니다.
- 에탄올과 원심분리기가 포함된 550μL의 세척 버퍼로 최대 속도로 2분 동안 세척합니다.
- 컬럼을 깨끗한 마이크로 원심분리기 튜브로 옮기고 최대 속도로 2분 동안 원심분리하여 잔여 세척 버퍼를 제거합니다.
- 컬럼을 새로운 마이크로 원심분리 튜브로 옮기고, 뉴클레아제가 없는 물 10μL를 첨가한 다음, 최대 속도로 1분 동안 회전하여 DNA를 용리시킵니다.
- 초기 PCR을 수행하여 epPCR용 템플릿을 증폭합니다.
- 효모 표면 디스플레이 벡터의 인서트 외부에 어닐링된 프라이머를 사용하여 PCR로 DNA를 증폭합니다.
참고: 효모 miniprep 중에 관심 플라스미드와 함께 많은 게놈 효모 DNA가 용출되므로 이 단계는 epPCR의 템플릿으로 사용할 깨끗한 DNA 단편을 얻는 데 권장됩니다. - pCTCON2의 경우 프라이머 fwd 5'- GGCTCTGGTGGAGGCGGTAGCGGAGGCGGG
GGTCGGCTAGC 및 rev 5'- CTATTACAAGTCC
표 3에 기재된 반응 성분 및 표 4에 기재된 사이클링 조건을 갖는 TCTTCAGAAATAAGCTTTTGTTCGGATCC. - PCR 및 DNA 클린업 키트로 PCR을 정제합니다. 20 μL의 뉴클레아제가 없는 H2O로 용리합니다.
- 효모 표면 디스플레이 벡터의 인서트 외부에 어닐링된 프라이머를 사용하여 PCR로 DNA를 증폭합니다.
- epPCR을 수행하여 무작위 돌연변이를 도입합니다.
- 표시된 성분(표 5) 및 조건(표 6)과 함께 뉴클레오티드 유사체를 사용하여 epPCR을 수행합니다.
- 분취용 1% 아가로스 겔에서 120분 동안 45분 동안 전체 epPCR 제품을 실행합니다. 마커를 포함하고 epPCR 산물과 ladder 사이에 하나의 차선을 남겨 둡니다. 인서트의 예상 크기에 해당하는 원하는 밴드를 절제하고 겔 추출 키트를 사용하여 정제한 다음 DNA 농도를 측정합니다.
- PCR로 무작위 DNA를 증폭합니다.
- Q5 중합효소로 DNA 50ng을 증폭합니다. 표시된 조건(표 8)으로 각 전기천공법(표 7)에 대해 2 x 100 μL의 PCR 반응을 설정합니다. 첫 번째 PCR에 사용된 것과 동일한 프라이머를 사용합니다.
참고: 효모로 전기천공을 하려면 많은 양의 DNA가 필요합니다. - 1% 아가로스 겔에서 5μL의 PCR 산물을 120V로 45분 동안 실행합니다.
- Q5 중합효소로 DNA 50ng을 증폭합니다. 표시된 조건(표 8)으로 각 전기천공법(표 7)에 대해 2 x 100 μL의 PCR 반응을 설정합니다. 첫 번째 PCR에 사용된 것과 동일한 프라이머를 사용합니다.
- 증폭된 DNA를 에탄올 침전으로 정제합니다.
- 2 x 100 μL를 함께 풀합니다. 멸균 여과된 3M 아세트산 나트륨 20μL(부피의 10%), pH 5.2를 추가합니다.
참고: 이 버퍼는 미리 준비하여 RT에서 몇 달 동안 저장할 수 있습니다. - 440 μL의 100% 에탄올(또는 최소 2 x 부피)을 첨가하고 RT에서 2분 동안 배양합니다.
- 상등액을 20,000 x g 에서 5분 동안 원심분리기합니다.상층액을 제거하고 500μL의 70% 에탄올을 첨가한 후 짧게 혼합합니다.
- 상등액을 20,000 x g 에서 5분 동안 원심분리기합니다.상층액을 제거하고 100% 에탄올 500μL를 첨가한 후 짧게 혼합합니다.
- RT에서 5분 동안 20,000 x g 에서 원심분리기. 상층액을 제거하고 모든 에탄올이 증발할 때까지 층류 후드 아래에서 DNA 펠릿을 건조시킵니다.
알림: 이 프로세스의 속도를 높이려면 48°C에서 열 블록을 사용하십시오. DNA 펠릿은 항상 보이지 않을 수 있습니다. - 건조된 DNA 펠릿을 10μL의 뉴클레아제가 없는 H2O에 용해시킵니다.
참고: DNA는 가능한 한 적은 부피로 용해되어야 합니다. DNA 농도를 측정하지 마십시오.
- 2 x 100 μL를 함께 풀합니다. 멸균 여과된 3M 아세트산 나트륨 20μL(부피의 10%), pH 5.2를 추가합니다.
| 부피 [μL] | 최종 집중 | |
| 5x Q5 인핸서 | 10 | 1배 |
| Q5 버퍼 5개 | 10 | 1배 |
| 프라이머 fwd 10 μM | 2.5 | 0.5 μM의 |
| 프라이머 회전 10μM | 2.5 | 0.5 μM의 |
| dNTP 10mM | 1 | 200 마이크로미터 |
| Q5 폴리메라이제 | 0.5 | 20U/mL |
| 효모 miniprep의 DNA | 10 | |
| 뉴클레아제가 없는 H2O | 13.5 |
표 3: 분리된 효모 miniprep에서 POI 유전자를 증폭하기 위한 1단계 PCR의 조건.
| 걸음 | 온도 | 시간 |
| 초기 변성 | 98 기음 | 30 초 |
| 25 사이클 | 98 기음 | 10 초 |
| 72 기음 | 30 초 | |
| 72 기음 | 30 초 | |
| 최종 확장 | 72 기음 | 2 분 |
| 들다 | 4 기음 |
표 4: 분리된 효모 miniprep에서 POI 유전자를 증폭하기 위한 1ststep PCR의 사이클링 조건.
| 부피 [μL] | 최종 집중 | |
| 뉴클레아제가 없는 H2O | 최대 50 | |
| 10x Thermopol 버퍼 | 5 | 1배 |
| Primer_fwd(10μM) | 2.5 | 0.5 μM의 |
| Primer_rev(10μM) | 2.5 | 0.5 μM의 |
| dNTP(10mM) | 1 | 200 마이크로미터 |
| 8-옥소-dGTP(100μM) | 1 | 2 μM |
| dPTP(100μM) | 1 | 2 μM |
| 1st PCR로부터의 PCR 산물 | XX | 50 ng |
| Taq DNA 중합효소 | 0.5 | 0.05U/μL |
표 5: 1단계 PCR로 POI DNA를 증폭한 후 수행되는 epPCR의 조건.
| 걸음 | 온도 | 시간 |
| 초기 변성 | 94 기음 | 30 초 |
| 15 사이클 | 94 기음 | 45 초 |
| 60 기음 | 30 초 | |
| 72 기음 | 1분 | |
| 최종 확장 | 72 기음 | 10분 |
| 들다 | 4 기음 |
표 6: epPCR의 사이클링 조건.
| 부피 [μL] | 최종 집중 | |
| 5x Q5 인핸서 | 20 | 1배 |
| Q5 버퍼 5개 | 20 | 1배 |
| 프라이머 fwd 10 μM | 5 | 0.5 μM의 |
| 프라이머 회전 10μM | 5 | 0.5 μM의 |
| dNTP 10mM | 1 | 200 마이크로미터 |
| Q5 폴리메라이제 | 1 | 20U/mL |
| DNA 50ng | XX | |
| DDH20 | 최대 100 |
표 7: EBY100 세포의 전기천공 전 epPCR 산물의 증폭을 위한 2단계 PCR의 조건.
| 걸음 | 온도 | 시간 |
| 초기 변성 | 98 기음 | 30 초 |
| 25 사이클 | 98 기음 | 10 초 |
| 72 기음 | 30 초 | |
| 72 기음 | 30 초 | |
| 최종 확장 | 72 기음 | 2 분 |
| 들다 | 4 기음 |
표 8: epPCR 산물의 증폭을 위한 2단계 PCR의 사이클링 조건.
8. electroporation을 위한 효모 디스플레이 벡터의 선형화
- pCTCON2 효모 표면 표시 벡터를 선형화합니다(Addgene plasmid #41843).
- 대규모 digest를 수행하고 이 digested linearized vector를 모든 추가 electroporations에 사용합니다.
- 초기 선형화를 위해 2 x 200 μg의 pCTCON2 플라스미드 DNA를 Sal I으로 37°C에서 24시간 동안 분해합니다(표 9).
- 인서트를 절제하기 위해 37°C에서 24시간 동안 NheI 및 BamHI로 분해합니다(표 10).
- 마지막으로 각 효소 5 μL를 튜브(Sal I, NheI, BamHI)에 넣고 37°C에서 24시간 동안 다시 배양합니다.
- 분취용 아가로스 겔에서 digested vector를 실행합니다. 밴드를 절제하고 젤 추출 키트로 정제합니다. 벡터를 20 μL로 용리시키고 DNA 농도를 측정한 후 약 2 μg/μL로 조정합니다.
참고: 장기간 플라스미드를 분해하면 insert의 충분한 절단이 보장되고 electroporation 후 vector의 background 가 줄어듭니다.
| 디에이지 | 200 μg의 |
| CutSmartBuffer 10개 | 50 마이크로L |
| 살 I-HF (NEB) | 30 μL (60 유) |
| H2O | 최대 500 μL |
표 9: 효모 표면 디스플레이 벡터 pCTCON2의 대규모 분해의 첫 번째 단계에 대한 조건.
| pCTCON2 (Sal I 소화) | 500 마이크로L |
| CutSmartBuffer 10개 | 37.5 μL |
| NHE-HF (네브) | 15 μL (30 유) |
| 밤하이-HF(NEB) | 15 μL (30 유) |
| H2O | 최대 875 μL |
표 10: 효모 표면 디스플레이 벡터 pCTCON2의 대규모 분해의 두 번째 단계에 대한 조건.
9. 무작위 DNA 및 선형화된 벡터를 사용한 EBY100의 전기천공법
- EBY100 효모 세포(ATCC, 재료 표 참조)를 전기천공법 3일 전에 글리세롤 스톡에서 타격하여 준비합니다. 비형질전환된 EBY100 세포에는 효모 표면 표시 벡터가 포함되어 있지 않으므로 전체 배지 YPD 플레이트를 사용합니다(표 1). 30 °C에서 배양하십시오.
- 전기천공법 하루 전에 EBY100 단일 콜로니 1개를 갓 준비한 30mL의 YPD 배지에 접종합니다(표 1). 180rpm으로 흔들면서 30°C에서 밤새 배양합니다.
참고: YPD 배지는 1개월 이상 되지 않아야 합니다. - 다음날 아침, EBY100 익일 배양을 YPD 배지 100mL에 0.2의 OD600 으로 희석하고 180rpm으로 진탕하면서 30°C에서 배양합니다.
참고: 50mL의 배양 부피는 2회의 전기천공에 충분합니다. 일반적으로 100mL는 대조군(세포만 및 세포와 벡터)을 위한 충분한 세포를 갖도록 준비됩니다. - 1.3-1.5의 OD600 에 도달하면 2000 x g 의 50mL 튜브에 3분 동안 세포를 펠렛화하고 상등액을 제거합니다.
- 펠릿을 100mM 멸균 여과 리튬 아세테이트 25mL에 재현탁합니다( 재료 표 참조). 갓 준비한 멸균 여과 DTT(1M)( 재료 표 참조)를 최종 농도 10mM에 추가합니다.
알림: 리튬 아세테이트 부피는 YPD의 원래 부피의 절반이어야 합니다. 필요한 경우 축소 또는 확대합니다. 리튬 아세테이트(100mM)는 미리 준비하고 RT에서 몇 달 동안 보관할 수 있지만 1M DTT 스톡은 사용 직전에 준비하고 얼음에 보관해야 합니다. - 10분 동안 흔들면서 30°C에서 세포를 배양합니다.
- 2000 x g 에서 3분 동안 원심분리기를 하고 상층액을 버리고 세포를 얼음 위에 놓습니다.
알림: 다음 모든 단계는 얼음과 냉각된 시약, 큐벳 및 미세 원심분리기 튜브를 사용하여 수행해야 합니다. - 펠릿을 25mL의 시원한 멸균 H2O. 원심분리기에 2000 x g 에서 3분 동안 재현탁합니다.
알림: 사용된 H2O 부피는 원래 YPD 부피의 절반이어야 합니다. - 350 μL의 멸균 H2O에 재현탁합니다.
참고: 재부유 후 총 부피는 500μL여야 합니다. 필요한 경우 H2O를 더 추가합니다. - DNA 라이브러리를 병렬로 준비: 에탄올 정제 삽입물이 들어 있는 마이크로 원심분리 튜브를 얼음 위에 놓고 4μg의 선형화된 pCTCON2 벡터를 추가합니다.
- 250 μL의 세포를 DNA에 넣고 잘라서 부드럽게 섞습니다. 미리 냉각된 2mm 전기천공법 큐벳으로 옮기고 전기천공이 이루어질 때까지 얼음에 보관합니다.
참고: 대조군(DNA가 없는 세포만 및 삽입 없이 선형화된 벡터만 있는 세포)을 추가하여 electroporation의 성공 여부를 확인합니다. - 다음 조건으로 2mm electroporation 큐벳( 재료 표 참조)을 사용하여 Electroporate: 구형파 프로토콜, 단일 펄스, 500V, 15ms 펄스 지속 시간.
참고: 일반적인 처짐은 3%-9%입니다. 다른 electroporators를 사용할 수 있지만 표시된 조건은 이 연구에 사용된 기기에 최적화되어 있습니다. - 예열된 YPD 1mL로 세포를 구조하고 예열된 마이크로 원심분리 튜브로 옮기고 30°C에서 1시간 동안 흔들지 않고 배양합니다.
알림: 가열 블록을 전기천공기 옆에 놓아 회수 튜브가 적절한 온도를 유지하도록 합니다. - SD-CAA 플레이트(표 1 참조)에서 세포의 10-3, 10-4, 10-5 및 10-6 희석액을 100μL로 플레이트화하고 30°C에서 형질전환체의 성장을 허용합니다.
참고: 이론적 라이브러리 다양성은 원래 배양 부피(여기서는 1mL)로 형질전환체의 수를 역으로 계산하여 얻습니다. 전형적으로, 이 형질전환 프로토콜은 대략 10개의7-10 8 개의 형질전환된 세포를 산출한다. - 나머지 세포를 2000 x g 에서 3분 동안 펠렛화하고 200mL SD-CAA에 재현탁한 다음 30°C에서 180rpm으로 밤새 진탕하면서 배양합니다. epPCR-randomized library를 포함하는 형질전환된 세포는 SD-CAA에서 배양하고 다음 날 SG-CAA에서 표면 발현을 유도하거나 추가 사용까지 4°C에서 보관할 수 있습니다. 다양성을 결정한 후 epPCR 후 라이브러리의 글리세롤 재고를 동결합니다. 다음 날에도 세포의 일부는 플라스미드를 얻지 않고 형질전환 절차에서 수행된 형질전환되지 않은 세포가 될 것이기 때문에 이러한 효과를 보상하기 위해 첫 번째 전달(passaging) 단계에서 라이브러리 다양성의 최소 20배를 계대배양하는 것이 좋습니다.
10. 여러 선택 라운드 후 효모 라이브러리의 염기서열분석
- 위와 같이 효모 miniprep을 수행하여 효모 DNA를 추출합니다.
- 분리된 DNA를 가진 상업용 E. coli 10-beta 전기 유능 세포( 재료 표 참조)를 Electroporate.
- 전기천공법 큐벳(1mm, 재료 표 참조)과 마이크로 원심분리기 튜브를 얼음 위에 놓습니다. 37°C에서 950μL의 성장 매체로 마이크로 원심분리 튜브를 예열하여 전기천공법 후 세포를 회수합니다.
- 얼음 위에서 세포를 해동하고 튜브를 부드럽게 튕겨 섞습니다. 50μL의 세포를 냉각된 마이크로 원심분리기 튜브로 옮깁니다. 효모 miniprep DNA 1 μL를 추가합니다.
- 세포-DNA 혼합물을 냉각된 전기천공법 큐벳에 조심스럽게 옮깁니다.
알림: 거품을 피하고 세포가 큐벳 바닥에 침전되도록 하십시오. - 뒤에 오는 조건을 사용하는 electroporation 장치를 가진 Electroporate: 2.0 kV; 200 Ω; 25μF입니다.
참고: 일반적인 시간 상수는 4.8-5.1ms입니다. 다른 electroporators를 사용할 수 있지만 표시된 조건은 이 연구에 사용된 기기에 최적화되어 있습니다. - 즉시 950μL의 37°C 예열 성장 배지( 재료 표 참조)를 큐벳에 추가하고 부드럽게 혼합한 다음 따뜻한 마이크로 원심분리기 튜브로 다시 옮깁니다.
알림: 가열 블록을 전기천공기 옆에 놓아 회수 튜브가 적절한 온도를 유지하도록 합니다. - 37°C 및 300rpm에서 40분 동안 배양합니다. 10 μL, 100 μL 및 나머지 세포(원심분리 후)를 예열된 선택적 LB 플레이트에 플레이트합니다( 표 1 참조). 플레이트를 37 ° C에서 밤새 배양하십시오.
참고: pCTCON2 벡터의 경우 항생제 선별에 암피실린이 사용됩니다. - LB 한천과 필요한 항생제(여기서는 암피실린이 사용됨)가 포함된 복사 플레이트를 준비합니다: 96웰 플레이트의 각 웰에 100μg/mL 암피실린이 함유된 LB 한천 200μL를 피펫팅하고 응고시킵니다.
- 웰당 100μg/mL 암피실린이 있는 LB 50μL가 포함된 96웰 염기서열분석 플레이트를 준비합니다.
- 플레이트에서 단일 콜로니를 선택하고 먼저 복사 플레이트와 시퀀싱 플레이트의 동일한 웰을 접종합니다.
참고: 복사 플레이트는 액체 배양을 접종하고 miniprep을 수행하여 원하는 플라스미드를 쉽게 얻을 수 있도록 합니다. - 복사 플레이트를 37°C에서 밤새 배양한 다음 4°C에서 보관합니다.
- 플라스틱 또는 알루미늄 호일로 덮인 쉐이킹(180rpm)하면서 37°C에서 3-4시간 동안 시퀀싱 플레이트를 배양합니다.
- 염기서열분석 플레이트를 밀봉하고 500μL의 10μM pCTCON2 염기서열분석 프라이머(5'-CGTTTGTCAGTAATTGCGGTTCTCTC)와 함께 염기서열분석을 위해 보냅니다.
- 염기서열 분석을 위해 얻어진 DNA 염기서열을 선택한 적절한 프로그램(예: EMBOSS Transeq38)을 사용하여 아미노산 염기서열로 번역합니다. 다음으로, MUSCLE(Multiple Sequence Alignment)39 또는 유사한 정렬 도구를 사용하여 변이 서열을 서로 정렬하고 부모 야생형 단백질에 정렬합니다.
참고: 서로 다른 선택 라운드의 라이브러리에 대한 염기서열분석을 수행함으로써(예: 이전 선택 라운드 후 및 높은 친화성 변이체를 강화하기 위해 더 낮은 항원 농도로 추가 선택 라운드 후) 다양한 변이체를 식별할 수 있습니다. 이것은 분류 라운드 중에 적용되는 게이트의 엄격함과 그에 따라 도서관의 다양성에 크게 좌우됩니다.
결과
G4 라이브러리는 소분자 약물 A1120에 결합된 항원 hRBP4에 대해 선택되었습니다. 유세포 분석을 위한 라이브러리의 염색은 Method 6에 설명된 대로 수행되었으며 적용된 게이팅 전략은 그림 2A에 나와 있습니다. 첫 번째 게이트에는 세포 형태에 따른 모든 세포가 포함되었고, 두 번째 게이트(FSC-Width의 히스토그램)는 단일 세포를 선택하고 세포 응집체를 제거하기 위해 적용된 엄격한 게이팅 전략을 보여주었습니다. 세 번째이자 마지막 게이트는 단백질 변이체(x축) 대 항원 결합(y축)의 표시를 보여주었습니다. 디스플레이 신호와 결합 신호를 모두 보여주는 효모 세포를 분류했습니다. 중요한 것은 sorting gate가 높은 결합 신호로 결합 도메인을 풍부하게 하여 높은 친화력을 갖도록 엄격한 방식으로 설정되었다는 것입니다. 이 엄격한 선택은 선택 캠페인 전반에 걸쳐 표적 항원에 특이적으로 결합하는 효모 세포를 표시하는 농축을 산출했습니다(그림 2B). 이후의 유세포 분석 분류 라운드에서 항원 농도는 10배 감소했습니다(100nM에서 10nM으로). 따라서 전체 결합 신호가 감소하고 친화도가 높은 바인더만 여전히 검출 및 분류되었습니다(그림 2C).
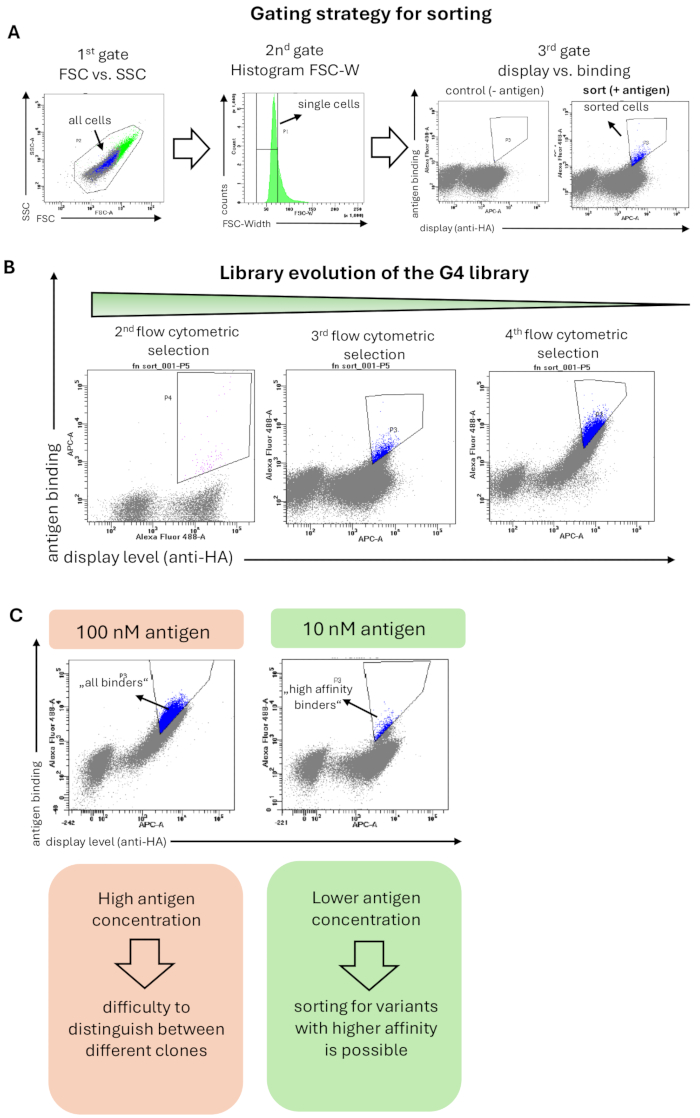
그림 2: 항원(A1120이 있는 경우 hRBP4)에 결합하기 위한 Fn3 기반 G4 라이브러리의 효모 표면 디스플레이 선택의 대표적인 결과. (A) 효모 라이브러리의 분류를 위한 일반적인 게이팅 전략. 첫 번째 게이트(FSC 대 SSC)는 모든 효모 세포를 선택하고 산란 이벤트를 제외하는 것입니다. 두 번째 게이트(FSC-W의 히스토그램)는 세포 응집체를 제거하고 단일 효모 세포만 선택하는 것을 목표로 합니다. 세 번째 게이트는 표면 표시 수준(HA- 또는 c-myc-태그의 검출) 대 항원에 대한 결합(여기서는 항-His 항체에 의해 검출된 5μM A1120의 존재 하에서 hRBP4)을 플롯합니다. 라이브러리는 항원 결합이 예상되지 않는 2차 항체(항원 없음)로만 추가로 염색되었습니다. 정렬된 셀은 파란색으로 강조 표시됩니다. (B) 3 라운드의 유세포 분석 분류를 통한 G4 라이브러리의 진화. 결합 개체군의 농축은 각 선택 라운드에서 관찰할 수 있습니다. (C) 더 낮은 항원 농도를 사용하면 표적 항원에 대한 친화도가 더 높은 단백질 변이체를 선택할 수 있습니다. 항원 농도(여기서는 hRBP4)가 10배 감소하면 서로 다른 대각선이 나타나며, 이는 친화도가 높거나(정렬된 세포, 파란색) 친화도가 낮은(분류된 세포, 파란색) 클론의 존재를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
효모 표면 디스플레이는 단백질 엔지니어링에 사용되는 핵심 방법 중 하나로 발전해 왔습니다. 친화력 1,18,40,41, 발현/안정성 24,27,42,43 및 활성28,44의 엔지니어링에 일반적으로 사용되지만, 항원결정기 매핑 45,46 또는 효모 세포 표면의 개별 돌연변이 특성화와 같은 추가 용도9 도 가능합니다. 이 프로토콜에서는 마그네틱 비드를 사용한 선택 및 유세포 분석적 분류를 통한 선택뿐만 아니라 친화성 성숙을 위한 epPCR에 의한 효모 라이브러리의 다양화를 포함하여 효모 표면 디스플레이 선택 캠페인을 시작하기 위한 기본 단계를 제공합니다.
기존 효모 표면 디스플레이 선택에 대한 한 가지 필수 요구 사항은 충분한 품질의 용해성 단백질을 사용할 수 있어야 한다는 것입니다. 순도가 높고 올리고머화 상태가 정의된 잘 접힌 표적 단백질(즉, 단량체 단백질은 단량체로만 존재해야 함)으로 시작하면 친화도가 높은 표적 항원에 결합하는 단백질 변이체를 선택할 수 있는 가장 높은 성공률을 제공합니다. 발현이 어려운 표적 단백질에 대한 대안은 세포 기반 선택이며, 이는 이러한 제한을 우회할 수 있는 합리적인 전략을 제시합니다47. 그러나 효모 표면 디스플레이는 힘들고 시간이 많이 소요되는 클로닝, 용해성 형식으로 발현 및 단백질 정제를 수행할 필요 없이 효모 표면에서 생성된 단백질 변형체를 직접 특성화할 수 있는 가능성과 같은 많은 이점을 제공합니다. 변이체의 친화력과 안정성은 모두 효모 표면9에서 직접 분석할 수 있습니다.
이 프로토콜에서는 단백질 변이체의 G4 라이브러리, 보다 구체적으로 인간 피브로넥틴의 10번째 유형 III 도메인의 G4 라이브러리가 소분자 A1120의 존재 하에서 항원 hRBP4에 결합하기 위해 어떻게 선택되었는지 보여줍니다. bead selection과 유세포 분석적 분류의 조합은 변형체의 농축을 산출했으며, 이는 selection round 전반에 걸쳐 표적 항원에 대한 결합이 증가한 것을 보여주었습니다(그림 2B). 더 낮은 농도의 항원을 사용하면 친화성이 높은 단백질 변이체를 선택할 수 있음을 보여주었습니다(그림 2C). 전형적으로, 효모 표시 선택으로 달성할 수 있는 친화도는 나노몰 또는 심지어 피코몰 범위(18)에 있다. 최종 친화도는 표적 항원, 선택 라운드 수 및 친화성 성숙, 사용된 결합 골격 및 적용된 게이팅 전략에 따라 달라집니다. 개별 단백질 변이체의 특성화는 이 프로토콜에서 다루지 않지만 이전 연구에서 자세히 설명했습니다9. 효모 디스플레이는 원래 scFvs 1,40과 같은 항체 단편의 엔지니어링에 사용되었지만, 이 방법은 비항체 기반 단백질에도 널리 사용되었습니다10.
요약하자면, 효모 표면 디스플레이는 거의 모든 표적 단백질에 대한 결합 및/또는 안정성 향상과 같은 새롭거나 향상된 특성을 가진 단백질 변형체를 생성할 수 있는 강력한 단백질 엔지니어링 도구입니다.
공개
M.W.T.는 Miltenyi Biotec으로부터 자금을 지원받습니다. 모든 저자는 효모 표면 디스플레이를 사용하여 개발된 기술 및 엔지니어링 단백질에 대한 특허 출원의 발명자입니다.
감사의 말
이 연구는 오스트리아 과학기금 (FWF Project W1224 - Doctoral Program on Biomolecular Technology of Proteins - BioToP and FWF Project ESP 465-B), 오스트리아 연방 디지털 경제부, 오스트리아 국립 연구, 기술 및 개발 재단, 크리스티안 도플러 연구 협회 (Christian Doppler Laboratory for Next Generation CAR T Cells)의 지원을 받았습니다. St. Anna Children's Cancer Research Institute(오스트리아 비엔나)에 대한 개인 기부금으로. E.S.는 St. Anna Children's Cancer Research Institute에서 Austrian Academy of Sciences의 DOC Fellowship을 수상했습니다. 피규어는 BioRender.com 으로 만들어졌습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
참고문헌
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210 (2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508 (2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유