Method Article
off-target 유전자 편집에 대한 조사를 위한 CIRCLE-Seq
요약
CRISPR과 같은 기술의 주요 장벽은 중요한 유전자를 파괴할 수 있는 off-target 이벤트입니다. 'Circularization for In Vitro Reporting of Cleavage Effects by Sequencing'(CIRCLE-seq)은 의도하지 않은 절단 부위를 식별하기 위해 고안된 기술입니다. 이 방법은 CRISPR-Cas9의 게놈 전체 활성을 높은 감도로 편향 없이 매핑합니다.
초록
CIRCLE-seq(Circularization for In Vitro Reporting of Cleavage Effects by Sequencing)는 CRISPR-Cas9 절단 DNA의 표적 염기서열분석을 통해 CRISPR-Cas9의 의도하지 않은 절단 부위를 공정하게 식별하기 위해 개발된 새로운 기술입니다. 이 프로토콜은 게놈 DNA(gDNA)를 순환화하는 것을 포함하며, 이 DNA는 Cas9 단백질과 관심 있는 가이드 RNA(gRNA)로 처리됩니다. 처리 후, 절단된 DNA는 정제되어 Illumina 염기서열분석을 위한 라이브러리로 준비됩니다. 염기서열분석 프로세스는 쌍단 판독을 생성하여 각 절단 부위에 대한 포괄적인 데이터를 제공합니다. CIRCLE-seq는 Cas9-cleaved gDNA에 대한 최소 염기서열분석 깊이 요구 사항, 낮은 백그라운드 및 높은 농축을 포함하여 다른 in vitro 방법에 비해 여러 가지 이점을 제공합니다. 이러한 장점은 의도된 분열 사건과 의도하지 않은 분열 사건을 모두 식별하는 데 있어 민감도를 향상시킵니다. 이 연구는 CIRCLE-seq를 사용하여 CRISPR-Cas9의 off-target 활성을 조사하기 위한 포괄적인 단계별 절차를 제공합니다. 예를 들어, 이 프로토콜은 AAVS1 유전자 자리가 변형되는 동안 CRISPR-Cas9의 게놈 전체 의도하지 않은 절단 부위를 매핑하여 검증됩니다. 전체 CIRCLE-seq 공정은 2주 이내에 완료할 수 있어 세포 성장, DNA 정제, 라이브러리 준비 및 Illumina 염기서열분석에 충분한 시간을 할애할 수 있습니다. CIRCLE-seq 파이프라인에 염기서열분석 데이터를 입력하면 절단 부위의 간소화된 해석 및 분석이 가능합니다.
서문
게놈 공학은 지난 20년 동안 상당한 발전을 이루었으며, 2012년 CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)-Cas9의 발견이 중요한 이정표가 되었습니다1. CRISPR-Cas9 기술은 박테리아 DNA 엔도뉴클레아제의 프로그래밍 가능한 특성을 활용하여 거의 모든 DNA 염기서열의 정확한 표적화 및 수정을 가능하게 합니다. 이 시스템은 처음부터 Cas9 엔도뉴클레아제와 가이드 RNA(gRNA)에만 의존하여 특정 게놈 영역을 편집하도록 최적화되었습니다. CRISPR-Cas9의 치료 치료제로서의 잠재력은 Leber의 선천성 무뇨증, 트랜스티레틴 아밀로이드증, 겸상 적혈구 빈혈과 같은 다양한 질환에 대한 임상 시험에서 입증되었습니다 2,3,4.
CRISPR-Cas9는 일반적으로 두 가지 메커니즘 중 하나에 의해 해결되는 이중 가닥 절단(DSB)을 유도합니다: 주형 DNA를 사용할 수 있는 경우 오류가 발생하기 쉬운 비상동 말단 결합(NHEJ) 또는 보다 정밀한 상동성 유도 복구(HDR) 중 하나에 의해 해결됩니다. CRISPR-Cas9가 의도하지 않은 게놈 부위에서 분열과 함께 NHEJ 관련 삽입 및 결실(indels)을 유발하는 경향으로 인해 임상 환경에서의 적용이 제한됩니다 5,6,7,8,9,10. 또한, 의도하지 않은 게놈 변형은 수수께끼 같은 스플라이스 부위, 넌센스 또는 미센스 돌연변이를 생성하거나, 염색체분증을 유도하거나, 세포에 발암 가능성을 부여할 수 있으며, 이는 여러 게놈 편집 시험에서 관찰된 결과입니다 11,12,13,14,15. 결론적으로, CRISPR-Cas9의 off-target 활성을 정확하게 식별하는 것은 특히 수십억 개의 세포를 변화시킬 수 있는 전신 유전자 치료에서 임상 적용에 매우 중요합니다.
CRISPR-Cas9 off-target cleavage site를 식별하기 위해 다양한 방법을 사용할 수 있으며, 여기에는 이중 가닥 oligodeoxynucleotide를 사용하여 살아있는 세포의 DSB를 태그하는 GUIDE(Genome-wide Unbiased Identification of Double-stranded break)-seq 16이 포함됩니다. 그럼에도 불구하고 이 방법에 대한 비판은 무작위 DSB 또는 PCR 아티팩트에서 위양성이 발생할 수 있으며, 이는 타겟 사이트와 유사성이 좋지 않은 캡처된 사이트를 제외하여 폐기해야 한다는 것입니다. Integrase-Defective Lentiviral Vector(IDLV)의 사용에 기반한 방법은 민감도가 낮고 많은 off-target site를 놓칠 가능성이 있습니다17. DSBCapture, BLESS 및 BLISS 18,19,20과 같은 다른 in situ 방법은 고정 세포를 포함하고 DSB를 직접 표지하지만 즉각적인 DSB 포획에 대한 의존성과 외인성 DNA의 부재로 인해 제약이 있습니다. in vitro 방법인 Digenome-seq21과 염기서열분석에 의한 태그된 게놈 DNA 말단의 선택적 농축 및 식별(SITE-seq)22은 모두 염기서열분석 솔루션을 제공하지만 각각 배경 잡음 및 단일 말단 분석에 한계가 있습니다. in situ Cas off-target의 발견 및 염기서열분석(Discover-Seq)23에 의한 검증은 MRE11 결합을 통한 Cas9 활성의 in vivo 및 in situ 식별을 제공하지만 샘플 준비24 시점에 존재하는 DSB만 검출합니다. 마지막으로, ICE(Inference of CRISPR Edits)는 생물정보학(bioinformatics) 접근법을 사용하여 Sanger 데이터25를 사용하여 CRISPR 편집을 강력하게 분석합니다.
이 논문은 관심 gRNA26이 있는 복합체에서 Cas9 nuclease의 게놈 전체 off-target 활성을 민감하고 공정하게 매핑하는 in vitro 기술인 CIRCLE-seq(In vitro Reporting of Cleavage Effects by Sequencing)를 위한 Circularization에 대한 자세한 절차를 설명합니다. 이 접근법은 관심 세포를 배양하고 DNA를 분리하는 것으로 시작하여 집속 초음파를 통한 무작위 전단, 엑소뉴클레아제 및 리가아제 처리로 시작합니다. 이 과정은 궁극적으로 원형 이중 가닥 DNA 분자를 생성한 다음 플라스미드에 안전한 DNase 처리를 통해 정제됩니다. 그런 다음 이 원형 DNA는 의도된 절단 부위와 의도하지 않은 절단 부위 모두에서 절단된 Cas9-gRNA 복합체에 노출되어 Illumina 어댑터 접합을 위한 기질 역할을 하는 노출된 DNA 말단을 남깁니다. 이 프로세스는 각 뉴클레아제 유도 DSB의 양쪽 말단을 포함하는 다양한 게놈 DNA(gDNA) 라이브러리를 생성하여 각 판독에 각 절단 부위에 필요한 모든 정보가 포함되도록 합니다. 이를 통해 더 낮은 염기서열분석 커버리지 요구 사항으로 Illumina 염기서열분석을 사용할 수 있으며, CIRCLE-seq는 위에서 언급한 다른 유사한 방법과 차별화됩니다. CIRCLE-seq는 in vitro 방법으로서 다른 프로토콜보다 더 높은 off-target sensitivity를 갖지만, GUIDE-seq16과 같은 다른 방법에 존재하는 후성유전학적 환경이 없기 때문에 더 높은 위양성 결과가 발생한다는 점에 유의하는 것이 중요합니다. 또한, DSB DNA 복구 및 관련 기전은 CIRCLE-seq에 존재하지 않으며, 그렇지 않으면 관찰될 수 있는 indels 또는 적절한 복구를 폐지합니다.
이 프로토콜은 CIRCLE-seq를 수행하기 위한 단계별 프로토콜을 설명하는 것 외에도 예를 들어 AAVS1 유전자 자리의 변형 중에 발생하는 CRISPR-Cas9의 게놈 전체의 의도하지 않은 절단 부위를 식별하여 검증됩니다. 따라하기 쉬운 이 프로토콜은 유도만능줄기세포(iPSC) 배양 및 gDNA 분리부터 gDNA 순환화, Cas9-gRNA 절단, 라이브러리 준비, 염기서열분석 및 파이프라인 분석에 이르기까지 자세한 지침을 제공합니다. 염기서열분석 적용 범위가 낮다는 점을 감안할 때, CIRCLE-seq는 차세대 염기서열분석에 액세스할 수 있는 모든 실험실에서 사용할 수 있습니다.
프로토콜
이 연구에 사용된 시약, 소모품 및 장비의 세부 정보는 재료 표에 나열되어 있습니다.
1. 세포 배양 (5일)
- 이 프로토콜 전체에 음수 컨트롤을 포함합니다. 추가로 25μg의 gDNA를 위한 충분한 세포를 준비합니다(샘플당 ~2.0e7 개의 세포).
- 확립된 프로토콜에 따라 iPSC를 배양합니다27. 세포를 수집하고 PBS 10mL에 재현탁합니다. 6μL 세포 샘플을 피펫팅하고 트리판 블루와 1:1 비율로 재현탁합니다. 자동화된 세포 계수기를 사용하여 샘플을 계수합니다.
- 튜브당 부분 표본 2 x 107 세포를 분취한 다음 25°C(실온(RT))에서 3분 동안 300 x g 로 스핀다운합니다. 이는 다중 반복실험에 충분합니다. 피펫을 끄고 상층액을 버립니다.
2. 게놈 DNA 분리 (1일)
- 제조업체의 지침에 따라 시중에서 판매되는 DNA 정제 키트를 사용하여 gDNA를 분리합니다.
- 세포 펠릿이 들어 있는 15mL 코니컬 튜브에 PBS 200μL를 추가하고 재현탁합니다. 그런 다음 세포 용해 완충액 3mL와 proteinase K 15μL를 튜브에 피펫팅합니다. 튜브를 25회 뒤집어 완전히 섞습니다. 최적의 DNA 수율을 위해 55°C 및 150rpm으로 설정된 수조 셰이커에 튜브를 3시간 동안 또는 하룻밤 동안 놓습니다.
- RNase A의 15 μL에 피펫을 넣으십시오. 25회 반전합니다. 37°C의 수조에 1시간 동안 넣습니다.
- 샘플을 얼음에 5분 동안 식힙니다. 그런 다음 단백질 침전 용액 1mL를 추가하고 20초 동안 고속으로 와류를 일으키고 실온에서 10분 동안 2000 x g 로 원심분리합니다.단백질은 튜브 바닥에 눈에 보이는 소형 펠릿을 형성해야 합니다. 펠릿이 보이지 않으면 얼음 위에서 샘플을 추가로 5분 동안 배양한 후 다시 원심분리합니다.
- 100% 이소프로판올 3mL를 새 15mL 코니컬 튜브에 추가합니다. 2.1.3 단계의 상등액을 튜브에 조심스럽게 피펫으로 넣습니다. 튜브를 50회 뒤집어 섞은 다음 2000 x g 에서 원심분리하고 실온에서 3분 동안 가열합니다. DNA 펠릿을 방해하지 않고 진공 트랩에 연결된 파스퇴르 피펫을 사용하여 상층액을 조심스럽게 흡인하고 깨끗하고 보푸라기가 없는 닦음으로 튜브를 뒤집습니다.
- 70%(v/v) 에탄올 3mL를 DNA 펠릿에 피펫팅하고 10회 반전시켜 세척합니다. 2,000 x g 에서 RT에서 3분 동안 원심분리기. 그런 다음 상등액을 조심스럽게 붓습니다.
- 튜브를 열어 두고 생성된 DNA 펠릿을 30분 동안 건조시켜 모든 에탄올이 완전히 증발하도록 합니다. 50 μL의 DNA 수화 용액에 피펫팅하고 부드러운 피펫팅으로 철저히 혼합합니다.
- 65°C의 수조 셰이커에 샘플을 넣고 1시간 동안 DNA를 용해한 다음 샘플을 RT에서 밤새 방치합니다. 실온 및 2,000 x g 에서 1분 동안 샘플을 원심분리하고 관련 튜브와 함께 dsDNA BR Assay Kit를 사용하여 형광계로 분리된 DNA를 정량화합니다.
3. gRNA 준비 (7일)
- 상업적인 출처에서 관심 있는 합성 gRNA를 주문합니다( Table of Materials(재료 표 참조). 이 프로토콜은 crRNA/tracrRNA와도 호환됩니다.
4. gRNA in vitro cleavage test
참고: 여기서는 AAVS1 유전자의 표적이 사용됩니다. 다른 관심 유전자를 표적으로 삼으려면 타겟 영역을 증폭하도록 프라이머(표 1)를 설계하고 다음 단계의 프라이머를 맞춤형 프라이머로 교체합니다.
- PCR 반응 준비: Phusion Hot Start Flex 2x 마스터 믹스 25μL(최종 농도 1x), AAVS1 F 프라이머 0.5μL(최종 농도 0.1μM), AAVS1 R 프라이머 0.5μL(최종 농도 0.1μM), gDNA 5μL(100ng, 20ng/μL, 단계 2.1.6) 및 19μL의 뉴클레아제가 없는 H2O(총 부피: 50 μL)입니다.
- 뒤에 오는 thermocycler 모수를 사용하십시오: 변성: 2 분 (1 주기) 동안 98 °C, 변성: 10 s (10 주기) 동안 98 °C, 어닐링: 15 s (10 주기) 동안 72-62 °C (- 1 °C/주기), 연장: 30 s (10 주기) 동안 72 °C, 변성, 10 s (30 주기) 동안 98 °C. 어닐링: 15초(30사이클) 동안 65°C, 확장: 30초(30사이클) 동안 72°C, 최종 확장: 5분(1사이클) 동안 72°C, 유지: 4°C 무기한.
- AMPure XP 비드를 사용하여 PCR 반응 산물을 정제합니다. 먼저, 1.8배 부피 또는 90μL의 XP 비드를 PCR 산물에 피펫으로 넣습니다. 철저하게 혼합하는 10회 피펫. 혼합물을 RT에서 5분 동안 그대로 두어 배양합니다.
- 마그네틱 랙을 사용하여 PCR 반응 플레이트를 자석에 3분 동안 올려 놓아 용액에서 비드를 분리합니다. 세척된 용액을 피펫으로 제거하고 폐기합니다. 비드에 80% 에탄올(v/v) 200μL를 추가하고 30초 동안 배양한 다음 에탄올을 제거합니다. 이 세척 단계를 두 번 반복하여 에탄올을 완전히 제거하십시오.
- 플레이트를 자석에 올려 샘플을 3분 동안 자연 건조시킵니다. 자석에서 플레이트를 제거하고 40μL의 TE 버퍼, pH 8.0을 추가합니다. 위아래로 10회 피펫팅하여 혼합합니다. 샘플을 2분 동안 RT에 그대로 둡니다.
- PCR 반응 플레이트를 자석에 1분 더 놓습니다. 1분 후 상등액을 새 플레이트로 옮깁니다. 분광광도계를 사용하여 정제된 PCR 수율을 측정하고, 제조업체의 지침에 따라 고감도 D1000 ScreenTape 및 고감도 D1000 시약(ladder 및 buffer)과 함께 광학 튜브 스트립 캡이 있는 광학 튜브 스트립을 사용하여 TapeStation에서 분석합니다. 준비된 샘플을 -20°C에서 최대 몇 개월 동안 보관하십시오.
- 다음과 같이 Cas9 nuclease 단백질을 1μM로 희석합니다: 총 부피 20μL에서 10x Cas9 완충액 2μL(최종 농도 1x), Cas9 Nuclease 1μL, S. pyogenes (최종 농도 1μM) 및 Nuclease-free H2O 17μL를 혼합합니다.
- gRNA 분해를 방지하기 위해 RNase-free 절차를 수행합니다. gRNA를 (step 3.1에서) H2O의 3 μM로 묽게 하여 총 부피 10 μL로 만듭니다.
참고: 다음 공식을 사용하여 gRNA의 분자량을 추정하십시오: ssRNA의 분자량(g/mol) = (ssRNA의 길이(nt) x 321.47g/mol) + 18.02g/mol. 참고로, 3 μM에서 104 nt 길이의 gRNA는 약 100 ng/μL입니다.
5. DNA 전단 (3 시간)
- 먼저 컨트롤 암을 원점으로 돌려 ME220을 준비합니다. 그런 다음 정제 된 탈 이온화 된 H2O로 저장소를 채 웁니다. 제어 스테이션 노트북에서 Water Works 에 액세스하고 Fill을 클릭합니다. 온도를 4.5 °C로 조정합니다.
- 25 μg의 gDNA를 마이크로튜브(microtube-130 AFA Fiber Screw-Cap)로 전달합니다. 그런 다음 1x TE로 튜브를 총 부피 130μL까지 채웁니다. 다음 조건을 사용하여 DNA를 약 300 bp의 평균 길이로 전단합니다. 최대 전력은 70입니다. 듀티 팩터 %에서 20으로; 50으로 사이클/버스트; 이 모든 것은 자동으로 평균 파워를 14.0으로 설정합니다.
6. 전단 게놈 DNA 정제 (1 시간)
- 전단된 게놈 DNA를 각각 65μL의 두 부분으로 나눕니다. 4.3-4.6단계에 설명된 절차에 따라 XP beads 부피의 1.8배(117μL)를 사용하여 정제합니다. 상층액을 새 PCR 플레이트로 옮기고 분광 광도계를 사용하여 양을 측정합니다.
- 제조업체의 지침에 따라 TapeStation에서 용출된 전단 gDNA의 1 μL를 실행하여 gDNA가 약 300 bp의 넓은 분포로 전단되었는지 확인합니다. 필요한 경우 전단된 gDNA를 -20°C에서 최대 몇 개월 동안 보관합니다.
7. CIRCLE-seq 라이브러리 준비(3일)
- 헤어핀 어댑터 어닐링
- 헤어핀 어댑터인 oSQT1288(표 1)을 1x TE에서 최종 농도 100μM로 재현탁합니다.
- 다음과 같이 어댑터 어닐링을 수행합니다: 총 부피 100μL에 대해 oSQT1288 40μL(최종 농도 40μM), 10x STE(최종 농도 1x) 10μL 및 50μL의 Nuclease-Free H2O를 혼합합니다.
- 다음 어닐링 매개변수를 사용하십시오: 95분 동안 5°C, 70주기 동안 분당 -1°C, 4°C에서 무기한 유지.
- 마침 복구를 수행합니다. PCR-Free HTP 라이브러리 준비 키트를 사용하여 End-Repair 마스터 믹스를 준비합니다.
- 8 μL의 Nuclease-Free H2O, 7 μL의 10x 말단 복구 완충액(최종 농도 1x) 및 5 μL의 말단 복구 효소 혼합물(최종 부피 20 μL의 총 말단 복구 마스터 믹스)을 혼합합니다.
- 말단 수리 마스터의 20μL를 4.3-4.6단계에서 전단된 gDNA 샘플에 혼합합니다. 20 μL의 End-Repair Master Mix와 50 μL의 전단 gDNA를 혼합하여 최종 부피 70 μL를 만듭니다.
- 혼합물을 20°C의 열순환기에 30분 동안 넣은 다음 4°C에서 무기한 유지합니다.
- 1.7x 부피 또는 120 μL의 XP 비드를 추가하고 4.3-4.6 단계의 정제 단계를 따릅니다. 42μL의 TE, pH 8.0으로 용리합니다. 다음 단계를 위해 비드가 용액에 남아 있는지 확인하십시오.
- A-테일링을 수행합니다. HTP Library Preparation (PCR-free) Kit를 사용하여 A-tailing Master Mix를 준비합니다.
- 5μL의 10x A-테일링 완충액(최종 농도 1x)과 3μL의 A-테일링 효소(총 8μL의 A-테일링 마스터 믹스)를 혼합합니다.
- 7.2.4단계에서 비드를 포함하는 각 DNA 샘플에 A-tailing Master Mix의 8μL를 피펫으로 주입하고, 8μL의 A-tailing Master Mix를 42μL의 End-Repaired DNA를 함유하는 비드(총 최종 부피 50μL)를 혼합합니다. 30°C의 열순환기에 30분 동안 놓습니다. 4 °C에서 무기한 유지하십시오.
- 1.8부피 또는 90μL의 PEG/NaCl SPRI 용액(HTP Library Preparation Kit의 구성 요소(PCR-free, 96회 반응))을 A-tailed DNA에 피펫으로 분사합니다. 4.3-4.6 단계에 따라 A-tailed DNA를 정제합니다. A-tailed DNA를 30μL의 TE, pH 8.0으로 용리합니다. 다음 단계를 위해 비드를 용액에 보관하십시오.
- 어댑터 접합을 수행합니다. HTP Library Preparation (PCR-free) Kit를 사용하여 Adapter Ligation Master Mix를 준비합니다.
- 7.1.3단계에서 10μL의 5x ligation buffer(최종 농도 1x), 5μL의 DNA Ligase 및 5μL의 어닐링된 헤어핀 어댑터(40μM)를 혼합합니다. 20 μL의 총 어댑터 결찰 마스터 믹스에 대해 최종 농도 4 μM를 보장합니다.
- 7.3.3단계의 비드를 포함하는 각 용리된 DNA 검체에 어댑터 결찰 마스터 믹스 20μL를 피펫팅합니다(시료당 총 최종 부피 50μL).
- 20°C의 열순환기에 1시간 동안 넣습니다. 4 °C에서 무기한 유지하십시오.
- 1배 부피의 PEG/NaCl SPRI 용액을 어댑터 결찰 DNA로 옮기고 4.3-4.6단계에 따라 정제합니다. 30μL의 TE, pH 8.0으로 용리하고 상층액을 새로운 semi-skirted PCR 플레이트로 디캔트합니다. dsDNA BR assay를 사용하여 DNA를 결합하고 정량화합니다. 필요한 경우 정제된 어댑터 결찰 DNA를 -20°C에서 최대 1개월 동안 보관합니다.
- Lambda Exonuclease/Exonuclease I (E. coli) Master Mix(양쪽 말단에 접합된 어댑터 없이 단일 또는 이중 가닥 DNA를 제거하는 기능)를 준비합니다.
- 7.4.4 단계에서 1 μg의 어댑터 결찰 DNA를 40 μL로 희석합니다. 10x 엑소뉴클레아제 I 반응 완충액 5μL(최종 농도 1x), 람다 엑소뉴클레아제 4μL(최종 농도 0.4 U/μL) 및 엑소뉴클레아제 I(E. coli) 1μL(최종 농도 0.4 U/μL)를 10 μL의 람다 엑소뉴클레아제/엑소뉴클레아제 I 마스터 믹스의 총 부피에 혼합합니다.
- Lambda 엑소뉴클레아제/엑소뉴클레아제 I 마스터 믹스 10 μL를 40 μL(1 μg)의 어댑터 결찰 DNA(총 부피 50 μL)에 피펫팅합니다. 37°C에서 1시간 동안 열순환기에 넣은 다음 75°C에서 10분 동안 가열합니다. 4 °C에서 무기한 유지하십시오.
- 1.8배 부피 또는 90 μL의 XP 비드를 Lambda Exonuclease/Exonuclease I-treated DNA에 피펫팅합니다. 4.3-4.6 단계에 제공된 지침에 따라 정화하십시오. 40μL의 TE, pH 8.0에서 용리합니다. 다음 효소 단계를 위해 비드가 용액에 남아 있는지 확인하십시오.
- USER 효소 및 T4 폴리뉴클레오티드 키나아제(PNK)로 치료합니다. USER/T4 PNK 마스터 믹스를 준비합니다(4-bp 돌출부를 분리하고 후속 ligation 반응에 필요한 ligation-ready DNA end를 준비하는 데 필요).
- 10 μL의 USER/PNK 마스터 믹스의 총 부피에 대해 5 μL의 10x T4 DNA 리가아제 완충액(최종 농도 1x), 3 μL의 USER 효소(최종 농도 0.05 U/μL) 및 2 μL의 T4 PNK(최종 농도 0.4 U/μL)를 혼합합니다.
- USER Enzyme/T4 PNK Master Mix 10μL를 7.5.3단계의 비드를 함유한 40μL의 lambda 및 exonuclease I 처리된 DNA 검체에 총 부피 50μL로 피펫팅합니다. 37°C의 열순환기에 1시간 동안 넣습니다. 4 °C에서 무기한 유지하십시오.
- USER/T4 PNK로 처리한 DNA에 1.8배 부피 또는 90μL의 PEG/NaCl SPRI 용액을 피펫팅하고 4.3-4.6단계에 따라 정제합니다. 35μL의 TE, pH 8.0에서 용리합니다. 상층액을 새로운 semi-skirted PCR 플레이트에 디캔팅합니다. dsDNA HS assay를 사용하여 DNA를 결합하고 정량화합니다.
- 분자 내 순환화를 수행합니다. Circularization Master Mix를 준비합니다.
- 8 μL의 Nuclease-free H2O, 10 μL의 10x T4 DNA 리가제 완충액(최종 농도 1x) 및 2 μL의 T4 DNA 리가제(최종 농도 8 U/μL)를 혼합하여 20 μL의 순환화 마스터 믹스의 총 부피를 만듭니다.
- 7.6.3단계에서 USER/PNK로 처리된 DNA 500ng에 Circularization Master Mix 20μL를 피펫팅합니다. USER/PNK로 처리된 DNA 500ng을 80μL에 희석한 다음 Circularization Master Mix 20μL(총 부피 100μL)를 추가합니다. 16°C의 열순환기에서 16시간(밤새) 동안 배양합니다.
- 원형 DNA에 1x 부피 또는 100 μL의 XP bead를 추가하고 4.3-4.6 단계에 따라 정제합니다. 38μL의 TE, pH 8.0에서 용리합니다. 상층액을 새로운 semi-skirted PCR 플레이트에 디캔팅합니다.
- Plasmid-Safe ATP-Dependent DNase로 치료합니다. Plasmid-Safe ATP-Dependent DNase Master Mix를 준비합니다(잔류 선형 DNA의 분해에 필요).
- 10x Plasmid-safe 반응 완충액 5μL(최종 농도 1x), ATP 2μL(최종 농도 1mM) 및 Plasmid-safe ATP-dependent DNase 5μL(최종 농도 1 U/μL)를 혼합하여 총 부피 12 μL의 Plasmid-safe Master Mix를 만듭니다.
- ATP 의존성 DNase 마스터 믹스 12 μL를 7.7.3 단계에서 원형 DNA 38 μL로 피펫팅합니다(총 부피 50 μL). 37°C에서 1시간 동안 thermocycler에서 배양한 다음 70°C에서 30분 동안 배양합니다. 4 °C에서 무기한 유지하십시오.
- 1x 부피 또는 50 μL의 XP 비드를 Plasmid-Safe ATP-Dependent DNase로 처리한 DNA에 피펫팅하고 4.3-4.6 단계에 따라 정제합니다. 15μL의 TE, pH 8.0에서 용리합니다. 상층액을 새로운 semi-skirted PCR 플레이트에 디캔팅합니다.
- DNA를 결합하고 dsDNA HS assay를 사용하여 정량화합니다. 필요한 경우 원형 DNA를 -20 °C에서 최대 몇 개월 동안 보관합니다.
8. 효소로 정제된 원형 gDNA 를 in vitro에서 절단(2시간)
- Cas9:gRNA complex를 이용한 in vitro cleavage를 수행합니다. in vitro Cleavage Master Mix를 준비합니다. 5 μL의 10x Cas9 완충액(최종 농도 1x), 4.5 μL의 S. pyogenes Cas9(최종 농도 90 nM) 및 1.5 μL의 gRNA(최종 농도 90 nM)를 혼합하여 총 11 μL의 분할 마스터 믹스 부피를 만듭니다.
- 분할 마스터 믹스를 RT에서 10분 동안 유지하여 Cas9:gRNA RNP complex를 형성합니다.
- 7.8.3단계에서 Plasmid-Safe DNase 처리된 DNA 125ng을 최종 부피 39μL까지 희석합니다. 그런 다음 11 μL의 Cleavage Master Mix를 39 μL Plasmid-Safe DNase 처리된 DNA에 총 50 μL의 부피로 추가합니다.
참고: 이 단계에서는 Cas9:gRNA 복합체 없이 Cas9 완충액과 혼합된 원형 DNA로 구성된 음성 대조군 검체를 포함합니다. - 37 °C에서 1 시간 동안 thermocycler에서 배양합니다. 무기한 4°C를 유지하십시오. in vitro-cleaved DNA에 50 μL(1x 부피)의 XP bead를 추가하고 4.3-4.6 단계에 따라 DNA를 정제합니다. 42μL의 TE 완충액, pH 8.0에서 용리합니다. 다음 단계를 위해 비드가 용액에 남아 있는지 확인하십시오.
9. 차세대 염기서열분석 라이브러리 준비(4 - 6시간)
- A-테일링을 수행합니다. A-테일링 마스터 믹스를 준비합니다.
- 5μL의 10x A-테일링 버퍼(최종 농도 1x)와 3μL의 A-테일링 효소(A-테일링 마스터 믹스의 총 부피는 8μL)를 혼합합니다.
- 8.4단계에서 비드를 함유한 용출된 DNA 샘플 42μL에 A-tailing Master Mix의 8μL를 피펫팅합니다(총 부피 50μL). 30°C에서 30분 동안 열순환기에 넣습니다. 4 °C에서 무기한 유지하십시오.
- 1.8배 부피의 PEG/NaCl SPRI 용액 또는 90μL를 A-tailed DNA에 피펫팅하고 4.3-4.6단계에 따라 DNA를 정제합니다. 25μL의 TE, pH 8.0에서 용리됩니다. 다음 단계를 위해 비드를 용액에 보관하십시오.
- 어댑터 접합을 수행합니다. 어댑터 결찰 마스터 믹스를 준비합니다.
참고: 3' T'의 동결-해동 유도 가수분해로 인한 어댑터 이량체의 형성을 방지하기 위해 NEB 어댑터의 일회용 부분 표본을 준비해야 합니다.- 10 μL의 5x Ligation Buffer(최종 농도 1x), 5 μL의 DNA Ligase 및 10 μL의 Adapter(최종 농도는 3 μM)를 혼합하여 총 25 μL를 만듭니다.
- 9.1.3 단계의 비드를 포함하는 25 μL의 A-tailed DNA 샘플에 어댑터 결찰 마스터 믹스 25μL를 피펫팅합니다. 1°C에서 20시간 동안 열순환기에 넣습니다. 4 °C에서 무기한 유지하십시오.
- 어댑터 결찰 DNA에 1배 부피 또는 50μL의 PEG/NaCl SPRI 용액을 피펫팅하고 4.3-4.6단계에 따라 DNA를 정제합니다. 47μL의 TE, pH 8.0에서 용리합니다. 다음 효소 단계를 위해 비드가 용액에 남아 있는지 확인하십시오.
- USER Enzyme으로 치료를 수행합니다(우라실 잔기에 단일 뉴클레오티드 갭 생성).
- 9.2.3단계의 비드가 포함된 어댑터 결찰 DNA 검체에 3μL의 USER 효소(Dual Index Primers Kit에 포함)를 추가합니다. 37°C에서 15분 동안 배양합니다.
- USER 효소 처리 DNA에 35μL(0.7배 용량)의 PEG/NaCl SPRI 용액을 추가하고 4.3-4.6단계에 따라 정제합니다. 20μL의 TE 완충액, pH 8.0에서 용리합니다. 상층액을 새로운 semi-skirted PCR 플레이트로 옮기고 dsDNA HS assay를 사용하여 DNA 농도를 측정합니다. 예상 농도는 약 2-5ng/μL여야 합니다.
- (선택 사항) 다음 단계로 진행하기 전에 PippinHT를 사용하여 DNA 크기를 선택할 수 있습니다. 250-850 bp 크기 범위의 1.5% PippinHT 카세트를 사용하십시오. 생성된 샘플은 다음 단계에서 PCR에서 직접 활용할 수 있습니다.
- 바코드 추가를 위한 PCR 수행
참고: 각 샘플에 대해 선택한 프라이머 염기서열 조합이 고유한지 확인하십시오. 가능한 경우 각 샘플에는 고유한 i5 및 i7 바코드가 있어야 합니다.- PCR Master Mix를 준비하여 paired-index 바코드를 추가합니다. 5 μL의 뉴클레아제가 없는 H2O, 2x 핫스타트 레디 믹스 25 μL(최종 농도 1x), 5 μL의 i5 프라이머(최종 농도 1 μM) 및 5 μL의 i7 프라이머(최종 농도 1 μM)를 혼합합니다(총 마스터 믹스 부피 40 μL).
- PCR Master Mix 40 μL를 9.3.2 단계(총 부피 50 μL)에서 USER 효소(약 20 ng)로 처리한 10 μL의 정제된 DNA에 피펫팅합니다.
- 다음 PCR 열순환 조건을 선택하십시오 : 변성 : 98 ° C에서 45 사이클 동안 1 초, 변성 : 98 ° C에서 15 초 동안 20 사이클, 어닐링 : 65 ° C에서 30 초 동안 20 사이클, 확장 : 72 ° C에서 30 초 동안 20 사이클, 최종 확장 : 72 ° C에서 1 사이클 동안 1 분, 유지 : 4 ° C를 무기한.
- PCR 산물에 0.7x 부피 또는 35 μL의 XP 비드를 추가하고 4.3-4.6 단계에 따라 정제합니다. 30μL의 TE, pH 8.0에서 용리합니다. 상층액을 새로운 semi-skirted PCR 플레이트에 디캔팅합니다. 필요한 경우 원형 DNA를 -20°C에서 최대 몇 개월 동안 보관합니다.
참고: Tapestation에서 PCR 샘플을 실행하여 라이브러리의 품질을 제어하고 어댑터 이량체의 형성을 평가합니다. 어댑터 이량체가 감지되면 9.5.4단계를 반복합니다.
10. 액적 디지털 PCR(dd_PCR)에 의한 CIRCLE-seq 라이브러리의 정량화(6시간)
참고: qPCR, Tapestation 또는 이와 유사한 방법을 사용하여 정량화를 수행할 수도 있습니다.
- 45 μL의 뉴클레아제-프리 TE와 잘 혼합된 라이브러리의 DNA(PCR step 9.5.4)로 시작한 다음, 10-1에서 10-8 희석 범위의 50 μL 부피에서 각 샘플을 1:10 연속 희석합니다.
- dd_PCR Master Mix 스톡 솔루션을 설정합니다. 프로브(최종 농도 1x), 프로브 oSQT1310 0.055μL(최종 농도 250nM), 프로브 oSQT1311 0.055μL(최종 농도 250nM), 프라이머 oSQT1274 0.099μL(최종 농도 450nM), 프라이머 oSQT1275 0.099μL(최종 농도 450nM) 및 뉴클레아제가 없는 H2O6.292μL를 dd_PCR혼합합니다. 총 dd_PCR 마스터 믹스 부피는 17.6μL입니다. 정확한 피펫팅을 위해 부피가 충분한지 확인하기 위해 모든 샘플에 대한 마스터 믹스를 준비합니다.
- 가장 낮은 희석액 3개(10-6,10-7 및 10-8)를 복제(96웰 플레이트에서)로 분석합니다. NTC(비 템플릿 컨트롤)를 사용해야 합니다.
- 다음과 같이 각 검체에 17.6 μL의 dd_PCR Master Mix를 피펫팅합니다: 17.6 μL를 시료 4.4 μL와 혼합하여(NTC를 사용하여 웰에 뉴클레아제가 없는 H2O 추가), 총 부피 22 μL를 만듭니다. 플레이트를 밀봉한 다음 RT에서 1분 동안 2000 x g 에서 원심분리합니다.
- 액적 생성, 열순환 및 분석을 수행합니다. Droplet Reader PCR 시스템을 사용하여 DG8 카트리지(8-well)를 카트리지 홀더에 넣습니다. 카트리지의 오일 행에 프로브용 액적 생성 오일 70μL를 분배합니다.
참고: 10.1.2단계에서 20μL의 샘플을 채취합니다. 8웰 카트리지의 샘플 행에 추가하고 카트리지를 DG8 고무 개스킷으로 덮고 액적 발생기에 넣은 다음 닫아 프로세스(자동)를 시작합니다. 완료되면 카트리지를 제거하고 8-well 카트리지의 액적 행에서 40 μL를 semi-skirted 96-well PCR 플레이트로 이동시켜 천천히 피펫팅합니다.- 열 블록을 PX1 PCR 플레이트 씰러에 놓습니다. 실러를 켜면 실러가 180°C까지 가열되기 시작합니다. 접시에 호일 히트 씰을 놓고 빨간색 선이 맨 위에 있는지 확인합니다. 플레이트를 PX1에 놓고 씰을 누릅니다.
- 다음 Thermocycler 조건을 선택하십시오 : 효소 활성화 : 95 사이클 동안 10 분 동안 1 °C, 변성 : 94 사이클 동안 30 초 동안 40 °C, 어닐링 / 확장 : 60 ° C 40 사이클 동안 1 분 동안, 효소 비활성화 : 98 ° C 1 사이클 동안 10 분 동안, 유지 : 4 ° C 무기한.
- 액적 판독기에서 호환 가능한 소프트웨어를 열고 읽을 웰을 선택합니다. 실험 유형으로 ABS 를 선택하고 프로브에 대해 Supermix를 dd_PCR . Target 1에 대해 Ch1 Unknown 을 선택하고 Target 2에 대해 Ch2 Unknown 을 선택합니다. 적용을 선택한 다음, 확인을 선택합니다. 플레이트를 액적 판독기에 넣습니다. 염료 세트의 경우 FAM/HEX 를 선택하고 실행을 클릭합니다.
- dd_PCR 결과를 분석합니다. negative control을 참조로 사용하여 double positive droplet population을 게이트합니다. 중복된 값의 평균을 계산한 다음 dd_PCR의 희석 계수와 5배 희석 계수를 곱합니다.
참고: 마이크로리터당 총 복제 수는 다음과 같이 계산됩니다: 마이크로리터당 총 복제 수 = 평균값 × 5 × 희석 계수, 여기서 '평균값'은 Ch1 및 Ch2의 평균 정량화 값을 나타냅니다. - 모든 시료를 등몰 농도의 단일 라이브러리로 결합합니다. 1x pooled library는 약 4.5 x 109 분자를 포함해야 하며 총 부피는 5μL여야 합니다.
11. 차세대 염기서열분석
- 염기서열분석을 위해 샘플을 외부 기관에 제출하여 올바른 어댑터 염기서열이 기록되었는지 확인합니다.
12. CIRCLE-seq 데이터 분석 (1 - 3 시간)
- Python 버전 2.7, BWA(Burrows-Wheeler Aligner) 및 SAMtools를 설치합니다. http://hgdownload.cse.ucsc.edu/goldenPath/hg38/bigZips/hg38.fa.gz 에서 참조 게놈(예: hg38)을 다운로드합니다.
참고: 타겟 종의 게놈을 사용할 수 없는 경우 CIRCLE-seq 계산 워크플로우를 참조 독립적 모드에서 실행할 수 있습니다. 이 경우 이 단계를 건너뛸 수 있습니다. - (1) git clone https://github.com/tsailabSJ/circleseq.git, (2) cd circleseq, (3) pip install -r requirements.txt 명령을 사용하여 CIRCLE-seq 파이프라인을 다운로드하고 설치합니다.
- YAML 형식(.yaml)으로 매니페스트 파일을 생성합니다. 다음은 워크플로를 테스트하기 위해 CIRCLE-seq 소프트웨어에 제공된 예제 데이터 세트와 함께 사용할 수 있는 샘플 매니페스트입니다.
참고: (1) 참조 게놈: data/input/CIRCLEseq_test_genome.fa; (2) analysis_folder: 데이터/출력; (3) BWA: BWA; (4) samtools: 샘툴즈; (5) read_threshold : <가치>; (6) window_size : <가치>; (7) mapq_threshold : <가치>; (8) start_threshold : <가치>; (9) gap_threshold : <가치>; (10) mismatch_threshold : <가치>; (11) merged_analysis: 참; (12) 샘플 : U2OS_EMX1; (13) 대상: GAGTCCGAGCAGAAGAAGAANGG; (14) read1 : 데이터 / 입력 / EMX1.r1.fastq.gz; (15) read2 : 데이터 / 입력 / EMX1.r2.fastq.gz; (16) ControlRead1 : 데이터 / 입력 / EMX1_control.r1.fastq.gz; (17) ControlRead2 : 데이터 / 입력 / EMX1_control.r2.fastq.gz; (18) 설명 : U2OS. 다음 매니페스트 값이 사용되었습니다: read_threshold: 4, window_size: 3, mapq_threshold: 50, start_threshold: 1, gap_threshold: 3, mismatch_threshold: 6 - 참조 게놈 FASTA 파일, 분석을 위한 출력 디렉터리, BWA 및 SAMtools 명령에 대한 경로를 정의합니다. nuclease-cleaved 샘플과 control 샘플 모두에 대해 타겟 염기서열과 역다중화된 FASTQ 파일의 경로를 정의합니다. 여러 실험을 단일 매니페스트 파일에 모두 포함하여 배치 모드에서 동시에 처리할 수 있습니다.
- 표준 참조 기반 분석을 위해 (1) python /path/to/circleseq.py all - manifest; (2) /path/to/manifest.yaml 명령을 실행합니다.
- 또는 표준 비참조 기반 분석을 위해 다음 명령을 실행합니다: (1) Python /path/to/circleseq.py reference-free - manifest; (2) /path/to/manifest.yaml입니다.
- 전체 파이프라인을 실행할 때 해당 특정 단계에 대해 지정된 고유한 output_folder 에서 각 단계의 출력 결과를 찾습니다.
결과
여기서, CIRCLE-seq는 유도만능줄기세포(iPSC)에서 분리된 DNA를 사용하여 아데노연관 바이러스 통합 부위 1(AAVS1)을 표적으로 하도록 설계된 gRNA가 있는 복합체에서 Cas9의 뉴클레아제 유도 절단 부위를 조사하는 데 사용됩니다. 이 gRNA는 이전에 본 출판물27에 설명되어 있습니다. 약 25μg의 gDNA를 iPSC에서 분리하고, 집속 초음파를 통해 전단하고, AMPure XP 비드 정제를 사용하여 크기를 선택하여 약 300bps의 단편을 생성했습니다. 이 25 μg의 DNA에서 약 2-5 ng의 DNA가 in vitro Cas9:gRNA 절단을 위해 성공적으로 순환되었습니다. 전체 절차는 그림 1에 나와 있습니다.
CIRCLE-seq 절차 및 계산 워크플로를 사용한 분석에 따라 감지된 모든 온-타겟 및 오프-타겟 분할 부위의 시각화가 그림 2A에 나와 있습니다. CIRCLE-seq 파이프라인은 또한 R 통계 소프트웨어를 통해 분석된 '병합 판독'을 제공하여 각 염색체를 따라 매핑된 검출된 뉴클레아제 유도 절단 부위를 보여주는 맨해튼 플롯을 생성했습니다(그림 2B).
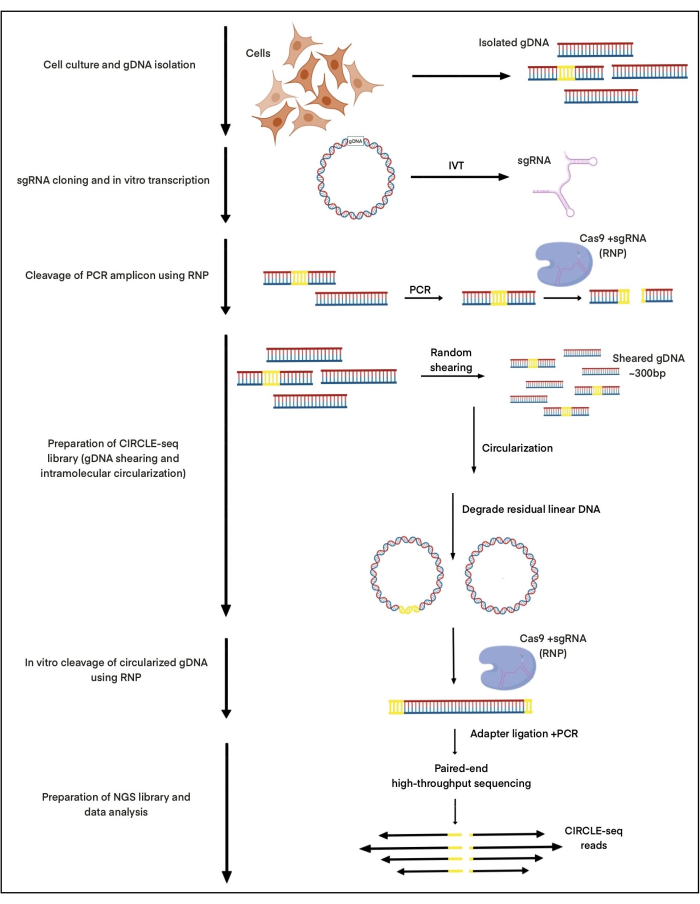
그림 1: CIRCLE-seq 워크플로우 회로도. 프로토콜의 주요 단계가 표시되어 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
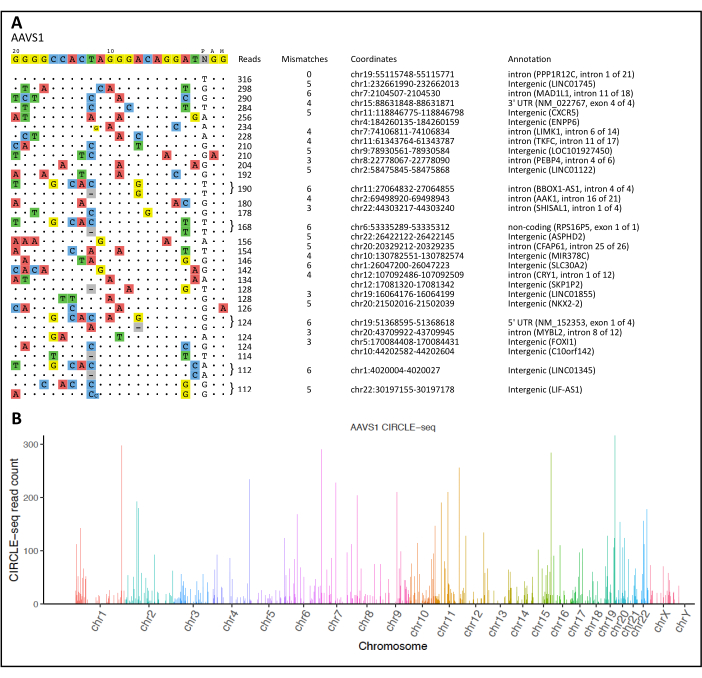
그림 2: CIRCLE-seq 시각화 및 맨해튼 플롯. (A) AAVS1 유전자 자리에 대한 의도된 표적에 대한 비표적 부위의 정렬. 대상 시퀀스는 맨 위에 표시되며, 여기서 off-target은 읽기 횟수에 따라 내림차순으로 순위가 매겨집니다. 원래 타겟 염기서열의 차이는 착색된 뉴클레오티드로 표시됩니다. AAVS1 유전자 자리에 대한 상위 의도하지 않은 절단 부위의 샘플이 표시됩니다. (B) AAVS1 유전자 자리에 대해 감지된 의도하지 않은 절단 부위를 보여주는 맨해튼 플롯. 막대 높이는 각 염색체 위치에 대한 판독 횟수를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 입문서 | 시퀀스 (5'-3') | 코멘트/설명 |
| AAVS1 단일 가이드 RNA (sgRNA) | GGGGCCACUAGGGACAGGAU | AAVS1 Locus의 형광 단백질 knock-in용 |
| AAVS1 포워드 프라이머 | GCTCTGGGCGGAGGAATG | gRNA in vitro cleavage 테스트용 |
| AAVS1 리버스 프라이머 | 아트씨씨그트그 | gRNA in vitro cleavage 테스트용 |
| 오SQT1288 | /5Phos/CGGTGGACCGATGATC /ideoxyU/ATCGGTCCACCGaT | CIRCLE-seq 헤어핀 어댑터 |
| 오SQT1274 | 아아가타크가그카카가그 | 트루시크 F1 |
| 오SQT1275 | CAAGCAGAAGACGGCATACGAGAT | 트루시크F2 |
| 오SQT1310 | /56-FAM/CCTACACGA/ZEN/CGCTCTTCCGATCT/3IABkFQ/ | TruSeq 프로브 |
| 오SQT1311 | /5HEX/TCGGAAGAG/ZEN/CACACGTCTGAACT/3IABkFQ/ | TruSeq 프로브 |
표 1: AAVS1 유전자 자리의 CIRCLE-seq 분석에 사용된 gRNA 및 primer의 염기서열.
토론
여기서, CIRCLE-seq는 iPSC에서 유래한 gDNA의 AAVS1 유전자 좌위를 표적으로 하여 게놈 전체에서 뉴클레아제 유도 DSB를 식별하기 위한 편향되지 않은 매우 민감한 기술임이 입증되었습니다. iPSC 내의 AAVS1 부위는 CRISPR-Cas928을 사용하여 외인성 유전자의 통합 부위로 자주 사용되는 세이프 하버 자리(safe harbor locus)로 잘 알려져 있습니다. 당사의 최근 보고서에서는 세포의 계통27 전반에 걸쳐 EGFP의 지속성으로 인해 iPSC와 분화된 iPSC의 라벨링 및 추적이 가능한 AAVS1 부위에 구성적으로 발현된 EGFP 리포터의 CRISPR 매개 통합을 수행하여 EGFP 표지 iPSC의 잠재력을 연구했습니다. 이 iPSC 라인은 이식 후 iPSC 유래 세포의 유기체 분포를 평가하기 위해 in vivo에 사용할 수 있습니다. 이 세포주는 CRISPR에 변형되어 iPSC의 임상 적용을 테스트하는 데에도 사용되고 있기 때문에 안전성과 효능을 보장하기 위해 잠재적인 AAVS1 off-target 부위를 파악하고 조사하여 CIRCLE-seq를 테스트하기에 이상적인 유전자 자리가 되어야 합니다.
이전에 발표된 연구27과 본 연구의 주목할 만한 차이점은 AAVS1 유전자 자리를 표적으로 하기 위해 변형된 gRNA를 사용했다는 것입니다. gRNA는 게놈 편집 정확도를 향상시키도록 설계될 수 있다24. 가이드 시퀀스는 온-타겟 및 오프-타겟 효율성에 영향을 미치는 가장 중요한 요소입니다. 따라서 선택한 gRNA는 여러 다른 가이드와 대조하여 테스트되었으며 우수한 충실도를 갖는 것으로 나타났습니다. 또한, 인간 섬유아세포를 재프로그래밍하는 방법에 관한 논문은 변형된 핵염기를 포함하는 합성 캡이 있는 mRNA가 항바이러스 반응의 낮은 활성화로부터 이익을 얻는다는 발견을 입증했습니다29,30. 낮은 면역원성은 체외 분석과 관련이 없을 수 있지만, 살아있는 세포에서 사용할 수 있는 임상적으로 관련된 치료법을 개발하는 것이 궁극적인 목표인 경우 매우 중요합니다.
CIRCLE-seq는 유사한 방법에 비해 많은 장점이 있습니다. 예를 들어, Digenome-seq 염기서열분석은 ~4억 개의 read를 사용하여 nuclease-cleaved gDNA와 unleaved gDNA를 모두 염기서열분석합니다21. 이로 인해 배경이 높아져 저주파의 선의의 절단 부위를 필터링하기가 어렵습니다. CIRCLE-seq는 nuclease-cleaved gDNA의 농축으로 인해 ~500만 번의 read만 사용하여 background가 낮습니다. 또한 Digenome-seq 및 유사한 방법인 SITE-seq는 단일 nuclease-cleaved DNA 말단의 염기서열분석에 의존합니다. 대조적으로, CIRCLE-seq 판독은 절단 부위의 양쪽 끝을 포함하므로 참조 21,22,26 없이 off-target 부위를 식별할 수 있습니다.
CIRCLE-seq의 장점은 GUIDE-seq와 같은 세포 배양에 의존하는 방법에 비해 감도가 더 높다는 것입니다. 두 가지 방법을 비교했을 때, CIRCLE-seq는 GUIDE-seq에 의해 감지된 모든 off-target 부위를 캡처할 수 있었고 GUIDE-seq가 놓친 의도하지 않은 추가 절단 부위를 발견할 수 있었습니다. 그러나 눈에 띄는 차이점은 GUIDE-seq는 후성유전학적 환경에 의해 방해를 받을 수 있는 반면 CIRCLE-seq는 전체 게놈에 액세스할 수 있다는 것입니다.
시험관 내 분석으로서 CIRCLE-seq는 몇 가지 제한 사항을 제시하며, 그 중 첫 번째는 위양성 검출입니다. 후성 유전학은 생체 내 특정 부위에서 뉴클레아제 활성을 방해하는 반면, 초음파는 체외에서 이러한 장애물을 제거하여 세포 맥락에서 일반적으로 접근할 수 없는 위치에서 표적을 벗어난 활동을 가능하게 합니다. 또한, Cas9는 이 in vitro 분석에서 고농도로 존재하여 in vivo 분석이 불가능한 절단을 가능하게 합니다. 이 분석은 또한 상대적으로 많은 양의 시작 gDNA를 필요로 하며, 이는 사용 가능한 자원에 따라 이 프로토콜의 사용을 무효화할 수 있습니다. 마지막으로, 일부 off-target 부위는 현재 차세대 염기서열분석 기술의 한계로 인해 검출되지 않을 수 있습니다.
최근 연구에서는 인실리코(in silico ) 접근법을 사용했으며, 이 접근법의 알고리즘은 CIRCLE-seq 및 GUIDE-seq31을 포함한 다양한 뉴클레아제 특성화 방법을 비교할 수 있는 여러 관련 매개변수를 식별했습니다. 관련 매개변수 중 두 가지는 '절단 부위 보강'과 '% 거짓 긍정'이었습니다. 흥미롭게도 CIRCLE-seq의 위양성률은 88%로 계산되었지만, 절단 부위 농축은 다른 in vitro 방법보다 더 높았습니다. 모든 방법을 비교 분석한 결과, GUIDE-seq가 중간 정도의 위양성률(false-positive rate)로 가장 큰 표적 특이도를 보였기 때문에 가장 우수한 성능을 보였다32. 이는 CIRCLE-seq를 무효화하는 것이 아니라 CIRCLE-seq와 GUIDE-seq를 동시에 활용할 수 있는 가능성을 암시하며, 전자는 감도가 더 높은 반면 후자는 절단 부위 농축이 높은 세포 기반 방법이기 때문에 GUIDE-seq와 함께 CIRCLE-seq의 결과를 검증합니다. 또한 데이터는 앰플리콘 기반 차세대 염기서열분석(NGS)이 잠재적인 후보 부위에서 진정한 off-target modification을 식별하기 위해 선호되는 방법이어야 함을 나타냅니다31. 이 데이터는 CIRCLE-seq, GUIDE-seq, 앰플리콘 기반 NGS를 차례로 사용하여 off-target 효과를 조사하는 잠재적 전략을 제안합니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
미국 국립보건원(National Institutes of Health, R01AR078551 및 T32AR007411), 오스트리아 수포성 표피박리증 연구 협회(Dystrophic Epidermolysis Bullosa Research Association, DEBRA), 게이츠 그럽스테이크 기금(Gates Grubstake Fund) 및 게이츠 프론티어 펀드(Gates Frontiers Fund)가 제공한 자금 지원에 깊은 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.2-mL Thin-walled Tubes and Flat Caps | ThermoFisher Scientific | AB1114 | |
| 1.5% PippinHT cassette | Sage Science | HTC1510 | |
| 10 mL Serological Pipettes | FisherScientific | 12567603 | |
| 15 mL Conical Tube | FisherScientific | 339651 | |
| 150 x 25 mm Tissue Culture Dish | FisherScientific | 877224 | |
| 25 mL Reagent Reservoir | FisherScientific | 2138127C | |
| 2x Kapa KiFi HotStart Ready Mix | Kapa Biosystems | KK2602 | |
| 5 mL Serological Pipettes | FisherScientific | 170355 | |
| 50 mL Conical Tube | FisherScientific | 339653 | |
| 50x TAE Electrophoresis Buffer | ThermoFisher Scientific | B49 | |
| 6 Well Cell Culture Plate | Corning | 3516 | |
| Agencourt AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | |
| Benchtop Microcentrifuge | Eppendorf | 5400002 | |
| BsaI-HF | New England BioLabs | ||
| Buffer QX1 | Qiagen | 20912 | |
| Cas9 nuclease, Streptococcus Pyogenes | New England BioLabs | M0386M | |
| CIRCLE-seq Library Preparation and NGS | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| ddPCR SuperMix for Probes | Bio-Rad | 1863010 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges | Bio-Rad | 1864008 | |
| DG8 Gaskets | Bio-Rad | 1863009 | |
| DPBS, no calcium, no magnesium | ThermoFisher Scientific | 14190144 | Made by Invitrogen |
| Droplet Generation Oil for Probes | Bio-Rad | 1863005 | |
| Droplet Reader Oil | Bio-Rad | 1863004 | |
| EDTA (0.5 M) | ThermoFisher Scientific | 15575020 | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher Scientific | AM9260G | Made by Invitrogen |
| Eppendorf ThermoMixer | Eppendorf | 5384000020 | |
| Equipment | |||
| Ethyl Alcohol, Pure | Sigma-Aldrich | E7023 | |
| Exonuclease I | New England BioLabs | M0293L | E. coli |
| Filter Unit | FisherScientific | FB0875713 | |
| Filtered Sterile Pipette Tips | |||
| Focused Ultrasonicator | Covaris | ME220 | |
| Genomic DNA Isolation | |||
| Genomic DNA Shearing | |||
| Gentra Puregene Cell Core Kit | Qiagen | 158043 | |
| gRNAs | Synthego | ||
| HCl | ThermoFisher Scientific | A144500 | |
| Heracell VIOS Tri-gas Humidified Tissue Culture Incubator | ThermoFisher Scientific | 51030411 | Need for culturing and expanding iPSCs (37 °C/5% CO2/5% O2) |
| High Sensitivity D1000 DNA ScreenTape | Agilent | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent | 5067-5585 | |
| HTP Library Preparation Kit | Kapa Biosystems | KK8235 | |
| HyClone Antibiotic Antimycotic Solution | Cytiva | SV30079.01 | |
| IDTE pH 8.0 (1x TE Solution) | Integrated DNA Technologies | 11050204 | |
| Inverted Microscope | Need for imaging iPS colonies in bright-field and fluorescent channels | ||
| iPSC Culture | |||
| Isopropanol | Sigma-Aldrich | 190764 | |
| Lambda Exonuclease | New England BioLabs | M0262L | |
| Loading Tips, 10 pack | Agilent | 5067-5599 | |
| Magnum FLX Enhanced Universal Magnet Plate | Alpaqua | A00400 | |
| Microcentrifuge Tube | Axygen | 31104051 | |
| Microtube AFA Fiber Pre-Slit Snap-Cap | Covaris | 520045 | |
| mTeSR-1 5x Supplement | StemCell Technology | 85852 | |
| mTeSR-1 Basal Medium (400 mL) | StemCell Technology | 85851 | Media for maintaining iPSC in culture |
| Nanodrop 8000 Spectrophotometer | ThermoFisher Scientific | ND-8000-GL | |
| NEBNext Multiplex Oligos for Illumina | New England BioLabs | E7600S | Dual Index Primers Set 1 |
| Optical tube strip caps, 8x strip | Agilent | 401425 | |
| Optical tube strips, 8x strip | Agilent | 401428 | |
| Other Reagents | |||
| PCR Plate Sealer | Bio-Rad | PX1 | Model Number PX1 |
| PEG/NaCl SPRI Solution | Kapa Biosystems | ||
| Phusion Hot Start Flex 2x Master Mix | New England BioLabs | M0536L | |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | |
| Plasmid-Safe ATP-dependent DNase | Epicentre | E3110K | |
| Primers, Adapters and Probes | IDT | Sequences are listed in Table 1 | |
| Proteinase K | Qiagen | 19131 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAxcel Gel Analysis System | Qiagen | 9001941 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32853 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32854 | |
| Qubit Fluorometer | ThermoFisher Scientific | Q33226 | |
| QX200 Droplet Digital PCR System | Bio-Rad | 1864001 | Contain a QX200 droplet generator and a QX200 droplet reader |
| RNase A | Qiagen | 19101 | |
| Semi-skirted PCR Plate | ThermoFisher Scientific | 14230244 | |
| SeqPlaque GTG Agarose | Lonza | 50110 | |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| T4 DNA Ligase | New England BioLabs | M0202L | |
| T4 Polynucleotide Kinase (PNK) | New England BioLabs | M0201L | |
| T-75 Flasks | FisherScientific | 7202000 | |
| Tapestation 4150 | Agilent | G2992AA | |
| Thermocycler with programmable temperature-stepping functionality | Bio-Rad C1000 Touch | ||
| Tris base | ThermoFisher Scientific | BP1521 | |
| Twin.tec PCR Plate | Eppendorf | E951020346 | 96 wells, semi-skirted, green |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977015 | |
| USER Enzyme | New England BioLabs | M5505L |
참고문헌
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Frangoul, H., et al. CRISPR-Cas9 gene editing for sickle cell disease and beta-thalassemia. N Engl J Med. 384 (3), 252-260 (2021).
- Ledford, H. CRISPR treatment inserted directly into the body for first time. Nature. 579 (7798), 185 (2020).
- Gillmore, J. D., et al. CRISPR-Cas9 in vivo gene editing for transthyretin amyloidosis. N Engl J Med. 385 (6), 493-502 (2021).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31 (9), 822-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Anzalone, A. V., et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature. 576 (7785), 149-157 (2019).
- Hong, S. A., et al. Therapeutic base editing and prime editing of COL7A1 mutations in recessive dystrophic epidermolysis bullosa. Mol Ther. 30 (8), 2664-2679 (2022).
- Zhang, G., et al. Enhancement of prime editing via xrRNA motif-joined pegRNA. Nat Commun. 13 (1), 1856 (2022).
- Lee, J., et al. Prime editing with genuine Cas9 nickases minimizes unwanted indels. Nat Commun. 14 (1), 1786 (2023).
- Howe, S. J., et al. Insertional mutagenesis combined with acquired somatic mutations causes leukemogenesis following gene therapy of SCID-X1 patients. J Clin Invest. 118 (9), 3143-3150 (2008).
- Hacein-Bey-Abina, S., et al. Insertional oncogenesis in 4 patients after retrovirus-mediated gene therapy of SCID-X1. J Clin Invest. 118 (9), 3132-3142 (2008).
- Has, C., Kiritsi, D. Therapies for inherited skin fragility disorders. Exp Dermatol. 24 (5), 325-331 (2015).
- Murnane, J. P., Yezzi, M. J., Young, B. R. Recombination events during integration of transfected DNA into normal human cells. Nucleic Acids Res. 18 (9), 2733-2738 (1990).
- Hsieh, M. M., et al. Myelodysplastic syndrome unrelated to lentiviral vector in a patient treated with gene therapy for sickle cell disease. Blood Advances. 4 (9), 2058-2063 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol. 33 (2), 187-197 (2015).
- Wang, X., et al. Unbiased detection of off-target cleavage by CRISPR-Cas9 and TALENs using integrase-defective lentiviral vectors. Nat Biotechnol. 33 (2), 175-178 (2015).
- Lensing, S. V., et al. DSBCapture: In situ capture and sequencing of DNA breaks. Nat Methods. 13 (10), 855-857 (2016).
- Crosetto, N., et al. Nucleotide-resolution DNA double-strand break mapping by next-generation sequencing. Nat Methods. 10 (4), 361-365 (2013).
- Yan, W. X., et al. BLISS is a versatile and quantitative method for genome-wide profiling of DNA double-strand breaks. Nat Commun. 8, 15058 (2017).
- Kim, D., et al. Digenome-seq: Genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods. 12 (3), 237-243 (2015).
- Cameron, P., et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods. 14 (6), 600-606 (2017).
- Wienert, B., Wyman, S. K., Yeh, C. D., Conklin, B. R., Corn, J. E. CRISPR off-target detection with DISCOVER-seq. Nat Protoc. 15 (5), 1775-1799 (2020).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, 1143157 (2023).
- Conant, D., et al. Inference of CRISPR edits from Sanger trace data. CRISPR J. 5 (1), 123-130 (2022).
- Lazzarotto, C. R., et al. Defining CRISPR-Cas9 genome-wide nuclease activities with CIRCLE-seq. Nat Protoc. 13 (11), 2615-2642 (2018).
- Butterfield, K. T., McGrath, P. S., Han, C. M., Kogut, I., Bilousova, G. Generation of an induced pluripotent stem cell line with the constitutive EGFP reporter. Methods Mol Biol. 2155, 11-21 (2020).
- Hayashi, H., Kubo, Y., Izumida, M., Matsuyama, T. Efficient viral delivery of Cas9 into human safe harbor. Sci Rep. 10 (1), 21474 (2020).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nat Commun. 9 (1), 745 (2018).
- Kariko, K., Buckstein, M., Ni, H., Weissman, D. Suppression of RNA recognition by Toll-like receptors: The impact of nucleoside modification and the evolutionary origin of RNA. Immunity. 23 (2), 165-175 (2005).
- Bao, X. R., Pan, Y., Lee, C. M., Davis, T. H., Bao, G. Tools for experimental and computational analyses of off-target editing by programmable nucleases. Nat Protoc. 16 (1), 10-26 (2021).
- Cromer, M. K., et al. Comparative analysis of CRISPR off-target discovery tools following ex vivo editing of CD34(+) hematopoietic stem and progenitor cells. Mol Ther. 31 (4), 1074-1087 (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유