JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
SV40 기반 인간 에피솜 내에서 구조가 형성되기 쉬운 DNA 반복을 통한 복제의 전기영동 분석
요약
여기에서는 2차원 겔 전기영동을 사용하여 병원성, 구조가 발생하기 쉬운 반복을 통한 복제 진행을 분석하는 절차를 간략하게 설명합니다.
초록
2차원 중성/중성 겔 전기영동(2DGE)은 자연적 장애를 통해 DNA 복제를 분석하기 위한 벤치마크 기법으로 부상했습니다. 이 프로토콜은 인간 세포의 유인원 바이러스 40(SV40) 기반 에피솜 내에서 구조가 발생하기 쉽고 확장 가능한 DNA 반복을 통해 복제 포크 진행을 분석하는 방법을 설명합니다. 간단히 말해서, 플라스미드가 인간 세포로 transfection되면 복제 중간체는 modified Hirt 프로토콜에 의해 분리되고 DpnI 제한 효소로 처리되어 복제되지 않은 DNA를 제거합니다. 그런 다음 중간체를 적절한 제한 효소에 의해 절단하여 3-5kb 길이의 DNA 단편의 기원 원위 절반 내에 관심 반복을 배치합니다. 복제 중간체는 두 개의 수직 차원으로 분리되는데, 처음에는 크기에 의해, 그 다음에는 모양에 따라 분리됩니다. 서던 블롯 교잡에 이어 이 접근 방식을 통해 연구자들은 복제 Y-아크의 하강 절반에서 다양한 구조 형성 반복에서 포크가 멈추는 것을 관찰할 수 있습니다. 또한, 스톨 사이트의 이러한 포지셔닝은 포크 반전, 수렴 포크의 출현 및 재조합 포크 재시작과 같은 반복 매개 포크 실속의 다양한 결과를 시각화할 수 있습니다.
서문
STR(Short Tandem Repeats)은 일반적으로 2-9개의 염기쌍(bp)으로 작으며, 인간 게놈의 약 3%를 구성하는 DNA의 반복적인 염기서열입니다1. STR은 유전자 조절2에서 중요한 역할을 합니다. 그러나 그들의 반복적인 구성은 비표준 DNA 2차 구조 형성 및 그에 따른 유전적 불안정성을 유발합니다 3,4. 왼손잡이 나선에서 머리핀/십자형, 3가닥 및 4가닥 나선에 이르기까지, 이러한 대체 DNA 구조는 레플리솜에 본질적인 문제를 일으킵니다. 2차 구조 형성을 위한 자연스러운 전제 조건은 DNA 풀림이며, 이는 DNA 복제의 전제 조건입니다. 이는 게놈 기능에 대한 독특한 수수께끼를 제시하는데, 이러한 구조 중 많은 부분이 복제 중에 형성되어 replisome 진행을 방해하고 궁극적으로 복제 포크 정지 5,6,7 또는 심한 경우 포크 붕괴 및 DNA 파손 8,9을 유발할 수 있습니다. 중단된 포크(stalled fork)의 재시작과 DNA 복구 경로 모두는 반복 확장(repeat expansions)10,11 및 복잡한 게놈 재배열(complex genome rearrangements, CGR)12,13과 같은 반복적인 불안정성을 유발하는 것으로 나타났습니다. 이러한 사건은 임마누엘 증후군(Emmanuel syndrome)16과 같은 CGR 질환뿐만 아니라 Fragile X 증후군, Huntington's disease, Friedreich's ataxia 등을 포함하여 반복 확장 장애로 알려진 약 60개의 인간 질병의 발병을 초래할 수 있습니다. 따라서 반복적인 불안정성으로 인한 인간 질병의 메커니즘을 더 잘 이해하려면 이러한 반복을 통한 복제 포크 진행의 세부 사항을 연구하는 것이 필수적입니다.
복제 진행을 연구하는 기술은 1980년대 중반에 Brewer와 Fangman이 Saccharomyces cerevisiae 의 복제 시작이 자율 복제 서열(일반적으로 ARS로 알려짐) 요소17에서 발생한다는 직접적인 증거를 제공하려고 했을 때 등장했습니다. 이를 통해 그들은 아가로스에서 효모 복제 중간체의 구조를 분리하여 Bell and Byers의 초기 방법인 2차원 중성/중성 겔 전기영동(2DGE)18을 적용했습니다. 이 기술은 비선형 DNA가 아가로스 겔에서 동일한 질량의 선형 등가물보다 다르게 이동한다는 사실을 활용했습니다. 좀 더 구체적으로 말하자면, 2DGE에서 분리된 DNA는 두 개의 수직 차원으로 분리되는데, 처음에는 주로 크기에 의해, 그 다음에는 주로 모양에 따라 분리되어 특정 관심 영역에서 포괄적인 복제 맵을 생성합니다. 그들의 원래 논문에서, Brewer와 Fangman은 이것을 "단순 Y" 구조 또는 복제되지 않은 DNA를 복제된 DNA와 복제된 DNA를 연결하는 복제 포크로 구성된 호로 보여주었습니다. 그들은 또한 관찰된 다른 중간체를 "거품"과 "이중 Y"로 설명하며, 이는 각각 복제 기원과 수렴 분기점을 나타냅니다.
2DGE는 주어진 시간에 DNA 복제 중간체의 상대적 집단을 연구하는 데 사용할 수 있습니다. 따라서 한 중간체 집단이 다른 집단보다 더 널리 퍼져 있으면 시각화 시 분명해집니다. 따라서 2DGE는 구조 형성 반복과 같은 까다로운 시퀀스를 통한 복제 진행을 연구하는 데 특히 유용한 도구입니다. 예를 들어, 분석된 영역에 복제 포크 정지를 유발할 수 있는 염기서열이 포함되어 있는 경우, 이는 아크에 돌출부로 나타나며(그림 1A), 이는 해당 궤적에 복제 포크가 누적되어 있음을 나타냅니다. 이것은 효모 19,20,21에서의 헤어핀 형성 반복 서열과 인간 세포 22,23,24에서의 삼중 형성 반복의 복제로 볼 수 있습니다. 스톨링 외에도 2DGE는 재조합 중간체(25)의 경우와 같이 복제 중에 형성된 표준 단순 Ys에 부합하지 않는 DNA 구조를 관찰하는 데 사용할 수 있습니다. 이러한 중간체는 더 무겁고 더 많은 분지된 X자형 구조를 가지고 있으므로 표준 복제 포크보다 1차원과 2차원 모두에서 더 느리게 이동합니다. 복제 포크 반전(replication fork reversal) 20,24,26에 대해서도 유사한 결과를 관찰할 수 있다. 강한 복제 스트레스에 대응하여 진핵 세포는 복제 포크 반전을 활용하여 중단된 포크를 구제하는 것으로 나타났습니다. 이 반전 된 포크는 스톨 포크와 비슷한 분자량을 갖습니다. 그러나 그들의 닭발 구조는 Y자형 보체에 비해 2차원에서 전기영동 이동성이 더 느려져 아크에서 위아래로 확장됩니다.
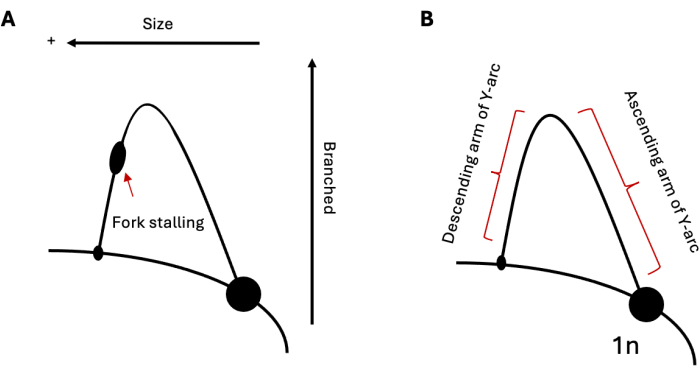
그림 1: DNA 복제의 2D 겔 전기영동 분석. (A) 포크 스톨링을 유발할 수 있는 구조 형성 반복을 통한 복제를 묘사하는 일반적인 2DGE의 개략도. 중간 크기와 구조는 전기영동 이동성에 영향을 미칩니다. (B) 각각 레이블이 지정된 오름차순 및 내림차순 암이 있는 샘플 Y-arc. 약어: 2DGE = 2차원 중성/중성 겔 전기영동. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
당연히 2DGE의 가장 중요한 측면 중 하나는 복제 중간체의 품질과 양에 관한 것입니다. 그러나 포유류 세포에서 내인성 유전자좌를 통한 복제에 대한 2DGE 분석의 해상도는 심하게 증폭된 DHFR 유전자좌27 또는 리보솜 RNA28과 같은 다중 복제 유전자에 대해 수행되었음에도 불구하고 6 × 109 bp 이배체 인간 게놈 내의 단일 복제 표적 염기서열에 대해 충분하지 않습니다. SV40 기반 복제는 진핵 세포에서 복제를 연구하는 효율적이고 잘 특성화된 수단이다29. 그것은 바이러스 게놈을 복제하기 위해 대부분의 숙주 replisome 기계를 사용하는 진핵 생물 복제의 신뢰할 수있는 모델을 제공하며, 이는 감염시 뉴클레오솜으로 포장됩니다30,31. 포유류 레플리솜의 두 가지 주목할 만한 예외는 숙주 CMG 복합체 대신에 T-항원(Tag)이 복제 DNA 헬리카제로 작용하고, DNA 중합효소 델타가 선행 및 후행 DNA 가닥을 모두 합성한다는 것이다(32). 우리는 원래 Massimo Lopes lab22에서 생성된 플라스미드 내에 SV40 복제 기원의 다운스트림에 구조 형성 반복의 병원성 스트레치를 배치하여 이 시스템을 활용했습니다. 중요한 것은 이 플라스미드는 Tag 자체에 대한 유전자 인코딩도 포함하고 있어 다양한 배양된 인간 세포에 transfection할 때 구성적이고 매우 강력한 복제를 초래한다는 것입니다. 이 특징은 인간 세포에서 병원성 반복의 복제 중 및 이에 대응하여 형성된 중간체의 2DGE 분석에 이상적인 많은 양의 제품을 생성합니다. 여기에서는 2차원 겔 전기영동을 사용하여 SV40 기반 인간 에피솜 내에서 구조 형성 반복의 복제를 시각화하는 자세한 방법을 설명합니다.
프로토콜
참고: 포유류 세포에서 간략하게 설명한 2DGE 분석을 위해 설계된 플라스미드에는 구조가 발생하기 쉬운 반복의 업스트림에서 수 kb 떨어진 SV40 복제 기원이 포함되어야 합니다(그림 2). 선행 및 후행 synthesis는 반복이 플라스미드에 클로닝되어야 하는 origin에 상대적인 방향을 선택할 때 염두에 두어야 합니다.

그림 2: 2DGE 분석을 위한 반복 함유 플라스미드의 분해. 구조가 발생하기 쉬운 반복은 오른쪽으로 이동하는 복제 포크에서 몇 kb 다운스트림으로 표시됩니다. 고유한 커터 1과 2를 사용한 분해는 분해된 단편의 중간 지점을 넘어서는 시퀀스가 주어지면 Y-아크의 하강하는 암에 반복 시퀀스를 배치합니다. 약어: 2DGE = 2차원 중성/중성 겔 전기영동. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 포유류 세포로의 플라스미드 transfection
- transfection하기 전에 10cm 조직 배양 플레이트에 600,000개의 HEK293T 세포를 파종합니다. 세포가 밤새 37°C에서 회복되도록 합니다.
참고: 이 실험에는 많은 세포주가 사용될 수 있지만 최적의 결과를 위해 SV40 태그를 포함하는 세포를 사용하는 것이 좋습니다. 주의: HEK293T 세포는 BSL-2로 간주되며 모든 배양 작업은 적절한 무균 기술과 적절한 PPE를 사용하여 생물 안전 캐비닛에서 수행해야 합니다. - 세포가 60%의 밀도에 도달하면 제조업체의 프로토콜에 따라 적절한 transfection 시약을 사용하여 8μg의 반복 함유 플라스미드 DNA를 파종된 세포에 transfection합니다.
- 이 시점에서 중간체를 분리하지 않는 경우 오래된 배지를 흡입하고 24시간 후에 10mL의 새 배지로 교체합니다.
- 형질주입 후 24-48시간 후에 세포 수집을 시작합니다.
- 배지를 흡입하고 10mL의 인산염 완충 식염수(PBS)로 조심스럽게 세척합니다. 0.5mL의 트립신을 사용하여 세포를 분리 및 수집하고 340× g 에서 4분 동안 스핀다운합니다.
- 상층액을 흡인하고 PBS로 세포 펠릿을 세척합니다. 340× g 에서 4분 동안 다시 스핀하고 상층액을 흡입합니다.
참고: 세포 펠릿을 -80°C에서 동결하여 실험을 일시 중지할 수 있습니다. 우리는 transfection 후 48시간 후에 복제 중간체를 분리하는 최상의 해결책을 찾았습니다. 그러나 24 H는 실행 가능한 결과를 산출했습니다.
2. 복제 중간체의 분리
- 50mL 코니컬 튜브에 1.5mL의 변형된 Hirt 용해 완충액[10mM tris-HCl(pH 7.5), 10mM 에틸렌디아민테트라아세트산(EDTA)]에 세포를 재현탁시키고 세포 용해를 시작합니다.
- 소듐 도데실 설페이트(SDS)를 최종 농도 0.6%(스톡 약 650μL, 2% SDS)로 추가하고 프로테나아제 K를 최종 농도 100μg/mL(스톡 약 10μL, 스톡 20mg/mL 프로테나아제 K)로 첨가하여 뉴클레아제를 제거합니다.
- 균질해질 때까지 피펫팅으로 부드럽게 혼합하고 혼합물을 37°C에서 최소 90분 동안 배양합니다.
- NaCl 농도를 1M(스톡 약 540μL, 5M NaCl)로 높이고 균질해질 때까지 부드럽게 혼합합니다. 4 °C에서 하룻밤(18-24시간) 배양하여 세포 파편, RNA 및 단백질이 염분을 제거하여 침전될 수 있도록 합니다.
알림: 혼합물은 점성이 높으므로 잘 혼합하는 동안 주의하고 인내심을 가지십시오. - 다음 날, 세포 파편, RNA 및 단백질에서 DNA를 분리합니다.
- 혼합물을 29,500× g 에서 4°C에서 45분 동안 원심분리합니다.
- DNA를 함유한 상층액을 옮기고 페놀:클로로포름:이소아밀 알코올 25:24:1(v/v) 한 부피를 넣고 균질해질 때까지 잠시 혼합합니다.
주의: 페놀:클로로포름:이소아밀 알코올은 유해 물질이므로 화학 흄 후드에서 적절한 PPE와 함께 취급해야 합니다. - 실온에서 15,000× g 에서 5분 동안 다시 원심분리합니다. 수성 층을 새로운 원추형 튜브로 옮깁니다.
- 분리된 DNA를 침전시키고 세척합니다.
- 한 부피의 순수 이소프로판올을 넣고 실온에서 최소 5분 동안 배양합니다. 15,000× g 에서 4°C에서 30분 동안 DNA를 스핀다운합니다.
- 상층액을 디캔팅하고 펠릿을 차가운 70% 에탄올로 세척하여 과도한 염분을 제거합니다.
- 15,000 × g 에서 4 ° C에서 30 분 동안 한 번 더 스핀하고 자연 건조 한 다음 펠릿을 Tris-EDTA (TE) 완충액 (10mM Tris-HCl, 1mM EDTA)에 부드럽게 재현탁시킵니다.
참고: 여기에서 실험을 일시 중지할 수 있으며 샘플은 -20°C에서 동결할 수 있습니다. 그러나 동결/해동 주기는 DNA 복제 중간체의 품질을 저하시킬 수 있으므로 피해야 합니다.
3. 시료 전처리 및 2차원 겔 전기영동
- plasmid replication의 분리된 중간체를 분해합니다.
- 적절한 제한 효소 100 단위를 샘플에 추가하여 플라스미드 DNA를 분해하고, 특히 반복 함유 서열을 선형 단편의 원위부 절반에 배치합니다(그림 2). 또한 DpnI를 첨가하여 메틸화된 DNA를 절단하여 배양된 인간 세포에서 완전히 복제되지 않은 플라스미드 DNA를 제거합니다.
참고: 최상의 결과를 위해, 제한 효소는 Y-아크의 descending arm에 구조가 발생하기 쉬운 sequence를 배치하는 3-5 kb fragment를 생성하는 고유한 커터여야 합니다. - 완전한 플라스미드 분해를 위해 37°C에서 6-10시간 동안 샘플을 배양합니다.
- 2.5 부피의 차가운 순수 에탄올로 DNA를 침전시키고 -20 °C에서 밤새 배양하거나 1 부피의 이소프로판올을 추가하고 실온에서 5분 동안 배양합니다.
- 분해 및 침전된 시료를 15,000× g 에서 4°C에서 30분 동안 원심분리합니다.
- 상층액을 디캔팅하고 샘플을 차가운 70% 에탄올로 세척합니다. 15,000× g 에서 4°C에서 30분 동안 다시 스핀합니다.
- 상층액을 디캔팅하고 10분 동안 자연 건조한 다음 15μL의 TE 완충액에 샘플을 재현탁시킵니다.
- 적절한 제한 효소 100 단위를 샘플에 추가하여 플라스미드 DNA를 분해하고, 특히 반복 함유 서열을 선형 단편의 원위부 절반에 배치합니다(그림 2). 또한 DpnI를 첨가하여 메틸화된 DNA를 절단하여 배양된 인간 세포에서 완전히 복제되지 않은 플라스미드 DNA를 제거합니다.
- 0.4x Tris-borate-EDTA(TBE) 0.4.5%(TBE) 1개(89mM 트리스 베이스, 89mM 붕산, 2mM EDTA)에서 1차원 아가로스 겔을 준비합니다. 용액이 최소 1시간 동안 응고되도록 합니다.
- 샘플을 첫 번째 차원으로 불러오기 시작합니다.
- 겔의 가장 왼쪽 가장자리를 기준으로 처음 3cm 이내에 사다리를 로드합니다. 그런 다음 준비된 샘플 전체를 로드하고 각 쌍 사이에 3cm를 확보합니다.
- 0.85V/cm에서 19-24시간 동안 1x TBE에 겔을 실행하여 크기에 따라 중간체를 분리합니다. DNA에 손상을 줄 수 있는 빛으로부터 샘플을 보호하기 위해 챔버가 덮여 있는지 확인하십시오.
- 다음 날, 완충액에서 겔을 제거하고 자를 사용하여 절단된 선형 단편의 위치를 추정합니다.
- 사다리가 들어있는 겔의 처음 3cm를 절제하고 0.3μg/mL 에티듐 브로마이드를 함유한 1x TBE에서 10-15분 동안 겔 세그먼트를 염색합니다. 겔 문서화 시스템을 사용하여 사다리를 시각화합니다.
- 추정된 위치에 1.3cm를 더하면 값 a가 생성됩니다. 그런 다음 값 a에서 7.5cm를 빼서 값 b를 산출합니다. 눈금자를 1차원 젤에 맞추고 값 a 와 b에서 가로로 자릅니다. 그런 다음 각 샘플에 대해 예약된 3cm 공간을 수직으로 자릅니다. 시각적 회로도는 그림 3 을 참조하십시오.
- 새 주조 트레이에서 세그먼트를 시계 방향으로 회전하여 시료 웰의 위치에 놓습니다(그림 3).
- 0.3μg/mL 브롬화 에티듐에서 1x TBE에 1-1.3% 농도로 2차원 아가로스 겔을 준비합니다.
- 약 55°C로 냉각한 후 회전된 1차원 세그먼트 위에 2차원 겔을 붓고 최소 1시간 동안 응고되도록 합니다.
- 2차원을 0.3μg/mL 에티듐 브로마이드에서 1x TBE가 있는 챔버로 옮기고 겔이 최소 30분 동안 평형을 이루도록 합니다.
- 4°C에서 4.23V/cm에서 9-10시간 동안 겔을 덮고 4°C에서 실행하여 모양에 대해 중간체를 분리합니다.
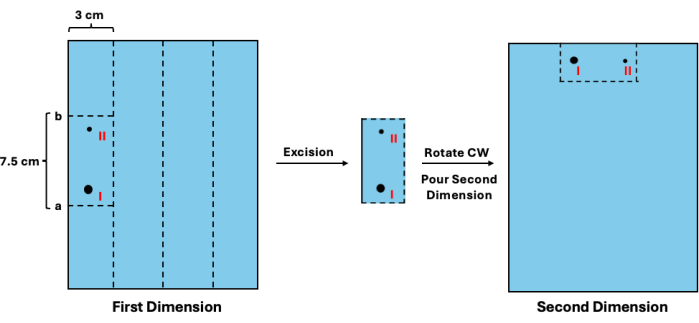
그림 3: 2차원 분리 전 1차원 중간체의 절제. 래더를 시각화한 후 복제되지 않은 단편의 이동성을 추정할 수 있습니다. (I) 그런 다음 이 값을 사용하여 적절한 절단 부위(a 및 b)를 결정하고 복제된 부위(II)를 절제할 수 있습니다. 그런 다음 겔 부분을 회전시켜 2차원 분리를 위해 웰 위치에 배치해야 합니다. 약어: CW = 시계 방향. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. radiolabeled 프로브를 사용한 Southern blotting 및 hybridization
- 챔버에서 2차원 겔을 제거하고 DNA 단편을 0.24M HCl 용액에서 부드럽게 흔들면서 10분 동안 탈피합니다. 젤을 탈이온수로 헹구고 0.4M NaOH에 10-15분 동안 담그십시오.
주의: HCl 및 NaOH는 부식성이므로 화학 흄 후드에서 적절한 PPE로 취급해야 합니다. - 겔에서 분리된 중간체가 멤브레인으로 쉽게 전달될 수 있도록 서던 블롯 조립을 시작합니다. 포괄적인 회로도는 그림 4 를 참조하십시오.
- 충분히 큰 용기에 1L의 0.4M NaOH를 채웁니다.
- 용기를 가로질러 긴 유리 시트를 정렬하고 유리 시트를 가로질러 수직으로 두 장의 긴 크로마토그래피 용지를 접어(길이를 따라) NaOH 용기로 확장합니다.
- 종이 상단을 NaOH로 적시고 표면 아래의 기포를 조심스럽게 제거합니다.
- 크로마토그래피 용지 3장에 NaOH를 적시고 접힌 종이 위에 놓고 기포를 다시 제거합니다.
- 2차원 젤을 거꾸로 뒤집어 종이 위에 옮깁니다.
- 양전하를 띤 나일론 멤브레인(0.45μm 기공 크기)을 DI 물로 적시고 젤 위에 놓습니다.
- 마지막으로 멤브레인 위에 DI 물에 적신 종이 3장을 더 추가합니다.
- 증발을 방지하기 위해 바닥 용기에 노출된 NaOH를 플라스틱 랩으로 덮습니다. 블롯 위에 냅킨이나 종이 타월 더미를 놓고 높이가 0.3-0.5m가 되도록 합니다. 상단에 무게를 놓고 전체 블롯을 압축하여 단단한 모세관 작용을 용이하게 합니다. DNA가 멤브레인으로 전달되는 데 최소 2일이 소요됩니다.
참고: 크로마토그래피 종이와 멤브레인의 길이와 너비는 2차원에 사용되는 아가로스 겔의 크기에 따라 다릅니다. 가장 효율적인 이송을 위해 겔과 동일한 치수의 종이와 멤브레인을 사용하십시오.
- 이식 후 120 μJ/cm2 에서 1분 동안 UV crosslinker를 사용하여 DNA를 멤브레인에 가교 결합합니다.
알림: 실온에서 멤브레인을 밀폐되고 건조하며 깨끗한 시트 프로텍터에 넣으면 여기에서 실험을 일시 중지할 수 있습니다. - 2x 식염수 구연산 나트륨 (SSC 완충액) (0.3M NaCl, 0.03M 구연산 나트륨)으로 멤브레인을 5 분 동안 2 회 세척합니다.
- 65°C에서 0.18mL/cm2 의 Church & Gilbert's hybridization buffer[1mM EDTA, 1% 소 혈청 알부민(BSA), 0.5M sodium phosphate, 7% SDS]로 멤브레인을 사전 교성화하고 혼성화 인큐베이터에서 최소 2시간 동안 회전합니다.
참고: 멤브레인은 며칠 동안 사전 교잡을 할 수 있습니다. - 제조업체의 프로토콜에 따라 α-32P dATP 또는 dCTP와 DNA 라벨링 키트를 사용하여 방사성 표지된 프로브를 준비합니다.
주의: 방사성 라벨이 부착된 dNTP는 위험하므로 취급 시 적절한 PPE를 착용해야 합니다. 모든 방사능 작업은 차폐 뒤에서 수행되어야 하며, 훈련된 개인은 선량계를 사용하여 방사선 흡수를 모니터링해야 합니다.- 제한 분해 염기서열(그림 2)을 보완하는 400-900bp 선형 DNA 단편을 설계하고 중합효소 연쇄 반응(PCR)을 사용하여 단편을 증폭합니다.
참고: 50-100 ng/μL의 스톡 PCR 단편을 보유하는 것이 좋습니다. - 100ng의 상보적 PCR 단편을 DNA Pol I, Klenow 단편(3' 5' exo-) 완충액 및 무작위 데카뉴클레오티드 올리고와 결합합니다.
- 100°C에서 10분 동안 단편을 변성시킵니다.
- DNA Pol I 5 단위, Klenow 단편(3' 5' exo-), 50 μCi의 α-32P dNTP, 방사성 표지된 dNTP 유형에 결핍된 30-50 μmol의 dNTP 혼합물을 샘플에 추가합니다.
- 37°C에서 10분 동안 배양하여 중합 및 방사성 표지된 dNTP 혼입을 가능하게 합니다.
- 이전에 결핍된 dNTP 유형 30-50 μmol을 샘플에 추가하고 37°C에서 10분 동안 배양합니다.
- 방사성 표지된 단편을 3,000× g 의 스핀 컬럼을 사용하여 2분 동안 정제합니다.
- 제한 분해 염기서열(그림 2)을 보완하는 400-900bp 선형 DNA 단편을 설계하고 중합효소 연쇄 반응(PCR)을 사용하여 단편을 증폭합니다.
- 50mL의 혼성화 완충액에 방사성 표지된 프로브를 추가하고 혼성화 인큐베이터에서 회전하면서 65°C에서 밤새 멤브레인과 함께 배양합니다.
- 다음 날, 프로브를 제거하고 42°C에서 세척 완충액 1(0.1x SSC, 0.1% SDS)로 멤브레인을 2회 세척하고 65°C에서 세척 완충액 2(2x SSC, 0.1% SDS)로 2회 세척합니다.
알림: 모든 세척은 인큐베이터에서 15분 동안 빠르게 회전하여 수행해야 합니다. - 멤브레인을 10분 동안 건조시키고 얇고 투명한 시트 보호대에 넣습니다. 밀봉된 멤브레인을 형광체에 민감한 스크린이 있는 제어된 방사선 저항성 카세트에 보관하십시오. 멤브레인이 1-10일 동안 스크린에 노출되도록 합니다.
- 형광체 이미징으로 설정된 생체 분자 이미저를 사용하여 결과를 시각화합니다. 필요한 경우 세척하고 다시 노출시키십시오.
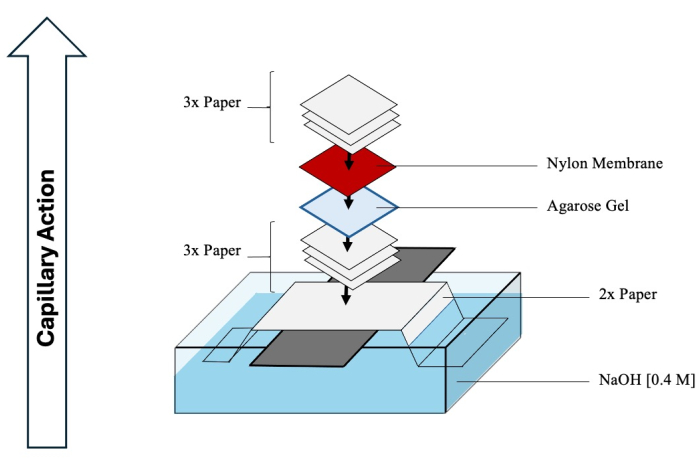
그림 4: 서던 블롯의 조립. Southern blot 중간체를 2차원에서 나일론 멤브레인으로 전달하는 데 사용되는 일반적인 장치의 포괄적인 개략도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
만약 성공한다면, 시각화 시 거대한 1n 지점에서 위아래로 뻗어 있는 복제 포크의 날카로운 호를 관찰할 수 있습니다(그림 5A). 프래그먼트의 크기 또는 복제되는 비율은 첫 번째 차원에서 프래그먼트의 이동성을 결정합니다. 중간체가 더 관절이 있는 구조를 개발함에 따라 2차원에서 더 천천히 이동하기 시작할 것입니다. 그러므로, 만약 중간체가 두...
토론
2DGE는 특정 염기서열의 복제 중에 발생하는 중간체의 상대적 집단에 대한 반정량적이고 포괄적인 이미지를 제공합니다. 복제 포크의 취약한 분자 구조가 이 절차 전반에 걸쳐 유지되어야 한다는 점을 감안할 때 물리적 전단 및 화학적 변성을 방지하기 위해 세심한 주의를 기울여야 합니다. 따라서 플라스미드 분리 중에는 알칼리 처리를 피하는 것이 매우 권장됩니다. 이?...
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
우리 실험실에서 이 접근법을 개발하기 시작한 Jorge Cebrian과 Anastasia Rastokina, pML113 플라스미드와 귀중한 조언을 제공한 Massimo Lopes, 통찰력 있는 토론을 제공한 Ylli Doksani, 그리고 지원을 해준 Mirkin 실험실 구성원들에게 감사드립니다. Mirkin 실험실의 연구는 National Institute of General Medical Sciences[R35GM130322] 및 NSF-BSF[2153071]의 지원을 받고 있습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
참고문헌
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -. Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -. M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863 (2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23 (2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L., Adam, M. P. Emanuel Syndrome. GeneReviews®. , (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M., Kohwi, Y. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. , (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994 (2012).
- . BLAST: Basic Local Alignment Search Tool Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024)
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유