É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Quantificação de γH2AX Focos em resposta à radiação ionizante
Neste Artigo
Resumo
Quantificação de DNA dupla fita-estrias usando formação γH2AX como um marcador molecular tornou-se uma ferramenta valiosa na biologia da radiação. Aqui mostramos o uso de um teste de imunofluorescência para a quantificação dos focos γH2AX após a exposição das células à radiação.
Resumo
DNA dupla fita-breaks (LAP), que são induzidos por qualquer processos metabólicos endógenos ou por fontes exógenas, são uma das lesões DNA mais críticos com relação à sobrevivência e preservação da integridade genômica. Uma resposta rápida à indução de LAP é fosforilação da variante histona H2A, H2AX, na serina-139 resíduos, na altamente conservada motivo SQEY C-terminal, formando γH2AX
Protocolo
Preparação de células
- Queratinócitos humanos (FEP-1811) foram cultivadas em queratinócitos-Serum Médio gratuito (K-SFM; Invitrogen) suplementado com fator de crescimento epidérmico, o extrato de hipófise de bovinos e 20 mcg / ml de gentamicina, a 37 ° CO C e 5% 2.
- A suspensão única célula foi preparado por destacando com tripsina-EDTA (0,05% v / v)
- Células foram semeadas em oito bem-Lab Tek slides II MicroChamber (10.000 células / poço) e as lâminas foram incubadas por 3 dias a 37 ° C e 5% de CO 2.
Irradiação
- As células foram irradiadas no gelo com 2 Gy usando uma fonte de 137 Cs (Gammacell irradiador 1000 Elite; Nordion International, ON, Canadá, 20,6 segundos / Gy)
- Controle não irradiado e 2 Gy células irradiadas foram incubadas por 1 hora a 37 ° C e 5% CO 2.
Imunofluorescência
- Mídia foi derrubado fora e as células foram lavadas com 300 ul de PBS (w / o Ca 2 + ou Mg 2 +) por poço e foram rodados em um misturador orbital por 5 minutos.
- O buffer foi derrubado fora e 100μl de recém-preparados 4% (v / v) paraformaldeído foi adicionado a cada poço e as lâminas foram incubadas em temperatura ambiente por 10 minutos.
Todas as incubações foram realizadas em um cocho de coloração umidificado - As células foram então lavadas com PBS (w / o Ca 2 + ou Mg 2 +). Lâminas foram colocadas em um frasco Coplin e rodado em um misturador orbital por 5 minutos. Esta etapa de lavagem foi repetido mais duas vezes.
- O buffer foi derrubado fora e excesso de PBS foi gentilmente apagados.
- Células foram permeabilizadas com 100ml Triton X-100 (0,1% v / v) por poço e uma incubação de 10 minutos em temperatura ambiente.
- Células foram lavadas com PBS (w / o Ca 2 + ou Mg 2 +), conforme descrito nos passos 3 e 4 acima.
- Ligação não específica de proteínas foi bloqueada com 100 ml de BSA (1% v / v) por poço e 20 minutos de incubação à temperatura ambiente.
- BSA excesso foi derrubado fora e 100μl do ensino primário monoclonal anti-anticorpo fosfo-histona H2AX (diluído 1:500 em BSA 1%; Millipore), foi adicionado a cada poço para a incubação de uma hora à temperatura ambiente.
- Células foram lavadas com PBS (w / o Ca 2 + ou Mg 2 +) conforme descrito nas etapas 3 e 4 acima e incubadas com 100μl de anticorpo secundário (Alexa Fluor 488 cabra anti-camundongo IgG diluído 1:500 em BSA 1% ; Invitrogen) por poço por 45 minutos em temperatura ambiente no escuro. (Anticorpo diluído foi mantido no escuro durante todo o procedimento)
- Células foram lavadas com PBS (w / o Ca 2 + ou Mg 2 +), conforme descrito nos passos 3 e 4 acima. No entanto, a exposição à luz foi minimizado usando papel alumínio.
- Contracoloração nuclear foi realizado com 100 ml TOPRO3 (diluído 1:500; Invitrogen) por poço e incubação de 10 minutos em temperatura ambiente
- Células foram lavadas com PBS (w / o Ca 2 + ou Mg 2 +) como descrito na etapa 10 acima.
- As câmaras foram cuidadosamente removido os slides, o excesso de umidade foi apagado e as lâminas foram secas ao ar.
- Uma gota de solução anti Prolongar-fade GOLD (Invitrogen) foi adicionado por poço e lâminas foram montadas (22x50 milímetros lamela) e qualquer excesso de líquido ao redor das bordas do slide foi apagado.
- As lâminas foram mantidas no escuro por mais 30 minutos em temperatura ambiente antes de selar com unha polonês.
- As lâminas foram armazenadas durante a noite a 4 ° C no escuro antes da análise.
Microscopia / Análise
- Zeiss LSM510 Meta Confocal Microscpe utilizados para adquirir imagens utilizando a GFP padrão (para γH2AX - Alexa Fluor 488 cabra anti-IgG de rato) e lasers vermelho distante (para TOPRO-3). Tipicamente, um óleo de 63 x lente objetiva de imersão é usado.
Imagens são adquiridas em um padrão Z-série com um tamanho de passo de 0,5 micron. A tamanho do passo de 0,5 m foi escolhida para minimizar a perda de apresentar focos em diferentes planos no núcleo. Durante a análise, planos individuais são deconvoluídos e empilhados para produzir uma imagem máxima projetada para minimizar a sobreposição de focos (Top hat-filtro aplicado). - Metamorph (dispositivos Molecular, EUA) foi utilizado para analisar o número de focos.
O programa quantitates o número de focos em cada célula após o limite tem sido aplicado para excluir fundo. As informações são registradas em uma planilha do Microsoft Excel para análise posterior.

Figura 1. Visualização de imunofluorescência γH2AX focos (verde) em queratinócitos humanos não tratados e em células irradiadas com 2 Gy e incubados por uma hora mais 1 a 37 ° C, 5% CO 2. O DNA foi corado com TOPRO-3 (azul). Imagens foram adquiridas utilizando um microscópio Zeiss LSM 510 Meta Confocal. Bar = 10 mM.
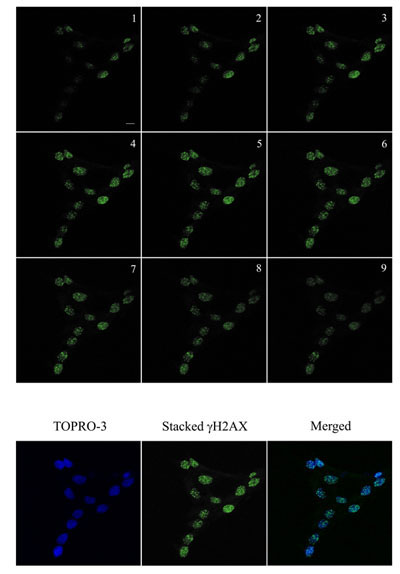
Figura 2. Visualização de imunofluorescência γH2AX focos (verde) em queratinócitos humanos e em células irradiadas com 2 Gy e incubados por uma hora mais 1 a 37 ° C, 5% CO 2. Imagens foram adquiridas utilizando um microscópio Zeiss LSM 510 Meta Confocal utilizando 0,5 m de corte Z-(1-9) para garantir que todos os focos foram adquiridas. As imagens foi então empilhados para quantificação usando Metamorph. O DNA foi corado com TOPRO-3 (azul). O γH2AX empilhados e imagens azuis foram empilhadas para visualização. Bar = 10 mM.
Discussão
Após a exposição à radiação ionizante (raios-γ), forma γH2AX focos rapidamente e os números de focos atingir um máximo entre 30-60 minutos 2. Portanto, nosso ponto de uma hora o tempo pós-irradiação reflete DSB formação inicial. Temos usado a dose de radiação clinicamente relevantes de 2 Gy para nosso experimento. No entanto, o método pode ser usado para as doses de radiação até 4 Gy para a detecção da formação inicial DSB; sobreposição significativa de focos impede a quantificaçã...
Divulgações
Agradecimentos
O apoio do Instituto Australiano de Ciência Nuclear e Engenharia é reconhecido. TCK foi o ganhador de prêmios AINSE. Lab Medicine epigenômico é suportado pelo National Health and Medical Research Council of Australia (566.559). LM é apoiada por Melbourne Research (University of Melbourne) e Biomedical Imaging bolsas complementares CRC. O apoio de Monash Micro Imaging (Drs. Stephen Cody e ISKA Carmichael) foi inestimável para este trabalho.
Materiais
| Name | Company | Catalog Number | Comments | |
| Keratinocyte-Serum Free Medium (K-SFM) | Media | Invitrogen | 17005042 | Keratinocyte media supplemented with human recombinant Epidermal Growth Factor 1-53 (EGF 1-53), and Bovine Pituitary Extract (BPE). |
| Lab Tek lI Chamber Slides (8-well) | Chamber Slides | Nalge Nunc international | NUN154534 | |
| Coverslips (22x50mm) | Coverslips | Menzel-Glaser | CS2250100 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906 | BSA (1%) is used to block any non-specific antibody binding. Primary and secondary antibodies are diluted in BSA. | |
| PBS (without Ca2+ and Mg2+) | Invitrogen | 17-517Q | ||
| 0.5% Trypsin-EDTA x10 | Invitrogen | 15400-054 | Trypsin-EDTA (0.05%) used to detach cells from culture flasks. | |
| Triton X-100 | Reagent | Sigma-Aldrich | T8787 | Triton X-100 (0.1%) used to permeabilise cells. |
| Paraformaldehyde | Reagent | Sigma-Aldrich | 158127 | Paraformaldehyde (4%) used to fix cells. |
| Mouse monoclonal anti-phospho histone-H2AX antibody | Primary Antibody | EMD Millipore | 16193 | Dilution of primary antibody (1:500), in 1% BSA. |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) | Secondary Antibody | Invitrogen | 11029 | Dilution of secondary antibody (1:500), in 1% BSA. |
| TOPRO3 | DNA Stain | Invitrogen | T3605 | DNA stain commonly used: 4,6-diamidino-2-phenylindole dihydrochloride (DAPI). Can only be used with microscopes with the appropriate excitation laser. |
| ProLong Gold | Anti-fade solution | Invitrogen | P36930 | Refractive index of 1.42 at 20°C. |
| Tissue Culture Flask, Vented Cap | Culture Flask | BD Biosciences | 353112 | |
| Tissue Culture Dish (150x25mm) | Petridish | BD Biosciences | 353025 | |
| Coplin Jar, glass | Grale Scientific P/L | 1771-OG | ||
| Staining Trough | Grale Scientific P/L | V1991.99 | ||
| Gammacell 1000 Elite Irradiator | Gamma Irradiator | Nordion International Inc. | ||
| Zeiss LSM 510 Meta Confocal | Confocal Microscope | |||
| Metamorph | Software for Imaging analysis | Molecular Devices |
Referências
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J. Cell Biol. 146, 905-916 (1999).
- Bonner, W. M. Gamma H2AX and cancer. Nature Reviews Cancer. 8 (12), 957-967 (2008).
- Savic, V. Formation of Dynamic [gamma]-H2AX Domains along Broken DNA Strands Is Distinctly Regulated by ATM and MDC1 and Dependent upon H2AX Densities in Chromatin. Molecular Cell. 34 (3), 298-310 (2009).
- Downs, J. Binding of chromatin-modifying activities to phosphorylated histone H2A at DNA damage sites. Mol Cell. 16 (6), 979-990 (2004).
- Chowdhury, D. [gamma]-H2AX Dephosphorylation by Protein Phosphatase 2A Facilitates DNA Double-Strand Break Repair. Molecular Cell. 20 (5), 801-809 (2005).
- Nakada, S., Chen, G., Gingras, A., Durocher, D. PP4 is a gamma H2AX phosphatase required for recovery from the DNA damage checkpoint. EMBO Rep. 9 (10), 1019-1026 (2008).
- Altaf, M., Auger, A., Covic, M., Côté, J. Connection between histone H2A variants and chromatin remodeling complexes. Biochem Cell Biol. 87 (1), 35-50 (2009).
- Kusch, T. Acetylation by Tip60 Is Required for Selective Histone Variant Exchange at DNA Lesions. Science. 306, 2084-2087 (2004).
- Hurlin, P. Progression of human papillomavirus type 18-immortalized human keratinocytes to a malignant phenotype. Proc Natl Acad Sci U S A. 88 (2), 570-574 (1991).
- Dickey, J. H2AX: functional roles and potential applications. Chromosoma. , (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados