É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Cromatina imunoprecipitação (CHIP), utilizando Drosophila Tecido
Neste Artigo
Resumo
Recentemente high-throughput tecnologia de seqüenciamento aumentou muito a sensibilidade da cromatina imunoprecipitação (CHIP) experiência e levou o seu aplicativo usando células purificadas ou tecido dissecado. Aqui nós delinear um método para usar a técnica de chip com Drosophila Tecido, que pode tratar o estado da cromatina endógeno em um sistema bem caracterizada biológica.
Resumo
Epigenética continua a ser um campo de rápido desenvolvimento que estuda como o estado de cromatina contribui para a expressão diferencial de genes em tipos celulares distintos em diferentes estágios de desenvolvimento. Regulação epigenética contribui para um amplo espectro de processos biológicos, incluindo a diferenciação celular durante o desenvolvimento embrionário e homeostase na idade adulta. Uma estratégia fundamental em estudos epigenéticos é examinar como várias modificações de histonas e fatores de cromatina regular a expressão gênica. Para resolver isso, cromatina imunoprecipitação (CHIP) é amplamente utilizado para obter um instantâneo da associação de fatores particulares com DNA nas células de interesse.
Técnica ChIP geralmente usa células cultivadas como material de partida, que pode ser obtido em abundância e homogeneidade para gerar dados reprodutíveis. No entanto, existem várias advertências: Primeiro, o ambiente para crescer as células em placa de Petri é diferente do que in vivo, assim, podenão reflectem o estado da cromatina endógena de células de um organismo vivo. Em segundo lugar, nem todos os tipos de células podem ser cultivadas ex vivo. Há apenas um número limitado de linhas de células, a partir do qual as pessoas podem obter material suficiente para ensaio ChIP.
Aqui nós descrevemos um método para fazer experiência chip usando tecidos de Drosophila. O material de partida é dissecado de tecido de um animal vivo, assim com precisão pode reflectir o estado da cromatina endógena. A adaptabilidade deste método com muitos tipos diferentes de tecido irá permitir aos pesquisadores abordar muito mais perguntas biologicamente relevantes sobre regulação epigenética in vivo 1, 2. Combinando este método com alto rendimento de seqüenciamento (CHIP seq-) continuará a permitir que os investigadores de obter uma paisagem epigenômico.
Protocolo
(O procedimento todo leva ChIP aproximadamente dois dias. Preparação de bibliotecas de Fichas para high-throughput seqüenciamento leva mais de 2-3 dias).
1. Dissecar e preparação de tecidos para o Experimento chip (~ 1 milhão de células)
- Dissecar tecido de interesse (por exemplo, 200 pares de Drosophila testículos) em PBS frio inibidor da protease + 1x (dissolver 1 pelete de protease cocktail inibidor em 1,5 mL de PBS 1x para se obter solução de reserva 7x) + PMSF (concentração final 100 ug / mL). Lavar duas vezes tecido e ressuspender o tecido em 200 uL da solução de PBS mesmo com inibidores.
- Para fixar a amostra, adicionar 5,5 uL de formaldeído 37% (formaldeído quente em 37 ° C banho de água durante 1 min antes de usar). Incubar a 37 ° C durante 15 minutos, de vórtice a cada 5 minutos entre os dois.
- Colocar a amostra em gelo para o tecido para resolver durante 2 minutos. Lavar 2x com 450 uL de PBS (com inibidores da protease e PMSF). Amostra já pode ser lojad a -20 ° C. Repita várias vezes dissecação para obter material suficiente para ChIP.
2. Preparar o sobrenadante com DNA-proteína Conjugação de Ensaio ChIP
- Combine amostra suficiente a partir do passo de 1,3 em um tubo eppendorf 1,75 mL.
- Remover PBS a partir de amostra de tecido. Adicionar 200 uL de tampão de lise (50 mM Tris-HCl, pH 7,6, 1 mM de CaCl2, 0,2% de Triton X-100 ou NP-40, 5 butirato mM, e 1 x cocktail inibidor de proteinase). Adicionar solução de reserva de fresco PMSF para tampão de lise (PMSF concentração final de 0,5 mM). Homogeneizar com azul homogeneizador (Fisher Cat. # 749.521-1590), até que os tecidos se dissociam completamente sem qualquer agregação, incubar a RT (temperatura ambiente) durante 10 minutos.
- Sonicação: sonicador uso microtip (Misonix HS-XL200) na potência 20. Sonifique por 10 segundos e repouso em gelo durante 50 seg. Repita 4-5 vezes (tempo de sonicação óptima deve ser determinada empiricamente, como já sonicação irá produzir fragmentos menores. Para chip usando anticorpos contra Histonmodificações e, nós normalmente sonicate cromatina para aproximadamente 200-300 pb; para o fator de transcrição ou regulador de cromatina (por exemplo, proteínas Polycomb), que normalmente sonicate cromatina de 200-1000 pb. No entanto, o tamanho do fragmento óptima deve ser testado empiricamente. Nota: Sempre mantenha o tubo em gelo MESMO DURANTE sonicando PARA EVITAR AMOSTRA DE AQUECIMENTO!
- , Diluir a amostra por adição de 1,8 mL de tampão RIPA (10 mM Tris-HCl, pH 7,6, EDTA 1 mM, SDS a 0,1%, 0,1% de Na-Desoxicolato, Triton a 1% X-100, com inibidores de protease e PMSF na mesma concentração, tal como descrito anteriormente). Guardar 40 uL como controle de entrada por adição de 2 uL NaCl 5M e incubar a 65 ° C durante a noite (O / N) para inverter reticulação.
- Para conjugar o anticorpo a esferas, adicionar 40 uL de Proteína A pérolas para um tubo Eppendorf de 1,5 ml, em seguida, adicionar 600 uL de PBS e rocha a 4 ° C durante 2 minutos, aplicar pérolas para magneto e remover o sobrenadante. Adicionar 100 uL de PBS e do anticorpo de interesse (quantidade de anticorpo deve ser determinada empircamente). Incubar a temperatura ambiente durante 1 hora ou 4 ° C durante 4 horas.
- Remover o sobrenadante a partir de grânulos por magneto e adicionar 1 mL de cromatina extracto a partir do passo 2,4 para os grânulos. Gire a 4 ° CO / N.
- Aplicar a amostra ChIP de ímã e remova o sobrenadante. Lavar os grânulos com a seguinte tampão a 4 ° C durante 10 minutos cada:
2x com 1 mL de tampão RIPA [1,89 'tampão RIPA' mL + 315 uL inibidores de protease PMSF 7x + 20 uL];
2x com 1 mL de tampão RIPA + 0,3 M de NaCl [1,89 mL de tampão RIPA + 220 uL 3 M de NaCl];
2x com 1 mL de tampão de LiCl (0,25 M LiCl, 0,5% NP40, NaDOC 0,5%);
1x com 1 mL de 1x TE + 0,2% de Triton X-100;
1x com 1 mL de 1x TE. - Para inverter reticulação, ressuspender as pérolas em 100 uL de tampão TE + 3 uL de 10% SDS + 5 ul de proteinase K (20 mg / mL). Incubar a 65 ° CO / N.
- Aplicar pérolas para íman e transferir o sobrenadante para um novo tubo. O sobrenadante contiver a sua amostra de DNA. Lavar pérolas com 100 uL de TE + 0,5 MNaCl e combinar o sobrenadante dois.
- Fenol-clorofórmio extrair a amostra de ADN. Adicionar 200 uL Fenol: Chlorofrom: IAA mix (25:24:1) e vortex. Girar em 14k durante 5 minutos à RT. Alternativamente, o DNA pode ser extraído usando Qiagen kit de purificação PCR.
- Transferir a camada sobrenadante / aquosa para um novo tubo, e adicionar acrilamida linear para uma concentração final de 20 ug / mL (Alternativamente 1 uL de glicogénio a 20 mg / mL para cada 1 mL de sobrenadante pode também ser usado. Glicogénio é utilizado para qPCR subsequente ou análises de PCR enquanto acrilamida linear é utilizada para a sequenciação ensaios.) Adicionar 20 ul de NaOAc 3M e 500 uL de EtOH a 100%. Misturar bem e incubar a 80 ° C durante 10 minutos. Girar à velocidade máxima durante 20 minutos a 4 ° C.
- Remover o sobrenadante e lava-se com 300 uL de EtOH a 70%. Ar amostra seca e ressuspender em 50 uL de tampão TE. A amostra pode agora ser armazenada a -20 ° C. A amostra pode ser utilizado para PCR quantitativo de ensaio (qPCR) (Figura 1).
3. Analisar ChIP-ed DNA
3a. Analisar ChIP-ed DNA usando qPCR
- Diluir o ADN genómico na concentração seguinte para fazer uma curva padrão: não diluído, 1/10, 1/100, 1/1000, 1/5000.
- Para cada par de primers, configurar qPCR em duplicado em uma placa de 96 poços com o ADN genómico a partir de 3A.1 passo, o controlo de entrada e Chip-ed ADN:
10 L 2x SYBR PCR mix (Fermentas, K0222);
1 ml de 10 mM para a frente do primer;
1 uL 10 iniciador reverso iM;
x DNA ChIP-ed uL (controle de entrada, ou ADN genómico);
uL y sem nuclease de H2O para ajustar o volume de reacção de 20 uL. - Girar a placa de 96 poços com Mini placa giratório (Labnet).
- Realizar qPCR com ABI 7300 PCR em tempo real do sistema (ou outros sistemas em tempo real de PCR), utilizando a seguinte condição:
Fase 1: 50 ° C durante 2 min, 1 ciclo;
Fase 2: 95 ° C durante 10 min, 1 ciclo;
Fase 3: 95 ° C durante 15 s, 60 ° C durante 1 min, 40 cycles;
Fase 4 (fase de dissociação): 95 ° C durante 15 s, 60 ° C durante 1 min, 95 ° C durante 15 s. - Verifique a curva de dissociação: Um pico no gráfico de dissociação térmica sugere um fragmento amplificado única a partir da reacção de PCR. Mais do que um pico indica não-específica do produto com o par de iniciadores, os dados qPCR não deve ser utilizado.
- Determinar a fase linear de amplificação exponencial de reacção de PCR para cada conjunto de iniciadores, através do cálculo da fórmula que resolve quantidade de DNA de acordo com o valor Ct (limiar de ciclo), que se baseia na curva padrão, utilizando a série de ADN genómico diluída em 3a. 1.
- Se o valor de Ct ChIP-ed DNA e de controlo de entrada estão dentro da gama linear do valor Ct, calcular o enriquecimento de ChIP-ed DNA versus entrada, de acordo com o factor de diluição de Chip-ed DNA e de controlo de entrada em 2,4.
3b. Amplify ChIP-ed DNA para o seqüenciamento de alto rendimento.
- Fim-de reparação de Chip-ed mistura DNA (UseEpicentro DNA Repair Kit END), criado no gelo:
1-34 uL de ADN genómico a partir do passo 2,12 (0,1 a 5 mg)
5 tampão reparação ul 10x Fim
5 ul 2,5 mM mistura de dNTP
5 ul de 10 mM ATP
x uL de H2O para ajustar o volume de reacção de 49 uL
1 mistura de enzimas ul End Reparação-(DNA polimerase T4 + polinucleótido quinase T4)
Incubar durante 45 minutos à RT. Purifica-se reacção utilizando Qiagen Reacção MinElute
Kit de limpeza. Eluir em 30 ul de tampão de eluição (EB). - Adicionar um overhangs-de-final 3 ':
30 ul de produto de ADN eluído a partir de 3B.1 passo
5 tampão NEB ul 10x # 2
10 uL de 1 mM de dATP
2 uL de dH2O
3 ul 5 fragmento de Klenow U / uL (3 '→ 5' exo-)
Incubar durante 30 minutos a 37 ° C. Purifique reação usando MinElute kit ReactionCleanup. Eluir em 10 EB uL. - Solexa ligadura vinculador:
10 ul de DNA eluído a partir de 3B.2 passo
10 uL ddH2O
2.5 μ l de tampão ligase 10x T4 DNA
Adaptador de 1 ml mistura de oligo Illumina (1/10-diluted do estoque)
2,5 DNA-ligase de T4 ul (400 unidades / ul)
Incubar durante 1 hora à RT. Purifique reação usando MinElute kit ReactionCleanup. Eluir em 20 EB uL. A amostra pode agora ser armazenada a -20 ° C. - Executar a amostra de DNA eluído a partir de 3B.3 passo usando o aparelho de E-gel. Isolar o 300-500 pb banda no gel e purificar usando o kit Qiagen Gel Extraction (este passo elimina os do ~ 125 pb auto-ligadura linkers a partir da ligadura adaptador oligo). Eluir em 12 EB uL.
- Amplify biblioteca usando emparelhado-end (PE) primers de Illumina:
10,5 ul de DNA eluído a partir de 3b.4 passo
12,5 ul de mistura principal (2X Phusion HF, Finnzymes)
1 uL de PE1 iniciador de PCR 1
1 uL de PE2 iniciador de PCR 2
Condição de PCR:
98 ° C durante 30 seg
(98 ° C 10 seg, 65 ° C 30 seg, 72 ° C 30 seg), repetem 20 ciclos
72 ° C durante 5 minutos. - Amostras de 3b.5 passo pode ser usado para ChIP seq-depois de seleccionar o DNA de tamanho adequado (300-500 pb), utilizando o padrão de 2% de gel de agarose. A amostra pode agora ser armazenada a -20 ° C. É melhor para isolar cada amostra num gel de ChIP único para evitar contaminação. As amostras podem ser submetidas para seqüenciamento de alto rendimento.
3c. Solexa análise gasoduto
- A sequenciação de 25-bp lê foram obtidos a partir do genoma Illumina Analyzer (GA) pipeline.
- Todas as leituras foram alinhados ao genoma da Drosophila (dm3), utilizando o ELAND (alinhamento local eficiente de dados de nucleotídeos), software, permitindo que até duas incompatibilidades com a seqüência de referência.
- Leituras exclusivamente mapeados foram retidas para análise a jusante. Para várias leituras idêntico, até três cópias foram retidos para reduzir a possibilidade de desvios de amplificação por PCR.
- A saída do Pipeline GA foi convertido em dados do navegador extensível (TCAP) arquivos.
- Para geraros arquivos wig para carregar para o navegador UCSC para visualização, utilizou-se um scrip python que tem sido descrita em uma publicação anterior 3 com 4 pb como o tamanho da janela e 160 bp como o tamanho do fragmento de DNA.
- Para classificar genes de Drosophila em genes silenciosos e expressa, usamos um número digital chamado RPKM (lê / por kilobase fundiu região exônico / por milhão mapeada lê). Genes com RPKM = 0 foram classificados como grupo silencioso e genes com RPKM ≥ 1 foram divididos em três grupos expressas, que podem ser classificados em três subgrupos de genes com baixos níveis de expressão, moderada e alta, e cada grupo tem sobre o mesmo número de genes.
- Para comparar o nível de modificação da histona e expressão do gene, foi utilizado um script python que tem sido descrito em uma publicação anterior 3.
4. Os resultados representativos
Exemplos de ChIP qPCR-bam resultados usando (saco de bolas de gude) mutante testículos são mostrados na Figura 1 4. Em bam testículos, há uma falha na transição da espermatogônias proliferativa para diferenciar espermatócitos 5, 6. Usamos bam testículos como uma fonte para as células germinativas indiferenciadas, que são enriquecidas neste tipo de tecido. Genes de diferenciação necessários para a diferenciação de espermatozóides, como macho-específico fator de transcrição 87 (mst87F), Don Juan (dj), e cebola fuzzy (fzo) não são expressos em bam testículos. Estes genes são altamente enriquecida com a modificação H3K27me3 repressor histona 7 (Figura 1A), mas são desprovidos da modificação H3K4me3 activa histona 8 (Figura 1B), uma assinatura cromatina chamamos 7 'monovalente'. Enriquecimento de uma das H3K27me3 ou H3K4me3 é determinada por normalização de uma ciclina expressa constitutivamente um gene (CYCA) 4.
conteúdo "análise> Chip-seq usando o mesmo conjunto de anticorpos (ie H3K27me3 repressiva e H3K4me3 ativa) com bam testículos mutantes (Figura 2) validou os resultados qPCR mostrados na Figura 1. Para os três genes de diferenciação testados terminais mst87F, dj e fzo , as suas regiões genómicas são altamente enriquecida com H3K27me3 mas não H3K4me3 (Figura 2A-2C). Em contraste, a expressão constitutiva do gene CYCA tem H3K4me3 significativo, mas pouco H3K27me3 ligação perto da sua local de início da transcrição (TSS) (Figura 2D).Além disso, os perfis de chip de H3K27me3 e H3K4me3 perto das TSSs de quatro classes de genes expressos diferencialmente são consistentes com a função de cada modificação histona. Como mostrado na Figura 3A, o enriquecimento de H3K27me3 a jusante da TSSs está inversamente correlacionada com o nível de expressão do gene. Os genes silenciosas tem a mais alta que a H3K27me3genes altamente expressos não têm ligação H3K27me3. Estes dados são consistentes com o papel repressivo do H3K27me3 sobre a expressão gênica. Em contraste, o enriquecimento de H3K4me3 em torno dos TSSs mostrou a correlação oposto, com a nível do gene de expressão (Figura 3B), consistente com o papel activo de H3K4me3 na expressão do gene.
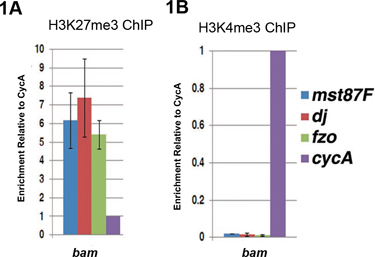
Figura 1. Análise qPCR de Chip-ed ADN, utilizando o anticorpo contra quer o H3K27me3 modificação repressor histona (A) ou modificação H3K4me3 activa histona (B) em indiferenciadas célula enriquecido bam testículos mutantes. (A) Em bam testículos, genes de diferenciação (Mst87F, dj, fzo) são enriquecidos com o H3K27me3 modificação repressiva das histonas. (B) genes de diferenciação estão esgotados com H3K4me3 marca ativa. O nível de DNA chip (DNA chip / entrada) com o gene alvo (Mst87F, dj ou fzo) foi primeiro normalizados para o lev el de DNA chip no gene CYCA controlo. As barras de erro indicam o desvio padrão de três biológica independente repetições.
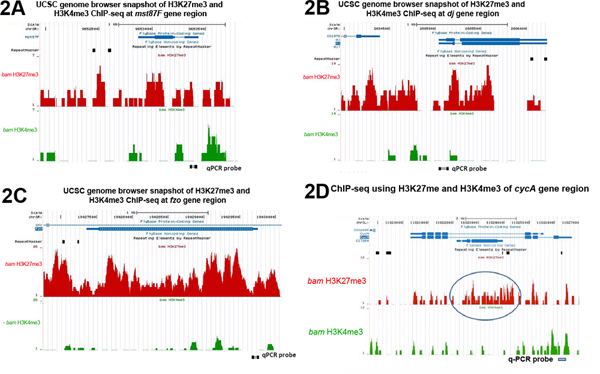
Figura 2. Instantâneos UCSC navegador do genoma de H3K27me3 e H3K4me3 enriquecimento em todas as regiões inteiras genómicos de (A) Mst87F (B) dj e (C) fzo, genes (D) CYCA. A circulado H3K27me3 enriquecido região em (D) reflecte o estado da cromatina de um gene sobreposição CG7264, que é expresso em humilde bam testículos (RPKM = 1), mas altamente expresso em testículos de tipo selvagem (RPKM = 130) 8 (ChIP-SEQ dados a partir de 7). Sondas utilizadas na análise de PCR quantitativa dos resultados ChIP na Figura 1 são identificados na parte inferior de cada trama. para ver figura maior .
tp_upload/3745/3745fig3.jpg "/>
Figura 3. CHIP-seq perfis usando H3K27me3 e H3K4me3 em bam testículos 7. Os quatro grupos de genes (7.509 genes) foram classificados de acordo com seus níveis de expressão gênica com base no RNA-seq resultados 8. As classes representativas dos genes são traçados para o enriquecimento de uma modificação especial histona, utilizando seqüências de 3kb a montante para jusante 3kb de seus sites de transcrição de início (TSSs). Isso gera um perfil de enriquecimento de (A) H3K27me3 (K27) e (B) H3K4me3 (K4) ChIP seq-análises em cada grupo. Clique aqui para ver maior figura .
Access restricted. Please log in or start a trial to view this content.
Discussão
A versatilidade do análises ChIP discutidos nesta protocolo pode ser utilizado em diferentes tecidos, o que proporciona uma oportunidade para estudar o estado de cromatina em um sistema biologicamente relevantes. Experimentos chip usando células a partir de sistemas de cultura são convenientes para realizar porque a grande quantidade de células pode ser facilmente obtida. No entanto, células de cultura não reflectem necessariamente células em um ambiente multi-celular. Ao desenvolver esta técnica usando tecido d...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não temos nada a divulgar.
Agradecimentos
Os autores gostariam de agradecer ao laboratório do Dr. Keji Zhao (NIH / NHLBI) por sua ajuda no fornecimento de resultados de sequenciamento. Gostaríamos também de agradecer ao projeto genoma UCSC para o uso do navegador para visualizar o seqüenciamento do genoma mapeado lê.
Este trabalho foi apoiado pela Pathway NIH R00HD055052 para Prêmio Independência e R01HD065816 do NICHD, a Lucile Parkard Foundation, ea Johns Hopkins University start-up financiamento para XC
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Coquetel inibidor de protease Mini completo | Roche | 11836153001 | |
| O formaldeído (37%) | Supelco | 47.083-U | |
| PMSF | Sigma | 78830 | |
| Kontes pellet pilão | Fischer Scientific | K749521-1590 | |
| O kit de purificação PCR | Qiagen | 28104 | |
| De poliacrilamida linear | Sigma | 56575-1ml | |
| Glicogênio | Qiagen | 158930 | |
| SYBR green / ROX qPCR Mix Master | Fermentas | K0223 | |
| Mini placa giratório | Labnet | Z723533 | |
| PCR em tempo real do sistema | Applied Biosystem | 4351101 | |
| Processador de Pequeno Volume Ultrasonic | Misonix | HS-XL2000 | Modelo descontinuado |
| Dynabeads proteína, uma | Invitrogen | 100-01D | |
| Dynamag ímã | Invitrogen | 123-21D | |
| Fenol: Chlorofrom: IAA | Invitrogen | 15593-049 | |
| Epicentro DNA END Repair Kit | Biotecnologias Epicentre | ER0720 | |
| MinElute reaction Kit de limpeza | Qiagen | 28204 | |
| Klenow Fragmento (3 '→ 5' exo-) | New England Biolabs | M0212S | |
| DNA-ligase de T4 | Promega Corporation | M1794 | |
| Oligonucleotídeos Adaptor | Illumina | PE-400-1001 | |
| Emparelhado Fim-Primer 1,0 e 2,0 | Illumina | 1001783 1001 784 | |
| E-Gel sistema Electorphoresis | Invitrogen | G6512ST | |
| 2X Phusion HF Mastermix | Finnzymes | F-531 |
Referências
- Kharchenko, P. V., Alekseyenko, A. A., Schwartz, Y. B., Minoda, A., Riddle, N. C., Ernst, J., Sabo, P. J., Larschan, E., Gorchakov, A. A., Gu, T. Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485 (2011).
- Filion, G. J., van Bemmel, J. G., Braunschweig, U., Talhout, W., Kind, J., Ward, L. D., Brugman, W., de Castro, I. J., Kerkhoven, R. M., Bussemaker, H. J., van Steensel, B. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Barski, A., Cuddapah, S., Cui, K., Roh, T. Y., Schones, D. E., Wang, Z., Wei, G., Chepelev, I., Zhao, K. High-resolution profiling of histone methylations in the human genome. Cell. 129, 823-837 (2007).
- Chen, X., Lu, C., Prado, J. R., Eun, S. H., Fuller, M. T. Sequential changes at differentiation gene promoters as they become active in a stem cell lineage. Development. 138, 2441-2450 (2011).
- Gonczy, P., Matunis, E., DiNardo, S. bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. 124, 4361-4371 (1997).
- McKearin, D. M., Spradling, A. C. bag-of-marbles: a Drosophila gene required to initiate both male and female gametogenesis. Genes Dev. 4, 2242-2251 (1990).
- Gan, Q., Schones, D. E., Eun, S. H., Wei, G., Cui, K., Zhao, K., Chen, X. Monovalent and unpoised status of most genes in undifferentiated cell-enriched Drosophila testis. Genome Biol. 11, 42-42 (2010).
- Gan, Q., Chepelev, I., Wei, G., Tarayrah, L., Cui, K., Zhao, K., Chen, X. Dynamic regulation of alternative splicing and chromatin structure in Drosophila gonads revealed by RNA-seq. Cell Res. 7, 763-783 (2010).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados