É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Duas e três dimensões imagens de células vivas de Proteínas de DNA resposta a danos
Neste Artigo
Resumo
Este protocolo descreve um método para a visualização de um DNA double-strand proteína quebra de sinalização activada em resposta a danos no ADN, bem como sua localização durante a mitose.
Resumo
Duplo-strand breaks (LAP) são o DNA mais deletéria lesões uma célula pode encontrar. Se deixado sem reparos, DSBs potencial grande porto para gerar mutações e aberrações cromossômicas 1. Para evitar este trauma de catalisar a instabilidade genômica, é crucial para que as células detectam DSBs, ativar a resposta de danos no DNA (DDR), e reparar o DNA. Quando estimulado, o DDR trabalha para preservar a integridade genómica, desencadeando paragem do ciclo celular para permitir a reparação para ter lugar ou forçar a célula a apoptose. Os mecanismos predominantes de reparação de DSB de ocorrer através da extremidade não homóloga-joining (NHEJ) e homóloga recombinação reparação (HRR) (revista em 2). Há muitas proteínas cujas atividades devem ser precisamente orquestrada para a DDR para funcionar corretamente. Relata-se um método para a 2 - e 3-dimensional de visualização (D) de uma destas proteínas, 53BP1.
A proteína p53 de ligação 1 (53BP1) localiza a áreas deDSBs ligando-se a histonas modificadas 3,4, formando focos dentro de 5-15 minutos 5. As modificações de histonas e recrutamento de proteínas e de outros 53BP1 DDR para locais DSB são acreditados para facilitar o rearranjo estrutural da cromatina em torno das áreas de danos e de contribuir para a reparação do ADN 6. Além da participação direta na reparação, funções adicionais têm sido descritos para 53BP1 no DDR, como regular um posto de controle intra-S, um checkpoint G2 / M, e ativar proteínas jusante DDR 7-9. Recentemente, descobriu-se que não se forma 53BP1 focos em resposta ao dano do DNA induzido durante a mitose, em vez de espera para as células antes de introduzir a localização de G1 para a vizinhança de DSBs 6. Proteínas, tais como DDR 53BP1 foram encontrados para associar com estruturas de mitose (por exemplo, cinetocoros), durante a progressão através da mitose 10.
Neste protocolo, descrevemos o uso de 2 - e 3-D imagens de células vivas para visualizara formação de focos 53BP1, em resposta ao agente danificador de ADN camptotecina (CPT), bem como o comportamento 53BP1 durante a mitose. A camptotecina é um inibidor da topoisomerase I, que provoca principalmente DSBs durante a replicação do ADN. Para conseguir isto, foi utilizado um anteriormente descrito 53BP1-mCherry proteína de fusão que consiste em fluorescente construir um domínio de proteína capaz de se ligar 53BP1 DSBs 11. Além disso, utilizou-se uma histona H2B-GFP fluorescente construir a proteína de fusão capaz de controlar a dinâmica da cromatina ao longo do ciclo celular, mas em particular durante a mitose 12. Imagens de células vivas em múltiplas dimensões é uma excelente ferramenta para aprofundar nossa compreensão da função de proteínas DDR em células eucarióticas.
Protocolo
A. Preparação celular
- Fibroblastos humanos normais primárias (GM02270) foram obtidos a partir do Repositório Coriell Cell, Camden, New Jersey, e imortalizadas com hTERT 6. As células foram cultivadas e expandidas em CellStar 6 cm de pratos no meio (4 ml) que consistem em MEM suplementado com 20% soro fetal bovino (GIBCO), non-essential/essential aminoácidos, vitaminas, piruvato de sódio, e penicilina / estreptomicina (HyClone ).
- Rim embrionário humano 293 (HEK293) as células foram obtidas a partir de American Type Culture Collection e cultivadas e expandidas em CellStar 6 centímetros-pratos. As células foram mantidas em meios de comunicação (4 ml), constituído por DMEM suplementado com 10% soro fetal de bovino, não-aminoácidos essenciais, L-glutamina e penicilina / estreptomicina.
- 53BP1 mCherry-(N-Myc-53BP1 WT pLPC-Puro; Addgene plasmídeo 19836) e H2B-GFP (pCLNR-H2BG; Addgene 17735 plasmídeo) construções de gene de fusão também expressar genes de resistência à puromicina ou G418, respectivamente, foram transduzidas (fibroblasts) ou transfectadas (HEK293) usando SuperFect (Qiagene) em células e mantidas sob selecção de droga. Manutenção de fluorescência foi verificada periodicamente.
- 24-48 h antes de aquisição de imagem, as células foram tripsinizadas para uma suspensão de célula única e semeadas a uma densidade baixa em 3,5 cm de placas de fundo FluroDish vidro.
B. Configuração Microscópio e Aquisição de Imagens
Este protocolo foi desenvolvido utilizando o celular Zeiss Observer SD microscópio confocal disco giratório equipado com um Z1 AxioObserver stand, um dual-channel Yokagawa CSU-X1A 5000 unidade de disco giratório, 2 Fotometria QuantEM câmeras 512SC EMCCD, um HXP iluminador à base de fibra 120C, 4 lasers (um Lasos 100mW multi-linha Argon [458, 488, 514 nm], 50 mW diodo de 405 nm, 40 mW diodo nm 561, e 30 mW diodo 635 nm), AOTF, um Pecon XL multiS1 sistema de incubação fase, a incubação Zeiss módulos (O 2 Módulo de S, CO 2 Módulo de S, S TempModule, Aquecimento Unidade XL S) e uma mo Antestorized estágio XY com uma inserção NanoScanZ Z piezo. Para minimizar a aberração esférica, enquanto as células vivas de imagem suportado num meio aquoso, um C-Apochromat 63x/1.20 água / objetivo Corr lente e Immersol Zeiss fluido de imersão W (com um índice de refracção n = 1,334) foram usados. Para 2 canais confocal imaging, um RQFT 405/488/568/647 espelho dicróico e BP525/50 (verde) e BP629/62 (vermelho) filtros de emissão foram utilizados. O software do sistema utilizado foi Zeiss Axiovision (ver. 4.8.2.0) com AV4 Multi-channel/Z/T, Imaging rápido, Fisiologia, MosaiX, Mark & Find, câmera dupla, Inside 4D, foco automático e os módulos 3-D Deconvolução.
- Certifique-se de que o gás de CO 2 está em execução para o Módulo 2 CO do sistema de incubação. Se o microscópio é suportado por uma mesa de ar anti-vibração, ligar o fornecimento de ar (ou gás de N 2) para a mesa de ar.
- Ligue a energia para o stand microscópio, unidade de disco giratório, câmeras, módulos de incubação, iluminador HXP, palco motorizado, Argonlaser, e do computador.
- Depois de um minuto de tempo de aquecimento, desligue a chave de ignição para o laser de argônio para "On".
- No painel de controle Zeiss laser, ligue os interruptores para as linhas de laser a ser utilizado.
- Comutar o interruptor para o controlador Lasos laser de Árgon de "espera" para "run laser" e ajustar o controlador de luz para o nível óptimo (isto é, logo abaixo do ponto em que o indicador verde passa a vermelho).
- Iniciar o software AxioVision Nota:. Interface do usuário para AxioVision pode ser personalizado com janelas e menus suspensos que são específicos para o microscópio especial e componentes que ele controla. Como tal, cada um dos sistemas tem uma interface potencialmente único. Portanto, instruções genéricas para manipulação de software são fornecidos em etapas subseqüentes, em vez de instruções para janelas de software específicos, guias e / ou menus suspensos.
- Cerca de 1 h antes da imagiologia, no software, localizar os controles para a incubadora eligar o aquecimento para a câmara superior e a placa de fase. Definir a temperatura a 37 ° C. Ligue o controle de CO 2 e definir o nível de 5%.
- Selecione a lente objetiva para imagens. Neste estudo, um 63x/1.20 NA C-Apochromat Água / Corr lente objectiva utilizada foi Nota:. Para esta lente, um meio de imersão com um índice de refracção semelhante ao da água (Zeiss Immersol fluido de imersão W) é necessária.
- Se várias posições separadas devem ser sujeitos a amostragem durante um longo período de tempo, ser determinada a aplicar uma quantidade suficiente de meio de imersão para assegurar que é feita a partir de uma posição para outra.
- Coloque o recipiente sobre o palco e trazer a lente objectiva para cima em contacto com o fundo.
- Usando os controles do microscópio ou o software, direcionar a luz emitida para as oculares e selecionar o apropriado Widefield filtro definido para o sinal fluorescente de interesse (para este estudo, "Red" do filtro de 53BP1 e um "GFP" do filtro de H2B ).
- Ver através das lentes oculares, focar a imagem, e localizar um campo adequado de células.
- Usando o software ou os controles do microscópio, direcionar a luz emitida longe das oculares para a porta com a unidade de disco confocal spinning.
- No software, ligar o laser apropriado (para este estudo, o laser nm 561 para 53BP1 ea linha 488 nm do laser de árgon durante H2B).
- Para cada canal, ajustar a intensidade do laser, ajustando a acústico-óptico sintonizável filtro de controlo (AOTF) para um nível apropriado.
- Selecione o espelho apropriado dicróico (RQFT 405/488/568/647) e filtros de emissão (PA 629/62 para 53BP1 e BP 525/50 para H2B). Abrir o obturador para a unidade de disco giratório.
- Selecione a janela "Live" para exibir o campo de vista atual.
- No software, abra a "Câmara" de controle, selecione a câmera para ser usada (se um sistema de câmera dupla) e definir o tempo de exposição para aproximadamente 100 ms. Ajuste o% e EM ganhar como neNota cessário:. A construção 53BP1-mCherry parece um pouco fraca. Descobrimos que é útil para aumentar o ganho de EM.
- No software, abra o controlo para a unidade de disco giratório confocal e ajustar a velocidade de rotação do disco, inserindo o tempo de exposição da câmara que foi definida para captar uma imagem adequada (por exemplo, ~ 100 ms). Clique em "Set" para travar a mudança.
- Abra "aquisição multi-dimensional". Selecione a guia de canais e carregar / selecionar os canais apropriados. Para este estudo, estamos usando canais definidos para DsRed (561 nm excitação laser e BP 629/62 filtro de emissão) e GFP (488 nm linha de laser para excitação e BP525/50 filtro para emissão).
- Para assegurar o registo de imagem, um espelho dicróico comum (RQFT 405/488/568/647) foi utilizado para ambos os canais. Ajustar o software para "Foco automático" Nota:. Vá em Ferramentas → Configurações editor para ajustar as configurações do MDA. Garantir que a energia do laser adequado é definido ("potência do laser Adequada" refere-se a uma configuração que permite que vocêpara excitar o fluoróforo adequado, minimizando foto-branqueamento).
- Na janela MDA, selecione a guia z-stack. Selecione "Z-stack na posição atual foco". Defina o intervalo de 10 m ~ z-stack e escolher "ótima" para o número de etapas para garantir amostragem de Nyquist através Z. Nota: O tamanho exato do z-stack dependerá da altura de suas células. Ajustar em conformidade.
- 23. Clique em "Iniciar" e analisar a imagem resultante z-stack para garantir as configurações são adequadas para o que você está investigando (por exemplo, neste estudo, foi importante para a imagem de todo o núcleo).
- Selecione a opção "T" (tempo) guia. Para a nossa experiência com a camptotecina, definimos o intervalo entre pontos de tempo de imagiologia de 5 minutos e a duração total da sessão durante 1 hora para um controlo (não tratadas células) de vídeo Nota:. Minimizar foto-branqueamento das células, o experimento devem ser configurados de tal forma que o AOTF blanks o laser entre pontos de tempo de imagem.
- Fou multi-ponto de imagem de várias células no prato, na janela do MDA, selecione a guia posição. Garantir "Aplicar" ajuste antes / depois ponto de tempo 'por posição "está marcada. Selecione "Mark_Find". Usando a visão "Live", mova o prato ao redor e selecionar campos apropriados do ponto de vista.
- Clique em "Iniciar" no menu de multi-dimensional-aquisição para começar a experimentar e gravar um vídeo de controle (de células não tratadas).
- Após o controlo de vídeo, adicione o tratamento adequado (neste caso, 10 camptotecina uM). Nós gravamos o vídeo (droga células tratadas) experimental em intervalos de 5-10 min para 2-4 horas Nota:. Uma questão importante a considerar é a velocidade com que um dado efeito ocorre após o tratamento. Como visto no vídeo, 53BP1 focos começam a partir de ~ 5 min após a adição de CPT. Apesar de ser um processo relativamente fácil, a adição de drogas ao prato manualmente e re-calibrar o microscópio não ter tempo. Consideração deve ser dada a este ao projetar experimentos.
- Para monitorar mitosis, definimos o intervalo de 7,5 min e registados durante 4-5 h (as configurações específicas dependem da linha celular utilizada e o comprimento do seu ciclo celular). Ajustamentos podem ter de ser tomadas medidas para evitar foto-branqueamento durante gravações mais longas.
C. Processamento e Análise de Imagem
Este protocolo foi desenvolvido usando o software Volocity (PerkinElmer). O software utilizado para adquirir estes dados (AxioVision) também tem a capacidade de processar e analisar imagens. Usuários são encorajados a utilizar o software disponível para eles, e consultar literatura apropriada quanto à sua aplicação.
- Abra o software Volocity. Criar e nomear uma nova biblioteca, e importar os arquivos de vídeo.
- Para visualizar os arquivos, que geralmente encontrá-lo mais útil usar o "Extended Foco" configuração. Este sobrepõe as fatias z-pilha e, por este protocolo, permite visualizar focos 53BP1 em diferentes áreas do núcleo.
- Ajuste vídeos, se necessário. Volocity está equipado com uma variedade de ferramentas para melhorar a qualidade das imagens obtidas. Ao garantir que as definições sobre o microscópio foram adequadas, muito tempo pode ser guardado na edição mais tarde. Muitas vezes, é útil para deconvolve suas imagens, e ajustar o brilho / contraste. Suas necessidades de edição específicos irão variar com base na experiência.
- Adicionar um carimbo de tempo relativo e barra de escala.
- Para ver as células em 3-D, mudar para o "3-D opacidade" configuração. Isto permite a rotação das células em 3-D fundidas no espaço, proporcionando assim várias perspectivas de estruturas de interesse no interior das células Nota:. É útil para visualizar as células em diferentes planos para determinar onde as estruturas de interesse de viagem. Por exemplo, no "Extended Focus" configuração, é difícil de discernir que o 53BP1 se, de facto, a partir de DNA dissociar durante a mitose. No entanto, este é prontamente aparente em 3-D.
- Filmes e imagens estáticas podem ser exportados para uma variedade de tipos de arquivo com base na preferência do usuário. i>
D. Representante Resultados
Um exemplo de formação de focos 53BP1 em resposta a CPT é mostrado na Figura 1. As células expostas a forma CPT focos dentro de 5-10 min e manter estes focos em toda a duração de gravação. Como mostrado na Figura 2, 53BP1 dissocia a cromatina no início da mitose, a formação de uma névoa fina em torno dos cromossomas de condensação. Como telófase ocorre a mitose e chega ao fim, 53BP1 mais uma vez agregados em focos distintos. Enquanto as células HEK293 não foram expostas a CPT, que, no entanto, formada abundante, focos de reparação espontânea 53BP1 gerada por danos no ADN endógeno. Esta observação permitiu concluir que 53BP1 não formar focos durante a mitose no início, de acordo com um relatório anterior que mostra um efeito semelhante após a exposição das células à radiação ionizante e as drogas radiomimético 5.
A Figura 1 "src =" files/ftp_upload/4251/4251fig1.jpg / "/>
Figura 1. Camptotecina (10 | iM) provoca DSBs e formação de focos em 53BP1 ciclismo fibroblastos dentro de 30 minutos da adição do fármaco ao meio.
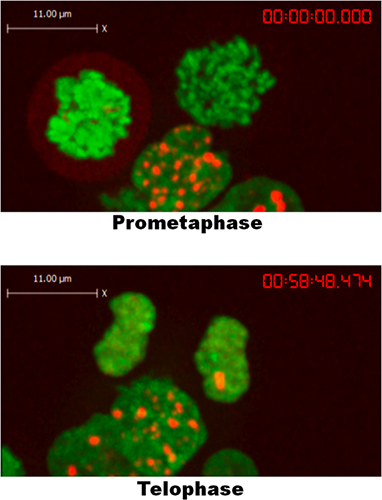
Figura 2. 53BP1 não formar focos durante a mitose até telophase/G1 em células HEK293.
Discussão
Manutenção da integridade do genoma é essencial para a sobrevivência da célula. A falta de preservar os resultados do genoma em envelhecimento prematuro, carcinogênese, a morte ou 8. Há um grande interesse em discernir como as funções de DDR, decorrentes da sua importância para a pesquisa básica e clínica. Muitas técnicas têm sido desenvolvidas ao longo dos anos para ajudar no estudo de como as células detectar e reparar danos no DNA. Métodos tradicionais, como imunocitoquímica e western blot...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Suportado em parte pela R01NS064593 e R21ES016636 (KV). Microscopia foi realizada no VCU - Departamento de Neurobiologia e Anatomia Facilidade Microscopia, suportado, em parte, com recursos do NIH-NINDS Centro núcleo concessão 5P30NS047463. O microscópio confocal disco giratório foi comprado com um prêmio NIH-NCRR (1S10RR027957).
Materiais
| Name | Company | Catalog Number | Comments |
| Produto | Companhia | ||
| Cultura pratos CellStar | Greiner Bio-One | ||
| Pratos de vidro de fundo FluroDish | Precision mundo Instruments, Inc. | ||
| MEM mídia | GIBCO | ||
| Não-aminoácidos essenciais | GIBCO | ||
| Aminoácidos | GIBCO | ||
| Vitaminas | GIBCO | ||
| Piruvato de sódio | Invitrogen | ||
| Penicilina / estreptomicina | HyClone | ||
| Soro Fetal Bovino | GIBCO | ||
| N-Myc-53BP1 WT pLPC-Puro; 19836 plasmídeo | Addgene | ||
| pCLNR-H2BG; 17735 plasmídeo | Addgene | ||
| SuperFect | Qiagen | ||
| Celular Zeiss Observer SD Imaging System | Zeiss | ||
| AxioVision (versão 4.8.2) | Zeiss | ||
| Zeiss Immersol W óleo | Zeiss | ||
| Volocity software (versão 6.0) | PerkinElmer |
Referências
- Botuyan, M. V., Lee, J., Ward, I. M., Kim, J. E., Thompson, J. R., Chen, J., Mer, G. Structural basis for the methylation state-specific recognition of histone H4-K20 by 53BP1 and Crb2 in DNA repair. Cell. 127, 1361-1373 (2006).
- Dimitrova, N., Chen, Y. C., Spector, D. L., de Lange, T. 53BP1 promotes non-homologous end joining of telomeres by increasing chromatin mobility. Nature. 456, 524-528 (2008).
- Feuerhahn, S., Egly, J. M. Tools to study DNA repair: what's in the box. Trends Genet. 24, 467-474 (2008).
- Giunta, S., Belotserkovskaya, R., Jackson, S. P. DNA damage signaling in response to double-strand breaks during mitosis. J. Cell Biol. 190, 197-207 (2010).
- Giunta, S., Jackson, S. P. Give me a break, but not in mitosis: the mitotic DNA damage response marks DNA double-strand breaks with early signaling events. Cell Cycle. 10, 1215-1221 (2011).
- Golding, S. E., Morgan, R. N., Adams, B. R., Hawkins, A. J., Povirk, L. F., Valerie, K. Pro-survival AKT and ERK signaling from EGFR and mutant EGFRvIII enhances DNA double-strand break repair in human glioma cells. Cancer Biol. Ther. 8, 730-738 (2009).
- Huyen, Y., Zgheib, O., Ditullio, R. A., Gorgoulis, V. G., Zacharatos, P., Petty, T. J., Sheston, E. A., Mellert, H. S., Stavridi, E. S., Halazonetis, T. D. Methylated lysine 79 of histone H3 targets 53BP1 to DNA double-strand breaks. Nature. 432, 406-411 (2004).
- Jackson, S. P., Bartek, J. The DNA-damage response in human biology and disease. Nature. 461, 1071-1078 (2009).
- Kanda, T., Sullivan, K. F., Wahl, G. M. Histone-GFP fusion protein enables sensitive analysis of chromosome dynamics in living mammalian cells. Curr. Biol. 8, 377-385 (1998).
- Massignani, M., Canton, I., Sun, T., Hearnden, V., Macneil, S., Blanazs, A., Armes, S. P., Lewis, A., Battaglia, G. Enhanced fluorescence imaging of live cells by effective cytosolic delivery of probes. PLoS One. 5, e10459 (2010).
- Nakamura, K., Sakai, W., Kawamoto, T., Bree, R. T., Lowndes, N. F., Takeda, S., Taniguchi, Y. Genetic dissection of vertebrate 53BP1: a major role in non-homologous end joining of DNA double strand breaks. DNA Repair (Amst). 5, 741-749 (2006).
- Schultz, L. B., Chehab, N. H., Malikzay, A., Halazonetis, T. D. p53 binding protein 1 (53BP1) is an early participant in the cellular response to DNA double-strand breaks. J. Cell. Biol. 151, 1381-1390 (2000).
- Valerie, K., Povirk, L. F. Regulation and mechanisms of mammalian double-strand break repair. Oncogene. 22, 5792-5812 (2003).
- Wang, B., Matsuoka, S., Carpenter, P. B., Elledge, S. J. 53BP1, a mediator of the DNA damage checkpoint. Science. 298, 1435-1438 (2002).
- Ward, I. M., Minn, K., Jorda, K. G., Chen, J. Accumulation of checkpoint protein 53BP1 at DNA breaks involves its binding to phosphorylated histone H2AX. J. Biol. Chem. 278, 19579-19582 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados