É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
RNA Secundária Previsão Estrutura Usando SHAPE de alta capacidade
Neste Artigo
Resumo
Alta capacidade seletiva 2 'acilação hidroxila analisados por extensão primer (SHAPE) utiliza um romance químico sondagem tecnologia, transcrição reversa, eletroforese capilar e software de predição de estrutura secundária para determinar as estruturas de RNAs de várias centenas a vários milhares de nucleotídeos em única resolução de nucleotídeos.
Resumo
Compreender a função do RNA envolvido em processos biológicos requer um conhecimento profundo da estrutura do RNA. Para este fim, o método denominado "2 selectiva high-throughput" acilação hidroxilo analisados por extensão de iniciador ", ou forma, permite a previsão da estrutura secundária de ARN com um único nucleótido resolução. Esta abordagem utiliza agentes químicos que preferencialmente sondagem acilato de regiões de cadeia simples de RNA ou flexível, em solução aquosa. Os locais de modificação química são detectados através de transcrição reversa do RNA modificada, e os produtos desta reacção são fraccionados por electroforese capilar automatizada (CE). Uma vez que a transcriptase reversa pausas naqueles nucleótidos de RNA modificados pelos reagentes de forma, a biblioteca de ADNc resultante indirectamente mapeia os ribonucleótidos que são de cadeia simples, no contexto do ARN dobrado. Usando o software ShapeFinder, os produzidos por electrofogramas automatizado CE são processados e convertidos em nutabelas reactividade cleotide que são eles próprios convertidos em constrangimentos pseudo-A energia utilizada no RNAStructure (v5.3) algoritmo de predição. As estruturas de RNA bidimensionais obtidas pela combinação de sondagem com FORMA na previsão da estrutura secundária de RNA in silico foram encontrados para ser muito mais precisas do que as estruturas obtidas usando qualquer um dos métodos sozinho.
Introdução
Para entender as funções dos RNAs catalíticos e não-codificantes envolvidos na regulação do splicing, tradução, replicação do vírus e câncer, um conhecimento detalhado da estrutura do RNA é necessária 1,2. Infelizmente, a previsão precisa de RNA dobrável apresenta um desafio formidável. Agentes de sondagem clássicas sofrem de muitas desvantagens, tais como a toxicidade, a cobertura incompleta de nucleótidos e / ou uma produção limitada de 100-150 nucleótidos por experimentação. Secundárias algoritmos de predição de estrutura nu são igualmente desvantajoso, devido a imprecisões resultantes da sua incapacidade de distinguir de forma eficaz entre as estruturas energeticamente semelhantes. Grande ARN, em particular, são também muitas vezes refractária aos métodos de determinação da estrutura 3D, tais como cristalografia de raios X e ressonância magnética nuclear (RMN), devido a sua flexibilidade conformacional e grandes quantidades de amostras altamente puros necessários para estas técnicas.
HSHAPE igh throughput resolve muitos desses problemas, fornecendo uma abordagem simples e eficaz para investigar as estruturas de grandes RNAs em resolução de nucleotídeo único. Além disso, os reagentes utilizados para a forma são seguras, fáceis de manusear e, em contraste com a maioria dos outros reagentes químicos de sondagem, reagir com todos os quatro ribonucleótidos. Estes reagentes também podem penetrar as membranas celulares, o que torna possível para sondar ARN na sua no contexto in vivo (s) 3. Desenvolvido originalmente nas semanas de laboratório 4, a forma tem sido usado para analisar uma grande variedade de RNAs, o exemplo mais notável sendo a determinação da estrutura secundária completa do ~ 9 kb de ARN do genoma do HIV-1 5. Outras realizações notáveis utilizando FORMA incluem elucidação das estruturas dos viróides infecciosas 6, RNAs humanos longos não codificantes 7, ribossomas de levedura Riboswitches 8 e 9, bem como para identificar os locais de ligação de proteína no virião associada ao HIV-1 RNA 3. While as variações originais e de alto rendimento do protocolo FORMA ter sido publicado anteriormente 10-12, o presente trabalho fornece uma descrição detalhada de determinação da estrutura secundária de RNA pela forma de alto rendimento, utilizando os oligonucleótidos fluorescentes, a Beckman Coulter CEQ 8000 Genetic Analyzer e (v5.3) software SHAPEfinder e RNAStructure. Detalhes técnicos inéditos e conselhos de resolução de problemas também estão incluídos.
Variações de SHAPE
A essência da SHAPE e suas variações, é a exposição de ARN, em solução aquosa em anidridos electrofílicos que acilar selectivamente 2'-2'-hidroxilo (OH) grupos ribose, produzindo adutos volumosos nos locais de modificação. Esta reacção química serve como um meio de interrogar a dinâmica estruturais do local de ARN, tal como os nucleótidos de cadeia simples são mais propensas a adoptar conformações propícios ao ataque electrof por estes reagentes, enquanto a base emparelhada ou arquitectura constrnucleótidos ained são menos reactivos ou 10. Os locais de formação de aduto são detectados por transcrição reversa a partir de primers iniciando fluorescente ou radiomarcado hibridizaram para um local específico no ARN modificado (a "(+)" reacção de extensão de iniciador). Quando a transcriptase reversa (RT) não atravessam os ribonucleótidos acilados, um conjunto de produtos de cDNA é produzida cujos comprimentos coincidem com os locais de modificação. Um controle, "(-)" iniciador de reacção de extensão, utilizando o RNA que não foram expostas ao reagente é também efectuada de modo que a terminação prematura da síntese de ADN (isto é, "stops"), devido à estrutura do RNA, RNA de cadeia inespecífica quebra, etc, podem. ser distinguidos pausando produzido por modificação química. Finalmente, duas reacções de sequenciação didesoxi-iniciadores a partir dos mesmos iniciadores são usados como marcadores para correlacionar nucleótidos reactivos com a sequência primária de ARN após electroforese.
Na aplicação original do SHAPE, o mesmo P-32 marcado na extremidade iniciador é utilizado para o (+), (-), e duas reacções de sequenciação. Os produtos destas reacções são carregadas em poços adjacentes de uma placa de gel de poliacrilamida a 5-8%, e fraccionados por electroforese em gel desnaturante de poliacrilamida (PAGE; Figura 1). A análise quantitativa de imagens de gel produzidas pela forma convencional, pode ser realizada utilizando SAFA, um software de análise de pegadas semi-automático 13.
Em contraste, a forma high-throughput utiliza primers marcados com fluorescência e eletroforese capilar automatizada. Especificamente, para cada região de ARN sob investigação, um conjunto de quatro iniciadores de ADN contendo uma sequência comum mas diferente 5 'marcadores fluorescentes deve ser sintetizado ou adquirido. Estes oligonucleotídeos diferente com rótulos servem para duas principais reações forma e duas reações de sequenciamento, os produtos de que são agrupados e fracionado / detectado por eletroforese capilar automatizada (CE). Whereas do perfil de reactividade de 100-150 nt de ARN pode ser obtida a partir de um conjunto de quatro reacções, usando a abordagem inicial, a forma de alta-resolução, de caudal permite 300-600 nt a partir de uma única amostra combinada 3. Até 8 conjuntos de reacções podem ser fraccionados em simultâneo, enquanto que até 96 amostras podem preparado para fraccionamento ao longo de 12 ensaios consecutivos, CE (Figura 2). Além disso, o software SHAPEfinder, desenvolvido para processar e analisar dados resultantes do CEQ e outros analisadores genéticos, é mais automatizado e requerem muito menos do que a intervenção do utilizador SAFA 13 ou outros pacotes de gel de análise.
Mais avançadas metodologias de alto rendimento surgiram recentemente, como PARS (análise paralela da estrutura RNA) 14 e Frag-seq (fragmento de seqüenciamento) 15, que utilizam enzimas específicas-estrutura, em vez de reagentes de alquilação em conjunto com a próxima geração de técnicas de sequenciamento para obter information sobre a estrutura do RNA. Apesar da atratividade dessas técnicas, as muitas limitações inerentes à nuclease sondagem ainda permanecem 16. Estes problemas podem ser contornados na sequenciação FORMA (FORMA-Seq) 17 de protocolo, em que a geração seguinte de sequenciação é precedido por modificação química e transcrição inversa do ARN de um modo semelhante ao realizado em forma convencional. Embora estes métodos podem representar o futuro da determinação da estrutura de RNA, é importante lembrar que a próxima geração de sequenciação é muito caro, e permanece disponível para muitos laboratórios.
SHAPE Análise de Dados
Os dados produzidos no analisador genético é apresentado sob a forma de um electroferograma, em que a intensidade de fluorescência da amostra (s) que flui através do detector capilar é representada em função de um índice de tempo de migração. Este lote converte a forma de vestígios de sobreposição correspondente a quatro canais de fluorescências usados para detectar os fluoróforos diferentes, e onde cada um dos traços é constituído por picos correspondentes aos produtos de cDNA ou de sequenciação individuais. Dados electroferograma é exportado a partir do analisador genético como um arquivo de texto delimitado por tabulação e importados para transformação ShapeFinder e software de análise 18.
ShapeFinder é inicialmente utilizada para realizar uma série de transformações matemáticas sobre os dados, para assegurar que os tempos de migração de pico e volumes refletem com precisão as identidades e as quantidades dos produtos de reacção, respectivamente. Os picos são então alinhados e integrados, e os resultados tabulados, juntamente com a sequência de ARN primário. Um "perfil de reactividade" para o segmento relevante de ARN é obtido por subtracção de valores de controlo a partir da (+) valores associados com cada ARN de nucleótidos, e normalizar os dados, conforme descrito abaixo. Este perfil é importado para RNAstructure (v5.3) 19,20 software, que converte o val reatividade normalizadaues em restrições pseudo-energia, que são incorporados ao algoritmo dobrável estrutura secundária do RNA. Combinando sondagem química e dobrando algoritmos desta forma melhora de forma significativa a precisão de previsão de estrutura em comparação com qualquer dos métodos isoladamente 12,21. A saída do RNAstructure (v5.3) inclui imagens de menor energia estruturas secundárias de ARN codificado por cores com o perfil de reactividade FORMA (s), bem como as mesmas estruturas em notação de ponto de colchete textual. Este último pode ser posteriormente transportados para o software dedicado à visualização gráfica da estrutura secundária de RNA tais como Varna PseudoViewer 22 e 23.

Figura 1. Fluxograma de RNA determinação da estrutura via SHAPE 4,10. (A) RNA may ser obtidas a partir de amostras biológicas ou por transcrição in vitro. (B) Dependendo da fonte, o RNA é dobrada ou de outra forma processados e modificado com o reagente FORMA. (C) A transcrição reversa utilizando iniciadores marcados com fluorescência ou radioactivamente. (D) são produtos de cDNA fracionado, quer através de eletroforese capilar à base de gel ou laje. (E) análise de fragmentos. (F) predição de estrutura de RNA. Clique aqui para ver a figura maior.
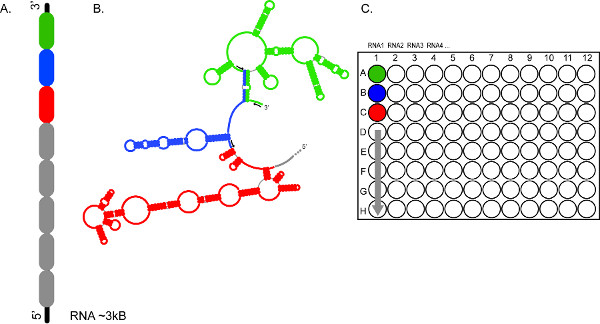
Figura 2. O carácter de elevado rendimento de SHAPE-Based permite a análise rápida de múltiplos ARN, e / ou múltiplos dos mesmos segmentos de RNA. (A) Representa como um RNA pode ser dividido em seções 300-600 NT (código de cores em verde, azul e vermelho) (B) Seções do RNA são detectados de forma independente usando diferentes conjuntos de primers fluorescentes (setas pretas) (C) conjuntos de As reacções foram reunidas e carregadas em poços A1, B1, C1, etc, respectivamente, proporcionando uma cobertura completa para a ~ 3 kb RNA1. Produtos de reacção de RNAs 2, 3, 4, etc, podem ser igualmente preparado para fracionamento em corridas eletroforéticas consecutivos. Clique aqui para ver a figura maior.
Protocolo
Desenho de primers e extensão do terminal do RNA 3 '
Para analisar longos RNAs pela forma de alto rendimento, uma série de sítios de hibridação do iniciador deve ser seleccionada de tal modo que (i) são separados por 300 nt ~, (ii) são de 20-30 nt de comprimento, e (iii) que o RNA / híbridos de DNA produzidos por tratamento térmico de ADN para estes locais têm uma temperatura de fusão esperada de> 50 ° C. Além disso, os segmentos de RNA que estão previstos para serem altamente estruturado deve ser evitado, embora fazer essa determinação requer algum conhecimento prévio da estrutura do ARN, que é frequentemente indisponível. Iniciadores de ADN que hibridizam com estes locais deveriam então ser concebido, tendo o cuidado de assegurar que não seria esperado para formar dímeros estáveis ou estruturas secundárias intracadeia.
Uma vez concebidos, os conjuntos de iniciadores deve ser ou comprados (por exemplo, a partir de DNA integrado Technologies, Ames, Iowa) ou sintetizados 24,25. Os iniciadores marcados com 5'-Cy5, Cy5.5,WellRedD2 (Beckman Coulter) e IRDye800 (Lycor) / WellRedD1 (Beckman Coulter) são os mais adequados para a Beckman Coulter 8000 CEQ, proporcionando boa intensidade de sinal, minimizando a interferência. Os oligonucleótidos marcados podem ser armazenados indefinidamente em pequenas aliquotas de 10 uM a -20 ° C; evitar congelamento / descongelamento repetidos.
Usando primers desenhados desta maneira, é possível obter os dados da forma de praticamente todo um ARN de qualquer comprimento. No entanto, a sequência em ou perto do terminal 3 'de um RNA é sempre inacessível para a forma, a menos que o ARN é modificado para conter a 3' com a extensão do terminal (por exemplo, uma "cassete de estrutura"), ao qual pode ser um iniciador hibridado 4.
RNA Preparação através de Eletroforese Capilar
Embora o ARN a partir de amostras biológicas podem ser utilizados para a forma de alto rendimento, o protocolo fornecido aqui é optimizada para o ARN produzido por transcrição in vitro. Comercial trakits nscription como MegaShortScript (Ambion) usado em conjunto com MegaClear colunas de purificação de RNA (Ambion) são bem adequados para a geração de grandes quantidades de RNA puros. RNAs deve ser armazenado em tampão TE compreendida entre -20 ° C e -80 ° C. Para melhores resultados, deve aparecer RNAs homogéneo por electroforese em gel tanto de poliacrilamida desnaturante e não desnaturante.
1. RNA Folding
- Num tubo de microcentrífuga de 0,5 ml, diluir 12 pmol de ARN a 18 ul com água e adicionar 2 mL de tampão de renaturação 10X. Misturar bem.
- Aquecer a 85 ° C durante 1 min, em seguida arrefecer a 4 ° C a uma taxa de 0,1 ° C / seg.
- Adicionam-se 100 ul de água e 30 ul de tampão de dobragem 5X.
- Incubar a 37 ° C durante 30-60 min, dependendo do RNA a ser dobradas. Em geral, Mg 2 + dependente de dobragem de maiores e mais estruturado RNAs requerem tempos de incubação mais longos.
- Transferir uma alíquota de 72 ul a cada um de dois tubos de 0.5 ml de microcentrífuga: modified (+) e de Controlo (-).
2. Modificação química de ARN
Bem caracterizados, reagentes SHAPE electrofílicos incluem anidrido isatóico (IA), de anidrido N-methylisatoic (NMIA), anidrido 1-metil-7-nitro-isatóico (1M7) 26, e cianeto de benzoílo (BzCN) 27. Destes, o mais comumente usado para a forma high-throughput são 1M7 e NMIA, e apenas este último está disponível comercialmente (Life Technologies). A concentração final do reagente de modificação deve ser optimizada para cada RNA para se obter "um único hit" cinética de modificação, isto é, a condição na qual a maioria dos RNAs em solução uma vez que são modificados na região de ARN a ser analisado 11. Esta concentração óptima pode ser determinada através da realização de múltiplas reacções em que a concentração de reagente é variada através do intervalo (s) indicado na tabela abaixo na Secção 2.1. Utilizar a concentração do reagente, que produz um sinal facilmente detectável enquanto minimizing a diferença na intensidade de sinal entre os produtos de síntese de ADN longas e curtas (por exemplo, Figura 3).
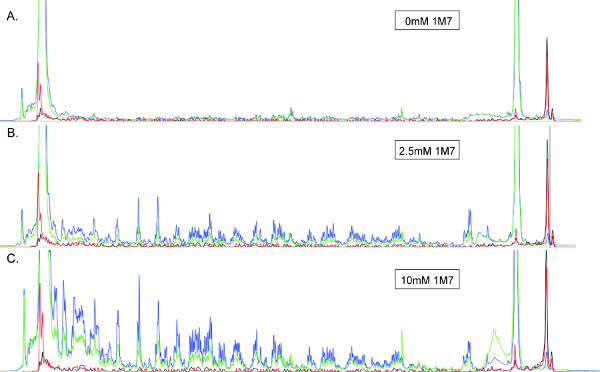
Figura 3. Electrofogramas SHAPE produzidos a partir de um RNA de 360 nt ~ tratada com (A) 0 (B) 2,5 mM ou (C) a 10 mM. 1M7 Todos electrofogramas são mostrados na mesma escala. Azul, vestígios verde, vermelho e negro correspondem ao (+) Os produtos de reacção (Cy5), (-) Os produtos de reacção (Cy5.5), e as duas escadas de sequenciação (D2 WellRed e IRDye800), respectivamente. Os RNA utilizados para produzir imagem (B) tem sido tratada com a quantidade ideal de 1M7, demonstrando boa resolução de pico e intensidade, com decaimento de sinal mínima em todo o trace (esquerda). Leia comprimento é máxima sob estas condições. Em contraste, a ausência de int médioensity, picos bem resolvidos em (A) sugere uma concentração sub-ótima de 1M7. Por outro lado, o decaimento do sinal evidente em (C) indica que a cinética hit único não é observada, e o RNA é sobre-modificados. Em tais casos, especialmente quando a TR não seria de esperar para encontrar o terminal 5 'do molde de ARN, leia comprimento será de qualidade inferior.
- Prepare estoque 10X de reagente SHAPE (NMIA ou 1M7). Este é melhor conseguido pela adição de uma pequena quantidade de reagente para um tubo de microcentrífuga de 1,5 ml, em seguida adição de DMSO para atingir a concentração desejada Atenção:. Soluções de reagentes de forma devem permanecer até anidro misturado com RNA. Loja DMSO num exsicador, à temperatura ambiente, e preparar soluções de reserva, imediatamente antes de usar, a fim de minimizar a exposição ao vapor de água ambiente.
Reagente 10X concentração óptima (em DMSO) Tempo paradegradação completa do reagente 27 NMIA 10-100 mM ~ 20 min 1M7 10-50 mM 70 seg
Tabela 1. Electrofílica reagentes usados para a modificação de ARN. - Adicionar 8 mL de 10X NMIA/1M7 ou DMSO anidro para modificado (+) e de controlo (-) mistura, respectivamente Nota:. 2,5 mM provou ser eficaz uma concentração de partida para ambos NMIA e 1M7, independentemente do ARN a ser analisado.
- Incubar a 37 ° C durante 50 min (NMIA) ou 5 minutos (1M7), conforme apropriado.
- RNA precipitado segundo adicionando 8 ul (0,1 vol) de NaOAc 3 M (pH 5,2), 8 uL de EDTA 100 mM, 240 ul (3 vol) de etanol frio e 1 ul de 10 mg / ml de glicogénio. Refrigerar durante 2 horas, e depois centrifugar a 14000 xg durante 30 min a 4 ° C. Lava-se a pelete duas vezes com etanol frio a 70% Atenção:. É importanteminimizar o tempo de refrigeração, o tempo e velocidade de centrifugação de forma a minimizar a co-precipitação do sal, porque isto pode adversamente afetar a resolução de pico durante a electroforese.
- Remover o sobrenadante com uma micropipeta, e secar ao ar sedimento durante 5 min à temperatura ambiente.
- Dissolve-se o RNA precipitado em 10 ul de tampão TE e incubar durante 5 min à temperatura ambiente. Isto é suficiente dissolvido RNA para duas reacções de transcrição reversa. Armazenar a parte não utilizada a -20 ° C. Atenção: ressuspensão mecânica do pelete não é geralmente necessário, e podem danificar o ARN.
3. Transcrição reversa
Este passo gera os produtos de cDNA marcadas com fluorescência que são usados para identificar indirectamente o grau em que os nucleótidos de RNA foram modificadas por um reagente FORMA. Para a forma, o desempenho do Superscript III (Invitrogen) RT foi superior a todos os outros RTs testado, e é a enzima escolhido para uso com o presenteprotocolo. Oligonucleotídeos marcados com Cy5 e Cy5.5 são usados para preparar o (+) e (-) reações, respectivamente. Para RNAs curtos, os iniciadores são hibridados com um iniciador 3 'de extensão terminal do ARN nativo (por exemplo, uma "cassete de estrutura"), a fim de obter informação sobre o terminal 3' 4 Atenção:. Deste ponto através do CE, as amostras devem ser protegidos contra luz.
- Prepare a (+) e (-) das amostras para a transcrição reversa em tubos de microcentrífuga de 0,5 ml. Para a reacção de RT (+), misturar com 5 ul de ARN modificado (+), 6 ul de água e 1 ul de iniciador marcado com Cy5 (10 uM), para o (-) reacção de RT, misturar 5 ul de RNA de controlo (-), . 6 mL de água e 1 ml Cy5 marcado primer (10 mM) Atenção: Sarstedt PCR tubos (REF 72.735.002) são recomendados para esta aplicação.
- Colocar os tubos num termociclador, e os iniciadores se ligam ao ARN e preparar para a transcrição reversa, aplicando o programa seguinte: 85 ° C, 1 min, 60 ° C, 5 min;35 ° C, 5 min, 50 ° C, manter.
- Durante a etapa de recozimento, a preparação da mistura de RT 2.5X suficiente para o número de reacções a serem realizadas, além de 50% (por exemplo, para dois (+) e dois (-) reacções, a escala de 4,5 vezes). Uma reacção requer 8 mL, como se segue: 4 ul de tampão RT 5x, 1 ul de DTT 100 mM, 1,5 mL de água, 1 ul de dNTPs 10 mM, 0,5 ul SuperScript RT III. Manter em gelo Atenção:. Tampão RT 5X e 100 mM DTT são fornecidos com o SuperScript III RT.
- Uma vez que a temperatura das misturas de recozimento atinge 50 ° C, adicionar 8 mL de mistura de RT para os 2.5X (+) e (-) reacções Recomendação:. Aqueça a mistura temperatura ambiente até 37 ° C durante 5 min antes da adição às reacções .
- Incubar durante 50 min a 50 ° C, depois arrefecer a 4 ° C, e / ou colocar em gelo Nota:. Incubação das reacções de RT por mais de 50 minutos pode resultar em produtos de cDNA aberrantes.
- Hidrolisar ARN por adição de 1 mL de NaOH 4 M e aquecimento a 95 ° C por 3 min. Reações interessantes sobre gelo e depois neutralizá-los por adição de 2 mL de HCl 2 M Atenção:. Omissão desta etapa resulta na separação má qualidade dos produtos de cDNA.
- Combinar (+) e (-) e reacções de precipitar o ADNc por adição de 0,1 volume de NaOAc 3 M, 0,1 vol de 100 mM EDTA, 1,5 vol de etanol frio e 1 ul de 10 mg / ml de glicogénio. Refrigerar durante 2 horas, depois centrifugar a 14000 xg durante 30 min a 4 ° C. Lave o pellet duas vezes com etanol a frio de 70% Atenção:. Centrifugação a taxas mais altas ou por um período mais longo resulta em dificuldades ressuspensão do pellet (s).
- Ressuspender sedimentadas de cDNA em 40 ul de formamida desionizada, por aquecimento a 65 ° C durante 10 min, seguido por vórtice vigoroso durante mais de 30 min Atenção:. Grânulos podem ser invisível. A falta de sinal ou sinal fraco seguinte electroforese pode ser o resultado de uma falha para dissolver adequadamente o sedimento nesta fase.
Seqüenciamento escadas servem como marcadores para a determinação da posição dos nucleotídeos durante o processamento de dados. Estes são gerados utilizando um kit de sequenciação do ciclo de USB (# 78500), ADN possuindo a mesma sequência que o ARN a ser estudada, e primers marcados com D2 WellRed ou D1/Lycor 800. Tipicamente, o ADN utilizado nesta reacção terá que ser utilizado como um modelo para a transcrição de RNA em causa. Embora o protocolo de reacção aqui apresentado se assemelha a recomendada pelo fabricante do kit, a reacção foi reduzida até várias vezes. Enquanto ddA e ddT são usados como terminadores de cadeia nas reacções descritas abaixo, qualquer par de terminais pode ser usada para gerar as escadas de sequenciação.
- Misturar 40 ul da mistura de terminação ddA, 5 pmol de ADN molde, 4,6 ul de tampão Sequenase 10X, 10 ul de iniciador marcado WellRed D2, 4,6 ul de Sequenase e água para perfazer um volume total de 82 ul. Adicione o Sequenase passado. Prepare uma segunda reacção de sequenciação do mesmo modo, utilizando o DDT e os Licor IR800 iniciador marcado em vez disso.
- Continuar a amplificação por PCR usando as condições recomendadas USB Atenção:. Adição de óleo mineral não é necessária nem recomendados para protocolos / termocicladores que utilizam uma tampa aquecida.
- Combine o ddA e ddT seqüenciamento reações em um microtubo de 1,5 ml (~ total de 164 mL).
- O precipitado de ADN como se segue: adicionar 16 uL de NaOAc 3 M (pH 5,2), 16 ul de EDTA 100 mM, 10 ul de 1 mg / ml de glicogénio, e 480 ul de etanol 95%. Misture bem, incubar a 4 ° C durante 30 min e centrifuga-se a 14.000 xg durante 30 min a 4 ° C.
- Ressuspender sedimentadas ADNc em 100 ul de formamida desionizada, por aquecimento a 65 ° C durante 10 min, seguido por vórtice vigoroso durante pelo menos 30 min.
5. Fracionamento de produtos de reacção por eletroforese capilar
A electroforese capilar permite simultâneaseparação dos produtos de síntese de cDNA a partir de quatro reacções combinadas numa única amostra. Oito amostras pode ser fraccionada em simultâneo, enquanto que até 96 amostras podem ser fraccionados durante um único ensaio (Figura 2).
- Misturar 40 ml de amostras SHAPE reunidas com 10 ul das escadas de sequenciação reunidas, e transferir para placas de 96 poços de amostra Atenção:. É imperativo que os reagentes da Beckman Coulter e placas (incluindo LPA-I de gel, tampão de corrida, em óleo mineral, a amostra carregamento da solução e da amostra e as placas de buffer) ser utilizado com a Beckman-Coulter CEQ 8000 Genetic Analyzer.
- Programa e preparar instrumento eletroforese capilar e iniciar executar conforme instruções do fabricante Nota:. Para uma melhor resolução de amostras, use os parâmetros do método CAFA publicados anteriormente 28.
Idealmente, fora de primer e forte-stop picos, os sinais para cada pico em todos os quatro electroferograma tcorridas deve estar na gama linear, uma queda gradual no sinal é aceitável. Às vezes, porém, os grandes picos (pontos) são evidentes mesmo na reação controle, e estes podem interferir com o processamento de dados subseqüente. CDNAs truncadas que dão origem a estes picos pode ser o resultado de um obstáculo natural durante a transcrição reversa (por exemplo, a estrutura secundária de RNA) ou a degradação de RNA. No primeiro caso, os aditivos, tais como a betaína pode melhorar a processabilidade e reduzir RT RT parando terminação / prematura.
Informática
ShapeFinder software permite ao usuário visualizar e transformar traços CE e convertê-los em perfis de reatividade SHAPE 18. Uma vez que os valores de reatividade são tabulados, eles são normalizados e importados para RNAStructure (v5.3) para gerar e refinar os modelos estruturais secundárias.
6. ShapeFinder Software
Uma extensão do BaseFinder traço processamento platformulário 29, a versão publicada ShapeFinder está disponível gratuitamente para uso não-comercial 18. Instruções detalhadas para manipulação de dados em ShapeFinder são fornecidos com a documentação do software.
- Eletroferogramas são importadas do CEQ em ShapeFinder, onde são ajustados para corrigir a (i) fundo fluorescente, (ii) sobreposição espectral entre canais fluorescentes, (iii) a mobilidade turnos transmitidas por primers de forma diferente com a tag, (iv) as diferenças na intensidade de fluorescência de produtos comuns marcados com diferentes fluoróforos, e (v) de decaimento do sinal resultante a partir de terminação prematura da transcrição reversa.
- A função "Setup" do "Alinhar e integrar" ferramenta ShapeFinder atribui automaticamente identidades picos individuais e correlaciona este para a seqüência do RNA, conforme definido pela entrada do usuário e as duas escadas de sequenciamento. Embora as atribuições iniciais são geralmente imperfeita, os erros podem ser corrigidos manualmente utilizando a função "Modificar" dea mesma ferramenta. Finalmente, a função de "Ajuste" calcula a área sob a alinhados (+) e (-) picos de reacção, e tabula estes valores de reactividade juntamente com o número de nucleótidos correspondente num ficheiro de texto por tabulação.
Nota: A análise dos dados é crítica para a exactidão de forma e, em algumas considerações são muito importantes durante esta análise, incluindo:
- Signal-to-noise: A relação sinal-ruído deve ser tal que os picos individuais devem ser facilmente identificáveis, mesmo para cargos com baixa reatividade. Embora ShapeFinder fornece uma opção de dados de alisamento; essa alternativa deve ser usado com cautela extrema, uma vez que pode análise subsequente inclinação.
- Região de análise: Normalmente, os dados fiáveis podem ser obtidas a partir de cDNAs 300-600 nt de comprimento, a partir de uma região de 40-80 nt removido do iniciador 3 'terminal e terminando como os decaimentos de sinal para níveis difíceis de distinguir do ruído de fundo. O uso de múltiplasconjuntos de iniciadores PLE serão necessários para analisar trechos longos de RNA. Neste caso, recomenda-se que a sobreposição do sinal fiável entre conjuntos de iniciadores é na gama de 30-50 nt. Em RNAs curtos, onde a transcriptase inversa frequentemente atinge o final do molde de ARN, deve ser tomado cuidado para excluir aqueles picos cuja relação sinal-ruído é afectada pela síntese de DNA de paragem forte.
- Decaimento do sinal: O decaimento do sinal está relacionada com a extensão da modificação de ARN durante a experiência bem como a processabilidade imperfeita da RT. Idealmente, a cinética de um único hit relativos à região do RNA a ser analisado deve ser conseguido de modo a maximizar o comprimento ler. Shapefinder contém uma ferramenta que é eficaz na correção para o decaimento do sinal, no entanto, porque isso tende a introduzir um erro na análise - especialmente quando cinética single não forem observadas, é melhor utilizado quando decadência sinal é mínima (ou seja, quando a distribuição de picos é consistente com um único hit vacastiques). Recentemente, algoritmos melhorados para transformar sinal sinal de decadência foram publicados 30 e deve ser investigado se a cárie é sinal de preocupação especial em uma experiência particular.
- Sinal de escala. Provavelmente o passo mais arbitrário no processamento de dados de formato, o perfil de controlo devem ser dimensionadas de modo a que as intensidades de pico entre minimamente reactivo (+) e (-) vestígios são iguais. Escalando o traço de controle para uma extensão muito grande resultará em uma abundância de valores de reatividade negativa no primeiro quartil (ver Normalização de dados abaixo). Neste caso, o fator de escala deve ser reduzido e os dados reintegrados.
- Peaks atribuição. Em geral, a versão automatizada da atribuição pico funciona bem. Quando o processo falha, no entanto, é imperioso que o utilizador se assegure de que todos os picos foram reconhecidos pelo software, em particular quando a relação sinal-ruído é baixo. Picos de ombro, por exemplo, nem sempre são detectados, e G-rico SEconsequências muitas vezes são compactados.
7. Normalização de dados
Para incorporar perfis de reatividade de nucleotídeos no algoritmo estrutura secundária utilizada por (v5.3) RNAStructure software, e / ou comparar os perfis de RNAs intimamente relacionados, Dados da Forma deve ser normalizado de forma padronizada 12. Isto envolve (i) a partir de valores extremos excluindo cálculos subsequentes, (ii) a determinação da reactividade "máxima eficaz" (ou seja, a média de 8%, o mais alto valor de reactividade, excluindo outliers), e (iii) dividindo normalização todos os valores de reactividade o "máximo efectivo", como se segue:
- Abra o arquivo de texto delimitado por tabulação gerada após o alinhamento e integração e copiar o seu conteúdo em uma planilha do Excel. A coluna mais à direita deste arquivo (RX.area-BG.area) contém os valores de reatividade SHAPE absolutos calculados para cada nucleotídeo do RNA. As colunas mais à esquerda relacionar esta reatividade ao sequ RNAcia.
- Calcular e armazenar o primeiro e terços quartil (ou seja, o percentil 25 e 75) os valores para (RX.area-BG.area) utilizando a função de Excel "= quartil (array, quart)"
- Calcular e armazenar a diferença interquartil "= quartil (array, 3)-quartil (array, 1)"
- Calcular e armazenar o "valor de corte outlier", usando a fórmula "= (QUARTILE, array, 3) 1,5 * ((quartil (array, 3)-quartil (array, 1))". Todos reatividade valores superiores a esse valor são ser excluídos dos cálculos subseqüentes.
- Copiar os valores de reactividade (RX.area-BG.area) e colá-las, uma coluna vazia adjacente, em seguida, separar estes valores tais que a maior estão no topo da coluna.
- Na coluna "valores ordenados" recém-criado, excluir valores maiores que o valor de corte outlier.
- Calcular e armazenar a média de 8% a maior reactividade dos valores restantes do "classificados valores column ". Este valor é o" reatividade máxima efetiva ".
- Divida o indiferenciados (RX.area-BG.area) de cada nucleotídeo (incluindo outliers) pelo valor "máximo eficaz" reatividade para obter os "valores de reatividade normalizado". Armazenar estes em uma coluna vazia, deixando uma coluna vazia para a esquerda. Em seguida, copiar os nucleótidos números à esquerda da tabela e colá-los na coluna vazia directamente à esquerda de "os valores de reactividade normalizados".
- Copie e cole os pares de valores de reatividade posição normalizados nucleotídeos em um editor de texto.
- Eliminar a valores abaixo de -0.09 (isto é, deixar o espaço em branco), uma vez que estes são provavelmente o resultado de RT parando durante a síntese de cDNA para outros fins que a modificação química do molde razões. Além disso, quaisquer valores de reactividade de nucleótidos em que se observa um forte parando no modelo não modificado (como determinado por inspecção visual de "alinham e Integrar" perfil ShapeFinder), deve ser excluída.
- Save o arquivo com a extensão. forma para uso em análise estrutural (v5.3) software RNAstructure.
8. Modelagem de Dados
(V5.3) RNAstructure software é utilizada para prever a estrutura secundária de RNA experimentalmente suportada (s), utilizando as restrições energéticas pseudo-livres derivados de análise de formas 19. O software proporciona representações gráficas das melhores estruturas de energia 2D de RNA, bem como representação textual destas estruturas em notação de ponto-suporte. Este último pode ser importado em um visualizador de estrutura RNA de preferência do usuário, por exemplo, Pseudoviewer 23 ou 22 de Varna, para produzir imagens com qualidade de publicação.
Nota: Cuidados devem ser tomados quando se considera as estruturas produzidas pelo (v5.3) software RNAstructure. Por exemplo, o software não pode resolver interações terciárias, tais como pseudoknots e loops de beijo, nem pode distinguir se a falta dereatividade em uma determinada região é devido à proteção emparelhamento de bases ou estérico por proteínas ligadas. Como consequência, estes factores, em conjunto com as energias reportados para as estruturas individuais, deve ser considerado quando se apresenta um modelo estrutural definitiva.
Resultados
RNA contendo o HIV-1 elemento de resposta rev (RRE) e uma extremidade 3 'da cassete estrutura do terminal 4 foi preparado a partir de um plasmídeo linearizado por transcrição in vitro, depois do que foi dobrado por meio de aquecimento, arrefecimento, e incubação a 37 ° C, na presença de MgCl 2. O ARN foi exposto a NMIA e depois a transcrição reversa a partir de um iniciador de ADN 5 '-fim-marcadas hibridadas com cassete de estrutura do terminal 3'. A bibliotec...
Discussão
Apresentamos aqui um protocolo detalhado para SHAPE high-throughput, uma técnica que permite a determinação da estrutura secundária a resolução de nucleotídeo único para RNAs de qualquer tamanho. Além disso, o acoplamento de dados experimentais com SHAPE secundárias algoritmos de previsão de estrutura facilita a geração de modelos de RNA 2D com um maior grau de precisão do que é possível com qualquer um dos métodos sozinho. A combinação de iniciadores marcados com fluorescência e automatizado CE prop...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch e SFJ Le Grice são apoiados pelo Programa do National Cancer Institute, National Institutes of Health, EUA Research intramuros.
Materiais
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
Referências
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados