É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagens de fluorescência com um nanômetros Precisão (Fiona)
Neste Artigo
Resumo
Fluorophores individuais podem ser localizados com precisão nanométrica usando FIONA. Aqui um resumo da técnica FIONA é relatado, e como a realização de experimentos Fiona é descrito.
Resumo
Imagens de fluorescência com uma precisão de um nanômetro (Fiona) é uma técnica simples, mas útil para localizar fluorophores individuais com precisão nanométrica no plano xy. Aqui um resumo da técnica FIONA é relatado e exemplos de pesquisas que têm sido realizados utilizando FIONA são descritas resumidamente. Em primeiro lugar, como configurar o equipamento necessário para experimentos de FIONA, ou seja, um total de microscopia de fluorescência de reflexão interna (TIRFM), com detalhes sobre o alinhamento da ótica, é descrito. Em seguida, como para realizar uma experiência simples fIONA em localizar moléculas imobilizadas com Cy3 de ADN individuais utilizando os protocolos apropriados, seguindo-se a utilização de fIONA para medir o tamanho do passo de 36 nm, de um único motor de miosina Va truncado marcado com um quantum dot, é ilustrado. Por fim, o esforço recente para estender a aplicação da FIONA para amostras espessas é relatado. Mostra-se que, utilizando uma objectiva de imersão em água e pontos quânticos embebido profundamente em sol-gel e córneas do olho de coelho (>200 mm), a precisão da localização 2-3 nm pode ser atingida.
Introdução
Por volta de 1882, Ernst Abbe descobriu que a resolução de um microscópio de luz visível é ~ λ / 2NA, ou ~ 200 nm (onde λ é o comprimento de onda e NA é a abertura numérica) 1,2. Por conseguinte, qualquer objecto mais pequeno do que esta dimensão que aparecem como uma mancha de difracção limitada, no microscópio óptico. No entanto, é possível determinar o centro do ponto, isto é, a localização do objecto, com uma precisão muito superior 3. Imagens de fluorescência com uma precisão de um nanômetro (Fiona) é uma técnica simples, mas útil para localizar fluorophores individuais com precisão nanométrica no plano xy 4. A precisão da localização, σ μ (ou seja, o erro padrão da média), depende do número total de fotões recolhidos, 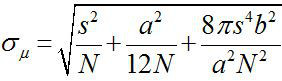 , Onde N é o número de fotões, s é o desvio padrão da mancha fluorescente, um éo tamanho do pixel do detector de imagens, e b é o desvio padrão do fundo de 3,4. Para um fluoróforo que emite fótons ~ 10.000, FIONA pode alcançar ~ 1 nm precisão 4.
, Onde N é o número de fotões, s é o desvio padrão da mancha fluorescente, um éo tamanho do pixel do detector de imagens, e b é o desvio padrão do fundo de 3,4. Para um fluoróforo que emite fótons ~ 10.000, FIONA pode alcançar ~ 1 nm precisão 4.
FIONA pode ser utilizada para determinar com precisão a posição de um emissor estacionário, ou um movimento (assumindo que as imagens podem ser feita suficientemente rápida). FIONA pode ser aplicado, sequencialmente, para os quadros do filme e, assim, controlar o movimento da única molécula 4 a 8. Reagentes foto-proteção pode ser necessário garantir que a amostra não photodegrade. Além disso, o próprio objecto fluorescente pode ser de qualquer tamanho, menor ou maior do que a difracção LIMIT- por exemplo, pode consistir de um organelo (~ 1 mm) com muitas proteínas fluorescentes dispersas sobre a sua membrana. Usando FIONA ainda pode render um (nanômetros) média muito precisos de sua média de centro de massa. A grande melhoria na precisão de localização por Fiona permite resolver nanometer movimentos escala ao longo do tempo. Isso levou microscopia na escala de comprimento molecular 4 a 8.
Desde a sua invenção, as variantes do fIONA têm sido desenvolvidos. Por exemplo, a imagem de campo claro com uma precisão de um nanômetro (bFIONA) 9, uma ligeira variante da FIONA, imagens e localiza objetos densos como melanossomas in vivo (objetos escuros contendo o pigmento melanina) com luz transmitida. Além disso, fIONA tem sido empregue para resolver vários corantes. Por exemplo, uma única molécula de alta resolução de imagem com fotodegradação (camarão) 10,11 ou uma única molécula de alta resolução co-localização (SHREC) 12 foram desenvolvidos para resolver dois corantes dentro de cerca de 10 nm. (Note-se que esta é a resolução, ou seja, a precisão com que se pode contar corantes idênticos separados.) Mais recentemente, a análise FIONA tem contribuído para o processo de localização de certos microscopia de super-resolução, como reco óptica estocásticamicroscopia nstruction (STORM) 13 e 15 e microscopia de localização ativados por foto (PALM) 16, em que fluorophores escuras temporárias está animado, e, em seguida, a fluorescência é localizada. Por várias vezes bastante interessante uma baixa densidade de corantes (menos do que um por difracção limitada local), e, em seguida, recolhendo a fluorescência, a análise de cada um deles por fIONA, pode-se construir um mapa de alta resolução. A resolução é, então, apenas limitado pelo número de fotões cada corante coloca fora, bem como coisas como manter a amostra estacionária (incluindo, por exemplo, a fase de microscópio), durante a aquisição.
Neste trabalho, um resumo da técnica FIONA e descrever brevemente exemplos de pesquisas que têm sido realizadas utilizando FIONA é relatado. Em primeiro lugar, como configurar o equipamento necessário para experimentos de FIONA, ou seja, um total de microscopia de fluorescência de reflexão interna (TIRFM), com detalhes sobre o alinhamento da ótica, é descrito. Então, comorealizar uma experiência simples FIONA em localizar moléculas imobilizadas Cy3-DNA único meio de protocolos apropriados, é ilustrado. Depois disso, o uso de fIONA para medir o tamanho do passo de 36 nm, de um único motor de miosina Va truncado marcado com um quantum dot é apresentado. Miosina Va é uma proteína motora processive essencial que transporta carga celular enquanto translocação ao longo dos filamentos de actina. Aqui uma miosina Va construir truncada é utilizado para remover os domínios irrelevantes para o tamanho do passo, e com um marcador FLAG adicionado ao terminal C para permitir a facilidade de marcação com quantum dots funcionalizados com anticorpos anti-FLAG. Esta experiência é realizada em condições de baixa ATP para abrandar a miosina e permitir a utilização de longos tempos de exposição suficiente para obter uma boa contagem de fotões em cada quadro. Qualquer marcador fluorescente suficientemente brilhante pode ser substituído no seguinte protocolo. Por fim, é relatado recente esforço de estender a aplicação da FIONA a amostras de espessura. Como princípio de prova de, pontos quânticos foram embebidosem sol-gel e córneas do olho de coelho e, em seguida, analisados e localizado com Fiona. Para imagem, uma objectiva de imersão em água 60X com NA = 1.2 foi usado porque este objectivo tem uma distância maior do que trabalhar utilizado anteriormente objetiva de imersão em óleo de 100X. Para compensar a perda na ampliação da objetiva, uma lente extra-ampliação (3.3X ou 4.0X) foi inserido no caminho de emissões. Além disso, a epi-fluorescência (não TIR), microscopia precisa de ser utilizado para acesso a regiões profundas nas amostras grossas. Mostra-se que os pontos quânticos embebidos no fundo do sol-gel e em córneas do olho de coelho (Z> 200 mM) podem ser localizados com precisão 2-3 nm.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Declaração de Ética: O tecido da córnea de coelhos foi coletado de acordo com a Universidade de Illinois Institutional Animal Care e Uso diretrizes.
Configuração 1 TIRFM
NOTA: Utilizar óculos de laser de segurança o tempo todo.
- Certifique-se de que todos os componentes ópticos necessários listados na Lista de Materiais estão disponíveis e prontos para o alinhamento. Se necessário, usar substitutos com funções equivalentes de outras empresas. Certifique-se de que os espelhos e lentes devem ter (AR) revestimentos anti-reflexo que correspondem o laser em uso.
- Definir as alturas de todos os elementos ópticos com a altura do centro do microscópio para trás da porta.
- Monte a laser, obturador laser e filtro ND (s). Uso de filtros ND para atenuar a energia do laser para baixo tão baixo quanto possível, mantendo o feixe visível. Apertar os parafusos com chaves sextavadas apropriadas.
- Planeje um caminho do feixe e marcá-lo com fita adesiva ou marcador na mesa óptica (dotted linhas azuis na Figura 1). Para simplificar, manter caminhos retos ao longo de linhas de furos na tabela óptica.
- Local espelho M1 (Figura 1), na primeira vez em ângulo recto. Coloque as duas íris ao longo da segunda seção reta do caminho do feixe planejado. Ajuste a posição ea inclinação do M1 tal que o laser atravessa a íris.
- Local espelho M2 (Figura 1) no segundo turno ângulo recto. Inserir o expansor de feixe de 10x (L1 e L2, a Figura 1) ao longo da terceira secção do percurso linear. Ajustar a inclinação tal que o expansor de feixe é paralelo tanto a tabela e o caminho óptico do feixe planeado.
- Ajustar M1 e M2 de forma iterativa de tal forma que o laser passa a direito através dos centros das duas lentes de o expansor de feixe. Repita este passo até que o perfil do feixe do feixe expandido não é cortada de Gauss.
- Ajuste M1 para centralizar o feixe em L1 (Fifigura 1) e M2 para centrar a viga na L2 (Figura 1) de forma iterativa. Um perfil de feixe mau geralmente significa que o laser é cortada; usar uma folha de papel branca para bloquear o feixe depois do expansor de feixe para verificar o perfil do feixe com os olhos. Para a análise de alta precisão, use um profiler feixe óptico.
- Ajustar a distância entre L1 e L2 de tal modo que o feixe é colimado. Repita o passo 1.8 e 1.9, se necessário.
NOTA: Quando o tamanho do feixe não muda com a distância, o feixe é colimado suficiente para uma instalação TIRFM. Para melhorar ainda mais a colimação do feixe, ferramentas como um interferômetro de corte poderia ser utilizado. Um tamanho típico feixe depois da expansão é de aproximadamente 20 mm. - Obturador do laser. Desenrosque a objetiva do microscópio e parafuso em um alvo alinhamento fluorescente. Colocar espelhos M3 e M4 (Figura 1) para direccionar o feixe expandido na porta microscópio e sobre o espelho dicróico dentro da torre. Certifique-se de que o feixe de laser rebate tele dicróicas e para o teto.
- Ajuste M3 para centralizar a parte mais brilhante do feixe no alvo fluorescente, e M4 para ajustar a inclinação do feixe a ser vertical.
- Obturador o laser e parafuso o objetivo novamente. Se o alinhamento na etapa anterior é bem feito deve haver um ponto simétrico de sair do objetivo. Ajuste as inclinações do M3 e M4 para otimizar a potência do laser e perfil do feixe do objetivo.
- Montar uma câmera EMCCD ao microscópio e conectar a câmera a um computador. Inicie o software para a câmera.
- Montar uma amostra fluorescente (solução de fluoróforos) sobre o microscópio. Olhe para a mancha fluorescente brilhante na câmara. Verifique se o local não se desloque na tela como o foco é alterado.
- Coloque a lente TIR (L3, Figura 1) na fase de tradução XYZ, a uma distância a partir do plano focal posterior da objectiva, que é igual ao comprimento focal de L3 (~ 30 cm). Ajuste a posição da L3de tal forma que o laser passa através do centro da lente.
- Traduzir L3 ao longo do percurso do feixe para ajustar a colimação de feixe. Certifique-se de que o feixe ainda está centrada no monitor e simétricos em forma.
NOTA: A área de iluminação no plano da amostra aumenta com a diminuição da lente TIR comprimento focal. Em geral, use o comprimento focal menor que poderia caber no set-up. - Traduzir L3 perpendicular à trajetória do feixe de inclinar a trave do objectivo. Manter traduzindo a lente de modo a que TIR TIR é alcançado. Observar uma amostra de partículas fluorescentes através da câmera EMCCD, e afinar L3 para obter um bom SNR.
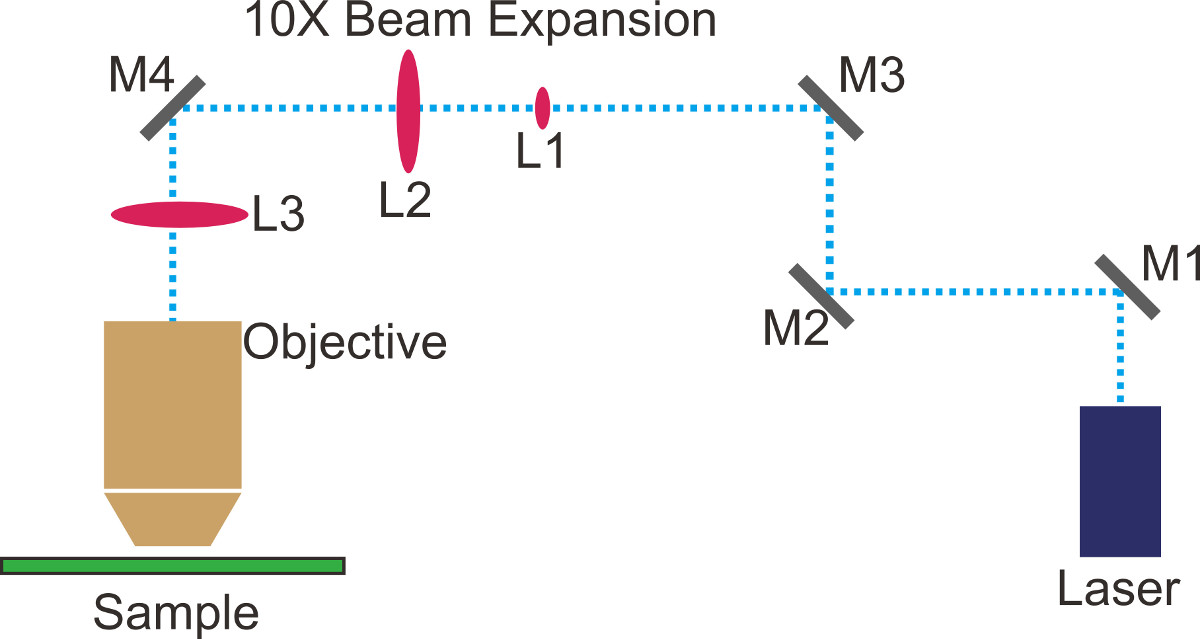
Figura 1 configuração óptica para microscopia de fluorescência de reflexão interna total (TIRFM).
2. fIONA em Cy3 ADN
- Clâminas de microscópio e magras lamelas: Lavar lâminas de microscópio e lamelas com DDH 2 O e isopropanol e seque-os com gás nitrogênio; coloque as lâminas e lamelas do limpador de plasma por 5 min sob plasma de argônio.
- Construir câmaras de amostras (como esboçado na Figura 2).
- Coloque um tecido sobre o banco e, em seguida, colocado um pedaço de papel de lente na parte superior do tecido. Coloque o slide no papel lente. Certifique-se de que o lado limpo do slide é para cima.
- Aplicar duas tiras de fita dupla face para o slide ao longo das bordas longas, deixando um espaço de 3-5 mm no centro. Coloque a lamela limpa em cima do slide. Certifique-se de que o lado limpo da lamela está voltada para o slide.
- Use uma ponteira para pressionar para baixo sobre a fita dupla face. Use uma navalha para remover o excesso de fita a partir da corrediça de modo que a fita se mantém apenas sob a lamela.
NOTA: As extremidades abertas da câmara são deixadas abertas e servem como entrada e para foradeixar. O volume da câmara é de vários microlitros.

Figura 2 Esboço de uma câmara de amostra típica (a) Vista superior.; (B) Vista lateral da direita; (C) Vista lateral da parte dianteira.
- Imobilizar Cy3 DNA em superfícies internas das câmaras de amostra.
- Preparar tampão de T-50 (10 mM Tris-HCl pH 8,0, 50 mM de NaCl). Prepare a BSA-biotina em T-50 a uma concentração final de 1 mg / ml. Preparar tampão de T50-BSA por dissolução de BSA em T-50 a uma concentração final de 10 mg / ml.
- Prepare a 0,5 mg / ml de neutravidina em tampão de BSA-T50. Prepare ADN biotinilado marcado com Cy3 (Cy3-ADN) para T50-BSA numa concentração final de 5-10 pM.
- Pipetar 10 ul de BSA-biotina (1 mg / ml) para dentro da câmara da amostra. Espere por 5 min.
- Lava-se a câmara com 40 ul de BSA-T50. Pipeta neutravidina 10 ul (0,5 mg / ml) para dentro da câmara da amostra. Incubar durante 5 min.
- Lava-se a câmara com 40 ul de BSA-T50. Pipetar 20 ul Cy3 ADN para dentro da câmara da amostra. Incubar durante 5 min e em seguida a câmara de lavagem com 80 ul de BSA-T50.
- Imagem Cy3-DNA moléculas individuais sob TIRFM.
- Preparar tampão de imagem (100 ul) por mistura de 1 mL Protocatecoate-3,4-dioxigenase (PCD, 5 mM), 4 uL de ácido protocatecuico (APC, 62,5 mM), 50 ul 6-hidroxi-2,5,7,8- ácido (Trolox, 2 mM em T-50) carboxílico tetramethylchromane-2-, e 45 ul de BSA-T50.
- Pipeta em tampão de imagem de 30 mL e esperar por 8-10 min.
- Montar a amostra para imagiologia numa TIRFM que é equipado com um laser verde (532 nm), um objectivo de imersão em óleo de 100X (NA 1,45), e uma câmara EMCCD.
- Defina o tempo de exposição a 100 a 500 ms e ganho de EM de 25 a 100 Adquirir um filme da amostra para 1,000 quadros.
- Realizar análise de dados FIONA nas imagens gravadas de Cy3-DNA.
- Determine o tamanho efetivo de pixels (ou seja, fator de conversão de pixels para nanômetros), dividindo o tamanho do pixel físico (leia-se a partir da folha de especificações da câmera EMCCD) pela ampliação total (a ampliação da objetiva do microscópio multiplicado por quaisquer aumentos extras).
- Determinar o fator de conversão de intensidades de pixel para fótons números dividindo sensibilidade CCD (ie, elétrons por contagem de A / D, lido a partir da folha de especificação da câmera CCD) pelo multiplicador de elétrons (EM) ganho utilizado na aquisição da imagem.
- Compilar e executar FIONA.pro para análise FIONA. Utilize este programa IDL para importar a imagem adquirida, para introduzir o tamanho do pixel eficaz (a partir do Passo 2.5.1) e do fator de conversão de intensidade ao número de fótons (a partir do Passo 2.5.2), e escolher os pontos para análise de FIONA.
NOTA: No final, o prograsaída m vontade os resultados de montagem, com funções 2D de Gauss, bem como o número total de fótons e precisão de localização. Um resultado típico é mostrado na seção de resultados representativos e Figuras 4c-4d. - Compilar e executar phcount.pro caracterizar contagem de fótons. Utilize este programa IDL para medir o número médio de fótons emitidos por um fluoróforo antes de fotodegradação, para importar a imagem adquirida e para introduzir o fator de conversão de intensidade ao número de fótons (a partir do Passo 2.5.2).
NOTA: O programa irá então detectar pontos fluorescentes automaticamente (seleção manual é uma opção), calcular contagens de fótons em função do número de quadro e saída dos vestígios de contagens de fótons.- Descarte traços ruins e especificar intervalos de quadros após a fotodegradação para correção de linha de base. No final, o programa irá exibir uma lista de números de fótons no total para todos os pontos não descartada. Em seguida, traçar a distribuição dos números de fótons e de ajustar a distrição com um decaimento exponencial para se obter o número médio de fotões. Um resultado típico é mostrado na seção de resultados representativos e Figuras 4e-4f.
3. FIONA aplicada para quantificação de Motor (por exemplo, a miosina na actina) Dynamics em nanômetro Scale
- Polimerizar a actina (por exemplo, preparar a F-actina) um dia antes da experiência fIONA.
- Reconstituir G-actina (monómero) e biotina G-actina (monómero) para 10 mg / ml com tampão de actina geral. Mexa para certificar-se de ambos são completamente dissolvido, e mantenha-os no gelo.
- Misturar 10 mL de G-actina (monómero) com 1,7 mL de biotina G-actina em um tubo de microcentrífuga de 1,5 ml. Adicionar 100 ul de tampão de polimerização de gelo frio de actina.
- Deixar a mistura durante a noite a 4 ° C (F-actina formados) e, em seguida, adicionar ddH2O para um volume total de 1 ml.
- Armazenar os filamentos de actina (actina F) a 4 ° C para posterior utilização em experiências.
NOTA: Filamentos irá desintegrar e diminuir ao longo do tempo, mas pode ser utilizado para, pelo menos, duas semanas.
- Prepare a amostra para a imagiologia.
- Faça uma câmara de amostra (conforme descrito no protocolo 2.1 e 2.2). Pipeta de 20 ul de BSA biotinilado a 1 mg / ml em ddH 2 O. Incubar durante 10 min. Lavar com 30 mL DDH 2 O.
NOTA: Isto bloqueia a superfície do vidro e estabelece biotina para a ligação dos filamentos de actina. Biotinilado poli-L-lisina - polietileno glicol (PEG-PLL) pode ter a mesma função. - Pipeta em 0,5 mg / ml neutravidina. Incuba-se durante 2 min e, em seguida, a câmara de lavagem com tampão 30 ul M5.
- Pipeta no preparado de F-actina diluído 25 vezes em tampão de actina geral, a concentração final de ~ 0,004 mg / ml. Aguarde 10 minutos, lave a câmara com 30 ul de tampão.
- Diluir miosina Va com etiquetas FLAG 30 vezes em tampão de M5 (20 mM de HEPES (pH 7,6), MgCl2 2 mM, KCl 25 mM, EGTA a 1 mM) a uma concentração final of 250 nM. Misture 1 ml miosina com 1 ml Anti-FLAG-Qdot705 (~ 1 PM, conjugado de anticorpos Anti-Flag e Qdot705 usando o Qdot705 Antibody Conjugação Kit de acordo com o manual de instruções do fabricante). Adicionar em 8 mL M5 para preencher a 10 ul. Pipeta cima e para baixo para misturar bem. Incubar durante 10 min em gelo.
NOTA: Isso produz uma mistura de 1 motor a 4 pontos quânticos, em ~ 25 nM concentração miosina.
- Faça uma câmara de amostra (conforme descrito no protocolo 2.1 e 2.2). Pipeta de 20 ul de BSA biotinilado a 1 mg / ml em ddH 2 O. Incubar durante 10 min. Lavar com 30 mL DDH 2 O.
- Imagem da miosina andando em actina.
- Preparar tampão de imagem (100 ul) por mistura de 84 ul M5-BSA (tampão M5 com 1 mg / ml de BSA), 1 ul de ATP (50 uM em ddH2O), 2 ul de DTT (500 mM em ddH2O), 1 CK ul (500 U / ml), 5 ul de PB (200 mM), 1 ul de PCD, 4 ul APC, 1 ul da miosina-Qdot depois diluído outra de 10 vezes a concentração de 2,5 nM de miosina, e 1 ul de BME.
- Pipeta em tampão de imagem de 20 mL de amostra de câmara e incubar por 8-10 min.
- Imagem da amostra em TIRF microscópio a 30 mseg exposição. Adquirir pelo menos 1.000 quadros. Ajuste o volume da miosina-Qdot no Passo 3.3.1, se necessário.
- Realizar análise de dados e encontrar o tamanho do passo de miosina andar.
- Abra o arquivo de vídeo no ImageJ 17 e recortar o vídeo em torno de um ponto em movimento. Cortar uma área grande o suficiente para que o local nunca fica dentro de 20 pixels da borda, e certificar-se de que não há outros pontos no vídeo. Certifique-se de que este local está se movendo em um caminho linear.
- Acompanhe o local através do vídeo para gerar coordenadas X e Y ao longo do tempo, em pixels, através da aplicação de análise FIONA (Passo 2.5.3) para cada quadro do vídeo.
- Converta pixels de nanômetros, como descrito na seção anterior.
- Calcular deslocamento a partir da posição inicial, em função do tempo.
- Executar o teste t sobre o deslocamento para obter os passos de miosina curta.
NOTA: O programa previsto t-teste (step_t_test.zip) é codificado em IDL e consiste de 14 sub-rotinas na pasta.- Abra todas as sub-rotinas em IDL e compilar tudo duas vezes. Em seguida, execute mtltyanalysis_ttest.pro e escolher um arquivo de texto contendo apenas os dados de distância em uma única coluna. Um arquivo de saída Excel serão gerados, contendo os dados brutos, o ajuste eo tamanho do passo.
- Exclua todos os valores nulos da coluna passo-size. Traça-se a distribuição dos tamanhos de passo utilizando Origem ou MATLAB. Coloque um Gaussian para o histograma.
NOTA: Passo tamanhos de valores de zero precisa ser suprimida por um passo de zero significa que nenhum passo é feita a partir do quadro anterior. O encaixe apresenta um pico de cerca de 36 nm (como mostrado na Figura 5).
4. Grosso Preparação de Amostras para FIONA
- Prepare quantum dots encapsulados em sol-gel.
- Misturar 4,5 ml de TMOS, 1 ml de ddH 2 O e 100 mL de HCl (120 mM). Sonicar a mistura em gelo durante 30 min no banho de ultra-sons specified na Lista de Materiais (frequência = 40 kHz, aquecedor = off). Misture a solução cada 10 minutos.
- Diluir 1,5 mL Qdot605 em 1,5 ml de HEPES (50 mM, pH 7,2). Misturar a solução com 1,5 ml de TMOS da etapa anterior.
- Despeje a mistura em um prato fundo de vidro. Selar o prato fundo de vidro com Parafilm e armazenar a 4 ° C durante 1 5 horas.
- Adicionar 2 ml de 1% de BME em PBS para o prato de amostra e incubar à temperatura ambiente durante 30 min antes da imagiologia.
- Prepare amostra córnea manchada com pontos quânticos.
NOTA: Coelho-olhos foram presentes de Dr. Marina Marjanovic.- Separe a córnea dos olhos e corte-o em 3 mm por 3 mm peças.
- Dilui-se 1 ml de estreptavidina-Qdot 605 em 1 ml de PBS. Incubar o tecido da córnea com uma solução a 4 nM Qdots C durante 1 hora. Lavar o tecido com PBS.
- Dê uma lâmina de vidro limpa e uma lamela # 1.5. Coloque 4 camadas de fita dupla face na lâmina ao longo do lado mais longo, umnd colocar mais de 4 camadas de fita de dupla face paralela à fita anterior e deixa um canal de cerca de 1 cm de entre. Colocar o tecido a partir da etapa anterior, no meio do canal, e cobri-lo com uma lamela.
- Pressione suavemente sobre os lados da lamela para fazer colar as fitas. Molhe o canal com 50 mL PBS antes de imagem.
- Quantum dots imagem em sol-gel e na córnea.
- Para imagens FIONA em amostras espessas, use uma objectiva 60X de imersão em água com distância de trabalho de 0,27 mm, ou um objetivo 60X de imersão em água com distância de trabalho de 0,28 mm.
- Monte a amostra no microscópio. Ajustar a lente TIRF tal que atinge o modo de epi-fluorescência (isto é, o feixe de laser de sai da objectiva com um ângulo contra a lamela). Coloque uma lente de ampliação adicional (3.3X ou 4.0X).
- Mova o plano focal da objectiva para a posição z desejado (por exemplo,> 200 mm). Grava imagens de imóvelpontos quânticos na amostra.
- Realizar análise FIONA como descrito na seção 2.5 do presente protocolo.
Access restricted. Please log in or start a trial to view this content.
Resultados
A configuração típica TIRFM tipo objetivo é mostrado na Figura 3. Primeiro, amostra Cy3-DNA imobilizado na superfície foi fotografada. Uma imagem típica é mostrada na Figura 4a. A imagem foi feita com tempo de exposição de 0,5 seg, com ganho EM = 50 e sensibilidade CCD = 12,13 para a câmera. A função de espalhamento-ponto (PSF) de uma única molécula de DNA com Cy3 é mostrado na Figura 4b (a partir do local indicado pela seta da Figura 4-A),
Access restricted. Please log in or start a trial to view this content.
Discussão
Fiona é uma técnica para localizar a posição de um emissor fluorescente (fluoróforo orgânico ou quantum dot) com precisão nanométrica e resolução temporal de até 1 ms 4 a 8. Quando os fótons suficientes são coletados, esta técnica permite determinar a posição de um emissor fluorescente com muito mais precisão do que o limite de difração (~ 200 nm) e, portanto, esta técnica abre uma maneira de observar o que não tem sido visto com microscopia óptica convencional / tradicional 4 - 8.<...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi financiado pelo NIH Grants 068.625, Subsídios NSF 1063188 e Centro de Física de células vivas 0822613. agradecimentos especiais vão para o Dr. Marina Marjanovic no Instituto Beckman para Ciência Avançada e Tecnologia para o dom de olhos de coelhos.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Double-sided tape | 3M | ~75 µm thick | |
| EMCCD camera | Andor Technology | DU-897E-CS0-#BV | |
| Ultrasonic cleaner | Branson | 2510 | |
| Fluorescence filter set | Chroma | 49016 | |
| Actin polymerization buffer | Cytoskeleton | BSA02 | |
| Biotin G-actin | Cytoskeleton | AB07 | |
| G-actin | Cytoskeleton | AKL95 | |
| General actin buffer | Cytoskeleton | BSA01 | |
| Laser shutter (with driver) | Electro-Optical Products Corp. | SH-10-MP | |
| IDL | Exelis Visual Information Solutions | ||
| Neutravidin | Fisher Scientific | PI-31000 | |
| Coverslip | Fisherbrand | 22X30-1.5 | 0.16-0.19 mm thick |
| Microscope slide | Gold Seal Microslides | 30103X1 | 0.93-1.05 mm thick |
| Plasma cleaner | Harrick Plasma | PDC-001 | |
| Glass bottom dish | In Vitro Scientific | D35-20-1.5-N | |
| Cy3-DNA oligos | Integrated DNA Technologies | 5'-Cy3/GCCTCGCTGCCGTCGCCA-3'Bio | |
| Fluorescent beads | Invitrogen | T-7280 | |
| Qdot 605-streptavidin | Invitrogen | Q10101MP | |
| Qdot605 | Invitrogen | Q21301MP | |
| Qdot705 | Invitrogen | Q22021MP | |
| Qdot705 Antibody Conjugation Kit | Invitrogen | Q22061MP | |
| MATLAB | MathWorks | ||
| Optical table | Newport Corp | RS4000 Series | |
| 60X Objective | Nikon | Plan Apo VC 60x WI | |
| 100X Objective | Olympus | PlanApo 100X/1.45 Oil ∞/0.17 | |
| 60X Objective | Olympus | UPlanApo 60X/1.20W | |
| Inverted microscope | Olympus | IX71/IX70/IX81 | |
| Origin | OriginLab | ||
| Anti-FLAG antibody | Sigma Aldrich | F7425-.2MG | |
| ATP | Sigma Aldrich | A7699 | |
| BME | Sigma Aldrich | 63689-25ML-F | |
| BSA | Sigma Aldrich | A7906 | |
| BSA-biotin | Sigma Aldrich | A8549-10MG | |
| CK | Sigma Aldrich | C3755 | Creatine Phosphokinase from rabbit muscle |
| CP | Sigma Aldrich | P1937 | Phosphocreatine di(tris) salt |
| DTT | Sigma Aldrich | 43815 | DL-Dithiothreitol |
| EGTA | Sigma Aldrich | E3889 | Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid |
| HCl | Sigma Aldrich | 93363-500G | |
| HEPES | Sigma Aldrich | H0887 | |
| KCl | Sigma Aldrich | P9333 | |
| MgCl2 | Sigma Aldrich | M1028 | |
| NaCl | Sigma Aldrich | S7653 | |
| PCA | Sigma Aldrich | 03930590 | Protocatechuic acid |
| PCD | Sigma Aldrich | P8279 | Protocatechuate-3,4-dioxygenase |
| TMOS | Sigma Aldrich | 341436-25G | Tetramethyl orthosilicate |
| Tris-HCl | Sigma Aldrich | 93363 | |
| Trolox | Sigma Aldrich | 238813 | 6-hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| 1” diameter broadband dielectric mirrors with mounts | Thorlabs | BB1-E02, KM100 | Quantity: 2 |
| ½” diameter posts | Thorlabs | TR4 | Quantity ≥ 6 |
| 10X beam expander | Thorlabs | BE10M-A | |
| 2” diameter f = 300 mm lens with mount | Thorlabs | LA1256-A, LMR2 | TIR lens |
| Fluorescent alignment target | Thorlabs | VRC2SM1 | |
| Laser safety goggles | Thorlabs | LG3 | |
| ND filter(s) | Thorlabs | FW1AND | |
| Optical beam profiler | Thorlabs | BP209-VIS | |
| Post-mounted iris diaphragm | Thorlabs | ID25 | Quantity: 2 |
| Shearing interferometer | Thorlabs | SI100 | |
| XYZ translation stage, ½” travel | Thorlabs | T12XYZ | |
| Laser | World Star Technologies | TECGL-30 | 532 nm, 30 mW |
Referências
- Abbe, E. The Relation of Aperture and Power in the Microscope. Journal of the Royal Microscopical Society. 2 (3), 300-309 (1882).
- Abbe, E. The Relation of Aperture and Power in the Microscope (continued). Journal of the Royal Microscopical Society. 2 (4), 460-473 (1882).
- Thompson, R. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Yildiz, A., et al. Myosin V Walks Hand-Over-Hand: Single Fluorophore Imaging with 1.5-nm Localization. Science. 300 (5628), 2061-2065 (2003).
- Yildiz, A., Tomishige, M., Vale, R. D., Selvin, P. R. Kinesin Walks Hand-Over-Hand. Science. 303 (5658), 676-678 (2004).
- Yildiz, A., et al. Myosin VI Steps via a Hand-over-Hand Mechanism with Its Lever Arm Undergoing Fluctuations when Attached to Actin. Journal of Biological Chemistry. 279 (36), 37223-37226 (2004).
- Yildiz, A., Selvin, P. R. Fluorescence Imaging with One Nanometer Accuracy: Application to Molecular Motors. Accounts of Chemical Research. 38 (7), 574-582 (2005).
- Toprak, E., Yildiz, A., Hoffman, M. T., Rosenfeld, S. S., Selvin, P. R. Why kinesin is so processive. Proceedings of the National Academy of Sciences. 106 (31), (2009).
- Kural, C., et al. Tracking melanosomes inside a cell to study molecular motors and their interaction. Proceedings of the National Academy of Sciences. 104 (13), 5378-5382 (2007).
- Gordon, M. P., Ha, T., Selvin, P. R. Single-molecule high-resolution imaging with photobleaching. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6462-6465 (2004).
- Qu, X., Wu, D., Mets, L., Scherer, N. F. Nanometer-localized multiple single-molecule fluorescence microscopy. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11298-11303 (2004).
- Churchman, L. S., Ökten, Z., Rock, R. S., Dawson, J. F., Spudich, J. A. Single molecule high-resolution colocalization of Cy3 and Cy5 attached to macromolecules measures intramolecular distances through time. Proceedings of the National Academy of Sciences of the United States of America. 102 (5), 1419-1423 (2005).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3 (10), 793-796 (2006).
- Bates, M., Huang, B., Dempsey, G. T., Zhuang, X. Multicolor Super-Resolution Imaging with Photo-Switchable Fluorescent Probes. Science. 317 (5845), 1749-1753 (2007).
- Betzig, E., et al. Imaging Intracellular Fluorescent Proteins at Nanometer Resolution. Science. 313 (5793), 1642-1645 (2006).
- Abramoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics international. 11 (7), 36-42 (2004).
- Enderlein, J., Toprak, E., Selvin, P. R. Polarization effect on position accuracy of fluorophore localization. Optics Express. 14 (18), 8111-8120 (2006).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Rasnik, I., McKinney, S. A., Ha, T. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3 (11), 891-893 (2006).
- Zhuang, X., et al. A Single-Molecule Study of RNA Catalysis and Folding. Science. 288 (5473), 2048-2051 (2000).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados