Method Article
Escala atômica estudos estruturais de conjuntos macromoleculares por espectroscopia de estado sólido da ressonância magnética Nuclear
Neste Artigo
Resumo
Estruturas de conjuntos supramoleculares proteína em resolução atômica são de alta relevância, por causa de seus papéis cruciais em uma variedade de fenômenos biológicos. Neste documento, apresentamos um protocolo para realizar estudos estruturais de alta resolução em assemblies de proteína macromolecular insolúvel e não cristalina por magia-ângulo girando espectroscopia da ressonância magnética nuclear Solid-State (MAS SSNMR).
Resumo
Conjuntos supramoleculares proteína papel fundamental nos processos biológicos variando de interação patógeno-hospedeiro, infecção viral para a propagação de doenças neurodegenerativas. Esses assemblies consistem em múltiplas subunidades proteicas, organizadas de forma não-covalente para formar objetos macromoleculares grandes que podem executar uma variedade de funções celulares ou causar consequências prejudiciais. Atômicas insights sobre os mecanismos de montagem e o funcionamento desses assemblies macromoleculares frequentemente permanecem escassos desde sua insolubilidade inerente e não-cristalinidade reduz muitas vezes drasticamente a qualidade dos dados obtidos da maioria das técnicas de usado em biologia estrutural, tais como a cristalografia de raio-x e ressonância magnética Nuclear (NMR) de solução. Aqui apresentamos a espectroscopia de RMN Solid-State de magia-ângulo de giro (SSNMR) como um método poderoso para investigar estruturas de conjuntos macromoleculares em resolução atômica. SSNMR podem revelar detalhes atômicos no complexo montado sem limitações de tamanho e solubilidade. O protocolo aqui apresentado descreve as etapas essenciais da produção de 13C / conjuntos de isótopo-etiquetou a proteína macromolecular15N à aquisição de padrão SSNMR de espectros e sua análise e interpretação. Como exemplo, mostramos o pipeline de uma análise estrutural do SSNMR de um conjunto de proteínas filamentosas.
Introdução
Avanços na magia-ângulo girando espectroscopia da ressonância magnética nuclear Solid-State (SSNMR) para oferecer uma ferramenta eficiente para a caracterização estrutural de assemblies de proteína macromolecular em uma resolução atômica. Esses assemblies de proteína são onipresentes sistemas que desempenham funções essenciais em muitos processos biológicos. Suas estruturas moleculares, interações e a dinâmica é acessível através de estudos SSNMR, como foi demonstrado por viral (capsids1) e mecanismos de infecção bacteriana (secreção sistemas2,3, pili4), a membrana proteína complexos5,6,7,8 e funcional amiloides 9,10,11. Este tipo de montagem molecular pode também provocar patologias tais como em doenças neurodegenerativas onde proteínas montar nos Estados de misfolded, amiloides e causam comportamento aberrante cela ou célula morte 12,13. Assemblies de proteína são construídos frequentemente pela oligomerização simétrica de cópias múltiplas de subunidades proteicas em grandes objetos supramoleculares de várias formas, incluindo fibras, filamentos, poros, tubos ou nanopartículas. A arquitetura do quaternária é definida por interações fracas entre subunidades proteicas para organizar a montagem espacial e temporal e para permitir a sofisticadas funções biológicas. Investigações estruturais em uma escala atômica sobre esses assemblies são um desafio para técnicas de alta resolução desde sua insolubilidade intrínseca e muitas vezes sua cristalinidade-não restringe o uso de cristalografia de raios x convencional ou solução NMR aproxima-se. Magia-ângulo girando SSNMR (MAS) é uma técnica emergente para obter dados de resolução atômica sobre conjuntos macromoleculares insolúveis e provou sua eficiência para resolver 3D modelos atômicos para um número crescente de sistemas biomoleculares complexas incluindo filamentos bacterianos, assemblies amiloides e partículas virais 14,15,16,17,18,19,20, 21,22. Avanços técnicos na altos campos magnéticos, desenvolvimentos metodológicos e preparação da amostra estabeleceu SSNMR MAS em um método robusto para investigar proteínas insolúveis em vários ambientes, nomeadamente na sua biologicamente relevantes macromoleculares montados estaduais ou nas membranas celulares, tornando a técnica altamente complementares para microscopia cryo-elétron. Em muitos casos, um elevado grau de simetria caracteriza tais conjuntos de proteína. MAS SSNMR explora esse recurso, como todas as subunidades de proteína em uma montagem de homomolecular teria a mesma estrutura local e, portanto, praticamente a mesma SSNMR assinatura, reduzindo drasticamente a complexidade da análise.
Um protocolo eficiente para estudos estruturais de assemblies de proteína macromolecular por MAS moderadas (< 25 kHz) SSNMR é apresentado neste vídeo e pode ser subdividido em diferentes etapas (Figura 1). Vamos demonstrar as fases críticas de fluxo de trabalho de um estudo estrutural de SSNMR, exemplificado em um conjunto de proteínas filamentosas (consulte destacada as etapas na Figura 1), com exceção de purificação de proteínas subunidade, diferindo para cada proteína montagem, mas de fundamental importância para estudos estruturais e sem entrar nos detalhes técnicos/metodológicos de cálculo de estrutura e espectroscopia SSNMR de que tutoriais especializados estão disponíveis online. Enquanto o presente protocolo terá como principal foco no estado sólido NMR experimentos, realizados em condições MAS, o uso de alinhado ambientes biológicos 23,24,25,26 , 27, tais como bicelles alinhados, permitem a investigação de conformação de proteínas e interação dinâmica da proteína-proteína na membrana, como mídia sem tecnologia MAS. Vamos mostrar a expressão da proteína e passos de montagem, bem como a gravação de espectros de SSNMR cruciais e sua análise e interpretação. O nosso objectivo é fornecer insights sobre o pipeline de análise estrutural, permitindo o leitor a realizar um estudo estrutural de resolução atômica de um assembly macromolecular por técnicas SSNMR.
O protocolo abrange 3 seções:
1. produção de amostra NMR Solid-State
Como um pré-requisito para uma análise de NMR Solid-State, os componentes proteicos da montagem macromolecular precisam ser expressos, isótopo-etiquetadas, purificada e montado em vitro no estado complexo como de um nativo (para um ver exemplo Figura 2) . Para garantir alta sensibilidade NMR, enriquecimento de isótopo em 13C e 15N rotulagem é necessário através da utilização de meios de expressão bacteriana mínimo suplementados com 13C e fontes de N 15, como uniformemente 13 C-rotulado glicose/glicerol e 15NH4Cl respectivamente. Em fase posterior do protocolo, 13C-rotulado amostras produziram seletivamente com seletivamente 13fontes C-rotulados como (1,3 -13C)- e (2 -13. C)-glicerol (ou (1 -13. C)- e (2 -13. C)- glicose) são usados para facilitar a análise de NMR. Mista rotulada amostra correspondente a uma mistura equimolar de 50% 13C-rotulados ou 50% e 50% 15N - (1,3 -13C)- e 50% (2 -13. C)-glicose são introduzidos para descrever a detecção de intermoleculares interações. Um alto grau de pureza da proteína, bem como condições rigorosas durante a etapa de montagem são factores-chave para garantir uma ordem estrutural homogênea da amostra final.
2. preliminar caracterização estrutural baseada em NMR Solid-State de unidimensional (1D)
Apresentamos as experiências essenciais para uma análise estrutural por SSNMR. Unidimensional (1D) polarização cruzada (CP) e INEPT / experimentos de28 RINEPT, detectados no 13C núcleos são usados para detectar segmentos de proteína rígida e flexível na montagem, respectivamente e para estimar o grau de estrutural homogeneidade e polimorfismo local (para um ver exemplo, Figura 3).
rong > 3. Determinação da estrutura de análise conformacional e 3D
Subseções 1 e 2 dizem respeito a análise conformacional, que baseia-se na atribuição de ressonância SSNMR de todos os resíduos rígidos da Assembleia de proteína, como as mudanças químicas são sondas muito sensíveis ao ambiente local e podem ser usadas para prever a phi/psi diedro ângulos e, assim, determinar a estrutura secundária. A Figura 4 ilustra um exemplo de uma atribuição de ressonância sequencial no núcleo rígida de um conjunto de proteínas. A determinação da estrutura 3D baseia-se a recolha de dados estruturais tais como restrições de distância codificação fechem proximidades (< Å 7-9), que contém ambos intrae intermoleculares informações. Subseções, 3 e 4 descrevem interpretação e coleção de contenção distante de longo alcance. Contatos de longo alcance são definidos como intramolecular 13C -13C proximidades provenientes de resíduos a j, com | i-j | ≥4, definindo assim a dobra de proteína terciária de subunidade monomérica, ou como intermoleculares 13C -13C proximidades, definir as interfaces intermoleculares entre subunidades proteicas no assembly. Intrae intermoleculares interfaces são ilustrados na Figura 5. Apoios SSNMR detectado através de 13C -13C e 15N -13C reassociação experimentos geralmente codificam para distâncias internuclear < 1 nm. Subsecção 4 explica a detecção de restrições de distância intermolecular. Nas assembleias de proteína simétrica, o uso de amostras homogeneamente rotulados (ou seja, 100% uniformemente ou seletivamente rotulado) para identificar as interações intermoleculares subunidade-subunidade é limitado, como ambos intrae inter - molecular contatos levam a sinais detectáveis. A detecção inequívoca de proximidades intermoleculares é conseguida usando amostras etiquetadas mistas, contendo uma mistura equimolar de duas amostras diferente rotuladas, combinado antes da agregação. Subseção 5 introduz brevemente a modelagem da estrutura.
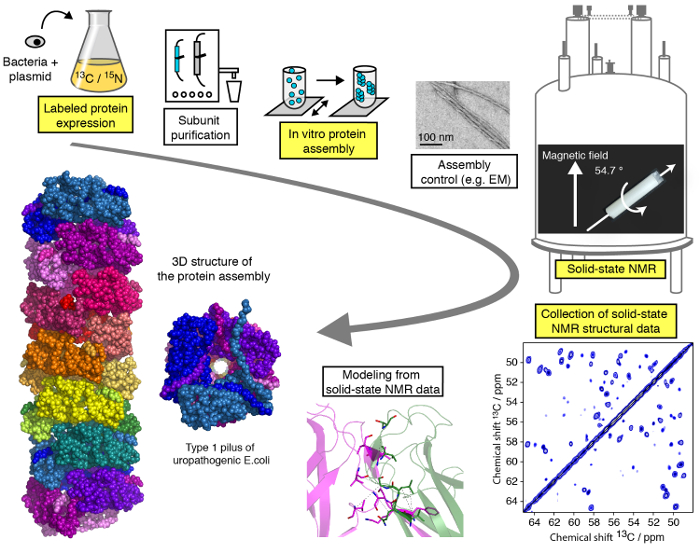
Figura 1 : Fluxo de trabalho de um estudo estrutural de resolução atômica por NMR Solid-State. 13 C, 15N isótopo rotulado de produção de proteína, purificação de subunidade, montagem de subunidade, controle de formação de montagem, SSNMR experiências, análise do experimento SSNMR e extração de restrições de distância, e modelagem da estrutura são mostrados. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. NMR Solid-State amostra productiona
- produção de uniformemente 13 C / subunidades de proteína N-rotulado de 15
- uso recém transformado, 6his-pET24-peptídeo contendo plasmídeo Escherichia coli BL21 (DE3) células, colhê-las da placa de ágar preparado.
- Inocular uma pre-cultura 15 mL de médio pré-aquecido de LB com uma colônia. Incubar a 37 ° C com 200 rpm agitação durante a noite.
- Transferir a pre-cultura para 1 L de cultura principal de pré-aquecido médio M9 (ver composição na tabela 1), contendo os necessário rotulado de isótopo carbono e nitrogênio fontes.
Nota: Isto pode ser uniformemente 13 C-rotulado glicose, seletivamente (1 - 13. C)- ou (2 - 13. C)-rotulado glicose 29 , 30 , 31 ou ( 1,3 - 13 C)- ou (2 - 13. C)-rotulado de 32 , de glicerol 33. - Isto, incubar a 37 ° C e medir o OD 600, assim como a cultura torna-se turva. Quando o OD 600 atingiu um valor de 0,8, induzir a expressão de proteínas com 0,75 mM IPTG para 4 h.
Nota: As condições de indução ideal podem variar de uma proteína para outra. - Recuperar as células por centrifugação por 30 min a 6000 x g e 4 ° C.
- Purificação sketch (não mostrado no vídeo protocolo)
- Lyse pilhas usando técnicas padrão, como tratamento sonication ou lisozima e purificar a subunidade de proteínas usando técnicas estabelecidas ou adaptados para o investigou a montagem da proteína.
Nota: A proteína pode ser expressa em corpos de inclusão, verifique para a localização da proteína por lise celular e posterior centrifugação. Se a proteína encontra-se no sedimento com os restos de células, tenha sido acumulado em corpos de inclusão. - Teste de expressão e a pureza da amostra da proteína por exemplo por um padrão de 12-15% Tris-Tricine SDS-PAGE, ver Figura 2A. Se a pureza é suficiente, a proteína pode ser montada, caso contrário, executar uma etapa de purificação complementar.
- Lyse pilhas usando técnicas padrão, como tratamento sonication ou lisozima e purificar a subunidade de proteínas usando técnicas estabelecidas ou adaptados para o investigou a montagem da proteína.
- In vitro de montagem dos filamentos da proteína
- Verifique a concentração da proteína na solução purificada. Se a proteína contém resíduos de absorventes, medir a absorção em um espectrofotômetro UV em 280 nm. Insira o tampão puro o espectrofotômetro como calibração e então medir a absorvância de proteína.
- Calcular a concentração de proteína usando o coeficiente de absorção de subunidade a proteína com a lei de Beer-Lambert adaptada: um λ = ε λ x l x c; aqui A é a ótica de densidade em um determinado comprimento de onda λ; ε é o coeficiente de absorção específico; l é o comprimento da trajetória do óptico em cm; c é a concentração em mol/L.
- Concentrar a proteína a uma concentração de 1 mM em uma unidade de filtro centrífugo. Introduzir a amostra na unidade de filtragem centrífuga e centrifugar a 4000 x g durante 30 minutos, misture a solução na unidade de filtro suavemente com uma pipeta para evitar a deposição de proteína no filtro. Repita o procedimento até atingir a concentração desejada.
Nota: A concentração ideal de montagem depende do sistema e precisa ser otimizado (geralmente entre 0,1 - 1 mM). Se um equimolar misturado rotuladas amostra de dois lotes diferentes proteínas etiquetados é preparada (por exemplo, (50/50) (U - 15 N)-/ (U - 13 C)-rotulados), mistura deve ocorrer antes ou logo após a etapa de concentração para garantir homogeneização da solução mista. - Transferir a amostra para um tubo e incube-lo sob agitação durante uma semana à temperatura ambiente.
- Geralmente a polimerização das subunidades em filamentos é acompanhada pela solução se tornar turva. A morfologia microscópica fibrila e homogeneidade por exemplo por microscopia electrónica (ampliação 6300 X - 45000 X), ver Figura 2B.
- Centrifugar a amostra por 1h em 20000 x g e 4 ° C. Remove a maioria do líquido sobrenadante, sair só líquido suficiente para cobrir a superfície para evitar a secagem da amostra. Verifique o sobrenadante para subunidades independentes sobre o espectrofotômetro UV.
- Lavar a amostra com a adição de H 2 O e cuidado resuspenda o conteúdo de uma pipeta. Fazer não o vórtice. Girar a amostra por 30 min em 22000 x g e 4 ° C. Repita a etapa de lavagem 3 vezes. Verifique durante todas as etapas de lavagem se o pellet preserva seu aspecto após a centrifugação e o sobrenadante para subunidades independentes sobre o espectrofotômetro UV.
- Adicionar 0,02% (p/v) de azida de sódio (NaN 3) para evitar a contaminação bacteriana e armazenar a amostra até a medição em 4 ° C.
- Rotor NMR Solid-State de enchimento
- centrifugar duas vezes durante 30 min a 22000 x g e 4 ° C. remover montante máximo do líquido sobrenadante; a consistência de amostra resultante depende da montagem da proteína e é geralmente uma gelatinosa depósito.
- Usar uma pipeta capilar para inserir a amostra para o rotor SSNMR.
Nota: Aqui, apresentamos o procedimento em um rotor de diâmetro de 4 mm. Centrífuga- centrífuga do rotor em uma tabela de introduzir a amostra suavemente o rotor. Repita o procedimento até que o rotor está cheia. Manter o mínimo espaço para fechar com a tampa do rotor o rotor. Se a quantidade de amostra não é suficiente, introduzir uma inserção comercialmente disponível para preencher o espaço restante para garantir a homogeneidade de distribuição de amostra no rotor.
Nota: Dependente sobre a consistência da amostra montado, pode ser mais adequado usar uma espátula fina, especialmente para amostras muito densas. Rotores com diâmetros de 2,5 a 4 mm são usados em frequências de MAS moderadas.
- centrífuga do rotor em uma tabela de introduzir a amostra suavemente o rotor. Repita o procedimento até que o rotor está cheia. Manter o mínimo espaço para fechar com a tampa do rotor o rotor. Se a quantidade de amostra não é suficiente, introduzir uma inserção comercialmente disponível para preencher o espaço restante para garantir a homogeneidade de distribuição de amostra no rotor.
- Adicionar traços de ácido 4,4-dimethyl-4-silapentane-1-sulfonic (DSS) para calibração de temperatura e mudança química interno.
- Fechar a tampa com um dispositivo de fecho.
- Verificar sob uma lupa, se a tampa está bem inserida e completamente fechada.
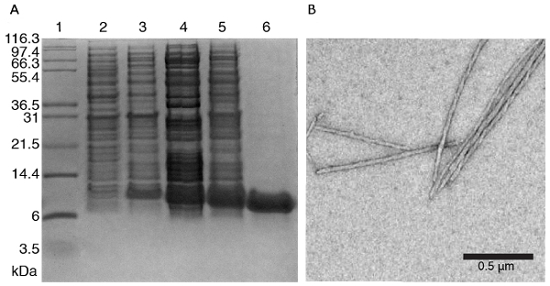
Figura 2: representante resultados para purificação de proteínas subunidade e montagem. A) 15% Tris-tricine SDS-PAGE de subunidade de proteína (incluindo seu 6-marca) em diferentes estágios de purificação. Pista 1 - marcador de peso molecular da proteína; pista 2 - Escherichia coli BL21 (DE3) células controle uninduced; faixa 3 - Escherichia coli BL21 (DE3) células induzidas com 0,75 mM IPTG; pista 4 - solubilizada corpos de inclusão faixa 5 - frações sobrenadante de célula lisada; faixa 6 - fração purificada depois níquel imobilizada cromatografia de afinidade de metal (IMAC) FPLC e dessalinização. B) negativamente coradas fibrilas de proteína por imagem TEM. clique aqui para ver uma versão maior desta figura.
2. caracterização estrutural preliminar baseada em NMR Solid-State de unidimensional (1D)
- set-up inicial e 1D polarização cruzada (CP)
- Inserir o rotor para o ímã NMR
- começar a girar o rotor em uma frequência de 5 kHz e esperar por uma estabilização de ± 10 Hz e, em seguida, acelerar até 7 kHz. Sintonize e corresponder a 1 H, 13 C e 15 N canais. Definir a temperatura de 2-10 ° C (275-283 K); amostra de aquecimento a 7 kHz MAS é baixa e ajuste de temperatura geralmente não é necessário nessa frequência MAS.
- Gravar um single-pulsado 1D 1 H espectro usando 16 varreduras.
Nota: Os níveis de energia devem anteriormente foram otimizados em um composto de referência (como um 13 C / < sup > 15 histidina N ou glicina), para fornecer valores de partida para otimizar os níveis de energia nos experimentos com a amostra de destino; Este procedimento é uma tarefa rotineira e, portanto, fora do âmbito do presente protocolo. - Manter a temperatura durante todos os experimentos entre 2-10 ° C, em amostras hidratados, que a temperatura é refletida na mudança relativa da ressonância em massa água 1 H no que diz respeito a DSS sinal 34. Medir a temperatura, seguindo a relação δ (H 2 O) = 7,83-T/96,9 com uma precisão de 1-2 ° C 35.
- Conjunto até o desejado MAS frequência e espere até a estabilização de ± 10 Hz.
Nota: Aqui está demonstrado por uma frequência de 11 kHz. - Tune e jogo 1 H e 13 C canais novamente e ajustar a temperatura com a ajuda de um experimento de 1 H 1 D.
- Configurar um D 1 1 H - 13 C CP 36.
Nota: O CP experimentos mostram os sinais provenientes de resíduos em uma conformação rígida. Calibração de pulso e dissociação parâmetros podem ser diretamente otimizados na amostra quando a sensibilidade é alta o suficiente para observar os sinais após menos de 32 ~ varreduras. Os valores iniciais de otimização são retirados a otimização padrão em um composto de referência. Tempo de contato do CP e os níveis de energia são escolhidos com base na intensidade de sinal máxima. Em amostras da proteína, o tempo de contato ideal CP é geralmente entre 200 µs e 2 ms. os parâmetros de dissociação também devem ser re-ajustados através da calibragem em um composto de referência.- Observar cuidadosamente a localização de fiação bandas laterais na frequência determinada MAS.
- Gravar um espectro CP C 13 D 1 referência que serve como impressão digital espectral 1D. Parâmetros típicos são 128 varreduras, 1 ms CP tempo de contato, 100kHz dissociação de força, um atraso de reciclagem de 3 s e 25 ms de aquisição.
- Calibrar a 1 H e 13 C mudanças químicas seguindo as recomendações de IUPAC 37. Determinar a frequência de ressonância do sinal 1 H DSS (shift química de 0 ppm); 13 C mudanças químicas são referenciadas para turnos de 1 H (normalmente ~ 0.25144, derivado a relação dos 13 C para o de frequência de ressonância de 1 H 37).
- Processo a 1D 1 H - 13 C CP experimentar sem apodização função (consulte a Figura 3A e B para a detecção de um assembly de proteína homogênea bem ordenada). Escolha um pico isolado para estimar o linewidth na amostra, indicativa de homogeneidade e de ordem estrutural. Típico de 13 C linewidths para assemblies bem-ordenado proteína biológica sob MAS em altos campos magnéticos variam entre 20-150 Hz (medido como a largura total a meia-altura (FWHH)).
- Estimar a relação sinal-ruído (S/N) no espectro CP.
Nota: A relação S/N depende de muitos fatores, incluindo principalmente a quantidade de amostra no rotor, o grau de rigidez da estrutura de proteínas e a presença de uma única conformação molecular. Como regra geral, uma amostra deve ser adequada para espectroscopia multidimensional SSNMR se um sinal é observado em um otimizado 1D 1 H - 13 C CP espectro gravado com 64 scans. - Zoom para a região espectral de carbonila de espinha dorsal da proteína ressonâncias (principalmente) localizadas entre 165-180 ppm. Estimar o conteúdo de estrutura secundária na assembleia pela posição das ressonâncias de carbonila de espinha dorsal da proteína com ressonâncias de resíduos em α-hélice ou conformação β-vertente deslocado para o campo ou o campo, respectivamente (ver Figura 3B para uma subunidade α-hélice principalmente) 38.
- D 1 INEPT experiência
- configurar um D 1 1 H - 13 experimento C INEPTO para sonda peças altamente móveis (sub-µs) da Assembleia proteína.
Nota: GARP dissociação de alguns kHz durante aquisição 39 é comumente usado, para permitir atrasos interscan curtos (tipicamente 1 s) sem danificar a sonda. - Gravar um espectro de referência INEPTO que serve como a impressão digital para os segmentos de proteína móvel; parâmetros típicos são 128 varreduras e um tempo de aquisição de 25 MS.
- Processo a 1 H - 13 C INEPTO experimentar. A quantidade e as posições dos sinais são indicativas da quantidade de resíduos móveis e a composição de aminoácidos, respectivamente.
Nota: Também gravar um 2D 1 H - 13 C INEPTO experimento se os sinais estiverem presentes no espectro INEPTO 1D (consulte a Figura 3, por exemplo) para permitir uma identificação mais específica de aminoácidos. Em casos de atribuição ambígua, recomendamos que verifique as posições de sinal de componente de reserva pela solução NMR. - Usar o banco de dados BMRB 40 e publicou dados 38 para atribuir as ressonâncias a sua correspondente aminoácido em perto de conformação de bobina aleatória; o espectro contém apenas componentes de móveis reserva se não resíduos são em um regime altamente móvel.
- configurar um D 1 1 H - 13 experimento C INEPTO para sonda peças altamente móveis (sub-µs) da Assembleia proteína.

Figura 3: resultados representativos de Aquisição de espectros SSNMR para um assembly de proteína bem estruturados. A) 13 C-detectado FID de um experimento de polarização cruzada 1 H - 13 C. Experimento de polarização cruzada 13 C, 1 H - B). C) 1 H - 13 C INEPTO experimento de um assembly de proteína rígida; somente componentes de reserva são visíveis. clique aqui para ver uma versão maior desta figura.
3. determinação da estrutura de análise conformacional e 3D
- atribuição de ressonância sequencial
- configurar um 2D 13 C - 13 C difusão de rotação de prótons (PDSD) 41 experimentos para detectar correlações de 13 C 13 C - intraresíduo, incluindo cadeias laterais. Assuma os valores para a etapa de polarização cruzada inicial 1 H - 13 C no 1 D 1 H 13 CP C experiência.
- Definir o tempo de mistura de 50 ms para uniformemente 13 amostras C-etiquetadas e 100 ms para seletivamente 13 amostras C-rotulados. Definir o tempo de aquisição indirecta entre 5-25 ms e a aquisição direta entre 15-25 ms dependentes a resolução espectral intrínseca, que pode ser estimada pelo sinal visível no comprimento (exemplificado em decadência de indução livre (FID) Figura 3A).
- Testar vários parâmetros de processamento para localizar valores ideais com relação sinal / ruído e resolução no espectro, tipicamente processamento adequado pode ser alcançado usando uma função de janela de qsine com um turno de sino de seno (SSB) de 2.5-4.
- Set up, registro e processo um 2D 13 C - 13 C PDSD com uma experiência de tempo de mistura intermediária para detectar sequencial 13 C - 13 C correlações conectando na maior parte i - i±1, mas também i±2 e i±3 resíduos, dependendo da rigidez de espinha dorsal da proteína e elementos de estrutura secundária. O tempo de mistura deve ser definido entre 100-200 ms para amostras uniformemente rotuladas. Todos os outros valores serão recolhidos o curta mistura tempo 2D 13 C - 13 PDSD C.
- Setup, registro e processo um 2D 15 N - 13 C N eu CA eu N eu CO eu experimento e usando um CP 1 H - 15 N e uma transferência de 13 C 15 N - CP específica 42. otimizar a transferência de polarização 13 C 15 N - de forma 1D diretamente na amostra de interesse, com base na intensidade máxima na região de CA ou carbonilo, respectivamente. Kinderdvalores de cal para o tempo de contato específico CP são entre 2-6 ms.
- Setup, registro e processo uma série de 2D 15 N-(13) C - 13 C experimentos com a finalidade de atribuição.
Nota: O primeira 15 N - 13 C polarização transferência o valor do parâmetro pode ser retomado do N eu CA eu / N eu CO eu experimentos. As correlações intraresíduo são estabelecidas de N eu N eu (CA eu) CO , usando respectivamente e (CA eu) CB eu SONHAM 43 e PDSD 13 C - 13 C transferências de polarização. A conectividade sequencial (i-1) de resíduo (i) resíduo é obtida a partir de N eu (CO i-1) CA i-1 e N eu (CO i-1) CX i-1 experiências, ambos usando um PDSD 13 C - 13 C transferência de polarização. - Programa de análise de escolher um NMR como CcpNmr análise 44 ou 45 de faísca
- Carregar os espectros 2D para o software (exemplificado com análise de CcpNmr) e carregar a sequência primária da proteína.
- Começa com a identificação dos tipos de aminoácido visíveis no curto-mistura 13 C - 13 espectro C PDSD. Conectar os átomos de carbono dos sistemas de rotação para permitir a " tipo de resíduo " atribuição específica; Ver Figura 4A para a atribuição de um resíduo de treonina. Identificar resíduos tantos quanto possível; Isso vai depender do tamanho de subunidade de proteína, a resolução espectral e dispersão.
- Sobrepor a curto-mistura com o espectro de PDSD C intermediário-mistura da 13 C - 13.
Nota: Os picos complementares visíveis no PDSD intermediário-mistura surgem principalmente a partir sequencial (resíduo eu - i±1 resíduo) contatos. Um exemplo para uma missão de dois-resíduo sequencial é ilustrado na Figura 4B.- Marcar os picos de ressonância de um sistema de rotação e encontrar correlações no PDSD intermediário-mistura com frequências de ressonância de outros sistemas de rotação. Em pequenas proteínas ou peptídeos, pode ser possível alcançar a atribuição de ressonância sequencial inteiro com base em experiências PDSD 2D 13 C - 13 C.
Nota: No resíduo de motivos de estrutura secundária de β-vertente contatos de i - resíduo i±2 13 C - 13 C podem mostrar sinais mais intensos do que contatos sequenciais devido à sua proximidade no espaço; no resíduo de motivos de estrutura secundária α-helicoidais contatos de i - resíduo i±3 13 C - 13 C também podem levar a sinais intensos.
- Marcar os picos de ressonância de um sistema de rotação e encontrar correlações no PDSD intermediário-mistura com frequências de ressonância de outros sistemas de rotação. Em pequenas proteínas ou peptídeos, pode ser possível alcançar a atribuição de ressonância sequencial inteiro com base em experiências PDSD 2D 13 C - 13 C.
- Se intermediário-mistura PDSD experimentos em seletivamente 13 C-rotulado amostras estão disponíveis, sobrepô-los para o espectro de curto-mistura (se estiver disponível na seletivamente 13 C-rotulado amostra) e atribuir a suplementação picos, tendo em conta a seletiva 13 C rotulagem padrão (1,3 - 13 C e 2 - 13 C glicerol 32 , 33 ou 1 - 13 C e 2 13 C glicose 29 , 30 , 31.
Nota: por exemplo, em um 2 - 13 C glicerol rotuladas amostra vários tipos de aminoácidos são etiquetados na posição Cα sem os carbonos adjacentes (Cβ e C ') rotulado, favorecendo, portanto, o Cα-Cα transferência entre resíduos adjacentes. Em amostras etiquetadas seletivamente, de longo alcance 13 C - 13 C correlações podem construir já no intermediário-mistura 13 C - 13 C PDSD experiências. Se um pico não pode ser explicado por uma correlação sequencial, isso poderia surgir de um contato de longo alcance. Para altamente ordenado estruturas subunidade homogênea na Assembleia macromolecular, apenas um conjunto de ressonâncias deverá ser visível no espectro. Proteína trechos de espinha dorsal, conformações de dois (ou mais) para o átomo ou o trecho de sequência primária na Assembleia, respectivamente, existirem ou se dobrou (ou ainda mais multiplicada) ressonâncias são visíveis para átomos de 13 C. - Usar o espectro NCA 2D contendo correlações de Cα intraresíduo 15 N - 13 para identificar as frequências de ressonância de 15 N para cada resíduo. Se a resolução na dimensão 13 C não for suficiente, usar as informações complementares sobre a ressonância Cβ o NCACB 2D para identificar a frequência de ressonância intraresíduo 15 N.
- Use os espectros NCACO contendo intraresíduo 15 N - 13 C - 13 C correlações e 2D NCACB (i) identificar a frequência de 15 N intraresíduo e (ii) para resolver ambiguidades na 13 C - 13 C PDSD com a ajuda da dimensão adicional 15 N. A inversão do sinal devido à transferência de sonho leva à observação de correlações de Cα-Cβ típicas para o qual o sinal Cα é positivo e o sinal Cβ negativo. Mais cadeia lateral 13 C, se visível do espectro, são positivo novamente.
- configurar um 2D 13 C - 13 C difusão de rotação de prótons (PDSD) 41 experimentos para detectar correlações de 13 C 13 C - intraresíduo, incluindo cadeias laterais. Assuma os valores para a etapa de polarização cruzada inicial 1 H - 13 C no 1 D 1 H 13 CP C experiência.
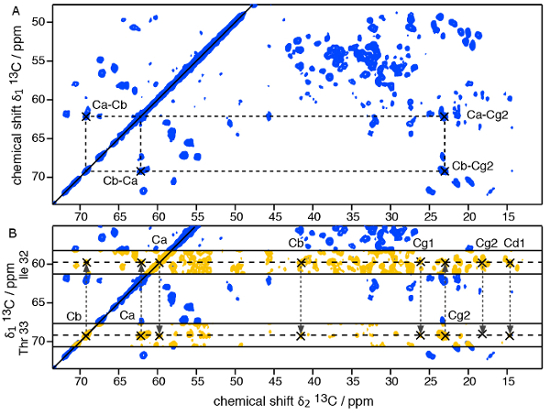
Figura 4: 2-dimensional 13 C - 13 C SSNMR PDSD experiências em um bem-ordenado, uniformemente 13 C, 15 N-etiquetou a proteína assembly. A) curto tempo PDSD de mistura (50 ms mistura). B) atribuição do trecho 2-resíduo Ile32 - Thr33 usando a sobreposição do curto tempo de mistura PDSD com um tempo de mistura tempo PDSD (200 ms mistura). clique aqui para ver uma versão maior desta figura.
- determinação de estrutura secundária
- usar 13 Cα, valores de deslocamento químico 13 Cβ para calcular o produto químico secundário shift 46 ΔδCα-ΔδCβ , indicativo da estrutura secundária. Calcular 13 Cα(assigned) - 13 Cα (bobina aleatória) e 13 Cβ(assigned) - 13 Cβ (bobina aleatória).
Nota: Valores de deslocamento químico de resíduos na conformação de bobina aleatória podem ser obtidos 38. Valores de- trama ΔδCα-ΔδCβ, positivo ou negativo para > 3 resíduos seguidas indicam β-strand ou conformação α-helicoidal respectivamente; resíduos de glicina e prolina podem mostrar valores de deslocamento químico incomum, como muitas vezes agem como " disjuntores de estrutura secundária ".
- Prever os ângulos de diedro de proteína das mudanças químicas atribuídos usando TALOS + 47 , 48 ou 49 , de PREDITOR 50. Os ângulos de diedro Phi/Psi previstos refletem a estrutura secundária da Rafaella da proteínaNIT e são utilizadas como estruturais restrições durante todo o processo de modelagem.
- usar 13 Cα, valores de deslocamento químico 13 Cβ para calcular o produto químico secundário shift 46 ΔδCα-ΔδCβ , indicativo da estrutura secundária. Calcular 13 Cα(assigned) - 13 Cα (bobina aleatória) e 13 Cβ(assigned) - 13 Cβ (bobina aleatória).
- Coleção de restrições estruturais
- definido acima e gravar um 2D 13 C - 13 experimento C PDSD com um tempo de mistura tempo de detectar de longo alcance 13 C - 13 C correlações. Mistura típica vezes variam de 400 ms a 1 s. usar seletivamente 13 C-rotulado de amostras, como a diluição de rotação melhora consideravelmente a transferência de polarização entre carbonos distantes.
- Sobreposição a long-mistura 2D 13 C - 13 C PDSD gravado na amostra etiquetada seletivamente para o intermediário-mistura 13 C - 13 C PDSD, se possível gravado na mesma amostra etiquetada seletivamente. Suplementares picos resultam em correlações entre átomos de 13 C mais distantes. Durante a atribuição de ressonância, levar em conta o regime de rotulagem seletivo.
- Avaliar cuidadosamente a resolução do espectro para definir uma janela de tolerância de atribuição, ou seja, dependente de ressonâncias resolução espectral em um determinado intervalo de ppm que isto pode contribuir para o sinal. O desvio padrão de precisão a atribuição é automaticamente calculado em programas de atribuição de NMR tais como análise de CcpNmr ou SPARKY.
- Se um sinal pode ser explicado por um sequencial (resíduo eu - resíduo i±1) ou um médio alcance 13 C - 13 C contato (resíduo-resíduo eu ± 2, 3 ou 4), manter esta missão desde polarização retransmitida transferência pode explicar o mesmo se a distância de 13 C 13 C - está acima da faixa de contato visível esperada correlação.
- Classificar as atribuições de longo alcance 13 C - 13 C contatos em sinais inequívocos e ambíguo de frequência. No caso de atribuições inequívocas de frequência, atribuição de único ressonância é possível em relação a janela atribuição de tolerância.
Nota: As atribuições ambíguas contêm todas as atribuições de ressonância possível dentro da janela de tolerância. As ambiguidades podem ser levantadas simultaneamente durante o cálculo de estrutura por rodadas iterativas de desambiguação com base nas estruturas preliminares calculadas usando apenas os apoios de inequívocos, como realizado em rotinas computacionais como ARIA 51 ou UNIO 52. Além disso, dados estruturais de diferentes fontes biofísicos (por exemplo, uma mutação ou truncado estrutura de subunidade por solução NMR ou cristalografia de raios x, massa por comprimento de medições, mapa de cryo-EM) também podem ser usados para reduzir o nível de ambiguidade, para exemplos ver 1 , 3 , 53 , 54 , 55 , 56 , 57 , 58.
- deteção de restrições de distância intermolecular em um assembly de proteína simétrica
- criada em um 2D 15 N - 13 experimento de 59 C dor-CP um misto (50/50) U - 15 - N / U - amostra 13 C-rotulados. Sinais detectados no espectro do dor-CP devem surgir de inter molecular 15 N - 13 C proximidades. Use as ressonâncias previamente atribuídas para realizar a atribuição dos contatos intermoleculares.
- Configurar um 2D 13 C - 13 C PDSD com um tempo de tempo de mistura (> 600 ms) em um (50/50) misturado (1,3 - 13 C)-/ (2 - 13 C)-glicerol ou (1 - 13 C)-/ (2 - 13. C)-amostra glicose.
Nota: Sinais detectados neste espectro de 13 C 13 C - surgem de molecular intra e inter molecular 13 C - 13 C proximidades. No entanto, a alta complementaridade dos dois regimes de rotulagem permite que pares de particular ressonância resultam inequivocamente inter moleculares contatos, por exemplo, uma CA de serina no (2 - 13. C)-glicose rotulado subunidades que correlacionam uma serina CB no (1 - 13 C)-rotulado subunidades de glicose.- Sobreposição do espectro da amostra mista para os espectros de 13 C 2D 13 C - gravado em homogeneamente rotulado amostras ((1,3-13C) - e (2 - 13. C)-glicerol ou (1 - 13. C)- e (2 - 13. C)-glicose rotulado de amostras). Sinais adicionais no espectro gravado na amostra mista devem surgir de interações moleculares inter.
- Estrutura de modelagem de dados SSNMR
- preparar as listas de retenção SSNMR necessárias para o cálculo de estrutura: sequência de proteína (1); (2) restrições de distância inequívoca intramoleculares; (3) restrições de distância ambíguo intramoleculares; (4) com base em TALOS de ângulo diedro restrições; (5) restrições de distância inequívoca inter-molecular; (6) restrições de distância ambíguo inter-molecular; (7) adicionais dados de outras técnicas biofísicas (por exemplo, parâmetros de massa-por-comprimento, simetria).
- Vários clientes fornecem insights sobre modelagem de estrutura com base em dados SSNMR e em conjunto com dados estruturais complementares 14 , 60 , 15 , 19 , 61 , 62 nota que as restrições de distância ambígua podem ser disambiguated durante o cálculo de estrutura com relação a preliminar modelos estruturais baseados em restrições de distância sem ambiguidades. Para assegurar a precisão da estrutura, observar atentamente se a estrutura converge para uma única dobra.
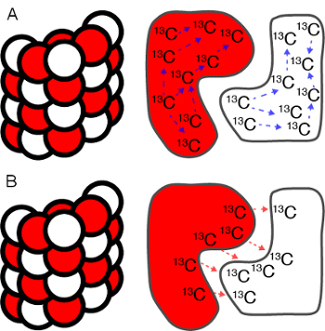
Figura 5 : Intrae intermoleculares contatos em assemblies de proteína simétrica. Representação esquemática de intravs intermoleculares 13 C - 13 C contatos de longo alcance em um assembly macromolecular helicoidal. As subunidades são coloridas em branco e vermelho para ilustrar a rotulagem misto das subunidades; ou seja, antes da montagem, realizou-se uma mistura 1:1 de dois regimes diferentes de rotulagem (por exemplo, 1 - 13 C glicose e 1 - 13 C glicose). A) Intramolecular 13 C - 13 C contatos de longo alcance (seta tracejada azul); B) intermoleculares 13 C - 13 C contatos de longo alcance (vermelho setas do tracejado). clique aqui para ver uma versão maior desta figura.
Resultados
O fluxo de trabalho típico de SSNMR inclui várias etapas, ilustradas na Figura 1. Geralmente as subunidades de proteína são produzidas por em vitro expressão heteróloga em e. coli, purificadas e montadas sob a tremer, mas às vezes também em condições estáticas. Expressão e purificação de subunidade proteína são seguidos por cromatografia em gel de SDS (Figura 2A). A formação de conjuntos macromoleculares então pode ser confirmada pela análise de microscopia de elétron (EM) (ver Figura 2B para obter um exemplo de um assembly filamentoso).
Após a introdução da assembleia para o rotor SSNMR de proteína, o rotor é inserido o espectrômetro, a frequência de MAS e a temperatura são regulamentadas e registam-se os espectros. Primeiras percepções podem ser obtidas por técnicas SSNMR 1, D. A Figura 3 mostra um típico FID SSNMR, detectado no canal 13C em uma amostra de proteína estruturalmente homogênea, uma 1H -13C CP espectro, revelando as ressonâncias13C presentes no núcleo rígido de subunidade em proteína a montagem e um espectro C INEPTO 2D 1H -13, que representa os resíduos móveis. Para atômicas insights sobre o núcleo rígido da estrutura de montagem, multidimensional SSNMR experiências precisam ser gravadas em uniformemente e seletivamente rotulado amostras para atribuir primeiro as ressonâncias SSNMR e então detectar proximidades de longo alcance (ver Figura 4 de ).
Todos os espectros são processados e analisados com o software adequado para atribuir as ressonâncias SSNMR e extrair intrae apoios de distância intermolecular (Figura 5). As restrições de distância SSNMR também são utilizadas isoladamente ou em conjunto com dados de técnicas complementares, que podem ser integrados ao programa de modelagem.
Para estruturas atômicas representativas dos conjuntos macromoleculares resolvidos por técnicas SSNMR Figura 6 ilustra vários assemblies filamentos de apêndices bacterianas e amiloides.

Figura 6:estruturas filamentosas macromoleculares, determinadas por uma abordagem de NMR Solid-State: filamentos bacterianos e fibrilas de proteína amiloide. A) tipo 1 pilus de uropathogenic e. coli, PDB código 2N7H 4; B) ASC filamento, PDB código 2N1F 63; C) agulhas de sistema de secreção tipo III, PDB códigos 2MME, 2LPZ e 2MEX 2,3,64; D) fibrilas de amiloide-beta AB42, PDB código 2NAO, 5KK3, 2MXU, 65,,66,67 e Osaka mutante PDB código 2MVX 57, Iowa mutante PDB código 2MPZ 58; E) fibrilas de alfa-synuclein, 2N0A de código PDB 68; F) domínio do prião HET-s, 2RNM de código PDB, 2KJ3 69,70. Clique aqui para ver uma versão maior desta figura.
Discussão
NMR Solid-State (SSNMR) é um método de escolha para caracterizar assemblies de proteína macromolecular no nível atômico. Uma das questões centrais na determinação da estrutura baseada em SSNMR é a qualidade espectral do sistema investigado, que permite estabelecer modelos estruturais 3D de precisão vários, geralmente variando de modelos de baixa resolução (contendo o secundário estrutura elementos e pouca informação 3D) para estruturas 3D pseudo atômicas. A quantidade e qualidade de informações estruturais, extraídas de experiências multidimensionais de SSNMR é a chave para calcular uma estrutura NMR de alta resolução da Assembleia.
O protocolo descrito baseia-se na detecção de 13C -13C e 15N -13C estruturais restrições que exigem a gravação dos vários espectros 2D (e às vezes 3D) com alto sinal-ruído. Em frequências de MAS moderadas (< 25 kHz), a amostra é introduzida em rotores com tamanhos de 3.2-4 mm de diâmetro permitindo a quantidades de proteína de até ~ 50 mg, dependente da hidratação de amostra. A quantidade de amostra no interior do rotor é diretamente proporcional a relação sinal-ruído em espectros SSNMR, um factor decisivo para a detecção de restrições de distância de longo alcance e sua atribuição inequívoca.
A resolução espectral é um parâmetro crucial durante a atribuição de ressonância sequencial e a coleção de restrições. Para obter melhores resultados, os parâmetros de preparação de amostra precisam ser otimizado, particularmente na purificação da subunidade e as condições de montagem (pH, tampão, agitação, temperatura, etc.). Para a otimização de amostra, é recomendável para preparar amostras sem rótulo para várias condições distintas para as quais tem sido observada a montagem e para gravar um 1D 1H -13C CP espectro (descrito no passo 2.1) em cada amostra preparada. Os espectros servem para comparar a resolução espectral e dispersão entre as preparações diferentes, com base em que as condições ideais podem ser determinadas.
A qualidade dos dados SSNMR depende fortemente da escolha dos parâmetros de aquisição NMR, especialmente para as etapas de transferência de polarização. O uso de alta intensidade de campo magnético (frequência de ≥600 MHz 1H) é essencial para a alta sensibilidade e resolução espectral, necessária quando encaramos o complexos alvos como assemblies de proteína macromolecular.
Em muitos casos, um factor limitativo é a disponibilidade do espectrômetro. Portanto, uma escolha criteriosa das amostras para ser preparado deve preceder a sessão espectrômetro. Em qualquer caso, um uniformemente 13C, 15amostra N-etiquetado é um pré-requisito para realizar a atribuição de ressonância sequencial e intra residual. Para proteínas atribuídas por NMR Solid-State técnicas consulte71. Determinação da estrutura de conjuntos macromoleculares em frequências de MAS moderados seletivamente requer 13C-rotulado de amostras; para a detecção de longo alcance 13C -13C e 13C -15N entra em contato com amostras baseadas em 1,3 -13C - e 2 -13C-gylcerol e/ou 1 -13C - e 2 -13C-glicose rotulagem são comumente utilizados, como descrito acima. A escolha entre os dois esquemas de rotulagem baseia o espectral relação sinal-ruído e resolução. Para distinguir entre intrae intermoleculares contatos de longo alcance, mistas etiquetadas e diluídas amostras revelaram-se eficientes.
Em suma, os passos críticos para um estudo estrutural de SSNMR atômico são: (i) a preparação das subunidades e a necessidade de montagem de ser otimizado para obter a amostra de excelente quantidade e qualidade, (ii) o espectrômetro campo força e aquisição parâmetros precisam ser escolhidas com cuidado; (iii) seletivas estratégias de rotulagem são necessárias para a determinação da estrutura 3D e a quantidade de dados necessárias depende da qualidade dos dados e a disponibilidade de dados complementares.
Apesar de sua aplicabilidade a uma ampla gama de sistemas supramoleculares variando de proteínas de membrana de homomultimeric nano-objetos, SSNMR é muitas vezes limitada pela necessidade de mg-quantidades de material desvendar rotulada. Os recentes desenvolvimentos tecnológicos no ultra rápido MAS (≥100 kHz) SSNMR aberta uma via para 1H-detectado NMR e empurrar o limite da quantidade de amostra mínima para sub-mg 72,73,74. No entanto, para estudos detalhados estruturais 13C-rotulado amostras são indispensável, que limita a aplicação de SSNMR para amostras montadas em vitro ou sistemas expressados em organismos que sobrevivem em meio mínimo onde na célula SSNMR é um método emergente (para comentários Veja 75,,76,7778).
Um fator importante na aplicação de SSNMR para obter estruturas 3D de alta resolução é a resolução espectral: heterogeneidade conformacional intrínseca em um assembly pode limitar a análise de espectros e resolução espectral. Resíduo específico 13C rotulagem pode em alguns casos fornecem uma alternativa para obter informações específicas de distância estratégicas resíduos a fim de obter modelos estruturais (para uma recente exemplos ver 79,,80).
SSNMR para determinação da estrutura 3D ainda requer a recolha de vários conjuntos de dados com tempos de coleção de dados muitas vezes longos em instrumentos sofisticados, dependendo da abordagem e o sistema de vários dias ou semanas em um 600-1000 MHz (frequência de1H) espectrômetro. Portanto, o acesso ao tempo de espectrômetro pode ser um fator limitante em um estudo aprofundado de SSNMR.
No caso de conjuntos de proteínas homomultimeric, levando a dados SSNMR de qualidade suficiente para identificar um elevado número de apoios estruturais, tais como em 3,57,64,70, SSNMR Ainda dá sem acesso às dimensões microscópicas. Portanto, em uma determinação de estrutura de novo SSNMR de um conjunto de homomultimeric, EM ou massa-por-comprimento (MPL) dados complementam idealmente SSNMR dados para derivar os parâmetros de simetria. SSNMR dados sozinhos fornecer o atômica intrae intermoleculares interfaces
SSNMR é altamente complementar com técnicas estruturais, tais como medições EM ou MPL, mas os dados também perfeitamente podem ser combinados com estruturas atômicas obtidas por cristalografia de raios x ou NMR do solução em subunidades mutadas ou truncadas. Um número crescente de estudos pode ser encontrado na literatura, onde o conjunto de dados estruturais diferentes permitiu determinar atômicos modelos 3D de conjuntos macromoleculares (consulte a Figura 6 para exemplos representativos).
No campo da biologia estrutural, SSNMR surge como uma técnica promissora para estudarinsolúveis e não cristalina assemblies nos atômico nível, ou seja, fornecendo dados estruturais na escala atômica. A este respeito, SSNMR é o pendente de solução NMR e cristalografia de raios x para assemblies moleculares, incluindo proteínas de membrana em seus conjuntos de ambiente e proteína nativos como envelopes virais, bacterianos filamentos ou amiloides, bem como a RNA e Complexos de RNA-proteína (ver, por exemplo,81). Suas aplicações altamente versátil em vitro e no contexto celular, tais como o acompanhamento de alterações estruturais secundárias, terciárias e quaternários, identificação de superfícies de interação com moléculas de parceiro na escala atômica (por exemplo, 82) e mapeamento de dinâmica molecular no contexto dos complexos montados, indicam o potencial importante de SSNMR em futuros estudos estruturais complexas biomolecular assemblies.
| Componente de | M9 médio |
| NaCl | 0,5 g/L |
| KH2PO4 | 3 g/L |
| Na2HPO4 | 6,7 g/L |
| MgSO4 | 1 mM |
| ZnCl2 | 10 ΜM |
| FeCl3 | 1 ΜM |
| CaCl2 | 100 ΜM |
| Mistura de vitamina MEM 100 X | 10 mL/L |
| 13 C-glicose | 2 g/L |
| 15 NH4Cl | 1 g/L |
Tabela 1: Composição do meio de expressão mínima para a proteína recombinante produção em Escherichia coli células de BL21.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho é financiado pela ANR (13-PDOC-0017-01 para B.H. e ANR-14-CE09-0020-01 para A.L.), "Os investimentos para o futuro" programa IdEx Bordeaux/CNRS (PPEs 2016 para B.H.) referência ANR-10-IDEX-03-02 para B.H., da Fondation pour la Recherche Médicale ( FRM-AJE20140630090 para A.L.), o programa do 7PQ (FP7-pessoas-2013-CIG para A.L.) e o Conselho Europeu de investigação (CEI) sob Horizonte 2020 investigação e inovação programa da União Europeia (ERC começando Grant para A.L., acordo n 639020) e o projeto " WEAKINTERACT."
Materiais
| Name | Company | Catalog Number | Comments |
| Instruments | |||
| NMR Spectrometer (> 11.7 Tesla) | Bruker | - | |
| triple resonance MAS SSNMR probehead | Bruker | - | |
| SSNMR rotors 4mm | Bruker | K1910 | |
| Centrifuge 5804 R | Eppendorf | 5805000629 | |
| GeneQuant 1300 spectrometer | Dutscher | 28-9182-13 | |
| IGS60 INCUBATEUR HERATHERM 75 L | Dutscher | 228001 | |
| MaxQ 4450 bench top orbital shaker | Dutscher | 78376 | |
| Tube Revolver Agitator | Dutscher | 79547 | |
| sonopuls HD 3100 | Bandelin | 3680 | |
| MicroPulser electroporator | Biorad | 165-2100 | |
| mini-PROTEAN tetra cell system | Biorad | 165-8000 | |
| AKTA pure system | GE Healthcare | 29-0182-24 | |
| capillary microman M25 pipet | Gilson | F148502 | |
| Name | Company | Catalog Number | Comments |
| Materials | |||
| amiconR ultra-15 | sigma | Z740199-8EA | |
| capillaries and pistons | Gilson | F148112 | |
| spatula | Fisher | 13263799 | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| D-glucose 13C6 | Sigma | 389374 | |
| Ammonium-15N-chloride | Sigma | 299251 | |
| 1,3 13C2 glycerol | Sigma | 492639 | |
| 2 13C glycerol | Sigma | 489484 | |
| Kanamycin | Sigma | K1876 | |
| Carbenicillin | Sigma | C3416 | |
| Sodium phosphate dibasic | Sigma | S7907 | |
| Potassium phosphate monobasic | Sigma | P5655 | |
| Sodium chloride | Sigma | 71380 | |
| calcium chloride | Sigma | C1016 | |
| Magnesium sulfate | Sigma | 208094 | |
| Iron Chloride | Sigma | 157740 | |
| Zinc chloride | Sigma | 793523 | |
| MEM Vitamin Solution (100×) | Sigma | M68954 | |
| IPTG | Fisher | BP1755 | |
| Trizma base | Sigma | T1503 | |
| Tricine | Sigma | T0377 | |
| SDS | Sigma | 436143 | |
| sodium azide | sigma | 71289 | |
| 4,4-dimethyl-4-silapentane-1-sulfonic acid | Sigma | 178837 | |
| Name | Company | Catalog Number | Comments |
| Softwares | |||
| Unicorn 6.3 | GE Healthcare | Akta systems | |
| ccpNMR | CCPN | spectrometer systems |
Referências
- Morag, O., Sgourakis, N. G., Baker, D., Goldbourt, A. The NMR-Rosetta capsid model of M13 bacteriophage reveals a quadrupled hydrophobic packing epitope. Proc Natl Acad Sci U S A. 112 (4), 971-976 (2015).
- Loquet, A., et al. Atomic model of the type III secretion system needle. Nature. 486 (7402), 276-279 (2012).
- Demers, J. P., et al. High-resolution structure of the Shigella type-III secretion needle by solid-state NMR and cryo-electron microscopy. Nat Commun. 5 (4976), (2014).
- Habenstein, B., et al. Hybrid Structure of the Type 1 Pilus of Uropathogenic Escherichia coli. Angew Chem Int Ed Engl. 54 (40), 11691-11695 (2015).
- Cady, S. D., et al. Structure of the amantadine binding site of influenza M2 proton channels in lipid bilayers. Nature. 463 (7281), 689-692 (2010).
- Park, S. H., et al. Structure of the chemokine receptor CXCR1 in phospholipid bilayers. Nature. 491 (7426), 779-783 (2012).
- Kaplan, M., et al. Probing a cell-embedded megadalton protein complex by DNP-supported solid-state NMR. Nat Methods. 12 (7), 649-652 (2015).
- Wang, S., et al. Solid-state NMR spectroscopy structure determination of a lipid-embedded heptahelical membrane protein. Nat Methods. 10 (10), 1007-1012 (2013).
- Daskalov, A., et al. Signal transduction by a fungal NOD-like receptor based on propagation of a prion amyloid fold. PLoS Biol. 13 (2), e1002059 (2015).
- Daskalov, A., et al. Identification of a novel cell death-inducing domain reveals that fungal amyloid-controlled cell death is related to necroptosis. Proc Natl Acad Sci U S A. , (2016).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nat Rev Mol Cell Biol. 15 (6), 384-396 (2014).
- Aguzzi, A., Lakkaraju, A. K. Cell Biology of Prions and Prionoids: A Status Report. Trends Cell Biol. 26 (1), 40-51 (2016).
- Habenstein, B., Loquet, A. Solid-state NMR: An emerging technique in structural biology of self-assemblies. Biophys Chem. , (2015).
- Meier, B. H., Bockmann, A. The structure of fibrils from 'misfolded' proteins. Curr Opin Struct Biol. 30, 43-49 (2015).
- Miao, Y., Cross, T. A. Solid state NMR and protein-protein interactions in membranes. Curr Opin Struct Biol. 23 (6), 919-928 (2013).
- Tang, M., Comellas, G., Rienstra, C. M. Advanced solid-state NMR approaches for structure determination of membrane proteins and amyloid fibrils. Acc Chem Res. 46 (9), 2080-2088 (2013).
- Weingarth, M., Baldus, M. Solid-state NMR-based approaches for supramolecular structure elucidation. Acc Chem Res. 46 (9), 2037-2046 (2013).
- Loquet, A., Habenstein, B., Lange, A. Structural investigations of molecular machines by solid-state NMR. Acc Chem Res. 46 (9), 2070-2079 (2013).
- Yan, S., Suiter, C. L., Hou, G., Zhang, H., Polenova, T. Probing structure and dynamics of protein assemblies by magic angle spinning NMR spectroscopy. Acc Chem Res. 46 (9), 2047-2058 (2013).
- Tycko, R., Wickner, R. B. Molecular structures of amyloid and prion fibrils: consensus versus controversy. Acc Chem Res. 46 (7), 1487-1496 (2013).
- Hong, M., Zhang, Y., Hu, F. Membrane protein structure and dynamics from NMR spectroscopy. Annu Rev Phys Chem. 63, 1-24 (2012).
- Jelinek, R., Ramamoorthy, A., Opella, S. J. High-Resolution Three-Dimensional Solid-state NMR Spectroscopy of a Uniformly 15N-Labeled Protein. J Am Chem Soc. 117, 12348-12349 (1995).
- Xu, J., et al. Bicelle-enabled structural studies on a membrane-associated cytochrome B5 by solid-state MAS NMR spectroscopy. Angew Chem Int Ed Engl. 47 (41), 7864-7867 (2008).
- Durr, U. H., Gildenberg, M., Ramamoorthy, A. The magic of bicelles lights up membrane protein structure. Chem Rev. 112 (11), 6054-6074 (2012).
- Yamamoto, K., et al. Probing the transmembrane structure and topology of microsomal cytochrome-p450 by solid-state NMR on temperature-resistant bicelles. Sci Rep. 3, 2556 (2013).
- Huang, R., et al. Probing the transmembrane structure and dynamics of microsomal NADPH-cytochrome P450 oxidoreductase by solid-state NMR. Biophys J. 106 (10), 2126-2133 (2014).
- Durr, U. H., Yamamoto, K., Im, S. C., Waskell, L., Ramamoorthy, A. Solid-state NMR reveals structural and dynamical properties of a membrane-anchored electron-carrier protein, cytochrome b5. J Am Chem Soc. 129 (21), 6670-6671 (2007).
- Hong, M. Determination of multiple phi-torsion angles in proteins by selective and extensive (13)C labeling and two-dimensional solid-state NMR. J Magn Reson. 139 (2), 389-401 (1999).
- Lundstrom, P., et al. Fractional 13C enrichment of isolated carbons using [1-13C]- or [2- 13C]-glucose facilitates the accurate measurement of dynamics at backbone Calpha and side-chain methyl positions in proteins. J Biomol NMR. 38 (3), 199-212 (2007).
- Loquet, A., Lv, G., Giller, K., Becker, S., Lange, A. 13C spin dilution for simplified and complete solid-state NMR resonance assignment of insoluble biological assemblies. J Am Chem Soc. 133 (13), 4722-4725 (2011).
- Castellani, F., et al. Structure of a protein determined by solid-state magic-angle-spinning NMR spectroscopy. Nature. 420 (6911), 98-102 (2002).
- Higman, V. A., et al. Assigning large proteins in the solid state: a MAS NMR resonance assignment strategy using selectively and extensively 13C-labelled proteins. J Biomol NMR. 44 (4), 245-260 (2009).
- Bockmann, A., et al. Characterization of different water pools in solid-state NMR protein samples. J Biomol NMR. 45 (3), 319-327 (2009).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G., Skelton, N. J. . Protein NMR spectroscopy, principles and practice. , (1996).
- Hartman, S. R., Hahn, E. L. Nuclear Double Resonance in the Rotating Frame. Phys Rev. 128 (5), 2042-2053 (1962).
- Harris, R. K., et al. Further conventions for NMR shielding and chemical shifts IUPAC recommendations 2008. Solid State Nucl Magn Reson. 33 (3), 41-56 (2008).
- Wang, Y., Jardetzky, O. Probability-based protein secondary structure identification using combined NMR chemical-shift data. Protein Sci. 11 (4), 852-861 (2002).
- Shaka, A. J., Baker, P. B., Freeman, R. Computer-Optimized Scheme for Wideband Applications and Low-Level Operation. J Magn Reson. 64, 547-552 (1985).
- Szeverenyi, N. M., Sullivan, M. J., Maciel, G. E. Observation of Spin Exchange by Two-Dimensional Fourier-Transform C-13 Cross Polarization-Magic-Angle Spinning. J Magn Reson. 47, 462-475 (1982).
- Baldus, M., Petkova, A. T., Herzfeld, J., Griffin, R. G. Cross polarization in the tilted frame: assignment and spectral simplification in heteronuclear spin systems. Mol Phys. 95 (5), 1197-1207 (1998).
- Verel, R., Ernst, M., Meier, B. H. Adiabatic dipolar recoupling in solid-state NMR: the DREAM scheme. J Magn Reson. 150 (1), 81-99 (2001).
- . Sparky - NMR Assignment and Integration Software Available from: https://www.cgl.ucsf.edu/home/sparky/ (2017)
- Luca, S., et al. Secondary chemical shifts in immobilized peptides and proteins: a qualitative basis for structure refinement under magic angle spinning. J Biomol NMR. 20 (4), 325-331 (2001).
- Shen, Y., Bax, A. SPARTA+: a modest improvement in empirical NMR chemical shift prediction by means of an artificial neural network. J Biomol NMR. 48 (1), 13-22 (2010).
- Berjanskii, M. V., Neal, S., Wishart, D. S. PREDITOR: a web server for predicting protein torsion angle restraints. Nucleic Acids Res. 34 (Web Server issue), W63-W69 (2006).
- Bardiaux, B., Malliavin, T., Nilges, M. ARIA for solution and solid-state NMR. Methods Mol Biol. 831, 453-483 (2012).
- Guerry, P., Herrmann, T. Comprehensive automation for NMR structure determination of proteins. Methods Mol Biol. 831, 429-451 (2012).
- Vasa, S., et al. beta-Helical architecture of cytoskeletal bactofilin filaments revealed by solid-state NMR. Proc Natl Acad Sci U S A. 112 (2), E127-E136 (2015).
- He, L., et al. Structure determination of helical filaments by solid-state NMR spectroscopy. Proc Natl Acad Sci U S A. 113 (3), E272-E281 (2016).
- Tang, M., et al. High-resolution membrane protein structure by joint calculations with solid-state NMR and X-ray experimental data. J Biomol NMR. 51 (3), 227-233 (2011).
- Paravastu, A. K., Leapman, R. D., Yau, W. M., Tycko, R. Molecular structural basis for polymorphism in Alzheimer's beta-amyloid fibrils. Proc Natl Acad Sci U S A. 105 (47), 18349-18354 (2008).
- Schutz, A. K., et al. Atomic-resolution three-dimensional structure of amyloid beta fibrils bearing the Osaka mutation. Angew Chem Int Ed Engl. 54 (1), 331-335 (2015).
- Sgourakis, N. G., Yau, W. M., Qiang, W. Modeling an in-register, parallel "iowa" abeta fibril structure using solid-state NMR data from labeled samples with rosetta. Structure. 23 (1), 216-227 (2015).
- Lewandowski, J. R., De Paepe, G., Griffin, R. G. Proton assisted insensitive nuclei cross polarization. J Am Chem Soc. 129 (4), 728-729 (2007).
- Carlon, A., et al. How to tackle protein structural data from solution and solid state: An integrated approach. Prog Nucl Magn Reson Spectrosc. 92-93, 54-70 (2016).
- Judge, P. J., Taylor, G. F., Dannatt, H. R., Watts, A. Solid-state nuclear magnetic resonance spectroscopy for membrane protein structure determination. Methods Mol Biol. 1261, 331-347 (2015).
- Wang, S., Ladizhansky, V. Recent advances in magic angle spinning solid state NMR of membrane proteins. Prog Nucl Magn Reson Spectrosc. 82, 1-26 (2014).
- Sborgi, L., et al. Structure and assembly of the mouse ASC inflammasome by combined NMR spectroscopy and cryo-electron microscopy. Proc Natl Acad Sci U S A. 112 (43), 13237-13242 (2015).
- Loquet, A., et al. Atomic structure and handedness of the building block of a biological assembly. J Am Chem Soc. 135 (51), 19135-19138 (2013).
- Walti, M. A., et al. Atomic-resolution structure of a disease-relevant Abeta(1-42) amyloid fibril. Proc Natl Acad Sci U S A. 113 (34), E4976-E4984 (2016).
- Colvin, M. T., et al. Atomic Resolution Structure of Monomorphic Abeta42 Amyloid Fibrils. J Am Chem Soc. 138 (30), 9663-9674 (2016).
- Xiao, Y., et al. Abeta(1-42) fibril structure illuminates self-recognition and replication of amyloid in Alzheimer's disease. Nat Struct Mol Biol. 22 (6), 499-505 (2015).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human alpha-synuclein. Nat Struct Mol Biol. 23 (5), 409-415 (2016).
- Wasmer, C., et al. Amyloid fibrils of the HET-s(218-289) prion form a beta solenoid with a triangular hydrophobic core. Science. 319 (5869), 1523-1526 (2008).
- Van Melckebeke, H., et al. Atomic-resolution three-dimensional structure of HET-s(218-289) amyloid fibrils by solid-state NMR spectroscopy. J Am Chem Soc. 132 (39), 13765-13775 (2010).
- Lamley, J. M., et al. Solid-state NMR of a protein in a precipitated complex with a full-length antibody. J Am Chem Soc. 136 (48), 16800-16806 (2014).
- Agarwal, V., et al. De novo 3D structure determination from sub-milligram protein samples by solid-state 100 kHz MAS NMR spectroscopy. Angew Chem Int Ed Engl. 53 (45), 12253-12256 (2014).
- Stanek, J., et al. NMR Spectroscopic Assignment of Backbone and Side-Chain Protons in Fully Protonated Proteins: Microcrystals, Sedimented Assemblies, and Amyloid Fibrils. Angew Chem Int Ed Engl. 55 (50), 15504-15509 (2016).
- Baker, L. A., Baldus, M. Characterization of membrane protein function by solid-state NMR spectroscopy. Curr Opin Struct Biol. 27, 48-55 (2014).
- Luchinat, E., Banci, L. In-cell NMR: a topical review. IUCrJ. 4 (Pt 2), 108-118 (2017).
- Freedberg, D. I., Selenko, P. Live cell NMR. Annu Rev Biophys. 43, 171-192 (2014).
- Selenko, P., Wagner, G. Looking into live cells with in-cell NMR spectroscopy. J Struct Biol. 158 (2), 244-253 (2007).
- Qiang, W., Yau, W. M., Luo, Y., Mattson, M. P., Tycko, R. Antiparallel beta-sheet architecture in Iowa-mutant beta-amyloid fibrils. Proc Natl Acad Sci U S A. 109 (12), 4443-4448 (2012).
- Bateman, D. A., Tycko, R., Wickner, R. B. Experimentally derived structural constraints for amyloid fibrils of wild-type transthyretin. Biophys J. 101 (10), 2485-2492 (2011).
- Marchanka, A., Simon, B., Althoff-Ospelt, G., Carlomagno, T. RNA structure determination by solid-state NMR spectroscopy. Nat Commun. 6, 7024 (2015).
- Schutz, A. K., et al. The amyloid-Congo red interface at atomic resolution. Angew Chem Int Ed Engl. 50 (26), 5956-5960 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados