É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Cromatografia de exclusão de tamanho para analisar heterogeneidade vesícula de membrana externa bacteriana
Neste Artigo
Resumo
As vesículas bacterianas desempenham papéis importantes na patogênese e têm aplicações biotecnológicas promissoras. A heterogeneidade das vesículas complica a análise e o uso; portanto, é necessário um método simples e reprodutível para separar diferentes tamanhos de vesículas. Aqui, demonstramos o uso da cromatografia de exclusão de tamanho para separar vesículas heterogêneas produzidas por Aggregatibacter actinomycetemcomitans.
Resumo
A parede celular de bactérias Gram-negativas consiste em uma membrana interna (citoplasmática) e externa (OM), separada por uma fina camada peptidoglycan. Ao longo do crescimento, a membrana externa pode bleb para formar vesículas de membrana externa esférica (OMVs). Esses OMVs estão envolvidos em inúmeras funções celulares, incluindo entrega de carga para células hospedeiras e comunicação com células bacterianas. Recentemente, o potencial terapêutico dos OMVs começou a ser explorado, incluindo seu uso como vacinas e veículos de entrega de medicamentos. Embora os OMVs sejam derivados do OM, há muito se aprecia que a carga lipídica e proteica do OMV difere, muitas vezes significativamente, da do OM. Mais recentemente, evidências de que as bactérias podem liberar vários tipos de OMVs foram descobertas, e existem evidências de que o tamanho pode impactar o mecanismo de sua absorção por células hospedeiras. No entanto, os estudos nessa área são limitados por dificuldades em separar eficientemente os OMVs de tamanho heterogêneo. A centrifugação de gradiente de densidade (DGC) tem sido tradicionalmente utilizada para este fim; no entanto, essa técnica é demorada e difícil de escalar. A cromatografia de exclusão de tamanho (SEC), por outro lado, é menos complicada e se presta à escala futura necessária para o uso terapêutico de OMVs. Aqui, descrevemos uma abordagem SEC que permite a separação reprodutível de vesículas de tamanho heterogêneo, usando como um caso de teste, OMVs produzidos por Aggregatibacter actinomycetemcomitans, que variam em diâmetro de menos de 150 nm a mais de 350 nm. Demonstramos separação de OMVs "grandes" (350 nm) e OMVs "pequenos" (<150 nm), verificados por dispersão dinâmica de luz (DLS). Recomendamos técnicas baseadas em SEC sobre técnicas baseadas em DGC para separação de vesículas heterogêneas devido à sua facilidade de uso, reprodutibilidade (incluindo usuário-para-usuário) e possibilidade de dimensionamento.
Introdução
Bactérias gram-negativas liberam vesículas derivadas de sua membrana externa, as chamadas vesículas de membrana externa (OMVs), durante todo o crescimento. Esses OMVs desempenham papéis importantes na comunicação célula-celular, tanto entre bactérias quanto hospedeiras, bem como entre células bacterianas, carregando uma série de biomoléculas importantes, incluindo DNA/RNA, proteínas, lipídios e peptidoglycans1,2. Em particular, o papel dos OMVs na patogênese bacteriana tem sido extensivamente estudado devido ao seu enriquecimento em certos fatores de virulência e toxinas3,4,5,6,7,8,9,10,11.
Os OMVs têm sido relatados de tamanho de 20 a 450 nm, dependendo das bactérias parentais e do estágio de crescimento, com vários tipos de bactérias liberando OMVs heterogêneas de tamanho heterogêneo8,12,13,14, que também diferem em sua composição proteica e mecanismo de entrada de células hospedeiras12. H. pylori liberou OMVs variando em diâmetro de 20 a 450 nm, com os OMVs menores contendo uma composição proteica mais homogênea do que os OMVs maiores. É importante ressaltar que as duas populações de OMVs foram observadas como internalizadas pelas células hospedeiras através de diferentes mecanismos12. Além disso, demonstramos que a Aggregatibacter actinomycetemcomitans libera uma população de pequenos OMVs (<150 nm) juntamente com uma população de Grandes (>350 nm) OMVs, com os OMVs contendo uma quantidade significativa de uma toxina proteica secretada, leukotoxina (LtxA)15. Embora o papel da heterogeneidade do OMV nos processos celulares seja claramente importante, as dificuldades técnicas na separação e análise de populações distintas de vesículas limitaram esses estudos.
Além de sua importância na patogênese bacteriana, os OMVs têm sido propostos para uso em diversas aplicações biotecnológicas, incluindo como vacinas e veículos de entrega de medicamentos16,17,18,19,20. Para seu uso translacional em tais abordagens, é necessária uma preparação limpa e monodispersa de vesículas. Assim, são necessários métodos eficazes e eficientes de separação.
Mais comumente, a centrífugação gradiente de densidade (DGC) é usada para separar populações de vesículas heterogêneas de detritos celulares, incluindo flagelae e proteínas secretadas21; o método também tem sido relatado como uma abordagem para separar subpopulações OMV de tamanho heterogêneo12,13,14. No entanto, o DGC é demorado, ineficiente e altamente variável de usuário para usuário22 e, portanto, não é ideal para scale-up. Em contraste, a cromatografia de exclusão de tamanho (SEC) representa uma abordagem escalável, eficiente e consistente para purificar os OMVs21,23,24. Descobrimos que uma longa (50 cm), fluxo de gravidade, coluna SEC, preenchida com meio de filtragem de gel é suficiente para purificar e separar eficientemente subpopulações de OMVs. Especificamente, usamos essa abordagem para separar os OMVs A. actinomycetemcomitans em subpopulações "grandes" e "pequenas", bem como para remover a contaminação de proteínas e DNA. A purificação foi concluída em menos de 4h, e foi realizada a separação completa das subpopulações OMV e a remoção dos detritos.
Protocolo
1. Preparação de tampões
- Para preparar o tampão de lavagem ELISA, adicione 3,94 g Tris-base, 8,77 g NaCl e 1 g de albumina de soro bovino (BSA) a 1 L de água desionizada (DI). Adicione 500 μL de polisorbato-20. Ajuste o pH para 7.2 usando HCl ou NaOH.
- Para preparar o buffer de bloqueio, adicione 3,94 g Tris-base, 8,77 g NaCl e 10 g BSA. Adicione 500 μL de polisorbato-20 a 1 L de água DI. Ajuste o pH para 7.2 usando HCl ou NaOH.
- Para preparar o buffer de elução (PBS), adicione 8,01 g NaCl, 2,7 g KCl, 1,42 g Na2HPO4e 0,24 g KH2PO4 a 1 L DI. Ajuste o pH para 7.4 usando HCl ou NaOH.
NOTA: Uma solução de 10x deste buffer pode ser feita e diluída com água DI conforme necessário.
2. Preparação da amostra de OMV
- Crescer células A. actinomycetemcomitans até a fase exponencial tardia (densidade óptica a 600 nm de 0,7). Pelota as células por centrifugação duas vezes a 10.000 x g a 4 °C por 10 min. Filtre o supernatante através de um filtro de 0,45 μm.
- Concentre o supernante livre de bactérias usando filtros de corte de peso molecular de 50 kDa. Ultracentrizar a solução concentrada a 105.000 x g a 4 °C por 30 min.
- Resuspengue a pelota em PBS e ultracentrifuuge novamente (105.000 x g a 4 °C por 30 min.) Resuspense a pelota em 2 mL de PBS.
3. Embalando a coluna S-1000
- Misture a garrafa de caldo do meio de filtragem de gel com uma haste de vidro e despeje em uma garrafa de vidro o volume necessário para encher a coluna, além de aproximadamente 50% de excesso (cerca de 135 mL). Deixe essas contas sentarem-se até que se acomodem, e depois decantar o excesso de líquido. Resuspenda as contas no buffer de eluição, de modo que a solução final seja aproximadamente 70% (em volume) gel, 30% tampão. Degas a solução sob vácuo.
- Monte a coluna de vidro verticalmente usando um suporte de anel e encha com tampão de eluição para molhar as paredes da coluna. Escorra o tampão até que haja apenas cerca de 1 cm de tampão restante na coluna.
- Sem criar bolhas, cuidadosamente pipeta contas na coluna, preenchendo a coluna até o topo. Continue a drenar o excesso de buffer durante todo esse processo. Certifique-se de não deixar as contas se acomodarem completamente antes de adicionar contas adicionais ao topo da coluna. A coluna deve ser embalada a uma altura de cerca de 2 cm abaixo da parte inferior do reservatório da coluna.
4. Carregar a amostra e coletar frações
- Degas o tampão de eluição sob vácuo. Lave a coluna com dois volumes de coluna (180 mL) de tampão de eluição.
- Deixe que o buffer restante entre totalmente na coluna. Uma vez que o buffer tenha atingido o topo da camada de gel, pipeta cuidadosamente uma amostra de 2 mL contendo OMVs (em uma concentração lipídica de aproximadamente 100 - 200 nmol/L) na superfície das contas, tomando cuidado para não perturbar nenhuma das contas na parte superior da coluna. Deixe a amostra entrar totalmente no gel, ou seja, quando nenhum líquido permanecer acima da camada de gel.
- Adicione com cuidado e lentamente o tampão de eluição em cima da coluna gel. Não perturbe a camada superior do gel, pois isso causará diluição amostral.
- Coloque um único tubo de 50 mL sob a coluna e abra a coluna. Colete os primeiros 20 mL do eluent. Adicione um tampão de eluição adicional na parte superior da coluna, com cuidado, conforme necessário para garantir que a coluna nunca esteja seca.
- Coloque uma série de tubos de 1,5 mL sob a coluna. Inicie a coluna e colete uma série de amostras de 1 mL em cada tubo. À medida que as amostras estão sendo coletadas, continue adicionando tampão de elução na parte superior da coluna, conforme necessário. Repita até que 96 frações tenham sido coletadas. Pare a coluna.
NOTA: As amostras devem ser armazenadas a -20 °C para armazenamento a longo prazo ou 4 °C para armazenamento a curto prazo até uma análise posterior. - Para limpar a coluna, execute um volume de coluna (90 mL) de 0,1 M NaOH através da coluna. Execute dois volumes de coluna (180 mL) de tampão de eluição através da coluna.
5. Análise amostral
- Para medir a concentração lipídica em cada fração, pipeta 50 μL de cada fração em um único poço de uma placa de 96 poços. Para cada poço, adicione 2,5 μL de corante lipofílico. Incubar por 15 s. Meça a intensidade da fluorescência em um leitor de placas com um comprimento de onda de excitação de 515 nm e um comprimento de onda de emissão de 640 nm. Para calcular a fração de todos os lipídios em cada amostra, somam todas as intensidades de emissão e dividem cada intensidade individual pelo total.
- Para medir a concentração de uma determinada proteína, pipeta 100 μL de cada fração em um único poço de uma placa imuno-placa ELISA. Incubar a 25 °C por 3 h.
- Decante as amostras. Adicione 200 μL de tampão de lavagem ELISA a cada poço e decante. Repita quatro vezes para um total de cinco lavagens.
- Adicione 200 μL de tampão de bloqueio a cada poço e incubar por 1h a 25 °C. Decantar.
- Incubar placas com tampão de bloqueio de 100 μL mais anticorpo primário (1:10.000 para anticorpo purificado; 1:10 para anticorpo não purificado) durante a noite a 4 °C. Decantar.
- Adicione 200 μL de tampão de lavagem ELISA a cada poço e decante. Repita quatro vezes para um total de cinco lavagens.
- Adicione 100 μL de tampão de lavagem ELISA mais anticorpo secundário (1:30.000) a cada poço. Incubar por 1h a 25 °C.
- Adicione 200 μL de tampão de lavagem ELISA a cada poço e decante. Repita quatro vezes para um total de cinco lavagens.
- Adicione 100 μL da solução de um passo de 3,3',5,5'-tetrameethylbenzidina (TMB) e incubar por 15-30 min ou até que uma cor azul se desenvolva. Pare a reação TMB com 50 μL da solução de parada.
- Em um leitor de placas, leia a absorvência de cada poço em um comprimento de onda de 450 nm.
- Para medir a concentração total de proteínas, registotam a absorvência em um comprimento de onda de 280 nm (A280) de cada fração, utilizando um espectrofotômetro UV-vis.
Um esquema do protocolo é mostrado na Figura 1.
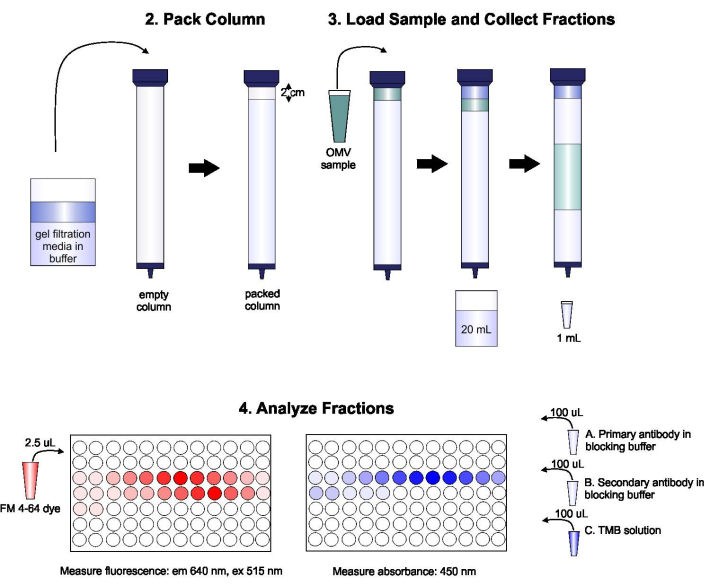
Figura 1: Esquema do procedimento SEC. A coluna é embalada com meio de filtragem de gel desgaseado cuidadosamente para evitar bolhas e descontinuidades, em seguida, lavada com dois volumes de coluna de tampão de eluição. Em seguida, a amostra é cuidadosamente escoada na parte superior do gel, sem interromper a embalagem de gel. A coluna é aberta e executada até que a amostra entre completamente no gel. Neste ponto, o buffer é colocado na parte superior da coluna, e os primeiros 20 mL de elunato são coletados. Em seguida, uma série de frações de 1 mL é coletada. Essas frações são então colocadas em uma placa de 96 poços ou 96-bem de imuno-placa para análise de conteúdo lipídico e proteico. Clique aqui para ver uma versão maior desta figura.
Resultados
A Figura 2 mostra resultados representativos deste método. Os OMVs produzidos por A. actinomycetemcomitans cep JP2 foram primeiro purificados da cultura sobrenante usando ultracentrifugação15. Anteriormente, descobrimos que esta cepa produz duas populações de OMVs, uma com diâmetros de cerca de 300 nm e outra com diâmetros de cerca de 100 nm15. Para separar essas populações de OMV, purificamos a amostra usando o protocolo SEC ...
Discussão
Aqui, fornecemos um protocolo para a separação simples, rápida e reprodutível das subpopulações bacterianas OMV. Embora a técnica seja relativamente direta, existem alguns passos que devem ser realizados com muito cuidado para garantir que ocorra uma separação eficiente na coluna. Primeiro, é essencial que o gel seja carregado na coluna com cuidado e lentamente para evitar bolhas de ar. Observamos que deixar o gel em temperatura ambiente por várias horas antes de carregar a coluna permite que o gel se equilibr...
Divulgações
Os autores não têm conflitos de interesse para relatar.
Agradecimentos
Este trabalho foi financiado pela Fundação Nacional de Ciência (1554417) e Institutos Nacionais de Saúde (DE027769).
Materiais
| Name | Company | Catalog Number | Comments |
| 1-Step Ultra TMB-ELISA | Thermo Scientific | 34028 | |
| Amicon 50 kDa filters | Millipore Sigma | UFC905024 | |
| Bovine Serum Albumin (BSA) | Fisher Scientific | BP9704-100 | |
| ELISA Immuno Plates | Thermo Scientific | 442404 | |
| FM 4-64 | Thermo Scientific | T13320 | 1.5 x 50 cm |
| Glass Econo-Column | BioRad | 7371552 | |
| Infinite 200 Pro Plate Reader | Tecan | ||
| Potassium Chloride (KCl) | Amresco (VWR) | 0395-500G | |
| Potassium Phosphate Monobasic Anhydrous (KH2PO4) | Amresco (VWR) | 0781-500G | |
| Sephacryl S-1000 Superfine | GE Healthcare | 17-0476-01 | |
| Sodium Chloride (NaCl) | Fisher Chemical | S271-3 | |
| Sodium Phosphate Dibasic Anhydrous (Na2HPO4) | Amresco (VWR) | 0404-500G | |
| Tris Base | VWR | 0497-1KG | |
| Tween(R) 20 | Acros Organics | 23336-2500 |
Referências
- Kuehn, M. J., Kesty, N. C. Bacterial outer membrane vesicles and the host-pathogen interaction. Genes and Development. 19, 2645-2655 (2005).
- Kulp, A., Kuehn, M. J. Biological functions and biogenesis of secreted bacterial outer membrane vesicles. Annual Reviews Microbiology. 64, 163-184 (2010).
- Kato, S., Kowashi, Y., Demuth, D. R. Outer membrane-like vesicles secreted by Actinobacillus actinomycetemcomitans are enriched in leukotoxin. Microbial Pathogenesis. 32 (1), 1-13 (2002).
- Nice, J. B., et al. Aggregatibacter actinomycetemcomitans leukotoxin is delivered to host cells in an LFA-1-independent manner when associated with outer membrane vesicles. Toxins. 10 (10), 414 (2018).
- Haurat, M. F., et al. Selective sorting of cargo proteins into bacterial membrane vesicles. Journal of Biological Chemistry. 286 (2), 1269-1276 (2011).
- Horstman, A. L., Kuehn, M. J. Enterotoxigenic Escherichia coli secretes active heat-labile enterotoxin via outer membrane vesicles. The Journal of Biological Chemistry. 275 (17), 12489-12496 (2000).
- Wai, S. N., et al. Vesicle-mediated export and assembly of pore-forming oligomers of the Enterobacterial ClyA cytotoxin. Cell. 115, 25-35 (2003).
- Balsalobre, C., et al. Release of the Type I secreted α-haemolysin via outer membrane vesicles from Escherichia coli. Molecular Microbiology. 59 (1), 99-112 (2006).
- Donato, G. M., et al. Delivery of Bordetella pertussis adenylate cyclase toxin to target cells via outer membrane vesicles. FEBS Letters. 586, 459-465 (2012).
- Kim, Y. R., et al. Outer membrane vesicles of Vibrio vulnificus deliver cytolysin-hemolysin VvhA into epithelial cells to induce cytotoxicity. Biochemical and Biophysical Research Communications. 399, 607-612 (2010).
- Maldonado, R., Wei, R., Kachlany, S. C., Kazi, M., Balashova, N. V. Cytotoxic effects of Kingella kingae outer membrane vesicles on human cells. Microbial Pathogenesis. 51 (1-2), 22-30 (2011).
- Turner, L., et al. Helicobacter pylori outer membrane vesicle size determines their mechanisms of host cell entry and protein content. Frontiers in Immunology. 9, 1466 (2018).
- Zavan, L., Bitto, N. J., Johnston, E. L., Greening, D. W., Kaparakis-Liaskos, M. Helicobacter pylori growth stage determines the size, protein composition, and preferential cargo packaging of outer membrane vesicles. Proteomics. 19 (1-2), 1800209 (2019).
- Rompikuntal, P. K., et al. Perinuclear localization of internalized outer membrane vesicles carrying active cytolethal distending toxin from Aggregatibacter actinomycetemcomitans. Infections and Immunity. 80 (1), 31-42 (2012).
- Nice, J. B., et al. Aggregatibacter actinomycetemcomitans leukotoxin is delivered to host cells in an LFA-1-independent manner when associated with outer membrane vesicles. Toxins. 10 (10), 414 (2018).
- Stevenson, T. C., et al. Immunization with outer membrane vesicles displaying conserved surface polysaccharide antigen elicits broadly antimicrobial antibodies. Proceedings of the National Academy of Sciences. 115 (14), 3106-3115 (2018).
- Gujrati, V., et al. Bioengineered bacterial outer membrane vesicles as cell-specific drug-delivery vehicles for cancer therapy. ACS Nano. 8 (2), 1525-1537 (2014).
- Huang, W., et al. Development of novel nanoantibiotics using an outer membrane vesicle-based drug efflux mechanism. Journal of Controlled Release. 317, 1-22 (2020).
- Chen, D. J., et al. Delivery of foreign antigens by engineered outer membrane vesicle vaccines. Proceedings of the National Academy of Sciences. 107 (7), 3099-3104 (2010).
- Chen, L., et al. Outer membrane vesicles displaying engineered glycotopes elicit protective antibodies. Proceedings of the National Academy of Sciences. 113 (26), 3609-3618 (2016).
- Singorenko, P. D., et al. Isolation of membrane vesicles from prokaryotes: A technical and biological comparison reveals heterogeneity. Journal of Extracellular Vesicles. 6 (1), 1324731 (2017).
- Zeringer, E., Barta, T., Li, M., Vlassov, A. V. Strategies for isolation of exosomes. Cold Spring Harbor Protocols. 2015 (4), 319-323 (2015).
- Benedikter, B. J., et al. Ultrafiltration combined with size exclusion chromatography efficiently isolates extracellular vesicles from cell culture media for compositional and functional studies. Science Reports. 7 (1), 15297 (2017).
- Mol, E. A., Goumans, M. J., Doevendans, P. A., Sluijter, J. P. G., Vader, P. Higher functionality of extracellular vesicles isolated using size-exclusion chromatography compared to ultracentrifugation. Nanomedicine. 13 (6), 2061-2065 (2017).
- Lally, E. T., Golub, E. E., Kieba, I. R. Identification and immunological characterization of the domain of Actinobacillus actinomycetemcomitans leukotoxin that determines its specificity for human target cells. Journal of Biological Chemistry. 269 (49), 31289-31295 (1994).
- Chang, E. H., Giaquinto, P., Huang, J., Balashova, N. V., Brown, A. C. Epigallocatechin gallate inhibits leukotoxin release by Aggregatibacter actinomycetemcomitans by promoting association with the bacterial membrane. Molecular Oral Microbiology. 35 (1), 29-39 (2020).
- Klimentová, J., Stulík, J. Methods of isolation and purification of outer membrane vesicles from gram-negative bacteria. Microbiological Research. 170, 1-9 (2015).
- Dauros Singorenko, P., et al. Isolation of membrane vesicles from prokaryotes: a technical and biological comparison reveals heterogeneity. Journal of Extracellular Vesicles. 6 (1), 1324731 (2017).
- Monguió-Tortajada, M., Gálvez-Montón, C., Bayes-Genis, A., Roura, S., Borràs, F. E. Extracellular vesicle isolation methods: rising impact of size-exclusion chromatography. Cellular and Molecular Life Sciences. 76 (12), 2369-2382 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados