É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Análise eletroforética da replicação por meio de repetições de DNA propensas à estrutura dentro do epissoma humano baseado em SV40
Neste Artigo
Resumo
Aqui, descrevemos o procedimento para analisar a progressão da replicação por meio de repetições patogênicas e propensas à estrutura usando eletroforese em gel bidimensional.
Resumo
A eletroforese em gel bidimensional neutra/neutra (2DGE) surgiu como uma técnica de referência para analisar a replicação do DNA por meio de impedimentos naturais. Este protocolo descreve como analisar a progressão do garfo de replicação por meio de repetições de DNA expansíveis e propensas à estrutura dentro do epissoma baseado no vírus símio 40 (SV40) em células humanas. Em resumo, após a transfecção do plasmídeo em células humanas, os intermediários de replicação são isolados pelo protocolo Hirt modificado e tratados com a enzima de restrição DpnI para remover o DNA não replicado. Os intermediários são então digeridos por enzimas de restrição apropriadas para colocar a repetição de interesse dentro da metade distal de origem de um fragmento de DNA de 3-5 kb de comprimento. Os intermediários de replicação são separados em duas dimensões perpendiculares, primeiro por tamanho e depois por forma. Após a hibridização Southern blot, essa abordagem permite que os pesquisadores observem o travamento do garfo em várias repetições de formação de estrutura na metade descendente do arco Y de replicação. Além disso, esse posicionamento do local de parada permite a visualização de vários resultados da parada do garfo mediada por repetição, como reversão do garfo, o advento de um garfo convergente e a reinicialização do garfo recombinacional.
Introdução
As repetições curtas em tandem (STR) são pequenas, normalmente de 2 a 9 pares de bases (pb), sequências repetitivas de DNA que constituem cerca de 3% do genoma humano1. STR desempenham um papel importante na regulação gênica2; no entanto, sua composição repetitiva os deixa propensos à formação de estruturas secundárias de DNA não canônicas e subsequente instabilidade genética 3,4. De hélices canhotas a grampos de cabelo / cruciformes, a hélices de três e quatro fitas, essas estruturas alternativas de DNA causam desafios intrínsecos para o replissoma. Um pré-requisito natural para a formação da estrutura secundária é o desenrolamento do DNA, que é um pré-requisito para a replicação do DNA. Isso apresenta um enigma único para o funcionamento do genoma, pois muitas dessas estruturas podem se formar durante a replicação, dificultando a progressão do replissoma e, em última análise, causando a paralisação do garfode replicação 5,6,7 ou, em casos graves, o colapso do garfo e a quebra do DNA 8,9. Tanto o reinício de garfos paralisados quanto as vias de reparo do DNA demonstraram levar à instabilidade repetida, como expansões repetidas10,11 e rearranjos genômicos complexos (CGR) 12 , 13 . Esses eventos podem resultar no desenvolvimento de cerca de 60 doenças humanas conhecidas como distúrbios de expansão de repetição, incluindo a síndrome do X frágil, a doença de Huntington, a ataxia de Friedreich e outras14,15, bem como doenças CGR, como a síndrome de Emmanuel16. Portanto, para entender melhor os mecanismos da doença humana impulsionada pela instabilidade de repetições, é imperativo estudar os detalhes da progressão do garfo de replicação por meio dessas repetições.
Uma técnica para estudar a progressão da replicação surgiu em meados da década de 1980, quando Brewer e Fangman procuraram fornecer evidências diretas de que o início da replicação em Saccharomyces cerevisiae ocorre em elementos de sequência de replicação autônoma (comumente conhecidos como ARS)17. Ao fazer isso, eles separaram estruturas de intermediários de replicação de levedura em agarose, adaptando um método anterior de Bell e Byers conhecido como eletroforese em gel neutra / neutra bidimensional (2DGE) 18 . Essa técnica utilizou o fato de que o DNA não linear viaja de maneira diferente no gel de agarose do que seu equivalente linear da mesma massa. Mais especificamente, em 2DGE, o DNA isolado é separado em duas dimensões perpendiculares, primeiro principalmente por tamanho e depois principalmente por forma, para criar um mapa abrangente de replicação em uma determinada região de interesse. Em seu artigo original, Brewer e Fangman demonstraram isso como um arco composto de estruturas "Y simples" ou garfos de replicação que unem o DNA não replicado às suas contrapartes replicadas. Eles descrevem ainda outros intermediários observados como "bolhas" e "Ys duplos", representando origens de replicação e bifurcações convergentes, respectivamente.
2DGE pode ser usado para estudar as populações relativas de intermediários de replicação de DNA em um determinado momento. Portanto, se uma população de intermediários é mais prevalente do que outra, isso seria evidente na visualização. Isso torna o 2DGE uma ferramenta especialmente útil para estudar a progressão da replicação por meio de sequências desafiadoras, como repetições de formação de estrutura. Por exemplo, se a região analisada contiver uma sequência capaz de induzir a paralisação da bifurcação de replicação, isso se apresentaria como uma protuberância no arco (Figura 1A), indicando um acúmulo de bifurcações de replicação naquele locus. Isso pode ser visto com a replicação de ambas as sequências de repetição formadoras de grampo de cabelo em levedura 19,20,21 e repetições de formação triplex em células humanas 22,23,24. Além de estol, o 2DGE pode ser usado para observar estruturas de DNA que não estão em conformidade com os Ys simples padrão formados durante a replicação, como no caso de intermediários recombinantes25. Esses intermediários têm uma estrutura em forma de X mais pesada e ramificada e, portanto, viajam mais devagar na primeira e na segunda dimensões do que os garfos de replicação padrão. Resultados semelhantes também podem ser observados em relação à reversão do garfo de replicação 20,24,26. Em resposta ao forte estresse de replicação, as células eucarióticas demonstraram utilizar a reversão do garfo de replicação para resgatar garfos parados. Esses garfos invertidos têm peso molecular semelhante aos garfos parados; no entanto, sua estrutura de pé de galinha resulta em mobilidade eletroforética mais lenta na segunda dimensão em relação aos seus complementos em forma de Y, resultando em uma extensão para cima e para fora do arco.
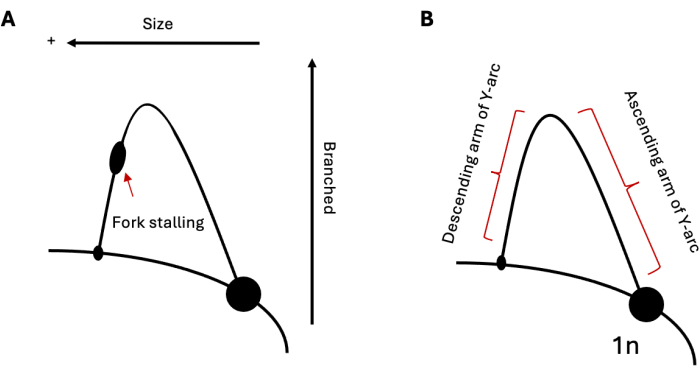
Figura 1: Análise de eletroforese em gel 2D da replicação do DNA. (A) Esquema de um 2DGE típico representando a replicação por meio de uma repetição de formação de estrutura capaz de induzir o travamento do garfo. O tamanho e a estrutura intermediários influenciarão a mobilidade eletroforética. (B) Amostra de arco Y com braços ascendentes e descendentes, respectivamente, rotulados. Abreviatura: 2DGE = Eletroforese em gel bidimensional neutra/neutra. Clique aqui para ver uma versão maior desta figura.
Naturalmente, um dos aspectos mais importantes do 2DGE diz respeito à qualidade e quantidade de intermediários de replicação. No entanto, a resolução da análise 2DGE da replicação através de loci endógenos em células de mamíferos é insuficiente para uma única sequência alvo de cópia dentro do genoma humano diplóide de 6 ×10 9 pb, embora tenha sido feita para genes de múltiplas cópias, como o locus DHFR27 fortemente amplificado ou o RNA ribossômico28. A replicação baseada em SV40 é um meio eficiente e bem caracterizado de estudar a replicação em células eucarióticas29. Ele fornece um modelo confiável de replicação eucariótica que utiliza a maior parte da maquinaria do replissoma do hospedeiro para replicar o genoma viral, que é parcelado em nucleossomos após a infecção30,31. Duas exceções notáveis do replissoma de mamíferos são que o antígeno T (Tag), em vez do complexo CMG hospedeiro, serve como DNA helicase replicativo, e a DNA polimerase delta sintetiza as fitas de DNA principais e atrasadas32. Aproveitamos esse sistema colocando trechos patogênicos de repetições formadoras de estrutura a jusante de uma origem de replicação SV40 dentro de um plasmídeo que foi originalmente criado no laboratório Massimo Lopes22. É importante ressaltar que este plasmídeo também contém o gene que codifica o próprio Tag, resultando em sua replicação constitutiva e extremamente potente após a transfecção em uma variedade de células humanas cultivadas. Esta característica dá origem a uma grande quantidade de produtos, ideal para a análise de 2DGE dos intermediários formados durante e em resposta à replicação de repetições patogênicas em células humanas. Aqui, descrevemos um método detalhado de visualização da replicação de repetições formadoras de estrutura dentro do epissoma humano baseado em SV40 usando eletroforese em gel bidimensional.
Protocolo
NOTA: O plasmídeo projetado para nossa análise 2DGE delineada em células de mamíferos deve conter uma origem de replicação SV40 vários kb a montante de repetições propensas à estrutura (Figura 2). A síntese principal e atrasada deve ser mantida em mente ao escolher a orientação em relação à origem em que as repetições devem ser clonadas no plasmídeo.

Figura 2: Digestão de plasmídeo contendo repetições para análise 2DGE. As repetições propensas à estrutura são representadas vários kb a jusante do garfo de replicação que se move para a direita. A digestão com os cortadores exclusivos 1 e 2 colocará a sequência de repetição no braço descendente do arco Y, dada a sequência além da metade do fragmento digerido. Abreviatura: 2DGE = Eletroforese em gel bidimensional neutra/neutra. Clique aqui para ver uma versão maior desta figura.
1. Transfecção de plasmídeo em células de mamíferos
- Semeie 600.000 células HEK293T em uma placa de cultura de tecidos de 10 cm antes da transfecção. Permitir que as células recuperem a 37 °C durante a noite.
NOTA: Muitas linhagens celulares podem ser usadas para este experimento, embora as células contendo a etiqueta SV40 sejam recomendadas para obter os melhores resultados. CUIDADO: HEK293T células são consideradas BSL-2 e todo o trabalho de cultura deve ser realizado em um gabinete de biossegurança usando técnica asséptica apropriada e EPI adequado. - Quando as células atingirem 60% de confluência, transfectar 8 μg de DNA plasmidial contendo repetições nas células semeadas usando reagentes de transfecção apropriados de acordo com o protocolo do fabricante.
- Se não isolar os intermediários neste momento, aspire os meios antigos e substitua por 10 mL de meios novos após 24 h.
- Comece a coletar células 24-48 h após a transfecção.
- Aspire o meio e lave cuidadosamente com 10 mL de solução salina tamponada com fosfato (PBS). Retire e colete as células usando 0,5 mL de tripsina e gire a 340 × g por 4 min.
- Aspirar o sobrenadante e lavar os grânulos celulares com PBS. Girar novamente a 340 × g durante 4 min e aspirar o sobrenadante.
NOTA: A experiência pode ser interrompida congelando os grânulos de células a -80 °C. Encontramos a melhor resolução isolando intermediários de replicação 48 h após a transfecção; no entanto, 24 h produziu resultados viáveis.
2. Isolamento de intermediários de replicação
- Ressuspenda as células em 1,5 mL de tampão de lise de Hirt modificado [10 mM de tris-HCl (pH 7,5), 10 mM de ácido etilenodiaminotetracético (EDTA)] em tubos cônicos de 50 mL e inicie a lise celular.
- Adicione dodecil sulfato de sódio (SDS) a uma concentração final de 0,6% (aproximadamente 650 μL de estoque 2% SDS) e proteinase K a uma concentração final de 100 μg / mL (aproximadamente 10 μL de estoque 20 mg / mL de proteinase K) para remover nucleases.
- Misturar delicadamente, pipetando até ficar homogéneo, e incubar a mistura a 37 °C durante, pelo menos, 90 min.
- Aumente a concentração de NaCl para 1 M (aproximadamente 540 μL de estoque 5 M NaCl) e misture suavemente até ficar homogêneo. Incubar durante a noite (18-24 h) a 4 °C para permitir a precipitação de detritos celulares, RNA e proteínas por salga.
NOTA: A mistura será altamente viscosa, portanto, tome cuidado e seja paciente enquanto mistura bem. - No dia seguinte, separe o DNA dos detritos celulares, RNA e proteína.
- Centrifugar a mistura a 29.500 × g durante 45 min a 4 °C.
- Transferir o sobrenadante contendo ADN, adicionar um volume de fenol:clorofórmio:álcool isoamílico 25:24:1 (v/v) e homogeneizar brevemente.
CUIDADO: Fenol: clorofórmio: álcool isoamílico é um material perigoso e deve ser manuseado com EPI apropriado em uma capela de exaustão química. - Centrifugue novamente a 15.000 × g por 5 min em temperatura ambiente. Transferir a camada aquosa para um novo tubo cónico.
- Precipitar e lavar o ADN isolado.
- Adicione um volume de isopropanol puro e incube em temperatura ambiente por pelo menos 5 min. Girar o DNA a 15.000 × g por 30 min a 4 °C.
- Transvase o sobrenadante e lave o pellet com etanol 70% frio para remover o excesso de sal.
- Gire mais uma vez a 15.000 × g por 30 min a 4 ° C, seque ao ar e ressuspenda suavemente o pellet em tampão Tris-EDTA (TE) (10 mM Tris-HCl, 1 mM EDTA).
NOTA: O experimento pode ser pausado aqui e as amostras podem ser congeladas a -20 °C; no entanto, o ciclo de congelamento / descongelamento deve ser evitado, pois isso pode diminuir a qualidade dos intermediários de replicação do DNA.
3. Preparação da amostra e eletroforese em gel bidimensional
- Digerir os intermediários isolados da replicação do plasmídeo.
- Adicione 100 unidades das enzimas de restrição apropriadas à amostra para digerir o DNA do plasmídeo, colocando especificamente a sequência contendo repetições na metade origens-distal do fragmento linear (Figura 2). Além disso, adicione DpnI para cortar o DNA metilado, removendo assim qualquer DNA de plasmídeo que não tenha sido totalmente replicado em células humanas cultivadas.
NOTA: Para obter melhores resultados, as enzimas de restrição devem ser cortadores exclusivos que produzem um fragmento de 3-5 kb que coloca a sequência propensa à estrutura no braço descendente do arco Y. - Incubar as amostras a 37 °C durante 6-10 h para permitir a digestão completa do plasmídeo.
- Precipitar o ADN com 2,5 volumes de etanol puro frio e incubar a -20 °C durante a noite, ou adicionar um volume de isopropanol e incubar à temperatura ambiente durante 5 min.
- Centrifugar as amostras digeridas e precipitadas a 15 000 × g durante 30 min a 4 °C.
- Decantar o sobrenadante e lavar a amostra com etanol a 70% a frio. Gire novamente a 15.000 × g por 30 min a 4 °C.
- Transvase o sobrenadante, seque ao ar por 10 min e ressuspenda as amostras em 15 μL de tampão TE.
- Adicione 100 unidades das enzimas de restrição apropriadas à amostra para digerir o DNA do plasmídeo, colocando especificamente a sequência contendo repetições na metade origens-distal do fragmento linear (Figura 2). Além disso, adicione DpnI para cortar o DNA metilado, removendo assim qualquer DNA de plasmídeo que não tenha sido totalmente replicado em células humanas cultivadas.
- Prepare o gel de agarose de primeira dimensão a 0,4-0,5% em 1x Tris-borato-EDTA (TBE) (89 mM tris base, 89 mM de ácido bórico, 2 mM EDTA). Deixe a solução solidificar por pelo menos 1 h.
- Comece a carregar as amostras na primeira dimensão.
- Carregue a escada nos primeiros 3 cm em relação à borda mais à esquerda do gel. Em seguida, carregue a totalidade das amostras preparadas, garantindo 3 cm entre cada par.
- Execute o gel em 1x TBE por 19-24 h a 0,85 V/cm para separar os intermediários em relação ao seu tamanho. Certifique-se de que a câmara esteja coberta para proteger as amostras da luz, que pode causar danos ao DNA.
- No dia seguinte, remova o gel do tampão e estime a localização do fragmento linear digerido usando uma régua.
- Extirpar os primeiros 3 cm do gel que contém a escada e corar o segmento de gel em 1x TBE contendo 0,3 μg/ml de brometo de etídio durante 10-15 min. Visualize a escada usando um sistema de documentação de gel.
- Adicione 1,3 cm ao local estimado, resultando no valor a. Em seguida, subtraia 7,5 cm do valor a, obtendo o valor b. Alinhe a régua com o gel de primeira dimensão e corte horizontalmente nos valores a e b. Em seguida, corte verticalmente o espaço de 3 cm reservado para cada amostra. Consulte a Figura 3 para obter um esquema visual.
- Em uma nova bandeja de fundição, gire os segmentos no sentido horário e coloque-os na posição dos poços de amostra (Figura 3).
- Prepare o gel de agarose de segunda dimensão a uma concentração de 1-1,3% em 1x TBE a 0,3 μg/mL de brometo de etídio.
- Após o resfriamento a aproximadamente 55 °C, despeje o gel de segunda dimensão sobre os segmentos de primeira dimensão girados e deixe-o solidificar por pelo menos 1 h.
- Transfira a segunda dimensão para uma câmara com 1x TBE a 0,3 μg/mL de brometo de etídio e deixe o gel se equilibrar por pelo menos 30 min.
- Execute o gel, novamente coberto, por 9-10 h a 4,23 V/cm a 4 °C para separar os intermediários em relação à sua forma.
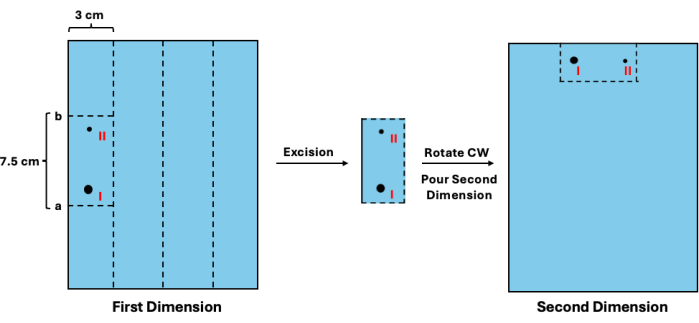
Figura 3: Excisão de intermediários de primeira dimensão antes da separação de segunda dimensão. Após a visualização da escada, a mobilidade de fragmentos não replicados pode ser estimada. (I) Este valor pode então ser usado para determinar os locais de corte apropriados (a e b) para excisá-lo e suas contrapartes replicadas (II). A seção do gel deve então ser girada e colocada na posição de poços para a separação de segunda dimensão. Abreviatura: CW = sentido horário. Clique aqui para ver uma versão maior desta figura.
4. Southern blotting e hibridização com sonda radiomarcada
- Remova o gel de segunda dimensão da câmara e depurine os fragmentos de DNA por 10 min em uma solução de HCl 0,24 M com balanço suave. Enxágue o gel com água deionizada e mergulhe-o em NaOH 0,4 M por 10-15 min.
CUIDADO: HCl e NaOH são corrosivos e devem ser manuseados com EPI apropriado em uma capela de exaustão química. - Comece a montar o Southern blot para facilitar a transferência dos intermediários separados do gel para a membrana. Consulte a Figura 4 para obter um esquema abrangente.
- Encha um recipiente de tamanho suficiente com 1 L de NaOH 0,4 M.
- Alinhe uma longa folha de vidro ao longo do recipiente e dobre (ao longo do comprimento) duas folhas compridas de papel para cromatografia perpendicularmente ao longo da folha de vidro, estendendo-se para dentro do recipiente de NaOH.
- Molhe a parte superior do papel com NaOH e remova cuidadosamente quaisquer bolhas de ar sob sua superfície.
- Mergulhe três folhas de papel cromatógrafo com NaOH e coloque-as sobre o papel dobrado, removendo novamente as bolhas.
- Vire o gel de segunda dimensão de cabeça para baixo e transfira-o sobre os papéis.
- Molhe uma membrana de náilon carregada positivamente (tamanho de poro de 0,45 μm) com água DI e coloque-a sobre o gel.
- Por fim, adicione mais três folhas de papel, molhadas com água DI, sobre a membrana.
- Cubra qualquer NaOH exposto no recipiente inferior com filme plástico para evitar a evaporação. Coloque uma pilha de guardanapos ou toalhas de papel sobre a mancha, certificando-se de que ela tenha 0,3-0,5 m de altura. Coloque um peso no topo, comprimindo todo o borrão para facilitar a ação capilar firme. Aguarde pelo menos 2 dias para que o DNA seja transferido para a membrana.
NOTA: O comprimento e a largura do papel de cromatografia e da membrana dependem do tamanho do gel de agarose usado para a segunda dimensão. Para uma transferência mais eficiente, use papel e membrana com as mesmas dimensões do gel.
- Após a transferência, reticule o DNA com a membrana usando um reticulador UV a 120 μJ/cm2 por 1 min.
NOTA: O experimento pode ser pausado aqui colocando a membrana em um protetor de lençol hermético, seco e limpo à temperatura ambiente. - Lave a membrana 2x por 5 min com 2x citrato de sódio salino (tampão SSC) (NaCl 0,3 M, citrato de sódio 0,03 M).
- Pré-hibridizar a membrana com 0,18 mL / cm2 de tampão de hibridização Church & Gilbert [1 mM EDTA, 1% de albumina de soro bovino (BSA), 0,5 M de fosfato de sódio, 7% SDS] a 65 ° C, girando em uma incubadora de hibridização por pelo menos 2 h.
NOTA: A membrana pode pré-hibridizar por vários dias. - Prepare a sonda radiomarcada usando α-32P dATP ou dCTP e um kit de marcação de DNA de acordo com o protocolo do fabricante.
CUIDADO: Os dNTPs radiomarcados são perigosos e o EPI apropriado deve ser usado ao manuseá-los. Todo o trabalho radioativo deve ser realizado por trás de blindagem, e indivíduos treinados devem ser monitorados quanto à absorção de radiação usando dosímetros.- Projete um fragmento de DNA linear de 400-900 pb complementar à sequência digerida de restrição (Figura 2) e amplifique o fragmento usando a reação em cadeia da polimerase (PCR).
NOTA: Recomendamos ter um fragmento de PCR de estoque de 50-100 ng/μL - Combine 100 ng do fragmento de PCR complementar com DNA Pol I, tampão de fragmento de Klenow (3 '5' exo-) e oligos de decanucleotídeos aleatórios.
- Desnaturar o fragmento a 100 °C durante 10 min.
- Adicione 5 unidades de DNA Pol I, fragmento de Klenow (3 '5' exo-), 50 μCi de α-32P dNTP e 30-50 μmol de mistura dNTP deficiente no tipo dNTP radiomarcado à amostra.
- Incubar a 37 °C por 10 min para permitir a polimerização e a incorporação de dNTP radiomarcado.
- Adicione 30-50 μmol do tipo dNTP anteriormente ausente à amostra e incube a 37 °C por 10 min.
- Purifique o fragmento radiomarcado usando uma coluna de rotação a 3.000 × g por 2 min.
- Projete um fragmento de DNA linear de 400-900 pb complementar à sequência digerida de restrição (Figura 2) e amplifique o fragmento usando a reação em cadeia da polimerase (PCR).
- Adicione a sonda radiomarcada a 50 mL de tampão de hibridização e incube com a membrana durante a noite a 65 °C girando em uma incubadora de hibridização.
- No dia seguinte, remova a sonda e lave a membrana 2x com tampão de lavagem 1 (0,1x SSC, 0,1% SDS) a 42 °C e 2x com tampão de lavagem 2 (2x SSC, 0,1% SDS) a 65 °C.
NOTA: Todas as lavagens devem ser realizadas com rotações rápidas por 15 min na incubadora. - Seque a membrana por 10 min e coloque-a em um protetor de folha fino e transparente. Armazene a membrana selada em um controlado e resistente à radiação com uma tela sensível ao fósforo. Deixe a membrana ser exposta à tela por 1 a 10 dias.
- Visualize os resultados usando um gerador de imagens biomolecular definido para imagens de fósforo. Lave e exponha novamente, se necessário.
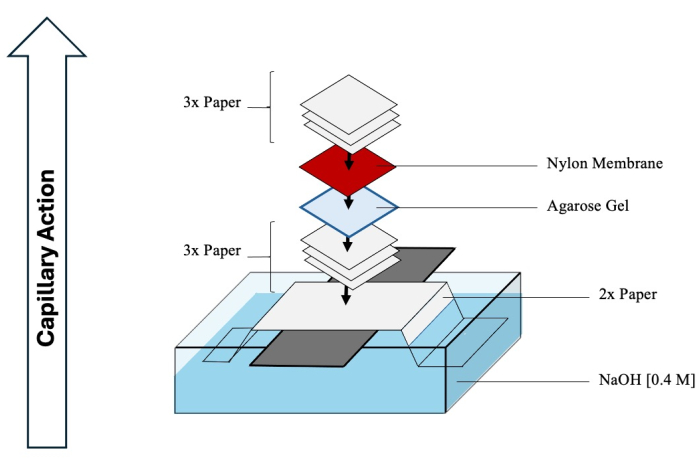
Figura 4: Montagem do Southern blot. Esquema abrangente de um aparelho típico usado para transferência de Southern blot de intermediários da segunda dimensão para uma membrana de nylon. Clique aqui para ver uma versão maior desta figura.
Resultados
Se for bem-sucedido, após a visualização, um arco agudo de garfos de replicação pode ser observado estendendo-se para cima e para fora do ponto maciço 1n (Figura 5A). O tamanho de um fragmento, ou porcentagem que é replicada, determina a mobilidade do fragmento na primeira dimensão. À medida que os intermediários desenvolvem uma estrutura mais articulada, eles começarão a viajar mais lentamente na segunda dimensão. Portanto, se um intermediário...
Discussão
O 2DGE fornece uma imagem semiquantitativa e abrangente das populações relativas de intermediários que surgem durante a replicação de uma sequência específica. Dado que as frágeis estruturas moleculares dos garfos de replicação devem ser mantidas durante todo este procedimento, deve-se tomar muito cuidado para evitar cisalhamento físico e desnaturação química. Portanto, é altamente recomendável que qualquer tratamento alcalino seja evitado durante o isolamento do plasmíd...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Agradecemos a Jorge Cebrian e Anastasia Rastokina que começaram a desenvolver essa abordagem em nosso laboratório, Massimo Lopes por nos fornecer o plasmídeo pML113 e conselhos inestimáveis, Ylli Doksani por discussões perspicazes e membros do laboratório Mirkin por seu apoio. O trabalho no laboratório de Mirkin é apoiado pelo Instituto Nacional de Ciências Médicas Gerais [R35GM130322] e NSF-BSF [2153071].
Materiais
| Name | Company | Catalog Number | Comments |
| 10x TBE Buffer | Bio Rad | 1610733 | |
| 20x SSC Buffer | Fisher Scientific | BP1325-1 | |
| 293T cells | ATCC | CRL-3216 | |
| a-32P dATP, 3000 Ci/mmol | Revvity | BLU512H250UC | |
| Agarose | Fisher Scientific | BP160-500 | |
| Amersham Hybond-N+ | Fisher Scientific | RPN303B | |
| BAS Storage Phosphor Screens | Fisher Scientific | 28956482 | |
| Church and Gibert's hybriddization buffer | Fisher Scientific | 50-103-5408 | |
| DecaLabel DNA labeling kit | ThermoFisher Scientific | K0622 | |
| DMEM, high gluctose, GltaMAX Supplement, pyruvate | ThermoFisher Scientific | 10569010 | |
| DpnI | New England Biolabs | R0176S | Additional restriction enzymes will need to be purchased as well |
| EDTA 0.5 M, pH 8 | Fisher Scientific | BP2482500 | |
| Ethanol, 70% | Fisher Scientific | BP82031GAL | |
| Fetal Bovine Serum | VWR | 97068-085 | |
| Hydrochloric acid solution, 12 M | Millipore Sigma | 13-1683 | |
| Isopropanol | Fisher Scientific | BP26184 | |
| jetPRIME DNA and siRNA Transfection Reagent with Buffer | VWR | 101000027 | |
| MycoZap Plus-CL | VWR | 75870-448 | |
| NaCl | Millipore Sigma | 746398-500G | |
| Nalgene Oak Ridge High-Speed Centrifuge Tubes | ThermoFisher Scientific | 3139-0050 | |
| Phosphate Buffer Saline, pH 7.4 | ThermoFisher Scientific | 10010023 | |
| Phosphate Buffer Saline, pH 7.5 | ThermoFisher Scientific | 10010024 | |
| Proteinase K | ThermoFisher Scientific | EO0491 | |
| Proteinase K | ThermoFisher Scientific | EO0492 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-4 | |
| Pure Cellulose Chromatography Paper | Fisher Scientific | 05-714-5 | |
| Ruler | Fisher Scientific | 09-016 | |
| Scalpel | Fisher Scientific | 12-460-451 | |
| Sodium dodecyl sulfate | Millipore Sigma | 436143-25G | |
| Sodium hydroxide | Fisher Scientific | S25548 | |
| Sorval LYNX 4000 Superspeed Centrifuge | ThermoFisher Scientific | 75006580 | |
| Sub-cell Horizontal Electrophoresis System | Bio Rad | 1704401 | |
| TH13-6 x 50 Swinging Bucket Rotor | ThermoFisher Scientific | 75003010 | |
| Tris-HCl 1 M, pH 7.5 | Fisher Scientific | BP1757-500 | |
| Trypsin-EDTA (0.25%), phenol red | ThermoFisher Scientific | 25200056 |
Referências
- Liao, X., et al. Repetitive DNA sequence detection and its role in the human genome. Commun Biol. 6 (1), 1-21 (2023).
- Fotsing, S. F., et al. The impact of short tandem repeat variation on gene expression. Nat Genet. 51 (11), 1652-1659 (2019).
- Fan, H., Chu, J. -. Y. A brief review of short tandem repeat mutation. GPB. 5 (1), 7-14 (2007).
- Khristich, A. N., Mirkin, S. M. On the wrong DNA track: Molecular mechanisms of repeat-mediated genome instability. J Biol Chem. 295 (13), 4134-4170 (2020).
- Samadashwily, G. M., Raca, G., Mirkin, S. M. Trinucleotide repeats affect DNA replication in vivo. Nat Genet. 17 (3), 298-304 (1997).
- Khristich, A. N., Armenia, J. F., Matera, R. M., Kolchinski, A. A., Mirkin, S. M. Large-scale contractions of Friedreich's ataxia GAA repeats in yeast occur during DNA replication due to their triplex-forming ability. Proc Natl Acad Sci USA. 117 (3), 1628-1637 (2020).
- Shishkin, A. A., et al. Large-scale expansions of Friedreich's ataxia GAA repeats in yeast. Mol Cell. 35 (1), 82-92 (2009).
- Sundararajan, R., Gellon, L., Zunder, R. M., Freudenreich, C. H. Double-strand break repair pathways protect against CAG/CTG repeat expansions, contractions and repeat-mediated chromosomal fragility in Saccharomyces cerevisiae. Genetics. 184 (1), 65-77 (2010).
- Kim, H. -. M., et al. Chromosome fragility at GAA tracts in yeast depends on repeat orientation and requires mismatch repair. EMBO J. 27 (21), 2896-2906 (2008).
- Polleys, E. J., House, N. C. M., Freudenreich, C. H. Role of recombination and replication fork restart in repeat instability. DNA Repair. 56, 156-165 (2017).
- Gold, M. A., et al. Restarted replication forks are error-prone and cause CAG repeat expansions and contractions. PLoS Genet. 17 (10), e1009863 (2021).
- Lambert, S., et al. Homologous recombination restarts blocked replication forks at the expense of genome rearrangements by template exchange. Mol Cell. 39 (3), 346-359 (2010).
- Burssed, B., Zamariolli, M., Bellucco, F. T., Melaragno, M. I. Mechanisms of structural chromosomal rearrangement formation. Mol Cytogenet. 15 (1), 23 (2022).
- Paulson, H. Repeat expansion diseases. Handb Clin Neurol. 147, 105-123 (2018).
- Malik, I., Kelley, C. P., Wang, E., Todd, P. Molecular mechanisms underlying nucleotide repeat expansion disorders. Nat Rev Mol Cell Biol. 22 (9), 589-607 (2021).
- Emanuel, B. S., Zackai, E. H., Medne, L., Adam, M. P. Emanuel Syndrome. GeneReviews®. , (1993).
- Brewer, B. J., Fangman, W. L. The localization of replication origins on ARS plasmids in S. cerevisiae. Cell. 51 (3), 463-471 (1987).
- Bell, L., Byers, B. Separation of branched from linear DNA by two-dimensional gel electrophoresis. Anal Biochem. 130 (2), 527-535 (1983).
- Voineagu, I., Narayanan, V., Lobachev, K. S., Mirkin, S. M. Replication stalling at unstable inverted repeats: Interplay between DNA hairpins and fork stabilizing proteins. Proc Natl Acad Sci USA. 105 (29), 9936-9941 (2008).
- Nguyen, J. H. G., et al. Differential requirement of Srs2 helicase and Rad51 displacement activities in replication of hairpin-forming CAG/CTG repeats. Nucleic Acids Res. 45 (8), 4519-4531 (2017).
- Krasilnikova, M. M., Mirkin, S. M., Kohwi, Y. Analysis of triplet repeat replication by two-dimensional gel electrophoresis. Trinucleotide Repeat Protocols. , (2004).
- Follonier, C., Oehler, J., Herrador, R., Lopes, M. Friedreich's ataxia-associated GAA repeats induce replication-fork reversal and unusual molecular junctions. Nat Struct Mol Biol. 20 (4), 486-494 (2013).
- Chandok, G. S., Patel, M. P., Mirkin, S. M., Krasilnikova, M. M. Effects of Friedreich's ataxia GAA repeats on DNA replication in mammalian cells. Nucleic Acids Res. 40 (9), 3964-3974 (2012).
- Rastokina, A., et al. Large-scale expansions of Friedreich's ataxia GAA•TTC repeats in an experimental human system: role of DNA replication and prevention by LNA-DNA oligonucleotides and PNA oligomers. Nucleic Acids Res. 51 (16), 8532-8549 (2023).
- Giannattasio, M., et al. Visualization of recombination-mediated damage-bypass by template switching. Nat Struct Mol Biol. 21 (10), 884-892 (2014).
- Hisey, J. A., et al. Pathogenic CANVAS (AAGGG)n repeats stall DNA replication due to the formation of alternative DNA structures. Nucleic Acids Res. 52 (8), 4361-4374 (2024).
- Kalejta, R. F., Lin, H. B., Dijkwel, P. A., Hamlin, J. L. Characterizing replication intermediates in the amplified CHO dihydrofolate reductase domain by two novel gel electrophoretic techniques. Mol Cell Biol. 16 (9), 4923-4931 (1996).
- Little, R. D., Platt, T. H., Schildkraut, C. L. Initiation and termination of DNA replication in human rRNA genes. Mol Cell Biol. 13 (10), 6600-6613 (1993).
- Fanning, E., Zhao, K. SV40 DNA replication: From the A gene to a nanomachine. Virology. 384 (2), 352-359 (2009).
- Sogo, J. M., Stahl, H., Koller, T., Knippers, R. Structure of replicating simian virus 40 minichromosomes: The replication fork, core histone segregation and terminal structures. J Mol Biol. 189 (1), 189-204 (1986).
- Weisshart, K., Taneja, P., Fanning, E. The replication protein A binding site in simian virus 40 (SV40) T antigen and its role in the initial steps of SV40 DNA replication. J Virol. 72 (12), 9771-9781 (1998).
- Sowd, G. A., Fanning, E. A Wolf in sheep's clothing: SV40 co-opts host genome maintenance proteins to replicate viral DNA. PLoS Pathog. 8 (11), e1002994 (2012).
- . BLAST: Basic Local Alignment Search Tool Available from: https://blast.ncbi.nlm.nih.gov/Blast.cgi (2024)
- Hirt, B. Selective extraction of polyoma DNA from infected mouse cell cultures. J Mol Biol. 26 (2), 365-369 (1967).
- Lopes, M., Foiani, M., Sogo, J. M. Multiple mechanisms control chromosome integrity after replication fork uncoupling and restart at irreparable UV lesions. Mol Cell. 21 (1), 15-27 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados