É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um fluxo de trabalho rápido e simples para quantificação de estruturas externas de Drosophila adulta
Neste Artigo
Resumo
Aqui, apresentamos um fluxo de trabalho rápido e de baixo custo para imagens de alta resolução de olhos adultos de Drosophila para quantificar defeitos de padronização e crescimento. Descrevemos nosso protocolo para preparação de amostras por montagem em ponto, aquisição de imagem de alta resolução e análise de imagem.
Resumo
O olho composto de Drosophila é um tecido padronizado com precisão que revelou mecanismos moleculares e processos biológicos que impulsionam a morfogênese. É uma estrutura simples de olhos unitários repetitivos, denominada omatídeos, que é usada para caracterizar interações genéticas e funções gênicas. Mutações que afetam a arquitetura do olho podem ser facilmente detectadas e analisadas; portanto, esse sistema é frequentemente usado em instituições com poucos recursos. A análise fenotípica adicional geralmente inclui um microscópio eletrônico de varredura (SEM) para gerar imagens de alta ampliação adequadas para análise quantitativa. No entanto, os SEMs são caros e requerem reagentes caros; a preparação da amostra se estende por dias; e, muitas vezes, eles precisam de pessoal em tempo integral para preparação de amostras e manutenção de instrumentos. Isso limita sua utilidade em instituições com poucos recursos ou durante a austeridade orçamentária. Em entomologia, o uso de tecnologia de imagem digital de alta resolução é uma prática comum para a identificação e caracterização de espécies. Este artigo descreve um método que combina estratégias e permite imagens digitais de alta resolução de estruturas adultas de Drosophila e análise quantitativa usando o software aberto ImageJ. O fluxo de trabalho é uma alternativa rápida e amigável ao aluno que corrige as limitações de instalações de pesquisa subfinanciadas e com poucos recursos com uma abordagem econômica e rápida para a análise fenotípica quantitativa.
Introdução
Drosophila melanogaster é um poderoso organismo modelo genético que tem sido usado há décadas para elucidar vias de sinalização molecular e comportamentos celulares. Muitas das vias de sinalização evolutivamente conservadas que são essenciais para o desenvolvimento multicelular foram identificadas pela primeira vez e seu mecanismo de ação definido em Drosophila. Cerca de 65-75% de todos os genes associados a doenças humanas têm ortólogos em Drosophila 1,2. O olho adulto de Drosophila é um modelo importante que permitiu triagens genéticas imparciais que facilitaram a descoberta de importantes genes conservados implicados em doenças humanas, incluindo câncer 3,4, neurodegeneração5 e distúrbios metabólicos6.
O olho da Drosophila é composto por ~ 800 olhos unitários, denominados omatídeos, que são precisamente dispostos em um padrão hexagonal na superfície do olho adulto7. Cada omatídio é composto por oito neurônios fotorreceptores que ocupam uma localização distinta dentro de um trapézio assimétrico. Estes são suportados por quatro células cone não neurais e duas células de pigmento primário que secretam lente e pseudo-cone para focalizar a luz nos rabdômeros sensíveis à luz dos neurônios fotorreceptores. Os omatídeos vizinhos são separados por uma única fileira de células de rede interommatidial, compostas por células pigmentares secundárias, células pigmentares terciárias e complexos de cerdas mecanossensoriais 8,9,10.
Perturbações no desenvolvimento ocular são visíveis em olhos adultos como aumento ou diminuição do tamanho do olho, abundância anormal ou estrutura de lentes ou cerdas, ou como um "olho áspero" onde o padrão hexagonal normalmente invariante é interrompido de tal forma que uma fileira de omatídeos não pode mais ser seguida pela superfície do olho. Esses fenótipos podem ser pontuados no nível do tecido macroscópico usando microscópios de dissecação. A análise detalhada dos fenótipos tradicionalmente inclui microscopia eletrônica de varredura seguida de análise quantitativa de imagens11. No entanto, a microscopia eletrônica de varredura requer instrumentação cara, reagentes caros, preparação de amostras que se estende por dias e, muitas vezes, equipe em tempo integral para manter.
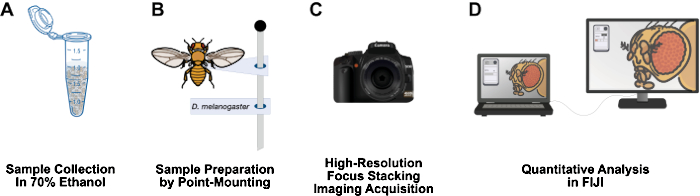
Figura 1: Fluxo de trabalho para imagens de estruturas de Drosophila adultas. (A) Colete e fixe Drosophila adulta em etanol 70%. (B) Prepare amostras para imagem por montagem pontual e fixação em pinos. (C) Adquira imagens de alta resolução por meio de empilhamento e integração de foco. (D) Quantificar imagens usando FIJI. Clique aqui para ver uma versão maior desta figura.
Este artigo apresenta um fluxo de trabalho relativamente barato, tem um tempo de preparação de amostra curto, pode ser facilmente configurado em uma bancada de laboratório de 3 pés, não requer materiais perigosos e pode ser uma adição de longa duração aos laboratórios de pesquisa de Drosophila (Figura 1). A montagem pontual é uma técnica entomológica usada para secar ao ar e preservar insetos pequenos e de corpo mole, como Drosophila12. Este método baseia-se na combinação de objetivas de microscópio com câmeras DSLR de alta resolução para ampliações efetivas de 10x a 1.000x. A profundidade de campo limitada inerente à macrofotografia é superada pelo empilhamento de foco: costurando uma série de imagens com o plano focal se movendo através da amostra de interesse13. Este método produz imagens de alta resolução adequadas para a quantificação de fenótipos e pode ser facilmente adaptado para outras estruturas de interesse, como asa, perna, tórax e abdômen. O fluxo de trabalho de análise de imagem usa o programa gratuito de análise de imagem FIJI (NIH ImageJ). Essa metodologia torna a preparação de amostras, imagens de alta resolução e análises acessíveis para estudantes de graduação e cientistas em instituições com poucos recursos.
Protocolo
1. Coleta e fixação de Drosophila adulta
- Configure cruzamentos de Drosophila ou cepas selecionadas e coloque-as em frascos contendo comida para moscas. Incubar os frascos para injetáveis à temperatura desejada (geralmente 25 °C) até que as moscas se desenvolvam e os adultos se fechem (~10-14 dias a 25 °C).
- Anestesie as moscas com CO2 e coloque-as em uma almofada de CO2 .
- Separe as moscas com uma pena e selecione os indivíduos com o fenótipo desejado (por exemplo, asas retas). Faça um classificador de moscas de penas aparando uma pena de ganso para caber na extremidade cônica de uma pipeta sorológica de 1 mL.
- Prepare um tubo de microcentrífuga de 1,7 mL com 1 mL de etanol a 70%. Coloque as moscas selecionadas no tubo de microcentrífuga e coloque no gelo. Armazene os tubos da microcentrífuga a 4 °C durante a noite (Figura 2A).
NOTA: Não conserve moscas em etanol por mais de 24 h. O armazenamento prolongado de moscas em etanol a 70% resultará em perda de pigmento dos olhos e do corpo.
2. Preparação da amostra por montagem pontual
NOTA: Drosophila são insetos de corpo mole que se tornam quebradiços e colapsam quando secos ao ar; portanto, este protocolo exige que as amostras sejam fotografadas no mesmo dia em que são montadas. Trabalhe em pequenos conjuntos de ~5 moscas de cada vez para evitar a perda de amostra. Aumente o número de amostras em um conjunto com base na eficiência. Espécimes que requerem mais tempo antes da imagem podem ser desidratados por meio de uma série de concentrações crescentes de hexametildisilazano (HMDS)14.
- Corte pequenas pontas triangulares (7,1 mm x 2,7 mm) de cartolina de arquivo de 65 lb usando um punção de ponta especializado. Prepare os pontos dobrando a ponta (25% mais estreitos) em um ângulo de 90° com uma pinça de ponta fina Dumont #5 (Figura 2B).
- Usando a pinça de ponta fina Dumont #5, remova as moscas dos tubos da microcentrífuga (etapa 1.4). Seque suavemente as moscas com tecido de laboratório sem fiapos para remover o excesso de etanol. Posicione cada mosca do lado esquerdo em um cartão de índice sob um microscópio de dissecação.
NOTA: Remova as moscas do tubo segurando uma estrutura anatômica que não seja a área de interesse - ao obter imagens da cabeça, seguramos as amostras pela asa ou perna. Não segure as amostras pelo abdômen, pois essa estrutura é usada para colar a mosca na ponta do cartão. - Prepare a cola para pele, ajustando sua consistência à viscosidade desejada. Misture 1-2 gotas de cola de couro com 1-2 gotas de água deionizada (DI) e misture com uma pipeta de transferência em um cartão de índice. Pegue uma ponta de cartão preparada na extremidade larga com uma pinça e coloque uma pequena quantidade de cola diluída na ponta dobrada da ponta, esfregando-a na mistura de cola e água (Figura 2C).
NOTA: A cola deve ser espalhada, mas não escorrendo. - Aplique a ponta dobrada da ponta na face anterior do abdômen direito ao redor dos segmentos abdominais 2-3 (Figura 2C). Antes que a cola seque, faça pequenos ajustes na mosca, de modo que o eixo ântero-posterior da mosca fique perpendicular à ponta dobrada da ponta.
- Insira um pino de montagem nº 3 na extremidade larga da ponta do cartão (Figura 2D) e prenda-o a um bloco de fixação de insetos (Figura 2E). Rotule cada alfinete ou linha de alfinetes com o genótipo correspondente.

Figura 2: Preparação da amostra. (A) Drosophila adulta são classificadas com base em marcadores fenotípicos e coletadas em tubos de microcentrífuga rotulados contendo 70% de etanol em gelo. As moscas são armazenadas a 4 ° C durante a noite. (B) As pontas do cartão de papel são preparadas dobrando a extremidade estreita 90 ° do resto do cartão usando um par de pinças # 5. (C) As moscas são recuperadas dos tubos e deixadas secar brevemente ao ar. A cola de couro é aplicada na extremidade pequena e dobrada da ponta do cartão preparada e colada na mosca adulta nos segmentos abdominais 2-3. (D) As amostras são montadas, com uma etiqueta de identificação, em um pino de inseto de aço inoxidável # 3. (E) As amostras fixadas são armazenadas em uma placa de amostra até que estejam prontas para aquisição de imagens. Clique aqui para ver uma versão maior desta figura.
3. Aquisição de imagens de empilhamento de foco de alta resolução
- Adquira fotografias de alta resolução de olhos de mosca com um sistema de imagem de empilhamento de foco montado e personalizado, montado e personalizado.
- Capture fotografias com um corpo de câmera DSLR com uma lente telefoto de 70-200 mm conectada a uma objetiva de microscópio Apo 20x por meio de um adaptador de lente de 77 mm.
- Certifique-se de que a amostra esteja iluminada com um flash através de um difusor (Figura 3).
- Controle o posicionamento Z usando um controlador Stackshot e um trilho macro.
- Conecte a câmera, o flash e o estágio motorizado a um tripé de alumínio anodizado resistente.
- Posicione cada amostra montada em um gimbal de platina universal com a cabeça orientada de forma que o olho fique voltado para a lente. Faça ajustes na posição da cabeça movendo suavemente a cabeça com a pinça.
CUIDADO: Ajustes grandes e rápidos podem resultar em decapitação acidental. - Com a câmera conectada a um laptop, ajuste as configurações de aquisição no software. Fotografe espécimes com ampliação de 20x com estas configurações: Potência do flash 1/32, Velocidade do obturador 1/200, Abertura F2.8 e ISO 400. Certifique-se de que todos os recursos de foco automático e estabilização de imagem estejam desativados.
NOTA: Essas configurações equilibram a iluminação ideal do flash, a velocidade do obturador e a profundidade de campo. Eles precisariam ser ajustados para outras ampliações e/ou combinações de lentes. - Defina o local para salvar a pilha de imagens resultante (10 a 50 imagens) na pasta de arquivos desejada. Garanta capacidade de armazenamento suficiente para as imagens (~8,5 MB por imagem).
- Ajuste as configurações da pilha de foco na unidade de controle Stackshot no modo Distância automática .
- Defina o tamanho do passo para 5 μm e calcule o número de passos definindo as posições inicial e final da pilha de foco.
- View a amostra no modo LiveView e com a câmera no modo de disparo automático para identificar as posições de início e parada.
- Mova o trilho para que a parte mais próxima da amostra esteja em foco (defina a posição inicial ) e, em seguida, mova-se para onde a característica de interesse mais distante esteja em foco (defina a posição final ).
- Retorne a câmera para o modo de disparo manual e inicie a aquisição de imagem a partir da unidade de controle Stackshot.
NOTA: O tempo de aquisição da imagem depende do tamanho da amostra. Quanto maior a profundidade de campo necessária para espécimes grandes, mais fatias são incluídas na pilha de imagens, o que estenderá o tempo geral de aquisição. - Abra arquivos no software de empilhamento de foco referenciado. Gere uma imagem empilhada clicando em Pilha | Alinhar & Empilhar Tudo (PMax).
- Salve a imagem final no disco rígido do computador como um arquivo .tif clicando em Arquivo | Salve a imagem de saída.
NOTA: Dependendo da resolução do arquivo de imagem empilhado e do número de espécimes fotografados, discos rígidos externos grandes (1 TB) podem ser necessários para o backup da imagem. Nesse protocolo, as imagens empilhadas têm aproximadamente 100 MB cada antes de serem compactadas.

Figura 3: Aquisição de imagem. (A) Aparelho de imagem com partes rotuladas da seguinte forma: a) Corpo da câmera DSLR; b) lente teleobjetiva; c) Objetiva e Adaptador para Microscópio Apo 20x; d) Flash; e) Difusores de Lentes e Dome; f) Controlador Stackshot, Macro Rail e Estágio Rotativo; g) Gimbal de Palco Universal; h) Tripé. (B) Aparelho de imagem com difusor de luz instalado. (C) Close-up da amostra montada em posição para imagem. Clique aqui para ver uma versão maior desta figura.
4. Fluxo de trabalho de análise FIJI para calcular a área dos olhos adultos
- Para análise de imagens, obtenha o software FIJI15 no site da ImageJ.net.
- Escolha imagens para análise em que o olho esteja centralizado e alinhado com iluminação adequada e desfoque periférico mínimo, indicando o alinhamento adequado com a câmera.
- Calibre a escala da imagem.
- Baixe a imagem da barra de escala para ampliação de 20x que se correlaciona com 500 μm. Como alternativa, no momento da aquisição da imagem, fotografe uma régua usando as mesmas configurações. Abra a imagem no FIJI Software.
- Meça o comprimento da barra de escala (Figura 4A). Use a ferramenta Linha reta para traçar exatamente a linha. Clique em Analisar | Medir. Esta distância de pixel é equivalente a 500 μm (Figura 4B).
- Calcule pixels por mícron. Use isso para converter medições de pixel em medições de micrômetro.
- Abra o arquivo de imagem empilhado em FIJI (Figura 4C).
- Selecione Lupa na barra de ferramentas para ampliar a área de foco. Tente preencher a tela com o olho e a cutícula da cabeça imediatamente ao redor (Figura 4D).
- Selecione Ferramenta de seleção à mão livre na barra de ferramentas. Contorne a área retiniana o mais próximo possível, seguindo os contornos da fileira mais externa de omatídeos (Figura 4E). Para remover parte da seleção, mantenha pressionado o botão de opção e selecione os pixels a serem removidos. Para adicionar à seleção, mantenha pressionados os botões option e shift e selecione os pixels a serem adicionados.
- Para calcular a área, selecione Analisar | Meça no menu superior (Figura 4F). Uma nova janela aparecerá com os parâmetros área, média, mínimo e máximo . Copie e cole esses dados em uma planilha para documentação e conversão de pixels para medições de micrômetros.
- Realize análises estatísticas apropriadas.
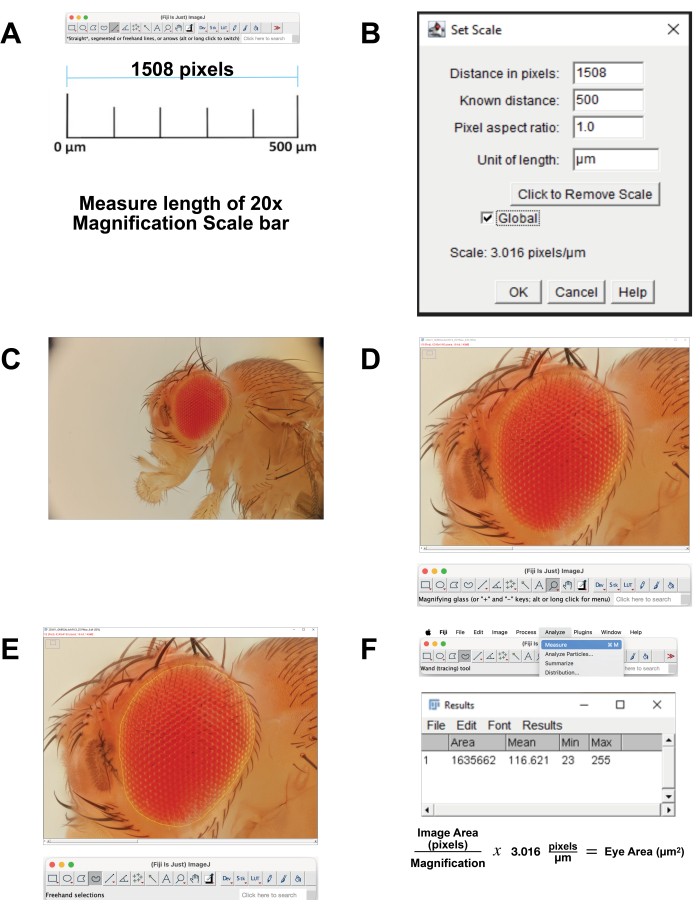
Figura 4: Análise de imagem em FIJI. (A) Dimensione a imagem original. Baixe a imagem de calibração e meça o comprimento da barra de 500 μm. (B) Ajuste a escala usando a função Set Scale . (C) Abra a imagem empilhada. (D) Amplie a imagem para que o olho fique centralizado e quase em tela cheia. (E) Use a ferramenta Seleção à mão livre para delinear o olho na borda entre a fileira mais externa de omatídeos e a cutícula circundante. (F) A medida da área dentro da região selecionada é calculada clicando em Analisar | Medida | Área. Clique aqui para ver uma versão maior desta figura.
Resultados
O olho de Drosophila é um excelente sistema modelo para estudar padrões de tecidos, controle de crescimento e morte celular. Recentemente, publicamos um estudo investigando como o pH intracelular (pHi) influencia o crescimento do tecido. Primeiro, estabelecemos um sistema genético em que a superexpressão do trocador de sódio-prótons DNhe2 (o ortólogo do NHE1 de mamíferos) no olho em desenvolvimento causa defeitos de padronização e aumento da proliferação
Discussão
Aqui descrevemos um método para preparação de amostras, imagens de alta resolução e análise de estruturas adultas de Drosophila . O olho de Drosophila é um sistema modelo geneticamente tratável que produziu insights críticos sobre os mecanismos moleculares subjacentes a doenças, incluindo câncer19, neurodegeneração20 e doenças metabólicas21. Em particular, os "avatares" de pacientes c...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores gostariam de agradecer aos membros do laboratório de pHly Grillo-Hill pelas discussões e apoio. Agradecemos a Tim Andriese, Randy Kirschner, Kitty (Ngoc-Huong) Nguyen, Marco Parent, Jonny Shaloub e Librado Veliz pelo excelente suporte técnico. Este trabalho foi apoiado pelos prêmios NIH SC3GM132049 e 1R16GM153640 (BKGH), um Prêmio de Pesquisa Docente-Estudantil de Biotecnologia da CSU (LM e BKGH) e fundos iniciais da Faculdade de Ciências e do Departamento de Ciências Biológicas da Universidade Estadual de San José (FJL). Menção especial vai para Bernd Becker por sua desenvoltura e assistência durante este processo. Agradecemos à comunidade BioIcons (https://bioicons.com/) por fornecer ícones de alta qualidade para nossas figuras e, especialmente, à Serviere pelo ícone de pipeta e DBCLS pelos ícones de Drosophila, fórceps e microscópio eletrônico de mesa usados na Figura 1 e Figura 2, que são licenciados sob CC-BY 4.0 Unported. Agradecemos também à comunidade SciDraw (https://scidraw.io/) por fornecer ícones de alta qualidade para nossas figuras, especialmente Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953), que estão licenciados sob a licença Creative Commons 4.0 (CC-BY).
Materiais
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
Referências
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados