Method Article
Микрокосмы компоста как микробно разнообразные, естественные среды для исследования микробиома в Caenorhabditis elegans
В этой статье
Резюме
Компостные микрокосмы приносят микробное разнообразие, обнаруженное в природе, в лабораторию, чтобы облегчить исследования микробиома у Caenorhabditis elegans. Здесь представлены протоколы для проведения экспериментов с микрокосмом, причем эксперименты демонстрируют способность модулировать микробное разнообразие окружающей среды для изучения взаимосвязей между микробным разнообразием окружающей среды и составом микробиома кишечника червей.
Аннотация
Нематода Caenorhabditis elegans становится полезной моделью для изучения молекулярных механизмов, лежащих в основе взаимодействий между хозяевами и их кишечными микробиомами. В то время как эксперименты с хорошо охарактеризованными бактериями или определенными бактериальными сообществами могут облегчить анализ молекулярных механизмов, изучение нематод в их естественном микробном контексте имеет важное значение для изучения разнообразия таких механизмов. В то же время выделение червей из дикой природы не всегда осуществимо, и, даже когда это возможно, отбор проб из дикой природы ограничивает использование генетического инструментария, доступного в противном случае для исследований C. elegans . Следующий протокол описывает метод исследований микробиома с использованием компостных микрокосм для роста в лаборатории в микробно разнообразных и естественных средах.
Почва из местных источников может быть обогащена продуктами для диверсификации микробных сообществ, в которых выращиваются черви и из которых они собираются, моются и стерилизуются для последующего анализа. Репрезентативные эксперименты демонстрируют способность модулировать микробное сообщество в общей почве, обогащая его различными продуктами, и дополнительно демонстрируют, что черви, выращенные в этих различных средах, собирают аналогичные кишечные микробиомы, отличные от их соответствующих сред, поддерживая идею видоспецифического основного кишечного микробиома. В целом, компостные микрокосмы обеспечивают естественную лабораторную среду для исследований микробиома в качестве альтернативы синтетическим микробным сообществам или изоляции диких нематод.
Введение
Нематода Caenorhabditis elegans становится полезной моделью для изучения взаимодействий между хозяевами и их кишечными микробиомами 1,2. Как модель, он предлагает несколько преимуществ. Во-первых, животные, свободные от микробов или гнотобиотические животные, легко получить и содержать; отбеливатель может быть использован для уничтожения гравидных червей и связанных с ними микробов, оставляя их устойчивые к отбеливанию яйца невредимыми для роста в качестве синхронизированных по возрасту популяций, которые могут быть колонизированы бактериями, представляющими интерес 3,4. Кроме того, при выращивании в присутствии бактерий C. elegans, бактеривора, проглатывает встречающиеся бактерии, при этом восприимчивые виды перевариваются или выводятся, в то время как устойчивые и стойкие виды стабильно колонизируют кишечник червя. Кроме того, C. elegans в основном гермафродитны, производя популяции генетически идентичного потомства, что уменьшает смешение генетических вариаций. В сочетании с наличием мутантных и трансгенных штаммов червей, работа с C. elegans предлагает исследователям гнотобиотическую и генетически поддающуюся обработке модель для исследования молекулярных основ взаимодействий хозяин-микроб 5,6,7,8.
В то время как эксперименты с хорошо охарактеризованными бактериями могут облегчить анализ молекулярных механизмов, выявление и изучение бактерий, с которыми черви взаимодействуют в природе, имеет важное значение для изучения разнообразия таких механизмов, разгадки естественного контекста для их функции и понимания селективных сил, которые сформировали их эволюцию. За пределами лаборатории C. elegans встречается во всем мире во влажном умеренном климате, где, как полагают, популяции подвергаются жизненному циклу «бума и спада», характеризующемуся быстрым ростом населения, когда ресурсы в изобилии, с последующим переходом развития к новаторским, стрессоустойчивым дауэрам, когда ресурсы истощаются9. Несмотря на то, что популяции C. elegans считаются почвенной нематодой, размножающиеся в дикой природе, чаще всего питаются разлагающимся органическим материалом, таким как гниющие цветы или фрукты, где бактериальные популяции многочисленны и разнообразны.
Исследования кишечного микробиома у нематод, выделенных из дикой природы, выявили разнообразные, но характерные бактериальные сообщества10,11, состав которых был дополнительно подтвержден исследованиями, проведенными с червями, выращенными в естественно-подобных микромировых средах12,13. Вместе такие исследования позволили очертить основной микробиом кишечника червя2. В то время как выборка популяций C. elegans в дикой природе представляет собой наиболее непосредственное исследование естественных взаимодействий червей и микробов, она не осуществима везде и в любое время, поскольку она ограничена регионами и сезонами с достаточным количеством осадков10,11. В качестве альтернативы, вместо выделения червей из их естественной среды обитания, эксперименты с использованием микрокосм приносят естественную среду обитания в лабораторию 6,8,12,13,14,15. Микромировые среды получают из почвы, компостированной различными фруктами или овощами, что позволяет дополнительно диверсифицировать исходное почвенное сообщество. Они предлагают поддающиеся обработке экспериментальные методы, которые сочетают микробное разнообразие и трехмерную дикую почвенную среду с экспериментальными преимуществами контролируемой лабораторной установки и генетически определенных штаммов червей. В приведенном ниже протоколе подробно описываются этапы, связанные с работой с компостными микрокосмами, демонстрируя их использование в понимании сборки характерного микробиома кишечника червя из различных сред.
протокол
1. Приготовление компоста
- Получите компост или садовую почву из любого удобного источника и храните внутри лаборатории в стандартном кухонном пластиковом контейнере с отверстиями, вырезанными в крышке, чтобы впустить воздух. Заткните отверстия ватой, чтобы не допустить плодовых мух и других беспозвоночных (рисунок 1А).
ПРИМЕЧАНИЕ: Пятьсот граммов почвы (помещаются в контейнер объемом 1,5 галлона, размеры: 30 см х 20 см х 10 см) обеспечат достаточно материала для 12 микрокосм. - Обогатите компост или почву измельченным продуктом или смесью различных продуктов в массовом соотношении продукта к почве 1:2.
- Инкубировать в течение 7-14 дней при 20-25 °C, смешивая один раз в день и добавляя среду M9 по мере необходимости для поддержания влаги, не делая ее грязной.
ПРИМЕЧАНИЕ: Почвы, не обогащенные продуктами, обычно не поддерживают рост C. elegans , но какой конкретный продукт использовать, зависит от исследователя, со многими типами и смесями, способными поддерживать рост червей. Обогащение различными продуктами будет способствовать диверсификации бактериального сообщества различными способами, позволяя изучать сборку кишечного микробиома из разных отправных точек (см. раздел обсуждения).
2. Приготовление микрокосм компоста
- Для каждого микромира добавьте 10 г обогащенного компоста в стеклянный стакан объемом 30 мл, покрытый оловянной фольгой и автоклавом (рисунок 1B).
- Чтобы приготовить микробный экстракт для пополнения автоклавного компоста, начните с добавления 30 г того же компоста, который использовался на этапе 2.1, в каждый из трех пробирок по 50 мл и заполните М9. Вихрь в течение 1 мин (рисунок 1С).
ПРИМЕЧАНИЕ: Это должно обеспечить достаточное количество бактерий для девяти микрокосм, каждая из которых поддерживает развитие сотен нематод. - Центрифугируют пробирки по 560 × г в течение 5 мин при комнатной температуре (RT).
- Обращая внимание на то, чтобы не потревожить гранулу, удалите супернатанты серологической пипеткой и соедините в новую трубку объемом 50 мл.
- Сконцентрируйте бактериальный экстракт путем центрифугирования на максимальной скорости (2000 × г) в течение 15 мин при RT. Повторно суспендируйте гранулы в достаточном количестве M9, чтобы иметь 200 мкл на каждый микрокосм и еще 200 мкл, чтобы добавить к пластине, которая будет служить видимым прокси развития червей внутри микрокосм.
ПРИМЕЧАНИЕ: Например, для девяти микрокосм повторно суспендировать микробную гранулу в 2 мл M9. - Добавьте 200 мкл концентрированного микробного экстракта в каждый стакан автоклавного компоста, а также в пластину NGM, которая будет служить видимой прокси-пластиной.
- Инкубировать микрокосмы и прокси-пластину в течение 24 ч при 20-25 °С перед добавлением червей.
3. Выращивание червей в компостных микрокосмах
- Добавьте 500-1000 яиц или личинок L13 к каждому микрокосму и к прокси-пластине (шаг 2.7), чтобы начать эксперимент (рисунок 1D).
ПРИМЕЧАНИЕ: В экспериментах, описанных здесь, используются черви дикого типа N2. Тем не менее, другие штаммы C. elegans (и, возможно, другие нематоды) могут быть использованы. - Поднимите червей до зрелого возраста при 20 °C (обычно 3 дня).
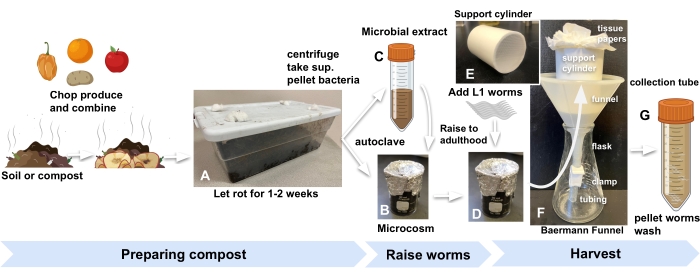
Рисунок 1: Подготовка микрокосмов компоста, выращивание червей и сбор урожая. (А) Обогатите местную почву или компост продуктами и инкубируйте в течение 2 недель. Смешайте (B) автоклавную обогащенную почву с (C) микробным экстрактом и (D) инкубируйте в течение не менее 24 ч, прежде чем добавлять синхронизированных червей L1s в микрокосмы, чтобы начать эксперимент. (Е, Ф) Когда будете готовы к сбору урожая, добавьте компост из микромира в цилиндр поддержки воронки Baermann и накройте M9. (G) Через 15 мин выпустите фильтрат в пробирку объемом 50 мл. Аббревиатура: суп. = сверхнатант. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
4. Подготовка воронки Baermann для заготовки червей
- Соберите воронку Baermann, прикрепив 5-8 см жесткой резиновой трубки к концу пластиковой воронки.
- Сдвиньте зажим на трубку и зажмите его.
- Поместите в воронку цилиндрическую трубу из ПВХ длиной 7 см, диаметром 5 см с приклеенной на ее дне нейлоновой сеткой 1 мм (рисунок 1Е). Выровняйте цилиндр двумя листами папиросной бумаги.
- Поместите воронку Baermann в колбу (рисунок 1F).
5. Сбор червей из микрокосмов и сбор соответствующих образцов почвы
- Добавьте 20 мл M9 в микромир, в котором были подняты черви, перемешайте смесь, а затем вылейте смесь из стакана в цилиндр с папиросной бумагой в воронке Baermann. Добавьте еще M9, чтобы полностью погрузить компост в воронку.
ПРИМЕЧАНИЕ: Популяции C. elegans (N2) достигают зрелого возраста в компостных микрокосмах с той же скоростью, что и на стандартных агаровых пластинах, засеянных кишечной палочкой. Для дальнейшей точности в сборе червей на определенной стадии развития обратитесь к прокси-пластине из шага 3.3. - Через 30 мин отстегните зажим, чтобы освободить фильтрат, содержащий собранных червей, в трубку объемом 50 мл (рисунок 1G).
- Добавьте больше M9 в цилиндр и повторите для второго раунда, чтобы собрать больше червей, и еще раз, если требуются дополнительные черви.
ПРИМЕЧАНИЕ: При повторении раундов сбора урожая будьте осторожны, чтобы не поставить под угрозу целостность папиросной бумаги, которая может позволить частицам почвы пройти. Максимум четыре раунда сбора урожая должны изолировать не менее 50% червей, первоначально добавленных в микромир, без ущерба для целостности папиросной бумаги. - Концентрировать червей путем центрифугирования при 560 × г в течение 2 мин (RT). Удалите 35 мл супернатанта серологической пипеткой.
- Переложите оставшиеся 15 мл в трубку объемом 15 мл и снова центрифугу при 560 × г в течение 1 мин для дальнейшей концентрации червей. Удалите 14 мл супернатанта серологической пипеткой.
- Параллельно соберите 1 г оставшейся почвы микромира в трубку объемом 1,5 мл. Немедленно обработайте образцы почвы, содержащие экологическое бактериальное сообщество, или храните их при -20 °C для последующей экстракции нуклеиновых кислот, как описано ниже для образцов червей.
6. Промывка и поверхностная стерилизация заготовленных червей
- Переложите 1 мл концентрированных, собранных червей со стадии 5,5 на трубку объемом 1,5 мл с помощью стеклянной пипетки. Инкубировать в течение 2 мин, чтобы черви осели на дне трубки. Удалите супернатант, оставив дно 100 мкл нетронутым.
- Промывайте 6x с 1,5 мл M9 + T (0,025% Triton-X в M9), позволяя червям каждый раз оседать на дне.
- Перенесите промытых червей в объеме 100 мкл в новую трубку объемом 1,5 мл с помощью стеклянной пипетки.
ПРИМЕЧАНИЕ: На этом этапе протокола должно было пройти примерно 30 минут с момента первой стирки (шаг 6.2). Позволяя червям оставаться без пищи в течение не менее 1 ч до добавления левамизола, рекомендуется обеспечить выведение преходящих бактерий и полное переваривание пищевых бактерий. - Добавьте 100 мкл 25 мМ гидрохлорида левамизола, чтобы парализовать червей. Инкубировать в течение 5 мин на РТ.
- Добавить 200 мкл 4% раствора отбеливателя. Инкубировать в течение 2 мин.
- Удалите супернатант, оставив самое нижнее 150 мкл нетронутым, и вымойте 3x с M9 + T, как указано выше.
ПРИМЕЧАНИЕ: Чтобы свести к минимуму загрязнение образцов, рекомендуется использовать фильтрованный M9 + T (через фильтр 0,2 мкм) и носить перчатки с этого момента. - После промывки возьмите 50 мкл из оставшихся 150 мкл от последней промывки и пластины на пластине LB, инкубируя при 25 °C в течение 48 ч, чтобы подтвердить эффективное удаление внешних бактерий.
ПРИМЕЧАНИЕ: Наблюдение до 30 колоний в последней промывке (представляющих 60 внешних бактериальных клеток, в общей сложности оставшихся в образце) разрешено и, как ожидается, будет иметь лишь незначительный вклад в анализируемый состав микробиома, учитывая тысячи бактерий, которые обычно колонизируют каждого взрослого червя15. Когда наблюдается больше, целостность образца может быть нарушена. - Немедленно используйте поверхностно-стерилизованных червей (например, для извлечения живых бактерий для культивирования [количество КОЕ]) или храните при -20 °C для последующей экстракции нуклеиновых кислот.
7. Извлечение ДНК
ПРИМЕЧАНИЕ: Следующие шаги описывают экстракцию ДНК собранных червей с использованием коммерческого набора, предназначенного для извлечения микробной ДНК из почвы (см. Таблицу материалов), с модификациями, описанными ниже, чтобы облегчить извлечение микробной ДНК из червей.
- Перенесите образец, содержащий поверхностно-стерилизованных червей (при необходимости размораживающий при RT), в трубки, предусмотренные комплектом, заменив предоставленные стеклянные шарики шариками диаметром циркония диаметром примерно 30-50 1 мм (см. Таблицу материалов).
ПРИМЕЧАНИЕ: Наблюдаемые выходы ДНК выше с шариками циркония, чем со стеклянными бусинами, предоставляемыми набором. Хотя причина этого наблюдения остается неопределенной, шарики циркония могут более эффективно разрушать кутикулу нематоды, высвобождая больше бактерий. - Для образцов компоста добавьте примерно 250 мг собранной почвы в трубки комплекта без замены стеклянных шариков.
- После добавления буферного раствора, предусмотренного комплектом, ко всем образцам гомогенизируют с помощью силового гомогенизатора на RT (см. Таблицу материалов) в течение двух раундов по 2000 об/мин в течение 30 с каждый, делая паузу на 30 с между ними.
- Завершите оставшиеся этапы очистки ДНК в соответствии с протоколом набора. Элюируют образцы червей не более чем в 50 мкл элюционного буфера, чтобы обеспечить концентрации ДНК, которые достаточно высоки для секвенирования.
Результаты
Чтобы изучить способность диверсифицировать сообщество почвенных микрокосмов, мы сравнили микробные сообщества в компостных микрокосмах, полученных путем обогащения одной и той же исходной почвы, компоста промышленного класса, доступного из города Беркли, штат Калифорния, различными продуктами: яблоками, болгарским перцем, апельсинами или картофелем (каждый набор в трех экземплярах). Мы также сравнили микробные сообщества каждой компостной среды с кишечным микробиомом диких C. elegans , выращенных в соответствующем микромире. Анализ проводился с образцами ДНК, извлеченными примерно из 500 поверхностно стерилизованных взрослых на микрокосм и из образцов компоста 250 мг соответствующих микрокосм.
Характеристика экологических микробиомов почвы и кишечника червей основывалась на секвенировании следующего поколения области V4 бактериального гена 16S рРНК. Подготовка библиотеки секвенирования осуществлялась с использованием стандартных комплектов и выполнялась в соответствии с инструкциями производителей, при этом секвенирование выполнялось на коммерческом секвенсоре (см. Таблицу материалов). Демультиплексированные последовательности обрабатывали с использованием DADA2, присваивали таксономию на основе справочной базы данных SILVA v132 и анализировали с помощью phyloseq 16,17,18 (см. Дополнительный файл 1, Дополнительный рисунок S1, Дополнительный рисунок S2, Дополнительный рисунок S3, Дополнительная таблица S1 и Дополнительная таблица S2 для подробного описания последовательности и анализа; полный вычислительный конвейер доступен в GitHub [https://github.com/kennytrang/CompostMicrocosms]). Необработанные данные доступны в архиве чтения последовательностей NCBI (идентификатор биопроекта PRJNA856419).
В среднем на выборку было получено 73 220 последовательностей. Эти последовательности представляют собой 15 027 вариантов последовательности ампликонов (ASV), охватывающих 27 типов и 216 семейств, включая семейства, считающиеся частью основного микробиома кишечника C. elegans 13, такие как Rhizobiaceae, Burkholderiaceae и Bacillaceae. Enterobacteriaceae и Pseudomonadaceae, которые ранее были признаны доминирующими членами, на этот раз были меньшинством, но все еще были обогащены (в 2-10 раз) по сравнению с их соответствующими почвенными средами. Сравнения, основанные как на невзвешенных, так и на взвешенных расстояниях UniFrac19,20, продемонстрировали хорошую воспроизводимость среди трипликатов микромира, обогащенных одним и тем же продуктом, о чем свидетельствует тесная кластеризация. Напротив, экологические почвенные микробиомы, обогащенные различными продуктами, сгруппированными вдали друг от друга, демонстрируя способность диверсифицировать исходное микробное сообщество путем добавления различных продуктов (рисунок 2).
При сравнении микробиомов кишечника червей и экологических сообществ анализ основных координат (PCoA) с невзвешенными или взвешенными расстояниями UniFrac показал отчетливую кластеризацию микробиомов кишечника червей вдали от их соответствующих сред для каждого типа микромира (рисунок 2). В то время как PCoA, основанный на невзвешенных расстояниях UniFrac, не проводил различий между микробиомами почвы и червей (рисунок 2A), кластеризация на основе взвешенных расстояний выявила четкое разделение микробиомов кишечника червя и компоста (рисунок 2B). Эти результаты поддерживают процесс, в котором фильтрация хозяина работает на экологическую доступность для формирования кишечного микробиома, который не полностью отличается от своего экологического источника в отношении присутствия таксонов, но модулирует их численность, обогащая подмножество доступных таксонов, что в конечном итоге приводит к основному микробиому кишечника червя, разделяемому между червями, выращенными в разных средах.

Рисунок 2: Микробиомы кишечника червей группируются вдали от их соответствующих пищевых разнообразных микробных сред. Состав микробиома определяли с помощью секвенирования 16S, и сообщества из микрокосм, обогащенных назначенным продуктом, или из червей, выращенных в них, группировали с использованием PCoA на основе (A) невзвешенных или (B) взвешенных расстояний UniFrac. Показаны оси, которые объясняют наибольшие различия в составе сообщества между образцами (N = 3 для каждого типа микромира). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный файл 1: Секвенирование и анализ данных нового поколения. Здесь представлены шаги по подготовке библиотеки, лабораторному секвенированию и анализу данных. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок S1: Пример графика контроля качества для обратного считывания из одного образца. Ось X (цикл) показывает положение нуклеотида вдоль считываемой последовательности. Левая ось Y показывает показатель качества. Тепловая карта в оттенках серого представляет частоту показателя качества в каждой нуклеотидной позиции; зеленая линия изображает медианный показатель качества в каждой нуклеотидной позиции; верхняя оранжевая линия изображает квартили распределения показателей качества; нижняя красная линия изображает процент показаний последовательности, которые расширили это положение нуклеотида (правая ось Y, здесь 100%). Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок S2: Частота ошибок для различных образцов. Частота ошибок в различных выборках (черные точки) должна уменьшаться с увеличением показателя качества для каждой возможной замены пары оснований, отражающей ожидаемую тенденцию. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный рисунок S3: Пример PCoA на основе взвешенных расстояний UniFrac. Названия групп, показанные в легенде, представляют продукт, используемый для обогащения компоста, используемого в различных микрокосмах. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица S1: Последовательная фильтрация последовательностей. a Количество последовательностей, считываемых перед фильтрацией. b-d Каждый столбец представляет количество показаний последовательностей, оставшихся после этапа фильтрации: фильтрация считываний низкого качества (шаг 2.5), алгоритм шумоподавления, выполняемый dada() (шаг 2.8), слияние прямого и обратного считывания (шаг 2.9) и удаление химер (шаг 2.11). Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица S2: Таблица метаданных. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Представленный здесь протокол описывает метод изучения кишечного микробиома нематод, выращенных в естественно-подобных средах, предлагая альтернативный подход к выделению червей от природы или к их выращиванию на синтетических сообществах.
Тысячи потенциальных видов бактерий, захваченных в эксперименте с репрезентативным микрокосмом, отражают микробное разнообразие, с которым развились черви, и демонстрируют способность конвейера микрокосма сочетать преимущества работы с модельным организмом-хозяином и преимущества работы с естественными, разнообразными микробными сообществами.
Репрезентативные результаты показывают, что обогащение общей почвы различными типами продуктов модулирует экологическое микробное разнообразие, подчеркивая диапазон микробного разнообразия, доступного для разведки с использованием этого трубопровода. Выбор продукции не особенно важен. Предыдущая работа использовала бананы, яблоки, апельсины, клубнику, листья зеленого чая и картофель для обогащения почвы, что привело к повышению эффективности подобных червей. Смешанная продукция также используется эффективно. Ключевая особенность заключается в том, что различные продукты будут диверсифицировать данную почву различными способами.
Несмотря на широкие различия в микробном разнообразии окружающей среды, микробиомы кишечника червей значительно меньше различаются, что подтверждает важность ниши кишечника червя и фильтрации хозяина для сборки основного микробиома кишечника, который отличается от его почвенной среды13. Эта картина лучше всего наблюдается при взвешенном анализе UniFrac PCoA, демонстрирующем, что различия между экологическими почвенными и кишечными микробиомами червей проистекают в основном из различий в относительном изобилии ключевых таксонов.
Хотя протокол, описанный здесь, фокусируется на сборе червей для секвенирования 16S, микрокосмы могут быть использованы для изучения дополнительных вопросов, представляющих интерес. Например, черви, собранные из микрокосм, могут быть впоследствии исследованы на предмет влияния разнообразного кишечного микробиома на устойчивость хозяина к различным неблагоприятным условиям, включая патогены или токсины. В качестве альтернативы, новые виды бактерий и штаммы могут быть выделены и культивированы из наземных червей, расширяя таксономическое и функциональное разнообразие бактерий, доступных для проведения экспериментов.
В то время как методы исследования в лабораторных условиях стремятся к согласованности и воспроизводимости, работа с микрокосмами использует естественные вариации для изучения взаимодействий хозяина и микроба в естественном контексте. Тем не менее, это изменение также создает некоторые проблемы. Некоторые почвы, которые включают высокие уровни эндогенных беспозвоночных, могут потребовать дополнительных этапов центрифугирования, фильтрации и обследования для эффективного устранения нежелательных организмов из препарата микромира. Кроме того, низкое микробное изобилие в почве может вызвать нежелательное образование дауэра в популяциях червей, требуя увеличения микробного экстракта или обогащения почвы большим количеством продукта. С каждым экспериментом с микрокосмом исследователи продолжают изучать полное таксономическое и функциональное разнообразие, обеспечиваемое природой, что позволяет открывать новые микробные таксоны и функциональные способности, начиная от устойчивости к инфекциям до защиты от ксенобиотиков окружающей среды.
Раскрытие информации
Авторы не имеют конфликта интересов, чтобы заявить, что имеют отношение к содержанию данной статьи.
Благодарности
Работа, описанная в этой рукописи, была поддержана грантами NIH R01OD024780 и R01AG061302. K.T. был также поддержан Летней исследовательской стипендией для студентов из Калифорнийского университета в Беркли, финансируемой Фондом Роуз-Хиллз. Мультяшные рисунки на рисунке 1 были получены от BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| AMPure XP Reagent, 60 mL | Beckman Coulter | A63881 | Supplementary File Step 1.3 |
| Bleach (Sodium Hypochlorite) | Sigma-Aldrich | 7681-52-9 | Step 6.5 |
| DNeasy PowerSoil Pro Kit | Qiagen | 47016 | Step 7 DNA extractions |
| dNTP set 10 mM | Invitrogen | 18427013 | Supplementary Step 1.2 |
| Easypet 3 Serological Pipette Controller | Eppendorf | 4430000018 | Used to remove supernatant when specified |
| Greiner Bio-One 25 mL Sterile Serological Pipets | Fisher Scientific | 07-000-368 | Used to remove supernatant when specified |
| KH2PO4 | Fisher Scientific | P285-500 | Used to make M9 |
| Levamisole Hydrochloride | Fisher Scientific | AC187870100 | Step 6.4 |
| M9 Minimal Media Solution | Prepared in-house | N/A | Recipe in wormbook.org |
| MgSO4 | Fisher Scientific | M63-500 | Used to make M9 |
| MiniSeq High Output Reagent Kit (150 cycles) | Illumina | FC-420-1002 | Supplementary Step 1.7 |
| MiniSeq System | Illumina | SY-420-1001 | Commercial sequencer used; Supplementary Step 1.7 |
| Na2HPO4 | Fisher Scientific | S374-500 | Used to make M9 |
| NaCl | Fisher Scientific | S271-3 | Used to make M9 |
| Nematode Growth Media (NGM) | Prepared in-house | N/A | Recipe in wormbook.org |
| Nextera XT DNA Library Preparation Kit (96 samples) | Illumina | FC-131-1096 | Library prep kit used; Supplementary Step 1.4 |
| PhiX Control v3 | Illumina | FC-110-3001 | Supplementary Step 1.7 |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | M0530L | Supplementary Step 1.2 |
| PowerLyzer 24 Homogenizer (110/220 V) | Qiagen | 13155 | Step 7.3 |
| Qubit dsDNA HS Assay Kit | Invitrogen | Q32851 | Supplementary Steps 1.1 & 1.6 |
| Qubit Fluorometer | Invitrogen | Q33238 | Supplementary Steps 1.1 & 1.6 |
| Triton X-100 | Fisher Scientific | BP-151 | Used to prepare M9+T |
| Zirconia/Silica Beads 1.0 mm diameter | Fisher Scientific | NC9847287 | Step 7.1 |
Ссылки
- Shapira, M. Host-microbiota interactions in Caenorhabditis elegans and their significance. Current Opinion in Microbiology. 38, 142-147 (2017).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 285(2017).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 7-8 (2006).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Ortiz, A., Vega, N. M., Ratzke, C., Gore, J. Interspecies bacterial competition regulates community assembly in the C. elegans intestine. ISME Journal. 15 (7), 2131-2145 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10, 604(2019).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Montalvo-Katz, S., Huang, H., Appel, M. D., Berg, M., Shapira, M. Association with soil bacteria enhances p38-dependent infection resistance in Caenorhabditis elegans. Infection and Immunity. 81 (2), 514-520 (2013).
- Frézal, L., Félix, M. A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: Gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Berg, M., Zhou, X. Y., Shapira, M. Host-specific functional significance of Caenorhabditis gut commensals. Frontiers in Microbiology. 7, 1622(2016).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. ISME Journal. 10 (8), 1998-2009 (2016).
- Slowinski, S., et al. Interactions with a complex microbiota mediate a trade-off between the host development rate and heat stress resistance. Microorganisms. 8 (11), 1-9 (2020).
- Pérez-Carrascal, O. M., et al. Host preference of beneficial commensals in a microbially-diverse environment. Frontiers in Cellular and Infection Microbiology. 12, 795343(2022).
- Callahan, B. J., et al. DADA2: High-resolution sample inference from Illumina amplicon data. Nature Methods. 13 (7), 581-583 (2016).
- Quast, C., et al. The SILVA ribosomal RNA gene database project: Improved data processing and web-based tools. Nucleic Acids Research. 41, Database issue 590-596 (2013).
- McMurdie, P. J., Holmes, S. phyloseq: An R package for reproducible interactive analysis and graphics of microbiome census data. PLoS One. 8 (4), 61217(2013).
- Lozupone, C., Knight, R. UniFrac: A new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology. 71 (12), 8228-8235 (2005).
- Lozupone, C., Lladser, M. E., Knights, D., Stombaugh, J., Knight, R. UniFrac: An effective distance metric for microbial community comparison. ISME Journal. 5 (2), 169-172 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены