Method Article
在关系 (MySQL) 和 NoSQL (MongoDB 和存在) 中执行复杂度增加的查询大小-增长的 ISO/EN 13606 标准化的电子病历数据库
摘要
本研究比较关系和非关系 (NoSQL) 标准化的医疗信息系统。使用双倍大小的数据库计算查询此类数据库管理系统 (DBMS) 的响应时间的计算复杂性。这些结果有助于讨论每个数据库方法对不同方案和问题的适用性。
摘要
本研究显示了一种评估查询关系和非关系 (NoSQL (不仅仅是结构化查询语言)) 标准化电子健康记录 (EHR) 医疗信息数据库系统 (DBMS) 的计算复杂性的协议。它使用了一套三倍大小的数据库,即存储5000、1万和2万实际标准化的电子病历提取物的数据库, 在三个不同的数据库管理系统 (DBMS) 中: 关系 MySQL 对象-关系映射 (ORM),基于文档的 NoSQL MongoDB 和本机可扩展标记语言 (XML) NoSQL 存在。
计算了六个复杂度增加查询的平均响应时间, 结果显示了 NoSQL 情况下的线性行为。在 NoSQL 领域, MongoDB 呈现出比存在更平坦的线性斜率。
由于医疗信息更新政策的特殊性, NoSQL 系统也可能更适合维持标准化的医疗信息系统, 这不应影响存储在 NoSQL 数据库中的数据的一致性和效率。
此协议的一个限制是, 缺少改进的关系系统 (如原型关系映射 (ARM)) 与相同数据的直接结果。然而, 将双倍大小的数据库结果插到文献和其他公布的结果中, 表明 NoSQL 系统在许多具体的场景和需要解决的问题上可能更合适。例如, NoSQL 可能适合于以文档为基础的任务, 如临床实践中使用的电子病历提取物、编辑和可视化, 或其目的不仅是查询医疗信息, 而且还能以完全的原始形式恢复电子病历的情况。
引言
NoSQL (不仅 SQL) dbms 最近成为传统关系 dbms (RDMBS) 的替代方法。在数据库系统中, RDBMS 已经占据了数十年的数据存储方式。经过良好研究和理解的关系代数和微积分, 保证了 RDBMS1的效率和一致性。NoSQL 系统不会成为关系系统的替代品, 但在某些情况下和在几个条件下它们可以表现得更有利。
在设计用于管理和存储医疗信息的电子健康记录 (EHR) 系统的数据库持久性时, 会出现这些特定的情况和条件。为了在实践中可互操作和可持续, 几个国际标准, 如 ISO/EN 13606, openEHR 和 HL72,3,4,5已被用来标准化的电子病历提取。
ISO/EN 13606 和 openEHR 等几个标准将信息和知识分成两个不同的抽象级别, 由参考模型 (RM) 和称为原型的特殊数据结构表示。这种分离通常称为双重模型, 它允许电子病历系统在语义上互操作和医学知识的进化, 而无需重新编程整个电子病历系统, 因此, 在实践中可维护和可持续性6.但是, 在标准化的电子病历系统中实现的双重模型要求信息的组织遵循特定的结构, 这对系统的数据库持久性级别设计为7的方式产生了深刻的影响。
对象关系映射 (ORM)8是使用关系数据库范式实现电子病历系统的一种方法。ORM 详尽地映射了系统中用于关系数据库的标准化的 EHR 提取 XML (可扩展标记语言) 文件的结构。ORM 构建了许多关系表, 它遵循标准化的 EHR 提取 XML 文件的结构。这些关系表通过许多外键关联, 结果系统可能效率不高。
对 ORM 的几个关系改进已经提出。openEHR 的节点 + 路径9通过将整个抽取 XML 文件的子树序列化为 blob (二进制大对象) 来减少关系表的数量。但是, 这种简化会导致复杂的检索逻辑, 破坏复杂的查询。原型关系映射 (ARM)10生成由原型驱动的数据库模型, 基于原型和关系表之间的映射构建新的关系架构。因此, 一些非医疗信息的电子病历提取物丢失。
许多基于文档的 NoSQL 数据库将整个文档存储为与原始 XML 或 JSON (JavaScript 对象表示法) 格式相同的整个 blob。这意味着不构造关系表。这些 NoSQL 数据库没有架构, 它们不支持联接或 (酸性) 属性11、即、原子性、一致性、隔离或耐久性。因此, 如果文档的某个元素使用间接链接引用相同或其他此类文档的元素, 则它们可能非常低效。发生这种情况的原因是, 为了保持一致性, 必须按顺序处理所引用文档的全部内容。但是, 如果 DBMS 执行的主要任务是基于文档的任务, 则非关系数据库可能仍然是适当的。这是因为数据可能保持在一个表单中, 它使用基于文档的 NoSQL 数据库更接近于其真实表示形式, 尽管这也是由于电子病历文档所完成的特殊持久性策略 (参见讨论部分)。
这些方法的目的是展示几个实验, 以比较使用三不同 DBMSs 的标准电子病历系统的持久性层的实现: 一个关系 (MySQL) 和两个 NoSQL (基于文档的 MongoDB 和本机 XML 存在)。他们的计算复杂性已经计算和比较使用三种不同的增加大小的数据库和六不同的复杂性增加查询。在已执行查询的同一台计算机上, 已在本地安装并配置了三数据库服务器。有关技术详细信息, 请参阅材料表。
为了比较关系 MySQL 和 NoSQL MongoDB DBMSs 的性能, 还进行了并发实验。还使用文献10中的相关适当结果比较了描述的 ORM 改进 (节点 + 路径和 ARM)。
数据库管理系统正在以加速速度不断发展。当唯一现存的范式是关系模型时, 没有人会想到这个指数的发展。如要举一个例子, 请参见12, 其中提出了一个模型来实现响应时间改进的关系数据库, 以保留 ACID 属性。
研究方案
1. 建立一个关系 MySQL DBMS 来存储三个双标标准的电子病历提取数据库
- 将与 ISO/EN13606 RM 和 ISO21090 数据类型相对应的 W3C (万维网联合体) XML 架构导入到 "Java IDE" (集成开发环境) 中。
注: ISO 代表国际标准组织。EN 代表欧洲规范。 - 在 IDE 中执行JAXB (Java XML 绑定) 插件。这会产生与电子病历的元素结构相对应的 Java 类文件。
- 手动标记使用 JPA 标签生成的 Java 类。这些标签指的是 MySQL 数据库的关系表之间的基数和其他关系。
- 将JPA (Java 持久性 API) 的库导入到 IDE 中, 并执行从标记的 Java 类中生成MySQL数据库的方法。
- 创建三个目录与 5000, 1万和2万现实的电子病历提取 XML 文件。
- 执行 JPA 方法, 在5000提取目录的所有提取物上将 XML 提取物加载到 MySQL DBMS 中。
- 重复步骤1.6 两次, 一次与1万提取目录, 一次与2万提取目录。
2. 建立一个 NoSQL MongoDB DBMS 来存储三个双标标准的电子病历提取数据库
- 处理包含5000、1万和2万真实的电子病历的三个目录中的每个都提取 xml 文件, 并使用标准程序将 xml 文件转换为 JSON 文件 (如 json.org.XML)。应生成三个具有5000、1万和 2万 JSON 文件的目录。
- 启动 MongoDB GUI (图形用户界面, 请参阅材料表)。
- 启动从 DOS (磁盘操作系统) 系统窗口执行mongod程序的 MongoDB 2.6服务器。
- 使用端口27017将 MongoDB GUI 连接到本地主机服务器。
- 选择 "连接" 菜单。
- 为连接编写名称 (例如 "第一")。
- 在 DB 服务器文本框中写入 localhost:27017。
- 按 "连接" 按钮。应在左侧显示具有当前数据库的树。
- 建立一个包含5000标准的电子病历提取物的数据库。
- 单击左侧树顶部的连接名称。
- 选择 "文件" 菜单。
- 选择 "添加数据库"。
- 在出现的对话框中输入数据库的名称。
- 单击 "确定"。
- 建立一个包含5000标准的电子病历提取物的集合。
- 单击左侧树中数据库的名称。
- 选择菜单 "数据库"。
- 选择 "AddCollection"。
- 在出现的对话框中输入集合的名称。
- 单击 "创建"。
- 单击集合的名称。
- 选择 "导入" 菜单。
- 选择单选按钮 "JSON-蒙壳//mongoexport"。
- 单击 "下一步"。
- 按 "添加源文件" 按钮。
- 使用对话框在计算机上导航。
- 打开包含5000个 JSON 提取文件的目录。
- 选择目录中的所有文件。
- 按 "打开"。JSON 文件的列表应显示在 "导入" 对话框中。
- 按 "下一步"。数据库中新集合的预览将显示在左侧。
- 按 "下一步"。
- 按 "开始导入"。导入的进度显示在左侧, 指示导入的文件数和运行时间。
- 重复步骤5和 6, 建立1万标准的电子病历提取物的集合。
- 重复步骤5和 6, 建立2万标准的电子病历提取物的集合。
3. 构建一个 NoSQL 存在的 DBMS 存储三个双标标准的电子病历提取数据库
- 启动存在数据库。
- 使用数据库的图标, 打开 Java 管理客户端。
- 输入管理员密码。
- 按 "连接" 按钮。
- 建立一个包含5000标准的电子病历提取物的集合。
- 在工具栏中, 选择菜单 "创建新集合"。
- 在出现的对话框中, 键入新集合的名称。
- 单击 "接受";将显示新的集合。
- 选择集合的名称。
- 在工具栏中, 选择菜单 "存储数据库中的文件"。
- 使用对话框在计算机上导航。
- 选择包含5000标准化 XML 提取文件的目录。
- 单击 "选择要存储的文件或目录" 按钮。请注意, 出现一个对话框, 显示进度、正在存储的文件以及创建的数据库的百分比。
- 重复步骤5构建包含1万标准的电子病历提取物的集合。
- 重复步骤5构建包含2万标准的电子病历提取物的集合。
4. 在3关系 MySQL 数据库中设计和执行6复杂度增加的查询
- 根据电子病历提取物所使用的原型设计六复杂度增加的查询。
- 使用 MySQL 数据库上的第一个查询编程 SQL 脚本。由于提取标准化 (原型), SQL 必须适应 MySQL 数据库的特殊结构。数据库映射提取物的整个结构。因此, SQL 查询相当复杂。
- 标识数据库的属性, 如果建立了索引, 则可以加快查询的响应时间, 然后构造此类索引, 尽管大多数索引是由 DBMS 自动生成的。
- 如果查询需要非自动生成的索引, 请手动生成它。
- 连接到 MySQL 服务器 (辅助图 1)。
- 选择并单击左侧的数据库名称。
- 选择并单击索引字段所在的关系表。
- 单击选项卡 "结构"。
- 选择并单击要生成索引的列。
- 单击 "索引"。请注意, 生成索引的 SQL 语句将出现, 并显示一条消息, 指出已成功生成该句子。
- 执行第一个查询。
- 选择并单击左侧的数据库名称。
- 单击 "SQL" 选项卡。
- 写入或粘贴第一个查询的 SQL 代码 (请参见辅助图 2)。
- 按 "继续"。请注意, 将显示结果列表的第一个屏幕, 以及带有查询执行时间的消息。
- 重复执行5次并计算平均响应时间。
- 重复步骤 5, 查询2到6。
- 执行整个过程三次, 其中5000、1万和2万提取数据库。
5. 在 3 NoSQL MongoDB 数据库中设计和执行6复杂度增加的查询
- 启动 MongoDB GUI (请参阅材料表)。
- 从 DOS 系统窗口启动执行mongod程序的 MongoDB 2.6 服务器 (请参阅辅助图 3)。
- 按照步骤 2.4, 使用端口27017将 MongoDB GUI 连接到本地主机服务器。
- 选择并展开左侧的 MongoDB 数据库。
- 选择集合。
- 单击工具栏中的 "收藏" 菜单。
- 执行第一个 MongoDB 查询。
- 双击 "查询生成器" 按钮。
- 双击右侧查询生成器的 "查询字段"。
- 在 "查询" 面板的 "字段" 文本框中写入 MongoDB 查询的字段 (请参见辅助图 4)。
- 在 "查询" 面板的 "值" 文本框中写入 MongoDB 查询的值。
注意: 此查询应类似 {"ns3:EHRExtract.allCompositions.content.items.parts.parts.name.ns2:originalText"。值 ":" Descripcion "}。字段和值用分号进行引用和分隔。 - 双击 "查询生成器" 的投影字段
- 在投影文本框中写入第一个投影 (请参见辅助图 5)。
- 双击 "投影" 字段以添加新的投影文本框。
- 在投影文本框中写入第二个投影。
注意: 投影选择查询检索到的文档的一部分。这些应该是类似 {"ns3:EHRExtract" 的东西。allCompositions.content.items.parts.parts.value.value ": 1} 和 {" ns3: EHRExtract. 全部 Compositions.content.items.parts.parts.value.nullFlavor ": 1} - 单击 "蓝色播放" 按钮执行查询。
- 在 "查询代码" 选项卡中可视化查询代码。
- 在 "解释" 选项卡中查看结果的详细信息: 结果数、执行时间 (以毫秒为单位)。
- 在 "结果" 选项卡中查看、展开和检查结果。
- 如果需要进一步处理查询, 请用 MongoDB java 驱动程序编写一个 java 程序, 并使用查询和处理结果的方法。
- 重复执行5次并计算平均响应时间。
- 对其余2到6查询执行步骤5.7。
- 重复在5000、1万和2万中提取 MongoDB 数据库的整个过程。
6. 设计和执行在 3 NoSQL 存在数据库6增长复杂性查询
- 启动存在DBMS。
- 打开 Java 管理客户端。
- 按按钮 "连接到数据库"。
- 选择该数据库并单击它。
- 单击菜单 "使用 XPath 查阅数据库";将出现 "咨询" 对话框。
- 执行第一个XPath 查询(请参见辅助图 6)。
- 在对话框的上部写入或粘贴第一个查询的 XPath 代码。
- 在对话框的工具栏上单击菜单 "执行"。
- 使用对话框下部的 "XML" 选项卡查看 XML 结果。
- 查看对话框底部的结果数和编译和执行时间。
- 重复执行5次并计算平均响应时间。
- 对查询2到6重复步骤6。
- 执行整个过程三次, 对于5000、1万和2万抽取存在数据库。
7. 使用 MySQL 和 MongoDB 5000 提取数据库设计和执行并发实验
注: 由于先前实验的性能较差, 存在的数据库在这个时刻已被从实验中删除。
- 使用5000抽取数据库 (通常在几秒钟内), 在以前的实验中选择具有三最短时间响应的查询。
- 如果需要, 为这些查询确定并手动生成相应的属性索引。
- 编程两个 Java 多线程应用程序, 一个用于 MySQL, 另一个用于 MongoDB;每个应用程序将有三个不同的优先级线程, 一个用于在步骤1中选择的每个查询。
- 执行和计算每个线程 (查询) 的CPU (中央处理单元)使用分发。
- 执行每个多线程应用程序, 在每个10分钟范围内单击执行按钮五次, 并计算最执行的 (最高优先级) 查询平均吞吐量和三查询的平均时间响应。
- 使用优先级和执行时间查看执行中的查询。
- 计算每三个查询的平均吞吐量和平均响应时间。
结果
在表 1中显示了六种不同的查询, 其中包含有关病人问题的信息, 包括其名称、初始日期和最终时间和严重性。
每个 DBMS 中三个加倍大小的数据库中六查询的平均响应时间显示在表 2-4中。图 1-6以图形方式显示相同的结果 (请注意, 垂直坐标轴在这些数字中使用的比例非常不同)。
在 NoSQL 数据库的所有查询中, 计算复杂度的强线性行为是显而易见的, 尽管由于使用的3数据集的大小相对较小, 因此需要适当的谨慎。但是, 关系 ORM 数据库显示了一个不明确的线性行为。MongoDB 数据库的斜率比存在的数据库要平坦得多。
在文献中发表的介绍中讨论的改进的关系系统的结果可以在表 5中找到。将表 3中的 MongoDB 结果与类似的查询和数据库大小相同的结果从表 5等同于 Q1 中的两个数据库系统, 但在 Q3 中有利于 MongoDB。
并发实验的结果可能在表 5和table6中找到。MongoDB 在吞吐量和响应时间上都能击败 MySQL。事实上, MongoDB 在并发性方面的表现比隔离的更好, 并且在并发执行中作为一个令人印象深刻的数据库。
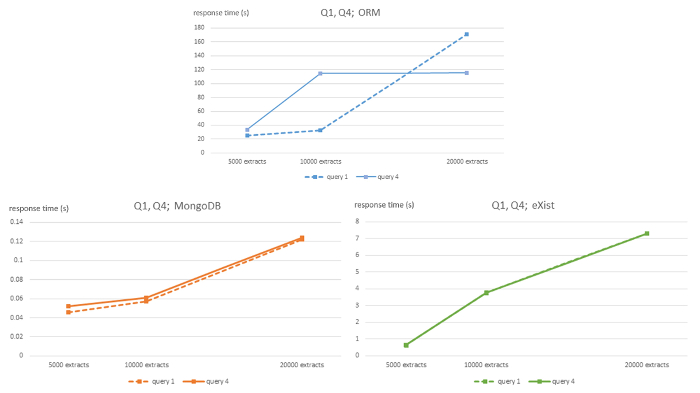
图 1: ORM MySQL、MongoDB 的算法复杂性和存在查询 Q1 和 Q4 的 DBMS。此数字已从7使用创意共享许可证 (http://creativecommons.org/许可证/4.0/) 进行了修改, 并显示了5000、1万和2万大小的电子数据库的响应时间 (以秒为单位) 为每个 DBMS 和查询 Q1 和 Q4。请单击此处查看此图的较大版本.
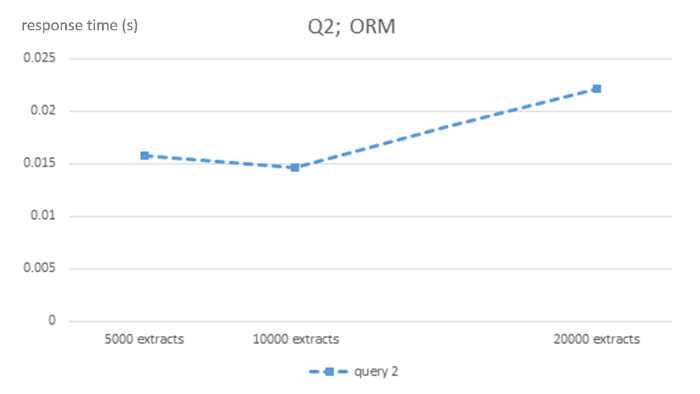
图 2:用于查询 Q2 的 ORM MySQL DBMS 的算法复杂性。此图显示5000、1万和2万大小的电子病历的响应时间 (以秒为单位) 提取 ORM MySQL 数据库以查询 Q2。请单击此处查看此图的较大版本.
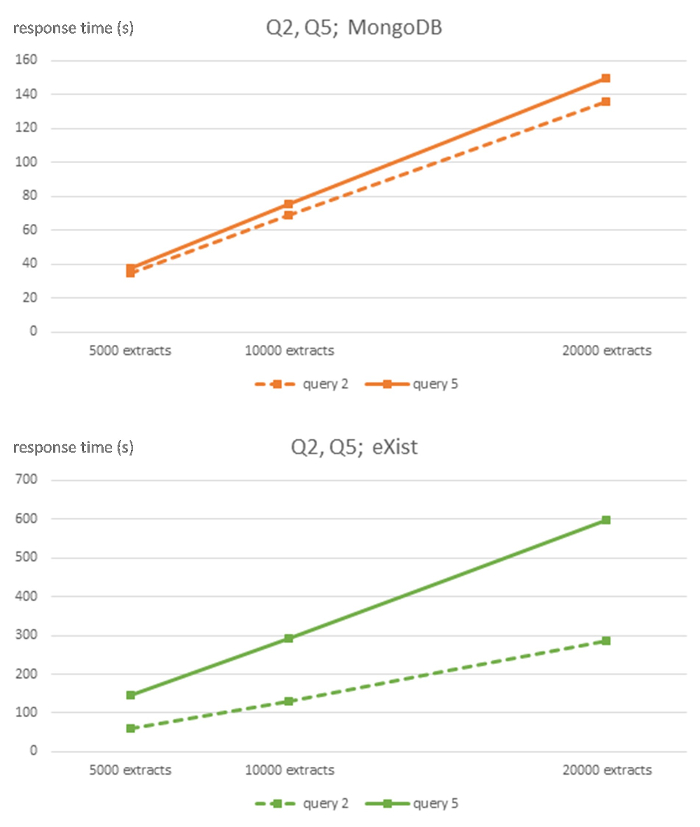
图 3:MongoDB 的算法复杂性和存在查询 Q2 和 Q5 的 DBMS。此数字已从7使用创造性共享许可 (http://creativecommons.org/licenses/4.0) 进行了修改, 并显示了5000、1万和2万大小的 EHR 的响应时间 (以秒为单位), 这些数据库用于每个 DBMS 和查询 Q2 和 Q5。请单击此处查看此图的较大版本.
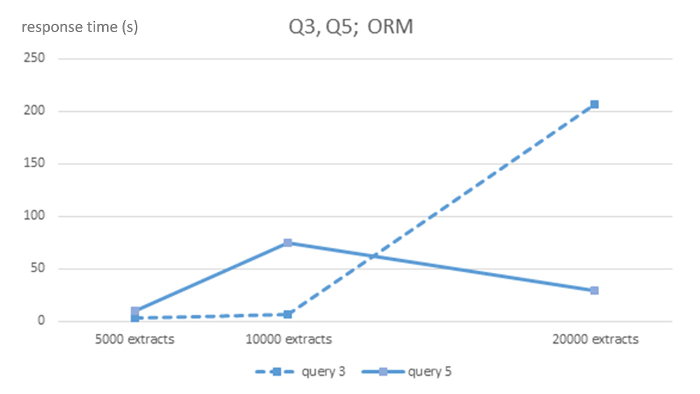
图 4: 用于查询 Q3 和 Q5 的 ORM MySQL DBMS 的算法复杂性。以秒为单位显示5000、1万和2万大小的 Q3 为每个 DBMS 提取数据库和查询和 Q5 的响应时间。请单击此处查看此图的较大版本.
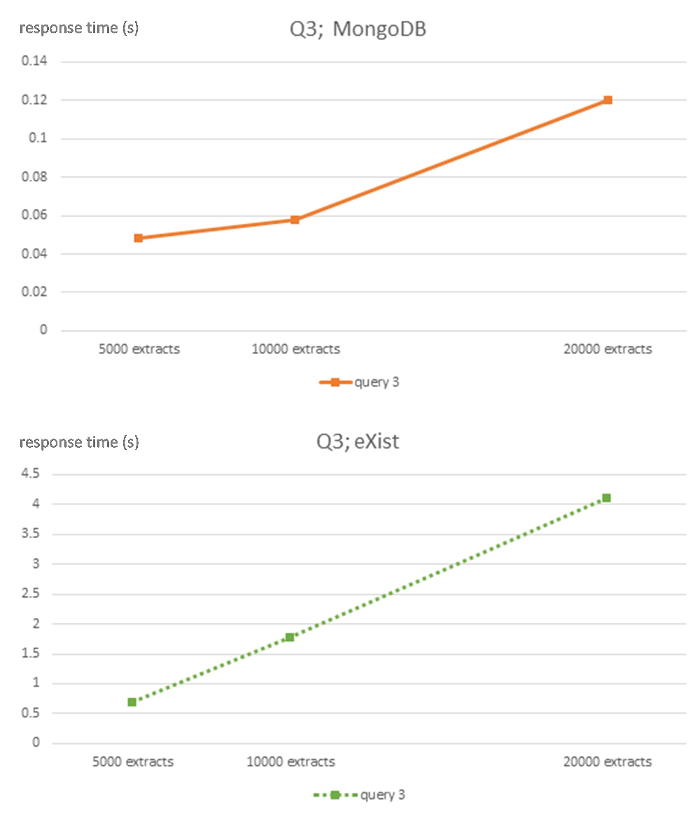
图 5:查询 Q3 的存在和 MongoDB DBMS 的算法复杂性。此数字已从7使用创造性共享许可证 (http://creativecommons.org/licenses//4.0/) 进行了修改, 并以秒为单位显示5000、1万和2万大小的 EHR 为每个 DBMS 和查询 Q3 提取数据库的响应时间。请单击此处查看此图的较大版本.
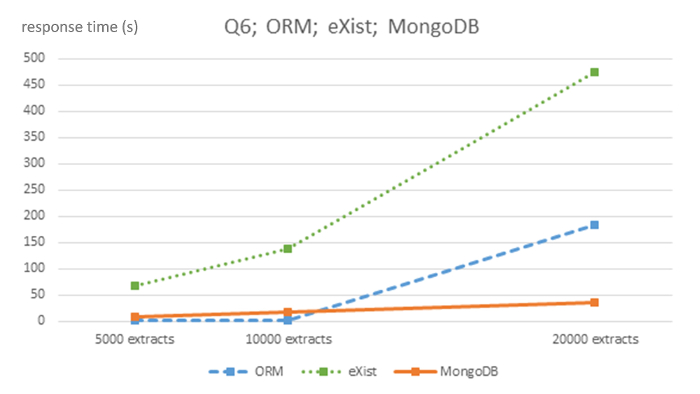
图 6:ORM MySQL 的算法复杂性, 存在和查询 Q6 的 MongoDB DBMS。此数字已从7使用创造性共享许可证 (http://creativecommons.org/licenses//4.0/) 进行了修改, 并以秒为单位显示5000、1万和2万大小的 EHR 为每个 DBMS 和查询 Q6 提取数据库的响应时间。请单击此处查看此图的较大版本.
| 查询 | |
| Q1 | 查找单个病人的所有问题 |
| Q2 | 查找所有病人的所有问题 |
| Q3 | 查找初始日期、解决日期和严重性 |
| 单个病人的单一问题 | |
| Q4 | 查找初始日期、解决日期和严重性 |
| 单个病人的所有问题 | |
| Q5 | 查找初始日期、解决日期和严重性 |
| 所有病人的问题 | |
| Q6 | 找到所有有问题的病人 ' 咽炎 ', |
| 初始日期 > = ' 16/10/2007 ', 决议日期 | |
| < = ' 06/05/2008 ' 和严重性 ' 高 ' |
表 1: 在关系和 NoSQL 数据库上执行的六个查询, 其中包含有关病人问题的标准的电子病历提取.此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/按/4.0/) 进行了修改, 并显示了在三个大小增长的数据库上执行的六个复杂增长的查询, 每个 DBMS 在自然语言.
| ORM/MySQL | 5000文档 | 1万文档 | 2万文档 |
| Q1 (s) | 25.0474 | 32.6868 | 170.7342 |
| Q2 (s) | 0.0158 | 0.0147 | 0.0222 |
| Q3 (s) | 3.3849 | 6.4225 | 207.2348 |
| Q4 (s) | 33.5457 | 114.6607 | 115.4169 |
| Q5 (s) | 9.6393 | 74.3767 | 29.0993 |
| Q6 (s) | 1.4382 | 2.4844 | 183.4979 |
| 数据库大小 | 4.6GB | 9.4GB | 19.4GB |
| 总提取物 | 5000 | 1万 | 2万 |
表 2: 在 ORM MySQL 关系 DBMS 的双倍大小数据库上六查询的平均响应时间 (以秒为单位).此表使用 ORM MySQL 关系 DBMS 和三数据库的内存大小, 为三个加倍大小的数据库的每个查询显示六响应时间.
| mongodb | 5000文档 | 1万文档 | 2万文档 | 斜坡 (* 10 exp (-6)) |
| Q1 (s) | 0.046 | 0.057 | 0.1221 | 5.07 |
| Q2 (s) | 34.5181 | 68.6945 | 136.2329 | 6780.99 |
| Q3 (s) | 0.048 | 0.058 | 0.1201 | 4.81 |
| Q4 (s) | 0.052 | 0.061 | o. 1241 | 4.81 |
| Q5 (s) | 38.0202 | 75.4376 | 149.933 | 7460.85 |
| Q6 (s) | 9.5153 | 18.5566 | 36.7805 | 1817.68 |
| 数据库大小 | 1.95GB | 3.95GB | 7.95GB | |
| 总提取物 | 5000 | 1万 | 2万 |
表 3: 对 MongoDB NoSQL DBMS 的双倍大小数据库的六查询中的平均响应时间 (以秒为单位).此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/4.0/) 进行了修改, 并显示了使用 NoSQL MongoDB DBMS 和内存大小的三个双倍大小数据库的每个查询的六响应时间。三数据库中。还显示了每个查询的线性斜率。
| 存在 | 5000文档 | 1万文档 | 2万文档 | 斜坡 (* 10 exp (-6)) |
| Q1 (s) | 0.6608 | 3.7834 | 7.3022 | 442.76 |
| Q2 (s) | 60.7761 | 129.3645 | 287.362 | 15105.73 |
| Q3 (s) | 0.6976 | 1.771 | 4.1172 | 227.96 |
| Q4 (s) | 0.6445 | 3.7604 | 7.3216 | 445.17 |
| Q5 (s) | 145.3373 | 291.2502 | 597.7216 | 30158.93 |
| Q6 (s) | 68.3798 | 138.9987 | 475.2663 | 27125.82 |
| 数据库大小 | 1.25GB | 2.54GB | 5.12GB | |
| 总提取物 | 5000 | 1万 | 2万 |
表 4: 在存在的 NoSQL DBMS 的双倍大小数据库上六查询的平均响应时间 (以秒为单位).此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/4.0/) 进行了修改, 并显示了使用 NoSQL 存在的 DBMS 和内存大小的三个双倍大小数据库的每个查询的六响应时间。三数据库。还显示了每个查询的线性斜率。
| 扶手纸 | 臂 (s) | 节点 + 路径 | |
| Q1 | 查询2。1 | 0.191 | 24.866 |
| Q3 | 查询3。1 | 0.27 | 294.774 |
| 数据库大小 | 2.90GB | 43.87GB | |
| 总提取物 | 29743 | 29743 |
表 5: 以秒为单位的查询的平均响应时间 (类似于所介绍的改进的关系系统的 Q1 和 Q3)10. 此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/按/4.0/) 进行了修改, 并显示在10中与两个改进的关系数据库系统对应的两个最相似的查询 Q1 和 Q3和他们的反应时间。同时还显示了两个数据库大小。
| ORM/MySQL | 吞吐量 | 响应时间 |
| Q1 (s) | 4711.60 | 0.0793 |
| Q3 (s) | 4711.60 | 0.1558 |
| Q4 (s) | 4711.60 | 0.9674 |
表 6: 在并发执行中, ORM MySQL 关系 DBMS Q1、Q3 和 Q4 查询的平均吞吐量和响应时间 (以秒为单位).此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/按/4.0/) 进行了修改, 显示了三个单病人查询的最高平均吞吐量及其在并发中的平均响应时间使用 ORM MySQL 关系系统执行实验。
| mongodb | 吞吐量 | 响应时间 |
| Q1 (s) | 178672.60 | 0.003 |
| Q3 (s) | 178672.60 | 0.0026 |
| Q4 (s) | 178672.60 | 0.0034 |
表 7: 并行执行中 MongoDB NoSQL DBMS 的查询 Q1、Q3 和 Q4 的平均吞吐量和响应时间 (以秒为单位).此表已从7使用创造性共享许可证 (http://creativecommons.org/许可证/按/4.0/) 进行了修改, 显示了三个单病人查询的最高平均吞吐量及其在并发中的平均响应时间使用 MongoDB NoSQL 系统执行实验。
辅助图 1: 屏幕截图显示了要连接到 MySQL 服务器的软件屏幕.请单击此处下载此图.
补充图 2: 屏幕截图显示了 sql 接口到 MySQL 服务器, 其中第一个 SQL 查询已写入.请单击此处下载此图.
补充图 3: 通过执行服务器 mongod, 使用 DOS 系统窗口启动 MongoDB 2.6 本地主机服务器.请单击此处下载此图.
辅助图 4: 屏幕截图显示了查询生成器文本框中写入的查询, 如步骤5.7.1 通过5.7.4 所示。屏幕截图说明步骤5.7.3。请单击此处下载此图.
辅助图 5: 屏幕截图显示步骤 5.7.6.请单击此处下载此图.
辅助图 6: 屏幕快照说明了在对话框的 theupper 部分中 XPath 查询的写入.请单击此处下载此图.
讨论
该协议表明, 纯关系 ORM 系统对于单病人查询 (Q1、Q3 和 Q4) 似乎并不实用, 因为响应时间较慢, 可能是由于大量的关系表执行了许多昂贵的连接操作, 而且由于特定类型的数据库所使用的存储系统。NoSQL 数据库以基于文档的方式存储数据, 而关系系统使用基于表的方式将每个文档传播到整个数据库中。NoSQL 系统显示一个线性斜率, MongoDB 的性能比存在的 DBMS 快得多。在并发中, MongoDB 的行为也比关系 MySQL ORM7要好得多。
此协议为7中提供的有关 ORM MySQL DBMS 的结果提供了一个疑难解答协议。MySQL 系统已经更新到最新版本, 结果也略有修改。此外, 基于文档的 NoSQL 系统 (如 MongoDB) 中的一个关键点是, 在存储医疗信息7时, 它们可能保持一致性, 因为当一个电子病历提取被更新时, 它不会被覆盖, 但新数据的整个新提取物是生成并存储在系统中, 并保留原始提取物。这是对医疗信息的严格要求, 因为一些医生可能根据原始数据做出了重要的医疗决策。
改进的关系 ARM 系统大大减少了关系表的数量, 提高了关系性能。但是, 由于它修改了关系模式, 提取的医学信息可能会被查询, 但提取物不能以其确切的原始形式恢复。
对于二次使用 (研究) 中的大型数据库, 目前尚不清楚哪个数据库系统更合适, 因为在 ORM 中, 所有病人的查询 (Q2 和 Q5) 的行为比在 NoSQL 系统中表现得更好, 但是这些系统的性能比简化的关系12中的系统。我们认为 Q6 在临床实践和二次使用之间的特殊查询, 其行为不能由这些实验产生的结果决定。
然而, 该方法的一个局限性是直接实验的出车前发生, 将改进的关系臂系统与 NoSQL MongoDB 对单病人、医学实践查询的比较, 与协议中使用的数据完全相同。我们在对单个病人查询进行了插值表 3和表 5的情况下, 直到执行了该协议中包括优化 ARM 的实验为止。我们将这些实验留给未来的应用。协议中的一个关键步骤是从最近几年中选择免费数据库, 类似的软件版本, 这样我们就可以比较三技术的精确状态。
这是第一次尝试使用实际的、现实的、标准化的医疗信息直接比较关系和 NoSQL 系统。但是, 要使用的特定系统在很大程度上取决于要解决的实际方案和问题8。
披露声明
作者没有什么可透露的。这些实验中使用的数据集是由几家西班牙医院根据这些实验的许可证提供的, 因此没有公开提供。ISO/EN 13606 RM XML 模式由伦敦大学大学健康信息学 & Multiprofessional 教育中心提供。
致谢
作者要感谢迪帕克卡尔拉博士, 该工作队的负责人, 它定义了 iso/en 13606 标准和他的团队从伦敦大学学院, 他们的亲切的许可使用 ISO/en 13606 W3C XML 模式。
这项工作得到了干杯 (PI15/00321、PI15/00003、PI1500831、PI15CIII/00010 和 RD16CIII) 的资助。
材料
| Name | Company | Catalog Number | Comments |
| MySQL 5.7.20 | MySQL experiments | ||
| Red Hat Enterprise Linux Server release 7.4 (Maipo), 2.60GHz, RAM 16GB | |||
| MongoDB 2.6 | MongoDB experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| eXist 3.0RC1 | eXist experiments | ||
| Windows 7, 2.66GHz, RAM 12GB | |||
| Studio 3T 5.5.1 | 3T Software Labs Gmbh | MongoDB GUI |
参考文献
- Codd, E. F. A relational model for large shared data banks. Comm ACM. 13 (6), 377-387 (1970).
- Kalra, D., Lloyd, D. . ISO 13606 electronic health record communication part 1: reference model. , (2008).
- Kalra, D., et al. . Electronic health record communication part 2: archetype interchange specification. , (2008).
- Kalra, D., Beale, T., Heard, S. The openEHR foundation. Stud. Health Technol. Inform. 115, 153-173 (2005).
- Beale, T. Archetypes: constraint-based domain models for future proof information systems. OOPSLA, Workshop Behav Semant. , (2002).
- Sánchez-de-Madariaga, R., et al. Examining database persistence of ISO/EN 13606 standardized electronic health record extracts: relational vs. NoSQL approaches. BMC Medical Informatics and Decision Making. 32 (2), 493-503 (2017).
- Ireland, C., Bowers, D., Newton, M., Waugh, K. Understanding object-relational mapping: a framework based approach. Int. J. Adv. Softw. 2, 202-216 (2009).
- . Node+Path Persistence Available from: https://openehr.atlassian.net/wiki/spaces/dev/pages/6553626/Node+Path+Persistence (2017)
- Wang, L., Min, L., Wang, R., et al. Archetype relational mapping - a practical openEHR persistence solution. BMC Medical Informatics and Decision Making. 15, 88 (2015).
- Kaur, K., Rani, R. Managing data in healthcare information systems: many models, one solution. Computer. March. , 52-59 (2015).
- Sabo, C., Pop, P. C., Valean, H., Danciulescu, D. An innovative approach to manage heterogeneous information using relational database systems. Advances in Intelligent Systems and Computing. 557, (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。
我们使用 cookie 来增强您在我们网站上的体验。
继续使用我们的网站或单击“继续”,即表示您同意接受我们的 cookie。
