È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Facile protocollo per la sintesi di auto-assemblaggio basato su poliammina Peptide anfifili (PPA) e biomateriali correlate
In questo articolo
Riepilogo
La sintesi delle poliammine-basato del peptide anfifili (PPA) è una sfida significativa per la presenza di multiple azoti ammina, che richiede l'uso giudizioso di proteggere i gruppi per mascherare queste funzionalità reattive. In questo articolo, descriviamo un metodo facile per la preparazione di questi nuova classe di molecole autoassemblanti.
Abstract
Basato su poliammina Peptide anfifili (PPA) sono una nuova classe di autoassemblanti anfifilici biomateriali-relazionati con il peptide anfifili (PAs). Tradizionale PAs possiedono caricate aminoacidi come solubilizzanti gruppi (lisina, arginina), che sono direttamente collegati ad un segmento del lipido o possono contenere una regione linker fatta di amminoacidi neutri. Messa a punto la sequenza del peptide di PAs può produrre diverse morfologie. Allo stesso modo, PPA possieda un segmento idrofobo e aminoacidi neutri, ma contenga anche molecole di poliammine come acqua solubilizzanti (idrofili) gruppi. Come è il caso con PAs, PPA anche possibile auto-assemblarsi in morfologie diverse, tra cui asticelle, nano-nastri intrecciati e nano-fogli di fuse, quando disciolto in acqua. Tuttavia, la presenza di ammine primarie e secondarie su una molecola singola poliammina pone una sfida significativa quando sintetizzando PPA. In questa carta, indichiamo un protocollo semplice, basato su precedenti di letteratura, di raggiungere una sintesi facile di PPA utilizzando sintesi del peptide di fase solida (SPPS). Questo protocollo può essere esteso alla sintesi di PAs e altri sistemi simili. Abbiamo anche illustrare i passaggi che sono necessari per la scissione da resina, identificazione e purificazione.
Introduzione
Autoassemblanti peptide anfifili (PAs) sono una classe di biomateriali in genere composto da segmenti seguenti: (a) idrofilo testa, regione linker (b) e (c) idrofobo. La maggior parte PAs descritti nella letteratura possiedono una testa idrofila composta da cariche o polare dell'amminoacido residui1,2,3,4. PAs hanno trovato una vasta gamma di applicazioni nel campo della biomedicina tra cui la consegna della droga, malattia diagnostica, medicina rigenerativa, ecc5. Base alla loro sequenza dell'amminoacido, PAs può formare un'ampia varietà di nanostrutture tra cui micelle sferiche e nano-filamenti. Recentemente abbiamo segnalato una classe di ibridi basati su poliammina peptide anfifili, chiamato PPA6. Le morfologie, autoassemblanti cinetica e degradazione metabolica, di questi biomateriali, sono stati trovati per essere collegati al loro gruppo testa solubilizzante. Inoltre, le nanostrutture di PPA non hanno mostrato tossicità verso le cellule di mammiferi (linee MiaPaCa2 e delle cellule HeLa) alle concentrazioni testate. Nanovettori basati su PPA sono veicoli di consegna della droga attraente perché: (1) della poliammina assorbimento ed il metabolismo è stato indicato per essere aumentato in cellule cancerogene, nanostrutture (2) cationico può raggiungere endosomal fuga7,8, che conduce alla più alta circolazione e soggiorno all'interno di una cella e (3) dovrebbero avere un distinto profilo metabolico se confrontato con il PA; ad esempio, essi saranno più stabile verso proteasi trovate nel corpo umano (anche se essi forse sensibili ad altri enzimi, quali ammino ossidasi)9,10. Inoltre, PPA è stati trovati per avere diverse morfologie, proprietà fisico-chimiche, rigidità delle nanoparticelle e cinetica di assemblaggio a seconda della lunghezza e carica di singoli PPA molecola6. Qui, descriviamo un protocollo dettagliato per la sintesi, la identificazione e la purificazione di PPA che può essere applicato anche alla preparazione di PAs o simili molecole del peptide ibride.
Perché poliamine non sono comunemente disponibili in commercio nelle loro forme protette, e perché proteggere le ammine primarie e secondarie delle poliamine è della massima importanza per loro coniugazione con amminoacidi e altre molecole, che abbiamo descritto il procedura sintetica per raggiungere la loro protezione. L'obiettivo generale del presente protocollo è quello di fornire un metodo semplice per coniugazione poliamine agli aminoacidi. Poliammine mancano un gruppo carbossilico; così, essi non possono essere accoppiate a Rink ammide o resine Wang. Invece, resine come cloruro di 2-chlorotrityl sono raccomandate per il protocollo sintetico. La sfida principale per la sintesi di PPA è la presenza di gruppi funzionali di ammina primaria e secondaria. Per i nostri scopi, abbiamo protetto tutte le ammine secondarie nella poliammina, mantenendo il gruppo amminico primario sulla poliammina libera per consentire la reazione di accoppiamento. La reazione è stato fatto su un supporto solido, seguendo i principi della sintesi del peptide di fase solida (SPPS) per facilitare il work-up dopo ogni passaggio di accoppiamento e deprotezione. Il seguente protocollo è per entrambi la sintesi manuale e automatizzata di PPA (anche se sarà difficile la verifica di alcuni passi in un sistema automatizzato). La sintesi di queste molecole possono anche essere effettuata su un sintetizzatore automatico o con l'ausilio di un reattore a microonde (automatico o semi-automatica). Lo schema di reazione è stata riassunta nella Figura 1.
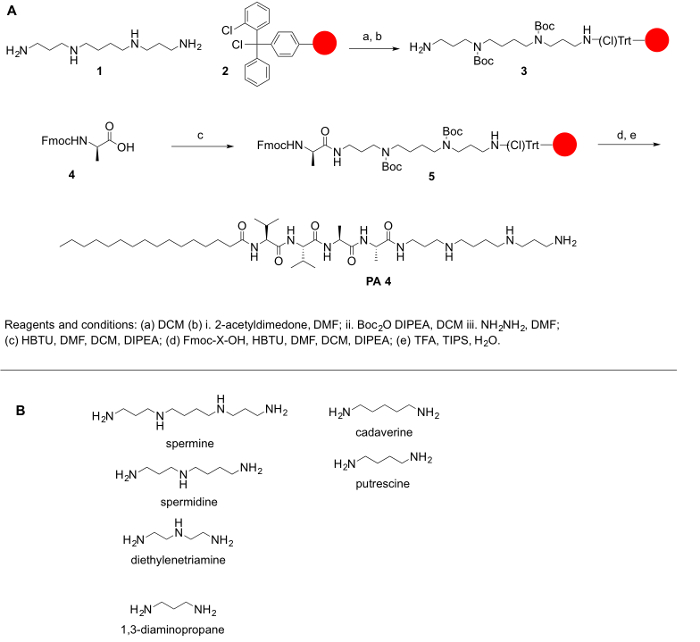
Figura 1: (A) A regime di reazione generale per la sintesi di PPA. Poliammine rappresentativo (B) che possono essere utilizzati per sintetizzato PPA descritto qui. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. generale protocollo per la sintesi di PPA
- Calcolare la scala di sintesi (solitamente mmol). Questa scala è basata sulla massa dell'importo-obiettivo PPA. Tenete a mente che l'efficienza di reazione di SPPS diminuisce gradualmente con un aumento di sequenza dell'amminoacido. Di conseguenza, l'efficienza di reazione esatta è difficile da calcolare.
- Calcolare il peso della resina da impiegare secondo il caricamento della resina. Il caricamento è trovato sul contenitore o il protocollo di analisi della resina ed espressa in mmol/g. La seguente formula può essere utilizzata per calcolare il peso della resina:
- Pesare con attenzione resina di cloruro di 2-chlorotrityl (nel nostro caso il caricamento è ~0.85 mmol)
- Versare la resina in un recipiente di sintesi sinterizzato con le seguenti specifiche: capacità – 50 mL, Fritted disco – 25 mm, porosità – media.
- Aggiungere 15 mL di diclorometano (DCM) alla resina.
- Appone alla nave di sintesi di un agitatore meccanico a velocità variabile (pallone shaker/agitatore) e girare i vasi per un angolo di 45° (o orizzontalmente) per massimizzare l'agitazione.
- Consentire le perle di resina a gonfiarsi per 15 min. gonfiore migliora il rendimento di reazione perché facilita la diffusione molecolare e l'accessibilità al sito attivo, aumentando l'efficienza di accoppiamento.
- Aggiungere 8 equivalenti (2 mmol per scala 0,25 mmol) della poliammina desiderata (spermina, spermidina, Diethyelenetriamine, 1,3-diaminopropane, ecc.) per la resina e lasciare reagire per 5 h.
Nota: Un minore periodo di reazione ridurrebbe la resa. - Fare un Kaiser prova (Vedi Tabella materiali) per confermare il successo accoppiamento del polyamine alla resina. Accoppiamento riuscito delle ammine primarie produce un colore viola/blu, che corrisponde all'ammina libera.
- Proteggere il gruppo di ammina primaria utilizzando 4 equivalenti di 1-(4,4-dimethyl-2,6-dioxocyclohex-1-ylidene)ethyl (Dde), disciolti in metanolo anidro (15 mL), agitando la miscela di reazione durante la notte11.
Nota: Un tempo di reazione minore riduce il rendimento. - Eseguire un test di Kaiser. Un'assenza di colore blu da resina perlina conferma protezione riuscita. In caso di esito negativo protezione dell'ammina primaria, ripetere il passaggio precedente.
- Scolare il DCM e lavare le resine due volte con una miscela di DCM e DMF (2:1, 15 mL).
- Sciogliere 20 equivalenti (5 mmol) di-tert butyl di-carbonato (Boc) in DCM (20 mL) e lasciare che la reazione procedere per 3 h.
- Fare un cloranile test (Vedi Tabella materiali) per confermare la completa protezione dell'ammina secondaria. Un test positivo (protezione) produce un colore giallo chiaro o incolore. Ammine secondarie gratis dare un colore verde/blu scuro, mentre le ammine primarie danno un colore rosso.
- Scolare la miscela solvente e lavare due volte con una miscela di DCM e DMF (2:1, 15 mL).
- Aggiungere una soluzione al 2% di idrazina in DMF (10 mL) e agitare per 1 h.
- Fare un Kaiser test per confermare il successo deprotezione dell'ammina primaria.
-
Aggiungere gli aminoacidi desiderati nell'ordine inverso in cui si sarebbe scrivere/disegnare loro. Peptidi sono attratti dalla termini N termini C ma sono sintetizzate in direzione opposta, C a N. Ad esempio, per un PPA che richiedono il peptide seguente nucleo - GLFD-, aggiungere l'acido aspartico (D), seguita da fenilalanina (F), leucina (L) e infine la glicina (G).
Nota: Per la maggior parte degli aminoacidi, il cocktail di accoppiamento seguente è adatto per allegato alle resine della poliammina caricata.- Mescolare 4 equivalenti (1 mmol) dell'amminoacido Fmoc-protetto, 3,95 equivalenti di 2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate (HBTU) e 15 equivalenti di ammina diisopropylethyl (DIPEA).
- Scioglierli in una miscela di DCM e DMF (1:1, 15 mL) e Sonicare il cocktail per 2 min (fino al completo scioglimento).
- Aspettare altri 3-5 min per garantire l'attivazione dell'acido carbossilico dell'aminoacido.
- Aggiungere la miscela di reazione alla nave. Svolgere la reazione per 2-4 h a temperatura ambiente.
Nota: Abbiamo trovato le seguenti alternative efficienti per HBTU (che solitamente richiedono minori quantità di aminoacidi e l'agente di accoppiamento): COMU, PyBOP, HATU, DIC/Oxima. - Fare un Kaiser test per confermare il successo accoppiamento.
- De-proteggere il Fmoc-gruppo dall'amminoacido aggiungendo una soluzione di 20% di 4-metil piperidine (10 mL) in DMF. Farlo due volte, ogni volta che agitando la miscela di reazione per 15 min.
Nota: Un'alternativa efficiente per la rimozione di Fmoc è piperazina/DBU12.- Eseguire un lavaggio di DCM (15 mL) tra aggiunte.
- Fare un Kaiser test per confermare il successo de-protezione dell'amminoacido.
- Lavare la resina accuratamente per rimuovere tutte le tracce di 4-metil. Lavare la resina due volte con DMF (10ml), ogni lavaggio dura per 5 min e infine con DCM (10 mL) per 10 min.
- Aggiungere tutti gli amminoacidi restanti successivamente ripetere l'accoppiamento e la de-protezione passaggi.
-
Infine, dopo l'accoppiamento di tutti gli aminoacidi necessari, coniugato la coda idrofobica per l'ultimo amminoacido del costrutto della poliammina-peptide:
- Aggiungere 10 equivalenti della funzionalità desiderata degli acidi carbossilici a 9,5 equivalenti di HBTU e 12 equivalenti di DIPEA disciolto in DCM e DMF miscela (1:1, 20 mL).
Nota: Tenere presente che alcune code potrebbero essere necessario una diversa proporzione di solvente (solitamente un maggiore % di DCM) o l'aggiunta o un altro solvente come N-metilpirrolidone (NMP). - Sonicare il cocktail per 5 – 10 min fino alla dissoluzione (abbiamo visto richiede più tempo per la coda sciogliere completamente) e aggiungere alla nave.
Nota: Per le code contenenti più siti reattivi, dovranno essere protetti prima di accoppiare li. - Eseguire questa reazione (accoppiamento coda idrofobica) per almeno 5 ore, sebbene si consiglia di portarlo fuori tutta la notte per il massimo rendimento.
- Aggiungere 10 equivalenti della funzionalità desiderata degli acidi carbossilici a 9,5 equivalenti di HBTU e 12 equivalenti di DIPEA disciolto in DCM e DMF miscela (1:1, 20 mL).
2. PPA scissione dal supporto solido
Lo scopo di questo passaggio è il clivaggio del PvP dalla resina e per rimuovere i gruppi di protezione di Boc dalla aminoacidi ed i residui delle poliammine.
- Lavare la resina con DMF (8 mL per 2 min) e due volte con DCM (8 mL, ogni volta per 5 min). Prima di ogni aggiunta, è necessario svuotare il solvente dalla nave. Una volta che viene eseguita l'ultimo lavaggio, asciugare la resina sotto vuoto per 15 min.
- Preparare la soluzione di clivaggio utilizzando il seguente rapporto di miscela: 28:1:1 acido trifluoroacetico (TFA): H2O: Triisopropyl Silano (suggerimenti). Per prenotare 15 mL della fenditura cocktail 14 mL di TFA, aggiungere 0,5 mL di H2O e 0,5 mL di suggerimenti.
- Aggiungere questa soluzione di fenditura alla resina e agitare per 2 – 4 h a temperatura ambiente.
- Raccogliere la soluzione in 25 o 50 mL pallone da....
- Concentrare il TFA nel vuoto a 1 – 2 mL utilizzando un evaporatore rotante a pressione ridotta mentre riscaldando la miscela a 40 ° C (non superare i 55 ° C per evitare la decomposizione del PvP).
- Dopo evaporazione, aggiungere la soluzione ottenuta TFA (goccia a goccia) in un matraccio da fondo tondo contenente etere anidro freddo (15 mL). Questo verrà immediatamente precipitare il PPA.
- Inoltre, è possibile aggiungere anidro freddo etere (5 mL) al pallone originale dove si concentrava il TFA.
- Sonicare questa boccetta per recuperare ulteriori solidi e si combinano con la soluzione di etere dal punto 2.6.
Nota: La pipetta di trasferimento può essere lavata con un piccolo volume di DCM. Evitare di introdurre DMF durante il processo, perché tende a formare una sostanza gelatinosa. - Posizionare il pallone (coperto) all'interno del frigorifero e lasciate riposare una notte per massimizzare la precipitazione.
- Raccogliere il materiale precipitato utilizzando un disco sinterizzato filtro imbuto di filtrazione sotto vuoto. Le dimensioni dei pori ideale sono fini o medio.
- Lavare il precipitato due volte con etere freddo (5 – 10 mL) per rimuovere eventuali sostanze organiche residue.
3. identificazione del prodotto grezzo utilizzando il metodo di goccia secchi MALDI
-
Preparazione della matrice per l'analisi in modalità positiva:
- Per iniziare a peso molecolare analisi mediante spettrometria di massa Matrix Assisted Laser Desorption/Ionization (MALDI), aggiungere 10-20 mg di acido α-ciano-4-idrossicinnamico ad un tubo del microcentrifuge.
- Aggiungere una soluzione di acqua/acetonitrile (1:1, 1,0 mL) con 0,1% acido trifluoroacetico (TFA). Mescolare nel Vortex.
Nota: Questa matrice deve essere utilizzata per peptidi caricati positivamente. Modalità negativa MALDI è simile ma richiede una matrice contenente 9-aminoacridine con 0,1% ammoniaca come solvente additivo. - Aggiungere 1-2 µ l del campione per la piastra in acciaio inox per MALDI e asciugare il campione in aria. Aggiungere 1-2 µ l della matrice e asciugare di nuovo.
Nota: Le piastre hanno marcature circolari griglia lungo la piastra di destinazione per individuare il punto particolare.
- Analizzare i campioni in strumento MALDI per verificare la loro identità. Ionizzazione electrospray (ESI) è una valida alternativa per confermare l'identità della PPA.
4. purificazione di PPA tramite purificazione preparativa inverso-fase ad alte prestazioni cromatografia liquida (HPLC)
-
Sciogliere il precipitato di PPA in una quantità minima di acqua e acetonitrile.
- Come regola generale, sciogliere 100 mg del greggio secco PPA in meno di 5 mL di acetonitrile e acqua. Più idrofobo PPA potrebbe richiedere una maggiore percentuale di acetonitrile per sciogliere e potrebbe inoltre richiedere un più grande volume di solvente in generale, a seconda della carica netta di PPA (tabella 1).
- Se la dissoluzione completa non ha luogo, aggiungere 1% TFA (ACN o H2O). È anche possibile aggiungere tracce di altri solventi compatibili tra cui dimetilsulfossido, metanolo o isopropanolo (limitare il loro contenuto al 5%).
| Solvente | Carica positiva PPAs | Carica negativa PPAs | ||
| 0,1 TFA % in acqua | 0,1% NH3 in acqua | |||
| 0,1% TFA in ACN | 0,1% NH3 in ACN | |||
Tabella 1: sistemi solvente. Proposto sistema solvente per PPA a carico positivamente e negativamente.
- Sonicare il PPA utilizzando un sonicatore corno per 20 min a 10 impulsi s con Amp1 del 48% o per 2-3 h in bagno ad ultrasuoni.
-
Quindi, di filtrare usando un filtro di 0,45 μm siringa seguito da filtrazione utilizzando un filtro di siringa di politetrafluoroetilene (PTFE) 0.20 μm. La soluzione di PPA deve essere limpida e priva di tutti i materiali in polvere.
- Se la filtrazione è troppo difficile, Sonicare per un periodo prolungato di tempo. Si consiglia vivamente che la soluzione di PPA è purificata immediatamente dopo filtrazione siringa, come stoccaggio prolungato potrebbe causare per aggregare o gelate, che richiederà un ri-sonicazione e, forse, ri-filtrazione.
- Durante e dopo la purificazione, utilizzare solventi HPLC grade o quelli che vengono filtrati attraverso un 0,25 μm siringa filtro/membrana.
Nota: Le condizioni più comuni solventi per purificare PPA consistono in una sfumatura di H2O e ACN. - Utilizzare uno strumento HPLC di inverso-fase standard per questo protocollo. Esso dovrebbe includere un programmatore di gradiente di eluizione, un sistema binario di consegna solvente, un rivelatore UV in grado di rilevare a 220 e 254 nm e un collezionista di frazione programmabile. La portata massima deve essere di 50 mL/min.
- Uso un C-18 invertito colonna in fase di separazione. Colonne delle dimensioni seguenti possono essere utilizzate in funzione della massa del solido viene purificato e rete carica del PvP (tabella 2).
| Carica di PPA | Dimensione delle particelle | Dimensione della colonna | Massa di greggio PPA |
| + ve cariche | 5 μm. | 150 x 30 mm | 170 mg |
| -ve cariche | 5 μm. | 150 x 30 mm | 170 mg |
| + ve cariche | 5 μm. | 150 x 21.2 mm | 90 mg |
| -ve cariche | 5 μm. | 150 x 21.2 mm | 90 mg |
Tabella 2: suggerito colonne: Dimensioni della colonna, dimensioni delle particelle e la capacità di carico massimo per iniezione per C18 reverse colonne HPLC fase
- Iniettare PPA con un volume determinato di sia la capacità della colonna e dal volume del ciclo iniezione di HPLC. Eseguire il metodo del gradiente di HPLC secondo le impostazioni visto in tabella 3 (tempo di eluizione di 40 min).
| Tempo | Solvente A (Acetonitrile) | Solvente B (acqua) | Portata (mL/min) |
| 0 | 5% | 95% | Portata dipende l'imballaggio di colonna e le sue dimensioni. |
| 2 | 5% | 95% | |
| 35 | 95% | 5% | |
| 38 | 100% | 0% | |
| 40 | 5% | 95% |
Tabella 3: gradiente suggerito: Gradiente di fase inversa suggerito la relativa composizione di acqua vs acetonitrile in un periodo di tempo. La portata dipende le specifiche di colonna.
-
Analizzare le frazioni raccolte sulle varie provette usando MALDI e determinare quale tubo (o tubi) contengono il PPA. Questo requisito viene valutato studiando il peso molecolare trovato in ogni provetta.
- Verificare la purezza delle frazioni su un HPLC analitica. Utilizzare un sistema di HPLC-gradiente dotato di un rivelatore UV impostato a 220 nm.
- Se sono presenti impurità, fare un ulteriore ciclo di purificazione mediante HPLC. Sfumatura sopra utilizzata per HPLC preparatorio può essere scalata verso il basso da utilizzare con l'HPLC analitica utilizzando il software online convertitore di colonna. Ogni frazione molto essere ≥ 95% puro per la caratterizzazione fisica o valutazione biologica.
- Raccogliere le frazioni in un pallone a fondo rotondo grande o il pallone di evaporazione e rimuovere tutti l'acetonitrile e la maggior parte dell'acqua. La soluzione finale di PPA dovrebbe essere non più di 10 mL.
- Trasferire questo concentrato di un tubo di centrifuga da 50 mL. Sappiate che PPA è sostanze detergenti simili; così, bolle verranno generate durante l'evaporazione del solvente. Abbiamo trovato che l'aggiunta di alcole etilico diminuisce la formazione di bolle.
- Concentrato di vuoto il concentrato. Durante l'evaporazione sotto vuoto, una grande quantità di PPA potrebbe aderire alle pareti del contenitore. Recuperare questo risciacquando ripetutamente il muro pallone con acqua di HPLC. Il volume totale di PPA concentrato e il rinsate dovrebbe essere non più di 30 mL.
- Posto la soluzione acquosa all'interno di un tubo da 50 mL.
-
Flash congelare questa soluzione PPA in azoto liquido:
Nota: Flash congelamento risultati più piccoli cristalli di ghiaccio che sarà sublime più velocemente nei liofilizzatori. Inoltre, più lento congelamento potrebbe consentire la formazione di strutture supramolecolari auto-assemblate. Un'altra alternativa (ma meno auspicabile) è gradualmente congelare in un congelatore a-80 ° C o di congelare utilizzando un ghiaccio secco + miscela di acetone- Prima di mettere la provetta da centrifuga nei liofilizzatori, perforare o allentare il tappo della provetta per permettere all'umidità di sfuggire il campione e ottenere raccolti nell'unità di refrigerazione. Impostare l'unità di liofilizzazione impostata-54 ° C e 0,016 mbar.
Nota: Un'altra opzione è di rimuovere il tappo e collocare un tampone imbevuto (con un elastico) sopra l'apertura. Inoltre, abbiamo scoperto che permette di congelare campioni ad angolo o rompere il ghiaccio in piccole porzioni per una più veloce e migliore liofilizzazione a causa di un incremento della superficie. - Se il solido inizia a sciogliersi, ciò potrebbe indicare che sono presenti tracce di un solvente organico (solitamente acetonitrile). Ripetere il passaggio di congelamento e valutare le condizioni della liofilizzazione.
- Se la fusione persiste, ci sono due possibili soluzioni:
- Split il campione in due porzioni (per essere inserito in tubi), diluire con acqua e congelare di nuovo. Non utilizzare questa soluzione spesso perché grandi quantità di solventi organici potrebbe danneggiare l'attrezzatura.
- In alternativa, posizionare il campione in una beuta e ulteriormente far evaporare il solvente organico sotto vuoto. Liofilizzazione un campione che è in forma liquida di solito porta a urtare e perdita del composto PPA.
- Prima di mettere la provetta da centrifuga nei liofilizzatori, perforare o allentare il tappo della provetta per permettere all'umidità di sfuggire il campione e ottenere raccolti nell'unità di refrigerazione. Impostare l'unità di liofilizzazione impostata-54 ° C e 0,016 mbar.
5. deposito di PPA
- Conservare la PPA in un da-20 ° C con i tappi serrati e sigillato con Parafilm su un tubo con un'etichetta adeguata.
- Prima dell'uso, portare il PPA torna a temperatura ambiente in una camera a vuoto per evitare la condensazione del vapore acqueo sulla PPA.
Risultati
Dopo la sintesi e la purificazione e prima valutazione fisico-chimiche o biologiche, è consigliabile le masse della PPA sono ri-controllate e la purezza accertato mediante HPLC analitica. Per la caratterizzazione di materiali o valutazione biologica, PPA necessario disporre di una purezza di > 95%. La figura 2 Mostra l'analisi HPLC (in alto) e spettri MALDI (in basso) che confermano la presenza del prodotto. Sistemi di analisi HPLC integrerà l'area sotto la...
Discussione
I protocolli descritti qui possono essere utilizzati per sintetizzare PPA come pozzi come PAs e relative molecole peptidici (ad esempio di ibrido PA-peptoids). Anche se la sintesi di peptidi utilizzando SPPS è una procedura semplice, la sintesi di peptidi contenenti molecole biologiche homing può essere particolarmente impegnativa. Poliammine come spermina, spermidina, diethyelenetriamine, ecc., possono funzionare come molecole homing per il targeting cancro cellule13. Il PPA possa asse...
Divulgazioni
Gli autori non hanno alcun conflitto di interessi per dichiarare
Riconoscimenti
Questo progetto è stato finanziato dall'Università del Nebraska Medical Center (fondi di Start-up, MC-S); NIH-COBRE, 5P20GM103480 (T. jakzrobic) e l'American Chemical Society, PRF # 57434-DNI7(MC-S).
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Chlorotrityl chloride resin | AappTec | RTZ001 | |
| SynthwareTM synthesis vessel | Aldrich | SYNP120050M | |
| Dichloromethane | Acros | AC406920250 | Fisher Sci. Catalogue # |
| Wrist Shaker | Boekel Scientific | 401000-2 | |
| Kaiser test kit | Sigma-Aldrich | 60017 | |
| 2-[(4,4-dimethyl-2,6-dioxocyclohex-1-ylidene)ethyl-amino]-ethanol | Sigma-Aldrich | CDS004772 | |
| Anhydrous Methanol | Acros | AC610981000 | Fisher Sci. Catalogue # |
| Chloranil test kit | TCI | TCC1771-KIT | VWR Catalogue # |
| Di-tert butyl di-carbonate | Acros | AC194670250 | Fisher Sci. Catalogue # |
| Dimethylformamide | Fisher Scientific | BP1160-4 | |
| Hydrazine | Acros | AC296815000 | FIsher Sci. Catalogue # |
| (2-(1H-benzotriazol-1-yl)-1,1,3,3-tetramethyluronium hexafluorophosphate) | p3biosystems | 31001 | |
| 4-methyl piperidine | Acros | AC127515000 | FIsher Sci. Catalogue # |
| Trifluoroacetic Acid | AappTec | CXZ035 | |
| Triisopropyl Silane | Sigma-Aldrich | 233781 | |
| Ether | Fisher Scientific | E138-1 | |
| α-Cyano-4-hydroxycinnamic acid | Sigma-Aldrich | C8982 | |
| 9-Aminoacridine | Sigma-Aldrich | 92817 | |
| Fisherbrand Syringe Filters: PTFE Membrane | Fisher Scientific | 09-730-21 |
Riferimenti
- Cui, H., Pashuck, E. T., Velichko, Y. S., Weigand, S. J., Cheetham, A. G., Newcomb, C. J., Stupp, S. I. Spontaneous and x-ray-triggered crystallization at long range in self-assembling filament networks. Science. 327, 555-559 (2010).
- Pashuck, E. T., Cui, H., Stupp, S. I. Tuning supramolecular rigidity of peptide fibers through molecular structure. Journal of the American Chemical Society. 132, 6041-6046 (2010).
- Stupp, S. I., Zha, R. H., Palmer, L. C., Cui, H., Bitton, R. Self-assembly of biomolecular soft matter. Faraday Discussions. 166, 9-30 (2013).
- Conda-Sheridan, M., Lee, S. S., Preslar, A. T., Stupp, S. I. Esterase-activated release of naproxen from supramolecular nanofibres. Chemical Communications. 50, 13757-13760 (2014).
- Mata, A., Palmer, L., Tejeda-Montes, E., Stupp, S. I. Design of biomolecules for nanoengineered biomaterials for regenerative medicine. Nanotechnology in Regenerative Medicine. , 39-49 (2012).
- Samad, M. B., Chhonker, Y. S., Contreras, J. I., McCarthy, A., McClanahan, M. M., Murry, D. J., Conda-Sheridan, M. Developing Polyamine-Based Peptide Amphiphiles with Tunable Morphology and Physicochemical Properties. Macromolecular bioscience. 17, (2017).
- Nel, A. E., Mädler, L., Velegol, D., Xia, T., Hoek, E. M., Somasundaran, P., Klaessig, F., Castranova, V., Thompson, M. Understanding biophysicochemical interactions at the nano-bio interface. Nature Materials. 8, 543 (2009).
- Gujrati, M., Malamas, A., Shin, T., Jin, E., Sun, Y., Lu, Z. -. R. Multifunctional cationic lipid-based nanoparticles facilitate endosomal escape and reduction-triggered cytosolic siRNA release. Molecular Pharmaceutics. 11, 2734-2744 (2014).
- Zhu, Y., Li, J., Kanvinde, S., Lin, Z., Hazeldine, S., Singh, R. K., Oupický, D. Self-immolative polycations as gene delivery vectors and prodrugs targeting polyamine metabolism in cancer. Molecular Pharmaceutics. 12, 332-341 (2014).
- Planas-Portell, J., Gallart, M., Tiburcio, A. F., Altabella, T. Copper-containing amine oxidases contribute to terminal polyamine oxidation in peroxisomes and apoplast of Arabidopsis thaliana. BMC Plant Biology. 13, 109 (2013).
- Nash, I. A., Bycroft, B. W., Chan, W. C. Dde - A selective primary amine protecting group: A facile solid phase synthetic approach to polyamine conjugates. Tetrahedron Letters. 37, 2625-2628 (1996).
- Ralhan, K., KrishnaKumar, V. G., Gupta, S. Piperazine and DBU: a safer alternative for rapid and efficient Fmoc deprotection in solid phase peptide synthesis. RSC Advances. 5, 104417-104425 (2015).
- Casero, R. A., Marton, L. J. Targeting polyamine metabolism and function in cancer and other hyperproliferative diseases. Nature Reviews Drug Discovery. 6, 373 (2007).
- Wuts, P. G. M., Greene, T. W. . Protection for the Amino Group. In Greene's Protective Groups in Organic Synthesis. , 696-926 (2006).
- Palasek, S. A., Cox, Z. J., Collins, J. M. Limiting racemization and aspartimide formation in microwave-enhanced Fmoc solid phase peptide synthesis. Journal of Peptide Science. 13, 143-148 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon