Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantitative [18F]-Naf-PET-MRI-Analyse zur Bewertung des dynamischen Knochenumsatzes bei Patienten mit facetogenen Rückenschmerzen
In diesem Artikel
Zusammenfassung
Bildgebende Verfahren, die den dynamischen Knochenumsatz widerspiegeln, können bei der Charakterisierung einer Vielzahl von Knochenpathologien helfen. Wir präsentieren detaillierte Methoden zur Durchführung und Analyse dynamischer [18F]-NaF-PET-MRT-Daten bei Patienten mit facetogenen Rückenschmerzen unter Verwendung der Lendenfacetgelenke als prototypische Interessensregion.
Zusammenfassung
Bildgebende Verfahren, die den dynamischen Knochenumsatz widerspiegeln, können bei der Charakterisierung einer Vielzahl von Knochenpathologien helfen. Knochen ist ein dynamisches Gewebe, das sich kontinuierlich umgestaltet, mit der konkurrierenden Aktivität von Osteoblasten, die die neue Knochenmatrix produzieren, und Osteoklasten, deren Funktion darin besteht, mineralisierte Knochen zu beseitigen. [18F]-NaF ist ein Pet-Radiotracer (Positronemission Tomography), der die Visualisierung des Knochenstoffwechsels ermöglicht. [18F]-NaF wird in der Knochenmatrix durch Osteoblasten chemisch in Hydroxyapatit aufgenommen und kann somit nichtinvasiv osteoblastische Aktivität erkennen, die bei herkömmlichen bildgebenden Verfahren okkult ist. Die kinetische Modellierung dynamischer [18F]-NaF-PET-Daten liefert detaillierte quantitative Messgrößen für den Knochenstoffwechsel. Herkömmliche semiquantitative PET-Daten, die standardisierte Aufnahmewerte (SUVs) als Maß für die Radiotracer-Aktivität verwenden, werden aufgrund ihrer Momentaufnahme der Traceraufnahme in der Zeit als statische Technik bezeichnet. Die kinetische Modellierung nutzt jedoch dynamische Bilddaten, bei denen Tracerebenen kontinuierlich erfasst werden, um eine zeitliche Auflösung der Traceraufnahme zu bieten. Aus der kinetischen Modellierung dynamischer Daten können quantitative Werte wie Blutfluss und Stoffwechselrate (d.h. potenziell informative Metriken der Tracerdynamik) extrahiert werden, alles in Bezug auf die gemessene Aktivität in den Bilddaten. In Kombination mit der dualen Modalität PET-MRT können regionsspezifische kinetische Daten mit anatomisch registrierten hochauflösenden strukturellen und pathologischen Informationen korreliert werden, die durch MRT zur Seite gelegt werden. Das Ziel dieses methodischen Manuskripts ist es, detaillierte Techniken zur Durchführung und Analyse dynamischer [18F]-NaF-PET-MRT-Daten zu skizzieren. Das Lendengelenk ist ein häufiger Ort der degenerativen Arthritis-Krankheit und eine häufige Ursache für axiale Rückenschmerzen. Neuere Studien deuten darauf hin, dass [18F]-NaF-PET als nützlicher Biomarker für schmerzhafte facetogene Krankheiten dienen kann. Das menschliche Lendengelenk wird daher als prototypische Interessensregion für die dynamische [18F]-NaF-PET-MRI-Analyse in diesem Manuskript verwendet.
Einleitung
Standard-klinische bildgebende Verfahren der Knochenpathologie beschränken sich in erster Linie auf die Charakterisierung struktureller Veränderungen, die unspezifisch sein können. Zum Beispiel können asymptomatische morphologische Anomalien im Zusammenhang mit der normalen Alterung nicht von degenerativen Veränderungen zu unterscheiden sein, die für starke Schmerzen und Behinderung verantwortlich sind1. Knochen ist ein dynamisches Gewebe, das sich kontinuierlich umgestaltet, mit der konkurrierenden Aktivität von Osteoblasten, die die neue Knochenmatrix produzieren, und Osteoklasten, deren Funktion darin besteht, mineralisierte Knochen zu beseitigen2. [18F]-NaF ist ein Pet-Radiotracer (Positronemission Tomography), der die Visualisierung des Knochengewebestoffwechsels ermöglicht. [18F]-NaF wird durch Osteoblasten chemisch in Hydroxyapatit in der Knochenmatrix absorbiert und kann somit osteoblastische Aktivität nicht invasiv erkennen und so einen Stoffwechselprozess erkennen, der bei herkömmlichen bildgebenden Verfahren okkult ist. Als Ergebnis, [18F]-NaF wurde zur Charakterisierung der Knochenpathologie in einer zunehmenden Anzahl von Knochenerkrankungen einschließlich Neoplasmen, entzündliche, und degenerative Erkrankungen der Knochen und Gelenke3,4,5 .
PET-Daten werden am häufigsten semiquantitativ analysiert, was in der klinischen Routinepraxis mit standardisierten Aufnahmewerten (SUVs) problemlos durchgeführt werden kann. Als Metrik sind SUVs für Ärzte nützlich, da sie die Gewebeaufnahme im Vergleich zum Rest des Körpers darstellen6. Werte aus nachfolgenden Scans können verwendet werden, um Veränderungen der Aufnahme infolge der Behandlung oder des Krankheitsverlaufs zu beobachten. Die numerische Natur von SUVs hilft auch im Vergleich zwischen Patienten und aufeinanderfolgenden Scans bei demselben Patienten. Der Algorithmus zur Berechnung von SUVs, Gleichung 1, geht davon aus, dass der Tracer gleichmäßig über den Körper verteilt ist und dass die schlanke Körpermasse das gesamte Körpervolumen genau darstellt. SuVs sind daher eine semiquantitative Messung. Für eine bestimmte Region von Interesse (ROI), SUVmax (der maximale SUV-Wert innerhalb eines ROI) undSUV-Mittelwert (der Mittelwert aller abgetasteten SUVs innerhalb eines ROI) sind häufig SUV-Metriken in der klinischen Praxis6verwendet.
Die kinetische Modellierung dynamischer PET-Daten kann auch für detailliertere quantitative Analysen durchgeführt werden. Während die SUV-Datenerfassung statisch ist, nutzt die kinetische Modellierung dynamische Bilddaten, bei denen kontinuierlich Tracer-Ebenen erfasst werden, die eine zeitliche Dimension bieten. Aus der komplexeren kinetischen Modellierung dynamischer Daten können quantitative Werte und informative Metriken der Tracerdynamik in Bezug auf die gemessene Aktivität in den Bilddaten extrahiert werden. Abbildung 17zeigt ein Mustermodell für zwei Gewebefächer, das für die dynamische kinetische Modellierung verwendet wird. Cp ist die Konzentration des Tracers im Blutplasma, während Ce und Ct die Konzentration im ungebundenen interstitiellen Raum bzw. gebundenen Tracer in der Zielknochenmatrix darstellen. K1, k2, k3, k4, sind 4 Rate-Parameter, die das kinetische Modell für Tracer-Ein-/Auswaschen und Binden beschreiben. K1 beschreibt den Tracer, der vom arteriellen Plasma in den interstitiellen Raum (Ct) aufgenommen wird, k2 beschreibt den Bruchteil des Tracers, der vom interstitiellen Raum zum Plasma zurückdiffusiver, k3 beschreibt den Tracer, der sich von interstitielle (Ce) Raum zu Knochen (Ct), und k4 beschreibt den Tracer, der sich vom Knochen (Ct) zurück zum interstitiellen Raum (Ce) bewegt.

Abbildung 1 . Ein Beispielmodell für zwei Gewebefächer für die dynamische kinetische Modellierung. Cp ist die Tracerkonzentration im Blutplasmakompartiment, die Ce freie und nicht spezifisch gebundene Tracerkonzentration im Gewebe und diespeziell gebundene Tracerkonzentration im Gewebe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Das kinetische Modell Patlak erzeugt Ki_Patlak als Maß für die Radiotracer-Zuflussrate (mL/ccm/min, kubisch cm = ccm) aus dem Blutpool in die Knochenmatrix. Die Tracerzuflussrate vom Blutpool zur Knochenmatrix kann dann mit Gleichung 2 und Gleichung 3 für Ki_Patlak bzw. Ki_NonLinear berechnet werden. Ki_Patlak und Ki_NonLinear sind die Raten, bei denen [18F]-NaF den arteriellen Blutpool verlässt und irreversibel an eine subsiteknochenmatrix bindet, mit den beiden Modellen. Ein Unterschied zwischen dem Patlak- und dem nichtlinearen kinetischen Modell liegt in der Nutzung der dynamischen Daten. Das Patlak-Modell erfordert das Erreichen des Gleichgewichts und berechnet dann die Zuflussrate aus der etablierten linearen Steigung. Das kinetische Modell Patlak erzeugt Ki_Patlak Zuflussraten, indem eine 24-minütige Zeit verwendet wird, um den Plasmapool Cpzum ungebundenen Pool Cuzu ausgrenzen. Die 24-Minuten-Zeit kann sich je nach der für alle Unterwebsites gefundenen Zeit ändern, um eine Gleichgewichtsmöglichkeit mit dem Plasmapool in der Probe zu erreichen. Das rechenintensivere nichtlineare Modell verwendet die Gesamtheit der Zeitdaten, um eine Kurve anzupassen.
Das Ziel dieses methodischen Manuskripts ist es, detaillierte Techniken zur Durchführung dynamischer [18F]-NaF-PET-MRT zu skizzieren. Das Lendengelenk ist ein häufiger Ort der degenerativen Arthritis-Krankheit und eine häufige Ursache für axiale Rückenschmerzen8. Neuere Studien deuten darauf hin, dass [18F]-NaF-PET-MRI als nützlicher Biomarker für schmerzhafte gesichtstogeneKrankheiten9 dienen kann. Die menschlichen Lendengelenke eines einzelnen Patienten mit facetogenen Rückenschmerzen werden somit als prototypischer ROI für die dynamische [18F]-NaF-PET-MRI-Analyse analysiert.
Protokoll
Diese prospektive Machbarkeitsstudie rekrutierte Patienten, nachdem sie die Human Study IRB-Zulassung erhalten und die HIPAA-Vorschriften eingehalten hatte.
1. Phantom
- Füllen Sie ein hohles zylindrisches Phantom mit einem Einsatz mit Hohlzylindern mit einem Durchmesserbereich von 5 - 38 mm mit 185 MBq von [18F]-NaF.
- Generieren Sie eine Dämpfungszuordnung des Phantoms mithilfe von CT oder einer Vorlage, die zuvor für dieses Phantom generiert wurde.
- Legen Sie das Phantom in die Mitte von PET/MR und erfassen Sie PET-Daten für 5-10 min Aufnahme des resultierenden Bildes mit der Bildgebungskonsole.
- Rekonstruieren Sie die Bildgebungskonsole mit der CT-basierten Dämpfungskarte mit einem Algorithmus, der mit demselben Rekonstruktionsalgorithmus übereinstimmt, der für die Abbildung menschlicher Probanden verwendet wird.
- Berechnen Sie die mittlere Aktivität in jedem Zylinder (links und rechts) gleicher Größe für alle Größen mit Freeware AMIDE.
- Tabuulieren Sie die mittlere Aktivität im Vergleich zur Zylindergröße.
- Berechnen Sie die Teilvolumenfehler (PVE), indem Sie die mittlere Aktivität jedes Zylinders durch die Mittelwertaktivität des Referenzzylinders dividieren.
- Plotten Sie die PVE nach der Größe des Zylinders.
- Verwenden Sie die lineare Gleichung zwischen Zweizylindergrößen, wenn Sie PVE in den Patientendaten korrigieren.
2. Patientenvorbereitung
- Bevor Sie Patienten rekrutieren, erhalten Sie die erforderliche Human Study IRB-Zulassung und halten Sie sich an die HIPPA-Vorschriften.
- Festlegung geeigneter Inklusions- und Ausschlusskriterien für die Untersuchung von Interesse.
- Die Kriterien für die Aufnahme waren wie folgt: Erwachsene, die mindestens 18 Jahre alt sind und über die Möglichkeit der Einwilligung in Kenntnis der Sachlage verfügen; eine berichtete Geschichte von axialen nicht-radikulären Rückenschmerzen; von den interventionellen Sapine-Radiologen empfohlen.
- Ausschlusskriterien waren wie folgt: die Geschichte der Fraktur oder Tumor der Wirbelsäule; Frauen, die schwanger sind oder stillen; Kontraindikationen für eine MRT oder die Verabreichung von Tracer oder Kontrast; vor der Lendenwirbelsäulenoperation oder -instrumentierung.
- Sammeln Sie die schriftliche Zustimmung des Patienten in Kenntnis der Sachlage, die vom Ausschuss für Humanforschung genehmigt wurde.
- Erhalten Sie alle relevanten klinischen Prüfungs- und/oder Patientenbefragungsdaten, die für Ihre Studie von Interesse sind.
- Lassen Sie sich in das Kleid wechseln, stellen Sie IV-Zugang her, verabreichen Sie einen Schwangerschaftstest, wenn der Patient weiblich ist und das gebärfähigkeitsgebärbte Alter hat, überprüfen Sie Kreatinin/GFR auf die sichere Verwendung von Kontrast, und rufen Sie [18F]-NaF-Dosis ab. Coachen Sie den Patienten darüber, wie wichtig es ist, während der gesamten Dauer der Untersuchung still zu bleiben.
- Positionieren Sie patientensupine und füße zuerst im PET/MRT.
3. Imaging-Protokoll
- Verwenden Sie einen 3,0 T PET/MRT-Scanner zur gleichzeitigen PET- und MR-Bildaufnahme.
- Verwenden Sie die zentrale molekulare Bildmaterial-Array-Spule des hinteren Arrays für die MR-Bildgebung.
- Stellen Sie sicher, dass der FOV der MR- und PET-Bildgebungsmodalitäten zentriert ist, um den unteren Wirbelsäulenbereich von T12 bis S3 abzudecken.
- Die klinischen MRT-Sequenzen für das Lumbar-Spine-Protokoll umfassen: Sagittal T1 (Wiederholungszeit/Echozeit (TR/TE) = 510/8,6 ms, In-Plane-Auflösung = 0,75 mm, Durchgangsebene Auflösung = 4mm), Sagittal T2 fettgesättigt (FS) (TR/TE = 4208/86,2 ms, in-plane Auflösung = 0,75mm, Durchgangsebene Auflösung = 4mm) Axial T2 Schnellentspannung Schnellspin Echo (FRFSE) mit und ohne Fettsättigung (TR/TE = 750/9,2 ms, In-Plane-Auflösung = 0,7mm, Durchgangsebene Auflösung = 4mm), Axial T1 Schnellspin Echo (FSE) Pre Gadolinium (TR/ TE = 575/8,9 ms, In-Plane-Auflösung = 0,65 mm, Durchgangsebenenauflösung = 4mm), Axial T1 FSE Post Gadolinium (TR/TE = 562/8,6 ms, In-Plane-Auflösung = 0,65 mm, Durchgangsebenenauflösung = 4mm).
- Injizieren Sie 0,1 mm/kg Gadbutrol (1M Gadavist) direkt vor dem Erwerb der MRT-Sequenzen, die sie erfordern, in die antecubitale Fossa IV des Patienten.
- Vor dem dynamischen PET-Scan injizieren Sie die radioaktive Dosis von [18F]-NaF in eine Konzentration von 2,96 MBq/kg [18F]-NaF.
- Führen Sie einen dynamischen PET-Scan von 60 min mit 3 separaten Zeitphasen aus, die über der unteren Wirbelsäule zentriert sind, T12 bis S3.
- Erwerben Sie die erste Phase des dynamischen Scans mit 12 Bildern zu je 10 s.
- Erwerben Sie die zweite Phase von 4 Bildern zu je 30 s.
- Erwerben Sie die letzte Phase von 14 Bildern zu je 4 min.
- Berechnen Sie die MR-Dämpfungskorrektur (MRAC) für den Lendenwirbelsäulenbereich mit der standardmäßigen Zweipunkt-Dixon-Methode. Die Dixon-Methode segmentiert die MR-Signale von Fett und Wasser in Luft, Weichgewebe, Lunge und Fett (wenn auch nicht Knochen).
- Stellen Sie sicher, dass PET-Daten gemeinsam in axiale T2-fettgesättigte FRFSE-Bilder registriert werden.
- Rekonstruieren Sie die PET-Daten auf der Konsole mit den folgenden Parametern: 60 cm Sichtfeld (FOV), 3 mm Nachfilter, Standard-Z-Achsenfilter, 256 x 256 Matrix, 28 Teilmengen und VPFX (Time of Flight - Ordered Subsets Expectation Maximization, TOF-OSEM) mit 4 Iterationen .
- Stellen Sie sicher, dass die Rekonstruktion eine Nachbearbeitung umfasst, um Zerfall, Dämpfung, Streuung und Totzeit zu korrigieren.
4. Bildanalyse
- Lassen Sie blinde Radiologen die klinischen MRT-Sequenzen interpretieren.
- Bewerten Sie fettunterdrückte T2- gewichtete und fett unterdrückte T1-gewichtete Postkontrastsequenzen zur Einstufung facet synovitis, wie zuvor von Czervionke und Fenton10beschrieben.
- Verwenden Sie die folgende Facette-Einstufung ist: MRT-Grad 0 = keine Anomalie des Facettengelenks, 1 = abnormale Verbesserung oder T2-Hyperintensität begrenzt auf Gelenkkapsel, 2 = abnormale extrakapsuläre Verbesserung oder T2-Hyperintensität mit < 50% des Y-Umfangs, 3 = abnormal extrakapsuläre Verbesserung oder T2-Hyperintensität mit >50% fj Perimeter, und 4 = Grad 3 mit Verlängerung des Ödems in das Neuroforamen, Ligementum flavum, Pedikle, Querprozess oder Wirbelkörper. Wie in der Ref: Czervionke LF, Fenton DS erklärt. Fettgesättigte MR-Bildgebung beim Nachweis von entzündlicher Facettenarthropathie (Facet Synovitis) in der Lendenwirbelsäule. 10
5. Datenanalyse
- Übertragen Sie PET- und MRT-Bilder an einen dedizierten Arbeitsplatz, der zur Analyse dynamischer PET-Daten wie der Software PMOD ausgestattet ist. Analysieren Sie die Facettengelenke der Lendenwirbelsäule von L1-L2 bis L5-S1.
- Suchen Sie Regionen, die für [18F]-NaF-Aufnahmemessungen ausgewertet werden: bilaterale Facettengelenke auf jeder Ebene. Wählen Sie Volumes of Interest (VOI) mithilfe anatomischer T2-MR-Bilder aus und übertragen Sie sie dann auf die PET-Bilder.
- Identifizieren Sie den Mittelpunkt jeder Lendenhülse, indem Sie visuell triangulierend mit sagittalen und axialen T2-MR-Bildern und die Scheibennummer des ungefähren Zentrums aufzeichnen.
- Wenn die Patientendaten auf der Registerkarte Ansichtgeöffnet sind, klicken Sie in der Seitenleiste auf die VOI-Schaltfläche und wählen Sie SPHERE (Objekt).
- Geben Sie im vordefinierten Fenster, das angezeigt wird, 7,5 mm als Radius ein, und klicken Sie auf Neue VOI erstellen.
- Platzieren Sie einen kugelförmigen VOI (7,5 mm Durchmesser) in der Mitte jeder Facettenverbindung, indem Sie mit der linken Maustaste auf die Facette klicken. Passen Sie die Kugel per Linksklick und Ziehen an, bis sie visuell auf der Facette zentriert ist.
- Wiederholen Sie dies bei Bedarf für alle Interessensgebiete, indem Sie auf Neue VOI erstellen klicken und Schritt 5.5 ausführen
- Platzieren Sie einen kugelförmigen VOI (5 mm Durchmesser) in den rechten Iliaskamm in der zentralen Markhöhle (um die Kortexbeteiligung auszuschließen) als Referenzbereich. Klicken Sie auf Neue VOI erstellen und klicken Sie mit der linken Maustaste in das Mark der rechten Ilias.
- Positionieren Sie VOI so Kanten sind innerhalb des Mark vollständig.
- Stellen Sie sicher, dass VOIs ähnlich wie das Bild platziert sind, das die VOIs des Wirbelkörpers (FJ) in Abbildung 2 zeigt, da sie die Mitte des Facettengelenks verkapseln.
6. SUV-Berechnungen und kinetische Daten
- Um die arterielle Eingangsfunktion zu berechnen, platzieren Sie einen zylindrischen VOI, der zwei axiale Scheiben der Bauchaorta abdeckt. Stellen Sie sicher, dass der Durchmesser dem Durchmesser der Aorta entspricht.
- Rechtsklick auf das Axialbild, wählen Sie Datenprüfung.
- Messen Sie den Durchmesser der Bauchaorta proximal zu ihrer Bifurkation.
- Klicken Sie mit der linken Maustaste auf die rechte Seite der Aortenwand und bewegen Sie den Cursor auf die linke Seite der Aortenwand.
- Zeichnen Sie den Abstand des Aortenwanddurchmessers im Fenster Dateninspektor auf. Dies wird verwendet, um den Koeffizienten der Teilvolumenkorrektur (PVC) zu berechnen.
- Klicken Sie in der Seitenleiste mit der linken Maustaste auf die SCHALTFLÄCHE VOI, wählen Sie CIRCLE (ROI).
- Erstellen eines Kreis-ROI mit einem angegebenen Radius von der Hälfte des zuvor gemessenen Durchmessers in Schritt 6.5
- Klicken Sie auf Neue VOI erstellen und klicken Sie mit der linken Maustaste in die Mitte der Aorta, positionieren Sie sie ggf. neu, um sicherzustellen, dass der Kreis die Aortenwandposition annähert.
- Eine Scheibe in der Axialebene absteigen und die Schritte 6.7-6.9 wiederholen, wodurch ein Zylinder aus den beiden kreisförmigen ROI s wird.
7. PET Partielle Volumenkorrektur
HINWEIS: Aufgrund von PVE wird die Traceraktivität im Verhältnis zur Größe des Ziels unterschätzt. Daher werden Schritte unternommen, um die PVE zu korrigieren.
- Verwenden Sie die zuvor abgeleiteten Rückgewinnungskoeffizienten mithilfe des PET/CT-Phantoms, indem Sie die Größe des Zylinderdurchmessers im Vergleich zum Verhältnis der wiedergewonnenen Aktivität zur tatsächlichen Aktivität zeichnen.
- Wenden Sie die Wiederherstellungskoeffizienten auf die bildbasierte Messung über die absteigende Aorta an, um eine teilvolumenkorrigierte arterielle Eingabe zu erstellen.
- Ersetzen Sie diesen teilvolumenkorrigierten arteriellen Eingang in PMOD für die Kinetikmodellierung und die genaue Quantifizierung der Tracerkinetik.
8. SUV-Berechnungen und kinetische Daten
Anmerkung: Der Algorithmus zur Berechnung des Standard-Aufnahmewerts (SUV), Gleichung 1, geht davon aus, dass der Tracer gleichmäßig über den Körper verteilt ist und dass die schlanke Körpermasse das gesamte Körpervolumen genau darstellt. Daher werden SUVs als semiquantitative Messung bezeichnet.
Gleichung 1: Standardaufnahmewert
- Berechnen Sie dieMITTELwerte SUVmaxund SUV für jede Unterwebsite mit dem 60-Minuten-Zeitpunkt.
Anmerkung: Das Zwei-Gewebe-Fachmodell, das für die kinetische Modellierung verwendet wird, ist in Abb. 1 dargestellt. Cp ist die Konzentration des Tracers im Blutplasma, während Ce und Ct die Konzentration im ungebundenen interstitiellen Raum bzw. gebundenen Tracer in der Zielknochenmatrix darstellen. K1, k2, k3, k4, sind 4 Parameter, die das kinetische Modell für Tracer-Ein-/Auswaschen und Binden beschreiben. - Verwenden Sie das Zwei-Gewebe-Fach irreversibel für Patlak Linear-Modell und nicht-lineare Regressionsmodelle während der kinetischen Analyse
Anmerkung: Ein irreversibles Zwei-Gewebe-Kompartimentmodell wird verwendet, um regionsspezifische Zuflussratenkonstanten (in min-1) für [18F]-NaF11zu berechnen. - Stellen Sie sicher, dass die Zeit bis zum Gleichgewicht auf 24 Minuten eingestellt ist, wenn Sie das kinetische Patlak-Modell verwenden
- Eingabe k4 = 0, wenn das nichtlineare Regressionsmodell verwendet wird, um Ki_NonLinear-Zuflussraten zu erzeugen.
- Berechnen Sie die Tracer-Zuflussrate vom Blutpool zur Knochenmatrix mit Gleichung 2 und Gleichung 3 für Ki_Patlak bzw. Ki_NonLinear. Ki_Patlak und Ki_NonLinear sind die Raten, bei denen [18F]-NaF den arteriellen Blutpool verlässt und irreversibel an eine subsiteknochenmatrix bindet, mit den beiden Modellen.
-
Gleichung 2: Patlak Graphical Kinetic Model
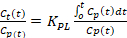 + Abfangen
+ Abfangen -
Gleichung 3: Nichtlineares Regressionskinetikmodell

-
Gleichung 2: Patlak Graphical Kinetic Model
9. Statistische Analyse
- Verwenden Sie eine lineare Regressionsanalyse, um zu bewerten, ob [18F]-NaF Ki_Patlak Zuflussrate korreliert war mit: SUVmittel, SUVmax, Ki_NonLinear, und alle klinischen Scoring-Grade spezifisch für die Studie.
- Verwenden Sie zweischwanzige t-Test- und Pearson-Korrelation, um auf statistische Signifikanz in früheren Korrelationen zu testen.
Ergebnisse
18 Die NaF-PET-Aufnahmewerte werden in den bilateralen Facettengelenken der Wirbelstufen L1-L2 bis L5-S1 für insgesamt 10 ROIs bei einem einzigen repräsentativen Patienten mit axialen Rückenschmerzen gemessen. Repräsentative [18F]-NaF-PET, axiales T2-Fett unterdrückt und axiale T1-nachkontrastsunterdrückte MR-Bilder durch den Pegel der L3-L4 Facettengelenke sind in Abbildung 2dargestellt. Die Ki_Patlak, SUVmittel, SUVmax, und MRT...
Diskussion
In diesem methodischen Manuskript haben wir Hintergrundinformationen zum potenziellen Nutzen von dynamischem [18F]-NaF-PET-MRT zur Bewertung einer Vielzahl von Knochenpathologien geliefert und die Techniken für dynamisches [18F]-NaF-PET-MRI-Bild skizziert. Erfassung und Analyse unter Verwendung der menschlichen Lendengelenke als prototypische Interessengebiete. Die duale Modalität PET-MRT ermöglicht die Erfassung dynamischer PET-Daten über einen Zeitraum, der dem für die MR-Datenerfassung allei...
Offenlegungen
Die Autoren haben nichts zu verraten
Danksagungen
Forschungsunterstützung wurde von NIH P50AR060752 und GE Healthcare geleistet. Wir möchten die Unterstützung von Vahid Ravanfar würdigen.
Materialien
| Name | Company | Catalog Number | Comments |
| Gadolinium Contrast agent (Gadovist) | Bayer | na | 1.0mmol/ml solution for IV injection. |
| [18F]-NaF Radiotracer | na | na | 2.96 MBq/kg |
| GE Signa PET-MRI Scanner | General Electric | na | 3.0Tesla 60cm Bore PET-MRI scanner |
| PMOD Kinetic Modeling Software | PMOD Technologies, LLC | na | Version 3.8 |
Referenzen
- Brinjikji, W., et al. Systematic literature review of imaging features of spinal degeneration in asymptomatic populations. AJNR American Journal of Neuroradiology. 36 (4), 811-816 (2015).
- Binder, D. S., Nampiaparampil, D. E. The provocative lumbar facet joint. Current Reviews in Musculoskeletal Medicine. 2 (1), 15-24 (2009).
- Spick, C., et al. Detection of Bone Metastases Using 11C-Acetate PET in Patients with Prostate Cancer with Biochemical Recurrence. Anticancer Research. 35 (12), 6787-6791 (2015).
- Brans, B., et al. Assessment of bone graft incorporation by 18 F-fluoride positron-emission tomography/computed tomography in patients with persisting symptoms after posterior lumbar interbody fusion. EJNMMI Research. 2 (1), 42 (2012).
- Jadvar, H., et al. Prospective evaluation of 18F-NaF and 18F-FDG PET/CT in detection of occult metastatic disease in biochemical recurrence of prostate cancer. Clinical Nuclear Medicine. 37 (7), 637-643 (2012).
- Kinahan, P. E., Fletcher, J. W. Positron emission tomography-computed tomography standardized uptake values in clinical practice and assessing response to therapy. Seminars in Ultrasound, CT, and MR. 31 (6), 496-505 (2010).
- Hawkins, R. A., et al. Evaluation of the skeletal kinetics of fluorine-18-fluoride ion with PET. Journal of Nuclear Medicine: Official Publication, Society of Nuclear Medicine. 33 (5), 633-642 (1992).
- Hancock, M. J., et al. Systematic review of tests to identify the disc, SIJ or facet joint as the source of low back pain. European Spine Journal: Official Publication of the European Spine Society, the European Spinal Deformity Society, and the European Section of the Cervical Spine Research Society. 16 (10), 1539-1550 (2007).
- Jenkins, N. W., et al. [18)F]-Sodium Fluoride PET MR-Based Localization and Quantification of Bone Turnover as a Biomarker for Facet Joint-Induced Disability. AJNR American Journal of Neuroradiology. 38 (10), 2028-2031 (2017).
- Czervionke, L. F., Fenton, D. S. Fat-saturated MR imaging in the detection of inflammatory facet arthropathy (facet synovitis) in the lumbar spine. Pain Medicine. 9 (4), 400-406 (2008).
- Phelps, M. E., et al. Tomographic measurement of local cerebral glucose metabolic rate in humans with (F-18)2-fluoro-2-deoxy-D-glucose: validation of method. Annals of Neurology. 6 (5), 371-388 (1979).
- Brenner, W., et al. Comparison of different quantitative approaches to 18F-fluoride PET scans. Journal of Nuclear Medicine: Official Publication, Society of Nuclear Medicine. 45 (9), 1493-1500 (2004).
- Schellinger, D., et al. Facet joint disorders and their role in the production of back pain and sciatica. Radiographics: A Review Publication of the Radiological Society of North America, Inc. 7 (5), 923-944 (1987).
- Schett, G. Joint remodelling in inflammatory disease. Annals of the Rheumatic Diseases. 66, 42-44 (2007).
- Baum, R., Gravallese, E. M. Impact of inflammation on the osteoblast in rheumatic diseases. Current Osteoporosis Reports. 12 (1), 9-16 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten