Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Bioengineering
Manipulation de cellules souches neurales et de neurones dans les tranches de cerveau à l’aide de microinjection robotique
Ce protocole démontre l’utilisation d’une plate-forme robotique pour la microinjection en cellules souches neurales simples et neurones en tranches de cerveau. Cette technique est polyvalente et offre une méthode de suivi des cellules dans les tissus à haute résolution spatiale.
Une question centrale dans la neurobiologie développementale est comment les cellules souches neurales et progénitrices forment le cerveau. Pour répondre à cette question, il faut étiqueter, manipuler et suivre les cellules individuelles dans le tissu cérébral à haute résolution au fil du temps. Cette tâche est extrêmement difficile en raison de la complexité des tissus dans le cerveau. Nous avons récemment mis au point un robot, qui guide une aiguille de microinjection dans le tissu cérébral en utilisant des images acquises à partir d’un microscope pour fournir des volumes de femtoliter de solution en cellules individuelles. L’opération robotique augmente résultant d’un rendement global qui est un ordre de grandeur supérieur à la microinjection manuelle et permet un étiquetage précis et une manipulation flexible des cellules individuelles dans les tissus vivants. Avec cela, on peut microinjecter des centaines de cellules dans une seule tranche organotypique. Cet article démontre l’utilisation du robot de microinjection pour la microinjection automatisée des cellules progénitrices neurales et des neurones dans les tranches de tissu cérébral. Plus largement, il peut être utilisé sur n’importe quel tissu épithélial comportant une surface qui peut être atteinte par la pipette. Une fois mis en place, le robot de microinjection peut exécuter 15 microinjections ou plus par minute. Le robot de microinjection en raison de son débit et de sa versalité fera de la microinjection une technique de manipulation cellulaire haute performance largement simple à utiliser dans la bioingénierie, la biotechnologie et la biophysique pour effectuer des analyses à cellules simples dans des tranches de cerveau organotypiques.
Ce protocole décrit l’utilisation d’un robot pour cibler et manipuler des cellules individuelles dans des tranches de tissu cérébral, en se concentrant en particulier sur les cellules souches neurales et les neurones. Le robot a été développé pour répondre à une question centrale en neurobiologie développementale, c’est-à-dire comment les cellules souches neurales et progénitrices contribuent à la morphogenèsecérébrale 1,2,3,4,5. Pour répondre à cette question, il faut étiqueter et suivre les cellules souches neurales individuelles et suivre leur progression de lignée au fil du temps pour corréler le comportement d’une seule cellule avec la morphogenèse tissulaire. Cela peut être réalisé de différentes façons, par exemple en électroporant le tissu cérébral in utero ou en étiquetant une seule cellule à l’aide de matrices lipophiles. Bien que puissantes, ces méthodes manquent de résolution précise d’une seule cellule (électroporation) et/ou de la possibilité de manipuler l’espace intracellulaire (colorant lipophile). La microinjection en cellules individuelles a été développée pour surmonter cedéfi 6,7,8. Pendant la microinjection, une pipette est brièvement insérée dans une seule cellule dans le tissu intact sous pression aux volumes de femtoliter de microinject des reagents9. Nous avons précédemment décrit une procédure manuelle pour microinjecter les cellules souches neurales simples dans le tissu organotypique (figure 1A)10,11. La microinjection dans les cellules souches neurales repose sur l’utilisation d’une micropipette insérée dans de simples cellules souches neurales pour injecter une solution contenant un colorant fluorescent, ainsi que d’autres molécules d’intérêt. Le ciblage sélectif des cellules souches neurales est atteint en approchant le télencéphale en développement par l’intermédiaire de la surface ventriculaire (ou ventricule, voir dessin animé dans la figure 1A), qui est formé par la membrane plasmatique apical des progéniteurs apiques (dessin animé dans la figure 1A). Ce processus doit être répété pour chaque cellule que l’expérimentateur souhaite injecter. En outre, le succès de la microinjection dépend du contrôle précis de la profondeur et de la durée de l’injection de micropipette dans le tissu. Ainsi, malgré les avantages uniques, la microinjection manuelle est extrêmement fastidieuse et nécessite une pratique considérable pour effectuer à un débit raisonnable et le rendement, ce qui rend cette technique difficile à utiliser d’une manière évolutive. Pour surmonter cette limitation, nous avons récemment développé un robot guidé par l’image, l’Autoinjector12 (ou robot de microinjection) qui peut automatiquement effectuer des microinjections en cellules individuelles.
Le robot de microinjection utilise des algorithmes d’imagerie microscopique et de vision par ordinateur pour cibler précisément des emplacements spécifiques dans l’espace 3D dans les tissus pour la microinjection (Figure 1B). Le robot de microinjection peut être construit en effectuant des modifications relativement simples à une configuration de microinjection existante. Le schéma global du robot de microinjection est indiqué dans la figure 1C. Une pipette est montée dans un porte-pipette attaché à un manipulateur à trois axes. Une caméra microscope est utilisée pour acquérir des images du tissu et de l’aiguille de microinjection. Un système de régulation de pression personnalisé est utilisé pour contrôler la pression à l’intérieur de la pipette et un micromanipulateur programmable est utilisé pour contrôler la position de la pipette microinjecteur. Les images de la caméra du tissu et de la pipette de microinjection sont utilisées pour déterminer l’emplacement spatial de la pointe de la pipette de microinjection et les endroits où des microinjections doivent être effectuées. Le logiciel calcule ensuite les trajectoires nécessaires pour déplacer la pipette dans le tissu. Tout le matériel est contrôlé par le logiciel que nous avons développé précédemment. Tous les logiciels sont écrits en langage de codage (par exemple, Python et Arduino) et peuvent être téléchargés à partir de https://github.com/bsbrl/Autoinjector avec des instructions. L’interface utilisateur graphique (GUI) permet à l’utilisateur d’imager le tissu et la micropipette, et de personnaliser la trajectoire de la microinjection. Notre système peut être établi à l’aide de modifications relativement simples à un microscope inversé équipé de filtres à champ lumineux et d’épi fluorescence.
Tout d’abord, nous fournissons des instructions sur la préparation des tranches de tissu organotypique du cerveau pour la microinjection. Ensuite, le protocole illustre le démarrage du robot de microinjection suivi d’étapes préparatoires, telles que l’étalonnage du mouvement pipette, qui doivent être faites avant la microinjection. Ceci est suivi par la définition des paramètres d’injection. Après cela, l’utilisateur peut définir la trajectoire utilisée par le robot de microinjection et commencer la procédure d’injection. Le tissu microinjecté (dans ce cas les tranches de tissu organotypique du cerveau) peut être maintenu en culture pendant différentes périodes de temps selon la conception expérimentale10,11. Le tissu peut être traité pour suivre et étudier l’identité et le sort des cellules injectées et leur progéniture. Alternativement, les cellules microinjectées peuvent être suivies à l’aide de l’imagerie en direct. Dans le cadre de ce protocole, nous démontrons l’utilisation du robot pour microinjection automatique des cellules progénitrices neurales dans des tranches organotypiques de souris E14.5 télencéphale dorsale. Le robot est en outre capable de microinjection dans les neurones nouveau-nés dans la souris telencéphale, ainsi que dans le télencéphale fœtal humain12.
En résumé, nous décrivons une plate-forme robotique qui peut être utilisée pour suivre et manipuler des cellules individuelles dans les tissus. La plate-forme fait usage de la pression et il est, par conséquent, extrêmement polyvalent quant à la nature chimique du composé à injecter. En outre, il peut être adapté aux cellules cibles autres que les cellules souches. Nous nous attendons à ce que notre système soit facilement adapté à d’autres systèmes modèles.
Toutes les études sur les animaux ont été menées conformément à la législation allemande sur le bien-être animal, et les licences nécessaires ont été obtenues auprès de la Commission régionale d’éthique pour l’expérimentation animale de Dresde, en Allemagne (Tierversuchskommission, Landesdirektion Dresde). Des tranches organotypiques ont été préparées à partir du télencéphale embryonnaire de souris E14.5 ou E16.5 C57BL/6 (Janvier Labs).
1. Installation de logiciels
- Suivez les instructions pour installer le logiciel à partir https://github.com/bsbrl/Autoinjector.
2. Préparation des reagents et des pipettes
- Agarose : Préparez 3 % d’agarose en dissolvant séparément 3 g d’agarose large gamme et 3 g d’agarose de point de faible fusion dans 100 mL de PBS de qualité culture cellulaire dans deux bouteilles en verre distinctes de 200 mL, respectivement. Conserver à température ambiante jusqu’à 3 mois.
- Solution tyrode : Dissoudre 1 g de bicarbonate de sodium et le sel de Tyrode (utiliser le contenu de toute la bouteille) et 13 ml de 1 M HEPES dans 1 L d’eau distillée. Réglez le pH à 7,4. Filtrer la solution à l’intermédiaire d’un filtre à bouteille de 0,2 μm.
- Milieu de culture de tranche (SCM) : Ajouter 10 mL de sérum de rat, 1 mL de glutamine de 2 mM, 1 mL de pénicilline-streptomycine (100x), 1 mL de supplément N-2 (100x), 2 mL de supplément B27 (50x) et 1 mL de tampon HEPES (pH 7,3) dans 84 mL de milieu neurobasal. Aliquot 5 mL de SCM en tubes de 15 mL. Conserver à -20 °C.
- CO2-Medium indépendant de microinjection (CIMM) : Préparer la solution 5x DMEM modifiée à faible teneur en glucose (sans rouge phénol) en dissolvant la poudre dans 200 mL d’eau distillée. Filtrez la solution à l’intermédiaire d’un filtre à bouteille de 0,2 μm (pour la poudre DMEM, utilisez le contenu de toute la bouteille). Pour préparer 100 mL de CIMM, mélanger 20 mL de solution modifiée 5x DMEM, 1 mL de tampon HEPES, 1 mL de supplément N2 (100x), 2 mL de supplément B27 (50x), 1 mL de pénicilline-streptomycine (100x), 1 mL de glutamine de 2 mM et 74 mL d’eau distillée. Conserver la solution à 4 °C.
- Tampon de reconstitution : Préparez le tampon de reconstitution en dissolvant 262 mM NaHCO3, 0,05 N NaOH, 200 mM HEPES dans de l’eau distillée. Stériliser la solution par filtration à travers un système de filtre de 0,22 μm en bouteille stérile. Aliquot 500 μL de tampon de reconstitution en tubes de microcentrifugeuse hermétiques. Conserver à 4 °C.
- Stock de colorant microinjection : Dissoudre le Dextran étiqueté fluorescent dans de l’eau distillée exempte de RNase (concentration finale de 10 μg/μL). Préparer 5 aliquots μL et les conserver à -20 °C jusqu’à utilisation.
- Tirez les pipettes de microinjection des capillaires en verre borosilicate (diamètre extérieur de 1,2 mm, diamètre intérieur de 0,94 mm) à l’aide du puller micropipette. Protégez les pipettes de la poussière. Ne rangez pas les pipettes pendant plus de 2 à 3 jours. Pour cette expérience, les paramètres de traction étaient HEAT: température de la rampe +1 - 5; PULL: 100; VEL: 110; DEL: 100. HEAT et VEL sont les paramètres qui affectent le plus la forme et la taille de la pipette.
REMARQUE : La pipette optimale de microinjection a une pointe longue et flexible, pour éviter des dommages de cellules pendant la microinjection.
3. Préparation de tranche de tissu
- Faire fondre l’agarose de 3 % à large portée à l’aide d’un four à micro-ondes avant la dissection des tissus cérébraux. Ne laissez pas l’agarose se solidifier en gardant dans un bain d’eau à 37 °C avant l’intégration. Assurez-vous que les pipettes sont protégées de la poussière. Ne rangez pas les pipettes pendant plus de 2 à 3 jours.
- Décongeler un aliquot de SCM et réchauffer 10 - 12 mL CIMM et 20 mL de solution tyrode à 37 °C à l’aide d’un bain d’eau.
- Mélanger le traceur fluorescent (Dextran-3000 ou Dextran-10000-Alexa conjugué; concentration finale 5 - 10 μg/μL) avec l’autre produit chimique à injecter. Centrifugeuse la solution de microinjection à 16 000 x g pendant 30 min à 4 °C. Recueillir le surnatant et transférer dans un nouveau tube. Conserver la solution de microinjection sur la glace jusqu’à utilisation.
- Utilisez les têtes des embryons de souris E13.5 – E16.5 pour préparer des tranches de tissu organotypique du télencéphale. Enlevez la peau et ouvrez le crâne à l’aide des forceps, en se déplaçant le long de la ligne médiane. Disséquer le cerveau embryonnaire du crâne ouvert et enlever les méninges qui recouvrent le tissu cérébral à partir du côté ventral du cerveau. Laissez le cerveau entier disséqué dans la solution de Tyrode sur un bloc chauffant de 37 °C.
REMARQUE : Toutes les étapes de dissection décrites dans 3.4 doivent être exécutées dans la solution préchaurée de Tyrode. - Verser l’agarose fondue large gamme dans un moule d’intégration jetable. Lorsque l’agarose est refroidie à 38 - 39 °C, transférez soigneusement le cerveau (un maximum de 4) en elle à l’aide d’une pipette Pasteur. Utilisez toujours des conseils coupés pour cette étape.
- Remuer l’agarose autour du tissu soit à l’aide d’une spatule ou d’une paire de #1 forceps sans toucher le tissu. Laisser l’agarose se solidifier à température ambiante. Une fois que l’agarose s’est solidifiée, coupez l’agarose excédentaire entourant le tissu.
- Remplissez le plateau tampon de PBS. Orientez le cerveau avec l’axe rostro-caudal du tissu perpendiculaire au plateau (utilisez comme repère les bulbes olfactifs, représentant la partie la plus rostrale du cerveau). Couper 250 tranches de μm à l’aide d’un vibratome.
- Remplir une boîte de Pétri de 3,5 cm de 2 mL de supports préchaurés. À l’aide d’une pipette Pasteur en plastique, transférer les tranches (10 à 15) dans ce plat. Une fois terminé, déplacez la boîte de Pétri avec les tranches dans l’incubateur de culture de tranche. Maintenir les tranches à 37 °C dans une atmosphère humidifiée contenant 40 % d’O2 / 5 % de CO2 / 55 % N2 jusqu’à utilisation.
4. Microinjection
- Allumez l’ordinateur, le microscope, la caméra microscope, les manipulateurs, la plate-forme de pression et le capteur de pression. Chargez l’application en cliquant sur le fichier "launchapp.py" dans le dossier principal téléchargé à partir de GitHub et spécifiez les paramètres de l’appareil dans l’écran popup (voir l’étape 1.1 pour les instructions d’installation).
- Créez une pression extérieure pour éviter l’engorgement indésirable avant d’entasser la pipette dans la solution. Pour appliquer une pression sur la pipette, faites glisser la barre de pression de compensation à 24 – 45 % et cliquez sur Valeurs définies. Ensuite, réglez la pression à une pression suffisante en tournant le bouton de soupape de pression mécanique à 1 - 2 PSI (69 - 138 mbar) comme indiqué par le capteur de pression.
- Transférer les tranches dans une boîte de Pétri de 3,5 cm contenant 2 mL de CIMM préchauffé. Placer les tranches à microinjecter au centre de la boîte de Pétri. Transférer la boîte de Pétri au stade de microinjection préchauffé (37 °C).
- Chargez la pipette de microinjection avec 1,4 à 1,6 μL de solution microinjectée (à partir de l’étape 3.3) à l’aide d’une pipette en plastique à longue pointe. Insérez la pipette de microinjection sur le porte-pipette.
- À l’aide du grossissement le plus bas au microscope, mettre la tranche au point et guider la micropipette vers ce champ de vision (FOV) afin qu’elle soit concentrée sur le même plan que la cible de tranche. Passez la sortie du microscope à la caméra pour voir le FOV dans l’application.
- Cliquez sur le bouton grossissement en haut à gauche de l’interface pour lancer l’étalonnage de l’appareil. Une fenêtre invitera à sélectionner le grossissement. Sélectionnez le grossissement 10x, ou tout grossissement de l’objectif est réglé à (par exemple, 4x, 10x, 20x, 40x) et appuyez sur Ok. Le logiciel suppose que l’objectif interne est 10x (le grossissement objectif le plus commun lentille).
- Recentrer la pointe pipette à l’aide de la roue micrométrique du microscope et cliquez sur la pointe pipette avec le curseur. Ensuite, appuyez sur le bouton étape 1.1 et appuyez sur OK dans la fenêtre popup. La pipette se déplacera dans la direction Y. Cliquez sur le bout de la pipette et appuyez sur le bouton étape 1.2. Enfin, entrez 45 dans la boîte d’angle Pipette et appuyez sur l’angle set.
- Entrez les paramètres souhaités dans le panneau automatisé de contrôle de microinjection. Pour la microinjection dans les progéniteurs apiques définir la distance d’injection à 20 - 40 μm et la profondeur à 10 - 15 μm. Pour la microinjection dans les neurones définir la distance d’injection 30 - 40 μm du côté basal, et la profondeur à 10 - 30 μm selon ce qui est ciblé. Réglez toujours la vitesse à 100%. Cliquez sur Définir les valeurs.
REMARQUE : La distance d’approche est la distance que la pipette tire hors du tissu avant de passer à la distance d’injection suivante, la profondeur est la profondeur dans le tissu que la microinjection va, l’espacement est la distance le long de la ligne entre les injections séquentielles, la vitesse est la vitesse de la pipette en μm/s. - Cliquez sur le bouton Dessiner le bord et faites glisser le curseur le long de la trajectoire désirée dans la fenêtre popup pour définir la trajectoire de l’injection. Pour les cellules souches progénitrices microinjectrices, le côté ventral de la surface du télencéphale est ciblé, comme le montre la figure 2A. Apportez la pipette au début de la ligne et cliquez sur le bout de la pipette. Cliquez sur Exécuter la trajectoire pour commencer le microinjection. Répétez cette étape pour chaque plan d’injection ciblé (généralement fait pour 3 - 4 avions avec 40 - 75 injections par avion).
5. Culture tissulaire et traitement des tranches tissulaires pour l’immunofluorescence
- Préparer le mélange de collagène (1,5 mg/mL) : À un tube ajouter 1,25 mL de la solution matricielle, 0,5 mL d’eau distillée, 0,5 mL de solution 5x DMEM-F12 et 0,25 mL de tampon de reconstitution. Gardez-le sur la glace jusqu’à utilisation.
- Obtenir la boîte de Pétri contenant les tranches microinjectées de la chambre d’incubation de la culture des tranches et plonger les tranches dans le mélange de collagène.
- Transférer les tranches avec 200 à 300 μL de mélange de collagène dans un puits de 14 mm d’un plat à fond de verre de 35 mm. Assurez-vous que les tranches sont recouvertes de très moins de collagène. Cette mise en place permet les conditions optimales pour les nutriments et l’absorption d’oxygène.
- Orientez les tranches tout en veillant à ce qu’il y ait suffisamment d’espace entre les tranches à l’aide de deux paires de forceps. Incuber la boîte de Pétri pendant 5 min à 37 °C à l’aide d’un bloc chauffant pour permettre au collagène de se solidifier. Considérez cette fois comme t = 0 de culture de tranche.
- Déplacez la boîte de Pétri vers l’incubateur de culture de tranche pour 40 min supplémentaires. Ajouter ensuite 2 mL de SCM préchauré. Les tranches sont conservées en culture jusqu’au moment désiré.
- Sortez les tranches de l’incubateur de culture en tranches et aspirez le SCM. Laver les tranches encastrées de collagène avec 1x PBS. Ajouter 4 % (wt/vol) de paraformaldéhyde (dans un tampon phosphaté de 120 mM, pH 7,4) et laisser le tissu à RT pendant 30 min. Déplacez-le ensuite à 4 °C pour permettre une fixation pendant la nuit.
- Aspirez la solution de paraformaldéhyde le lendemain et effectuez des lavages 1x PBS. Pour enlever les tranches du collagène, utilisez deux paires de forceps pour extraire délicatement les tranches sous un stéréomicroscope.
- Utilisez un four à micro-ondes pour faire fondre l’agarose à faible point de fusion de 3 % (wt/vol) pour le traitement des tranches microinjectées. Verser l’agarose fondue dans un moule d’intégration jetable et laisser refroidir à environ 38 - 39 °C.
- Transférer les tranches de tissu de l’étape 5.7 dans ce moule contenant une agarose à faible fusion à l’aide d’une pipette Pasteur en plastique. Assurez-vous que le côté pial de la tranche est en place et que la surface ventriculaire est en baisse. Si nécessaire orienter en conséquence. Laissez l’agarose refroidir à RT pour solidifier.
- Couper l’agarose supplémentaire entourant les tranches. Orientez le bloc d’agarose pour s’assurer que la surface coupée est parallèle à la lame de coupe du vibratome. À l’aide d’un vibratome, couper 50 sections de μm d’épaisseur.
- Remplir un plat de 24 puits avec 1x PBS. Transférer les sections dans ce plat à l’aide d’un pinceau à pointe fine. Effectuez l’immunofluorescence selon les protocoles standard.
La microinjection a pour but de suivre et de manipuler les cellules souches neurales individuelles et leur descendance dans les tissus vivants et de suivre leur progression de lignée dans un environnement physiologique. Dans cet article, nous avons démontré l’utilisation du robot de microinjection pour cibler et microinjecter automatiquement les tranches organotypiques du télescopique de souris. La figure 2 illustre des images représentatives de cellules progénitrices injectées avec succès et la figure 3 illustre les neurones nouveau-nés injectés. Lorsqu’on leur injecte du colorant Dextran Alexa-488 (ou Alexa-A555), les cellules semblent complètement remplies de colorant. En ce qui concerne les progéniteurs apiques (Figure 2) l’imagerie confocale permet de reconstruire avec haute résolution spatiale la morphologie cellulaire, la présence - ou l’absence - de l’attachement apical et basal, et de combiner l’enquête morphologique avec l’expression marqueur. En combinant ces critères, l’utilisateur peut attribuer un sort cellulaire spécifique aux cellules microinjectées et à leur progéniture. Quant à l’injection de neurones, l’utilisateur peut reconstruire la morphologie neuronale, y compris la structure et les caractéristiques du dendrite apical et de l’axone. La microinjection automatisée peut fournir un débit significativement plus élevé par rapport à la microinjection manuelle( figure 2B). De plus, l’étiquetage EdU confirme que la viabilité cellulaire n’est pas affectée par l’automatisation( figure 2C). Garder la tranche organotypique en culture permet de suivre la progression de la lignée des cellules microinjectées (nous avons montré 4 - 24h dans la figure 2D). Si la solution de microinjection contient du matériel génétique (ADN, ARNm, guides CRISPR-Cas9) ou des protéines recombinantes, cela permet d’étudier si et comment la progression de la lignée est affectée par la manipulation.
La microinjection en cellules souches neurales individuelles dans les tissus fournit une excellente résolution cellulaire unique et pour cette raison, elle a été utilisée pour disséquer la biologie cellulaire de la progression des cellules souches neurales et de la transition du destin (Figure 3A). La microinjection permet la livraison d’un mélange complexe de produits chimiques. Nous avons précédemment fait usage de cette fonctionnalité pour étudier le couplage jonctionnel dans les cellules progénitrices neurales en mélangeant perméable gap-junctional avec des colorants fluorescents imperméables jonctionnels12. Nous avons prolongé les travaux antérieurs en étudiant le couplage jonctionnel dans les neurones nouveau-nés, en injectant Lucifer Yellow avec Dextran-A555 (Figure 3B). Comme le montre la figure 3B, une proportion de neurones pyramidaux nouveau-nés sont couplés par des jonctions d’écart aux neurones voisins. Cette observation est compatible avec l’idée que les neurones immatures communiquent par gap-junction13,14. En outre, le ciblage des neurones montre que l’utilisation du robot de microinjection peut être généralisée à plusieurs types de cellules dans le cerveau mammifère en développement. Cette configuration expérimentale sera utile pour disséquer la biologie cellulaire des neurones dans les tissus, par exemple en fournissant des oligopéptides spécifiques pour interférer avec les interactions protéines-protéines.
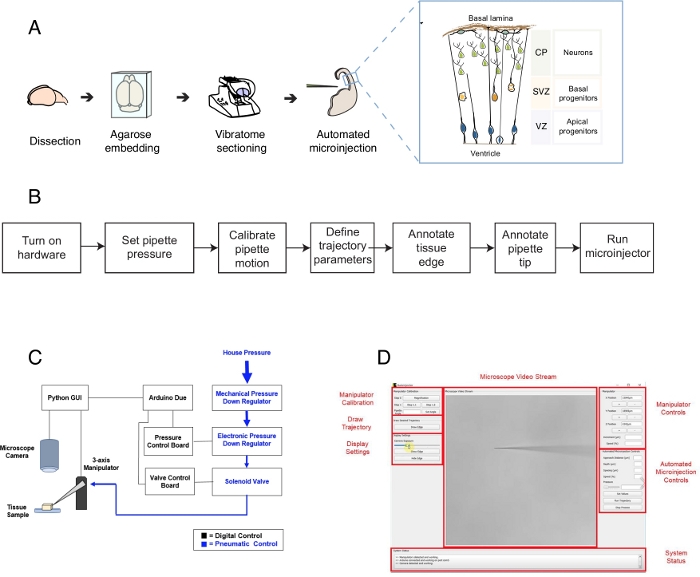
Figure 1 : Configuration et protocole automatisés de microinjection. (A) Protocole global pour la préparation des tissus et les microinjections automatisées à l’aide du robot de microinjection. Inset droit : Schéma de dessin animé de la souris Telencephalon visé pour la microinjection dans ce protocole. (B) Flowchart des étapes automatisées de microinjection. (C) Schéma du matériel robotisé de microinjection. (D) Interface utilisateur graphique (GUI) du logiciel utilisé pour contrôler et faire fonctionner le robot de microinjection. Ce chiffre est adapté de l’arbitre12. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
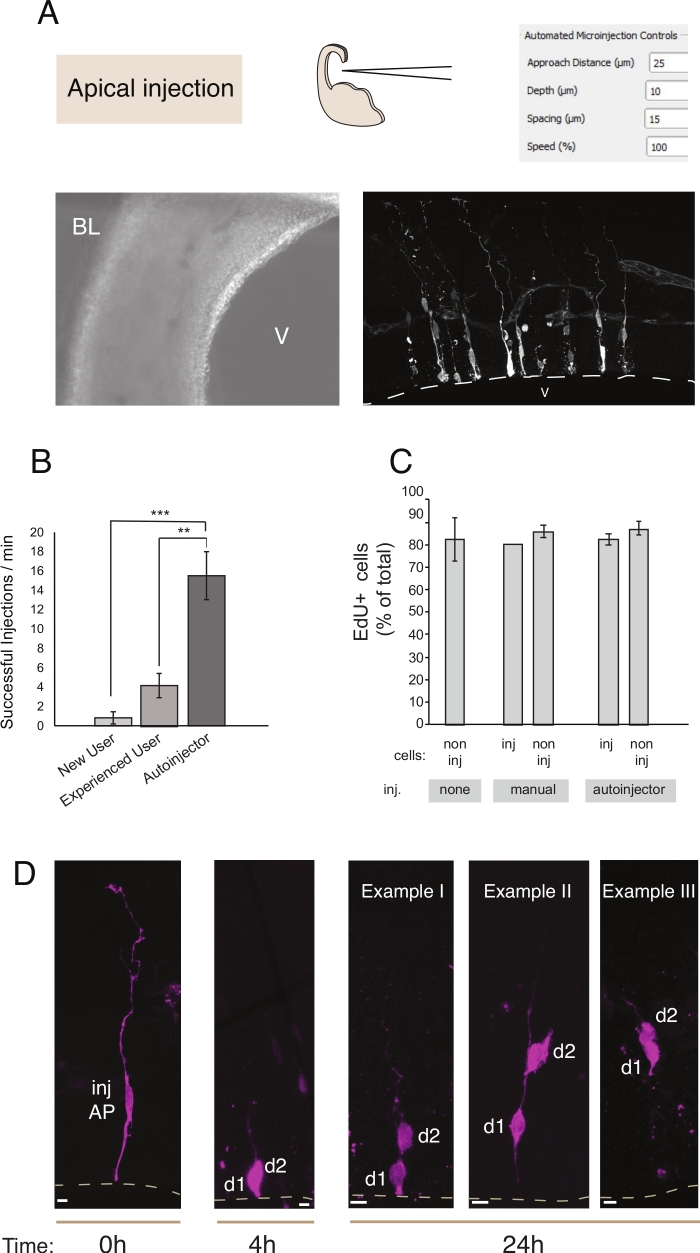
Figure 2 : Microinjection robotique en progéniteurs apiques. Résultats schématiques et attendus lors de l’utilisation du robot de microinjection pour cibler les progéniteurs apiques (AP) via la surface apical (injection apical). (A) Rangée supérieure. A gauche : schéma du processus. Sur la droite: GUI avec des paramètres pertinents pour l’injection apical. Rangée du bas. Sur la gauche : image de contraste de phase prise pendant la procédure d’injection (V : ventricule ; BL: lamina basale). À droite : résultats représentatifs montrant des AP microinjectés. La ligne pointillée représente le ventricule (V). Barre d’échelle : 10 μm. (B) Injections réussies par minute pour un utilisateur novice sur le système manuel de microinjection, un utilisateur expérimenté sur le système manuel de microinjection, et le robot de microinjection. (C) Incorporation d’EdU dans les cellules microinjectées et dans les cellules non injectées dans la zone injectée. Les tranches organotypiques de souris E14,5 télencéphale dorsale ont été soit (i) non injectées, soit (ii) soumises à une microinjection manuelle ou automatisée (tranche injectée) à l’aide de Dextran-A488 (pour l’injection manuelle et automatique). Les tranches ont été conservées en culture en présence d’EdU pendant 24 h, puis elles ont été fixées et tachées pour le DAPI et l’EdU. Des cellules injectées et non injectées dans la zone injectée ont été notées pour la positivité d’EdU. (D) Utilisation du robot de microinjection Lineage traçage. Un colorant fluorescent (Dx3-A555, magenta) est injecté dans une seule cellule souche neurale (t = 0 h). Le colorant fluorescent est divisé en cellules fille (d1, d2) pendant la mitose. Cela permet de suivre la descendance de la cellule injectée (t = 4 h et 24 h) et de révéler la progression de la lignée au fil du temps. Pour t = 24 h, nous montrons plusieurs exemples de la descendance que l’on s’attend à trouver. Barres d’échelle : 10 μm. Les graphiques en B et C sont pris à partir de l’arbitre12Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
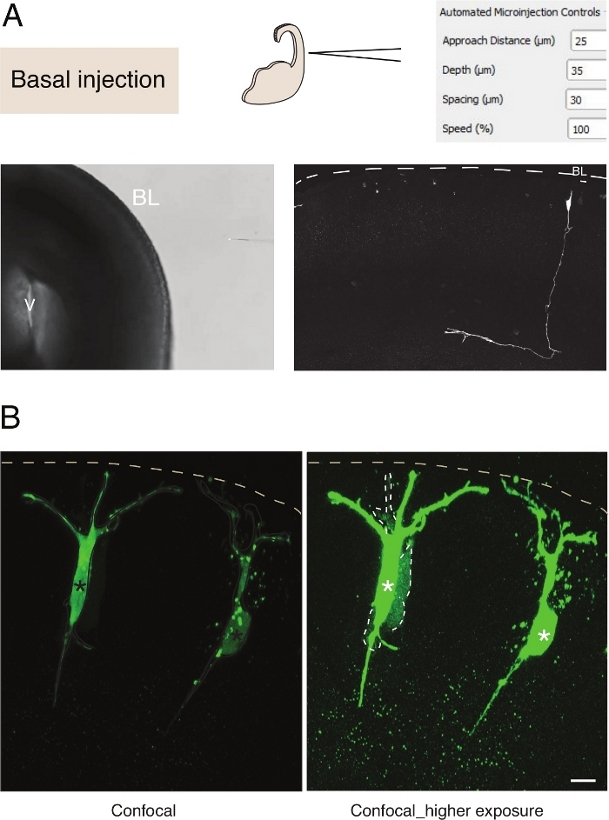
Figure 3 : Microinjection robotique dans les neurones. Résultats schématiques et attendus lors de l’utilisation du robot de microinjection pour cibler les neurones pyramidaux (N) via la surface basale (injection basale). (A) Rangée supérieure. A gauche : schéma du processus. Sur la droite: GUI avec des paramètres pertinents pour l’injection basale. Rangée du bas. Sur la gauche : image de contraste de phase prise pendant la procédure d’injection (V : ventricule ; BL: lamina basale). À droite : les résultats représentatifs montrant une ligne microinjectée N. Dashed représentent le lamina basal (BL). Barre d’échelle : 10 μm. (B) Utilisation de l’autoinjecteur pour étudier la communication jonctionnelle d’écart dans les tissus. Les neurones pyramidaux ont été injectés avec une solution contenant deux teintes: l’écart junctional-imperméable Dx-A555 (non montré) et l’écart-jonction perméable Lucifer Jaune (vert). Le Dx-A555 est confiné à la cellule ciblée (astérisques), tandis que le LY se diffuse aux cellules qui sont reliées par jonction d’écart à la cellule ciblée (lignes pointillées). Panneau gauche : Faible exposition, seules les cellules microinjectées sont visibles. Panneau droit : Une exposition élevée permet la visualisation des cellules injectées ainsi que des cellules couplées (lignes pointillées). Barre d’échelle : 10 μm. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Fichier supplémentaire : Dépannage de plusieurs erreurs courantes qui surviennent lors de la microinjection. S’il vous plaît cliquez ici pour télécharger ce fichier.
La microinjection en cellules souches neurales individuelles dans les tissus fournit une excellente résolution cellulaire unique et pour cetteraison,elle a été utilisée pour disséquer la biologie cellulaire de la progression des cellules souches neurales et de la transition du destin (figure 2;voir aussi 10,11,12). La procédure automatisée de microinjection peut être effectuée sur d’autres types de cellules chez les souris embryonnaires et les tissus cérébraux humains. Les résultats représentatifs de la microinjection des neurones nouveau-nés en ciblant la surface basale du télencéphale sont indiqués dans la figure 3.
Le principe établi ici peut être appliqué pour cibler plusieurs types de cellules différentes dans les cerveaux embryonnaires de souris et les cerveaux humains. Nous avons déjà montré que le robot de microinjection peut également être utilisé pour cibler les cellules progénitrices simples dans l’arrière-cerveau de la souris et les neurones télencéphales et nouveau-nés chez la souris et l’homme en développement néocortex12. Pour obtenir les meilleurs résultats de la procédure d’injection, il faut optimiser toutes les étapes avant de commencer l’injection. Il est important d’examiner et d’optimiser soigneusement la préparation de tranches de tissu organotypique viables et bien conservées à partir de tissus cérébraux (figure 1). Il est crucial d’être rapide dans la procédure de dissection et de tranchage illustrée à la figure 1. Pour l’injection apical ciblant les AP, il faut choisir les tranches montrant l’orientation idéale de la surface apical. Pour l’injection d’APs, l’orientation idéale est la surface apical perpendiculaire au fond de la boîte de Pétri. Toute autre orientation sera permissive ainsi, cependant, la surface apical perpendiculaire à la boîte de Petri fournit une surface plus large pour l’injection, augmentant ainsi le succès de l’injection. Pour l’injection dans les neurones, l’orientation de la tranche joue peu ou pas d’effet.
Une fois que les tranches à injecter sont sélectionnées, la procédure d’injection par tranche prend environ 5 minutes. Considérant que l’on travaille avec des tissus vivants, il est fortement recommandé d’accélérer la procédure d’injection. À cette fin, nous vous recommandons de définir tous les paramètres d’injection via l’interface graphique (Figure 1D) avant que le tissu est prêt, afin de réduire tout temps d’attente inutile. Pour le dépannage s’il vous plaît se référer au fichier supplémentaire.
En cas de culture de tranche à long terme, les étapes après la procédure automatisée de microinjection peuvent affecter la santé des cellules et donc l’expérience. Par conséquent, il est fortement recommandé d’exécuter un test de contrôle de la qualité et d’optimiser les conditions de culture des tranches. Pour évaluer la viabilité cellulaire après la procédure de tranchage et d’injection, nous avons effectué l’étiquetage EdU pendant la culture et nous avons quantifié le nombre de noyaux pyknotiques (un proxy pour les cellules apoptotiques) dans les cultures et les tissus injectés12. Ces quantifications n’ont révélé aucun impact significatif de la microinjection sur la viabilité des tissus (figure 2C). Nous recommandons d’exécuter des contrôles de qualité similaires tout en établissant le pipeline de tranchage et de microinjection des tissus organotypiques en laboratoire.
Par rapport à la microinjection manuelle, le robot de microinjection offre plusieurs avantages. Tout d’abord, la courbe d’apprentissage pour l’utilisateur est moins raide par rapport à l’injection manuelle: un nouvel utilisateur atteindra une grande compétence après un nombre limité de sessions, généralement 1 ou 2. Deuxièmement, dans le cas de la microinjection manuelle, une compétence comparable nécessite des mois de formation. La procédure d’injection est plus rapide et plus efficace( Figure 2B). Nous avons quantifié ces paramètres et constaté que le robot de microinjection surpassait un utilisateur manuel qualifié en ce qui concerne le succès de l’injection (% de l’injection réussie / nombre total d’injections) et dans le nombre total d’injections par temps unitaire12. Il en résulte une augmentation globale de 300 % de l’efficacité de l’injection (% de l’injection réussie/min) pour le robot de microinjection par rapport à un utilisateur qualifié. L’augmentation de l’efficacité a été encore plus prononcée lors de la comparaison du robot de microinjection avec un utilisateur débutant et a atteint 700%. Last but not the least, le robot de microinjection peut être facilement programmé pour explorer systématiquement tous les paramètres spatiaux. Ceci est particulièrement avantageux lors de l’adaptation du robot de microinjection pour cibler de nouvelles cellules ou tissus, ou lors de l’utilisation du robot de microinjection à des fins nécessitant une résolution spatiale différente.
La construction du robot de microinjection exige des changements minimaux à un microscope existant d’épi-fluorescence12. Nous avons déjà fourni des instructions pour cette adaptation à https://github.com/bsbrl/Autoinjector. Une fois que le matériel est configuré, ce protocole fournit des détails méthodologiques clés pour entreprendre avec succès des microinjections automatisées. Dans l’ensemble, le robot de microinjection a un taux d’injection réussi de 15,52 + 2,48 injections/min, ce qui est 15 fois plus élevé qu’un utilisateur inexpérimenté (1,09 ± 0,67 injections/min), et 3 fois plus grand qu’un utilisateur expert (4,95 ± 1,05 injections/min)12. Cette amélioration du taux d’injection réussie permet aux utilisateurs novices et experts d’injecter plus de cellules en moins de temps, ce qui est essentiel pour préserver la viabilité des tissus. En outre, le robot de microinjection est personnalisable et la trajectoire, la profondeur de l’injection, le nombre d’injections, l’espacement entre les injections peuvent tous être réglés à l’aide de l’interface graphique. Ces caractéristiques permettent au robot de microinjection d’être utilisé comme un outil pour optimiser les expériences auparavant laborieuses, et d’explorer des expériences fondamentalement nouvelles qui nécessitent un rendement plus élevé que possible auparavant.
Les principales limitations de la procédure de microinjection que nous avons décrite ici sont liées à la préparation des tranches de tissu, une étape cruciale qui nécessite une optimisation étendue. En outre, la microinjection repose sur la présence d’une surface qui peut être approchée par la pipette en verre. Cette fonctionnalité limite le type de tissus et d’emplacements tissulaires qui peuvent être ciblés par microinjection à l’aide de la configuration actuelle.
Le robot de microinjection utilise actuellement l’imagerie brightfield et a été utilisé dans les préparations in vitro de tranches de cerveau. À l’avenir, le robot de microinjection pourrait être combiné avec l’imagerie à 2 photons pour augmenter la spécificité du ciblage cellulaire unique in vivo pour le marquage moléculaire ou colorant. De tels efforts ont déjà été faits pour l’électrophysiologie à celluleunique 15,16. L’appareil actuel nécessite une observation manuelle de la procédure de microinjection. Les versions futures pourraient inclure des stratégies de nettoyage des pipettes de microinjectionobstruées 17 ou l’intégration de robots de manutentiondes fluides 18 pour les microinjections multiplexées et entièrement autonomes. Ces dispositifs pourraient augmenter l’échelle de microinjection par ordre de grandeur. L’adaptation d’algorithmes pour le contrôle parallèle de plusieurs pipettes de microinjection19 pourrait permettre la livraison multiplexée de dizaines de teintures et de réachemins moléculaires dans les mêmes cellules dans les mêmes expériences. Cela a le potentiel d’ouvrir de nouvelles voies pour le dépistage moléculaire dans les tissus.
Le robot de microinjection pourrait être utilisé pour étiqueter les cellules fonctionnellement identifiées à l’aide de codes à barres d’ADN ou d’ARN. Ceci pourrait à son tour être combiné avec d’autres techniques d’analyse de cellules simples, telles que le séquençage d’ARN à cellule unique (scRNAseq) et la microscopie électronique. Nos résultats préliminaires montrent que les cellules microinjectées et leur progéniture peuvent être récupérées et isolées à l’aide de la dissociation tissulaire suivie d’un tri FACS (Taverna, résultats non publiés). Les cellules triées FACS peuvent ensuite être utilisées pour scRNAseq. En outre, les résultats préliminaires montrent que les capacités de résolution d’une seule cellule du robot de microinjection peuvent être utilisées en combinaison avec l’analyse microscopique électronique pour explorer la biologie cellulaire sur les cellules souches neurales dans les tissus à haute résolution spatiale (Taverna et Wilsch-Bräuninger, résultats non publiés). Ces données suggèrent que le robot de microinjection peut être employé comme outil pour la lumière corrélative et la microscopie électronique dans le tissu et dans le sens plus large, pour l’analyse multimodale de l’identité et du comportement cellulaires dans le tissu.
La microinjection repose sur l’utilisation de la pression et on peut se permettre d’injecter des solutions à haute complexité moléculaire (p. ex., un transcriptome entier). Cette caractéristique de microinjection a été exploitée dans le passé pour isoler et clonage des récepteurs à portail ligand20. Le long de cette ligne, le robot de microinjection pourrait être utilisé pour la modélisation et l’étude des traits multigéniques au niveau cellulaire. Combiné à une stratégie de sous-mise en commun, le robot de microinjection pourrait également être utilisé comme plate-forme pour identifier l’ensemble minimum de gènes conduisant un certain comportement trait/cellulaire. Jusqu’à présent, le robot de microinjection a été utilisé pour manipuler la biochimie de la cellule par la livraison de l’ARNm, de l’ADN ou des protéines recombinantes10,21,22. Nous prévoyons une application du robot de microinjection dans l’exploration de la biophysique de l’espace intracellulaire, par exemple en fournissant des nanomatériaux ou des nanomachines qui permettent la détection et/ou la manipulation des propriétés biophysiques de l’espace intracellulaire.
Les auteurs n’ont rien à divulguer.
Les auteurs aimeraient remercier la Fondation Nomis (ET). SBK reconnaît les fonds du département de génie mécanique, College of Science and Engineering, MnDRIVE RSAM initiative de l’Université du Minnesota, Minnesota département de l’enseignement supérieur, National Institutes of Health (NIH) 1R21NS103098-01, 1R01NS111028, 1R34NS11654, 1R21NS112886 et 1R21 NS111196. GS a reçu l’appui de la National Science Foundation Graduate Research Fellowship et de la subvention de formation du NSF IGERT.
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose, Low Melt | Carl Roth | Cat# 6351.2 | |
| Agarose, Wild Range | Sigma-Aldrich | Cat# A2790 | |
| Best-CA 221 Glue | Best Klebstoffe GmbH & Co.KG | Cat# CA221-10ml | |
| B-27 Supplement | Thermo Fisher Scientific | Cat# 17504044 | |
| Cellmatrix Type-IA (Collagen, Type !) | FUJIFILM Wako Chemicals | Cat# 637-00653 | |
| Distilled Water | |||
| DMEM-F12, CO2 independent (w/o Phenol red) | Sigma-Aldrich | Cat# D2906 | |
| DMEM-F12, CO2 independent (with Phenol red) | Sigma-Aldrich | Cat# D8900 | |
| HEPES-NAOH, pH 7.2, 1M (HEPES buffer) | Carl Roth | Cat# 9105.3 | |
| L-Glutamine, 200 mM | Thermo Fisher Sientific | Cat# 25030024 | |
| Mowiol 4-88 | Sigma-Aldrich | Cat# 81381 | |
| N-2 Supplement | Thermo Fisher Scientific | Cat# 17502048 | |
| Neurobasal Medium | Thermo Fisher Scientific | Cat# 21103049 | |
| Nuclease-free water | Thermo Fisher Scientific | Cat# AM9937 | |
| O2 (40%), CO2 (5%), N2 (55%) Mix, 50 liters | |||
| Paraformaldehyde | Merck | Cat# 818715 | |
| PBS | |||
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | Cat# 15140122 | |
| Rat serum | Charles River Laboratories | ||
| Japan | |||
| Sodium bicarbonate (NaHCO3) | Merck | Cat# 106323 | |
| Sodium hydroxide (NaOH) | Merck | Cat# 106482 | |
| Tyrode’s salt | Sigma | Cat# T2145-10x1L) | |
| Equipment | |||
| Borosilicate glass capillaries, 1.2 mm outer diameter x 0.94 mm inner diameter | Sutter Instruments | Cat# BF-120-94-10 | |
| Bottle-top filter system, 500 mL | Corning | Cat# 430769 | |
| Computer PC | |||
| Custom pressure rig | Custom pressure rig | ||
| Electronic pressure regulator | Parker Hannifin | Cat# 990-005101-002 | |
| Falcon tubes, 15 mL | Corning | Cat# 430791 | |
| Falcon tubes, 50 mL | Corning | Cat# 430829 | |
| Fine-tip paintbrush | |||
| Flaming/ Brown micropipette puller | Sutter Instruments | Cat# P-97 | |
| Forceps, Dumont no. 3 | Fine Science Tools | Cat# 11231-30 | |
| Forceps, Dumont no. 5 | Fine Science Tools | Cat# 11255-20 | |
| Forceps, Dumont no. 55 | Fine Science Tools | Cat# 11252-20 | |
| Heating block | Labtech International | Cat # Dri block Digi2 | |
| Inverted fluorescence microscope | Zeiss | Cat# Axiovert 200 | |
| Light source | Olympus | Cat# Highlight 3100 | |
| Manual pressure regulator | McMaster Carr | Cat# 0-60 PSI 41795K3 | |
| Microloader Tips | Eppendorf | Cat# 5242956.003 | |
| Microcontroller | Arduino | Cat# Arduino Due | |
| Microscope camera Hamamatsu Orca Flash 4.0 V3 | |||
| Motorized stage XY for microscope | |||
| Multiwell plate, 24 wells | Nunc | Cat# 142475 | |
| Pasteur pipettes, plastic | |||
| Petri dish, 60 x 15 mm | Greiner | Cat# 628102 | |
| Petri dish, 35 x 10 mm | Nunc | Cat# 153066 | |
| Petri dish, 34 x 14 mm, including Microwell no. 1.5 cover glass | MatTek | Cat# P35G-1.5-14-C | |
| Pipette holder | Warner Instruments | Cat# 64-2354 MP-s12u | |
| Pipette and tips | |||
| Puller filament, 3.0-mm square box filament | Sutter Instrument | Cat# FB330B | |
| Slice culture incubation box | MPI-CBG | Cat# custom made | |
| Solenoid valve | Cat# LHDA053321H-A | ||
| Stereomicroscope | Olympus | Cat# SZX12 | |
| Tabletop centrifuge | Heraeus | Cat# 5431622 | |
| Thermometer | |||
| Three-axis Manipulator | Sensapex Inc | Cat# tree-axis uMP | |
| Vibratome | Leica | Cat# VT1000s | |
| Whole-embryo-culture-system incubator | Ikemoto Company | Cat# RKI-10-0310 | |
| Waterbath | |||
| Software and Algorithms | |||
| Arduino | Arduino | ||
| Fiji | RRID: SCR_002285 | ||
| Python | Python Software foundation | Python 2.7.12 | |
| ZEN | RRID: SCR_013672 |
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Götz, M., Huttner, W. B. The cell biology of neurogenesis. Nature Reviews Molecular Cell Biology. 6 (10), 777-788 (2005).
- Di Lullo, E., Kriegstein, A. R. The use of brain organoids to investigate neural development and disease. Nature Reviews Neuroscience. 18 (10), 573-584 (2017).
- Lancaster, M. A., Knoblich, J. A. Organogenesisin a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Kretzschmar, K., Clevers, H. Organoids: Modeling Development and the Stem Cell Niche in a Dish. Developmental Cell. 38 (6), 590-600 (2016).
- Pepperkok, R. et al. Automatic microinjection system facilitates detection of growth inhibitory mRNA. Proceedings of the National Academy of Sciences of the United States of America. 85 (18), 6748-6752 (1988).
- Pepperkok, R., Lowe, M., Burke, B., Kreis, T. E. Three distinct steps in transport of vesicular stomatitis virus glycoprotein from the ER to the cell surface in vivo with differential sensitivities to GTPγS. Journal of Cell Science. 111 (13), 1877-1888 (1998).
- Pepperkok, R. et al. β-COP is essential for biosynthetic membrane transport from the endoplasmic reticulum to the Golgi complex in vivo. Cell. 74 (1), 71-82 (1993).
- Ansorge, W., Pepperkok, R. Performance of an automated system for capillary microinjection into living cells. Journal of Biochemical and Biophysical Methods. 16 (4), 283-292 (1988).
- Taverna, E., Haffner, C., Pepperkok, R., Huttner, W. B. A new approach to manipulate the fate of single neural stem cells in tissue. Nature Neuroscience. 15 (2), 329-337 (2012).
- Wong, F. K., Haffner, C., Huttner, W. B., Taverna, E. Microinjection of membrane-impermeable molecules into single neural stem cells in brain tissue. Nature Protocols. 9 (5), 1170-1182 (2014).
- Shull, G., Haffner, C., Huttner, W. B., Kodandaramaiah, S. B., Taverna, E. Robotic platform for microinjection into single cells in brain tissue. EMBO Reports. 20 (10), e47880 (2019).
- Jabeen, S., Thirumalai, V. The interplay between electrical and chemical synaptogenesis. Journal of Neurophysiology. 120 (4), 1914-1922 (2018).
- Nagy, J. I., Pereda, A. E., Rash, J. E. Electrical synapses in mammalian CNS: Past eras, present focus and future directions. Biochimica et Biophysica Acta - Biomembranes. 1860 (1), 102-123 (2018).
- Suk, H.J. et al. Closed-loop real-time imaging enables fully automated cell-targeted patch-clamp neural recording in vivo. Neuron. 95 (5), 1037-1047 (2017).
- Annecchino, L. A. et al. Robotic automation of in vivo two-photon targeted whole-cell patch-clamp electrophysiology. Neuron. 95 (5), 1048-1055 (2017).
- Kolb, I. et al. Cleaning patch-clamp pipettes for immediate reuse. Scientific Reports. 6, 35001 (2016).
- Holst, G. L. et al. Autonomous patch-clamp robot for functional characterization of neurons in vivo: development and application to mouse visual cortex. Journal of Neurophysiology. 121 (6), 2341-2357 (2019).
- Kodandaramaiah, S. B. et al. Multi-neuron intracellular recording 1 in vivo via interacting autopatching 2 robots. ELife. 7, 24656 (2018).
- Lubbert, H. et al. cDNA cloning of a serotonin 5-HT1c receptor by electrophysiological assays of mRNA-injected Xenopus oocytes (RNA fractionation/hybrid depletion/hybrid selection/choroid plexus/voltage clamp). Neurobiology. 84 (2) 4332-4336 (1987).
- Florio, M. et al. Human-specific gene ARHGAP11B promotes basal progenitor amplification and neocortex expansion. Science. 347 (6229), 1465-1470 (2015).
- Kalebic, N. et al. CRISPR/Cas9-induced disruption of gene expression in mouse embryonic brain and single neural stem cells in vivo. EMBO Reports. 17 (3), 338-348 (2016).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved