Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Bioengineering
Manipulation von einzelnen neuronalen Stammzellen und Neuronen in Gehirnscheiben mit Robotik-Mikroinjektion
Dieses Protokoll demonstriert den Einsatz einer Roboterplattform für die Mikroinjektion in einzelne neuronale Stammzellen und Neuronen in Hirnscheiben. Diese Technik ist vielseitig und bietet eine Methode zur Verfolgung von Zellen im Gewebe mit hoher räumlicher Auflösung.
Eine zentrale Frage in der Entwicklungsneurobiologie ist, wie neuronale Stamm- und Vorläuferzellen das Gehirn bilden. Um diese Frage zu beantworten, muss man einzelne Zellen im Gehirngewebe mit hoher Auflösung im Laufe der Zeit beschriften, manipulieren und folgen. Diese Aufgabe ist aufgrund der Komplexität des Gewebes im Gehirn äußerst anspruchsvoll. Wir haben vor kurzem einen Roboter entwickelt, der eine Mikroinjektionsnadel in das Gehirngewebe führt, nachdem Bilder aus einem Mikroskop verwendet wurden, um Femtoliter-Lösungsvolumina in einzelne Zellen zu liefern. Die Roboteroperation erhöht sich zu einer Gesamtausbeute, die eine Größenordnung größer ist als die manuelle Mikroinjektion und eine präzise Etikettierung und flexible Manipulation einzelner Zellen im lebenden Gewebe ermöglicht. Damit kann man Hunderte von Zellen in eine einzige organotypische Scheibe mikroinjizieren. Dieser Artikel zeigt die Verwendung des Mikroinjektionsroboters für die automatisierte Mikroinjektion von neuronalen Vorläuferzellen und Neuronen in den Hirngewebescheiben. Im weiteren Sinne kann es auf jedem Epithelgewebe mit einer Oberfläche verwendet werden, die von der Pipette erreicht werden kann. Nach der Einrichtung kann der Mikroinjektionsroboter 15 oder mehr Mikroinjektionen pro Minute ausführen. Der Mikroinjektionsroboter wird aufgrund seines Durchsatzes und seiner Versalität die Mikroinjektion zu einer weitgehend unkomplizierten Hochleistungs-Zellmanipulationstechnik machen, die in Bioengineering, Biotechnologie und Biophysik zur Durchführung einzelliger Analysen in organotypischen Gehirnscheiben eingesetzt werden kann.
Dieses Protokoll beschreibt die Verwendung eines Roboters, um einzelne Zellen in Hirngewebescheiben zu zielen und zu manipulieren, wobei der Schwerpunkt insbesondere auf einzelnen neuronalen Stammzellen und Neuronen liegt. Der Roboter wurde entwickelt, um eine zentrale Frage in der Entwicklungsneurobiologie zu behandeln, das ist, wie neuronale Stamm- und Vorläuferzellen zur Hirnmorphogenesebeitragen 1,2,3,4,5. Um diese Frage zu beantworten, muss man einzelne neuronale Stammzellen beschriften und verfolgen und deren Abstammungsprogression im Laufe der Zeit verfolgen, um das Verhalten einzelner Zellen mit der Gewebemorphogenese zu korrelieren. Dies kann auf unterschiedliche Weise erreicht werden, z.B. durch Elektroporating von Hirngewebe in der Gebärmutter oder durch Kennzeichnung von Einzelzellen mit lipophilen Matrizen. Obwohl diese Methoden leistungsstark sind, fehlt es ihnen an präziser Einzelzellauflösung (Elektroporation) und/oder der Möglichkeit, den intrazellulären Raum (lipophiler Farbstoff) zu manipulieren. Mikroinjektion in einzelne Zellen wurde entwickelt, um diese Herausforderung zu überwinden6,7,8. Während der Mikroinjektion wird eine Pipette unter Druck, Femtoliter-Volumen von Reagenzien9. Wir haben zuvor ein manuelles Verfahren zur Mikroinjektion einzelner neuronaler Stammzellen in organotypisches Gewebe beschrieben (Abbildung 1A)10,11. Die Mikroinjektion in neuronale Stammzellen beruht auf der Verwendung einer Mikropipette, die in einzelne neuronale Stammzellen eingeführt wird, um eine Lösung zu injizieren, die einen Fluoreszenzfarbstoff enthält, zusammen mit anderen Molekülen von Interesse. Die selektive Ausrichtung neuronaler Stammzellen wird erreicht, indem das sich entwickelnde Telencephalon über die ventrikuläre Oberfläche (oder Ventrikel, siehe Karikatur in Abbildung 1A) nähert, die durch die apikale Plasmamembran von apikalen Vorläufern gebildet wird (Cartoon in Abbildung 1A). Dieser Prozess muss für jede Zelle wiederholt werden, die der Experimentator injizieren möchte. Darüber hinaus hängt der Erfolg der Mikroinjektion von der genauen Kontrolle der Tiefe und Dauer der Mikropipetteninjektion im Gewebe ab. Daher ist die manuelle Mikroinjektion trotz der einzigartigen Vorteile äußerst mühsam und erfordert eine beträchtliche Praxis, um bei angemessenem Durchsatz und Ausbeute zu arbeiten, was die Verwendung dieser Technik in skalierbarer Weise erschwert. Um diese Einschränkung zu überwinden, haben wir vor kurzem einen bildgesteuerten Roboter entwickelt, den Autoinjektor12 (oder Mikroinjektionsroboter), der automatisch Mikroinjektionen in einzelne Zellen durchführen kann.
Der Mikroinjektionsroboter nutzt mikroskopische Bildgebungs- und Computer-Vision-Algorithmen, um bestimmte Positionen im 3-D-Raum im Gewebe für die Mikroinjektion präzise anzusprechen (Abbildung 1B). Der Mikroinjektionsroboter kann durch relativ einfache Änderungen an einem vorhandenen Mikroinjektions-Setup konstruiert werden. Der Gesamtschema des Mikroinjektionsroboters ist in Abbildung 1Cdargestellt. Eine Pipette ist in einem Pipettenhalter montiert, der an einem dreiachsigen Manipulator befestigt ist. Eine Mikroskopkamera wird verwendet, um Bilder des Gewebes und der Mikroinjektionsnadel zu erfassen. Zur Steuerung des Drucks in der Pipette wird ein kundenspezifisches Druckregelungssystem verwendet und zur Steuerung der Position der Mikroinjektorpipette ein programmierbarer Mikromanipulator. Die Kamerabilder des Gewebes und der Mikroinjektionspipette werden verwendet, um die räumliche Position der Mikroinjektionspipettespitze und die Orte zu bestimmen, an denen Mikroinjektionen durchgeführt werden müssen. Die Software berechnet dann Diekbahnen, die benötigt werden, um die Pipette innerhalb des Gewebes zu bewegen. Die gesamte Hardware wird von der Software gesteuert, die wir zuvor entwickelt haben. Die gesamte Software ist in Codierungssprache (z.B. Python und Arduino) geschrieben und kann von https://github.com/bsbrl/Autoinjector mit Anweisungen heruntergeladen werden. Die grafische Benutzeroberfläche (GUI) ermöglicht es dem Benutzer, das Gewebe und die Mikropipette abzubilden und die Flugbahn der Mikroinjektion anzupassen. Unser System kann mit relativ einfachen Modifikationen an einem invertierten Mikroskop mit Hellfeld- und Epifluoreszenzfiltern aufgebaut werden.
Zuerst geben wir Anweisungen zur Vorbereitung von organotypischen Gewebescheiben des Gehirns für die Mikroinjektion. Dann zeigt das Protokoll den Start des Mikroinjektionsroboters, gefolgt von vorbereitenden Schritten, wie z. B. der Pipettenbewegungskalibrierung, die vor der Mikroinjektion durchgeführt werden müssen. Anschließend werden die Injektionsparameter definiert. Danach kann der Anwender die vom Mikroinjektionsroboter verwendete Flugbahn definieren und den Injektionsvorgang starten. Das mikroinjizierte Gewebe (in diesem Fall organotypische Gewebescheiben des Gehirns) kann je nach experimentellem Design10,11für unterschiedliche Zeiträume in Kultur gehalten werden. Das Gewebe kann verarbeitet werden, um die Identität und das Schicksal der injizierten Zellen und ihre Nachkommen zu verfolgen und zu untersuchen. Alternativ können die mikroinjizierten Zellen mittels Live-Bildgebung verfolgt werden. Im Rahmen dieses Protokolls demonstrieren wir den Einsatz des Roboters zur automatischen Mikroinjektion neuronaler Vorläuferzellen in organotypische Scheiben der Maus E14.5 dorsaltelencephalon. Der Roboter ist ferner in der Lage, Neugeborenen Neuronen im Maustelencephalon sowie in das menschliche fetale Telencephalon12zu mikroinziert zu haben.
Zusammenfassend beschreiben wir eine Roboterplattform, mit der einzelne Zellen im Gewebe verfolgt und manipuliert werden können. Die Plattform nutzt Druck und ist daher äußerst vielseitig in Bezug auf die chemische Natur der Verbindung zu injizieren. Darüber hinaus kann es an andere Zielzellen als Stammzellen angepasst werden. Wir erwarten, dass unser System auch an andere Modellsysteme leicht angepasst werden kann.
Alle Tierversuche wurden in Übereinstimmung mit dem deutschen Tierschutzrecht durchgeführt, und die erforderlichen Lizenzen wurden von der regionalen Ethikkommission für Tierversuche dresden (Tierversuchskommission, Landesdirektion Dresden) eingeholt. Organotypische Scheiben wurden aus E14.5 oder E16.5 C57BL/6 Maus embryonalem Telencephalon (Janvier Labs) hergestellt.
1. Installation von Software
- Befolgen Sie die Anweisungen, um die Software von https://github.com/bsbrl/Autoinjector zu installieren.
2. Herstellung von Reagenzien und Pipetten
- Agarose: Bereiten Sie 3% Agarose durch getrenntes Auflösen von 3 g Breitbereichs-Agarose und 3 g Niederschmelzpunkt-Agarose in 100 ml zellkulturtauglichen PBS in zwei separaten 200 ml Glasflaschen vor. Bei Raumtemperatur bis zu 3 Monate lagern.
- Tyrode-Lösung: 1 g Natriumbicarbonat und Tyrodes Salz (verwenden Sie den Inhalt der gesamten Flasche) und 13 ml 1 M HEPES in 1 L destilliertem Wasser auflösen. Stellen Sie den pH-Wert auf 7,4 ein. Filtern Sie die Lösung durch einen 0,2 m-Flaschen-Top-Filter.
- Slice Culture Medium (SCM): Fügen Sie 10 ml Rattenserum, 1 ml 2 mM Glutamin, 1 ml Penicillin-Streptomycin (100x), 1 ml N-2-Beilage (100x), 2 ml B27-Beilage (50x) und 1 ml HEPES (pH 7,3) Puffer in 84 ml Neurobasaler Medium hinzu. Aliquot 5 ml SCM in 15 ml Rohre. Bei -20 °C lagern.
- CO2-Unabhängiges Mikroinjektionsmedium (CIMM): Bereiten Sie 5x DMEM modifizierte Glukosearme Lösung (ohne Phenolrot) vor, indem Sie das Pulver in 200 ml destilliertem Wasser auflösen. Filterlösung durch einen 0,2 m Flaschen-Top-Filter (für das DMEM-Pulver den Inhalt der gesamten Flasche verwenden). Zur Vorbereitung von 100 ml CIMM 20 ml 5x DMEM modifizierte Lösung, 1 ml HEPES Puffer, 1 ml N2-Ergänzung (100x), 2 ml B27-Ergänzung (50x), 1 ml Penicillin-Streptomycin (100x), 1 ml N2-Ergänzung (100x), 2 ml B27-Ergänzung (50x), 1 ml Penicillin-Streptomycin (100x), 1 ml L von 2 ml Glutamin und 74 ml destilliertem Wasser mischen. Bewahren Sie die Lösung bei 4 °C auf.
- Rekonstitutionspuffer: Bereiten Sie den Rekonstitutionspuffer vor, indem Sie 262 mM NaHCO3, 0,05 N NaOH, 200 mM HEPES in destilliertem Wasser auflösen. Sterilisieren Sie die Lösung durch Filtration durch ein flaschentop0,22 m Filtersystem in eine sterile Glasflasche. Aliquot 500 l Rekonstitutionspuffer in luftdichte Mikrozentrifugenrohre. Bei 4 °C lagern.
- Mikroinjektionsfarbstoffbestand: Lösen Sie den fluoreszierend bezeichneten Dextran in RNase-freiem destilliertem Wasser (Endkonzentration 10 g/l). Bereiten Sie 5 L Aliquots vor und lagern Sie bei -20 °C bis zur Verwendung.
- Ziehen Sie die Mikroinjektionspipetten aus Borosilikatglaskapillaren (1,2 mm Außendurchmesser, 0,94 mm Innendurchmesser) mit dem Mikropipette-Zieher. Schützen Sie die Pipetten vor Staub. Pipettes nicht mehr als 2 – 3 Tage aufbewahren. Für dieses Experiment waren die Zugparameter HEAT: Rampentemperatur +1 – 5; ZUG: 100; VEL: 110; DEL: 100. HEAT und VEL sind die Parameter, die die Form und Größe der Pipette am meisten beeinflussen.
HINWEIS: Die optimale Mikroinjektionspipette hat eine lange und flexible Spitze, um Zellschäden während der Mikroinjektion zu vermeiden.
3. Gewebescheibenpräparation
- Schmelzen Sie die 3% breit reichende Agarose mit einer Mikrowelle vor der Hirngewebesektion. Lassen Sie die Agarose nicht verfestigen, indem Sie sie vor dem Einbetten bei 37 °C in einem Wasserbad halten. Stellen Sie sicher, dass die Pipetten vor Staub geschützt sind. Pipettes nicht mehr als 2 – 3 Tage aufbewahren.
- Mit einem Wasserbad ein Aliquot aus SCM und warme 10 – 12 ml CIMM und 20 ml Tyrodes Lösung auf 37 °C auftauen.
- Mischen Sie den fluoreszierenden Tracer (Dextran-3000 oder Dextran-10000-Alexa konjugiert; Endkonzentration 5 – 10 g/l) mit den anderen zu injizierenden Chemikalien. Zentrifugieren Sie die Mikroinjektionslösung bei 16.000 x g für 30 min bei 4 °C. Sammeln Sie den Überstand und übertragen Sie in eine neue Röhre. Halten Sie die Mikroinjektionslösung bis zur Anwendung auf Eis.
- Verwenden Sie die Köpfe von E13.5 – E16.5 Mausembryonen, um organotypische Gewebescheiben des Telencephalons zuzubereiten. Entfernen Sie die Haut und öffnen Sie den Schädel mit der Zange, entlang der Mittellinie bewegen. Sezieren Sie das embryonale Gehirn aus dem offenen Schädel und entfernen Sie die Hirnhirngewebe von der ventralen Seite des Gehirns. Lassen Sie das sezierte ganze Gehirn in Tyrodes Lösung auf einem 37 °C Heizblock.
HINWEIS: Alle in 3.4 beschriebenen Sezierschritte müssen in der vorgewärmten Tyrodes Lösung durchgeführt werden. - Gießen Sie die weiträumige geschmolzene Agarose in eine Einweg-Einbettform. Wenn die Agarose auf 38 – 39 °C abgekühlt ist, übertragen Sie die Gehirne (maximal 4) vorsichtig mit einer Pasteurpipette hinein. Verwenden Sie für diesen Schritt immer Schnitttipps.
- Rühren Sie die Agarose um das Gewebe entweder mit einem Spachtel oder einem Paar Dumont #1 Zange, ohne das Gewebe zu berühren. Lassen Sie die Agarose bei Raumtemperatur erstarren. Sobald sich die Agarose verfestigt hat, trimmen Sie die überschüssige Agarose, die das Gewebe umgibt.
- Füllen Sie das Pufferfach mit PBS. Richten Sie das Gehirn mit der rostro-kaudalen Achse des Gewebes senkrecht zum Tablett aus (verwenden Sie als Wahrzeichen die olfaktorischen Glühbirnen, die den rostral-größten Teil des Gehirns darstellen). Mit einem Vibratome 250 m Scheiben schneiden.
- Füllen Sie eine 3,5 cm Petrischale mit 2 ml vorgewärmten Medien. Mit einer Pasteur-Kunststoff-Pipette Scheiben (10 - 15) auf diese Schale übertragen. Sobald fertig, verschieben Sie die Petrischale mit den Scheiben in die Scheiben Kultur Inkubator. Halten Sie Scheiben bei 37 °C in einer befeuchteten Atmosphäre, die 40% O2 / 5% CO2 / 55%N2 enthält, bis zur Anwendung.
4. Mikroinjektion
- Schalten Sie Computer, Mikroskop, Mikroskopkamera, Manipulatoren, Druckgerät und Drucksensor ein. Laden Sie die Anwendung, indem Sie auf die Datei "launchapp.py" im Hauptordner klicken, der von GitHub heruntergeladen wurde, und geben Sie die Geräteeinstellungen im Popup-Bildschirm an (installationsanweisungen finden Sie unter Schritt 1.1).
- Erzeugen Sie einen äußeren Druck, um unerwünschte Verstopfungen zu verhindern, bevor Sie die Pipette in die Lösung eintauchen. Um Druck auf die Pipette auszuüben, schieben Sie den Kompensationsdruckbalken auf 24 – 45 % und klicken Sie auf Werte festlegen. Als nächstes stimmen Sie den Druck auf einen ausreichenden Druck ab, indem Sie den mechanischen Druckventilknopf auf 1 – 2 PSI (69 – 138 mbar) drehen, wie durch den Drucksensor angegeben.
- Die Scheiben in eine 3,5 cm lange Petrischale mit 2 ml vorgewärmten CIMM geben. Die zu mikroinjizierenden Scheiben in die Mitte der Petrischale geben. Die Petrischale auf die vorgewärmte (37 °C) Mikroinjektionsstufe geben.
- Beladen Sie die Mikroinjektionspipette mit einer Langspitzen-Kunststoffpipette mit 1,4 –1,6 l Mikroinjizlösung (ab Schritt 3.3). Setzen Sie die Mikroinjektionspipette auf den Pipettenhalter.
- Mit der niedrigsten Vergrößerung auf dem Mikroskop, bringen Sie die Scheibe in den Fokus und führen Sie die Mikropipette zu diesem Sichtfeld (FOV), so dass es auf die gleiche Ebene wie das Scheibenziel konzentriert ist. Schalten Sie den Ausgang des Mikroskops auf die Kamera, um den FOV in der Anwendung zu sehen.
- Klicken Sie auf die Vergrößerungsschaltfläche oben links in der Benutzeroberfläche, um die Gerätekalibrierung zu initiieren. In einem Fenster wird die Vergrößerung angezeigt. Wählen Sie die 10-fache Vergrößerung oder die Vergrößerung der Linse (z. B. 4x, 10x, 20x, 40x) und drücken Sie Ok. Die Software geht davon aus, dass die interne Objektivlinse das 10-fache (die häufigste Objektivvergrößerung) ist.
- Richten Sie die Pipettenspitze mit dem mikrometrischen Rad des Mikroskops neu aus und klicken Sie mit dem Cursor auf die Pipettenspitze. Drücken Sie als Nächstes die Taste Schritt 1.1 und im Popup-Fenster auf OK. Die Pipette bewegt sich in Y-Richtung. Klicken Sie auf die Spitze der Pipette und drücken Sie die Taste Schritt 1.2. Geben Sie schließlich 45 in die Pipette-Winkelbox ein und drücken Sie den Einstellungswinkel.
- Geben Sie die gewünschten Parameter in das Bedienfeld "Automatisierte Mikroinjektion" ein. Für die Mikroinjektion in apikale Vorläufer stellen Sie den Injektionsabstand auf 20 – 40 m und die Tiefe auf 10 – 15 m ein. Für die Mikroinjektion in Neuronen stellen Sie den Injektionsabstand von der Basalseite von 30 – 40 m und die Tiefe auf 10 – 30 m, je nachdem, was angestrebt wird. Stellen Sie die Geschwindigkeit immer auf 100 % ein. Klicken Sie auf Werte festlegen.
ANMERKUNG: Der Anflugabstand ist der Abstand, den die Pipette aus dem Gewebe zieht, bevor sie zum nächsten Injektionsabstand bewegt, Tiefe ist die Tiefe in gewebe, die die Mikroinjektion geht, Abstand ist der Abstand entlang der Linie zwischen sequentiellen Injektionen, Geschwindigkeit ist die Geschwindigkeit der Pipette in m/s. - Klicken Sie auf die Schaltfläche Kante zeichnen, und ziehen Sie den Cursor entlang der gewünschten Flugbahn im Popupfenster, um die Injektionsbahn zu definieren. Bei mikroinjizierenden Vorläuferstammzellen wird die ventrale Seite der Telencephalonoberfläche wie in Abbildung 2Adargestellt. Bringen Sie die Pipette an den Anfang der Linie und klicken Sie auf die Spitze der Pipette. Klicken Sie auf Flugbahn ausführen, um mit der Mikroinjektion zu beginnen. Wiederholen Sie diesen Schritt für jede zielgerichtete Injektionsebene (in der Regel für 3 – 4 Flugzeuge mit 40 – 75 Injektionen pro Ebene).
5. Gewebekultur und Gewebescheibenverarbeitung zur Immunfluoreszenz
- Kollagengemisch (1,5 mg/ml) vorbereiten: Zu einem Rohr 1,25 ml der Matrixlösung, 0,5 ml destilliertes Wasser, 0,5 ml 5x DMEM-F12-Lösung und 0,25 ml Rekonstitutionspuffer hinzufügen. Halten Sie es auf Eis, bis es verwendet wird.
- Erhalten Sie die Petrischale mit den mikroinjizierten Scheiben aus der Scheibenkultur-Inkubationskammer und tauchen Sie die Scheiben in die Kollagenmischung ein.
- Die Scheiben zusammen mit 200 – 300 l Kollagenmischung in einen 14 mm Brunnen einer 35 mm Glasbodenschale geben. Stellen Sie sicher, dass die Scheiben mit sehr weniger Kollagen bedeckt sind. Diese Einrichtung ermöglicht optimale Bedingungen für Nährstoffe und Sauerstoffaufnahme.
- Richten Sie die Slices aus, während Sie sicherstellen, dass genügend Platz zwischen den Slices vorhanden ist, indem Sie zwei Zangenpaare verwenden. Inkubieren Sie die Petrischale für 5 min bei 37 °C mit einem Heizblock, damit das Kollagen verfestigt werden kann. Betrachten Sie diese Zeit als t = 0 der Slice-Kultur.
- Bewegen Sie die Petrischale für weitere 40 min zurück in den Slice Culture Inkubator. Fügen Sie dann 2 ml des vorgewärmten SCM hinzu. Slices werden in der Kultur bis zum gewünschten Zeitpunkt aufbewahrt.
- Nehmen Sie die Scheiben aus dem Slice Culture Inkubator und aspirieren Sie den SCM. Waschen Sie die kollageneingebetteten Scheiben mit 1x PBS. 4% (wt/vol) Paraformaldehyd (in 120 mM Phosphatpuffer, pH 7,4) hinzufügen und das Gewebe 30 min bei RT lassen. Dann bewegen Sie es auf 4 °C, um eine nächtliche Fixierung zu ermöglichen.
- Saugen Sie die Paraformaldehydlösung am nächsten Tag an und führen Sie 1x PBS-Wärungen durch. Um die Scheiben aus dem Kollagen zu entfernen, verwenden Sie zwei Zangenpaare, um die Scheiben unter einem Stereomikroskop sanft zu extrahieren.
- Verwenden Sie eine Mikrowelle, um die 3% (wt/vol) niedrige Schmelzpunkt-Agarose für die Verarbeitung der mikroinjizierten Scheiben zu schmelzen. Gießen Sie die geschmolzene Agarose in eine Einweg-Einbettform und lassen Sie sie auf ca. 38 – 39 °C abkühlen.
- Übertragen Sie die Gewebescheiben aus Schritt 5.7 mit niedrigschmelzender Agarose in diese Form mit einer Pasteur-Kunststoffpipette aus Kunststoff. Stellen Sie sicher, dass die Pialseite der Scheibe nach oben und die ventrikuläre Oberfläche nach unten zeigt. Bei Bedarf entsprechend orientieren. Lassen Sie die Agarose auf RT abkühlen, um sie zu erstarren.
- Trimmen Sie die zusätzliche Agarose, die die Scheiben umgibt. Richten Sie den Agaroseblock aus, um sicherzustellen, dass die Schnittfläche parallel zur Schneidklinge des Vibratom ist. Mit Vibratome 50 'm dicke Abschnitte schneiden.
- Füllen Sie eine 24 Brunnenschale mit 1x PBS. Übertragen Sie die Abschnitte in diese Schale mit einem feinspitzen Pinsel. Führen Sie die Immunfluoreszenz gemäß den Standardprotokollen durch.
Mikroinjektion dient dem Zweck der Verfolgung und Manipulation einzelner neuronaler Stammzellen und ihrer Nachkommen in lebendem Gewebe und deren Abstammungsprogression in einer physiologischen Umgebung. In diesem Artikel haben wir den Einsatz des Mikroinjektionsroboters zur gezielten und automatischen Mikroinjektion organotypischer Scheiben des Maustelecephalons demonstriert. Abbildung 2 zeigt repräsentative Bilder von erfolgreich injizierten Vorläuferzellen und Abbildung 3 zeigt injizierte Neugeborene. Bei Injektion mit Dextran Alexa-488 (oder Alexa-A555) Farbstoff, Zellen erscheinen vollständig mit dem Farbstoff gefüllt. Was die apikalen Vorläufer (Abbildung 2) betrifft, so ermöglicht die konfokale Bildgebung die Rekonstruktion der Zellmorphologie, das Vorhandensein -oder Fehlen - der apikalen und basalen Anhaftung mit hoher räumlicher Auflösung und die Kombination der morphologischen Untersuchung mit markerexpression. Durch die Kombination dieser Kriterien kann der Benutzer den mikroinjizierten Zellen und deren Nachkommen ein bestimmtes Zellschicksal zuordnen. Wie für die Neuron-Injektion, der Benutzer kann die neuronale Morphologie rekonstruieren, einschließlich der Struktur und Merkmale von apikalen Dendrit und Axon. Automatisierte Mikroinjektion kann einen deutlich höheren Durchsatz im Vergleich zur manuellen Mikroinjektion liefern (Abbildung 2B). Darüber hinaus bestätigt die EdU-Kennzeichnung, dass die Zelllebensfähigkeit durch die Automatisierung nicht beeinträchtigt wird (Abbildung 2C). Das Halten der organotypischen Scheibe in der Kultur ermöglicht die folgende Lineage-Progression der mikroinjizierten Zellen (wir zeigten 4 - 24h in Abbildung 2D). Wenn die Mikroinjektionslösung genetisches Material (DNA, mRNA, CRISPR-Cas9-Leitfäden) oder rekombinante Proteine enthält, dann ermöglicht dies die Untersuchung, ob und wie die Abstammungsprogression durch die Manipulation beeinflusst wird.
Mikroinjektion in einzelne neuronale Stammzellen im Gewebe bietet eine ausgezeichnete Einzelzellauflösung und aus diesem Grund wurde es verwendet, um die Zellbiologie der neuronalen Stammzellprogression und Schicksalsübergang zu sezieren (Abbildung 3A). Microinjection ermöglicht die Lieferung von komplexen Mischungen von Chemikalien. Wir nutzten diese Funktion zuvor, um die Junctional-Kopplung in neuronalen Vorläuferzellen zu untersuchen, indem wir lückenjunctional durchlässig mit lückenkreuzenden, undurchlässigen Fluoreszenzfarbstoffen12vermischten. Wir erweiterten frühere Arbeiten, indem wir die Junctional-Kopplung in neugeborenen Neuronen untersuchten, indem wir Lucifer Yellow zusammen mit Dextran-A555 injizierten (Abbildung 3B). Wie in Abbildung 3Bdargestellt, wird ein Anteil der neugeborenen pyramidalen Neuronen über Spaltknoten mit benachbarten Neuronen gekoppelt. Diese Beobachtung steht im Einklang mit der Idee, dass unreife Neuronen über Gap-Junction13,14kommunizieren. Darüber hinaus zeigt das Targeting von Neuronen, dass der Einsatz des Mikroinjektionsroboters auf mehrere Zelltypen im sich entwickelnden Säugetiergehirn verallgemeinert werden kann. Dieses experimentelle Setup wird nützlich sein, um die Zellbiologie von Neuronen im Gewebe zu sezieren, zum Beispiel durch die Bereitstellung spezifischer Oligopeptide, um Protein-Protein-Wechselwirkungen zu stören.
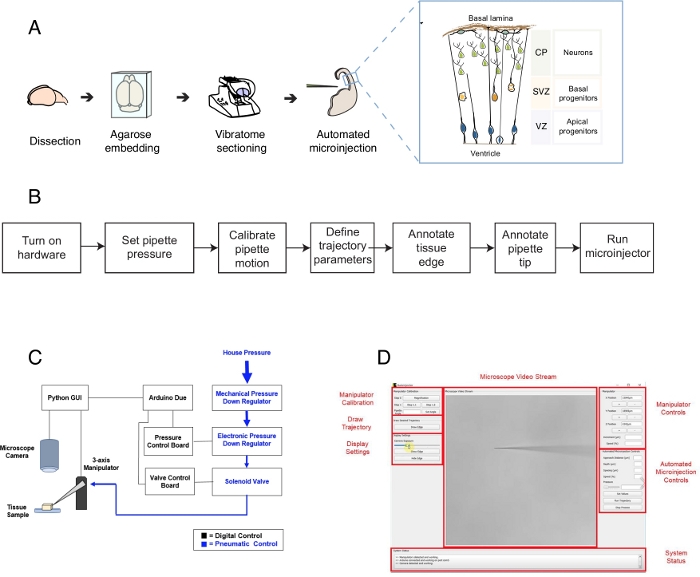
Abbildung 1: AutomatisierteS Microinjection-Setup und Protokoll. (A) Gesamtprotokoll für Gewebevorbereitung und automatisierte Mikroinjektionen mit dem Mikroinjektionsroboter. Right inset: Cartoon Schaltplan der Maus Telencephalon gezielt für Mikroinjektion in diesem Protokoll. (B) Flussdiagramm der automatisierten Mikroinjektionsschritte. (C) Schematic der Mikroinjektionsroboter-Hardware. (D) Grafische Benutzeroberfläche (GUI) der Software zur Steuerung und Bedienung des Mikroinjektionsroboters. Diese Zahl ist ab Ref.12angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
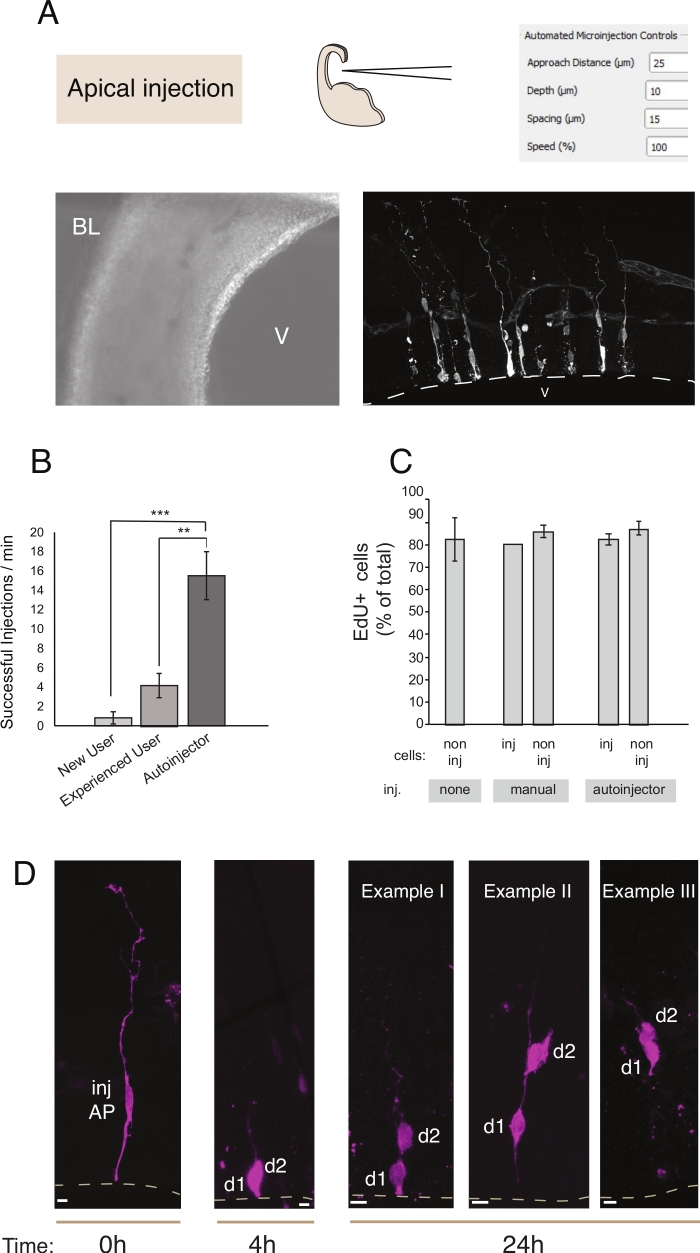
Abbildung 2: Robotische Mikroinjektion in apikale Vorläufer. Schematische und erwartete Ergebnisse bei verwendung des Mikroinjektionsroboters zur Zielscheibe von apikalen Vorläufern (APs) über die apikale Oberfläche (apikale Injektion). (A) Obere Zeile. Auf der linken Seite: Schema des Prozesses. Auf der rechten Seite: GUI mit relevanten Parametern für die apikale Injektion. Untere Zeile. Links: Phasenkontrastbild, das während des Injektionsvorgangs aufgenommen wurde (V: Ventrikel; BL: basal lamina). Auf der rechten Seite: repräsentative Ergebnisse, die mikroinjizierte APs zeigen. Die gestrichelte Linie stellt den Ventrikel (V) dar. Scale bar: 10 m. (B) Erfolgreiche Injektionen pro Minute für einen unerfahrenen Benutzer auf dem manuellen Mikroinjektionssystem, einen erfahrenen Benutzer auf dem manuellen Mikroinjektionssystem und den Mikroinjektionsroboter. (C) EdU-Einbindung in mikroinjizierte Zellen und in nicht injizierte Zellen im injizierten Bereich. Organotypische Mausscheiben E14.5 dorsal telencephalon wurden entweder (i) nicht injiziert oder (ii) manueller oder automatisierter Mikroinjektion (injizierte Scheibe) mit Dextran-A488 (für manuelle und Autoinjektor) unterzogen. Slices wurden in der Kultur in Gegenwart von EdU für 24 h gehalten, dann wurden sie fixiert und für DAPI und EdU gebeizt. Injizierte und nicht injizierte Zellen im injizierten Bereich wurden für EdU-Positivität bewertet. (D) Verwendung des Mikroinjektionsroboters Lineage Tracing. Ein fluoreszierender Farbstoff (Dx3-A555, Magenta) wird in einzelne neuronale Stammzelle injiziert (t = 0 h). Der Fluoreszenzfarbstoff wird während der Mitose in die Tochterzellen (d1, d2) partitioniert. Dies ermöglicht es, der Nachkommenschaft der injizierten Zelle (t = 4 h und 24 h) zu folgen und die Abstammungsprogression im Laufe der Zeit zu offenbaren. Für t = 24 h zeigen wir mehrere Beispiele der Nachkommenschaft, die man zu finden erwartet. Skalenbalken: 10 m. Grafiken in B und C stammen aus Ref.12Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
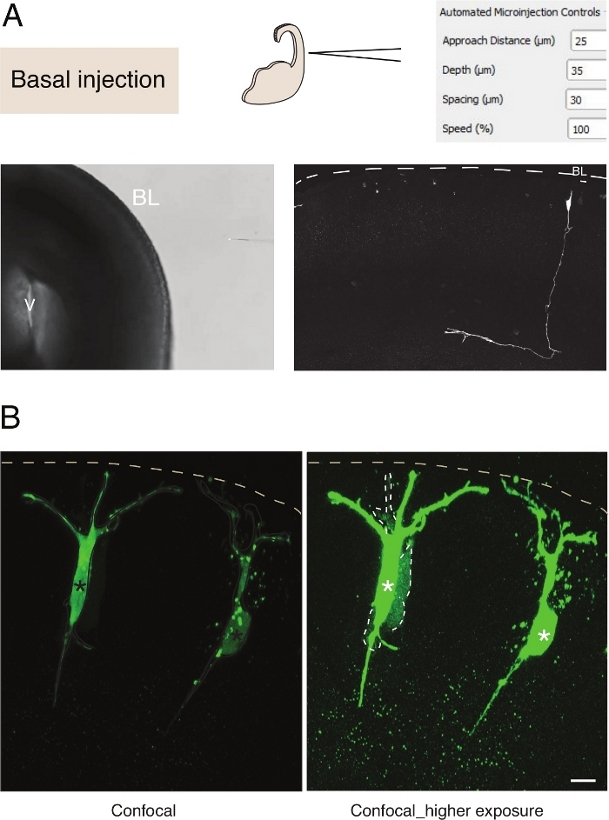
Abbildung 3: Robotische Mikroinjektion in Neuronen. Schematische und erwartete Ergebnisse bei Verwendung des Mikroinjektionsroboters, um pyramidale Neuronen (N) über die Basaloberfläche (Basalinjektion) zu zielen. (A) Obere Zeile. Auf der linken Seite: Schema des Prozesses. Auf der rechten Seite: GUI mit relevanten Parametern für die Basalinjektion. Untere Zeile. Links: Phasenkontrastbild, das während des Injektionsvorgangs aufgenommen wurde (V: Ventrikel; BL: basal lamina). Auf der rechten Seite: repräsentative Ergebnisse, die eine mikroinjizierte N. Dashed Linie zeigen, stellen die Basallamina (BL) dar. Skala bar: 10 m. (B) Verwendung des Autoinjektors zur Untersuchung der Lückenknotenkommunikation im Gewebe. Pyramidenneuronen wurden mit einer Lösung injiziert, die zwei Farbstoffe enthält: dem Spalt-Kreuzungs-undurchlässigen Dx-A555 (nicht gezeigt) und dem spalt-junctionaldurchlässigen Luzifergelb (grün). Dx-A555 ist auf die Zielzelle (Sternchen) beschränkt, während die LY zu Zellen diffundiert, die über einen Lückenknoten mit der Zielzelle verbunden sind (gestrichelte Linien). Linkes Panel: Geringe Belichtung, nur die mikroinjizierten Zellen sind sichtbar. Rechtes Panel: Hohe Belichtung ermöglicht die Visualisierung der injizierten Zellen sowie der gekoppelten Zellen (gestrichelte Linien). Maßstabsleiste: 10 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei: Fehlerbehebung bei mehreren häufigen Fehlern, die während der Mikroinjektion auftreten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Mikroinjektion in einzelne neuronale Stammzellen im Gewebe bietet eine ausgezeichnete Einzelzellauflösung und aus diesem Grund wurde es verwendet, um die Zellbiologie der neuronalen Stammzellprogression und Schicksalsübergang zu sezieren (Abbildung 2; siehe auch10,11,12). Das automatisierte Mikroinjektionsverfahren kann an anderen Zelltypen sowohl in embryonalen Mäusen als auch an menschlichem Hirngewebe durchgeführt werden. Repräsentative Ergebnisse der Mikroinjektion von neugeborenen Neuronen durch gezielte Stolperung der Basaloberfläche des Telencephalons sind in Abbildung 3dargestellt.
Das hier etablierte Prinzip kann auf verschiedene Zelltypen in embryonalen Mausgehirnen und menschlichen Gehirnen angewendet werden. Wir haben bereits gezeigt, dass der Mikroinjektionsroboter auch verwendet werden kann, um einzelne Vorläuferzellen im Maus-Hindhirn und Telencephalon und neugeborenen Neuronen in der Maus und human entwickelnden Neocortex12zu zielen. Um die besten Ergebnisse des Injektionsverfahrens zu erhalten, sollte man alle Schritte vor Beginn der Injektion optimieren. Es ist wichtig, die Vorbereitung lebensfähiger und gut erhaltener organotypischer Gewebescheiben aus Hirngewebe sorgfältig zu prüfen und zu optimieren (Abbildung 1). Es ist von entscheidender Bedeutung, bei der in Abbildung 1dargestellten Sezier- und Schneidungsprozedur schnell zu sein. Für die apikale Injektion, die auf die APs abzielt, sollte man die Scheiben auswählen, die die ideale Ausrichtung der apikalen Oberfläche aufweisen. Bei der APs-Injektion ist die ideale Ausrichtung die apikale Oberfläche senkrecht zum Boden der Petrischale. Jede andere Ausrichtung wird auch freizügig sein, aber die apikale Oberfläche senkrecht zur Petrischale bietet eine breitere Oberfläche für die Injektion, wodurch der Erfolg der Injektion erhöht wird. Bei der Injektion in Neuronen spielt die Ausrichtung der Scheibe wenig bis gar nichts.
Sobald die zu injizierenden Scheiben ausgewählt sind, dauert der Injektionsvorgang pro Scheibe ca. 5 Minuten. Wenn man bedenkt, dass man mit lebendem Gewebe arbeitet, wird dringend empfohlen, die Injektion zu beschleunigen. Zu diesem Zweck empfehlen wir, alle Injektionsparameter über die GUI (Abbildung 1D) einzustellen, bevor das Gewebe bereit ist, um unnötige Wartezeiten zu reduzieren. Zur Fehlerbehebung lesen Sie bitte die Zusatzdatei.
Bei langfristiger Scheibenkultur können Schritte nach dem automatisierten Mikroinjektionsverfahren die Gesundheit der Zellen und damit das Experiment beeinflussen. Daher wird dringend empfohlen, einen Qualitätskontrolltest durchzuführen und die Slice-Kulturbedingungen zu optimieren. Um die Zelllebensfähigkeit nach dem Slicing- und Injektionsverfahren zu bewerten, führten wir während der Kultur eine EdU-Kennzeichnung durch und quantifizierten die Anzahl der pyknotischen Kerne (ein Proxy für apoptotische Zellen) in den Kulturen und das injizierte Gewebe12. Diese Quantifizierungen ergaben keine signifikanten Auswirkungen der Mikroinjektion auf die Gewebelebensfähigkeit (Abbildung 2C). Wir empfehlen, ähnliche Qualitätskontrollen durchzuführen, während die organotypische Gewebeschneide- und Mikroinjektionspipeline im Labor etabliert wird.
Im Vergleich zur manuellen Mikroinjektion bietet der Mikroinjektionsroboter mehrere Vorteile. Erstens ist die Lernkurve für den Benutzer weniger steil im Vergleich zur manuellen Injektion: Ein neuer Benutzer erreicht nach einer begrenzten Anzahl von Sitzungen, in der Regel 1 oder 2, eine hohe Kompetenz. Zweitens erfordert eine vergleichbare Befähigung bei der manuellen Mikroinjektion eine monatelange Ausbildung. Das Injektionsverfahren ist schneller und effizienter (Abbildung 2B). Wir quantifizierten diese Parameter und fanden heraus, dass der Mikroinjektionsroboter einen erfahrenen manuellen Anwender in Bezug auf den Injektionserfolg (% der erfolgreichen Injektion/Gesamtanzahl der Injektionen) und in der Gesamtzahl der Injektionen pro Zeiteinheit12übertraf. Dies führt zu einer Gesamtsteigerung der Injektionseffizienz (% der erfolgreichen Injektion/min) für den Mikroinjektionsroboter im Vergleich zu einem erfahrenen Anwender. Noch ausgeprägter war die Effizienzsteigerung beim Vergleich des Mikroinjektionsroboters mit einem Anfänger und erreichte 700%. Zu guter Letzt lässt sich der Mikroinjektionsroboter einfach so programmieren, dass er systematisch alle räumlichen Parameter erforscht. Dies ist besonders vorteilhaft, wenn der Mikroinjektionsroboter an neue Zellen oder Gewebe angepasst wird oder wenn der Mikroinjektionsroboter für Zwecke verwendet wird, die eine unterschiedliche räumliche Auflösung erfordern.
Der Bau des Mikroinjektionsroboters erfordert minimale Änderungen an einem vorhandenen Epifluoreszenzmikroskop12. Wir haben bereits Anweisungen für diese Anpassung bei https://github.com/bsbrl/Autoinjector. Sobald die Hardware eingerichtet ist, liefert dieses Protokoll wichtige methodische Details für die erfolgreiche Durchführung automatisierter Mikroinjektionen. Insgesamt hat der Mikroinjektionsroboter eine erfolgreiche Injektionsrate von 15,52 + 2,48 Injektionen/min, was 15x größer ist als ein unerfahrener Benutzer (1,09 ± 0,67 Injektionen/min) und 3x größer als ein erfahrener Benutzer (4,95 ± 1,05 Injektionen/min)12. Diese Verbesserung der erfolgreichen Injektionsrate befähigt sowohl Anfänger als auch erfahrene Anwender, mehr Zellen in kürzerer Zeit zu injizieren, was wichtig ist, um die Lebensfähigkeit des Gewebes zu erhalten. Darüber hinaus ist der Mikroinjektionsroboter anpassbar und die Flugbahn, die Tiefe der Injektion, die Anzahl der Injektionen, der Abstand zwischen den Injektionen können alle mit der GUI abgestimmt werden. Diese Eigenschaften ermöglichen es, den Mikroinjektionsroboter als Werkzeug zur Optimierung bisher mühsamer Experimente zu verwenden und grundlegend neue Experimente zu erforschen, die eine höhere Ausbeute als bisher möglich erfordern.
Die Haupteinschränkungen des mikroinjektionsverfahrens, das wir hier beschrieben haben, beziehen sich auf die Herstellung von Gewebescheiben, ein entscheidender Schritt, der einer umfassenden Optimierung bedarf. Darüber hinaus beruht die Mikroinjektion auf dem Vorhandensein einer Oberfläche, die von der Glaspipette angefahren werden kann. Diese Funktion begrenzt die Art von Geweben und Gewebepositionen, die über Mikroinjektion mit dem aktuellen Setup gezielt werden können.
Der Mikroinjektionsroboter verwendet derzeit Brightfield-Bildgebung und wurde in In-vitro-Gehirnscheibenpräparaten eingesetzt. In Zukunft könnte der Mikroinjektionsroboter mit 2-Photonen-Bildgebung kombiniert werden, um die Spezifität des Einzelzell-Targetings in vivo für molekulare oder Farbstoff-Tagging zu erhöhen. Solche Anstrengungen wurden bereits für die Einzelzellelektrophysiologie15,16unternommen. Das aktuelle Gerät erfordert eine manuelle Beobachtung des Mikroinjektionsverfahrens. Zukünftige Versionen könnten Strategien für die Reinigung verstopfter Mikroinjektionspipetten17 oder die Integration von Fluidhandling-Robotern18 für multiplexige, vollautonome Mikroinjektionen umfassen. Diese Geräte könnten den Umfang der Mikroinjektion um Größenordnungen erhöhen. Die Anpassung von Algorithmen zur parallelen Steuerung mehrerer Mikroinjektionspipetten19 könnte die multiple Verabreichung dutzender Farbstoffe und molekularer Reagenzien in dieselben Zellen innerhalb derselben Experimente ermöglichen. Dies hat das Potenzial, neue Wege für das molekulare Screening im Gewebe zu eröffnen.
Der Mikroinjektionsroboter könnte verwendet werden, um funktionell identifizierte Zellen mit DNA- oder RNA-Barcodes zu markieren. Dies könnte wiederum mit anderen einzelzelligen Analysetechniken wie der Einzelzell-RNA-Sequenzierung (scRNAseq) und der Elektronenmikroskopie kombiniert werden. Unsere vorläufigen Ergebnisse zeigen, dass mikroinjizierte Zellen und ihre Nachkommen mit Hilfe der Gewebedissoziation, gefolgt von der FACS-Sortierung (Taverna, unveröffentlichte Ergebnisse), zurückgewonnen und isoliert werden können. Die FACS-sortierten Zellen können dann für scRNAseq verwendet werden. Darüber hinaus zeigen vorläufige Ergebnisse, dass die Einzelzellauflösungsfähigkeiten des Mikroinjektionsroboters in Kombination mit elektronenmikroskopischer Analyse genutzt werden können, um die Zellbiologie an neuronalen Stammzellen im Gewebe mit hoher räumlicher Auflösung zu erforschen (Taverna und Wilsch-Bräuninger, unveröffentlichte Ergebnisse). Diese Daten deuten darauf hin, dass der Mikroinjektionsroboter als Werkzeug für die korrelative Licht- und Elektronenmikroskopie im Gewebe und im weiteren Sinne für die multimodale Analyse der Zellidentität und des Zellverhaltens im Gewebe eingesetzt werden kann.
Die Mikroinjektion setzt auf den Einsatz von Druck und man kann sich Injektionslösungen mit hoher molekularer Komplexität (z.B. ein ganzes Transkriptom) leisten. Diese Funktion der Mikroinjektion wurde in der Vergangenheit für das Isolieren und Klonen von Liganden-Gated-Rezeptoren20genutzt. Entlang dieser Linie könnte der Mikroinjektionsroboter für die Modellierung und Untersuchung multi-genicrischer Merkmale auf zellulärer Ebene verwendet werden. In Kombination mit einer Subpooling-Strategie könnte der Mikroinjektionsroboter auch als Plattform verwendet werden, um den minimalen Satz von Genen zu identifizieren, die ein bestimmtes Merkmal/Zellverhalten antreiben. Bisher wurde der Mikroinjektionsroboter verwendet, um die Biochemie der Zelle durch die Lieferung von mRNA, DNA oder rekombinanten Proteinen10,21,22zu manipulieren. Wir sehen eine Anwendung des Mikroinjektionsroboters bei der Untersuchung der Biophysik des intrazellulären Raumes vor, beispielsweise durch die Bereitstellung von Nanomaterialien oder Nanomaschinen, die die Erfassung und/oder Manipulation der biophysikalischen Eigenschaften des intrazellulären Raumes ermöglichen.
Die Autoren haben nichts zu verraten.
Die Autoren möchten die Nomis Foundation (ET) würdigen. SBK erkennt Mittel aus dem Maschinenbau an, College of Science and Engineering, MnDRIVE RSAM Initiative der University of Minnesota, Minnesota Department of higher education, National Institutes of Health (NIH) 1R21NS103098-01, 1R01NS111028, 1R34NS1111654, 1R21NS112886 und 1R21 NS111196. GS wurde von der National Science Foundation Graduate Research Fellowship und dem NSF IGERT Ausbildungsstipendium unterstützt.
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose, Low Melt | Carl Roth | Cat# 6351.2 | |
| Agarose, Wild Range | Sigma-Aldrich | Cat# A2790 | |
| Best-CA 221 Glue | Best Klebstoffe GmbH & Co.KG | Cat# CA221-10ml | |
| B-27 Supplement | Thermo Fisher Scientific | Cat# 17504044 | |
| Cellmatrix Type-IA (Collagen, Type !) | FUJIFILM Wako Chemicals | Cat# 637-00653 | |
| Distilled Water | |||
| DMEM-F12, CO2 independent (w/o Phenol red) | Sigma-Aldrich | Cat# D2906 | |
| DMEM-F12, CO2 independent (with Phenol red) | Sigma-Aldrich | Cat# D8900 | |
| HEPES-NAOH, pH 7.2, 1M (HEPES buffer) | Carl Roth | Cat# 9105.3 | |
| L-Glutamine, 200 mM | Thermo Fisher Sientific | Cat# 25030024 | |
| Mowiol 4-88 | Sigma-Aldrich | Cat# 81381 | |
| N-2 Supplement | Thermo Fisher Scientific | Cat# 17502048 | |
| Neurobasal Medium | Thermo Fisher Scientific | Cat# 21103049 | |
| Nuclease-free water | Thermo Fisher Scientific | Cat# AM9937 | |
| O2 (40%), CO2 (5%), N2 (55%) Mix, 50 liters | |||
| Paraformaldehyde | Merck | Cat# 818715 | |
| PBS | |||
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | Cat# 15140122 | |
| Rat serum | Charles River Laboratories | ||
| Japan | |||
| Sodium bicarbonate (NaHCO3) | Merck | Cat# 106323 | |
| Sodium hydroxide (NaOH) | Merck | Cat# 106482 | |
| Tyrode’s salt | Sigma | Cat# T2145-10x1L) | |
| Equipment | |||
| Borosilicate glass capillaries, 1.2 mm outer diameter x 0.94 mm inner diameter | Sutter Instruments | Cat# BF-120-94-10 | |
| Bottle-top filter system, 500 mL | Corning | Cat# 430769 | |
| Computer PC | |||
| Custom pressure rig | Custom pressure rig | ||
| Electronic pressure regulator | Parker Hannifin | Cat# 990-005101-002 | |
| Falcon tubes, 15 mL | Corning | Cat# 430791 | |
| Falcon tubes, 50 mL | Corning | Cat# 430829 | |
| Fine-tip paintbrush | |||
| Flaming/ Brown micropipette puller | Sutter Instruments | Cat# P-97 | |
| Forceps, Dumont no. 3 | Fine Science Tools | Cat# 11231-30 | |
| Forceps, Dumont no. 5 | Fine Science Tools | Cat# 11255-20 | |
| Forceps, Dumont no. 55 | Fine Science Tools | Cat# 11252-20 | |
| Heating block | Labtech International | Cat # Dri block Digi2 | |
| Inverted fluorescence microscope | Zeiss | Cat# Axiovert 200 | |
| Light source | Olympus | Cat# Highlight 3100 | |
| Manual pressure regulator | McMaster Carr | Cat# 0-60 PSI 41795K3 | |
| Microloader Tips | Eppendorf | Cat# 5242956.003 | |
| Microcontroller | Arduino | Cat# Arduino Due | |
| Microscope camera Hamamatsu Orca Flash 4.0 V3 | |||
| Motorized stage XY for microscope | |||
| Multiwell plate, 24 wells | Nunc | Cat# 142475 | |
| Pasteur pipettes, plastic | |||
| Petri dish, 60 x 15 mm | Greiner | Cat# 628102 | |
| Petri dish, 35 x 10 mm | Nunc | Cat# 153066 | |
| Petri dish, 34 x 14 mm, including Microwell no. 1.5 cover glass | MatTek | Cat# P35G-1.5-14-C | |
| Pipette holder | Warner Instruments | Cat# 64-2354 MP-s12u | |
| Pipette and tips | |||
| Puller filament, 3.0-mm square box filament | Sutter Instrument | Cat# FB330B | |
| Slice culture incubation box | MPI-CBG | Cat# custom made | |
| Solenoid valve | Cat# LHDA053321H-A | ||
| Stereomicroscope | Olympus | Cat# SZX12 | |
| Tabletop centrifuge | Heraeus | Cat# 5431622 | |
| Thermometer | |||
| Three-axis Manipulator | Sensapex Inc | Cat# tree-axis uMP | |
| Vibratome | Leica | Cat# VT1000s | |
| Whole-embryo-culture-system incubator | Ikemoto Company | Cat# RKI-10-0310 | |
| Waterbath | |||
| Software and Algorithms | |||
| Arduino | Arduino | ||
| Fiji | RRID: SCR_002285 | ||
| Python | Python Software foundation | Python 2.7.12 | |
| ZEN | RRID: SCR_013672 |
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Götz, M., Huttner, W. B. The cell biology of neurogenesis. Nature Reviews Molecular Cell Biology. 6 (10), 777-788 (2005).
- Di Lullo, E., Kriegstein, A. R. The use of brain organoids to investigate neural development and disease. Nature Reviews Neuroscience. 18 (10), 573-584 (2017).
- Lancaster, M. A., Knoblich, J. A. Organogenesisin a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Kretzschmar, K., Clevers, H. Organoids: Modeling Development and the Stem Cell Niche in a Dish. Developmental Cell. 38 (6), 590-600 (2016).
- Pepperkok, R. et al. Automatic microinjection system facilitates detection of growth inhibitory mRNA. Proceedings of the National Academy of Sciences of the United States of America. 85 (18), 6748-6752 (1988).
- Pepperkok, R., Lowe, M., Burke, B., Kreis, T. E. Three distinct steps in transport of vesicular stomatitis virus glycoprotein from the ER to the cell surface in vivo with differential sensitivities to GTPγS. Journal of Cell Science. 111 (13), 1877-1888 (1998).
- Pepperkok, R. et al. β-COP is essential for biosynthetic membrane transport from the endoplasmic reticulum to the Golgi complex in vivo. Cell. 74 (1), 71-82 (1993).
- Ansorge, W., Pepperkok, R. Performance of an automated system for capillary microinjection into living cells. Journal of Biochemical and Biophysical Methods. 16 (4), 283-292 (1988).
- Taverna, E., Haffner, C., Pepperkok, R., Huttner, W. B. A new approach to manipulate the fate of single neural stem cells in tissue. Nature Neuroscience. 15 (2), 329-337 (2012).
- Wong, F. K., Haffner, C., Huttner, W. B., Taverna, E. Microinjection of membrane-impermeable molecules into single neural stem cells in brain tissue. Nature Protocols. 9 (5), 1170-1182 (2014).
- Shull, G., Haffner, C., Huttner, W. B., Kodandaramaiah, S. B., Taverna, E. Robotic platform for microinjection into single cells in brain tissue. EMBO Reports. 20 (10), e47880 (2019).
- Jabeen, S., Thirumalai, V. The interplay between electrical and chemical synaptogenesis. Journal of Neurophysiology. 120 (4), 1914-1922 (2018).
- Nagy, J. I., Pereda, A. E., Rash, J. E. Electrical synapses in mammalian CNS: Past eras, present focus and future directions. Biochimica et Biophysica Acta - Biomembranes. 1860 (1), 102-123 (2018).
- Suk, H.J. et al. Closed-loop real-time imaging enables fully automated cell-targeted patch-clamp neural recording in vivo. Neuron. 95 (5), 1037-1047 (2017).
- Annecchino, L. A. et al. Robotic automation of in vivo two-photon targeted whole-cell patch-clamp electrophysiology. Neuron. 95 (5), 1048-1055 (2017).
- Kolb, I. et al. Cleaning patch-clamp pipettes for immediate reuse. Scientific Reports. 6, 35001 (2016).
- Holst, G. L. et al. Autonomous patch-clamp robot for functional characterization of neurons in vivo: development and application to mouse visual cortex. Journal of Neurophysiology. 121 (6), 2341-2357 (2019).
- Kodandaramaiah, S. B. et al. Multi-neuron intracellular recording 1 in vivo via interacting autopatching 2 robots. ELife. 7, 24656 (2018).
- Lubbert, H. et al. cDNA cloning of a serotonin 5-HT1c receptor by electrophysiological assays of mRNA-injected Xenopus oocytes (RNA fractionation/hybrid depletion/hybrid selection/choroid plexus/voltage clamp). Neurobiology. 84 (2) 4332-4336 (1987).
- Florio, M. et al. Human-specific gene ARHGAP11B promotes basal progenitor amplification and neocortex expansion. Science. 347 (6229), 1465-1470 (2015).
- Kalebic, N. et al. CRISPR/Cas9-induced disruption of gene expression in mouse embryonic brain and single neural stem cells in vivo. EMBO Reports. 17 (3), 338-348 (2016).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved