Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Bioengineering
Manipulación de células madre y neuronas neuronales únicas en las rebanadas cerebrales mediante la microinyección robótica
Este protocolo demuestra el uso de una plataforma robótica para la microinyección en células madre y neuronas neuronales individuales en las rebanadas cerebrales. Esta técnica es versátil y ofrece un método de seguimiento de células en tejido con alta resolución espacial.
Una pregunta central en la neurobiología del desarrollo es cómo el tallo neural y las células progenitoras forman el cerebro. Para responder a esta pregunta, uno necesita etiquetar, manipular y seguir células individuales en el tejido cerebral con alta resolución a lo largo del tiempo. Esta tarea es extremadamente difícil debido a la complejidad de los tejidos en el cerebro. Recientemente hemos desarrollado un robot, que guía una aguja de microinyección en el tejido cerebral al utilizar imágenes adquiridas a partir de un microscopio para entregar volúmenes de solución femtoliter en células individuales. La operación robótica aumenta el resultado de un rendimiento general que es un orden de magnitud mayor que la microinyección manual y permite un etiquetado preciso y una manipulación flexible de células individuales en el tejido vivo. Con esto, se pueden microinyectar cientos de células dentro de una sola rebanada organotípica. Este artículo demuestra el uso del robot de microinyección para la microinyección automatizada de células progenitoras neuronales y neuronas en las rebanadas de tejido cerebral. En términos más generales, se puede utilizar en cualquier tejido epitelial con una superficie a la que pueda llegar la pipeta. Una vez configurado, el robot de microinyección puede ejecutar 15 o más microinyeccións por minuto. El robot de microinyección debido a su rendimiento y versalidad hará de la microinyección una técnica de manipulación celular de alto rendimiento ampliamente sencilla que se utilizará en bioingeniería, biotecnología y biofísica para realizar análisis de una sola célula en rodajas cerebrales organotípicas.
Este protocolo describe el uso de un robot para apuntar y manipular células individuales en las rebanadas de tejido cerebral, centrándose en particular en las células madre y neuronas neurales individuales. El robot fue desarrollado para abordar una cuestión central en la neurobiología del desarrollo, que es cómo el tallo neural y las células progenitoras contribuyen a la morfogénesis cerebral1,2,3,4,5. Para responder a esta pregunta, uno necesita etiquetar y rastrear células madre neurales individuales y seguir su progresión del linaje a lo largo del tiempo para correlacionar el comportamiento de una sola célula con la morfogénesis tisular. Esto se puede lograr de diferentes maneras, por ejemplo, electroporando el tejido cerebral en el útero o etiquetando una sola célula usando matrices lipofílicas. Aunque son potentes, estos métodos carecen de una resolución precisa de una sola célula (electroporación) y/o la posibilidad de manipular el espacio intracelular (colorante lipofílico). La microinyección en células individuales fue desarrollada para superar este desafío6,7,8. Durante la microinyección, se inserta brevemente una pipeta en una sola célula dentro del tejido intacto bajo presión para microinyecciones de femtoliter volúmenes de reactivos9. Hemos descrito previamente un procedimiento manual para microinyección de células madre neurales individuales en tejido organotípico (Figura 1A)10,11. La microinyección en las células madre neurales se basa en el uso de una micropipeta que se inserta en células madre neurales individuales para inyectar una solución que contiene un tinte fluorescente, junto con otras moléculas de interés. La focalización selectiva de las células madre neurales se logra acercándose al telencéfalo en desarrollo a través de la superficie ventricular (o ventrículo, ver dibujos animados en la Figura 1A),que está formada por la membrana plasmática apical de los progenitores apicales (dibujos animados en la Figura 1A). Este proceso debe repetirse para cada célula que el experimentador desea inyectar. Además, el éxito de la microinyección depende del control preciso de la profundidad y duración de la inyección de micropipeta en el tejido. Por lo tanto, a pesar de las ventajas únicas, la microinyección manual es extremadamente tediosa y requiere una práctica considerable para funcionar con un rendimiento y rendimiento razonables, lo que hace que esta técnica sea difícil de usar de manera escalable. Para superar esta limitación, recientemente hemos desarrollado un robot guiado por imágenes, el Autoinyector12 (o robot de microinyección) que puede realizar automáticamente microinyeccións en células individuales.
El robot de microinyección hace uso de imágenes microscópicas y algoritmos de visión por computadora para apuntar con precisión a ubicaciones específicas en el espacio 3D dentro del tejido para la microinyección (Figura 1B). El robot de microinyección se puede construir haciendo modificaciones relativamente simples a una configuración de microinyección existente. El esquema general del robot de microinyección se muestra en la Figura 1C. Una pipeta se monta en un soporte de pipeta unido a un manipulador de tres ejes. Una cámara de microscopio se utiliza para adquirir imágenes del tejido y la aguja de microinyección. Se utiliza un sistema de regulación de presión personalizado para controlar la presión dentro de la pipeta y se utiliza un micromaniprógrafo programable para controlar la posición de la pipeta microinyector. Las imágenes de la cámara del tejido y la pipeta de microinyección se utilizan para determinar la ubicación espacial de la punta de la pipeta de microinyección y las ubicaciones en las que se deben realizar microinyección. A continuación, el software calcula las trayectorias necesarias para mover la pipeta dentro del tejido. Todo el hardware está controlado por el software que hemos desarrollado anteriormente. Todo el software está escrito en lenguaje de codificación (por ejemplo, Python y Arduino) y se puede descargar desde https://github.com/bsbrl/Autoinjector con instrucciones. La interfaz gráfica de usuario (GUI) permite al usuario crear imágenes del tejido y la micropipeta, y personalizar la trayectoria de la microinyección. Nuestro sistema se puede establecer utilizando modificaciones relativamente simples a un microscopio invertido equipado con filtros de campo brillante y epifluorescencia.
En primer lugar, proporcionamos instrucciones sobre la preparación de rebanadas de tejido organotípico cerebral para la microinyección. A continuación, el protocolo ilustra el inicio del robot de microinyección seguido de pasos preparatorios, como la calibración de movimiento de la pipeta, que deben realizarse antes de la microinyección. Esto es seguido por la definición de los parámetros de inyección. Después de esto, el usuario puede definir la trayectoria utilizada por el robot de microinyección e iniciar el procedimiento de inyección. El tejido microinyecte (en este caso se pueden mantener en rodajas de tejido organotípico cerebral) durante diferentes períodos de tiempo dependiendo del diseño experimental10,11. El tejido se puede procesar para seguir y estudiar la identidad y el destino de las células inyectadas y su progenie. Alternativamente, las células microinyeccionado se pueden seguir usando imágenes en vivo. Dentro del alcance de este protocolo, demostramos el uso del robot para microinyección células progenitoras neuronales en rodajas organotípicas de ratón E14.5 telencefallo dorsal. El robot es además capaz de microinyección en las neuronas de los recién nacidos en el ratón telencéfalo, así como en el telencefallo fetal humano12.
En resumen, describimos una plataforma robótica que se puede utilizar para seguir y manipular células individuales en el tejido. La plataforma hace uso de la presión y, por lo tanto, es extremadamente versátil en cuanto a la naturaleza química del compuesto a inyectar. Además, se puede adaptar a las células diana que no sean las células madre. Esperamos que nuestro sistema también se adapte fácilmente a otros sistemas modelo.
Todos los estudios en animales se llevaron a cabo de conformidad con la legislación alemana de bienestar animal, y las licencias necesarias se obtuvieron de la Comisión ética regional para la experimentación animal de Dresde, Alemania (Tierversuchskommission, Landesdirektion Dresden). Se prepararon rodajas organotípicas a partir de telencefalollón embrionario de ratón E14.5 o E16.5 C57BL/6 (Janvier Labs).
1. Instalación de software
- Siga las instrucciones para instalar el software desde https://github.com/bsbrl/Autoinjector.
2. Preparación de reactivos y pipetas
- Agarosa: Preparar una agarosa al 3% disolviendo por separado 3 g de agarosa de amplio alcance y 3 g de agarosa de punto de fusión baja en 100 ml de PBS de grado de cultivo celular en dos botellas de vidrio separadas de 200 ml, respectivamente. Conservar a temperatura ambiente durante un máximo de 3 meses.
- Solución de Tyrode: Disolver 1 g de bicarbonato sódico y sal de Tyrode (utilizar el contenido de toda la botella) y 13 ml de 1 M HEPES en 1 L de agua destilada. Ajuste el pH a 7.4. Filtrar la solución a través de un filtro de tapa de botella de 0,2 m.
- Medio de cultivo de rebanadas (SCM): Añadir 10 ml de suero de rata, 1 ml de 2 mM de glutamina, 1 ml de penicilina-estreptomicina (100x), 1 mL de suplemento N-2 (100x), 2 ml de suplemento B27 (50x) y 1 ml de HEPES (pH 7.3) buffer en 84 mL de neurobasal medio. Alícuota 5 mL de SCM en tubos de 15 ml. Conservar a -20oC.
- CO2-Medio de microinyección independiente (CIMM): Preparar 5x DMEM solución de baja glucosa modificada (sin rojo fenol) disolviendo el polvo en 200 ml de agua destilada. Filtrar la solución a través de un filtro de tapa de botella de 0,2 m (para el polvo DMEM, utilice el contenido de toda la botella). Para preparar 100 mL de CIMM, mezclar 20 mL de 5x DMEM solución modificada, 1 ml de tampón HEPES, 1 ml de suplemento N2 (100x), 2 ml de suplemento B27 (50x), 1 ml de penicilina-estreptomicina (100x), 1 ml de 2 mM de glutamina y 74 ml de agua destilada. Almacene la solución a 4oC.
- Tampón de reconstitución: Preparar el tampón de reconstitución disolviendo 262 mM NaHCO3, 0.05 N NaOH, 200 mM HEPES en agua destilada. Esterilice la solución filtrando a través de un sistema de filtro de 0,22 m en una botella de vidrio estéril. Alícuota 500 l de tampón de reconstitución en tubos herméticos de microcentrífuga. Conservar a 4oC.
- Material de tinte de microinyección: Disolver el Dextran con la etiqueta fluorescente en agua destilada libre de RNase (concentración final de 10 g/l). Preparar alícuotas de 5 l y conservar a -20 oC hasta su uso.
- Tire de las pipetas de microinyección de los capilares de vidrio de borosilicato (1,2 mm de diámetro exterior, 0,94 mm de diámetro interior) utilizando el tirador de micropipetas. Proteja las pipetas del polvo. No almacene pipetas durante más de 2 – 3 días. Para este experimento, los parámetros de tracción fueron HEAT: temperatura de rampa +1 – 5; PULL: 100; VEL: 110; DEL: 100. HEAT y VEL son los parámetros que más afectan a la forma y el tamaño de la pipeta.
NOTA: La pipeta de microinyección óptima tiene una punta larga y flexible, para evitar daños celulares durante la microinyección.
3. Preparación de la rebanada de tejido
- Derretir la agarosa de amplio alcance del 3% usando un horno microondas antes de la disección del tejido cerebral. No deje que la agarosa se solidifique manteniendo en un baño de agua a 37 oC antes de la incrustación. Asegúrese de que las pipetas estén protegidas contra el polvo. No almacene pipetas durante más de 2 – 3 días.
- Descongelar una alícuota de SCM y calentar 10 – 12 ml de CIMM y 20 ml de la solución de Tyrode a 37 oC utilizando un baño de agua.
- Mezclar el trazador fluorescente (Dextran-3000 o Dextran-10000-Alexa conjugado; concentración final 5 – 10 g/L) con los otros productos químicos que se van a inyectar. Centrifugar la solución de microinyección a 16.000 x g durante 30 min a 4oC. Recoger el sobrenadante y transferir a un nuevo tubo. Mantenga la solución de microinyección sobre hielo hasta su uso.
- Utilice las cabezas de los embriones de ratón E13.5 – E16.5 para preparar rebanadas de tejido organotípico del telencéfalo. Retire la piel y abra el cráneo usando los fórceps, moviéndose a lo largo de la línea media. Diseccionar el cerebro embrionario del cráneo abierto y eliminar las meninges que cubren el tejido cerebral a partir del lado ventral del cerebro. Deje el cerebro entero diseccionado en la solución de Tyrode en un bloque de calentamiento de 37 oC.
NOTA: Todos los pasos de disección descritos en 3.4 deben realizarse en la solución de Tyrode precalentada. - Verter la amplia gama de agarrose derretida en un molde de incrustación desechable. Cuando la agarosa se enfríe a 38 – 39 oC, transfiera cuidadosamente los cerebros (un máximo de 4) en él utilizando una pipeta Pasteur. Utilice siempre las puntas de corte para este paso.
- Revuelva la agarosa alrededor del tejido usando una espátula o un par de fórceps de Dumont #1 sin tocar el tejido. Dejar que la agarosa se solidifique a temperatura ambiente. Una vez que la agarosa se ha solidificado, recortar el exceso de agarosa que rodea el tejido.
- Llene la bandeja del buffer con PBS. Orientar el cerebro con el eje rostro-caudal del tejido perpendicular a la bandeja (utilizar como punto de referencia las bombillas olfativas, que representan la parte más rostral del cerebro). Cortar rodajas de 250 m con un vibratome.
- Llene un plato de Petri de 3,5 cm con 2 ml de medios precalentados. Con una pipeta Pasteur de plástico, transfiera las rodajas (10 - 15) a este plato. Una vez hecho esto, cambia el plato de Petri con las rodajas a la incubadora de cultivo de rebanadas. Mantener las rodajas a 37oC en una atmósfera humidificada que contenga 40% O2 / 5% CO2 / 55% N2 hasta su uso.
4. Microinyección
- Encienda el ordenador, el microscopio, la cámara del microscopio, los manipuladores, la plataforma de presión y el sensor de presión. Cargue la aplicación haciendo clic en el archivo "launchapp.py" en la carpeta principal descargada de GitHub y especifique la configuración del dispositivo en la pantalla emergente (consulte el paso 1.1 para obtener instrucciones de instalación).
- Cree una presión externa para evitar obstrucciones no deseadas antes de sumergir la pipeta en la solución. Para aplicar presión a la pipeta, deslice la barra de presión de compensación a 24 – 45% y haga clic en Establecer valores. A continuación, ajuste la presión a una presión suficiente girando la perilla de la válvula de presión mecánica a 1 – 2 PSI (69 – 138 mbar) como lo indica el sensor de presión.
- Transfiera las rodajas a un plato petri de 3,5 cm que contenga 2 ml de CIMM precalentado. Coloque las rodajas a microinyeccionar en el centro de la placa Petri. Transfiera el plato Petri a la etapa de microinyección precalentada (37oC).
- Cargue la pipeta de microinyección con una pipeta de plástico de punta larga de 1,4 –1,6 ml (del paso 3.3) utilizando una pipeta de plástico de punta larga. Inserte la pipeta de microinyección en el soporte de la pipeta.
- Usando el aumento más bajo en el microscopio, ponga la rebanada en foco y guíe la micropipeta a este campo de visión (FOV) para que se centre en el mismo plano que el objetivo de la rebanada. Cambie la salida del microscopio a la cámara para ver el FOV en la aplicación.
- Haga clic en el botón de ampliación en la parte superior izquierda de la interfaz para iniciar la calibración del dispositivo. Una ventana le pedirá que seleccione la ampliación. Seleccione el aumento de 10x, o cualquier aumento en el que esté configurado la lente (por ejemplo, 4x, 10x, 20x, 40x) y pulse Ok. El software asume que la lente objetivo interna es 10x (el aumento objetivo de lente más común).
- Reenfoque la punta de la pipeta con la rueda micrométrica del microscopio y haga clic en la punta de la pipeta con el cursor. A continuación, pulse el botón paso 1.1 y pulse OK en la ventana emergente. La pipeta se moverá en la dirección Y. Haga clic en la punta de la pipeta y pulse el botón paso 1.2. Por último, introduzca 45 en el cuadro ángulo de pipeta y pulse el botón Definir ángulo.
- Introduzca los parámetros deseados en el panel Controles automatizados de microinyección. Para la microinyección en progenitores apicales, ajuste la distancia de inyección a 20 – 40 m y la profundidad a 10 – 15 m. Para la microinyección en las neuronas, establezca la distancia de inyección de 30 – 40 m desde el lado basal, y la profundidad a 10 – 30 m dependiendo de lo que se está apuntando. Ajuste siempre la velocidad al 100%. Haga clic en Establecer valores.
NOTA: La distancia de aproximación es la distancia que la pipeta extrae del tejido antes de pasar a la siguiente distancia de inyección, la profundidad es la profundidad en el tejido que va la microinyección, el espaciado es la distancia a lo largo de la línea entre las inyecciones secuenciales, la velocidad es la velocidad de la pipeta en m /s. - Haga clic en el botón Dibujar borde y arrastre el cursor a lo largo de la trayectoria deseada en la ventana emergente para definir la trayectoria de la inyección. Para las células madre progenitoras microinyección, el lado ventral de la superficie del telencéfalo está dirigido como se muestra en la Figura 2A. Lleve la pipeta al inicio de la línea y haga clic en la punta de la pipeta. Haga clic en Ejecutar trayectoria para iniciar la microinyección. Repita este paso para cada plano de inyección dirigido (normalmente para 3 – 4 planos con 40 – 75 inyecciones por plano).
5. Cultivo de tejidos y procesamiento de rebanadas de tejido para inmunofluorescencia
- Preparar la mezcla de colágeno (1,5 mg/ml): A un tubo añadir 1,25 ml de la solución matriz, 0,5 ml de agua destilada, 0,5 ml de solución de 5x DMEM-F12 y 0,25 ml de reconstitución. Manténgalo en hielo hasta su uso.
- Obtener el plato de Petri que contiene las rodajas microinyectedas de la cámara de incubación de cultivo de la rebanada y sumergir las rodajas en la mezcla de colágeno.
- Transfiera las rodajas junto con 200 – 300 l de mezcla de colágeno en un pozo de 14 mm de un plato de fondo de vidrio de 35 mm. Asegúrese de que las rodajas estén cubiertas de muy menos colágeno. Esta configuración permite las condiciones óptimas para la absorción de nutrientes y oxígeno.
- Oriente los sectores mientras se asegura de que haya suficiente espacio entre los sectores utilizando dos pares de fórceps. Incubar el plato de Petri durante 5 min a 37oC utilizando un bloque de calentamiento para permitir que el colágeno se solidifique. Considere esta vez como t - 0 de la referencia cultural de la porción.
- Mueva el plato Petri de vuelta a la incubadora de cultivo de rebanadas durante 40 minutos adicionales. A continuación, agregue 2 ml del SCM precalificado. Los sectores se mantienen en el cultivo hasta el punto de tiempo deseado.
- Saque las rebanadas de la incubadora de cultivo de rebanadas y aspire el SCM. Lave las rodajas incrustadas en colágeno con 1x PBS. Añadir 4% (wt/vol) paraformaldehído (en tampón de fosfato de 120 mM, pH 7.4) y dejar el tejido en RT durante 30 min. A continuación, muévalo a 4 oC para permitir la fijación durante la noche.
- Aspirar la solución de paraformaldehído al día siguiente y realizar lavados 1x PBS. Para eliminar las rebanadas del colágeno, utilice dos pares de fórceps para extraer suavemente las rodajas debajo de un estereomicroscopio.
- Utilice un microondas para derretir la agarosa del punto de fusión bajo del 3% (wt/vol) para procesar las rodajas microine inyectadas. Vierta la agarosa derretida en un molde de incrustación desechable y déjela enfriar a alrededor de 38 – 39 oC.
- Transfiera las rodajas de tejido del paso 5.7 a este molde que contiene baja agarosa de fusión utilizando una pipeta pasteur de plástico. Asegúrese de que el lado pial de la rebanada esté hacia arriba y la superficie ventricular hacia abajo. Si es necesario orientar en consecuencia. Deje que la agarosa se enfríe a RT para solidificarse.
- Recorta la agarosa extra que rodea las rodajas. Oriente el bloque de agarosa para asegurarse de que la superficie de corte es paralela a la cuchilla de corte del vibratomo. Con el vibratome, corte las secciones de 50 m de espesor.
- Llene un plato de 24 pozos con 1x PBS. Transfiera las secciones a este plato usando un pincel de punta fina. Realizar inmunofluorescencia según los protocolos estándar.
La microinyección sirve para rastrear y manipular células madre neurales individuales y su progenie en el tejido vivo y seguir su progresión del linaje en un entorno fisiológico. En este artículo, hemos demostrado el uso del robot de microinyección para apuntar y microinyección microinyección de las rebanadas organotípicas del ratón telecephalon. La Figura 2 ilustra imágenes representativas de las células progenitoras inyectadas con éxito y la Figura 3 ilustra las neuronas recién nacidas inyectadas. Cuando se inyecta con el tinte Dextran Alexa-488 (o Alexa-A555), las células aparecen completamente llenas con el tinte. En cuanto a los progenitores apicales (Figura 2) la imagen confocal permite reconstruir con alta resolución espacial la morfología celular, la presencia -o ausencia- del accesorio apical y basal, y combinar la consulta morfológica con la expresión del marcador. Mediante la combinación de estos criterios, el usuario puede asignar un destino celular específico a las células microinyeccionado y su progenie. En cuanto a la inyección de neuronas, el usuario puede reconstruir la morfología neuronal, incluyendo la estructura y características de la dendrita apical y el axón. La microinyección automatizada puede proporcionar un rendimiento significativamente mayor en comparación con la microinyección manual(Figura 2B). Además, el etiquetado EdU confirma que la viabilidad de la célula no se ve afectada por la automatización(Figura 2C). Mantener la rebanada organotípica en el cultivo permite seguir la progresión del linaje de las células microinyeccionadas (se muestran 4 - 24h en la Figura 2D). Si la solución de microinyección contiene material genético (ADN, ARNm, guías CRISPR-Cas9) o proteínas recombinantes, esto permite estudiar si y cómo la progresión del linaje se ve afectada por la manipulación.
La microinyección en células madre neurales individuales en el tejido proporciona una excelente resolución de una sola célula y por esa razón se ha utilizado para diseccionar la biología celular de la progresión de las células madre neurales y la transición del destino (Figura 3A). La microinyección permite la entrega de mezcla compleja de productos químicos. Anteriormente hemos hecho uso de esta característica para estudiar el acoplamiento de unión en células progenitoras neuronales mediante la mezcla de brecha-unión permeable con la brecha de colorantes fluorescentes impermeables de unión12. Ampliamos el trabajo previo mediante el estudio del acoplamiento de unión en neuronas recién nacidas, inyectando Lucifer Yellow junto con Dextran-A555 (Figura 3B). Como se muestra en la Figura 3B,una proporción de neuronas piramidales recién recién nacidas se acoplan a través de uniones de separación a las neuronas vecinas. Esta observación es consistente con la idea de que las neuronas inmaduras se comunican a través de laseparación-unión 13,14. Además, la orientación a las neuronas muestra que el uso del robot de microinyección se puede generalizar a varios tipos de células en el cerebro de mamífero en desarrollo. Esta configuración experimental será útil para diseccionar la biología celular de las neuronas en el tejido, por ejemplo mediante la entrega de oligopéptidos específicos para interferir con las interacciones proteína-proteína.
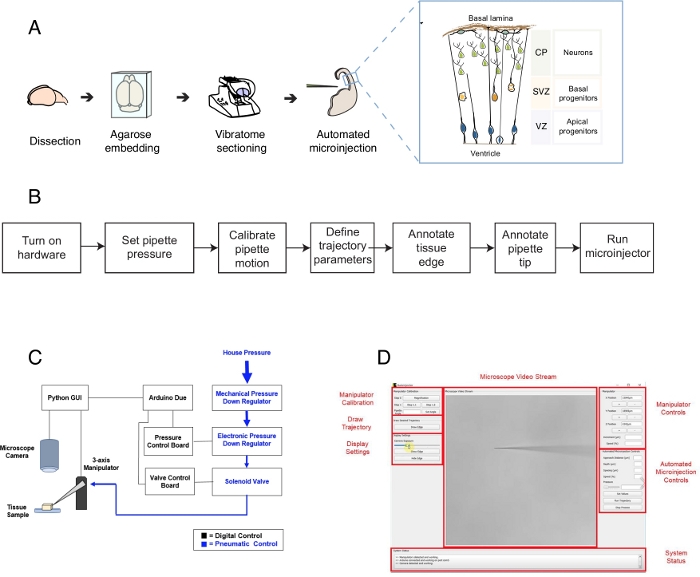
Figura 1: Configuración y protocolo automatizados de microinyección. (A) Protocolo general para la preparación de tejidos y microinyeccións automatizadas utilizando el robot de microinyección. Inserción derecha: Esquema de dibujos animados del ratón Telencephalon dirigido a la microinyección en este protocolo. (B) Diagrama de flujo de los pasos automatizados de microinyección. (C) Esquema del hardware del robot de microinyección. (D) Interfaz gráfica de usuario (GUI) del software utilizado para controlar y operar el robot de microinyección. Esta cifra se adapta a partir de la referencia12. Haga clic aquí para ver una versión más grande de esta figura.
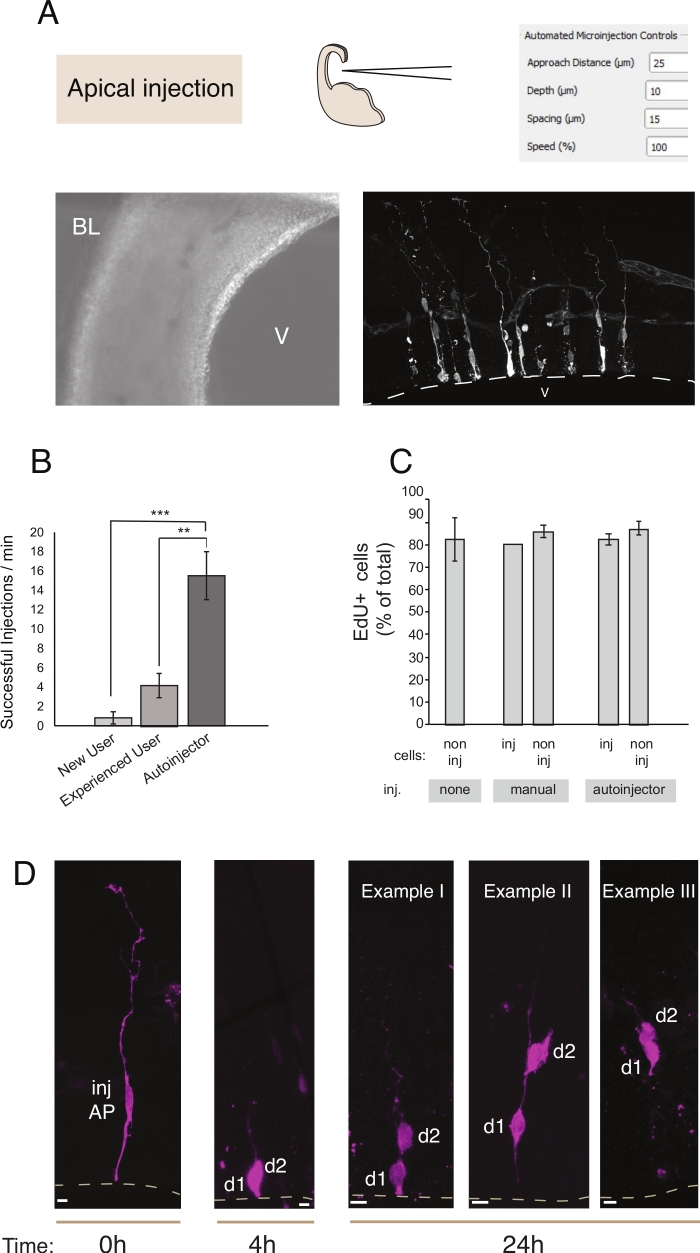
Figura 2: Microinyección robótica en progenitores apicales. Resultados esquemáticos y esperados cuando se utiliza el robot de microinyección para apuntar a los progenitores apicales (AP) a través de la superficie apical (inyección apical). (A) Fila superior. A la izquierda: esquema del proceso. A la derecha: GUI con parámetros relevantes para la inyección apical. En la fila inferior. A la izquierda: imagen de contraste de fase tomada durante el procedimiento de inyección (V: ventrículo; BL: lámina basal). A la derecha: resultados representativos que muestran los AP microinyecados. La línea discontinua representa el ventrículo (V). Barra de escala: 10 m. (B) Inyecciones exitosas por minuto para un usuario principiante en el sistema de microinyección manual, un usuario experimentado en el sistema de microinyección manual y el robot de microinyección. (C) Incorporación de EdU en células microinyeccionadas y en células no inyectadas en el área inyectada. Las rebanadas organotípicas del ratón E14.5 ortófalo dorsal fueron (i) no inyectadas o (ii) sometidas a microinyección manual o automatizada (rebanada inyectada) utilizando Dextran-A488 (para manual y autoinyector). Las rebanadas se mantuvieron en cultura en presencia de EdU durante 24 horas, luego se fijaron y se mancharon para DAPI y EdU. Las células inyectadas y no inyectadas en el área inyectada se puntuaron para la positividad de EdU. (D) Uso del robot de microinyección Trazado de linaje. Un tinte fluorescente (Dx3-A555, magenta) se inyecta en una sola célula madre neural (t a 0 h). El tinte fluorescente se divide en las células hijas (d1, d2) durante la mitosis. Esto permite seguir la progenie de la célula inyectada (t s 4 h y 24 h) y revelar la progresión del linaje a lo largo del tiempo. En el caso de las 24 horas, mostramos varios ejemplos de la progenie que se espera encontrar. Barras de escala: 10 m. Los gráficos en B y C se toman de la referencia12Haga clic aquí para ver una versión más grande de esta figura.
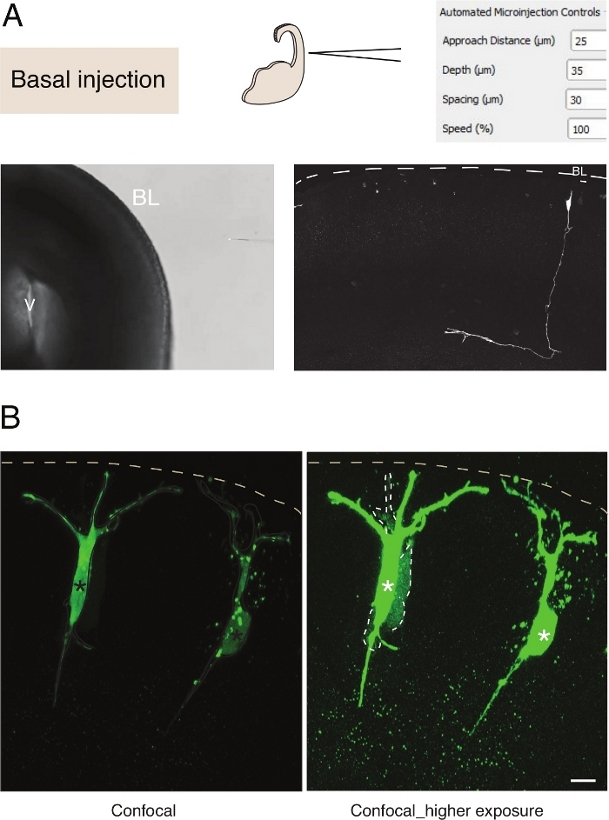
Figura 3: Microinyección robótica en las neuronas. Resultados esquemáticos y esperados cuando se utiliza el robot de microinyección para apuntar a las neuronas piramidales (N) a través de la superficie basal (inyección basal). (A) Fila superior. A la izquierda: esquema del proceso. A la derecha: GUI con parámetros relevantes para la inyección basal. En la fila inferior. A la izquierda: imagen de contraste de fase tomada durante el procedimiento de inyección (V: ventrículo; BL: lámina basal). A la derecha: los resultados representativos que muestran una línea N. Dashed microinyec inyectada representan la lámina basal (BL). Barra de escala: 10 m. (B) Uso del autoinyector para estudiar la comunicación de unión de brechas en el tejido. Las neuronas piramidales se inyectaron con una solución que contiene dos tintes: la brecha unión-impermeable Dx-A555 (no se muestra) y la brecha-unión permeable Lucifer Amarillo (verde). Dx-A555 se limita a la celda de destino (asteriscos), mientras que el LY se difunde a las celdas que están conectadas a través de la unión de separación a la celda de destino (líneas discontinuas). Panel izquierdo: Baja exposición, sólo las células microinyeccionadas son visibles. Panel derecho: La alta exposición permite la visualización de las celdas inyectadas, así como de las celdas acopladas (líneas discontinuas). Barra de escala: 10 m. Haga clic aquí para ver una versión más grande de esta figura.
Archivo suplementario: Solución de problemas de varios errores comunes que surgen durante la microinyección. Haga clic aquí para descargar este archivo.
La microinyección en células madre neurales individuales en el tejido proporciona una excelente resolución de una sola célula y por esa razón se ha utilizado para diseccionar la biología celular de la progresión de las células madre neurales y la transición del destino (Figura 2; véase también10,11,12). El procedimiento automatizado de microinyección se puede realizar en otros tipos de células tanto en ratones embrionarios como en tejido cerebral humano. Los resultados representativos de la microinyección de las neuronas recién nacidas apuntando a la superficie basal del telencéfalo se muestran en la Figura 3.
El principio establecido aquí se puede aplicar para apuntar a varios tipos de células diferentes en cerebros embrionarios de ratón y cerebros humanos. Hemos demostrado previamente que el robot de microinyección también se puede utilizar para apuntar a células progenitoras individuales en el ratón hindbrain y telencephalon y neuronas recién nacidas en el ratón y humano desarrollo neocórtex12. Para obtener los mejores resultados del procedimiento de inyección, se deben optimizar todos los pasos antes de iniciar la inyección. Es importante considerar y optimizar cuidadosamente la preparación de rebanadas de tejido organotípico viables y bien conservadas del tejido cerebral (Figura 1). Es crucial ser rápido en el procedimiento de disección y corte ilustrado en la Figura 1. Para la inyección apical dirigida a los AP, uno debe elegir los sectores que muestran la orientación ideal de la superficie apical. Para la inyección de PUNTOS de acceso, la orientación ideal es la superficie apical perpendicular a la parte inferior de la placa Petri. Cualquier otra orientación será permisiva, sin embargo, la superficie apical perpendicular a la placa Petri proporciona una superficie más amplia para la inyección, aumentando así el éxito de la inyección. Para la inyección en las neuronas, la orientación de la rebanada juega poco o ningún efecto.
Una vez seleccionados los sectores a inyectar, el procedimiento de inyección por rebanada tarda aproximadamente 5 minutos. Teniendo en cuenta que uno trabaja con tejido vivo, es muy recomendable acelerar el procedimiento de inyección. Para ello recomendamos establecer todos los parámetros para la inyección a través de la GUI (Figura 1D) antes de que el tejido esté listo, para reducir cualquier tiempo de espera innecesario. Para solucionar problemas, consulte el archivo suplementario.
En el caso del cultivo de cortes a largo plazo, los pasos posteriores al procedimiento automatizado de microinyección pueden afectar a la salud de las células y, por lo tanto, al experimento. Por lo tanto, se recomienda ejecutar una prueba de control de calidad y optimizar las condiciones de cultivo de sectores. Para evaluar la viabilidad celular después del procedimiento de corte e inyección, realizamos el etiquetado de EdU durante el cultivo y cuantificamos el número de núcleos pignóticos (un proxy para las células apoptóticas) en los cultivos y el tejido inyectado12. Estas cuantificaciones no revelaron ningún impacto significativo de la microinyección en la viabilidad tisular (Figura 2C). Recomendamos ejecutar controles de calidad similares al establecer el corte de tejido organotípico y la tubería de microinyección en el laboratorio.
En comparación con la microinyección manual, el robot de microinyección proporciona varias ventajas. En primer lugar, la curva de aprendizaje para el usuario es menos empinada en comparación con la inyección manual: un nuevo usuario alcanzará una alta competencia después de un número limitado de sesiones, normalmente 1 o 2. En segundo lugar, en el caso de la microinyección manual, una competencia comparable requiere meses de entrenamiento. El procedimiento de inyección es más rápido y eficiente(Figura 2B). Cuantificamos estos parámetros y descubrimos que el robot de microinyección superó a un usuario manual experto con respecto al éxito de la inyección (% de inyección exitosa/número total de inyecciones) y en el número total de inyecciones por unidad de tiempo12. Esto se traduce en un aumento general del 300% de la eficiencia de la inyección (% de inyección exitosa / min) para el robot de microinyección en comparación con un usuario experto. El aumento en la eficiencia fue aún más pronunciado al comparar el robot de microinyección con un usuario principiante y alcanzó el 700%. Por último, pero no menos importante, el robot de microinyección se puede programar fácilmente para explorar sistemáticamente todos los parámetros espaciales. Esto es particularmente ventajoso al adaptar el robot de microinyección para apuntar a nuevas células o tejidos, o cuando se utiliza el robot de microinyección para fines que requieren una resolución espacial diferente.
La construcción del robot de microinyección requiere cambios mínimos en un microscopio de epifluorescencia existente12. Anteriormente hemos proporcionado instrucciones para esta adaptación en https://github.com/bsbrl/Autoinjector. Una vez configurado el hardware, este protocolo proporciona detalles metodológicos clave para realizar con éxito microinyeccións automatizadas. En general, el robot de microinyección tiene una tasa de inyección exitosa de 15,52 + 2,48 inyecciones/min, que es 15 veces mayor que un usuario inexperto (1,09 ± 0,67 inyecciones/min), y 3 veces mayor que un usuario experto (4,95 ± 1,05 inyecciones/min)12. Esta mejora en la tasa de inyección exitosa permite a los usuarios principiantes y expertos inyectar más células en un período de tiempo más corto que es esencial para preservar la viabilidad del tejido. Además, el robot de microinyección es personalizable y la trayectoria, la profundidad de la inyección, el número de inyecciones, el espaciado entre las inyecciones se pueden ajustar mediante la GUI. Estas características permiten que el robot de microinyección se utilice como una herramienta para optimizar experimentos previamente laboriosos, y para explorar experimentos fundamentalmente nuevos que requieren un mayor rendimiento de lo que era posible anteriormente.
Las principales limitaciones del procedimiento de microinyección que describimos aquí están relacionadas con la preparación de rodajas de tejido, un paso crucial que necesita una optimización extensa. Además, la microinyección se basa en la presencia de una superficie que puede ser abordada por la pipeta de vidrio. Esta característica limita el tipo de tejidos y ubicaciones de tejido que se pueden dirigir a través de la microinyección utilizando la configuración actual.
El robot de microinyección actualmente utiliza imágenes de campo brillante y se ha utilizado en preparaciones in vitro de sectores cerebrales. En el futuro, el robot de microinyección podría combinarse con imágenes de 2 fotones para aumentar la especificidad de la segmentación de una sola célula in vivo para el etiquetado molecular o de tinte. Estos esfuerzos ya se han realizado para la electrofisiología de una sola célula15,16. El dispositivo actual requiere la observación manual del procedimiento de microinyección. Las versiones futuras podrían incluir estrategias para limpiar pipetas de microinyección obstruidas17 o la integración de robots de manipulación de fluidos18 para microinycciones múltiples y totalmente autónomas. Estos dispositivos podrían aumentar la escala de microinyección por órdenes de magnitud. La adaptación de algoritmos para el control paralelo de múltiples pipetas de microinyección19 podría permitir la entrega multiplexada de docenas de tintes y reactivos moleculares en las mismas células dentro de los mismos experimentos. Esto tiene el potencial de abrir nuevas vías para el cribado molecular en el tejido.
El robot de microinyección podría utilizarse para etiquetar células identificadas funcionalmente utilizando códigos de barras de ADN o ARN. Esto podría combinarse a su vez con otras técnicas de análisis de células individuales, como la secuenciación de ARN de una sola célula (scRNAseq) y la microscopía electrónica. Nuestros resultados preliminares muestran que las células microinyegadas y su progenie se pueden recuperar y aislar mediante disociación tisular seguida de clasificación FACS (Taverna, resultados inéditos). Las celdas ordenadas FACS se pueden utilizar para scRNAseq. Además, los resultados preliminares muestran que las capacidades de resolución de células únicas del robot de microinyección se pueden utilizar en combinación con el análisis microscópico electrónico para explorar la biología celular de células madre neurales en tejido a alta resolución espacial (Taverna y Wilsch-Br-uninger, resultados inéditos). Estos datos sugieren que el robot de microinyección se puede utilizar como una herramienta para la microscopía de luz correlativa y electrones en el tejido y en un sentido más amplio, para el análisis multimodal de la identidad celular y el comportamiento en el tejido.
La microinyección se basa en el uso de la presión y uno puede permitirse inyectar soluciones con alta complejidad molecular (por ejemplo, un transcriptoma completo). Esta característica de la microinyección ha sido explotada en el pasado para aislar y clonar receptores ligando-gated20. A lo largo de esta línea, el robot de microinyección podría utilizarse para modelar y estudiar rasgos multigénicos a nivel celular. Combinado con una estrategia de subcomulsión, el robot de microinyección también podría utilizarse como plataforma para identificar el conjunto mínimo de genes que impulsan un cierto comportamiento de rasgo/celular. Hasta ahora, el robot de microinyección se ha utilizado para manipular la bioquímica de la célula a través de la entrega de ARNm, ADN o proteínas recombinantes10,21,22. Prevemos una aplicación del robot de microinyección en el sondeo de la biofísica del espacio intracelular, por ejemplo, mediante la entrega de nanomateriales o nanomáquinas que permitan la detección y/o manipulación de las propiedades biofísicas del espacio intracelular.
Los autores no tienen nada que revelar.
Los autores quieren reconocer la Fundación Nomis (ET). SBK reconoce los fondos del departamento de Ingeniería Mecánica, Facultad de Ciencia e Ingeniería, iniciativa MnDRIVE RSAM del departamento de educación superior de la Universidad de Minnesota, Minnesota, Institutos Nacionales de Salud (NIH) 1R21NS103098-01, 1R01NS111028, 1R34NS11654, 1R21NS12886 y 1R21 NS111196. GS fue apoyado por la Beca de Investigación de Posgrado de la Fundación Nacional de Ciencias y la beca de formación NSF IGERT.
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose, Low Melt | Carl Roth | Cat# 6351.2 | |
| Agarose, Wild Range | Sigma-Aldrich | Cat# A2790 | |
| Best-CA 221 Glue | Best Klebstoffe GmbH & Co.KG | Cat# CA221-10ml | |
| B-27 Supplement | Thermo Fisher Scientific | Cat# 17504044 | |
| Cellmatrix Type-IA (Collagen, Type !) | FUJIFILM Wako Chemicals | Cat# 637-00653 | |
| Distilled Water | |||
| DMEM-F12, CO2 independent (w/o Phenol red) | Sigma-Aldrich | Cat# D2906 | |
| DMEM-F12, CO2 independent (with Phenol red) | Sigma-Aldrich | Cat# D8900 | |
| HEPES-NAOH, pH 7.2, 1M (HEPES buffer) | Carl Roth | Cat# 9105.3 | |
| L-Glutamine, 200 mM | Thermo Fisher Sientific | Cat# 25030024 | |
| Mowiol 4-88 | Sigma-Aldrich | Cat# 81381 | |
| N-2 Supplement | Thermo Fisher Scientific | Cat# 17502048 | |
| Neurobasal Medium | Thermo Fisher Scientific | Cat# 21103049 | |
| Nuclease-free water | Thermo Fisher Scientific | Cat# AM9937 | |
| O2 (40%), CO2 (5%), N2 (55%) Mix, 50 liters | |||
| Paraformaldehyde | Merck | Cat# 818715 | |
| PBS | |||
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | Cat# 15140122 | |
| Rat serum | Charles River Laboratories | ||
| Japan | |||
| Sodium bicarbonate (NaHCO3) | Merck | Cat# 106323 | |
| Sodium hydroxide (NaOH) | Merck | Cat# 106482 | |
| Tyrode’s salt | Sigma | Cat# T2145-10x1L) | |
| Equipment | |||
| Borosilicate glass capillaries, 1.2 mm outer diameter x 0.94 mm inner diameter | Sutter Instruments | Cat# BF-120-94-10 | |
| Bottle-top filter system, 500 mL | Corning | Cat# 430769 | |
| Computer PC | |||
| Custom pressure rig | Custom pressure rig | ||
| Electronic pressure regulator | Parker Hannifin | Cat# 990-005101-002 | |
| Falcon tubes, 15 mL | Corning | Cat# 430791 | |
| Falcon tubes, 50 mL | Corning | Cat# 430829 | |
| Fine-tip paintbrush | |||
| Flaming/ Brown micropipette puller | Sutter Instruments | Cat# P-97 | |
| Forceps, Dumont no. 3 | Fine Science Tools | Cat# 11231-30 | |
| Forceps, Dumont no. 5 | Fine Science Tools | Cat# 11255-20 | |
| Forceps, Dumont no. 55 | Fine Science Tools | Cat# 11252-20 | |
| Heating block | Labtech International | Cat # Dri block Digi2 | |
| Inverted fluorescence microscope | Zeiss | Cat# Axiovert 200 | |
| Light source | Olympus | Cat# Highlight 3100 | |
| Manual pressure regulator | McMaster Carr | Cat# 0-60 PSI 41795K3 | |
| Microloader Tips | Eppendorf | Cat# 5242956.003 | |
| Microcontroller | Arduino | Cat# Arduino Due | |
| Microscope camera Hamamatsu Orca Flash 4.0 V3 | |||
| Motorized stage XY for microscope | |||
| Multiwell plate, 24 wells | Nunc | Cat# 142475 | |
| Pasteur pipettes, plastic | |||
| Petri dish, 60 x 15 mm | Greiner | Cat# 628102 | |
| Petri dish, 35 x 10 mm | Nunc | Cat# 153066 | |
| Petri dish, 34 x 14 mm, including Microwell no. 1.5 cover glass | MatTek | Cat# P35G-1.5-14-C | |
| Pipette holder | Warner Instruments | Cat# 64-2354 MP-s12u | |
| Pipette and tips | |||
| Puller filament, 3.0-mm square box filament | Sutter Instrument | Cat# FB330B | |
| Slice culture incubation box | MPI-CBG | Cat# custom made | |
| Solenoid valve | Cat# LHDA053321H-A | ||
| Stereomicroscope | Olympus | Cat# SZX12 | |
| Tabletop centrifuge | Heraeus | Cat# 5431622 | |
| Thermometer | |||
| Three-axis Manipulator | Sensapex Inc | Cat# tree-axis uMP | |
| Vibratome | Leica | Cat# VT1000s | |
| Whole-embryo-culture-system incubator | Ikemoto Company | Cat# RKI-10-0310 | |
| Waterbath | |||
| Software and Algorithms | |||
| Arduino | Arduino | ||
| Fiji | RRID: SCR_002285 | ||
| Python | Python Software foundation | Python 2.7.12 | |
| ZEN | RRID: SCR_013672 |
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Götz, M., Huttner, W. B. The cell biology of neurogenesis. Nature Reviews Molecular Cell Biology. 6 (10), 777-788 (2005).
- Di Lullo, E., Kriegstein, A. R. The use of brain organoids to investigate neural development and disease. Nature Reviews Neuroscience. 18 (10), 573-584 (2017).
- Lancaster, M. A., Knoblich, J. A. Organogenesisin a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Kretzschmar, K., Clevers, H. Organoids: Modeling Development and the Stem Cell Niche in a Dish. Developmental Cell. 38 (6), 590-600 (2016).
- Pepperkok, R. et al. Automatic microinjection system facilitates detection of growth inhibitory mRNA. Proceedings of the National Academy of Sciences of the United States of America. 85 (18), 6748-6752 (1988).
- Pepperkok, R., Lowe, M., Burke, B., Kreis, T. E. Three distinct steps in transport of vesicular stomatitis virus glycoprotein from the ER to the cell surface in vivo with differential sensitivities to GTPγS. Journal of Cell Science. 111 (13), 1877-1888 (1998).
- Pepperkok, R. et al. β-COP is essential for biosynthetic membrane transport from the endoplasmic reticulum to the Golgi complex in vivo. Cell. 74 (1), 71-82 (1993).
- Ansorge, W., Pepperkok, R. Performance of an automated system for capillary microinjection into living cells. Journal of Biochemical and Biophysical Methods. 16 (4), 283-292 (1988).
- Taverna, E., Haffner, C., Pepperkok, R., Huttner, W. B. A new approach to manipulate the fate of single neural stem cells in tissue. Nature Neuroscience. 15 (2), 329-337 (2012).
- Wong, F. K., Haffner, C., Huttner, W. B., Taverna, E. Microinjection of membrane-impermeable molecules into single neural stem cells in brain tissue. Nature Protocols. 9 (5), 1170-1182 (2014).
- Shull, G., Haffner, C., Huttner, W. B., Kodandaramaiah, S. B., Taverna, E. Robotic platform for microinjection into single cells in brain tissue. EMBO Reports. 20 (10), e47880 (2019).
- Jabeen, S., Thirumalai, V. The interplay between electrical and chemical synaptogenesis. Journal of Neurophysiology. 120 (4), 1914-1922 (2018).
- Nagy, J. I., Pereda, A. E., Rash, J. E. Electrical synapses in mammalian CNS: Past eras, present focus and future directions. Biochimica et Biophysica Acta - Biomembranes. 1860 (1), 102-123 (2018).
- Suk, H.J. et al. Closed-loop real-time imaging enables fully automated cell-targeted patch-clamp neural recording in vivo. Neuron. 95 (5), 1037-1047 (2017).
- Annecchino, L. A. et al. Robotic automation of in vivo two-photon targeted whole-cell patch-clamp electrophysiology. Neuron. 95 (5), 1048-1055 (2017).
- Kolb, I. et al. Cleaning patch-clamp pipettes for immediate reuse. Scientific Reports. 6, 35001 (2016).
- Holst, G. L. et al. Autonomous patch-clamp robot for functional characterization of neurons in vivo: development and application to mouse visual cortex. Journal of Neurophysiology. 121 (6), 2341-2357 (2019).
- Kodandaramaiah, S. B. et al. Multi-neuron intracellular recording 1 in vivo via interacting autopatching 2 robots. ELife. 7, 24656 (2018).
- Lubbert, H. et al. cDNA cloning of a serotonin 5-HT1c receptor by electrophysiological assays of mRNA-injected Xenopus oocytes (RNA fractionation/hybrid depletion/hybrid selection/choroid plexus/voltage clamp). Neurobiology. 84 (2) 4332-4336 (1987).
- Florio, M. et al. Human-specific gene ARHGAP11B promotes basal progenitor amplification and neocortex expansion. Science. 347 (6229), 1465-1470 (2015).
- Kalebic, N. et al. CRISPR/Cas9-induced disruption of gene expression in mouse embryonic brain and single neural stem cells in vivo. EMBO Reports. 17 (3), 338-348 (2016).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved