Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Bioengineering
डीएनए का उपयोग कर कोशिकाओं के सरल, सस्ती, और मॉड्यूलर पैटर्निंग
यहां हम डीएनए-प्रोग्राम किए गए आसंजन का उपयोग करके एकल-कोशिका संकल्प पर माइक्रोपैटर्न कोशिकाओं के लिए एक प्रोटोकॉल प्रस्तुत करते हैं। यह प्रोटोकॉल एक ग्लास स्लाइड पर डीएनए ओलिगोन्यूक्लियोटाइड्स के पैटर्न बनाने के लिए एक बेंचटॉप फोटोलिथोग्राफी प्लेटफॉर्म का उपयोग करता है और फिर व्यावसायिक रूप से उपलब्ध पूरक ओलिगोन्यूक्लियोटाइड्स के साथ सेल झिल्ली को लेबल करता है। ओलिगोस के संकरण के परिणामस्वरूप प्रोग्राम किए गए कोशिका आसंजन होतेहैं ।
कोशिकाओं की सापेक्ष स्थिति माइक्रोएनवायरमेंट की एक प्रमुख विशेषता है जो सेल-सेल इंटरैक्शन का आयोजन करती है। एक ही या विभिन्न प्रकार की कोशिकाओं के बीच बातचीत का अध्ययन करने के लिए, माइक्रोपैटर्निंग तकनीक उपयोगी साबित हुई है। कोशिकाओं की डीएनए प्रोग्रामेड असेंबली (डीपीएसी) एक माइक्रोपैटर्निंग तकनीक है जो डीएनए संकरण का उपयोग करके कोशिकाओं के आसंजन को सब्सट्रेट या अन्य कोशिकाओं में लक्षित करती है। डीपीएसी में सबसे बुनियादी संचालन लिपिड-संशोधित ओलिगोन्यूक्लियोटाइड के साथ सेल झिल्ली को सजाने के साथ शुरू होता है, फिर उन्हें एक सब्सट्रेट पर बहता है जिसे पूरक डीएनए दृश्यों के साथ पैटर्न किया गया है। कोशिकाएं चुनिंदा रूप से सब्सट्रेट का पालन करती हैं जहां उन्हें एक पूरक डीएनए अनुक्रम मिलता है। गैर-अनुयायी कोशिकाओं को धोया जाता है, जो अनुयायी कोशिकाओं के पैटर्न का खुलासा करते हैं। अतिरिक्त संचालन में सेल-सब्सट्रेट या सेल-सेल आसंजन के आगे के दौर शामिल हैं, साथ ही डीपीएसी द्वारा बनाए गए पैटर्न को दीर्घकालिक संस्कृति के लिए एम्बेडिंग हाइड्रोगेल में स्थानांतरित करना शामिल है। इससे पहले, सतहों पर ओलिगोन्यूक्लियोटाइड पैटर्निंग के तरीकों और डीएनए दृश्यों के साथ सजाने वाली कोशिकाओं को क्रमशः विशेष उपकरण और कस्टम डीएनए संश्लेषण की आवश्यकता होती है। हम प्रोटोकॉल के एक अद्यतन संस्करण की रिपोर्ट करते हैं, एक मॉड्यूलर प्रारूप का उपयोग करके तैनात एक सस्ती बेंचटॉप फोटोलिथोग्राफी सेटअप और व्यावसायिक रूप से उपलब्ध कोलेस्ट्रॉल संशोधित ओलिगोनॉलियोटाइड्स (सीएमओ) का उपयोग करते हैं। सीएमओ लेबल कोशिकाएं डीएनए-पैटर्न वाले सब्सट्रेट्स के लिए उच्च दक्षता का पालन करती हैं। इस दृष्टिकोण का उपयोग उच्च परिशुद्धता के साथ एक बार में कई सेल प्रकारों को पैटर्न करने और एक बाह्य मैट्रिक्स के भीतर एम्बेडेड माइक्रोटिस्यूस की सरणी बनाने के लिए किया जा सकता है। इस विधि के फायदे में इसका उच्च संकल्प, कोशिकाओं को माइक्रोपैटर्न को बाधित किए बिना त्रि-आयामी माइक्रोएनवायरमेंट में एम्बेड करने की क्षमता और किसी भी कोशिका प्रकार को पैटर्न करने में लचीलापन शामिल है।
ऊतक में एक - दूसरे के संबंध में कोशिकाओं की स्थिति माइक्रोएनवायरमेंट 1 , 2 ,3,4की एक महत्वपूर्ण विशेषता है । जीवित कोशिकाओं को स्थानिक रूप से नियंत्रित व्यवस्थाओं में पैटर्न करने के लिए उपयोग की जाने वाली तकनीकें भेदभाव4,5,6,7,8,सेल मोटिविटी9,मॉर्मोजेनेसिस10,11,12,मेटाबॉलिज्म13,और सेल-सेल इंटरैक्शन7,14 का अध्ययन करने के लिए मूल्यवान प्रयोगात्मक उपकरणहैं। . कोशिकाओं को पैटर्न करने के लिए विभिन्न प्रकार के तरीके मौजूद हैं, प्रत्येक अपने स्वयं के फायदे और कमियां3,4के साथ। एक्सट्रासेलुलर मैट्रिक्स (ईसीएम) प्रोटीन के चिपकने वाले द्वीप बनाने वाले तरीके, जैसे माइक्रोकॉन्टैक्ट प्रिंटिंग और लेजर-कट स्टेंसिल, सरल और स्केलेबल हैं। हालांकि , एक समय में एक या दो से अधिक कोशिका प्रकारों को पैटर्न करना मुश्किल होता है क्योंकि विभिन्न ईसीएम अणुओं के लिए विभिन्न कोशिका प्रकारों के चिपकने वाले गुण अक्सर15 , 16,17समान होते हैं । अधिक जटिल माइक्रोपैटर्न प्रकाश-प्रेरित आणविक सोखने (लिम्प) के साथ बनाया जा सकता है, एक तकनीक जो खूंटी-लेपित क्षेत्रों को अवशोषित करने के लिए यूवी प्रकाश का उपयोग करती है और बाद में प्रोटीन सोखने के लिए अनुमति देती है18,19। इस प्रक्रिया को कई सेल प्रकारों के साथ उच्च-रिज़ॉल्यूशन माइक्रोपैटर्न बनाने के लिए दोहराया जा सकता है। हालांकि, विभिन्न प्रोटीन पैच के लिए कोशिकाओं के क्रॉस-बाइंडिंग हो सकते हैं, जिसके परिणामस्वरूप खराब पैटर्न विशिष्टता19होती है। माइक्रोमैकेनिकल रीकॉन्फिगरेबल कल्चर उपकरणों पर कोशिकाओं को सीडिंग करने जैसे भौतिक तरीके गतिशील नियंत्रण के साथ संरचित सह-संस्कृतियों का निर्माण कर सकते हैं, लेकिन माइक्रोकॉन्टैक्ट प्रिंटिंग या लिम्प14,8के पैटर्न डिजाइन में लचीलेपन के बिना। अन्य तकनीकों के विपरीत, बायोप्रिंटिंगहाइड्रोगेल 20, 21के भीतर कोशिकाओं की त्रि-आयामी व्यवस्था बनासकताहै। हालांकि, बायोप्रिंटेड स्ट्रक्स्ट्रेशन में अन्य माइक्रोपैटर्निंग तकनीकों की तुलना में बहुत कम रिज़ॉल्यूशन होता है, जिसमें सैकड़ों माइक्रोन22के क्रम पर औसत फीचर आकार होता है। एक आदर्श सेल पैटर्निंग विधि में उच्च संकल्प, पैटर्न मल्टीपल सेल प्रकार, उपकरण और रिएजेंट का उपयोग करना होगा जो आसानी से सुलभ हैं, और तीन आयामी (3 डी) सेल संस्कृति के लिए एक हाइड्रोगेल में सफल पैटर्न को एम्बेड करने की क्षमता रखते हैं। इस लेख में, हम सीएमओ-डीपीएसी, एक सेल माइक्रोपैटर्निंग तकनीक पेश करते हैं जो एक सब्सट्रेट के लिए सेल आसंजन को लक्षित करने के लिए डीएनए संकरण के लचीलेपन और गति का उपयोग करता है। इस विधि को अधिक किफायती, मॉड्यूलर और सुलभ बनाने के लिए हमारे पिछले प्रोटोकॉल23,24 से अनुकूलित किया गया है। वर्तमान प्रोटोकॉल का उपयोग करके, किसी भी प्रयोगशाला को किसी भी विशेष उपकरण या विशेषज्ञता के बिना पूरी तरह से कार्यात्मक प्रणाली स्थापित करने में सक्षम होना चाहिए।
कोशिकाओं की डीएनए प्रोग्राम असेंबली (डीपीएसी) एक शक्तिशाली ऊतक इंजीनियरिंग तकनीक है जो कोशिका-कोशिका अंतर और ऊतक ज्यामिति पर सटीक नियंत्रण के साथ एकल-कोशिका संकल्प पर कोशिकाओं को पैटर्न करती है। डीपीएसी में, कोशिका झिल्ली को कोशिका झिल्ली पर संकरित करने के लिए डिज़ाइन किए गए दो लिपिड-संशोधित ओलिगोस का उपयोग करके डीएनए ओलिगोन्यूक्लियोटाइड्स (ओलिगोस) से सजाया जाता है। क्योंकि ओलिगोस हाइड्रोफोबिक लिपिड के लिए संयुग्मित होते हैं, वे तेजी से कोशिका झिल्ली25 में विभाजित होते हैं जहां वे संकरण करते हैं, गैर-सहसंबद्ध अणुओं की शुद्ध हाइड्रोफोबसिटी में वृद्धि करते हैं, और इस तरह सेल सतह26पर अपने जीवनकाल को बढ़ाते हैं। ओलिगोस को सेल की सतह पर इस तरीके से प्रस्तुत किया जाता है जहां वे निर्धारित संरचना, सेल-सेल अंतर औरज्यामिति23,24के साथ परिभाषित 2D या 3 डी सेल पैटर्न बनाने के लिए अन्य कोशिकाओं या डीएनए-कार्यात्मक ग्लास स्लाइड पर पूरक ओलिगोस के साथ संकरण कर सकते हैं। पैटर्न वाले माइक्रोटिस्यूज़ को सतह से एंजाइमेटिक रूप से बंद किया जा सकता है और लंबे समय तक 3 डी संस्कृति के लिए हाइड्रोगेल में एम्बेडेड किया जा सकता है। प्राथमिक कोशिकाओं या स्टेम कोशिकाओं के संयोजन में उपयोग किए जाने पर, कोशिकाओं के परिणामस्वरूप संग्रह मॉर्फोजेनेसिस से गुजर सकते हैं औरऑर्गेनॉइड 23,27, 28में बनसकतेहैं। DPAC को प्रतिस्पर्धी संकेतों6, 29के जवाब में वयस्क तंत्रिका स्टेम सेल भाग्य की गतिशीलता की जांच करने के लिए लागू किया गया है, जो23, 28, 28के स्तन एपीथेलियल कोशिकाओं के स्व-संगठन का अध्ययन करने के लिए और मेसेंचिमल संघनन27के माध्यम से "ऊतक ओरिगेमी" उत्पन्न करने के लिए लागू किया गया है।
DPAC कई सेल आबादी के सटीक प्लेसमेंट के लिए अनुमति देता है और एक्सट्रूज़न-आधारित बायोप्रिंटर (माइक्रोन के आदेश पर)22, 23की तुलना में काफी बेहतर संकल्प है। इसके अलावा, माइक्रोकॉन्टैक्ट प्रिंटिंग जैसे ईसीएम-आधारित पैटर्निंग विधियों के विपरीत, डीपीएसी को ईसीएम-लेपित सतह15,23में विभिन्न सेल प्रकारों के अंतर आसंजन की आवश्यकता नहीं होती है। यह इस बारे में सवालों के जवाब देने के लिए आदर्श है कि ऊतक की संरचना उसके व्यवहार को कैसे प्रभावित करती है, कैसे कोशिकाएं निर्णय लेते समय कई सेलुलर और माइक्रोएनवायरमेंटल संकेतों को एकीकृत करती हैं6,29,और कोशिकाओं के जोड़े एक दूसरे के साथ कैसे बातचीत करते हैं। अन्य माइक्रोपैटरिंग विधियों पर इस विधि का एक लाभ यह है कि इसका उपयोग एक इमेजिंग विमान में 3 डी सेल संस्कृति के लिए किया जा सकता है, जिससे ऊतक स्व-संगठन और ऑर्गेनॉइड मॉर्फोजेनेसिस23,27,30के समय-चूक अध्ययनों को सुविधाजनक बनाया जा सकता है।
इन फायदों के बावजूद, डीपीएसी के सफल कार्यान्वयन के लिए कस्टम ओलिगोन्यूक्लियोटाइड रिएजेंट्स के संश्लेषण और डीएनए पैटर्निंग23,24के लिए विशेष उपकरणों तक पहुंचकीआवश्यकता है, जो व्यापक गोद लेने को सीमित करता है। उदाहरण के लिए, मूल प्रोटोकॉल में उपयोग किए जाने वाले इष्टतम लिपिड-संशोधित ओलिगोस (एलएमओ) को कस्टम संश्लेषित किया जाना चाहिए, जो लिग्नोस्रिक एसिड या पामिटिक एसिड के साथ संशोधित किया जाना चाहिए, औरशुद्ध 26होना चाहिए। इस प्रक्रिया में डीएनए सिंथेसाइज़र और एक उच्च प्रदर्शन वाले तरल क्रोमेटोग्राफी उपकरण के उपयोग के साथ-साथ मिथाइलामाइन जैसे संबद्ध अभिकर्णों की खरीद की आवश्यकता होती है, जो एक नियंत्रित पदार्थ है जो संस्थागत और संघीय दोनों नियमों के अधीन है। एक विकल्प के रूप में, एलएमओ थोक में खरीदा कस्टम किया जा सकता है, लेकिन यह प्रौद्योगिकी में एक महत्वपूर्ण अप-फ्रंट निवेश की आवश्यकता है ।
इन सीमाओं को दूर करने के लिए, हमने डीपीएसी का एक संशोधित संस्करण विकसित किया है जो कस्टम-संश्लेषित एलएमओ के स्थान पर वाणिज्यिक रूप से उपलब्ध कोलेस्ट्रॉल-संशोधित ओलिगोस (सीएमओ) का उपयोग करता है। लागत को और कम करने और मंच के लचीलेपन को बढ़ाने के लिए, हम एक मॉड्यूलर, तीन ओलिगो प्रणाली में बदल गए हैं । प्रत्येक अद्वितीय सेल आबादी के लिए एक नए कोलेस्ट्रॉल-संशोधित ओलिगो का आदेश देने के बजाय, इस प्रोटोकॉल का एक उपयोगकर्ता हर कोशिका आबादी के लिए एक ही कोलेस्ट्रॉल-संशोधित ओलिगो ("यूनिवर्सल एंकर" और "यूनिवर्सल को-एंकर") का उपयोग कर सकता है और फिर एक सस्ती, असंशोधित ओलिगो ("एडाप्टर स्ट्रैंड") को रोजगार देता है जो सार्वभौमिक एंकर और या तो सतह या किसी अन्य सेल प्रकार के एडाप्टर स्ट्रैंड पर समान डीएनए दोनों के साथ संकरित करता है।
मूल डीपीएसी प्रोटोकॉल की एक और सीमा यह थी कि इसने उच्च-रिज़ॉल्यूशन तरल प्रिंटर (जैसे, नैनो ईनबलर, बायोफोर्स नैनोसाइंसेज)23, 24का उपयोग करके डीएनए-पैटर्न वाली स्लाइड बनाई। हालांकि यह उपकरण असाधारण संकल्प और कम रिएजेंट आवश्यकताओं को समेटे हुए है, यह अधिकांश संस्थानों के लिए उपलब्ध नहीं है और इसमें अपेक्षाकृत कम मुद्रण दर (लगभग 1 सुविधा प्रति सेकंड पैटर्न) है। हाल ही में, सतहों पर डीएनए सुविधाओं को पैटर्न करने के लिए दो फोटोलिथोग्राफिक तरीके विकसित किए गए हैं। वायोला और उनके सहयोगियों ने एक पॉलीएक्रीलामाइड और बेंजोफेनोन कोटिंग का उपयोग किया जो यूवी लाइट30के संपर्क में आने पर एकल-फंसे डीएनए ओलिगोस को सहसंबद्ध करता था। इस विधि का उपयोग करके, वे ऊतक मचान बनाने में सक्षम थे जो सेल संकुचन और आत्म-संगठन के परिणामस्वरूप बड़े पैमाने पर, प्रोग्राम किए गए आकार में परिवर्तन करते थे। Scheideler एट अल एक विधि है कि एक सकारात्मक फोटोरेसिस्ट के यूवी जोखिम का उपयोग करता है चुनिंदा एक एल्डिहाइड-कार्यात्मक स्लाइड29के लिए अमीन संशोधित डीएनए ओलिगोस का पर्दाफाश विकसित की है । बेकिंग और अपचय के बाद, अमीन-संशोधित डीएनए को सतह पर सहसंयोजक रूप से बांधा जाता है। इस विधि का उपयोग वयस्क तंत्रिका स्टेम कोशिकाओं की प्रतिक्रिया की जांच करने के लिए किया गया था ताकि स्व-नवीकरण और भेदभाव संकेत प्रस्तुत किए जा सके। यह लेख डीएनए पैटर्न बनाने के लिए Scheideler एट अल के प्रोटोकॉल को अनुकूलित करता है जो सीएमओ-लेबल कोशिकाओं को कैप्चर करेगा। यह फोटोपैटर्निंग प्रोटोकॉल एक साफ कमरे का उपयोग किए बिना किया जा सकता है। यह सस्ती और व्यावसायिक रूप से उपलब्ध उपकरणों का उपयोग करता है जो आसानी से बेंचटॉप या धूम हुड पर तैनात किया जाता है। सस्ती या DIY (यह अपने आप को) फोटोलिथोग्राफी उपकरण का उपयोग साफ कमरे की सुविधाओं के लिए उपयोग के बिना शोधकर्ताओं तक पहुंच बढ़ जाती है और शोधकर्ताओं को समय या संसाधनों का एक बड़ा निवेश के बिना तकनीक की कोशिश करने की अनुमति देता है31,३२। हालांकि, बेहतर संकल्प और कई डीएनए सुविधाओं के संरेखण को आमतौर पर क्लीनरूम सुविधाओं में पाए जाने वाले वाणिज्यिक स्पिन कोटर और मास्क एलाइनर का उपयोग करके प्राप्त किया जा सकता है।
यहां, हम डीएनए-आधारित आसंजन का उपयोग करके एकल-कोशिका संकल्प पर कोशिकाओं को पैटर्न करने के लिए एक विधि का वर्णन करते हैं। सबसे पहले, एक सकारात्मक फोटोरेसिस्ट के साथ फोटोपेटर्निंग का उपयोग एल्डिहाइड-संशोधित ग्लास सब्सट्रेट पर अमीन-संशोधित डीएनए के उच्च-संकल्प पैटर्न बनाने के लिए किया जाता है। इसके बाद, गैर-विशिष्ट सेल अटैचमेंट को कम करने के लिए स्लाइड का इलाज किया जाता है और नमूनों वाले क्षेत्रों में कोशिकाओं को सीमित करने के लिए पीडीएमएस प्रवाह कोशिकाएं बनाई जाती हैं । कोशिकाओं को तब छोटे डीएनए ओलिगोन्यूक्लियोटाइड के साथ लेबल किया जाता है जो कोलेस्ट्रॉल के साथ कार्यात्मक होते हैं और परिणामस्वरूप कोशिका झिल्ली में सम्मिलित होते हैं। इसके बाद कोशिकाओं को डीएनए माइक्रोपैटर्न के ऊपर प्रवाहित किया जाता है । कोशिका-सतह डीएनए और कांच की सतह पर डीएनए के बीच संकरण डीएनए पैटर्न के लिए कोशिकाओं के विशिष्ट आसंजन में परिणाम है । गैर-अनुयायी कोशिकाओं को धोया जाता है, जो अनुयायी कोशिका पैटर्न का खुलासा करते हैं। इस प्रक्रिया को कई सेल प्रकारों के पैटर्न या बहुस्तरीय संरचनाओं को बनाने के लिए दोहराया जा सकता है। यदि वांछित है, तो कोशिकाओं को पूरी तरह से 3 डी सेल संस्कृति के लिए ईसीएम में एम्बेडेड किया जा सकता है।
1. डिजाइन प्रयोग
- सुविधा आकार, सुविधा रिक्ति, शामिल सेल प्रकारों की संख्या, और एक दूसरे के संबंध में कोशिकाओं की व्यवस्था को ध्यान में रखते हुए वांछित प्रयोग की योजना बनाएं। पूरक फ़ाइल 1,प्रयोगात्मक डिजाइन के लिए एक गाइड, और पूरक फ़ाइल 2को देखें, जिसमें उदाहरण ओलिगो दृश्य शामिल हैं।
- कंप्यूटर-एडेड डिजाइन सॉफ्टवेयर का उपयोग करके फोटोमास्क डिजाइन करें। पूरक फाइल 3में एक उदाहरण फोटोमास्क प्रदान किया जाता है।
- एक मानक माइक्रोस्कोप स्लाइड (25 मिमी x 75 मिमी) के आयामों का आयत बनाएं।
- स्लाइड में समान रूप से वितरित चार आयताकार क्षेत्रों को 10 मिमी चौड़ा और 10 मिमी लंबा देखें।
- प्रत्येक क्षेत्र के भीतर, ऐसी विशेषताएं आकर्षित करें जो प्रयोग के लिए वांछित आकार, आकार और अंतर हैं। कोशिकाएं प्रयोग में केवल इन विशेषताओं का पालन करेंगी।
- कई सेल प्रकारों के लिए गठबंधन फोटोमास्क बनाने के लिए, सभी सुविधाओं के सेट के साथ एक मास्टर ड्राइंग बनाएं, फिर प्रत्येक सेल प्रकार के अनुरूप संस्करणों को सहेजें।
- 1.2.3 पारदर्शी और बड़े क्षेत्रों में तैयार की गई सुविधाओं के साथ इस सीएडी ड्राइंग से एक उच्च-रिज़ॉल्यूशन (कम से कम 20,000 डॉट्स प्रति इंच) पारदर्शिता फोटोमास्क ऑर्डर करें।
2. एल्डिहाइड-कार्यात्मक स्लाइडों पर फोटोपैटर्न डीएनए (Scheideler एट अल से अनुकूलितप्रोटोकॉल)
- यदि कई सेल प्रकारों को पैटर्न करते हैं, तो सुविधाओं के संरेखण को सुविधाजनक बनाने के लिए किसी भी डीएनए पैटर्निंग से पहले एल्डिहाइड-कार्यात्मक स्लाइड पर फिड्यूशियल मार्कर बनाएं। पूरक फाइल 1में प्रत्ययी मार्कर बनाने के लिए वैकल्पिक तरीके सुझाए गए हैं ।
- धातु प्रत्ययी मार्कर बनाने के लिए, S1813 सकारात्मक फोटोरेसिस्ट लागू करें जैसा कि चरण 2.3 - 2.11 में वर्णित है। एक फोटोमास्क का उपयोग करें जिसमें बड़ी विशेषताएं हैं जिन्हें बाद में संरेखित करना आसान होगा। इन सुविधाओं को फोटोमास्क के डिजाइन में शामिल करें जिसका उपयोग डीएनए पैटर्निंग के लिए किया जाएगा।
- इलेक्ट्रॉन-गन वाष्पीकरण29का उपयोग करके स्लाइड पर टाइटेनियम की एक पतली फिल्म (100 एंग्स्ट्रॉम) जमा करें। एसीटोन का उपयोग करके अतिरिक्त धातु और फोटोरेसिस्ट को हटा दें, और फिर डीएनए फोटोपैटर्निंग में आगे बढ़ें।
- डीएनए बफर में 5'-अमीन-संशोधित ओलिगो (पानी में सोडियम फॉस्फेट का 50 mm, पीएच = 8.5) का 20 माइक्रोन घोल तैयार करें। सुझाए गए ओलिगो दृश्यों के लिए पूरक फ़ाइल 2 देखें।
नोट: कुछ पैटर्न और अनुप्रयोगों के लिए माइन-संशोधित ओलिगो के 5 माइक्रोन का उपयोग करना संभव है, इसलिए सतह डीएनए एकाग्रता को अनुकूलित करने की आवश्यकता हो सकती है। - एक गर्म प्लेट को 100 डिग्री सेल्सियस तक प्री-हीट करें।
- स्पिन कोटर के रोटर में एल्डिहाइड-कार्यात्मक ग्लास स्लाइड संलग्न करने के लिए डबल-तरफा टेप या वैक्यूम का उपयोग करें।
सावधानी: स्पिन-कोटिंग के दौरान स्लाइड टुकड़ी एक सुरक्षा जोखिम है। हमेशा एक ढक्कन के साथ एक संलग्न कंटेनर में स्पिन कोटर का उपयोग करें, जैसे कि एक्रेलिक बॉक्स।
नोट: कांच को खरोंच करने के लिए हीरे के मुंशी या इसी तरह के कार्यान्वयन का उपयोग करके स्लाइड का एक कोने लेबल करें। यह फोटोरेसिस्ट के बह जाने के बाद स्लाइड पहचान और अभिविन्यास के साथ मदद करता है। - एल्डिहाइड स्लाइड पर सकारात्मक फोटोरेसिस्ट को छोड़ने के लिए डिस्पोजेबल पिपेट का उपयोग करें। यहां तक कि कोटिंग्स के लिए, बीच में एक बड़ी बूंद(पूरक चित्रा 1A)के बजाय स्लाइड में फोटोरेसिस्ट की छोटी बूंदें जोड़ें।
- स्पिन कोटर का उपयोग करके, 30 एस के लिए 3000 आरपीएम पर स्लाइड स्पिन करें।
- स्लाइड को क्रॉसलिंक फोटोरेसिस्ट के लिए 1.5 मिनट (सॉफ्ट बेक) के लिए 100 डिग्री सेल्सियस हॉटप्लेट पर रखें।
- हॉटप्लेट से स्लाइड निकालें। स्लाइड के शीर्ष पर इस प्रयोग के लिए वांछित सुविधाओं के साथ एक फोटोमास्क रखें और फोटोमास्क को कांच के एकटुकड़े (पूरक चित्रा 1B, C)के साथ नीचे तौलें। पूरे सेटअप को एक अपारदर्शी बॉक्स(पूरक चित्रा 1D)में कवर करें। एक यूवी लैंप के साथ बेनकाब (३६५ एनएम तरंगदैर्ध्य, ३६० एमडब्ल्यू, स्लाइड से 5 इंच, कुल उज्ज्वल ऊर्जा घनत्व १०० mJ/सेमी2)2 मिनट के लिए ।
नोट: यूवी लाइट फोटोमास्क के पारदर्शी क्षेत्रों के नीचे फोटोरेसिस्ट में बहुलक बांड तोड़ देगा, उन क्षेत्रों का निर्माण करेगा जहां डीएनए बाद में पालन करने में सक्षम होगा। - 3-5 मिनट(पूरक चित्रा 1E)के लिए डेवलपर समाधान में विसर्जित करके स्लाइड विकसित करें।
- पानी के साथ अतिरिक्त डेवलपर समाधान को कुल्ला करें। हवा या नाइट्रोजन की धारा के नीचे सूखी। (पूरकचित्रा 1F)।
- पुष्टि करें कि माइक्रोस्कोप के नीचे स्लाइड को देखकर फोटोलिथोग्राफी सफल हुई। क्योंकि फोटोरेसिस्ट यूवी-लाइट सेंसिटिव है, इसलिए इस स्टेप को जल्दी करें और फिर अन्य स्लाइड्स (यदि लागू हो) तैयार करते समय स्लाइड को अंधेरे में स्टोर करें ।
नोट: एक सफलतापूर्वक पैटर्न स्लाइड प्रत्येक सुविधा के लिए तेजी से परिभाषित किनारों होना चाहिए, कोई खुर, और किनारों पर कोई सुविधा विरूपण । पूरक चित्रा 2 एमें सही और गलत फोटोलिथोग्राफी के उदाहरण दिए गए हैं। यदि फोटोलिथोग्राफी वांछित सुविधा गुणवत्ता प्रदान नहीं करती है तो समस्या निवारण सुझावों के लिए तालिका 1 देखें। - स्लाइड के प्रत्येक फोटोपैटर्न्ड क्षेत्र पर 20 माइक्रोन-संशोधित ओलिगो समाधान (चरण 2.1) की एक बूंद जोड़ें। धीरे-धीरे पूरे क्षेत्र में बूंद फैलाने के लिए एक पिपेट टिप का उपयोग करें, स्लाइड को खरोंच न करने के लिए सावधान रहें। (पूरकचित्रा 1G)।
- एक 65-70 डिग्री सेल्सियस ओवन में स्लाइड सेंकना जब तक डीएनए समाधान पूरी तरह से स्लाइड सतह पर सूख गया है (के बारे में 1 घंटे) ।
- एक 15 सेमी सेल-कल्चर डिश में पैटर्न, बेक्ड स्लाइड रखकर अपचय अमीनिंग करें और एक शेकर के शीर्ष पर एक धुएं के हुड में रखें। 100 मिलीग्राम सोडियम बोरोहाइड का वजन करें। एक धूम हुड में, फॉस्फेट-बफर खारा (पीबीएस) के 40 एमएल जोड़ें, धीरे-धीरे मिलाएं, और पैटर्न वाली स्लाइड वाले पकवान में जोड़ें। प्रतिक्रिया कोमल मिलाते हुए के साथ 15 मिनट के लिए आगे बढ़ने दें ।
नोट: ओलिगो पर अमीन पहले स्लाइड सतह पर एल्डिहाइड के साथ एक शिफ बेस बनाता है। यह एक प्रतिवर्ती सहसंयोजक बंधन है जिसे डीपीएसी में उपयोग करने से पहले एक अपरिवर्तनीय बंधन में परिवर्तित किया जाना चाहिए। एक कम करने वाले एजेंट (सोडियम बोरोहाइड्राइड) के अलावा शिफ बेस को अपचय अमीन द्वारा एक माध्यमिक अमीन में परिवर्तित करता है।
सावधानी: पानी के साथ सोडियम बोरोहाइड्राइड की प्रतिक्रिया हाइड्रोजन गैस बनाती है और प्रतिक्रिया शुरू होने के बाद घंटों या दिनों तक ऐसा करती रहेगी। एक धूम हुड में अपचयी अमीनेशन चरण करें और सभी सोडियम बोरोहाइड्राइड समाधान अपशिष्ट को कम से कम 24 घंटे के लिए धूम हुड में खुले या शिथिल कैप्ड कंटेनर में रखें। - पानी में 0.1% सोडियम डॉडेक्सिल सल्फेट (एसडीएस) के साथ दो बार धोने से अप्रतिबद्ध डीएनए निकालें, फिर आसुत पानी के साथ तीन बार। नाइट्रोजन या हवा की धारा के नीचे स्लाइड सुखाएं।
- शेष फोटोरेसिस्ट को हटाने के लिए एसीटोन के साथ स्लाइड कुल्ला।
नोट: इस बिंदु पर, डीएनए अपरिवर्तनीय और सहसंयोजक स्लाइड से जुड़ा हुआ है और सभी unreacted aldehyde कार्यात्मक समूहों शराब में बदल दिया गया है । फोटोरेसिस्ट की अब जरूरत नहीं है। - यदि कई ओलिगो पैटर्न किए जाएंगे, तो चरण 2.4 पर लौटें, फोटोमास्क को प्रत्ययी अंकों के साथ संरेखित करें, और दोहराएं।
नोट: प्रयोग यहां रुका जा सकता है । एक वैक्यूम डेसिकेटर में स्लाइड स्टोर करें। शुष्क परिस्थितियों में, स्लाइड गुणवत्ता के नुकसान के बिना 3 महीने तक संग्रहीत किया जा सकता है।
3. स्लाइड हाइड्रोफोबिक (वैकल्पिक) (टोडहंटर एट अल से अनुकूलितप्रोटोकॉल) बनाएं)
नोट: यह लाभप्रद है, लेकिन आवश्यक नहीं है, स्लाइड की सतह रसायन विज्ञान को संशोधित करने के लिए इसे और अधिक निष्क्रिय और हाइड्रोफोबिक प्रदान करने के लिए । इन सतहों पर गैर-विशिष्ट कोशिका लगाव कम होजाता है,जिससे कोशिकाओं के गैर-विशिष्ट बाध्यकारी को स्लाइड के गैर-पैटर्न वाले क्षेत्रों में समाप्त किया जाता है। इसके अतिरिक्त, यदि पैटर्न वाली कोशिकाओं को अंततः हाइड्रोगेल के भीतर एम्बेडेड किया जाएगा और स्लाइड से स्थानांतरित कर दिया जाएगा, तो सतह उपचार विरूपण या फाड़ के बिना स्लाइड में सेल से लदे हाइड्रोगेल के विश्वसनीय आंदोलन के लिए आवश्यक है। (ट्राइडेफल्लोरो-1, 1, 2-टेट्राहाइड्रोक्टिल) डाइमेथाइल्च्लोरोसिलीन के साथ सिलनाइजिंग स्लाइड सतह पर हाइड्रोफोबिक फ्लोरोल्किल समूहों की उपस्थिति में परिणाम देता है।
सावधानी: एसिटिक एसिड और मेथिलीन क्लोराइड धुएं के संपर्क को रोकने के लिए रासायनिक धुएं हुड में 3.1 से सभी चरणों को करें।
- कुल्ला 10% एसिटिक एसिड के साथ स्लाइड और फिर एक हवा धारा के तहत सूखी।
- ग्लास कॉप्लिन जार में, 60 एमएल मेथिलीन क्लोराइड (डाइक्लोरोमेथेन), ट्राइथाइलमाइन के 0.6 एमएल और 0.6 एमएल (ट्राइडेफाफ्लोरो-1, 1, 2, 2-टेट्राहाइड्रोक्टिल) डिमेथाइलक्लोरोसिलीन का समाधान तैयार करें। मिश्रण करने के लिए एक धातु स्पैटुला के साथ हिलाओ।
नोट: ये अभिकर् ता पानी के प्रति संवेदनशील होते हैं। उन्हें शुष्क परिस्थितियों में संग्रहित किया जाना चाहिए और यथासंभव ताजा उपयोग किया जाना चाहिए। - सिलेन समाधान युक्त कोप्लिन जार में स्लाइड जोड़ें। एक कक्षीय शेखर पर Coplin जार प्लेस (60-80 आरपीएम के लिए सेट) और silane की प्रतिक्रिया और स्लाइड 15 मिनट के लिए प्रगति करने के लिए अनुमति देते हैं ।
- सिलेन समाधान से स्लाइड को हटाने के लिए धातु संदंश का उपयोग करें। स्लाइड से अतिरिक्त सिलेन को हटाने के लिए 1 मिनट के लिए मेथिलीन क्लोराइड युक्त कोप्लिन जार में स्मूज स्लाइड।
- इथेनॉल युक्त 50 एमएल शंकु नली में स्लाइड विसर्जित करें। आंदोलन करना। स्लाइड को 50 एमएल शंकु नली में विसर्जित करें जिसमें आसुत पानी होता है। आंदोलन करना।
नोट: मेथिलीन क्लोराइड और पानी गलत नहीं हैं, इसलिए अंतिम पानी कुल्ला से पहले अतिरिक्त मेथिलीन क्लोराइड को हटाने के लिए इथेनॉल कुल्ला की आवश्यकता होती है। - स्लाइड को पानी से निकालकर निरीक्षण करें। स्लाइड काफी शुष्क होना चाहिए, किसी भी पानी की बूंदों के साथ 90 डिग्री से अधिक का संपर्क कोण होता है। स्लाइड को पूरी तरह से सूखने दें और उपयोग होने तक वैक्यूम डेसिकेटर में स्टोर करें।
नोट: प्रयोग यहां रुका जा सकता है । सूखी परिस्थितियों में स्लाइड स्टोर करें।
4. प्रयोग के लिए पीडीएमएस प्रवाह कोशिकाओं और स्लाइड तैयार करें
नोट: आयताकार पीडीएमएस प्रवाह कोशिकाओं का उपयोग स्लाइड के पैटर्न वाले क्षेत्रों पर कोशिकाओं को केंद्रित करने के लिए किया जाता है। 3 डी में सुसंस्कृत प्रयोगों के लिए, प्रवाह कोशिकाएं हाइड्रोगेल के लिए एक मोल्ड बनाती हैं।
- पीडीएमएस प्रवाह कोशिकाओं के लिए मोल्ड के रूप में उपयोग करने के लिए एसयू-8 मास्टर बनाएं।
- प्री-हीट हॉटप्लेट 95 डिग्री सेल्सियस तक।
- एक सिलिकॉन वेफर में एसयू-8 2075 का 5 एमएल जोड़ें।
- स्पिन कोट 10s के लिए ५०० आरपीएम पर वेफर पर एसयू-8, 30 के लिए १,००० आरपीएम के बाद । इससे 34 की ऊंचाई34में 240 माइक्रोन तक की विशेषताएं पैदा होनी चाहिए .
- सॉफ्ट कम से कम 45 मिनट के लिए हॉटप्लेट पर वेफर बेक करें।
- हॉटप्लेट से वेफर निकालें। वेफर के शीर्ष पर फोटोमास्क (पूरक फ़ाइल 4)(पायस साइड डाउन देखें) रखें और फोटोमास्क और स्लाइड के बीच संपर्क सुनिश्चित करने के लिए इसे ग्लास डिस्क के साथ तौलें।
- 350 mJ/सेमी2के एक उज्ज्वल ऊर्जा घनत्व के लिए यूवी प्रकाश (365 एनएम) के साथ बेनकाब करें।
- 12-15 मिनट के लिए हॉटप्लेट पर वेफर सेंकना।
- चौड़े कांच के कंटेनर में वेफर रखें। एसयू-8 डेवलपर समाधान के साथ कवर वेफर। एक शेखर पर रखें और कम से कम 15 मिनट के लिए आंदोलन करते हुए विकसित करें।
- डेवलपर समाधान से वेफर को हटाने के लिए संदंश का उपयोग करें। एक धार की बोतल से अधिक डेवलपर समाधान छिड़ककर 5 एस के लिए कुल्ला। कुल्ला करने के लिए आइसोप्रोपिल अल्कोहल के साथ स्प्रे करें। यदि कोई सफेद तेज़ दिखाई देता है, तो डेवलपर समाधान पर वेफर वापस करें और लंबे समय तक विकसित करें।
- हवा या नाइट्रोजन की धारा के नीचे सूखी वेफर।
- 5 मिनट के लिए स्लाइड सेंकना।
नोट: एक बार मास्टर वेफर बनाया गया है, जब तक सुविधाओं को बरकरार रखा गया है, तब तक इसे अनिश्चित काल के लिए पुन: इस प्रकार पुन: इस् याकि किया जा सकता है।
- पीडीएमएस तैयार करें।
- एक वजन नाव में, 10:1 अनुपात (द्रव्यमान द्वारा) में पॉलीडिमिथाइलसिलोक्सेन इलास्टोमर और क्रॉसलिंकर जोड़ें। यहां तक कि मिश्रण सुनिश्चित करने के लिए सख्ती से हिलाओ।
- डी-गैस पीडीएमएस एक वैक्यूम डेसिकेटर में 15-30 मिनट के लिए जब तक कोई और बुलबुले दिखाई देते हैं।
- मास्टर वेफर को 15 सेमी टिश्यू कल्चर डिश में रखें। वेफर के ऊपर पीडीएमएस डालो। यदि बुलबुले दिखाई देते हैं, तो कुछ मिनटों के लिए वैक्यूम डेसिकेटर में डी-गैस।
- 3 घंटे के लिए 60 डिग्री सेल्सियस ओवन में सेंकना।
नोट: बेकिंग के बाद, पीडीएमएस प्रवाह कोशिकाओं को अनिश्चित काल के लिए बेंचटॉप पर संग्रहीत किया जा सकता है।
- प्रयोग के लिए पीडीएमएस फ्लो सेल तैयार करें।
- सीएमओ-डीपीएसी प्रयोग शुरू करने से कुछ ही समय पहले, मास्टर वेफर से पीडीएमएस प्रवाह कोशिकाओं की आवश्यक संख्या में कटौती करें। प्लाज्मा सतह हाइड्रोफिलिक प्रदान करने के लिए 90 एस के लिए 10 सीसी/मिनट कमरे हवा के साथ ऑक्सीकरण।
- प्रत्येक व्यक्ति प्रवाह सेल को काट लें ताकि प्रत्येक तरफ 1-2 मिमी पीडीएमएस शेष रहे, फिर एक इनलेट और आउटलेट बनाने के लिए प्रवाह सेल के ऊपर और नीचे को खोलें।
- चरण 2 और 3 में बनाई गई नमूनों की स्लाइड को पुनः प्राप्त करें। फोटोमास्क के शीर्ष पर संरेखित करें।
- संदर्भ के रूप में फोटोमास्क का उपयोग करना, प्रत्येक नमूनों वाले क्षेत्र के स्थान में स्लाइड पर पीडीएमएस प्रवाह कोशिकाओं को रखें।
- प्रत्येक प्रवाह सेल के इनलेट में फॉस्फेट बफर नमकीन (पीबीएस) + 1% गोजातीय सीरम एल्बुमिन (बीएसए) के 50 माइक्रोल जोड़ें, जैसा कि पूरक चित्रा 1Hमें दिखाया गया है। इस बात की पुष्टि करें कि फ्लो सेल पूरी तरह से पीबीएस +1% बीएसए से भरा हुआ है और कोई बड़े बुलबुले नहीं हैं। चरण 5 और 6 पर तुरंत आगे बढ़ें।
नोट: बीएसए के साथ अवरुद्ध स्लाइड सतह के लिए गैर विशिष्ट सेल आसंजन को कम करता है ।
5. कोलेस्ट्रॉल संशोधित डीएनए के साथ लिफ्ट और लेबल कोशिकाओं
- कोलेस्ट्रॉल संशोधित डीएनए समाधान तैयार करें।
- प्रयोग में कोशिकाओं के प्रत्येक सेट के लिए, एक एडाप्टर स्ट्रैंड के 100 माइक्रोन स्टॉक समाधान के 3 माइक्रोन के साथ कोलेस्ट्रॉल-संशोधित यूनिवर्सल एंकर स्ट्रैंड के 100 माइक्रोन स्टॉक समाधान के 3 माइक्रोन को एक साथ मिलाएं। 1 मिनट के लिए इनक्यूबेट। इससे ओलिगोस को पहले से संकरित किया जा सकेगा। 4 माइक्रोन यूनिवर्सल एंकर + एडाप्टर समाधान बनाने के लिए फॉस्फेट-बफर नमकीन (पीबीएस) के 69 माइक्रोन जोड़ें।
- प्रयोग में कोशिकाओं के प्रत्येक सेट के लिए, पीबीएस के 12 माइक्रोन में 100 माइक्रोन यूनिवर्सल कोलेस्ट्रॉल-संशोधित सह-एंकर स्ट्रैंड स्टॉक समाधान के 3 माइक्रोन जोड़ें, जो 20 माइक्रोन घोल बनाते हैं।
- सिंगल-सेल सस्पेंशन (एस) तैयार करें।
- अनुयायी कोशिकाओं के लिए, संस्कृति फ्लास्क से कोशिकाओं को हटाने के लिए ट्राइप्सिन या अन्य वियोजन एजेंट का उपयोग करें। कोशिकाओं को गोली मारने के लिए ट्राइप्सिन और अपकेंद्रित्र को बेअसर करने के लिए संस्कृति मीडिया जोड़ें। गैर-पालनकारी कोशिकाओं के लिए, कोशिका निलंबन और कोशिकाओं को गोली मारने के लिए अपकेंद्रित्र इकट्ठा करें।
- बर्फ-ठंडे पीबीएस या सीरम-मुक्त मीडिया के 1 मिलील में सेल पेलेट को फिर से रीसुस्ट करें। 1-3 मिलियन कोशिकाओं को 1.5 एमएल माइक्रोसेंट्रफ्यूज ट्यूब में स्थानांतरित करें। 4 मिनट के लिए 160 x ग्राम पर सेंट्रलाइज।
नोट: यदि उपयोग किए जा रहे सेल प्रकार का उपयोग किया जा रहा है, तो अवांछित सेल एकत्रीकरण को कम करने के लिए सभी धोने के चरणों के लिए कैल्शियम और मैग्नीशियम आयनों के बिना पीबीएस का उपयोग करें। यदि व्यवहार्यता सेल प्रकार के उपयोग के लिए एक विशेष चिंता का विषय है, तो पीबीएस के बजाय सीरम-मुक्त मीडिया का उपयोग करें। भ्रूण गोजातीय सीरम युक्त मीडिया सेल लेबलिंग के लिए सिफारिश नहीं की जाती है क्योंकि यह लिपिड-संशोधित ओलिगोस के समावेश में बाधा डाल सकता है। 35
- कोशिकाओं को कोलेस्ट्रॉल-संशोधित ओलिगोस के साथ लेबल करें।
- बर्फ-ठंडे पीबीएस या सीरम-मुक्त मीडिया के 75 माइक्रोल में सेल पेलेट को फिर से रीसस्ट करें। कोशिका व्यवहार्यता को अधिकतम करने और कोशिका की सतह से कोलेस्ट्रॉल-संशोधित ओलिगोस के नुकसान को कम करने के लिए पूरे लेबलिंग और धोने की प्रक्रिया में कोशिकाओं को एक बर्फ बाल्टी में रखें।
नोट: डीएनए जोड़ने से पहले कोशिकाओं को फिर से खर्च करना यह सुनिश्चित करता है कि डीएनए का वितरण कोशिका आबादी में एक समान है । - सेल सस्पेंशन युक्त माइक्रोसेंट्रफ्यूज ट्यूब में चरण 5.1.1 में बनाए गए 4 माइक्रोनएम यूनिवर्सल एंकर + एडाप्टर सॉल्यूशन के 75 माइक्रोल जोड़ें। पाइपिंग करके अच्छी तरह मिलाएं। बर्फ पर 5 मिनट के लिए इनक्यूबेट।
- माइक्रोसेंट्रफ्यूज ट्यूब के लिए यूनिवर्सल सह एंकर समाधान के 15 μL जोड़ें । पाइपिंग करके अच्छी तरह मिलाएं। बर्फ पर 5 मिनट के लिए इनक्यूबेट।
- सेल निलंबन से अतिरिक्त ओलिगोस निकालें। माइक्रोसेंट्रफ्यूज ट्यूब में बर्फ-ठंडे पीबीएस या सीरम-मुक्त मीडिया का 1 एमएल जोड़ें। एक P1000 पिपेट के साथ मिलाएं। 4 डिग्री सेल्सियस पर 4 मिनट के लिए 160 x ग्राम पर सेंट्रलाइज। सुपरनेट को त्याग दें। दो बार और दोहराएं।
नोट: यदि कोशिकाओं को झुरमुट होने का खतरा है, तो अंतिम धोने से पहले 40 माइक्रोन फ़िल्टर के माध्यम से सेल निलंबन पारित करें। यदि कोशिकाओं को माइक्रोसेंट्रफ्यूज ट्यूब के किनारे पर सोखने का खतरा है, तो केसिन के साथ ट्यूब को पूर्व-अवरुद्ध करने पर विचार करें।
- बर्फ-ठंडे पीबीएस या सीरम-मुक्त मीडिया के 75 माइक्रोल में सेल पेलेट को फिर से रीसस्ट करें। कोशिका व्यवहार्यता को अधिकतम करने और कोशिका की सतह से कोलेस्ट्रॉल-संशोधित ओलिगोस के नुकसान को कम करने के लिए पूरे लेबलिंग और धोने की प्रक्रिया में कोशिकाओं को एक बर्फ बाल्टी में रखें।
6. डीएनए लेबल कोशिकाओं पैटर्न
- कम से कम 25 मिलियन कोशिकाओं/एमएल का सेल-सघन समाधान बनाने के लिए बर्फ-ठंडे पीबीएस या सीरम-मुक्त मीडिया में कोशिकाओं को फिर से खर्च करें।
नोट: स्टेप 4 में वर्णित 10 मिमी x 15 मिमी x 200 माइक्रोन पीडीएमएस फ्लो कोशिकाओं में से चार का उपयोग करके एक स्लाइड के लिए, इस घने सेल निलंबन के बारे में 100 माइक्रोन की आवश्यकता है। यद्यपि इनमें से अधिकांश कोशिकाएं पैटर्न का पालन नहीं करेंगी और अंततः खारिज कर दी जाएंगी, पैटर्न पर कोशिकाओं का एक अत्यंत केंद्रित समाधान होने से नाटकीय रूप से सेल पैटर्निंग की दक्षता में सुधार होता है। - स्लाइड उठाओ और इसे थोड़ा झुकाएं। पैटर्न वाली स्लाइड पर प्रत्येक प्रवाह कोशिका के इनलेट में सेल सस्पेंशन के 25 माइक्रोन जोड़ें। आउटलेट से पीबीएस + 1% बीएसए समाधान निकालें, सेल निलंबन को पीडीएमएस फ्लो सेल भरने की अनुमति देता है। बर्फ पर या कमरे के तापमान पर 30 एस के लिए इनक्यूबेट।
नोट: इस बिंदु पर, एक माइक्रोस्कोप के तहत प्रवाह कोशिका को देखते हुए कोशिकाओं के बीच दिखाई अंतराल के लिए थोड़ा के साथ घनी पैक कोशिकाओं को दिखाना चाहिए । पूरक चित्रा 2Bदेखें । - स्लाइड के आउटलेट से सेल निलंबन के 5 माइक्रोन को एस्पिरेट करें और इसे वापस इनलेट में जोड़ें। प्रति प्रवाह कोशिका 10 बार दोहराएं।
नोट: डीएनए पैटर्न स्लाइड करने के लिए सीएमओ लेबल कोशिकाओं के आसंजन लगभग तात्कालिक है । पैटर्न पर कोशिकाओं को कई बार बहने की संभावना बढ़ जाती है कि एक कोशिका एक दिए गए डीएनए स्थान पर प्रवाहित होगी और कब्जा कर लिया जाएगा । - अतिरिक्त कोशिकाओं को धोने के लिए प्रत्येक प्रवाह कोशिका के प्रवेश में धीरे-धीरे पीबीएस या सीरम-मुक्त मीडिया पिपेट करें। आउटलेट से सेल निलंबन ले लीजिए। 2-4 बार दोहराएं या जब तक माइक्रोस्कोप के नीचे स्लाइड का एक दृश्य निरीक्षण इस बात की पुष्टि नहीं करता है कि कोई अतिरिक्त कोशिकाएं शेष नहीं हैं।
नोट: यह पहले धोने से अतिरिक्त कोशिकाओं को बचाने के लिए लाभप्रद हो सकता है । यदि पैटर्निंग दक्षता असंतोषजनक है, तो अतिरिक्त कोशिकाओं को अधिक सेल-सघन समाधान बनाने के लिए पीबीएस की कम मात्रा में अपकेंद्री और पुनर्कृतीय किया जा सकता है, और फिर प्रक्रिया को चरण 6.2 से दोहराया जा सकता है। - पैटर्न में कोशिकाओं के प्रत्येक सेट के लिए चरण 6.1-6.4 दोहराएं। पैटर्न के लिए जिसमें कई सेल प्रकार सीधे सतह टेम्पलेट द्वारा पैटर्न किए जाते हैं, पैटर्न के कम से कम प्रचुर मात्रा में सेल प्रकार के साथ शुरू करें और सबसे प्रचुर मात्रा में सेल प्रकार के साथ समाप्त करें।
नोट: कोशिकाओं को पूल करने के बजाय सेलुलर असेंबली के प्रत्येक दौर को क्रमिक रूप से करने की सलाह दी जाती है, यहां तक कि उन परिस्थितियों में भी जहां कोशिकाओं को ऑर्थोगोनल डीएनए दृश्यों के साथ लेबल किया जाता है। कोशिकाओं को पूल करना प्रत्येक कोशिका की आबादी को प्रभावी ढंग से पतला करता है और पैटर्निंग दक्षता को कम करता है। - सेल असेंबली का अंतिम दौर पूरा होने के बाद, अगले चरण विशिष्ट प्रयोग के आधार पर भिन्न होंगे। यदि कोशिकाओं को गिलास पर रहने का इरादा कर रहे हैं, एक पेट्री स्लाइड युक्त पकवान के लिए मीडिया जोड़ें, और फिर धीरे से pdMS प्रवाह कोशिकाओं को नज करने के लिए संदंश का उपयोग स्लाइड से दूर । यदि कोशिकाओं को हाइड्रोगेल में एम्बेडेड किया जाएगा और 3 डी में सुसंस्कृत किया जाएगा, तो चरण 7 पर आगे बढ़ें।
7. 3 डी संस्कृति के लिए हाइड्रोगेल में स्थानांतरण (वैकल्पिक)
- 2% DNase युक्त एक हाइड्रोगेल अग्रदूत समाधान तैयार करें।
नोट: समाधान की संरचना प्रायोगिक सेटअप के आधार पर भिन्न होगी। मैट्रिक्सेल और मैट्रिगेल और कोलेजन के मिश्रण मैं इस प्रोटोकॉल में अच्छी तरह से काम करता हूं, लेकिन अन्य हाइड्रोगेल भी संभव हैं। - प्रत्येक प्रवाह सेल के इनलेट में 2% DNase युक्त हाइड्रोजेल समाधान के 50 माइक्रोन जोड़ें। आउटलेट से अतिरिक्त तरल पदार्थ को एस्पिरेट करें, हाइड्रोगेल समाधान को प्रवाह कोशिका में चलाएं। चिपचिपा हाइड्रोगेल अग्रदूत के लिए, स्लाइड को थोड़ा झुकाने के लिए हाइड्रोगेल प्रवाह को प्रवाह कोशिका में मदद करने की आवश्यकता हो सकती है।
- हाइड्रोगेल को सेट करने और कोशिकाओं और सतह के बीच डीएनए-आधारित आसंजन को क्लीव करने की अनुमति देने के लिए 30-45 मिनट (हाइड्रोजेल जेलेशन काइनेटिक्स के आधार पर) के लिए 37 डिग्री सेल्सियस पर स्लाइड को इनक्यूबेट करें।
- स्लाइड से प्रत्येक प्रवाह सेल निकालें और हाइड्रोगेल अग्रदूत समाधान के शीर्ष पर जगह है।
- 2-वेल चैंबर स्लाइड या 6-वेल प्लेट के कुएं में हाइड्रोजेल अग्रदूत का 50 माइक्रोन जोड़ें।
- प्रत्येक प्रवाह सेल के दोनों ओर पीबीएस के पिपेट 10 माइक्रोन।
- प्रवाह सेल की पूरी लंबाई के साथ पीबीएस वितरित करने के लिए एक रेजर ब्लेड या फाइन-पॉइंट चिमटी का उपयोग करें, फिर धीरे-धीरे प्रवाह सेल के किनारों को उठाएं ताकि पीबीएस हाइड्रोगेल के नीचे भागते हैं।
नोट: यह स्लाइड भर में हाइड्रोगेल को "फ्लोट" करेगा, जो विरूपण या फाड़ के बिना हस्तांतरण के लिए अनुमति देगा। - धीरे-धीरे ग्लास स्लाइड के किनारे करने के लिए प्रवाह सेल को स्थानांतरित करने के लिए एक रेजर ब्लेड का उपयोग करें।
- स्लाइड उलटा। रेजर ब्लेड के साथ, स्लाइड से प्रवाह सेल नज इतना है कि यह उस्तरा ब्लेड के शीर्ष पर भूमि ।
- घुमावदार संदंश का उपयोग करके रेजर ब्लेड से प्रवाह सेल चुनें। प्रवाह कोशिका को उलटें ताकि कोशिकाएं नीचे की ओर हों, और फिर हाइड्रोगेल अग्रदूत समाधान की बूंद के शीर्ष पर रखें।
- प्रत्येक प्रवाह कोशिका के लिए चरण 7.4.1 - 7.4.6 दोहराएं।
- कम से कम 30 मिनट के लिए इनक्यूबेट ताकि पैटर्न वाली कोशिकाओं वाले हाइड्रोगेल हाइड्रोगेल अंडरले से बांध सकें, जिसके परिणामस्वरूप पैटर्न वाली कोशिकाओं का पूरा एम्बेड हो।
- पीडीएमएस फ्लो सेल निकालें।
- पीडीएमएस प्रवाह सेल को विसर्जित करने के लिए पर्याप्त मीडिया जोड़ें।
नोट: मीडिया की आमद हाइड्रोगेल और पीडीएमएस फ्लो सेल के बीच आसंजन को ढीला कर देगी । - प्रवाह कोशिका की लंबी धुरी के साथ उन्मुख घुमावदार संदंश का उपयोग करें, प्रवाह कोशिका को धीरे से नज करने के लिए जब तक यह बंद चबूतरे और मीडिया में तैरता है । प्रवाह कोशिका को संदंश के साथ एकत्र करें और त्यागें।
नोट: इष्टतम परिणामों के लिए, घुमावदार संदंश फैलाएं और पीडीएमएस प्रवाह सेल की दीवारों पर कोमल दबाव लागू करें। प्रवाह कोशिका की लंबी धुरी की दिशा में बल लगाएं।
- पीडीएमएस प्रवाह सेल को विसर्जित करने के लिए पर्याप्त मीडिया जोड़ें।
8. सीएमओ के साथ कोशिकाओं की सफल लेबलिंग की पुष्टि करें (वैकल्पिक, समस्या निवारण के लिए)
- एक फ्लोरोसेंटली संशोधित (एफएएम या AF647) ओलिगोन्यूक्लियोटाइड ऑर्डर करें जो प्रयोग में उपयोग किए जा रहे एडाप्टर स्ट्रैंड के सतह आसंजन अनुक्रम के पूरक है।
- सीएमओ डीएनए के साथ लेबल कोशिकाओं और बाहर अतिरिक्त डीएनए धोने के रूप में चरण 5 में वर्णित है । बर्फ ठंडा PBS के 200 μL में resuspend.
- पीबीएस में फ्लोरोसेंटी लेबल वाले पूरक ओलिगोन्यूक्लियोटाइड का 4 माइक्रोन घोल बनाएं। सेल निलंबन के लिए इस समाधान के 200 μL जोड़ें। 5 मिनट के लिए बर्फ पर इनक्यूबेट।
- बर्फ-ठंडे पीबीएस का 1 मिलील जोड़ें। मिक्स करें। कोशिकाओं को उन्हें गोली मारने के लिए अपकेंद्रित्र करें। सुपरनिट निकालें। संकरण नहीं किए गए किसी भी डीएनए को धोने के लिए इस प्रक्रिया को दो बार और दोहराएं।
- कोशिका की सतह पर डीएनए की उपस्थिति की मात्रा निर्धारित करने के लिए विश्लेषणात्मक प्रवाह साइटोमेट्री करें।
- एक प्रवाह साइटोमीटर पर, नियंत्रण कोशिकाओं का विश्लेषण करें जिन्हें डीएनए के साथ लेबल नहीं किया गया है। इस आबादी के आधार पर गेट स्थापित करें।
- सीएमओ-लेबल कोशिकाओं का विश्लेषण करें जिनका इलाज फ्लोरोसेंटी लेबल वाले पूरक ओलिगोन्यूक्लियोटाइड के साथ किया गया है।
- मतलब फ्लोरेसेंस तीव्रता की गणना करें।
यह प्रोटोकॉल उच्च परिशुद्धता के साथ 2डी और 3 डी में और कस्टम रिएजेंट्स या महंगे क्लीनरूम उपकरण के उपयोग के बिना कोशिकाओं को पैटर्न करना संभव बनाता है। चित्रा 1 प्रोटोकॉल का अवलोकन दिखाता है। सबसे पहले, फोटोलिथोग्राफी के माध्यम से डीएनए-कार्यात्मक स्लाइड बनाई जाती हैं। इसके बाद, कोशिकाओं को सीएमओ के साथ लेबल किया जाता है। कोशिकाओं को तब स्लाइड पर प्रवाहित किया जाता है, जहां वे केवल स्लाइड के डीएनए-कार्यात्मक क्षेत्रों से जोड़ते हैं। अतिरिक्त कोशिकाओं को बह जाने के बाद, कोशिकाओं के वांछित पैटर्न का पता चलता है। इन कोशिकाओं को स्लाइड पर सुसंस्कृत किया जा सकता है या DNase युक्त हाइड्रोगेल में एम्बेडेड किया जा सकता है और 3 डी सेल संस्कृति के लिए स्लाइड को स्थानांतरित कर दिया जा सकता है।
सीएमओ के साथ कोशिकाओं की लेबलिंग डीएनए नमूनों स्लाइड(चित्रा 2)के लिए उनके लगाव के लिए अनुमति देता है । सबसे पहले, कोलेस्ट्रॉल-संशोधित यूनिवर्सल एंकर स्ट्रैंड एडाप्टर स्ट्रैंड के साथ पूर्व-संकरित है। इसके बाद, यूनिवर्सल एंकर + एडाप्टर समाधान सेल निलंबन के साथ 1:1 मिश्रित है। यूनिवर्सल एंकर + एडाप्टर कॉम्प्लेक्स पर कोलेस्ट्रॉल कोशिका झिल्ली में सम्मिलित करता है। कोलेस्ट्रॉल-संशोधित यूनिवर्सल सह-एंकर स्ट्रैंड के अलावा, जो यूनिवर्सल एंकर स्ट्रैंड के साथ संकरण करता है, परिसर26की शुद्ध हाइड्रोफोबसिटी को बढ़ाकर कोशिका झिल्ली में सीएमओ परिसर की स्थिरता में सुधार करता है। कोशिका निलंबन से अतिरिक्त डीएनए धोने के बाद, कोशिकाओं को स्लाइड पर प्रवाहित किया जाता है। एडाप्टर स्ट्रैंड और सतह डीएनए स्ट्रैंड के बीच संकरण के परिणामस्वरूप स्लाइड के डीएनए-पैटर्न वाले क्षेत्रों में कोशिकाओं की लगाव होती है।
कोशिकाओं का पैटर्न फोटोलिथोग्राफी का उपयोग करके बनाया गया है ताकि एल्डिहाइड-संशोधित ग्लास स्लाइड29 (चित्रा 3 ए)के विशिष्ट क्षेत्रों में अमीन-संशोधित डीएनए ओलिगोस के लगाव को प्रतिबंधित किया जा सके। सकारात्मक फोटोरेसिस्ट एक एल्डिहाइड-कार्यात्मक स्लाइड पर स्पिन-लेपित है। एक पारदर्शिता फोटोमास्क तो स्लाइड के शीर्ष पर रखा जाता है और स्लाइड यूवी प्रकाश के संपर्क में है । विकसित होने के बाद, यूवी प्रकाश के संपर्क में आने वाले स्लाइड के क्षेत्र अब फोटोरेसिस्ट में लेपित नहीं हैं और इस प्रकार एल्डिहाइड समूहों को उजागर किया है। माइन-संशोधित डीएनए ओलिगोस का 20 माइक्रोन समाधान फिर स्लाइड पर गिरा दिया जाता है और पैटर्न वाले क्षेत्रों को कवर करने के लिए फैल जाता है। पाक के बाद अपचय अमीन के परिणामस्वरूप अमीन-संशोधित डीएनए और स्लाइड के बीच एक सहसंयोजक बंधन होता है । उल्लेखनीय रूप से, इस प्रक्रिया को पहले पैटर्न वाले ओलिगोस(चित्रा 3 बी)की कार्यक्षमता के किसी भी नुकसान के बिना कई ओलिगो पैटर्न करने के लिए दोहराया जा सकता है। हालांकि, ओवरलैपिंग पैटर्न से बचने के लिए देखभाल की जानी चाहिए, जिसके परिणामस्वरूप कम एकाग्रता(पूरक चित्र 3)पर दोनों ओलिगोस की उपस्थिति होती है। एडाप्टर स्ट्रैंड्स का उपयोग करके कई सेल आबादी को क्रमिक रूप से पैटर्न किया जा सकता है जो उनके मॉड्यूलर डोमेन (3' के अंत के निकटतम 20 ठिकानों) में भिन्न होते हैं।
यद्यपि यह फोटोपैटर्निंग प्रोटोकॉल एक साफ कमरे के संदर्भ में शेइडेलर एट अल द्वारा विकसित किया गया था, हमने दिखा दिया है कि एक सस्ती, "होम-काढ़ा" फोटोलिथोग्राफी सेटअप के साथ समान परिणाम प्राप्त करना संभव है जो रासायनिक धुएं हुड के भीतर आसानी से फिट बैठता है। सेटअप में डीसी मोटर, डिजिटल कंट्रोलर और सीडी केक बॉक्स से बना $ 400 स्पिन कोटर, साथ ही एक यूवी लैंप भी शामिल है जिसे व्यक्तिगत घटकों से इकट्ठा किया गया था और एक पुनर्निर्मित शार्प्स कंटेनर(पूरक चित्रा 1)में रखा गया था। घर-काढ़ा फोटोलिथोग्राफी सेटअप का मुख्य लाभ यह है कि यह बहुत सस्ती है (सभी उपकरणों के लिए $ 1000 <) जबकि अभी भी एकल-सेल-आकार की विशेषताएं बनाने में सक्षम हैं। हालांकि, सस्ती उपकरणों के उपयोग की इसकी सीमाएं हैं - उदाहरण के लिए, मास्क एलाइनर के उपयोग के बिना कई डीएनए ओलिगो पैटर्न के लिए प्रत्ययी मार्कर को ठीक से संरेखित करना अधिक चुनौतीपूर्ण है। हम उन प्रयोगशालाओं के लिए इस सस्ती फोटोलिथोग्राफी सेटअप की सलाह देते हैं जिनके पास साफ कमरे तक सुविधाजनक पहुंच नहीं है या जो बड़े निवेश के बिना इस विधि को आज़माना चाहते हैं।
डीएनए-प्रोग्राम किए गए कोशिका आसंजन के लिए इष्टतम स्थितियों की पहचान करने के लिए, हमने व्यवस्थित रूप से कोशिका सतहों पर डीएनए किस्में की सांद्रता को अलग किया और डीएनए-संशोधित ग्लास सतहों के लिए कोशिका आसंजन की दक्षता को मापा। लेबलिंग समाधानों में यूनिवर्सल एंकर + एडाप्टर स्ट्रैंड और यूनिवर्सल को-एंकर की एकाग्रता परिमाण के कई आदेशों(चित्रा 4ए,बी)में भिन्न थी, जिसके परिणामस्वरूप प्रति सेल 104 -106 डीएनए कॉम्प्लेक्स(पूरक चित्रा 4)थे। सेल आसंजन खुराक निर्भर था, डीएनए पैटर्न के लिए न्यूनतम सेल आसंजन के साथ जब कोशिकाओं को 0.05 माइक्रोन या उससे कम की एकाग्रता पर सीएमओ के साथ लेबल किया गया था, और 2.5 माइक्रोन और उससे अधिक की एकाग्रता पर उच्च अधिभोग। इसलिए, हमने अधिकांश प्रयोगों में यूनिवर्सल एंकर + एडाप्टर स्ट्रैंड के 2 माइक्रोन समाधान और यूनिवर्सल सह-एंकर के 2 माइक्रोनएम समाधान का उपयोग किया। यदि कांच की सतह पर उपयोग किए जाने वाले डीएनए की मात्रा29 कम हो जाती है या यदि एडाप्टर स्ट्रैंड और सतह स्ट्रैंड के बीच बेमेल बढ़ जाती है तो सेल आसंजन में भी कमी आने की उम्मीद होगी । एडाप्टर स्ट्रैंड सीक्वेंस डिजाइन के बारे में अधिक जानकारी पूरक फ़ाइल 2में प्रदान की गई है। सीपीजी दोहराए बिना एडाप्टर किस्में का उपयोग कर सीएमओ लेबलिंग एचईके कोशिकाओं में TLR9 को उत्तेजित नहीं किया माउस TLR9 (पूरक चित्रा 5)व्यक्त ।
हम कई प्रदर्शन प्रदान करते हैं कि संशोधित प्रोटोकॉल प्रजनन योग्य और कुशल डीएनए-प्रोग्राम सेल आसंजन प्रदान करता है। उदाहरण के लिए, सीएमओ के साथ लेबल की गई मानव गर्भनाल नस एंडोथेलियल कोशिकाएं (एचयूवीसी) ने उच्च दक्षता के साथ डीएनए पैटर्न का पालन किया। सीएमओ लेबल HUVECs का पालन किया और साथ ही LMO लेबल HUVECs(चित्रा 5A)। सीएमओ-डीपीएसी का उपयोग करके पैटर्न वाली कोशिकाओं ने उनकी व्यवहार्यता और कार्यक्षमता को बनाए रखा। सीएमओ के साथ लेबल कोशिकाओं को व्यवहार्यता(पूरक चित्रा 6)का आकलन करने के लिए कैल्सीन एएम और एथिडियम होमोमर द्वारा दाग दिया गया था। अवेलेबल कंट्रोल सेल्स की तुलना में व्यवहार्यता में अंतर छोटे थे (94% बनाम 97%)। एकल एमडीसीके सीएमओ-डीपीएसी के माध्यम से पैटर्न और मैट्रिक्स में स्थानांतरित करने के लिए संस्कृति के 5 दिनों के बाद सही ढंग से पैदा करने और ध्रुवीकरण करने में सक्षम थे(चित्रा 5B)। डीपीएसी तीसरे आयाम(चित्रा 5C)में कोशिकाओं के पैटर्न को विस्तारित करने का एक साधन भी प्रदान करता है। उदाहरण के लिए, पूरक सीएमओ(चित्रा 5 सी)के साथ लेबल की गई कोशिकाओं की परतों को बदलकर बहुस्तरीय, बहुकोशिकीय समुच्चय बनाया जा सकता है। ये प्रयोग प्रदर्शित करते हैं कि प्रोटोकॉल पुन: उत्पन्न है, सेल व्यवहार्यता या कार्यक्षमता को नकारात्मक रूप से प्रभावित नहीं करता है, और सेलुलर पैटर्न पैदा करता है जिसे 3 डी ईसीएम में एक इमेजिंग विमान के भीतर सफलतापूर्वक सुसंस्कृत किया जा सकता है।
प्रत्यक्ष कोशिका आसंजन के लिए ऑर्थोगोनल डीएनए दृश्य प्रदान करके, DPAC एक ही सतह पर कई सेल प्रकार पैटर्निंग का एक साधन प्रदान करता है । डीपीएसी की इस सुविधा को लागू करने के लिए, फोटोलिथोग्राफी द्वारा उत्पन्न डीएनए पैटर्न को एक-दूसरे के संबंध में गठबंधन किया जाना चाहिए। कई फोटोमास्क के संरेखण के लिए अनुमति दी स्लाइड पर जमा धातु प्रत्ययी मार्कर और इसलिए एक बार में कई सेल प्रकार के पैटर्निंग । एमसीएफ 10As विभिन्न अद्वितीय रंगों के साथ दाग को ऑर्थोगोनल सीएमओ के साथ लेबल किया गया था और यूसी बर्कले और यूसीएसएफ लोगो(चित्र 6)का दृश्य बनाने के लिए पैटर्न किया गया था। यह प्रयोग दर्शाता है कि कई अद्वितीय कोशिका आबादी को उच्च परिशुद्धता के साथ और क्रॉस-संदूषण के बिना एक साथ पैटर्न किया जा सकता है।
सीएमओ-डीपीएसी का उपयोग करके कोशिकाओं के सफल पैटर्न में उच्च गुणवत्ता वाली फोटोलिथोग्राफी, कोशिका की सतह पर ओलिगो की पर्याप्त एकाग्रता, पैटर्न पर कोशिकाओं का उच्च घनत्व और पर्याप्त धोने की आवश्यकता होती है। इनमें से किसी एक चरण की विफलता अंतिम परिणाम को प्रभावित करती है। पूरक चित्रा 2 में सही और गलत फोटोलिथोग्राफी(पूरक चित्रा 2 ए),पूरी तरह से कब्जे वाले पैटर्न(पूरक चित्रा 2B)बनाने के लिए पैटर्न पर वांछित सेल घनत्व, डीपीएसी(पूरक चित्रा 2 सी)के बाद के चरणों के दौरान पीढ़ी जोरदार पिप्टिंग के कारण पैटर्न वाली कोशिकाओं का नुकसान और कोशिकाओं की अवांछित झुरमुट(पूरक चित्रा 2D)शामिल हैं। तालिका 1 में सामान्य विफलता बिंदुओं और सुझाए गए समस्या निवारण की एक सूची शामिल है। फ्लोरोसेंट पूरक ओलिगोस के उपयोग को स्लाइड पर पैटर्न वाले डीएनए की उपस्थिति और प्रवाह साइटोमेट्री द्वारा सेल सतह पर सीएमओ की उपस्थिति की पुष्टि करने के लिए समस्या निवारण के लिए एक उपकरण के रूप में सिफारिश की जाती है (प्रोटोकॉल के चरण 8 देखें)।
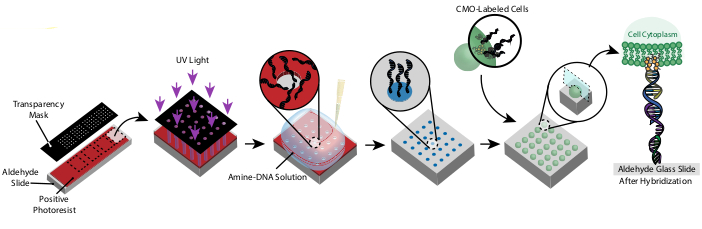
चित्रा 1:सीएमओ-डीपीएसी प्रोटोकॉल का अवलोकन। सबसे पहले, एक डीएनए-पैटर्न वाली स्लाइड एक सकारात्मक फोटोरेसिस्ट के साथ एक एल्डिहाइड-कार्यात्मक ग्लास स्लाइड को कोटिंग करके बनाई गई है, इसे वांछित पैटर्न में पारदर्शिता मास्क के साथ कवर किया जाता है, और इसे यूवी प्रकाश में उजागर करता है। यूवी-उजागर फोटोरेसिस्ट डेवलपर के साथ बह जाता है, एल्डिहाइड स्लाइड के उजागर क्षेत्रों को छोड़ देता है और सतह पर अमीन-कार्यात्मक डीएनए के बाध्यकारी की अनुमति देता है। कोशिकाओं को तो CMOs के साथ लेबल और सतह पर प्रवाहित कर रहे हैं । कोशिका झिल्ली पर डीएनए सतह पर डीएनए को संकरित करता है, जिसके परिणामस्वरूप आसंजन होता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
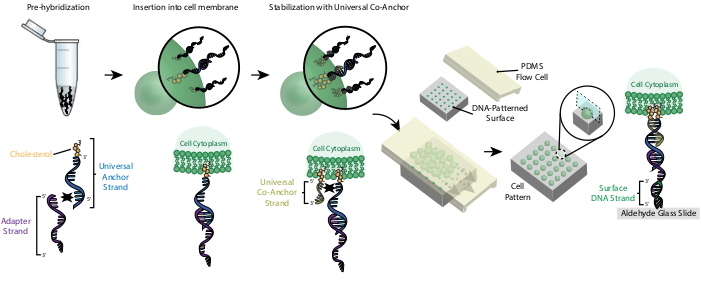
चित्रा 2:कोशिकाओं को एक कदम की प्रक्रिया में सीएमओ के साथ लेबल कर रहे हैं । सबसे पहले, कोलेस्ट्रॉल-संशोधित यूनिवर्सल एंकर स्ट्रैंड एडाप्टर स्ट्रैंड के साथ पूर्व-संकरित है। इसके बाद, यूनिवर्सल एंकर + एडाप्टर समाधान सेल निलंबन के साथ मिलाया जाता है। यूनिवर्सल एंकर + एडाप्टर कॉम्प्लेक्स पर कोलेस्ट्रॉल कोशिका झिल्ली में सम्मिलित करता है। इनक्यूबेशन के बाद, कोलेस्ट्रॉल-संशोधित यूनिवर्सल सह-एंकर स्ट्रैंड को सेल निलंबन में जोड़ा जाता है, जहां यह यूनिवर्सल एंकर स्ट्रैंड के साथ संकरण करता है और कोशिका झिल्ली में सम्मिलित करता है। कोलेस्ट्रॉल के दूसरे अणु को जोडऩे से डीएनए परिसर की शुद्ध हाइड्रोफोबसिटी बढ़ती है और वह झिल्ली26के भीतर स्थिर हो जाता है . अतिरिक्त डीएनए को धोने के बाद, कोशिकाओं को केंद्रित किया जाता है और पैटर्न वाली सतह के शीर्ष पर पीडीएमएस प्रवाह कोशिका में जोड़ा जाता है। एडाप्टर स्ट्रैंड का 3' अंत ग्लास स्लाइड पर सतह डीएनए स्ट्रैंड के साथ संकरित होता है, जिसके परिणामस्वरूप पूरक डीएनए के साथ कार्यात्मक क्षेत्रों में विशेष रूप से स्लाइड से आसंजन होता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
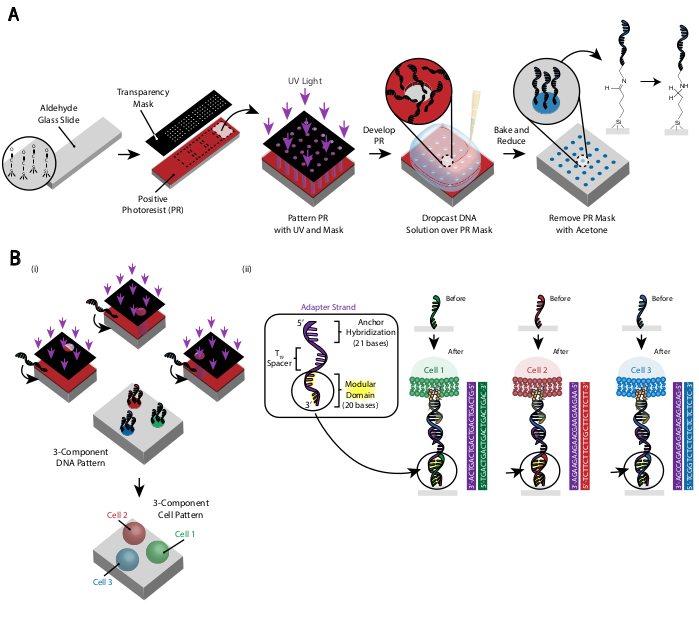
चित्र 3:फोटोलिथोग्राफी का उपयोग डीएनए-पैटर्न वाली स्लाइड बनाने के लिए किया जाता है जो अंततः कोशिकाओं के प्लेसमेंट को निर्देशित करेगा। (A)फोटोलिथोग्राफी प्रक्रिया का अवलोकन। एक एल्डिहाइड-कार्यात्मक स्लाइड एक सकारात्मक फोटोरेसिस्ट के साथ स्पिन-लेपित है। यूवी प्रकाश एक पारदर्शिता फोटोमास्क के माध्यम से स्लाइड पर चमकता है जो पारदर्शी है जहां सेल आसंजन वांछित है। स्लाइड विकसित होने के बाद, जिन क्षेत्रों को पहले यूवी लाइट के संपर्क में लाया गया था, उन्होंने अब एल्डिहाइड समूहों को उजागर किया है। एक माइन-कार्यात्मक डीएनए ओलिगो का 20 माइक्रोन समाधान फिर स्लाइड पर छोड़ दिया जाता है और पैटर्न वाले क्षेत्रों में फैल जाता है। इसके बाद स्लाइड को अमीन और एल्डिहाइड समूहों के बीच शिफ बांड (सी = एन) के गठन के लिए प्रेरित करने के लिए बेक किया जाता है, जो एक प्रतिवर्ती सहसंयोजक बांड29है । पीबीएस में 0.25% सोडियम बोरोहाइड्राइड के साथ बाद में अपचय अमीन शिफ बेस को अपचय अमीन द्वारा एक माध्यमिक अमीन में परिवर्तित करता है, जिसके परिणामस्वरूप डीएनए और स्लाइड के बीच एक अपरिवर्तनीय बंधन होता है। शेष फोटोरेसिस्ट को एसीटोन के साथ कुल्ला करके हटाया जा सकता है। (ख)इस प्रक्रिया को बहु-घटक डीएनए पैटर्न बनाने के लिए दोहराया जा सकता है और इसलिए कई सेल आबादी के साथ प्रयोग करते हैं । (i) पहले ओलिगो के पैटर्न के बाद, स्लाइड को फिर से फोटोरेसिस्ट में लेपित किया जाता है और प्रोटोकॉल पहले की तरह आगे बढ़ता है । कई डीएनए किस्में पैटर्निंग के लिए प्रत्ययी मार्कर का उपयोग कर फोटोमास्क का संरेखण आवश्यक है। (ii) प्रत्येक कोशिका प्रकार पैटर्न एडाप्टर स्ट्रैंड के 20-बेस मॉड्यूलर डोमेन में अलग होता है। पूरक ओलिगोस के ऑर्थोगोनल सेट का उपयोग करके, क्रॉस-आसंजन के बिना कई सेल प्रकारों को पैटर्न किया जा सकता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
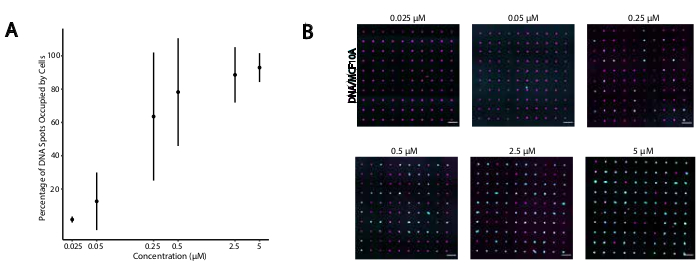
चित्र 4:सीएमओ लेबल कोशिकाओं के डीएनए पैटर्न के लिए आसंजन लेबलिंग के दौरान सीएमओ एकाग्रता के एक समारोह के रूप में बढ़ जाती है । इस प्रयोग में यूनिवर्सल एंकर + एडाप्टर स्ट्रैंड (पूर्व संकरित) और यूनिवर्सल सह-एंकर का उपयोग समान सांद्रता पर किया गया था । एकाग्रता कोशिकाओं के सीएमओ लेबलिंग के दौरान सेल निलंबन में सीएमओ की एकाग्रता को संदर्भित करता है। (क)15 माइक्रोन व्यास डीएनए स्पॉट के प्रतिशत का परिमाणीकरण जो सीएमओ लेबल वाले एमसीएफ 10ए कोशिकाओं द्वारा कोशिका लेबल वाले एमसीएफ 10ए कोशिकाओं द्वारा कोशिका लेबल के दौरान सीएमओ एकाग्रता के एक समारोह के रूप में किया गया था । डेटा तीन प्रयोगों से मानक विचलन ± मतलब के रूप में प्रतिनिधित्व किया । (ख)डीएनए पैटर्न (मजेंटा) की प्रतिनिधि छवियां और सीएमओ की विभिन्न सांद्रता पर MCF10As (सियान) का पालन किया । स्केल बार = 100 माइक्रोन. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
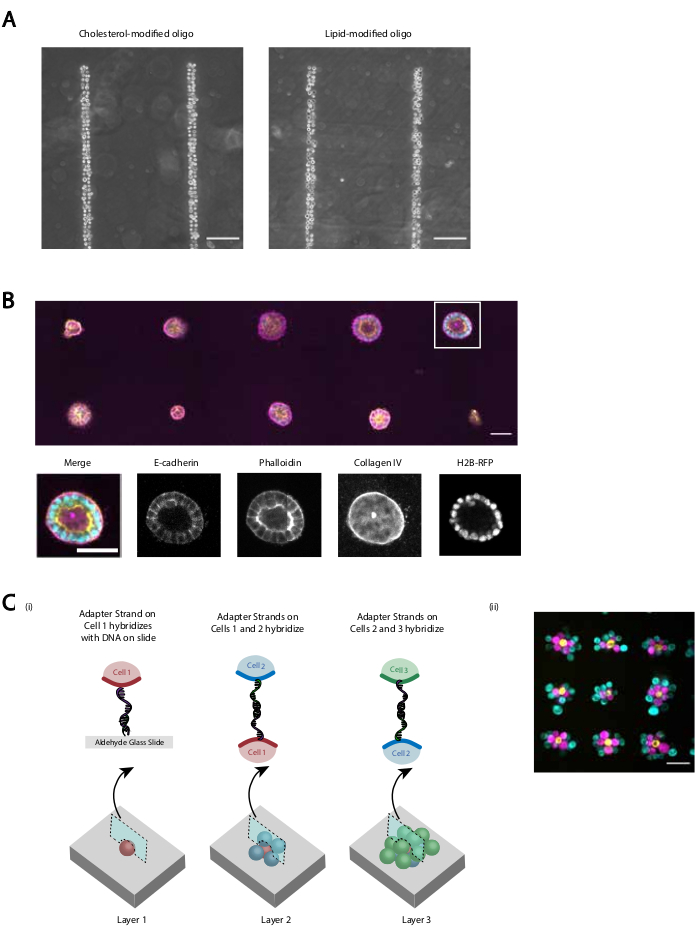
चित्र 5:सीएमओ-डीपीएसी का उपयोग दो-आयामी सेल पैटर्न बनाने के लिए किया जा सकता है जिसे बाद में संस्कृति के लिए त्रि-आयामी हाइड्रोगेल में एम्बेडेड किया जा सकता है और/या बहुस्तरीय संरचनाएं बनाने के लिए स्तरित किया जा सकता है । (क)सीएमओ लेबल वाली मानव नाभि नस एंडोथेलियल कोशिकाओं (एचयूवीसी) और एलएमओ-लेबल वाले एचयूवीसी के बीच सीधी तुलना एक रैखिक डीएनए पैटर्न का पालन करती है । सेल लेबलिंग के दोनों तरीकों के परिणामस्वरूप डीएनए पैटर्न की लगभग 100% अधिभोग होती है। (ख)एच2बी-आरएफपी को व्यक्त करने वाली सिंगल मैडीन-डार्बी कैनाइन किडनी सेल्स (एमडीसीके) को 15 माइक्रोन व्यास के स्थानों पर पैटर्न किया गया था जो २०० माइक्रोन के अलावा और बाद में मैट्रिक्स में एम्बेडेड थे । संस्कृति के 120 एच के बाद, परिणामस्वरूप एपिथेलियल अल्सर को ई-कैडेरिन, ऐक्टिन और कोलेजन चतुर्थ के लिए तय किया गया था और दाग दिया गया था। सफेद बॉक्स में गोलाकार विस्तार से दिखाया गया है। स्केल बार = 50 माइक्रोन(C)बहुस्तरीय सेलुलर संरचनाओं को पूरक एडाप्टर स्ट्रैंड्स और पैटर्निंग के साथ अलग सेल आबादी लेबल करके बनाया जा सकता है ताकि कोशिकाओं का प्रत्येक नया जोड़ इससे पहले सेल परत का पालन कर सके। (i)बहुस्तरीय संरचनाओं को बनाने के लिए सेल आबादी के अनुक्रमिक पैटर्निंग का एक योजनाबद्ध। } इस प्रक्रिया का उपयोग करके MCF10As (रंगों का उपयोग करके कल्पना) के तीन-स्तरित सेल समुच्चय बनाए गए थे। स्केल बार = 50 माइक्रोन. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।

चित्र 6:कई सेल प्रकार पार संदूषण या आसंजन की हानि के बिना पैटर्न किया जा सकता है। कई अमीन-संशोधित डीएनए ओलिगोस को एक एल्डिहाइड स्लाइड पर क्रमिक रूप से पैटर्न किया गया था और धातु प्रत्ययी मार्कर के उपयोग के माध्यम से गठबंधन किया गया था। MCF10As (सियान, मजेंटा, पीला) की तीन आबादी पूरक सीएमओ के साथ लेबल अद्वितीय रंगों के साथ दाग रहे थे, और स्लाइड पर नमूनों, UC बर्कले और UCSF लोगो की एक छवि में जिसके परिणामस्वरूप । स्केल बार 1 मिमी. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
पूरक चित्रा 1: बेंचटॉप फोटोलिथोग्राफी सेटअप की उदाहरण छवियां। (A)स्पिन कोटर पर स्लाइड, स्पिन कोटिंग से पहले सकारात्मक फोटोरेसिस्ट के साथ कवर किया गया । (ख)पारदर्शिता फोटोमास्क की तस्वीर । (ग)एक्सपोजर के दौरान फोटोमास्क को फोटोरेसिस्ट-कोटेड स्लाइड और ग्लास डिस्क के बीच सैंडविच किया जाता है ।(D)यूवी लैंप के लिए आवास एक फिर से उद्देश्य वाले शार्प्स कंटेनर से बाहर किया गया था । (ई)स्लाइड डेवलपर समाधान में डूबे । (च)विकसित स्लाइड। (जी)अमीन संशोधित डीएनए समाधान स्लाइड के नमूनों वाले क्षेत्रों पर फैल गया । (एच)पीडीएमएस फ्लो सेल स्लाइड के नमूनों वाले क्षेत्रों के शीर्ष पर रखा गया है । इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक चित्रा 2: इस प्रोटोकॉल की सामान्य विफलताओं के कुछ उदाहरण। (ए)(i)यूवी एक्सपोजर या ओवर-डेवलपिंग फीचर्स से पहले अंडर-बेकिंग के परिणामस्वरूप ऐसी विशेषताएं हो सकती हैं जिनमें दांतेदार किनारे हैं और आकार में अनियमित हो सकते हैं । (ii)एक सही ढंग से फोटोपैटर्न्ड स्लाइड का एक उदाहरण जिसमें सुविधाओं के आसपास साफ किनारे हैं, एक समान सुविधा आकार, और पैटर्न में कोई स्पष्ट दरारें नहीं हैं। स्केल बार = 50 माइक्रोन (बी)सेल घनत्व पैटर्निंग दक्षता के लिए महत्वपूर्ण है। माइक्रोस्कोप के तहत पैटर्न के शीर्ष पर कोशिकाओं को देखते समय, कोशिकाओं के बीच कुछ अंतराल मौजूद होने चाहिए, जैसा कि बाईं ओर उदाहरण छवि से सबूत है। स्केल बार = 50 माइक्रोन(सी)पैटर्न वाली कोशिकाएं पीढ़ी जोरदार पिपटिंग से उत्पन्न होने वाली तरल पदार्थ ताकतों के प्रति संवेदनशील हो सकती हैं, जो पैटर्न वाली कोशिकाओं को नुकसान पहुंचा सकती हैं और उन्हें उखाड़ फेंक सकती हैं। बहुस्तरीय कोशिका समुच्चय विशेष रूप से कमजोर होते हैं, क्योंकि नीचे एक कोशिका कई कोशिकाओं की संरचना का समर्थन कर रही है। (i)कोशिका की एक सरणी सफलतापूर्वक मैट्रिक्स में मैट्रिकेल में एम्बेडेड होती है। (ii) सेल का एक ग्रिड जो बहुत सख्ती से चिपचिपा मैट्रिगेल के परिणामस्वरूप उखाड़ दिया जाता है। (घ)कोशिकाओं का झुरमुट हो सकता है, विशेष रूप से एपिथेलियल कोशिकाओं के साथ। ये झुरमुट आमतौर पर होमोसिप्टिक होते हैं लेकिन यदि कोशिकाएं विशेष रूप से चिपचिपी होती हैं तो विषमतापिक (एक अलग प्रकार की पहले से पैटर्न वाली कोशिकाओं का पालन करने वाली कोशिकाएं) हो सकती हैं। छवि से पता चलता है MCF10As के तीन अलग आबादी तीन अलग एकल सेल आकार डीएनए स्पॉट (15 μm) से बना सरणी पर नमूनों थे । ज्यादातर डीएनए स्पॉट में 2-4 कोशिकाएं जुड़ी होती हैं। झुरमुट को ईडीटीए उपचार द्वारा या पैटर्निंग से पहले झुरमुटों को फ़िल्टर करके हल किया जा सकता है। स्केल बार = 100 माइक्रोन। इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक चित्र 3: कम एकाग्रता पर दोनों ओलिगोस की उपस्थिति में ओवरलैपिंग फोटोपैटर्न परिणाम देते हैं। दो ऑर्थोगोनल अमीन-संशोधित ओलिगोस को क्रमिक रूप से फोटोपैटर्न किया गया था, पहले एक ऊर्ध्वाधर रेखा (स्ट्रैंड 1), जिसके बाद एक क्षैतिज रेखा थी जो इसे ओवरलैप (स्ट्रैंड 2) से घिरा हुआ था। ओलिगोस को तब फ्लोरोसेंट पूरक ओलिगोस के साथ संकरण द्वारा कल्पना की गई थी। (A)स्ट्रैंड 1 की फ्लोरेसेंस इमेज । (ख)ओवरलैप में फैले 100 माइक्रोन वर्टिकल लाइन पर स्ट्रैंड 1 के फ्लोरेसेंस प्रोफाइल का मात्राकरण। (ग)स्ट्रैंड 2 की फ्लोरेसेंस इमेज । (घ)ओवरलैप में फैले 100 माइक्रोन क्षैतिज लाइन पर स्ट्रैंड 2 के फ्लोरेसेंस प्रोफाइल का मात्राकरण। स्केल बार = 50 माइक्रोन। इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक चित्र 4: सीएमओ लेबलिंग एकाग्रता के एक समारोह के रूप में कोशिका की सतह पर डीएनए परिसरों की मात्राकरण। HUVECs सीएमओ समाधान की विभिन्न सांद्रता के साथ लेबल किया गया था, धोया, और फिर एक फ्लोरोसेंट पूरक कतरा के साथ इनक्यूबेटेड । एक एमईएसएफ (समकक्ष घुलनशील फ्लोरोक्रोम के अणु) माइक्रोस्फीयर किट का उपयोग मात्रात्मक प्रवाह साइटोमेट्री करने और लेबलिंग के दौरान सीएमओ एकाग्रता के एक समारोह के रूप में कोशिका की सतह पर डीएनए परिसरों की संख्या का अनुमान लगाने के लिए किया गया था। इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक चित्रा 5: सीएमओ लेबलिंग TLR9 प्रतिक्रिया को प्रोत्साहित नहीं करता है। यह देखने के लिए एक प्रयोग किया गया था कि क्या सीएमओ लेबलिंग टीएलआर9 के डीएनए-डिटेक्शन तंत्र को ट्रिगर करेगी और क्या यह एडाप्टर स्ट्रैंड अनुक्रम में सीपीजी से प्रभावित होगा । एचईके कोशिकाओं माउस TLR9 व्यक्त या तो ODN १८२६ (एक CPG युक्त TLR9 agonist), सीएमओ यूनिवर्सल एंकर + यूनिवर्सल सह एंकर के ०.२ μM के साथ रातोंरात इनक्यूबेटेड थे + एडाप्टर स्ट्रैंड जिसमें ओडीएन 1826 (सीएमओ-सीपीजी), या सीएमओ यूनिवर्सल एंकर + यूनिवर्सल सह-एंकर + एडाप्टर स्ट्रैंड के समान अनुक्रम होते हैं, लेकिन जीपीसी (सीएमओ-जीपीसी) के साथ सीपीजी के प्रतिस्थापन के साथ। टीएलआर 9 उत्तेजना के परिणामस्वरूप सीईईपी (स्रावित भ्रूण क्षारीय फॉस्फेट) का उत्पादन होगा। सीईपी स्राव को एक कोलोरिमेट्रिक परख (अवशोषण) द्वारा निर्धारित किया गया था। उपचार की स्थिति की तुलना आराम करने वाली कोशिकाओं से की जाती थी जिन्हें केवल पीबीएस के साथ इलाज किया जाता था। सीएमओ-जीपीसी के साथ इनक्यूबेशन ने टीएलआर9 अभिव्यक्ति को प्रोत्साहित नहीं किया । सीएमओ-सीपीजी के साथ इनक्यूबेशन आराम कोशिकाओं की तुलना में थोड़ा अधिक था, लेकिन ODN-१८२६ की तुलना में बहुत कम है । इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक चित्रा 6: सीएमओ लेबलिंग प्रक्रिया के बाद कोशिकाओं की व्यवहार्यता। यह आकलन करने के लिए कि प्रोटोकॉल व्यवहार्यता को कैसे प्रभावित करता है, HUVECs को चार आबादी में विभाजित किया गया था: एक 1 घंटे के लिए बर्फ पर बना रहा, एक को पीबीएस के साथ नकली लेबल किया गया था, लेकिन अन्यथा सभी अपकेंद्रित्र और धोने के कदमों के माध्यम से लिया गया था, एक को सीएमओ के साथ लेबल किया गया था, और एक को सीएमओ के साथ लेबल किया गया था और झुरमुटों को हटाने के लिए 40 माइक्रोन फ़िल्टर के माध्यम से फ़िल्टर किया गया था। इसके बाद कोशिकाओं को जीवित और मृत कोशिकाओं की संख्या का आकलन करने के लिए कैल्सीन एएम और एथिडियम होमाडिमर से दाग दिया गया । सभी उपचारों के परिणामस्वरूप बर्फ नियंत्रण (तुकी पोस्ट-हॉक विश्लेषण के साथ एक-तरफा ANOVA) की तुलना में व्यवहार्यता में काफी कमी आई, लेकिन सीएमओ-लेबलिंग (फ़िल्टरिंग के साथ या बिना) के लिए औसत व्यवहार्यता लगभग 94% थी। तीन स्वतंत्र प्रयोगों से एकत्र किए गए डेटा। * = पी < 0.05। = पी < 0.0001 कृपया इस फ़ाइल को डाउनलोड करने के लिए यहां क्लिक करें।
| परिणाम | संभावित कारण (ओं) | सुझाए गए फिक्स |
| फोटोलिथोग्राफी - विशेषताएं फटा रहे हैं | असंगत या अपर्याप्त नरम सेंकना | 3 मिनट तक नरम सेंकना का समय बढ़ाएं; हॉटप्लेट के वास्तविक तापमान को सत्यापित करें और आवश्यक के रूप में तापमान बढ़ाएं |
| फोटोलिथोग्राफी - विशेषताएं तेज नहीं हैं या उनके भीतर फोटोरेसिस्ट शेष हैं | अल्प विकास | समय बढ़ाएं कि स्लाइड डेवलपर समाधान में खर्च करता है; सौम्य आंदोलन को शामिल करना |
| फोटोलिथोग्राफी - स्लाइड में असंगत विशेषताएं | यूवी प्रकाश केंद्रित नहीं किया जा सकता है या ठीक से ध्यान केंद्रित नहीं किया जा सकता है | यूवी लाइट सेटअप को समायोजित करें ताकि समान तीव्रता के कोलिमेटेड प्रकाश को सुनिश्चित किया जा सके |
| कोशिकाएं उच्च दक्षता वाले पैटर्न वाले स्थानों का पालन नहीं करती हैं | सतह पर पर्याप्त डीएनए नहीं | पुष्टि करें कि डीएनए फ्लोरोसेंट पूरक ओलिगोस के साथ स्लाइड को संकरण करके सतह पर मौजूद है और फिर माइक्रोस्कोप के नीचे इमेजिंग |
| सीएमओ के साथ कोशिकाओं को अपर्याप्त रूप से लेबल किया जाता है | सेल निलंबन के लिए फ्लोरोसेंट पूरक ओलिगोस जोड़ें और प्रवाह साइटोमेट्री के माध्यम से फ्लोरेसेंस की पुष्टि करें | |
| पैटर्न पर पर्याप्त कोशिकाएं नहीं | कोशिकाओं को ध्यान केंद्रित करने के लिए पीडीएमएस फ्लो सेल, अपकेंद्रित्र, और कम मात्रा में फिर से निलंबित करके कोशिकाओं को इकट्ठा करें | |
| सेल निलंबन में बहुत ज्यादा शेष सीएमओ, स्लाइड पर डीएनए के साथ संकरण | एक और वॉश स्टेप जोड़ें। प्रत्येक धोने के साथ जितना संभव हो उतना सुपरनेट को हटाना सुनिश्चित करें। | |
| समय और तापमान के कारण सीएमओ का बहुत अधिक आंतरिककरण | सीएमओ के साथ कोशिकाओं को लेबल करने के बाद जल्दी से काम करें; कोशिकाओं और बर्फ पर स्लाइड रखने के लिए और बर्फ ठंड अभिकर्दकों का उपयोग करें | |
| कोशिकाओं झुरमुट | ट्राइसाइनाइजेशन के दौरान कोशिकाओं को पर्याप्त रूप से अलग नहीं किया गया | सेल वॉश के दौरान पीबीएस + 0.04% ईडीटीए का उपयोग करें; अंतिम धोने से पहले 35 माइक्रोन फिल्टर के माध्यम से सेल निलंबन पास करें |
| कोशिकाएं गैर-विशेष रूप से पालन करती हैं | यदि एक विशिष्ट क्षेत्र में - स्लाइड पर खरोंच, पीडीएमएस प्रवाह कोशिकाओं के गलत संरेखण, या पैटर्न क्षेत्र के बाहर डीएनए के बिखराव के कारण हो सकता है | खरोंच से बचें, पैटर्न क्षेत्र में पीडीएमएस प्रवाह कोशिकाओं को संरेखित करने के लिए सावधान रहें |
| यदि कोशिकाएं हर जगह पालन कर रही हैं - अपर्याप्त अवरुद्ध या धोने | कोशिकाओं को पैटर्न करने के बाद अधिक वॉश में जोड़ें; वॉश के दौरान अधिक सख्ती से पिप्ट; सेल पैटर्निंग शुरू करने से पहले 1% बीएसए के साथ ब्लॉक करें; सीलनाइज स्लाइड (वैकल्पिक चरण 3) या पुष्टि सीलनीकरण पानी की बूंद के संपर्क कोण को मापने से सफल रहा था | |
| प्रवाह सेल के भीतर बुलबुले बनते हैं | प्लाज्मा ऑक्सीकरण के दौरान बनाई गई त्रुटिएं, असमान हाइड्रोफिलिक सतह | यदि बुलबुले छोटे हैं, तो प्रवाह सेल के इनलेट में पीबीएस जोड़ें और उन्हें धोया जा सकता है। यदि बुलबुले बड़े हैं, तो पीडीएमएस प्रवाह सेल पर कोमल दबाव लागू करें, इनलेट या आउटलेट की ओर बुलबुले को नज करें। |
| कोशिकाएं शुरू में पैटर्न का पालन करती हैं लेकिन वॉश के दौरान हटा दी जाती हैं, अन्य सेल प्रकारों की पैटर्निंग, या हाइड्रोगेल अग्रदूत को जोड़ना | बहुत सख्ती से कतरनी बलों कोशिकाओं को सतह से अलग करने के लिए पैदा कर सकता है | बाद के वॉश, सेल पैटर्निंग के दौर, या हाइड्रोजेल अग्रदूतों को जोड़ने के दौरान पिपेट अधिक धीरे-धीरे। क्योंकि हाइड्रोगेल अग्रदूत चिपचिपा हैं, वे पैटर्न को उखाड़ फेंकने के कारण अधिक संभावना है, इसलिए अतिरिक्त सावधानी बरतें। बहुस्तरीय संरचनाएं शीर्ष-भारी होती हैं और उखाड़ फेंकने के लिए अधिक संवेदनशील होती हैं। |
| 3डी ट्रांसफर के दौरान टिश्यू विकृत | हाइड्रोगेल स्लाइड करने के लिए चिपक जाती है | संपर्क कोण माप का उपयोग कर स्लाइड की हाइड्रोफोबसिटी की पुष्टि करें |
| दोनों किनारों पर पूरी तरह से पीडीएमएस उठाने के लिए रेजर ब्लेड का उपयोग करें, पीबीएस ऊतक के नीचे तैरने की अनुमति | ||
| यह शुद्ध कोलेजन हाइड्रोगेल के साथ हो सकता है - प्रोटीन एकाग्रता या हाइड्रोगेल की संरचना को समायोजित करने पर विचार करें | ||
| कोशिकाएं हाइड्रोगेल के साथ स्थानांतरित नहीं होती हैं और स्लाइड पर रहती हैं | टर्बो DNAse एकाग्रता बढ़ाएं या इनक्यूबेशन समय बढ़ाएं | |
| हाइड्रोगेल पर्याप्त ठोस नहीं है | प्रश्न में हाइड्रोगेल के लिए इनक्यूबेशन समय और/या जेलेशन तंत्र बढ़ाएं (उदाहरण के लिए कोलेजन के लिए, सुनिश्चित करें कि पीएच सही है) | |
| पीडीएमएस हटाने पर हाइड्रोजेल आंसू | प्रयोग शुरू करने से पहले प्लाज्मा ऑक्सीकरण का उपयोग कर पीडीएमएस प्रवाह कोशिकाओं हाइड्रोफिलिक बनाओ ताकि वे मीडिया को जोड़ने पर आसानी से अलग हो जाएं । पीडीएमएस को अलग करने के लिए बहुत धीरे-धीरे संदंश का उपयोग करें। |
तालिका 1: इस प्रोटोकॉल से उत्पन्न होने वाली संभावित विफलताओं की पहचान करने और उन्हें हल करने के लिए एक समस्या निवारण गाइड। विशेष रूप से, पैटर्न के लिए कोशिकाओं के खराब आसंजन कई मूल कारण हो सकते हैं और इस गाइड को उन मुद्दों की पहचान और समाधान में मदद करनी चाहिए।
पूरक फाइल 1. इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक फाइल 2. इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक फाइल 3. इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
पूरक फाइल 4. इस फाइल को डाउनलोड करने के लिए यहां क्लिक करें।
इस लेख में, हम इन विट्रो सेल कल्चर प्रयोगों के लिए 2डी और 3डी में कोशिकाओं के उच्च-रिज़ॉल्यूशन पैटर्निंग के लिए एक विस्तृत प्रोटोकॉल प्रस्तुत करते हैं। इस विधि के पहले प्रकाशित संस्करणों के विपरीत, यहां प्रस्तुत प्रोटोकॉल उपयोगिता पर केंद्रित है: इसके लिए अत्यधिक विशिष्ट उपकरणों की आवश्यकता नहीं है और सभी अभिकर्षकों को कस्टम संश्लेषण की आवश्यकता के बजाय विक्रेताओं से खरीदा जा सकता है। अन्य सेल माइक्रोपैटर्निंग विधियों के विपरीत, यह विधि तेजी से और सेल-प्रकार नास्तिक है: इसके लिए एक्सट्रासेलुलर मैट्रिक्स प्रोटीन15के लिए विशिष्ट आसंजन की आवश्यकता नहीं है। सीएमओ-डीपीएसी द्वारा पैटर्न वाली कोशिकाओं को एक एक्सट्रासेलुलर मैट्रिक्स जैसे मैट्रिक्स जैसे मैट्रिक्स या कोलेजन के भीतर एम्बेडेड किया जा सकता है, जिसके परिणामस्वरूप 3 डी संस्कृतियों में बहुत अधिक स्थानिक संकल्प के साथ वर्तमान में एक्सट्रूज़न प्रिंटिंग-आधारित विधियों22के साथ संभव है। सीएमओ-डीपीएसी का उपयोग प्रति स्लाइड सैकड़ों से हजारों सूक्ष्म सुविधाओं को बनाने के लिए किया जा सकता है, जिससे एक ही समय में कई प्रतिकृति का प्रदर्शन किया जा सकता है।
इस प्रोटोकॉल की सफलता में सबसे महत्वपूर्ण मापदंडों में से एक पैटर्न स्लाइड के शीर्ष पर प्रवाह कोशिकाओं में जोड़ा कोशिकाओं का घनत्व है। आदर्श रूप में, घनत्व कम से कम 25 मिलियन कोशिकाओं/एमएल होना चाहिए। जब प्रवाह कोशिकाओं में लोड किया जाता है, तो कोशिकाओं के इस घनत्व के परिणामस्वरूप पैटर्न(पूरक चित्रा 2B)के ऊपर कोशिकाओं की लगभग करीब-करीब पैक मोनोलेयर होती है। ये उच्च कोशिका घनत्व इस संभावना को अधिकतम करते हैं कि एक कोशिका सीधे डीएनए स्थान के शीर्ष पर बस जाएगी और पालन करेगी। सेल घनत्व को कम करने से समग्र पैटर्निंग दक्षता कम हो जाएगी। इस प्रोटोकॉल में एक और महत्वपूर्ण कदम सीएमओ समाधान जोड़ने से पहले पीबीएस या सीरम मुक्त मीडिया में कोशिकाओं को अच्छी तरह से निलंबित करना है। सीएमओ विभाजन बहुत तेजी से कोशिका झिल्ली में और सीएमओ समाधान को सीधे सेल पेलेट में जोड़ने से कोशिकाओं की विषम लेबलिंग होगी। सेल निलंबन के लिए सीएमओ समाधान जोड़ने के बाद, यह अच्छी तरह से पाइपिंग द्वारा मिश्रण करने के लिए महत्वपूर्ण है ताकि कोशिकाओं को समान रूप से सीएमओ के साथ लेबल किया जाता है। इन्क्यूबेशन के बाद, कई अपकेंद्रित्र के माध्यम से अतिरिक्त सीएमओ को अच्छी तरह से धोना और चरणों को धोना आवश्यक है। सेल निलंबन में मौजूद अतिरिक्त मुक्त सीएमओ कांच की स्लाइड पर पैटर्न वाले अमीन-संशोधित डीएनए से बांधेंगे, निलंबन में सीएमओ-संशोधित कोशिकाओं के संकरण और आसंजन को अवरुद्ध करेंगे। इस प्रोटोकॉल के लिए समय भी एक महत्वपूर्ण विचार है । सीएमओ का उपयोग करते समय जितनी जल्दी हो सके काम करना और कोशिकाओं को बर्फ पर रखना महत्वपूर्ण है ताकि सीएमओ के आंतरिककरण को कम किया जा सके और सेल व्यवहार्यता को अधिकतम किया जा सके। फ्लो साइटोमेट्री प्रयोगों से पता चला है कि सीएमओ एलएमओ के रूप में सेल की सतह पर लंबे समय तक नहीं रहते हैं, जिसमें बर्फ३६पर दो घंटे से अधिक इनक्यूबेशन सीएमओ परिसरों का 25% नुकसान होता है । इसके अलावा, कोशिकाओं की व्यवहार्यता कम हो जाएगी क्योंकि सेल हैंडलिंग समय बढ़ जाता है। व्यवहार्यता को जल्दी से काम करके, बर्फ पर कोशिकाओं को रखने, बर्फ-ठंडे अभिकर् ती का उपयोग करके, और कुछ पोषक तत्व प्रदान करने के लिए सीरम-मुक्त मीडिया का उपयोग करके अधिकतम किया जा सकता है।
हालांकि सीएमओ-DPAC उच्च परिशुद्धता के साथ कोशिकाओं पैटर्निंग द्वारा सेल जीव विज्ञान का अध्ययन करने का एक शक्तिशाली तरीका हो सकता है, यह अपनी सीमाएं है । सीएमओ-डीपीएसी प्रयोग चुनौतीपूर्ण हो सकते हैं, विशेष रूप से प्रयोगात्मक जटिलता को कई सेल प्रकारों, परतों, या 3 डी सेल संस्कृति(पूरक फ़ाइल 1)के साथ जोड़ा जाता है। इस प्रोटोकॉल को शुरू करते समय प्रयोगात्मक विफलताएं आम हो सकती हैं, जैसा कि तालिका 1में वर्णित है। इसलिए, हम अनुशंसा करते हैं कि उपयोगकर्ता गुणवत्ता नियंत्रण जांच संस्थान (पुष्टि करते हैं कि डीएनए स्लाइड पर मौजूद है, इस बात की पुष्टि करता है कि कोशिकाओं को डीएनए (चरण 8) के साथ पर्याप्त रूप से लेबल किया गया है, यह पुष्टि करते हुए कि अतिरिक्त कोशिकाओं को अच्छी तरह से धोया गया है, आदि) यह सुनिश्चित करने के लिए कि प्रयोग सफल होता है और उन चरणों की पहचान करने के लिए जिन्हें आगे अनुकूलन की आवश्यकता हो सकती है। हमें उम्मीद है कि इस पांडुलिपि में दी गई जानकारी और इसकी पूरक फाइलों से किसी भी आवश्यक समस्या निवारण को सुगम बनाने में मदद मिलेगी।
कोलेस्ट्रॉल एक जैव सक्रिय अणु है जिसका आंतरिककरण कोशिका चयापचय, जीन अभिव्यक्ति और झिल्ली तरलता37, 38को प्रभावित कर सकता है। एक पिछले अध्ययन में सीएमओ और एलएमओ-लेबल कोशिकाओं की जीन अभिव्यक्ति पर पड़ने वाले प्रभावों की तुलना एकल कोशिका आरएनए अनुक्रमण का उपयोग करके की गई थी । सीएमओ लेबल एचईके कोशिकाओं ने अवेलेबल और एलएमओ लेबल वाली कोशिकाओं३६की तुलना में जीन अभिव्यक्ति को बदल दिया था । सीएमओ के साथ लेबलिंग कोशिकाओं के परिणामस्वरूप अवेलेबल नियंत्रणों के सापेक्ष आठ जीनों की अंतर अभिव्यक्ति (> 1.5 गुना) हुई, जिसमें एपी 2बी1 शामिल है, जिसे कोलेस्ट्रॉल और स्फिंगोलिपिड परिवहन (जेनकार्ड्स) और मालट1 से जोड़ा गया है, जो कोलेस्ट्रॉल संचय39को नियंत्रित करने वाला एक लंबा गैर-कोडिंग आरएनए है। जबकि मामूली, इन प्रतिलेखन प्रतिक्रियाओं फिर भी चिंता का विषय हो सकता है अगर प्रश्न में प्रयोग चयापचय, झिल्ली गतिशीलता, या कोशिकाओं में अंय कोलेस्ट्रॉल से जुड़े रास्ते का अध्ययन कर रहा है ।
यह प्रोटोकॉल लचीला है और प्रत्येक प्रयोग की जरूरतों को पूरा करने के लिए समायोजित किया जा सकता है। क्योंकि सीएमओ किसी भी विशिष्ट रिसेप्टर का उपयोग करने के बजाय लिपिड झिल्ली में खुद को सम्मिलित करता है, विधि सेल प्रकार नास्तिक है (एचयूवीसी, एमसीएफ 10ए, एचईके, और एमडीकेएस यहां प्रदर्शित किए गए हैं)। हालांकि कोलेस्ट्रॉल हमारे पहले प्रकाशित एलएमओ की तुलना में एक अलग हाइड्रोफोबिक लंगर है, हम इस प्रकार अब तक उन्हें इसी तरह व्यवहार करने के लिए पाया है। इस प्रकार, हम उम्मीद करेंगे कि सीएमओ एलएमओ के साथ पहले प्रकाशित किए गए किसी भी प्रकार के सेल प्रकारों के साथ काम करें, जिसमें तंत्रिका स्टेम कोशिकाओं, फाइब्रोब्लास्ट, परिधीय रक्त मोनोन्यूक्लियर कोशिकाओं, ट्यूमर कोशिकाओं और प्राथमिक स्तन एपिथेलियल कोशिकाओं6,23, 27,29,36 तक सीमित नहीं है। . सीएमओ लेबलिंग TLR9 को प्रोत्साहित नहीं करता है, सुझाव है कि प्रोटोकॉल प्रतिरक्षा कोशिकाओं के साथ संगत है । सीएमओ का झिल्ली समावेश कुल कोशिका आकार का कार्य है और सेल ग्लाइकोकलिक्स35में नकारात्मक आवेश की डिग्री है। इस प्रकार, हमने झिल्ली निगमन की सीमा का परीक्षण करने के लिए एक प्रोटोकॉल (चरण 8) शामिल किया है जो तेजी से अनुकूलन के लिए उत्तरदायी है। प्रत्येक सेल पैटर्न की विशिष्ट विशेषताएं अनिवार्य रूप से प्रयोगात्मक डिजाइन के आधार पर भिन्न होंगी (अधिक मार्गदर्शन के लिए पूरक फ़ाइल 1 देखें)। हालांकि डीएनए पैटर्निंग के लिए ऊपर वर्णित फोटोपेटर्निंग प्रोटोकॉल की सिफारिश की जाती है, लेकिन अमीन-डीएनए समाधान की बूंदों को सीमित करने की किसी भी विधि को काम करना चाहिए, जैसे कि उच्च-संकल्प ड्रॉपलेट प्रिंटर का उपयोग। पैटर्न रिज़ॉल्यूशन और न्यूनतम सुविधा रिक्ति उपयोग की गई विधि के आधार पर भिन्न होगी। इस प्रोटोकॉल के डीएनए-फोटोपैटर्निंग वर्गों को अन्य तरीकों के साथ जोड़ना भी सैद्धांतिक रूप से संभव है, जिनका उपयोग डीएनए के साथ कोशिकाओं को लेबल करने के लिए किया गया है, जैसे कि डीएनए के साथ डीएनए को अप्रासंगिक-व्यक्त जिंक उंगलियों40के लिए संकरित डीएनए के साथ, एनएचएस-संयुग्मित डीएनए41का उपयोग करके, और कोशिका की सतह पर अज़िडो सियालिक एसिड अवशेषों को फॉस्फिन-कॉन्जुगेट डीएनए42 के साथ प्रतिक्रिया करना . सीएमओ-डीपीएसी को विभिन्न प्रकार के प्रयोगों पर लागू किया जा सकता है जिनके लिए सेल-सेल अंतर पर तंग नियंत्रण की आवश्यकता होती है, जिसमें कोशिकाओं के जोड़े के बीच बातचीत का अध्ययन, "प्रेषक" कोशिकाओं से "रिसीवर" कोशिकाओं के संकेतों के हस्तांतरण को देखते हुए सह-संस्कृति प्रयोग, और स्टेम सेसियभेद6,29 पर पास के बाह्राशक संकेतों के प्रभाव की जांच शामिलहै। . इस विधि का उपयोग माइक्रोटिस्यूज बनाने के लिए भी किया जा सकता है जिसका उपयोग तीन आयामों में सेल माइग्रेशन का अध्ययन करने, ऊतकों में कोशिकाओं के आत्म-संगठन23,27और कोशिकाओं और ईसीएम27के बीच गतिशील परस्पर क्रिया के लिए किया जा सकता है। हमें उम्मीद है कि यह प्रोटोकॉल शोधकर्ताओं को अपनी प्रयोगशालाओं में उच्च-रिज़ॉल्यूशन डीएनए-आधारित सेल पैटर्निंग के नए अनुप्रयोगों का पता लगाने के लिए एक सुलभ मंच प्रदान करेगा।
Z.J.G. प्रोवेंस बायोसाइंसेज में एक सलाहकार और इक्विटी धारक है।
लेखक इस प्रोटोकॉल का परीक्षण करने के लिए जेरेमी गार्सिया और यूसीएसएफ बायोमेडिकल माइक्रो एंड नैनोटेक्नोलॉजी कोर में उपकरणों पर प्रशिक्षण प्रदान करने के लिए भूषण खार्बिकर का शुक्रिया अदा करना चाहेंगे । इस शोध के हिस्से में रक्षा स्तन कैंसर अनुसंधान कार्यक्रम (W81XWH-10-1-1023 और W81XWH-13-1-0221), NIH (U01CA19315, DP2 HD080351-01, से अनुदान द्वारा समर्थित किया गया था, 1R01CA190843-01, 1R21EB019181-01A, और 1R21CA182375-01A1), एनएसएफ (MCB1330864), और सेलुलर निर्माण के लिए UCSF केंद्र (DBI-1548297), एक NSF विज्ञान और प्रौद्योगिकी केंद्र । O.J.S. एक एनएसएफ ग्रेजुएट रिसर्च फैलोशिप, एक सिबेल छात्रवृत्ति, और एक पीओ छात्रवृत्ति द्वारा वित्त पोषित किया गया था । जेडजेजी और ए.आर.ए. चान-जुकरबर्ग बायोहब जांचकर्ता हैं ।
| Name | Company | Catalog Number | Comments |
| 2-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155379 | |
| Acetic Acid | Sigma-Aldrich | A6283 | |
| Adapter with External SM1 Threads and Internal SM3 Thread | ThorLabs | SM3A1 | |
| Aldehyde Functionalized Slides | Schott | Nexterion Slide AL | Store under dry conditions after opening. |
| All Plastic Syringes, 1 mL | Fisher Scientific | 14-817-25 | |
| Amine-Modified DNA Oligo | IDT | n/a | See Supplemental File 1 for suggested sequences. |
| Aspheric Condenser Lens | ThorLabs | ACL7560 | |
| Borosilicate Disc, 6in Diameter X 1/2in Thick | Chemglass | CG-1906-23 | |
| Cell Culture Dishes 60x15 mm style | Corning | 353002 | |
| Cholesterol-Modified Oligo | IDT | n/a | See Supplemental File 1 for suggested sequences. |
| Diamond Scribe | Excelta | 475B | |
| DNA Oligonucleotide | IDT | n/a | See Supplemental File 1 for suggested sequences. |
| DPBS, no calcium, no magnesium | Thermo Fisher Scientific | 14190250 | |
| Isopropyl Alcohol | Sigma-Aldrich | 278475 | |
| Matrigel Matrix, Growth Factor Reduced | Corning | 354230 | |
| Methylene Chloride (Stabilized/Certified ACS) | Fisher Scientific | D37-4 | |
| MF-321 Developer | Kayaku Advanced Materials | n/a | |
| Microposit S1813 Positive Photoresist | Kayaku Advanced Materials | n/a | |
| Ø3" Adjustable Lens Tube, 0.81" Travel | ThorLabs | SM3V10 | |
| Oven | Thermo Scientific | 51-028-112H | |
| PE-50 Compact Benchtop Plasma Cleaning System | Plasma Etch | PE-50 | |
| Photomask (custom) | CAD/Art Services | n/a | Minimum feature size guaranteed by CAD/Art Services is 10 microns. |
| Razor Blades | Fisher Scientific | 12-640 | |
| RCT Basic Hot Plate | IKA | 3810001 | |
| Silicon Wafer (100 mm) | University Wafer | 590 | |
| Sodium Borohydride, 98%, granules | Acros Organics | 419471000 | |
| Spin Coater Kit | Instras | SCK-200 | This is a low cost option, but any spin coater that can maintain a speed of 3000 rpm will suffice. |
| SU-8 2075 | Microchem | Y111074 0500L1GL | |
| SU-8 Developer | Microchem | Y020100 4000L1PE | |
| Sylgard 184 Silicone Elastomer Kit | Dow | 2646340 | |
| Syringe Needles | Sigma-Aldrich | Z192341 | |
| T-Cube LED Driver, 1200 mA Max Drive Current | ThorLabs | LEDD1B | |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl dimethylchlorosilane | Gelest | SIT8170.0 | |
| Triethylamine | Sigma-Aldrich | 90335 | |
| Turbo DNase | Thermo Fisher Scientific | AM2238 | |
| Tweezers Style N7 | VWR | 100488-324 | The curved shape of these tweezers is essential for delicately picking up the PDMS flow cells containing patterned tissues. |
| UV LED (365 nm, 190 mW (Min) Mounted LED, 700 mA) | ThorLabs | M365L2 | |
| Wafer Tweezers | Agar Scientific | T5063 | |
| WHEATON Dry-Seal vacuum desiccator | Millipore Sigma | W365885 |
- Kreeger, P. K., Strong, L. E., Masters, K. S. Engineering approaches to study cellular decision-making. Annual Review of Biomedical Engineering. , 49-72 (2018).
- Goubko, C. a., Cao, X. Patterning multiple cell types in co-cultures: A review. Materials Science and Engineering C. 29 (6), 1855 (2009).
- Sun, W., et al. The bioprinting roadmap. Biofabrication. 12 (2), 022002 (2020).
- Liu, W. F., Chen, C. S. Cellular and multicellular form and function. Advanced Drug Delivery Reviews. 59 (13), 1319-1328 (2007).
- Duffy, R. M., Sun, Y., Feinberg, A. W. Understanding the role of ECM protein composition and geometric micropatterning for engineering human skeletal muscle. Annals of Biomedical Engineering. 44 (6), 2076-2089 (2016).
- Chen, S., et al. Interrogating cellular fate decisions with high-throughput arrays of multiplexed cellular communities. Nature Communications. 7, 10309 (2016).
- Shaya, O., et al. Cell-cell contact area affects notch signaling and notch-dependent patterning. Developmental Cell. 40 (5), 505-511 (2017).
- Rao, N., et al. A co-culture device with a tunable stiffness to understand combinatorial cell-cell and cell-matrix interactions. Integrative Biology. 5 (11), 1344 (2013).
- Sriraghavan, V., Desai, R. A., Kwon, Y., Mrksich, M., Chen, C. S. Micropatterned dynamically adhesive substrates for cell migration. Langmuir. 26 (22), 17733-17738 (2010).
- Wong, L., Pegan, J. D., Gabela-Zuniga, B., Khine, M., McCloskey, K. E. Leaf-inspired microcontact printing vascular patterns. Biofabrication. 9 (2), 021001 (2017).
- Chen, T. H., et al. Directing tissue morphogenesis via self-assembly of vascular mesenchymal cells. Biomaterials. 33 (35), 9019-9026 (2012).
- Laurent, J., et al. Convergence of microengineering and cellular self-organization towards functional tissue manufacturing. Nature Biomedical Engineering. 1 (12), 939-956 (2017).
- Lin, C., Khetani, S. R. Micropatterned co-cultures of human hepatocytes and stromal cells for the assessment of drug clearance and drug-drug interactions. Current Protocols in Toxicology. 2017, 1-23 (2017).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proceedings of the National Academy of Sciences of the United States of America. 104 (14), 5722-5726 (2007).
- D'Arcangelo, E., McGuigan, A. P. Micropatterning strategies to engineer controlled cell and tissue architecture in vitro. BioTechniques. 58 (1), 13-23 (2015).
- Martinez-Rivas, A., González-Quijano, G. K., Proa-Coronado, S., Séverac, C., Dague, E. Methods of micropatterning and manipulation of cells for biomedical applications. Micromachines. 8 (12), (2017).
- Lee, S., et al. Simple lithography-free single cell micropatterning using laser-cut stencils. Journal of Visualized Experiments. (158), e60888 (2020).
- Strale, P. O., et al. Multiprotein printing by light-induced molecular adsorption. Advanced Materials. 28 (10), 2024-2029 (2016).
- Melero, C., et al. Light-induced molecular adsorption of proteins using the primo system for micro-patterning to study cell responses to extracellular matrix proteins. Journal of Visualized Experiments. (152), e60092 (2019).
- Reid, J. A., Mollica, P. M., Bruno, R. D., Sachs, P. C. Consistent and reproducible cultures of large-scale 3D mammary epithelial structures using an accessible bioprinting platform. Breast Cancer Research. , 1-13 (2018).
- Wang, Z., Lee, S. J., Cheng, H. -. J., Yoo, J. J., Atala, A. 3D bioprinted functional and contractile cardiac tissue constructs. Acta Biomaterialia. 70, 48-56 (2018).
- Miri, A. K., et al. Effective bioprinting resolution in tissue model fabrication. Lab on a Chip. 19 (11), 2019-2037 (2019).
- Todhunter, M. E., et al. Programmed synthesis of three-dimensional tissues. Nature Methods. 12 (10), 975-981 (2015).
- Todhunter, M. E., Weber, R. J., Farlow, J., Jee, N. Y., Gartner, Z. J. Fabrication of 3D microtissue arrays by DNA programmed assembly of cells. Current Protocols in Chemical Biology. 8 (3), 147-178 (2016).
- Csizmar, C. M., Petersburg, J. R., Wagner, C. R. Programming cell-cell interactions through non-genetic membrane engineering. Cell Chemical Biology. 25 (8), 931-940 (2018).
- Weber, R. J., Liang, S. I., Selden, N. S., Desai, T. A., Gartner, Z. J. Efficient targeting of fatty-acid modified oligonucleotides to live cell membranes through stepwise assembly. Biomacromolecules. 15 (12), 4621-4626 (2014).
- Hughes, A. J., et al. Engineered tissue folding by mechanical compaction of the mesenchyme. Developmental Cell. 44 (2), 165-178 (2018).
- Weber, R. J., et al. Rapid organoid reconstitution by chemical micromolding. ACS Biomaterials Science & Engineering. 2 (11), 1851-1855 (2016).
- Scheideler, O. J., et al. Recapitulating complex biological signaling environments using a multiplexed, DNA-patterning approach. Science Advances. 6 (12), (2020).
- Viola, J. M., et al. Guiding cell network assembly using shape-morphing hydrogels. Advanced materials (Deerfield Beach, Fla.). , 2002195 (2020).
- Mohammad, A., Davis, M., Aprelev, A., Ferrone, F. A. Note: Professional grade microfluidics fabricated simply. Review of Scientific Instruments. 87 (10), 1-4 (2016).
- Lee, O. J., Chuah, H. S., Umar, R., Chen, S. K., Yusra, A. F. I. Construction of cost effective homebuilt spin coater for coating amylose-amylopectin thin films. Journal of Fundamental and Applied Sciences. 9 (2), 279 (2018).
- Webb, K., Hlady, V., Tresco, P. A. Relative importance of surface wettability and charged functional groups on NIH 3T3 fibroblast attachment, spreading, and cytoskeletal organization. Journal of Biomedical Materials Research. 41 (3), 422-430 (1998).
- Processing Guidelines for: SU-8 2025, SU-8 2035, SU-8 2050, SU-8 2075. Microchem SU-8 2000 Permanent Expoxy Negative Photoresist Available from: https://kayakuam.com/wp-content/uploads/2019/09/SU-82000DataSheet2025thru2075Ver4.pdf (2019)
- Palte, M. J., Raines, R. T. Interaction of nucleic acids with the glycocalyx. Journal of the American Chemical Society. 134 (14), 6218-6223 (2012).
- McGinnis, C. S., et al. MULTI-seq: sample multiplexing for single-cell RNA sequencing using lipid-tagged indices. Nature Methods. 16 (7), 619-626 (2019).
- Maxfield, F. R., van Meer, G. Cholesterol, the central lipid of mammalian cells. Current Opinion in Cell Biology. 22 (4), 422-429 (2010).
- Luo, J., Yang, H., Song, B. L. Mechanisms and regulation of cholesterol homeostasis. Nature Reviews Molecular Cell Biology. 21 (4), 225-245 (2020).
- Liu, L., Tan, L., Yao, J., Yang, L. Long non-coding RNA MALAT1 regulates cholesterol accumulation in ox-LDL-induced macrophages via the microRNA-17-5p/ABCA1 axis. Molecular Medicine Reports. 21 (4), 1761-1770 (2020).
- Mali, P., Aach, J., Lee, J. H., Levner, D., Nip, L., Church, G. M. Barcoding cells using cell-surface programmable DNA-binding domains. Nature Methods. 10 (5), 403-406 (2013).
- Hsiao, S. C., et al. Direct cell surface modification with DNA for the capture of primary cells and the investigation of myotube formation on defined patterns. Langmuir. 25 (12), 6985-6991 (2009).
- Gartner, Z. J., Bertozzi, C. R. Programmed assembly of 3-dimensional microtissues with defined cellular conductivity. Proceedings of the National Academy of Sciences. (17), 1-5 (2009).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved