A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
أداة تحليلية يحدد مقدار التغييرات الخلوية الصرف من التقاط صور ثلاثية الأبعاد الإسفار
In This Article
Summary
وضعنا منصة البرمجيات التي تستخدم Imaris العلوم العصبية، وImarisXT MATLAB لقياس التغيرات في مورفولوجية شكل غير معروف مأخوذ من ثلاثي الأبعاد من الخلايا مبائر مضان واحد. ويمكن استخدام هذا النهج الجديد لقياس التغييرات في شكل الخلية بعد تفعيل مستقبلات وبالتالي يمثل أداة ممكنة إضافية لاكتشاف المخدرات.
Abstract
The most common software analysis tools available for measuring fluorescence images are for two-dimensional (2D) data that rely on manual settings for inclusion and exclusion of data points, and computer-aided pattern recognition to support the interpretation and findings of the analysis. It has become increasingly important to be able to measure fluorescence images constructed from three-dimensional (3D) datasets in order to be able to capture the complexity of cellular dynamics and understand the basis of cellular plasticity within biological systems. Sophisticated microscopy instruments have permitted the visualization of 3D fluorescence images through the acquisition of multispectral fluorescence images and powerful analytical software that reconstructs the images from confocal stacks that then provide a 3D representation of the collected 2D images. Advanced design-based stereology methods have progressed from the approximation and assumptions of the original model-based stereology1 even in complex tissue sections2. Despite these scientific advances in microscopy, a need remains for an automated analytic method that fully exploits the intrinsic 3D data to allow for the analysis and quantification of the complex changes in cell morphology, protein localization and receptor trafficking.
Current techniques available to quantify fluorescence images include Meta-Morph (Molecular Devices, Sunnyvale, CA) and Image J (NIH) which provide manual analysis. Imaris (Andor Technology, Belfast, Northern Ireland) software provides the feature MeasurementPro, which allows the manual creation of measurement points that can be placed in a volume image or drawn on a series of 2D slices to create a 3D object. This method is useful for single-click point measurements to measure a line distance between two objects or to create a polygon that encloses a region of interest, but it is difficult to apply to complex cellular network structures. Filament Tracer (Andor) allows automatic detection of the 3D neuronal filament-like however, this module has been developed to measure defined structures such as neurons, which are comprised of dendrites, axons and spines (tree-like structure). This module has been ingeniously utilized to make morphological measurements to non-neuronal cells3, however, the output data provide information of an extended cellular network by using a software that depends on a defined cell shape rather than being an amorphous-shaped cellular model. To overcome the issue of analyzing amorphous-shaped cells and making the software more suitable to a biological application, Imaris developed Imaris Cell. This was a scientific project with the Eidgenössische Technische Hochschule, which has been developed to calculate the relationship between cells and organelles. While the software enables the detection of biological constraints, by forcing one nucleus per cell and using cell membranes to segment cells, it cannot be utilized to analyze fluorescence data that are not continuous because ideally it builds cell surface without void spaces. To our knowledge, at present no user-modifiable automated approach that provides morphometric information from 3D fluorescence images has been developed that achieves cellular spatial information of an undefined shape (Figure 1).
We have developed an analytical platform using the Imaris core software module and Imaris XT interfaced to MATLAB (Mat Works, Inc.). These tools allow the 3D measurement of cells without a pre-defined shape and with inconsistent fluorescence network components. Furthermore, this method will allow researchers who have extended expertise in biological systems, but not familiarity to computer applications, to perform quantification of morphological changes in cell dynamics.
Protocol
1. تحليل ثلاثي الأبعاد من المظهرية وحيدة الخلية التغييرات المظهرية
- وتقاس الكلى الجنينية البشرية (HEK293) مع خلايا هيماغلوتينين (HA)-الموسومة كورتيكوتروبين الإفراج عن عامل مستقبلات-2 (CRF-R2)، وهو بروتين G-يقترن مستقبلات (GPCR) كما هو موضح سابقا 4، 5.
- تركت دون علاج الخلايا (أي علاج، NT)، مع حفز يجند الذاتية CRF-R2، كورتيكوتروبين عامل الإفراج، CRF (1 ميكرومتر، 30 دقيقة)، أو سابقة التجهيز مع خصم CRF-R2 انتقائية، ومكافحة sauvagine 30 (AS -30، 1 ميكرومتر، 30 دقيقة) قبل العلاج ناهض.
- تم إصلاحها ثم الخلايا، وتعامل مع permeabilized HA للسامية. كان CRF-R2 تصور باستخدام اليكسا 594 نانومتر المتقارن المضادة للماوس (مفتش 1) الأجسام المضادة. وقد استخدم دابي لتصور المرحلة التفتلي نوى.
- للحد من الذاتية مجرب، لم تكن معروفة في الظروف التجريبية حتى بعد تم الحصول عليها من الصور وتحليلها.
- حصلنا على صورةS من الخلايا HEK293 الثابتة باستخدام خطة لامزيغ-63x/1.4 الهدف DIC النفط وزايس LSM المجهر متحد البؤر META 510 متصلة مترابطة متكاملة نظام ليزر ثنائي الفوتون تتكون من الليزر فيردي-V5 و900-F ميرا نظام ليزر.
- أثناء عملية الحصول على البيانات، ومجزأة الخلايا كل من باجتزاء متعددة الأطياف، NM 488، و 790 نانومتر (نانومتر ~ تحويلة 350 2ph.) وZ-تقسيم (0.5 ميكرومتر الزيادات) لتشمل البيانات من الغشاء النووي لمستقبلات للخارج الخلية الخارجي الأطراف.
- تمت معالجة البيانات أولا باستخدام مضان Imaris، والذي يسمح التصور وتجزئة بيانات المجهر 3D، وتم إنشاء نموذج 3D تتألف من voxels مكعب للتحليل المظهرية.
ثم، تم استخدام وحدة لImaris XT Imaris التفاعل مع الكمبيوتر MATLAB لغة البرنامج لتحديد إحداثيات المكان ملحقات GPCR.
على أن تأخذ في الاعتبار التباين الخلوية، وحصلنا على تحليل الصور مضان تاكEN من 22 الخلايا: يوجد علاج (NT) (ن = 7)، ناهض (CRF) العلاج (ن = 8) وقبل العلاج مع خصم (AS-30) قبل العلاج ناهض (ن = 7) (الشكل 2) .- يجب المنطقة الاهتمام (ROI) تشمل خلية واحدة غير موجود في المرحلة التفتلي بالموقع وليس على مقربة من الخلايا الأخرى. بهذه الطريقة، سوف تشمل تحليل الخلايا لا مع نواة واحدة فقط وملحقات مستقبلات مضطرب من جراء قربها من الخلايا الأخرى.
- أعيد بناؤها أولا هيكل 3D من البيانات الخلوية متعددة الأطياف باستخدام مضان Imaris (v.7.1.1).
- بعد الخوارزمية التي صممها Imaris، واستخدم لأول مرة لتمثيل تقديم السطحية الغشاء النووي. سوف Imaris تحديد إذا كان هناك أكثر من واحد في النواة ROI.
- ثم تم استخدام خوارزمية البقع-إنشاء ملحق لتحديد موقع CRF-R2. واستخدم البقع لأنه كشف يعوض عن الضوضاء الخلفية وكثافة غير النظامية للشبكة معقدة غير متبلور على شكل الخلايا.
- لتحقيق أقصى قدر من إدراج كل وحدة من كشف مضان من CRF-R2، أنشئت قطر للبقع 'إلى 0.2 ميكرون، وهو أصغر وحدة في الصورة لاستقراء المعلومات متميزة في شكل كثافة يقاس باستخدام فلتر التمويه. تأسست تصفية بقعة في إنشاء المواقع عملية آلية. البرنامج، ومع ذلك، يعطي المستخدم المرونة اللازمة لاستخدام الفلاتر لتحديد المعلمات.
- لتجنب اقتطاع البيانات، تم تحويل مجموعة من البيانات 8 بت (غير موقعة) نقطة ثابتة، إلى 32 بت عشرية.
- وتبادل البيانات فوكسل شدة على الفور باستخدام إحداثيات البيانات Imaris XT حدة تفاعل مع MATLAB والمكان المحدد المكاني للكل بقعة تم تحديد عن طريق إجراء التحول المسافة باستخدام الغشاء النووي كنقطة مرجعية (الشكل 3).
- يمكن كميا البيانات الناتجة وعرضها في شكل رسوم بيانية لياليtatistical التحليل. وأجريت مقارنات بين المجموعات باستخدام اتجاهين ANOVA والبعدي Bonferroni. يتم عرض البيانات كما يعني ± SD. وتعتبر فروق ذات دلالة إحصائية في * 0،05 وأدلى حسابات مع بريزم GraphPad 5.02 (الشكل 4).
2. ممثل النتائج
للتدليل على قوة نهجنا، ونحن كميا التغيرات الخلوية التي تنتج عن التفاعل بين مستقبلات البروتين يقترن G (GPCRs) والموجهة القشرية عامل الإفراج عن مستقبلات-2 (CRF-R2) مع يجند لها CRF الذاتية في الخلايا HEK293 بالنقل.
نعرض التي تقع CRF-R2 المستقبلات في الغشاء البلازمي والمشروع من مناطق محدودة من غشاء الخلايا (الشكل 2A والفيلم 1). باستخدام التحليل التقليدي 2D، فمن الممكن الكشف عن هذه المجموعة الفرعية من مستقبلات CRF-R2 خارج الخلية إلا إذا حللنا مستقبلات الالتصاق بورجات على زجاج يغطي. وبالتالي نفقد أي معلومات أخرى مستمدة من البيانات Z-مكدسة متعددة الأطياف (الشكل 5).
عندما يتم التعامل مع الخلايا CRF، وتقلص إلى حد كبير المستقبلات خارج الخلية، ويتضح ذلك من انخفاض في المسافة من البقع من غشاء البلازما. وإعادة توزيعها أيضا أنها من مواقع محدودة في المقام الأول إلى عدد من المواقع المتميزة (2B الشكل والفيلم 2).
يتم منع تأثير على توزيع CRF الغشاء مستقبلات من المعالجة المسبقة مع خصم CRF-R2 محددة، antisavagine 30 (AS-30) ونجد أن ملحقات CRF-R2 لا تتغير (الشكل 2C والفيلم 3).
توزيع البعيدة من البقع، وتآمر في فترات 5 ميكرومتر لون الطيف مشفرة، ويستخدم لتصور المسافة بين voxels من الغشاء النووي. ويوجد علاج مضاد pretreatmالأنف والحنجرة (AS-30، 1 ميكرومتر، 30 دقيقة) قبل بدء العلاج ناهض (CRF، 1 ميكرومتر، 30 دقيقة) لا تظهر أي مهمة (م) الفرق في الانكماش GPCR. علاج الخلايا مع ناهض (CRF، 1 ميكرومتر، 30 دقيقة) يقلل تدريجيا من عدد CRF-R2 المحتوية على voxels بالمقارنة مع أي علاج، 0-5 ميكرومتر (م)، 6-15 ميكرون (** p < 0.01) و> 15 ميكرون (*** p <0.005)، أو مقارنة AS-30 معاملة، 0-10 ميكرون (NS)، 11-15 ميكرومتر (** p <0.01) و> 15 ميكرون (*** p <0.005) (الشكل 4).
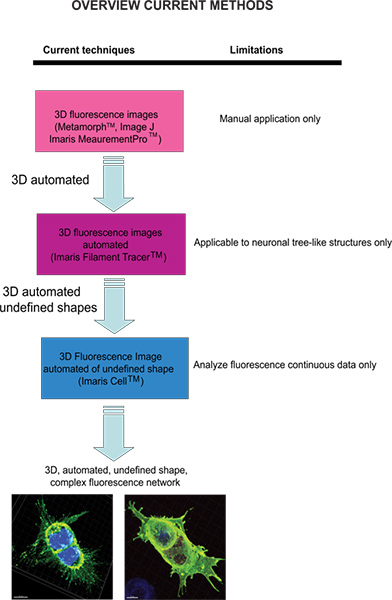
الشكل 1. الرسم البياني من التقنيات المتاحة حاليا والحد من أجل تحليل الصور مضان. اضغط هنا لمشاهدتها بشكل اكبر شخصية .
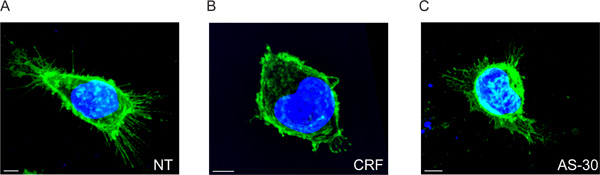
الشكل 2. 3D الصور المتعددة الأطياف مضان من الخلايا المصابة بالعدوى بالنقل مع HEK293 HA-CRF-R2 قبل وبعد العلاج ناهض وخصم. الصور المدمجة تبين HA-CRF-R2-بحثها استخدام الألغام المضادة للHA وتصور باستخدام الأجسام المضادة مترافق اليكسا 488 الثانوية المضادة للماوس (مفتش 1)، وكان يستخدم لتصور دابي نوى. تم الحصول على الصور مبائر المجهر مع الليزر (CLS) المسح. مقياس بار 5 ميكرون.
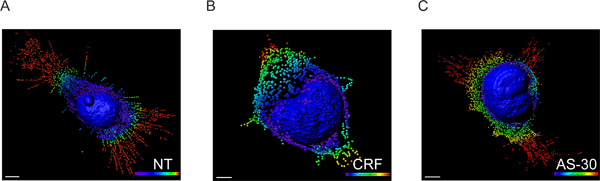
الشكل 3. إعادة بناء نموذج 3D من الخلايا المصابة بالعدوى بالنقل مع HEK293 HA-CRF-CLS R2 من الصور باستخدام برامج Imaris. تقديم سطح نواة وخلق بؤر تصف ملحقات GPCR تحويلها إلى حويصلات صغيرة. تمت معالجة البيانات أولا باستخدام مضان Imaris الذي يسمح للتجزئة والتصور 3D المجهر من مجموعة البيانات. ثم، تم استخدام Imaris XT لImaris التفاعل مع MATLAB. تم تبادل شدة فوكسل فيبقعة الإحداثيات. البقع الطيف ترميز الألوان (الأزرق 0-5 ميكرون، الأخضر، 6-10 ميكرون، ميكرومتر 11-15 الأصفر والأحمر> ميكرون 15) تمثل النموذج المسافة الغشاء النووي. مقياس تقييم من 5 ميكرون.
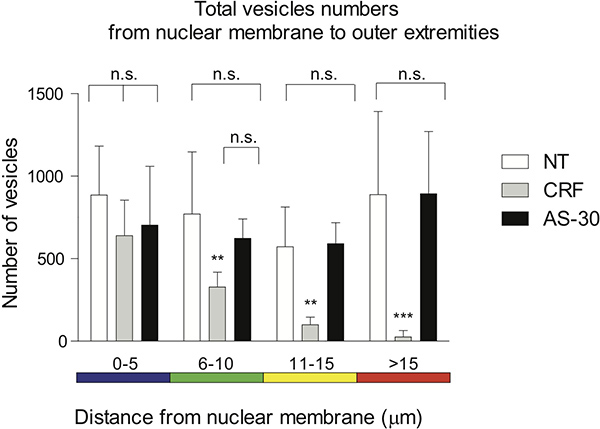
ويستخدم الشكل 4. تمثيل بياني لتوزيع البعيدة من البقع المرسومة في لون الطيف مشفرة في 5 ميكرومتر فترات لتصور المسافة بين voxels من الغشاء النووي. يوجد علاج والمعالجة المسبقة مع خصم (AS-30، 1 ميكرومتر، 30 دقيقة) قبل بدء العلاج ناهض (CRF، 1 ميكرومتر، 30 دقيقة) لا تظهر أي مهمة (م) الفرق في الانكماش GPCR. علاج الخلايا مع ناهض (CRF، 1 ميكرومتر، 30 دقيقة) يقلل تدريجيا المسافة بين عدد من CRF-R2 المحتوية على voxels بالمقارنة مع أي علاج 0-5 ميكرون (NS)، 6-15 ميكرون (ع ** <0.01) و> 15 ميكرون (*** p <0.005)، أو AS-30 ميكرومتر العلاج 0-10 (م)، 11-15 ميكرومتر (** p <0.01) و> 15 ميكرون (*** P <0.005).
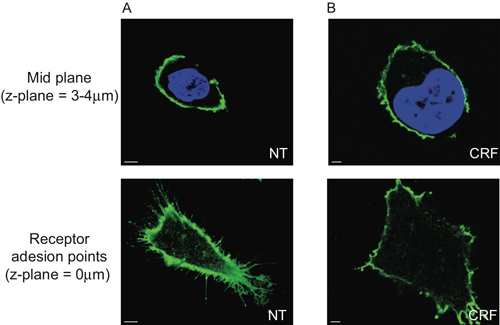
الشكل 5. الحد من تحليل المظهرية 2D من الخلايا transfected مع HEK 293 HA-CRF-R2. تصور المقطع منتصف الطائرة من الخلايا (3-4μm فوق ساترة الزجاج) تظهر وسط النوى مع دابي وHA-R2-CRF بحثها استخدام الألغام المضادة للHA وتصور باستخدام اليكسا 488nm مترافق المضادة للماوس (مفتش 1) الثانوية والأجسام المضادة المكتسبة مع CLS لا يظهر أي فرق بين عدم المعالجة (A) (NT) و (B) ناهض (CRF، 1 ميكرومتر، 30 دقيقة)، في حين أن نقطة التصاق مستقبلات مختلفة بشكل كبير.
الفيلم 1. الدورية بحرية نموذج 3D في وضع "فق" من HEK 293 الخلايا transfected مع CRF-HA-R2، أي علاج، لتقييم الاختلافات بين النمط الظاهري الخلوية البروتينات المستقبلة. مقياس بار 5 حتي 20 ميكرون./ files/ftp_upload/4233/4233movie1.avi "الهدف =" _blank "> اضغط هنا لمشاهدة الفيلم.
فيلم 2. الدورية بحرية نموذج 3D في وضع "فق" من HEK 293 الخلايا transfected مع HA-CRF-R2، والعلاج ناهض، CRF (CRF، 1 ميكرومتر، 30 دقيقة) لتقييم الاختلافات بين النمط الظاهري الخلوية البروتينات المستقبلة. شريط نطاق 5 حتي 20 ميكرون. انقر هنا لمشاهدة الفيلم.
الفيلم 3. بحرية 3D الدورية في وضع "فق" من HEK 293 الخلايا transfected مع CRF-HA-R2، المعالجة مع خصم (AS-30، 1 ميكرومتر، 30 دقيقة) قبل بدء العلاج ناهض (CRF، 1 ميكرومتر، 30 دقيقة ) لتقييم الاختلافات بين النمط الظاهري الخلوية البروتينات المستقبلة. شريط نطاق 5 حتي 20 ميكرون. انقر هنا لمشاهدة الفيلم.
Discussion
أثبتنا أن يسببها العلاج CRF تغييرا كبيرا في التشكل وموقع CRF-R2. وقد تحول دون التغيير في CRF-R2 عن طريق العلاج خصم انتقائية. أثبتنا أن التعديلات لم يتم اكتشاف مستقبلات ويمكن أن لا تقاس باستخدام معيار تقنيات متعددة الأطياف 2D. القدرة على دراسة الصور 3D المعقدة أمر بالغ الأهمية ?...
Disclosures
الإعلان عن أي تضارب في المصالح.
Acknowledgements
نشكر مركز التنمية التصوير البيولوجي (BIDC) جامعة كاليفورنيا في سان فرانسيسكو لاستخدام Imaris، XT Imaris ومطلب. نشكر V. Kharazia للمساعدة التقنية وAT هنري، LK Floren، L. Daitch لمساهماتها في التحرير من المخطوطة. وأيد هذا العمل من خلال تمويل من الدولة للأبحاث الطبية في كاليفورنيا الإسراف في تناول الكحول والمخدرات من خلال UCSF لSEB، والمعاهد الوطنية للصحة: 1R21DA029966 NIH-01 وجائزة المسار السريع لفحص جمع MLSMR لUCSF SEB مدرسة، الصيدلة ( مكتب العميد والصيدلة الإكلينيكية) وكلية الطب (علم الأدوية السريري والعلاج التجريبي) لCLHK.
Materials
| Name | Company | Catalog Number | Comments |
| اسم كاشف | شركة | كتالوج رقم | التعليقات (اختياري) |
| الجنينية البشرية الكلى (HEK293) | كوكتيل الأمريكية نوع الثقافة | CRL-1573 | |
| Dulbecco في معدلة النسر متوسطة (DMEM) | إينفيتروجن | 11965118 | |
| الجنين المصل البقري (FBS) | إينفيتروجن | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | إينفيتروجن | A-11001 | |
| وحيدة النسيلة المضادة للHA.11 (مفتش 1) | كوفانس | 16B12 | |
| دابي | ناقلات المختبرات | H-1200 | ؛ |
| CRF | سيجما | C2917 | |
| Antisauvagine-30 (AS-30) | سيجما | A4727 |
References
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved