Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein analytisches Werkzeug, das zelluläre Morphologie Änderungen von Dreidimensionale Fluorescence Images Quantifiziert
In diesem Artikel
Zusammenfassung
Wir entwickelten eine Software-Plattform, Imaris Neuroscience, ImarisXT und MATLAB, um die Änderungen in der Morphologie einer undefinierten Form von dreidimensionalen konfokale Fluoreszenz einzelner Zellen aufgenommen zu messen verwendet. Dieser neuartige Ansatz kann verwendet werden, um Veränderungen in der Zellform nach Aktivierung des Rezeptors zu quantifizieren und stellt somit eine mögliche zusätzliches Werkzeug für die Wirkstoffforschung.
Zusammenfassung
Die häufigsten Software-Analyse-Tools für die Messung der Fluoreszenz Bilder sind für zweidimensionale (2D) Daten, die auf manuelle Einstellungen für die Aufnahme und den Ausschluss von Datenpunkten und computergestützte Mustererkennung, um die Interpretation und die Ergebnisse der Analyse zu unterstützen verlassen. Es wird zunehmend wichtig, in der Lage sein Fluoreszenzbilder von dreidimensionalen (3D) ausgebildet Datensätze zu messen, um in der Lage sein, die Komplexität der zellulären Dynamik zu erfassen und verstehen, die aufgrund von zellulären Plastizität innerhalb biologischer Systeme. Sophisticated Mikroskopie Instrumente der Visualisierung von 3D-Fluoreszenz-Aufnahmen durch den Erwerb von multispektralen Fluoreszenz-Bildern und leistungsstarke Analysesoftware, die die Bilder rekonstruiert aus konfokalen Stacks, die dann eine 3D-Darstellung der gesammelten 2D-Bildern erlaubt. Fortschrittliches Design-based Stereologie Methoden aus der Angleichung und Annahmen des origina fortgeschrittenl Modell-basierte Stereologie 1 auch in komplexen Gewebeschnitten 2. Trotz dieser wissenschaftlichen Fortschritte in der Mikroskopie, bleibt ein Bedarf an einem automatisierten Verfahren, das analytische schöpft die intrinsische 3D-Daten für die Analyse und Quantifizierung der komplexe Veränderungen der Zellmorphologie, Proteinlokalisierung und Rezeptortransport ermöglichen.
Aktuelle Techniken zur Verfügung Fluoreszenz-Bildern zu quantifizieren sind Meta-Morph (Molecular Devices, Sunnyvale, CA) und Image J (NIH), die manuelle Analyse liefern. Imaris (Andor Technology, Belfast)-Software bietet das Merkmal MeasurementPro, welche die manuelle Erzeugung von Messpunkten, die in einem Volumen Bild platziert werden können oder auf einem gezogenen Serie von 2D-Slices, um ein 3D-Objekt zu erstellen. Diese Methode eignet sich für Ein-Klick-Punkt-Messungen, um eine Linie zwischen zwei Objekten zu messen oder ein Polygon, das einen Bereich von Interesse umschließt schaffen, aber es ist schwierig, kompl geltenex Zellularnetz Strukturen. Filament Tracer (Andor) ermöglicht die automatische Erkennung des 3D neuronalen fadenähnlicher aber dieses Modul wurde entwickelt, um definierte Strukturen, wie Neuronen, die von Dendriten, Axonen und Stacheln (Baumstruktur) enthalten sind zu messen. Dieses Modul wurde raffiniert Verwendete zu morphologischen Messungen nicht-neuronalen Zellen 3 machen jedoch vorsehen, die Ausgangsdaten mit Informationen von einer ausgefahrenen Zellularnetz unter Verwendung einer Software, die auf einem definierten Zellform anstatt eine amorphe-förmigen zellularen Modell abhängt. Um das Problem zu analysieren amorphe-förmigen Zellen und Bereitstellung der Software besser geeignet, um eine biologische Anwendung zu überwinden, entwickelte Imaris Imaris Cell. Dies war ein wissenschaftliches Projekt mit der Eidgenössischen Technischen Hochschule, die entwickelt wurde, um die Beziehung zwischen Zellen und Organellen zu berechnen. Während die Software ermöglicht die Detektion von biologischen Zwänge, durch Erzwingen einen Kern pro Zelleund Verwenden Zellmembranen Zellen zu segmentieren, kann er nicht verwendet, um Fluoreszenz-Daten, die nicht kontinuierlich, da idealerweise Zelloberfläche baut ohne Hohlräume zu analysieren. Nach unserem Kenntnisstand derzeit kein Benutzer änderbare automatisierten Ansatz, morphometrische Informationen liefert aus 3D-Fluoreszenz-Bildern entwickelt worden, erzielt zellulären räumliche Information einer undefinierten Form (Abbildung 1).
Wir haben eine analytische Plattform mit dem Imaris Kern Softwaremodul und Imaris XT Schnittstelle zu MATLAB (Mat Works, Inc.) entwickelt. Diese Tools ermöglichen die 3D-Messung von Zellen ohne eine vordefinierte Form und mit inkonsistenten Fluoreszenz-Netzwerk-Komponenten. Darüber hinaus wird diese Methode können die Forscher die Know-how erweitert haben in biologischen Systemen, aber nicht Vertrautheit EDV-Anwendungen, um eine Quantifizierung der morphologischen Veränderungen in der Zelle Dynamik durchzuführen.
Protokoll
Ein. Dreidimensionale morphometrische Analyse von Single-cell phänotypischen Veränderungen
- Menschliche embryonale Nieren-(HEK293) Zellen wurden mit Hämagglutinin (HA)-Tag Corticotropin Releasing Factor-Rezeptor-2 (CRF-R2), einen G-Protein-gekoppelten Rezeptor (GPCR), wie zuvor beschrieben, 4, 5.
- Die Zellen wurden unbehandelt gelassen (keine Behandlung, NT), stimuliert mit dem CRF-R2 endogener Ligand, Corticotropin Releasing Factor, CRF (1 pM, 30 min) oder vorbehandelt mit einem selektiven CRF-R2-Antagonisten, Anti-Sauvagin 30 (AS -30, 1 pM, 30 min) vor dem Agonist Behandlung.
- Die Zellen wurden dann fixiert, permeabilisiert und mit Anti-HA. CRF-R2 wurde unter Verwendung Alexa 594 nm Konjugat anti-Maus (IgG 1) Antikörper. DAPI wurde verwendet, um die Kerne mitotischen Phase visualisieren.
- Um den Experimentator Subjektivität zu begrenzen, wurden die experimentellen Bedingungen erst nach wurden die Bilder erfasst und analysiert bekannt.
- Wir aufgenommenen Bildess aus dem Festnetz HEK293 Zellen unter Verwendung eines Plan-Apochromat 63x/1.4 Öl DIC objektive und Zeiss LSM 510 META konfokalen Mikroskop mit einem Coherent integrierten Zwei-Photonen-Laser-System eines Verdi-V5 Laser und einem Mira 900-F Lasersystem besteht.
- Während der Datenaufnahme Verfahren wurden die Zellen sowohl durch multispektrale Sektionieren, 488 nm und 790 nm (~ 350 nm 2ph Ex.) Und z-Partitionierung (0,5 um Inkremente), um Daten aus der Kernmembran zu dem äußeren extrazellulären Rezeptor umfassen kompartimentiert Extremitäten.
- Die Fluoreszenz-Daten wurden zunächst verarbeitet Imaris, die die Visualisierung und Segmentierung von 3D-Mikroskopie Datensatz ermöglicht, und ein 3D-Modell von kubischen Voxeln komponiert wurde für morphometrische Analyse erstellt.
Dann wurde Imaris XT Modul-Schnittstelle Imaris mit MATLAB Computerprogramm Sprache genutzt werden, um die Stelle Koordinaten der GPCR-Erweiterungen zu bestimmen.
Zur Berücksichtigung zelluläre Variabilität nehmen, erhalten wir und analysiert Fluoreszenzbilder taken aus 22 Zellen: keine Behandlung (NT) (n = 7), Agonisten (CRF)-Behandlung (n = 8) und die Vorbehandlung mit Antagonisten (AS-30) vor dem Agonist Behandlung (n = 7) (Figur 2) .- Die Region of Interest (ROI) sollte eine Zelle, die nicht aktiv in mitotischen Bühne und nicht auf andere Zellen zu schließen. Auf diese Weise wird die Analyse die Zellen mit nur einem Kern und den Rezeptor Verlängerungen nicht gestört durch Nähe zu anderen Zellen.
- Die zelluläre 3D-Struktur wurde zuerst aus multispektralen Fluoreszenz-Daten mit Imaris (v.7.1.1) rekonstruiert.
- Nach dem Algorithmus durch Imaris gestalten, wurde zunächst die Oberfläche Rendering verwendet, um die Kernmembran repräsentieren. Imaris wird bestimmen, ob es mehr als einen Kern in der ROI.
- Dann wird der Flecken-Erstellung Algorithmus wurde verwendet, um die CRF-R2-Erweiterung finden. Spots Detektion verwendet wurde, weil es für Hintergrundgeräusche und unregelmäßigen Intensität des kompensiertkomplexes Netzwerk aus amorphem geformten Zellen.
- Um die Aufnahme von jeder Einheit der Fluoreszenzdetektion von CRF-R2 zu maximieren, wurde die Flecken 'Durchmesser bis 0,2 um, die die kleinste Einheit innerhalb des Bildes, um unterschiedliche Informationen in Form einer gemessenen Intensität mit einem Gauß-Filter extrapolieren gesetzt. Spot-Filterung in der spots automatisierte Erstellung einbezogen. Die Software, jedoch gibt dem Benutzer die Flexibilität, Filter zu verwenden, um die Parameter zu definieren.
- Um Daten abgeschnitten zu vermeiden, wurde der Datensatz von 8-Bit (unsigned) festen Punkt, auf 32-Bit Dezimalzahl umgewandelt.
- Die Voxel-Intensitäten Daten ausgetauscht wurden, um Koordinaten unter Verwendung Imaris XT-Modul eine Schnittstelle mit MATLAB und die exakte räumliche Position jedes Spots durch Ausführen einer Transformation unter Verwendung der Entfernung Kernmembran als Bezugspunkt (Abbildung 3) bestimmt erkennen.
- Die resultierenden Daten können quantifiziert und dargestellt werden in einem grafischen Format für sTATISTISCHE Analyse. Vergleiche zwischen den Gruppen wurden mit Zwei-Wege-ANOVA und Bonferroni Posttest. Die Daten sind als Mittelwert ± Standardabweichung präsentiert. Unterschiede werden als signifikant bei * p <0,05. Berechnungen wurden mit GraphPad Prism 5,02 (Abbildung 4).
2. Repräsentative Ergebnisse
Um die Kraft des Ansatzes zu demonstrieren, haben wir die zelluläre Veränderungen quantifiziert, die sich aus der Interaktion von G-Protein-gekoppelten Rezeptoren (GPCRs) und Corticotropin Releasing Factor-Rezeptor-2 (CRF-R2) mit seinem endogenen Liganden CRF in transfizierten HEK293-Zellen.
Wir zeigen, dass CRF-R2-Rezeptoren in der Plasmamembran und ragen aus endlichen Bereichen der Membran der Zellen (2A und Movie 1) befinden. Verwendung herkömmlicher 2D-Analyse ist es möglich, diese Untermenge von extrazellulären CRF-R2-Rezeptoren nur erkennen, wenn man den Rezeptor zu analysieren Adhäsion points auf dem Glas bedeckt. Folglich verlieren wir weitere Informationen aus z-stacked multispektralen Daten (Abbildung 5) abgeleitet.
Wenn die Zellen mit CNI behandelt werden, werden die extrazellulären Rezeptoren stark reduziert, wie durch die Abnahme des Abstandes der Flecken von der Plasmamembran gezeigt. Sie werden auch von vorwiegend endliche Orten in eine Anzahl diskreter Orte (2B und Film 2) umverteilt.
Die Wirkung von CRF auf Rezeptormembranpräparation Verteilung wird durch Vorbehandlung mit dem CRF-R2 spezifischer Antagonist, antisavagine 30 (AS-30) verhindert, und wir finden, dass die CRF-R2-Erweiterungen nicht ändern (2C und Movie 3).
Das distale Verteilung der Flecken, aufgetragen in den 5 um Spektrumspreizung farbcodiert Abständen wird genutzt, um den Abstand der Voxel aus der Kernmembran visualisieren. Keine Behandlung und Antagonist pretreatment (AS-30, 1 M, 30 min) vor Agonisten-Behandlung (CRF, 1 M, 30 min) zeigen keinen signifikanten (ns) Unterschied in der GPCR Kontraktion. Behandlung der Zellen mit dem Agonisten (CRF, 1 pM, 30 min) fortschreitend verringert die Anzahl der CRF-R2-haltigen Voxeln wenn die keiner Behandlung, 0-5 um (ns), 6-15 um (Vergleich ** p < 0,01) und> 15 um (*** p <0,005), oder im Vergleich zu AS-30 Behandlung, 0-10 um (ns), 11-15 um (** p <0,01) und> 15 um (*** p <0,005) (Figur 4).
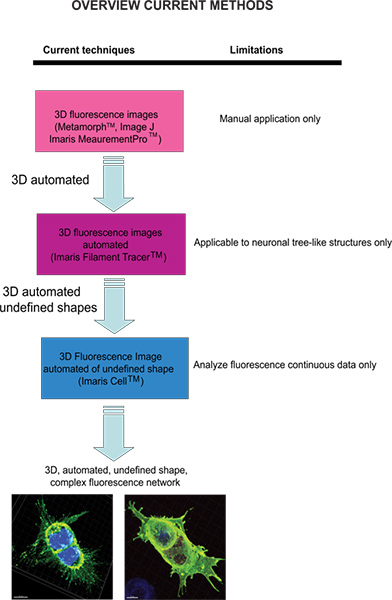
Abbildung 1. Schematische Darstellung der aktuell verfügbaren Techniken und deren Beschränkung auf Fluoreszenz-Bildern analysieren. Klicken Sie hier für eine größere Abbildung zu sehen .
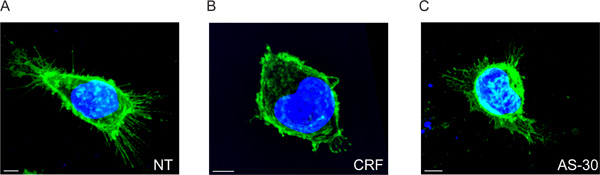
Abbildung 2. 3D multispektralen Fluoreszenz-Aufnahmen von HEK293 transfizierten Zellen mit HA-CRF-R2 vor und nach Agonisten und-Antagonisten. Fusionierten Bildern zeigt HA-CRF-R2-sondiert Verwendung von Anti-HA und visualisiert Alexa 488-konjugierte anti-Maus (IgG 1) sekundären Antikörper; DAPI wurde verwendet, um die Kerne zu visualisieren. Die Bilder wurden mit einem konfokalen Laser-Scanning (CLS)-Mikroskop erworben. Maßstab 5 um.
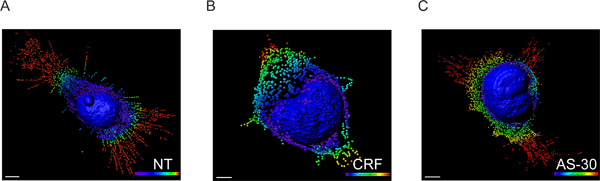
Abbildung 3. 3D-Modell der HEK293 transfizierten Zellen mit HA-CRF-R2 rekonstruiert aus CLS Bilder mit Imaris Software. Oberflächen-Rendering des Kerns und Flecken Erstellung beschreiben GPCR-Erweiterungen umgewandelt kleine Bläschen. Die Fluoreszenz-Daten wurden zunächst verarbeitet Imaris die Visualisierung und Segmentierung von 3D-Mikroskopie Datensatz ermöglicht. Dann wurde Imaris XT-Schnittstelle Imaris mit MATLAB genutzt. Die Voxel Intensitäten wurden in ausgetauschtOrt koordiniert. Die Spots Spektrum-codierte Farbe (blau 0-5 um, grün, 6-10 um, gelb 11-15 um und rot> 15 um) geben den Abstand bilden die Kernmembran. Skalieren 5 um.
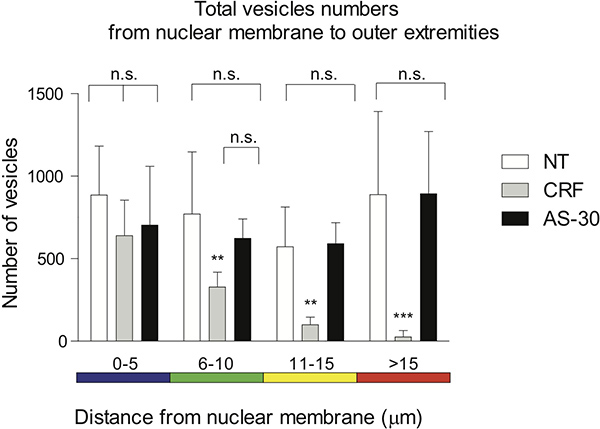
Abbildung 4. Graphische Darstellung der Verteilung von distalen Spots aufgetragen im Spektrum-Farbe in 5 um Intervallen codiert wird verwendet, um den Abstand der Voxel aus der Kernmembran visualisieren. Keine Behandlung und Vorbehandlung mit einem Antagonisten (AS-30, 1 M, 30 min) vor Agonisten-Behandlung (CRF, 1 M, 30 min) zeigen keinen signifikanten (ns) Unterschied in der GPCR Kontraktion. Behandlung der Zellen mit einer Agonisten (CRF, 1 pM, 30 min) fortschreitend verringert sich der Abstand der Anzahl an CRF-R2-haltigen Voxeln, wenn keine Behandlung 0-5 um (ns), 6-15 um (** p Vergleich <0,01) und> 15 um (*** p <0,005) oder AS-30 Behandlung 0-10 um (ns), 11-15 um (** p <0,01) und> 15 um (*** p <0,005).
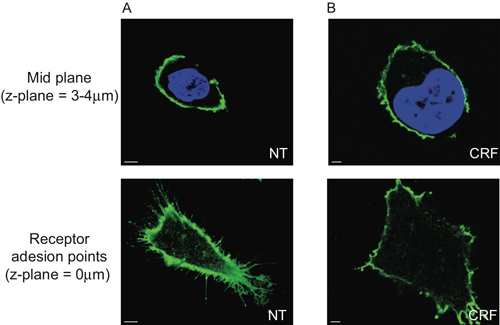
Abbildung 5. Begrenzung der 2D morphometrische Analyse von HEK 293 transfizierten Zellen mit HA-CRF-R2. Die Midplane Abschnitt von Zellen (3-4 um über dem Deckglas) zeigt den Mittelpunkt der Kerne sichtbar gemacht mit DAPI und HA-CRF-R2 sondiert Verwendung von Anti-HA und visualisiert Alexa 488nm konjugiertem anti-Maus (IgG 1) sekundäre Antikörper und erwarb mit CLS zeigt keinen Unterschied zwischen (A) keiner Behandlung (NT) und (B)-Agonisten (CRF, 1 M, 30 min), während die Rezeptor-Klebepunkten dramatisch unterschiedlich sind.
Movie 1. Frei rotierende 3D-Modell in der "Surpass"-Modus der HEK 293 transfizierten Zellen mit HA-CRF-R2, keine Behandlung, um zelluläre Phänotyp Unterschiede zwischen Rezeptorproteine zu bewerten. Maßstab von 5 bis 20 um./ Files/ftp_upload/4233/4233movie1.avi "target =" _blank "> Klicken Sie hier, um Film zu sehen.
Film 2. Freilaufend 3D-Modell in der "Surpass" Modus der HEK 293-Zellen transfiziert mit HA-CRF-R2,-Agonisten, CRF (CRF, 1 pM, 30 min) auszuwerten zelluläre Phänotyp Unterschiede zwischen Rezeptorproteine. Maßstab von 5 bis 20 um. Klicken Sie hier, um Film zu sehen.
Film 3. Freilaufend 3D im "Surpass" Modus der HEK 293-Zellen transfiziert mit HA-CRF-R2, Vorbehandlung mit einem Antagonisten (AS-30, 1 pM, 30 min), bevor Agonist Behandlung (CRF, 1 pM, 30 min ) zur Auswertung zelluläre Phänotyp Unterschiede zwischen Rezeptorproteine. Maßstab von 5 bis 20 um. Klicken Sie hier, um Film zu sehen.
Diskussion
Wir zeigten, dass CRF Behandlung eine signifikante Veränderung in der Morphologie und Lage der CRF-R2 induziert. Die Veränderung der CRF-R2 wurde durch selektive Antagonisten Behandlung gehemmt. Wir haben gezeigt, dass Rezeptor Modifikationen, die nicht erkannt wurden und kann nicht gemessen werden mit den Standard-2D-multispektralen Techniken. Die Fähigkeit, komplexe 3D-Bilder zu studieren ist kritisch, um die Komplexität der biologischen Parameter für morphometrische Analyse aufzunehmen. Wir waren in der Lage, 3D...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir danken dem Biological Imaging Development Center (BIDC) University of California, San Francisco für die Nutzung des Imaris, Imaris XT und Matlab. Wir danken V. Kharazia für die technische Unterstützung und AT Henry, LK Floren, L. Daitch für ihre Beiträge zur Bearbeitung des Manuskripts. 1R21DA029966-01 und NIH Fast Track Award an die MLSMR Sammlung SEB, UCSF School of Pharmacy screenen (: Diese Arbeit wurde durch Mittel aus dem US-Bundesstaat Kalifornien Medical Research zum Thema Alkohol & Substance Abuse durch UCSF SEB, die National Institutes of Health unterstützt Dekanat und Klinische Pharmazie) und die School of Medicine (Clinical Pharmacology & Experimental Therapeutics), um CLHK.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
| Menschlichen embryonalen Nierenzellen (HEK293) | American Type Culture Collection | CRL-1573 | |
| Dulbeccos modifiziertem Eagle Medium (DMEM) | Invitrogen | 11965118 | |
| Fötales Rinderserum (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | A-11001 | |
| monoklonalen Anti-HA.11 (IgG 1) | Covance | 16B12 | |
| DAPI | Vector Laboratories | H-1200 | ; |
| CRF | Sigma | C2917 | |
| Antisauvagine-30 (AS-30) | Sigma | A4727 |
Referenzen
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten