Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Аналитический инструмент, который количественно клеточные изменения морфологии от трехмерного изображения флуоресценции
В этой статье
Резюме
Мы разработали программную платформу, которая использует Imaris Neuroscience, ImarisXT и MATLAB для измерения изменений в морфологии неопределенная форма взята из трехмерного конфокальной флуоресцентной отдельных клеток. Этот новый подход может быть использован для количественной оценки изменений формы клеток после активации рецепторов и, следовательно, представляет собой возможный дополнительный инструмент для обнаружения наркотиков.
Аннотация
The most common software analysis tools available for measuring fluorescence images are for two-dimensional (2D) data that rely on manual settings for inclusion and exclusion of data points, and computer-aided pattern recognition to support the interpretation and findings of the analysis. It has become increasingly important to be able to measure fluorescence images constructed from three-dimensional (3D) datasets in order to be able to capture the complexity of cellular dynamics and understand the basis of cellular plasticity within biological systems. Sophisticated microscopy instruments have permitted the visualization of 3D fluorescence images through the acquisition of multispectral fluorescence images and powerful analytical software that reconstructs the images from confocal stacks that then provide a 3D representation of the collected 2D images. Advanced design-based stereology methods have progressed from the approximation and assumptions of the original model-based stereology1 even in complex tissue sections2. Despite these scientific advances in microscopy, a need remains for an automated analytic method that fully exploits the intrinsic 3D data to allow for the analysis and quantification of the complex changes in cell morphology, protein localization and receptor trafficking.
Current techniques available to quantify fluorescence images include Meta-Morph (Molecular Devices, Sunnyvale, CA) and Image J (NIH) which provide manual analysis. Imaris (Andor Technology, Belfast, Northern Ireland) software provides the feature MeasurementPro, which allows the manual creation of measurement points that can be placed in a volume image or drawn on a series of 2D slices to create a 3D object. This method is useful for single-click point measurements to measure a line distance between two objects or to create a polygon that encloses a region of interest, but it is difficult to apply to complex cellular network structures. Filament Tracer (Andor) allows automatic detection of the 3D neuronal filament-like however, this module has been developed to measure defined structures such as neurons, which are comprised of dendrites, axons and spines (tree-like structure). This module has been ingeniously utilized to make morphological measurements to non-neuronal cells3, however, the output data provide information of an extended cellular network by using a software that depends on a defined cell shape rather than being an amorphous-shaped cellular model. To overcome the issue of analyzing amorphous-shaped cells and making the software more suitable to a biological application, Imaris developed Imaris Cell. This was a scientific project with the Eidgenössische Technische Hochschule, which has been developed to calculate the relationship between cells and organelles. While the software enables the detection of biological constraints, by forcing one nucleus per cell and using cell membranes to segment cells, it cannot be utilized to analyze fluorescence data that are not continuous because ideally it builds cell surface without void spaces. To our knowledge, at present no user-modifiable automated approach that provides morphometric information from 3D fluorescence images has been developed that achieves cellular spatial information of an undefined shape (Figure 1).
We have developed an analytical platform using the Imaris core software module and Imaris XT interfaced to MATLAB (Mat Works, Inc.). These tools allow the 3D measurement of cells without a pre-defined shape and with inconsistent fluorescence network components. Furthermore, this method will allow researchers who have extended expertise in biological systems, but not familiarity to computer applications, to perform quantification of morphological changes in cell dynamics.
протокол
1. Трехмерная морфометрического анализа одной ячейке фенотипические изменения
- Эмбриональной почки человека (HEK293) клетки трансфицировали гемагглютинина (HA)-отмеченном кортикотропин рилизинг-фактор рецептора-2 (CRF-R2), G-белком рецептор (GPCR), как описано ранее 4, 5.
- Клетки лечить (без лечения, NT), стимулировали CRF-R2 эндогенных лигандов, кортикотропин рилизинг-фактор, CRF (1 мкМ, 30 мин), или предварительно с селективным CRF-R2 антагониста, анти-саувагин 30 (AS -30, 1 мкМ, 30 мин) до агонистов.
- Затем клетки фиксированы, проницаемость и обрабатывали анти-HA. CRF-R2 были визуализированы с помощью Alexa 594 нм сопряженных анти-мышиных (IgG 1) антител. DAPI был использован для визуализации сцены ядер митотической.
- Чтобы ограничить экспериментатор субъективности, условия эксперимента не были известны только после Изображения были получены и проанализированы.
- Мы приобрели изображенияа с фиксированной НЕК293 помощью план-апохромат 63x/1.4 масла ДВС объективных и Zeiss LSM 510 META конфокальной микроскопии связан с когерентной интегрированной двухфотонной лазерной системы, состоящей из Верди-V5 лазерных и Мира, 900-F лазерной системы.
- В процессе сбора данных, клетки были разграничены как по мультиспектральных срезов, 488 нм и 790 нм (~ 350 нм 2ph Ex.), Г-разбиения (0,5 мкм шагом), чтобы включить данные из ядерной мембраны к внешнему внеклеточных рецепторов конечностях.
- Флуоресценции данные впервые были обработаны с помощью Imaris, которая позволяет визуализировать и сегментации 3D наборов данных микроскопии и 3D-модели состоят из кубического вокселей был создан для морфометрического анализа.
Тогда, Imaris XT модуль был использован для интерфейса Imaris с MATLAB язык компьютерную программу для определения координат места GPCR расширения.
Для того чтобы учесть сотовой изменчивости, мы получили и проанализировали флуоресцентных изображений Takан из 22 клеток: отсутствие лечения (NT) (п = 7), агонистов (CRF) лечение (n = 8) и предварительной обработки с антагонистом (AS-30) до агонистов (п = 7) (рис. 2) .- Область интереса (ROI) должна включать в себя одну ячейку, которая не находится в активной стадии митотического и не близко к другим клеткам. Таким образом, анализ будет включать в себя клетки с одним ядром и рецепторов расширения не возмущенной близости от других клеток.
- Ячеистая структура 3D впервые был реконструирован из мультиспектральных флуоресценции данных с использованием Imaris (v.7.1.1).
- Следуя алгоритму разработана Imaris, в первую поверхность рендеринга была использована для представления ядерной мембраны. Imaris будет определить, есть ли более одного ядра в ROI.
- Тогда пятна создания алгоритма была использована, чтобы найти CRF-R2 расширения. Места обнаружения была использована, поскольку она компенсирует фоновый шум и нерегулярные интенсивностисложная сеть аморфной формы клеток.
- Для максимального включения каждого блока флуоресценции обнаружения CRF-R2, диаметр пятна "был установлен до 0,2 мкм, что является наименьшей единицей в пределах изображения для различных экстраполировать информацию в форме измеряются интенсивности с использованием фильтра Гаусса. Пятно фильтрация была включена в местах автоматизированный процесс создания. Программное обеспечение, однако, дает пользователю возможность использовать фильтры, чтобы определить параметры.
- Чтобы избежать усечения данных, набор данных был преобразован из 8-битных (без знака) неподвижная точка, в 32-разрядные десятичные.
- Воксела данных интенсивности были обменены определить координаты данных с использованием Imaris XT модуля сопряжена с MATLAB и точное пространственное расположение каждого места была определена путем проведения расстояние преобразование с использованием ядерной мембраны в качестве точки отсчета (рис. 3).
- Полученные данные могут быть определены и представлены в графическом формате сТАТИСТИЧЕСКИЕ анализа. Сравнения между группами проводили с использованием двустороннего ANOVA и Bonferroni посттестовых. Данные представлены как среднее значение ± SD. Различия являются значительными в * р <0,05. Расчеты были сделаны с GraphPad Prism 5.02 (рис. 4).
2. Представитель Результаты
Чтобы продемонстрировать силу нашего подхода, мы количественно клеточные изменения, которые возникают в результате взаимодействия G-белком рецепторы (GPCRs) и кортикотропин рилизинг-фактор рецептора-2 (CRF-R2) с эндогенным лигандом CRF в трансфицированных НЕК293.
Мы показываем, что CRF-R2 рецепторы расположены в мембране плазмы и проект из конечных областей мембраны клетки (рис. 2A и кино 1). Использование обычных 2D анализ, можно обнаружить это подмножество внеклеточной CRF-R2 рецепторы только тогда, когда мы анализируем рецепторы адгезии роИнтс на стекло крышки. Следовательно, мы теряем любую другую информацию, полученную от г-уложены мультиспектральных данных (рис. 5).
Когда клетки обрабатывают с ХПН, внеклеточный рецепторов значительно снижается, как показано уменьшение расстояния пятна от плазматической мембраны. Они также перераспределяется преимущественно из конечных местах в ряде отдельных местах (2B рисунок и кино 2).
Эффект CRF на рецепторы мембраны распределения препятствует предварительной обработки с CRF-R2 специфического антагониста, antisavagine 30 (AS-30), и мы обнаружили, что CRF-R2 расширения не изменится (рис. 2C и кино 3).
Дистального распределения пятен, построенная в 5 мкм спектра цветом интервалов, используется для визуализации расстояния вокселей от ядерной мембраны. Никакого лечения и антагониста pretreatmЛОР (AS-30, 1 мкМ, 30 мин) до агонистов (CRF, 1 мкМ, 30 мин) не показывают значительное (нс) разница в GPCR сокращения. Лечение клеток с агонистом (CRF, 1 мкМ, 30 мин) постепенно уменьшается количество CRF-R2-содержащих вокселов по сравнению с отсутствием лечения, 0-5 мкм (нс), 6-15 мкм (** р < 0,01) и> 15 мкм (*** р <0,005), или по сравнению с AS-30 лечения, 0-10 мкм (нс), 11-15 мкм (** р <0,01) и> 15 мкм (*** р <0,005) (рис. 4).
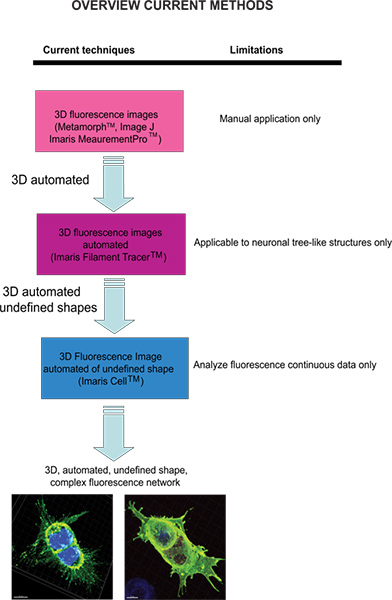
Рисунок 1. Схема имеющихся в настоящее время методов и их ограничений для анализа флуоресцентных изображений. Нажмите, чтобы увеличить показатель .
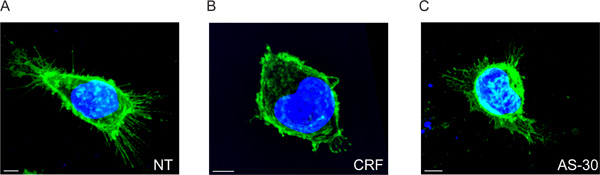
Рисунок 2. 3D мультиспектральных изображений флуоресценции HEK293 трансфицированных клеток с HA-CRF-R2 до и после агонистов и антагонистов лечение. Объединенные изображения, показывающие HA-CRF-R2-исследовали с использованием анти-HA и визуализированы с помощью Alexa 488 сопряженных анти-мышь (IgG 1) вторичные антитела; DAPI был использован для визуализации ядер. Изображения были получены с конфокальной лазерной сканирующей (CLS) микроскопом. Шкала бар 5 мкм.
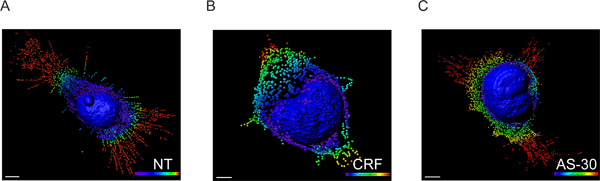
Рисунок 3. 3D-модель HEK293 трансфицированных клеток с HA-CRF-R2 восстанавливается CLS изображения с помощью Imaris программного обеспечения. Поверхность оказания ядра и места создания описания GPCR расширения превращается в мелкие пузырьки. Флуоресценции данные впервые были обработаны с помощью Imaris которая позволяет визуализировать и сегментации 3D набор данных микроскопии. Тогда, Imaris XT был использован для интерфейса Imaris с MATLAB. Воксела интенсивности были обменены наКоординаты места. Пятна спектр кодированием цвета (синий 0-5 мкм, зеленый, 6-10 мкм, желтый 11-15 мкм и красный> 15 мкм) представляют собой дистанционную форму ядерной мембраны. Масштаб 5 мкм.
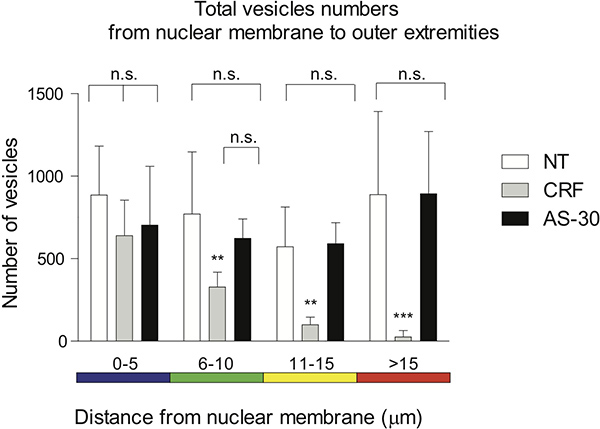
Рисунок 4. Графическое представление дистального распределения пятен построены в спектре цветов кодируется в 5 мкм интервалов используется для визуализации расстояния вокселей от ядерной мембраны. Никакого лечения и предварительной обработки с антагонистом (AS-30, 1 мкМ, 30 мин) до агонистов (CRF, 1 мкМ, 30 мин) не показывают значительное (нс) разница в GPCR сокращения. Лечение клеток с агонистом (CRF, 1 мкМ, 30 мин) постепенно сокращает расстояние число CRF-R2-содержащих вокселов по сравнению с отсутствием лечения 0-5 мкм (нс), 6-15 мкм (** р <0,01) и> 15 мкм (*** р <0,005), или AS-30 лечения 0-10 мкм (нс), 11-15 мкм (** р <0,01) и> 15 мкм (*** р <0,005).
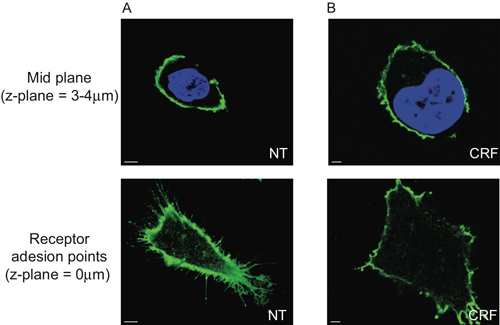
Рисунок 5. Ограничение 2D морфометрического анализа НЕК 293 трансфицированных клеток с HA-CRF-R2. В середине плоского сечения клеток (3-4 мкм над стеклом покровное), показывающий центре ядра визуализируются с DAPI и HA-CRF-R2 исследовали с использованием анти-HA и визуализированы с помощью Alexa 488nm сопряженных анти-мышь (IgG 1) вторичный антитело и полученные с CLS показывает никакой разницы между (A) без лечения (NT) и (B) агонисты (CRF, 1 мкМ, 30 мин), а точки рецепторов адгезии значительно отличаются.
Фильм 1. Свободно вращающаяся 3D-модель в "превзойти" режим НЕК 293 трансфицированных клеток с HA-CRF-R2, никакого лечения, для оценки сотовой различия между фенотипом рецепторных белков. Шкала бар от 5 до 20 мкм./ Files/ftp_upload/4233/4233movie1.avi "целевых =" _blank "> Щелкните здесь для просмотра фильмов.
Фильм 2. Свободно вращающаяся 3D-модель в "превзойти" режим НЕК 293 трансфицированных клеток с HA-CRF-R2, агонистов, ХПН (CRF, 1 мкМ, 30 мин), чтобы оценить сотовой различия между фенотипом рецепторных белков. Шкала бар от 5 до 20 мкм. Щелкните здесь для просмотра фильмов.
Фильм 3. Свободно вращающаяся 3D в "превзойти" режим НЕК 293 трансфицированных клеток с HA-CRF-R2, предварительная обработка с антагонистом (AS-30, 1 мкМ, 30 мин) до агонистов (CRF, 1 мкМ, 30 мин ), чтобы оценить сотовой различия между фенотипом рецепторных белков. Шкала бар от 5 до 20 мкм. Щелкните здесь для просмотра фильмов.
Обсуждение
Мы показали, что CRF лечении индуцированных значительные изменения в морфологии и расположение CRF-R2. Изменения в CRF-R2 ингибируется селективной очистки антагониста. Мы показали, что рецепторы изменений обнаружено не было и не может быть измерена с помощью стандартного 2D мультиспектральн?...
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Мы благодарим биологических изображений Центр развития (BIDC), Университет Калифорнии, Сан-Франциско для использования Imaris, Imaris XT и Matlab. Мы благодарим В. Харазия для оказания технической помощи и на Генри, LK Флорен, Л. Саундэкс Дэйча за их вклад в редактирование рукописи. Эта работа была поддержана финансирование от штата Калифорния медицинских исследований по алкоголю и наркомании через UCSF в SEB, Национальные Институты Здоровья: 1R21DA029966-01 и NIH Быстрый награду трек на экран MLSMR коллекции SEB, UCSF школы фармации ( Деканат и клинической фармации) и Школы медицины (клинической фармакологии и экспериментальной терапии), чтобы CLHK.
Материалы
| Name | Company | Catalog Number | Comments |
| Название реагента | Компания | Номер в каталоге | Комментарии (по желанию) |
| Эмбриональной почки человека (HEK293) | Американская коллекция типовых культур | CRL-1573 | |
| Дульбекко изменения Eagle Medium (DMEM) | Invitrogen | 11965118 | |
| Эмбриональной телячьей сыворотки (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | -11001 | |
| моноклональные анти-HA.11 (IgG 1) | Covance | 16B12 | |
| DAPI | Vector Laboratories | H-1200 | ; |
| CRF | Сигма | C2917 | |
| Antisauvagine-30 (AS-30) | Сигма | A4727 |
Ссылки
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены