A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
على صعيد رسم الخرائط الجينوم من المخدرات-DNA التفاعلات في الخلايا مع COSMIC (يشابك من الجزيئات الصغيرة لعزل لونين)
In This Article
Summary
تحديد الأهداف المباشرة للجزيئات التي تستهدف الجينوم لا يزال يشكل تحديا كبيرا. لفهم كيفية تفاعل جزيئات الحمض النووي ملزم الجينوم، وضعنا الطريقة التي تعتمد على يشابك من الجزيئات الصغيرة إلى عزل لونين (COSMIC).
Abstract
الجينوم هو الهدف من بعض مبحث المعالجة الكيميائية الأكثر فعالية، ولكن معظم هذه الأدوية تفتقر إلى الدقة تسلسل الحمض النووي، الأمر الذي يؤدي سمية والعديد من الآثار الجانبية السلبية للحد الجرعة. تستهدف الجينوم مع تسلسل محدد جزيئات صغيرة قد تمكن الجزيئات مع زيادة مؤشر العلاجي وعدد أقل من خارج الهدف آثار. N -methylpyrrole / N البلاستيك -methylimidazole هي الجزيئات التي يمكن أن تكون مصممة بشكل عقلاني لاستهداف تسلسل الحمض النووي محددة بدقة رائعة. وخلافا لمعظم عوامل النسخ الطبيعية، ويمكن البلاستيك ربط الحمض النووي الميثيلي وchromatinized دون خسارة في التقارب. خصوصية سلسلة من البلاستيك وقد درس على نطاق واسع في المختبر مع تحديد المواقع وما شابه ذلك (CSI) ومع النهج البيوكيميائية والفيزيائية الحيوية التقليدية، ولكن لا يزال دراسة مادة البولي أميد ملزمة لأهداف الجينومية في الخلايا بعيد المنال. نحن هنا الإبلاغ عن الأسلوب، يشابك من الجزيئات الصغيرة إلى ايزولاالشركة المصرية للاتصالات لونين (الكونية)، أن يحدد مادة البولي أميد ملزمة المواقع في جميع أنحاء الجينوم. COSMIC يشبه مناعي لونين (رقاقة)، ولكنه يختلف في ناحيتين هامتين: (1) يعمل على photocrosslinker لتمكين انتقائي، والتقاط تسيطر عليها مؤقتا من مادة البولي أميد الأحداث ملزمة، و (2) يستخدم مؤشر تقارب البيوتين لتنقية مادة البولي أميد تقارن -DNA في ظل ظروف تغيير طبيعة شبه لتقليل الحمض النووي التي هي غير ملزمة تساهميا. COSMIC هو الاستراتيجية العامة التي يمكن استخدامها للكشف عن أحداث الجينوم على نطاق ملزمة من البلاستيك وغيرها من عوامل العلاج الكيميائي تستهدف الجينوم.
Introduction
المعلومات لجعل يتم ترميز كل خلية في جسم الإنسان في الحمض النووي. الاستخدام الانتقائي لتلك المعلومات يحكم مصير خلية. عوامل النسخ (TFS) هي البروتينات التي تربط لتسلسل الحمض النووي محددة للتعبير عن مجموعة فرعية معينة من الجينات في الجينوم، وترتبط خلل في TFS إلى ظهور مجموعة واسعة من الأمراض، بما في ذلك العيوب التنموية، والسرطان، ومرض السكري 1،2 لقد كنا مهتمة بتطوير الجزيئات التي يمكن أن تربط بشكل انتقائي على الجينوم وتعدل شبكات تنظيم الجينات.
البلاستيك تتكون من N -methylpyrrole وN -methylimidazole والجزيئات التي يمكن أن تستهدف DNA مع الخصوصيات والانتماءات التي ينافس عوامل النسخ الطبيعية. 3-6 هذه الجزيئات تربط لتسلسل محددة في أخدود صغير من الحمض النووي بعقلانية مصممة. 4،5،7 وقد استخدمت -11 البلاستيك على حد سواء قمع وتفعيل التعبير عن ز محددأنيس. 4،12-19 لديهم أيضا مثيرة للاهتمام المضادة للفيروسات 20-24 والمضادة للسرطان 12،13،25-30 خصائص. ميزة واحدة جذابة من البلاستيك هي قدرتها على الوصول إلى تسلسل الحمض النووي التي تم ميثليته 31،32 وملفوفة حول البروتينات هيستون 9،10،33.
لقياس الخصائص ملزمة شاملة من جزيئات الحمض النووي ملزم، خلقت مختبرنا معرف الموقع وما شابه ذلك (CSI) الأسلوب. 34-39 وقوع توقع من مواقع الربط على أساس خصوصيات في المختبر (genomescapes) يمكن عرضها على الجينوم، لأن في المختبر شدة ملزمة ويتناسب طرديا مع الثوابت جمعية (K أ). 34،35،37 هذه genomescapes توفير نظرة ثاقبة الإشغال مادة البولي أميد عبر الجينوم، إلا أن قياس مادة البولي أميد ملزمة في الخلايا الحية يشكل تحديا. يتم حزم DNA بإحكام في النواة، والتي يمكن أن تؤثر على إمكانية الوصول إلى مواقع الربط. وعلىccessibility هذه تسلسل الحمض النووي لchromatinized البلاستيك لا يزال لغزا.
في الآونة الأخيرة، العديد من الأساليب لدراسة التفاعلات بين الجزيئات الصغيرة والأحماض النووية ظهرت. 40-48 القبض تقارب الكيميائية واسع مواز تسلسل الحمض النووي (الكيميائي وما يليها) هي واحدة من هذه التقنية. يستخدم علم وما يليها الفورمالديهايد لتشعبي جزيئات صغيرة إلى هدف الجيني من الفائدة ومشتقات المعقدة البيروكسيديز من جزيء صغير من الفائدة للقبض على التفاعل يجند المستهدفة. 48،49
الفورمالديهايد يشابك يؤدي إلى تفاعلات غير المباشرة التي يمكن أن تنتج ايجابيات كاذبة. 50 طورنا طريقة جديدة، ويشابك من جزيئات صغيرة لعزل لونين (الكونية)، 51 مع photocrosslinker للقضاء على ما يسمى قمم "الوهمية". 50 لتبدأ، قمنا بتصميم وتوليفها المشتقات trifunctional من البلاستيك. تحتوي هذه الجزيئات لبول ملزم DNAyamide، وهو photocrosslinker (السورالين)، والتعامل مع تقارب (البيوتين، الشكل 1). مع البلاستيك trifunctional، يمكننا التقاط تساهميا التفاعلات DNA مادة البولي أميد مع 365 نانومتر الأشعة فوق البنفسجية، والطول الموجي التي لا تضر DNA أو حث غير السورالين على أساس يشابك 51 المقبل، ونحن تفتيت الجينوم وتنقية DNA أسر تحت صرامة ونصف شروط -denaturing لتقليل الحمض النووي التي هي غير ملزمة تساهميا. وهكذا، نرى الكونية كوسيلة المتعلقة كيم وما يليها، ولكن مع قراءات أكثر مباشرة من DNA الاستهداف. الأهم من ذلك، ضعف (K 10 3 -10 4 M -1) تقارب من السورالين لDNA لا detectably تأثير مادة البولي أميد خصوصية. 51،52 يمكن تحليل شظايا الحمض النووي المخصب إما عن طريق البلمرة الكمي سلسلة من ردود الفعل 51 (COSMIC-QPCR) أو عن طريق الجيل التالي من التسلسل 53 (الكونية وما يليها). وتمكن هذه البيانات غير منحازة، موجهة الجينوم تصميم بروابط أن بينالتصرف المطلوب مواضع بهم الجيني وتقليل بعيدا عن الهدف الآثار.
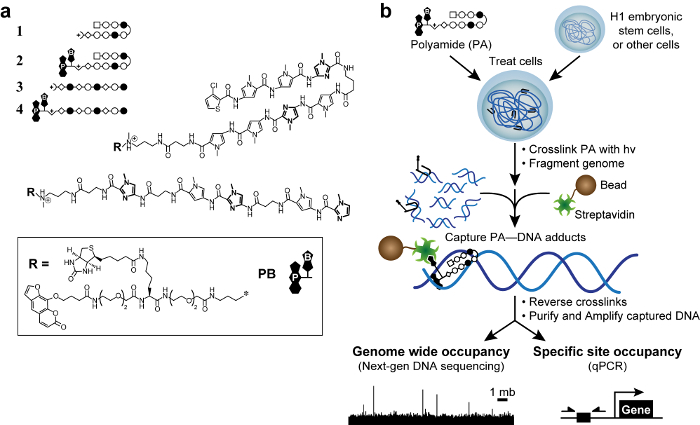
الشكل 1. البلاستيك الحيويه والمخطط الكوني. (A) البلاستيك دبوس 1-2 الهدف تسلسل الحمض النووي 5'-WACGTW-3 ". البلاستيك الخطية 3-4 الهدف 5'-AAGAAGAAG-3 ". والغامق حلقات من N-methylimidazole لوضوح. دوائر مفتوحة ومليئة تمثل N -methylpyrrole وN -methylimidazole، على التوالي. ساحة يمثل 3-chlorothiophene، والماس تمثل بيتا ألانين. يتم الرمز السورالين والبيوتين التي كتبها P و B على التوالي. (B) المخطط الكوني. يتم التعامل مع الخلايا المشتقات trifunctional من البلاستيك. بعد يشابك مع 365 نانومتر الأشعة فوق البنفسجية، والخلايا هي لوالمنفصمة DNA ysed والجيني. تضاف حبات streptavidin المغلفة المغناطيسي لالتقاط adducts-DNA مادة البولي أميد. يتم تحرير DNA ويمكن تحليلها بواسطة PCR الكمي (QPCR) أو عن طريق الجيل التالي من التسلسل (خ ع). الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
1. يشابك في الخلايا الحية
- بادئ ذي بدء ~ 2.5x10 7 خلايا H1، أو الخلايا المستزرعة الأخرى.
ملاحظة: هذا العدد من الخلايا H1 يتوافق مع خمسة أطباق 10 سم (حوالي 40٪ confluency). - تنمو الخلايا في E8 وسائل الإعلام على الأطباق المغلفة على سطح تدعم الخلايا الجذعية المحفزة (انظر المواد القائمة)، واحتضان لهم عند 37 درجة مئوية في جو مرطب 5٪ CO 2. حصاد الخلايا إنزيمي (انظر قائمة المواد). ملاحظة: لا تسمح للخلايا H1 تتجاوز 90٪ confluency. confluency يدفع التمايز عفوية من الخلايا H1. لحساب الخلايا، تنمو 10 سم اضافية طبق من الخلايا، ورفع الخلايا إنزيمي كما هو موضح في أقسام 1،8 حتي 1،10، ونعدهم مع عدادة الكريات.
- إعداد E8 سائل الإعلام والثقافة كما هو موضح في تشن وآخرون. 54
- قبل إضافة مادة البولي بروبيلين، وإزالة سائل الإعلام والثقافة الذي يقضيه مع ماصة باستور تعلق فخ الفراغ. إضافة وسائط جديدة مع النحوماصة erological وماصة موزع (8 مل لكل 10 سم طبق).
ملاحظة: إضافة وسائل الإعلام إلى جانب الطبق من أجل تجنب تعطيل الخلايا. من هذه النقطة فصاعدا حماية الخلايا من الضوء لتجنب سابق لأوانه الصور يشابك. - إضافة مادة البولي أميد مع ماصة (8 ميكرولتر من 400 ميكرومتر مادة البولي أميد في DMSO، 400 نانومتر تركيز النهائي) مباشرة إلى وسائل الإعلام ثقافة كل طبق. دوامة طبق لتفريق مادة البولي أميد بالتساوي في وسائل الإعلام.
ملاحظة: يمكن أن يكون تركيز متنوعة، ولكن ضمان عدم وجود سمية الخلوية لوحظ لعلاج المحدد. - احتضان الخلايا 24 ساعة 37 درجة مئوية في جو مرطب 5٪ CO 2، والتأكد من أنها محمية من الضوء.
ملاحظة: فترة حضانة يمكن أن تختلف لقياس مادة البولي أميد ملزم عبر الزمن. - يغسل كل 10 سم الطبق مع 4 مل PBS (1.05 ملي أحادى فوسفات البوتاسيوم، 155.17 ملي كلوريد الصوديوم، 2.97 ملي ثنائي القاعدة فوسفات الصوديوم) باستخدام ماصة المصلية وماصة ديسpenser. نضح PBS وإضافة 3 مل E8 سائل الإعلام والثقافة.
- مع أضواء خافتة، وإزالة الغطاء من 5 أطباق الثقافة ووضع الخلايا على سطح مستو خارج غطاء محرك السيارة. وضع مرشح الزجاج فوق أطباق ثقافة 5 لتصفية الضوء مع λ <300 نانومتر. وضع مصدر الأشعة فوق البنفسجية على رأس المرشح. عينات تشعبي 30 دقيقة مع 365 نانومتر الأشعة فوق البنفسجية (2.4 ميغاواط / سم 2).
ملاحظة: يشابك الوقت ينبغي أن تحدد تجريبيا. - نقل الأطباق ثقافة إلى غطاء محرك السيارة. نضح في وسائل الاعلام مع ماصة باستور تعلق فخ الفراغ. يغسل كل 10 سم الطبق مع 4 مل PBS. نضح في برنامج تلفزيوني. إضافة 3 مل انزيم لتفكك خلية (انظر قائمة المواد) في 10 سم طبق لفصل الخلايا. احتضان 5 دقائق عند 37 ° C.
ملاحظة: لا قبل تدفئة الانزيم. - إخماد الإنزيم مع 3 مل E8 وسائل الإعلام في الطبق. نقل فصل الخلايا في أنبوب واحد 15 مل المخروطية.
ملاحظة: مكان الخلايا على الجليد من هذه النقطة فصاعدا. - Centrifuge فصل الخلايا 5 دقائق، 500 x ج في 4 درجات مئوية. نضح طاف لإزالة الانزيم وسائل الإعلام.
ملاحظة: نقطة وقفة. الخلايا يمكن أن يكون فلاش المجمدة في النيتروجين السائل، وتخزينها في -80 ° C لمعالجتها لاحقا في وقت لاحق.
2. عزل لونين
- إضافة 1.2 مل العازلة الكونية (20 ملي تريس، حمض الهيدروكلوريك [الرقم الهيدروجيني 8،1]، 2 ملي EDTA، 150 ملي كلوريد الصوديوم، 1٪ تريتون-X100، 0.1٪ SDS) وماصة صعودا وهبوطا عدة مرات لresuspend الكرية خلية لكل عينة في 1.2 مل العازلة الكونية.
- إضافة 133 ميكرولتر من كل 100 ملي phenylmethylsulfonyl الفلورايد (PMSF)، و 100 ملي benzamidine، و 150 ميكرومتر pepstatin مثبطات الأنزيم البروتيني جديدة لتركيز النهائي من 1 ملم لPMSF وbenzamidine و 1.5 ميكرومتر لpepstatin. حل الانقسام من المحللة الخلية إلى قسمين العنبر 1.7 مل أنابيب microcentrifuge.
- يصوتن مع sonicator 35 دقيقة (10 ثانية في 10 ثانية قبالة والطاقة 60٪) لتفتيت الجينوم إلى ما بين 100 إلىالثاني 500 نقطة أساس.
- الحفاظ على مستوى حل لونين في موازاة أنبوب microcentrifuge لمستوى المياه في الخزان. تأكيد هذا المستوى عن طريق التفتيش البصري.
ملاحظة: تأكد من أن الحمض النووي والمنفصمة مع هلام الاغاروز 1.5٪ 55 - تحسين الوقت صوتنة تجريبيا. استخدام كمية ضئيلة من الجليد في خزان للبرد والعينات، والتأكد من الجليد لا تتدخل بين العينات والقرن الكأس.
- الحفاظ على مستوى حل لونين في موازاة أنبوب microcentrifuge لمستوى المياه في الخزان. تأكيد هذا المستوى عن طريق التفتيش البصري.
- الطرد المركزي العينة 12000 x ج 10 دقيقة. حفظ محلول مائي يحتوي على لونين للذوبان التي كتبها نقلها إلى أنبوب جديد العنبر microcentrifuge لمع ماصة. تجاهل بيليه.
- نقل 110 ميكرولتر (10٪) من عينة لأنبوب microcentrifuge جديد وتسميته DNA الإدخال. تخزين في -80 ° C. حفظ ما تبقى من عينة لونين على الجليد لاستخدامها في الخطوة 3.3.
3. لقطة من يجند-DNA Crosslinks
- استخدام ماصة للاستغناء 60 ميكرولتر streptaviالمغلفة الدين حبات مغناطيسية في أنبوب microcentrifuge. إضافة 1 مل عازلة الكونية وتخلط على nutator 5 دقائق على RT. ضع حبات مغناطيسية على الفصل المغناطيسي الرف 2 دقيقة لالتقاط حبات. إزالة العازلة الكونية مع ماصة.
ملاحظة: لا تسمح للالخرز لتجف. الشروع فورا في الخطوة التالية. - إضافة نموذج لونين (~ 1 مل) لحبات (60 ميكرولتر) و resuspend الخرز. احتضان لونين مع streptavidin المغلفة الخرز المغناطيسي لا يقل عن 4 ساعة على الدورية، خلاط هزاز في 4 درجات مئوية. احتضان العينات O / N إذا رغبت في ذلك.
4. عزل DNA الانجذاب تنقية-
- تغسل حبات مع 7 دقائق الفاصل الزمني RT مع مخازن غسل التالية لإزالة التفاعلات غير محددة. استخدام الحامل الفصل المغناطيسي لالتقاط حبات بعد كل غسل. resuspend والخرز بعد كل تغيير في غسل العازلة.
- إعداد مخازن يغسل في الماء المقطر منزوع الأيونات وفلتر (0.2 ميكرومتر) قبل الاستخدام. المخازن مخزن غسل 1 و 2 في 4 درجة مئوية لمدة الصورةأشهر everal. إعداد الاحتياطي اغسل 3 جديدة كل يوم. إضافة وإزالة مخازن غسل من العينة مع ماصة. ل2 و 4، إضافة 1 و 3 (5 ملم)، على التوالي، في يغسل. بالإضافة إلى ذلك يمكن عينات غسلها مرتين مع العازلة الكونية (مرة واحدة 12 ساعة، مرة واحدة 4 ساعة) قبل يغسل المدرجة أدناه.
- يغسل مرة واحدة مع الاحتياطي اغسل 1 (10 ملي تريس الكلورين [الرقم الهيدروجيني 8.0]، 1 ملم EDTA، 3٪ SDS). يغسل مرة واحدة مع الاحتياطي اغسل 2 (10 ملي تريس الكلورين [الرقم الهيدروجيني 8.0]، 250 ملي LiCl، 1 ملم EDTA، 0.5٪ NP40، 1٪ deoxycholate الصوديوم). يغسل مرتين مع الاحتياطي اغسل 3 (4 M اليوريا، و 10 ملي تريس الكلورين [7.5 درجة الحموضة]، 1 ملم EDTA، 0.1٪ NP-40). يغسل مرتين مع تريس EDTA (TE) العازلة (10 ملي تريس الكلورين [الرقم الهيدروجيني 8.0]، 1 ملم EDTA).
- resuspend والخرز في 200 ميكرولتر TE العازلة مع ماصة. ويشار إلى هذه عينة من الحمض النووي القبض على أنها (AP) DNA-تنقية تقارب.
- تكملة المدخلات وAP DNA مع 10x تشعبي عكس العازلة (100 ملي تريس الكلورين [الرقم الهيدروجيني 8.0] (1)، M KOH، 4 ملمEDTA) ل1x أخرى التركيز النهائي. 56،57 احتضان 30 دقيقة في 90 ° C.
ملاحظة: يمكن أيضا 254 نانومتر أشعة فوق البنفسجية يمكن استخدامها لعكس تشعبي السورالين، 58 ولكن dimers بيريميدين cyclobutyl يمكن أن تتكون من التشعيع في هذا الطول الموجي وتتداخل مع التجهيز النهائي من الحمض النووي. - لAP DNA، ووضع أنبوب microcentrifuge تحتوي العينة على الفصل المغناطيسي الرف 2 دقيقة في RT وعزل السائل (DNA) مع ماصة. نقل AP DNA (الذي أفرج عنه من الخرز) إلى أنبوب جديد microcentrifuge لالعنبر.
ملاحظة: يتم في السائل، لم يعد يرد DNA AP المطلوب إلى الخرز. - تحييد المدخلات وDNA AP مع حمض الهيدروكلوريك المركز لدرجة الحموضة 7 عن طريق إضافة ما يقرب من 1 ميكرولتر 6 N حمض الهيدروكلوريك في 100 عينة ميكرولتر مع ماصة. تأكد من أن يتم تحييد العينات عن طريق إضافة ~ 0.5 ميكرولتر على ورقة الرقم الهيدروجيني مع ماصة تحذير: حمض الهيدروكلوريك المركز هو حمض تآكل قوية. التعامل وفقا لمؤسستكم217؛ ق مبادئ توجيهية لمعدات الوقاية الشخصية المناسبة.
- إضافة ريبونوكلياز A (100 ملغ / مل) إلى 0.2 ميكروغرام / ميكرولتر تركيز النهائي على كل من المدخلات وAP DNA. احتضان 1 ساعة عند 37 ° C. إضافة بروتين K (20 ملغ / مل) إلى 0.2 ميكروغرام / ميكرولتر تركيز النهائي على كل من المدخلات وAP DNA، إضافة. احتضان 1 ساعة عند 55 ° C.
- تنقية الحمض النووي مع مجموعة تنظيف العمود DNA (انظر قائمة المواد). 55 أزل الحمض النووي في 58 ميكرولتر DNA الصف H 2 O. متجر المدخلات وAP العينات في -20 أو -80 درجة مئوية
- تحليل DNA AP التي كتبها الكمي تفاعل البلمرة المتسلسل (QPCR) مع الاشعال مكان معين، و / أو عن طريق الجيل التالي من التسلسل. لQPCR، استخدم 2 ميكرولتر AP DNA في مكان مع المعلمات التالية: 1 دورة من 95 درجة مئوية 10 دقيقة و 40 دورات من 95 درجة مئوية و 20 ثانية و 54 درجة مئوية أو 56 درجة مئوية و 20 ثانية، 72 ° C 40 ثانية.
ملاحظة: يجب أن يتم تعديل درجة الحرارة الصلب وفقا لدرجة حرارة انصهار أزواج التمهيدي المستخدمة. اختر من درجات الحرارة الصلبإعادة تقلل من دورة الكميات مع أي التضخيم غير محددة.
ملاحظة: لتحليلها من قبل الجيل القادم التسلسل، والهدف لا يقل عن 10 مليون معين يقرأ. يقرأ ويمكن زيادة عدد معين عن طريق زيادة كمية الحمض النووي AP وعن طريق الجمع بين عينات أقل إلى لخوض انتخابات التسلسل. أكثر يقرأ تحسين حساسية. حزم القياسية لتحليل الشذرة تسلسل (على سبيل المثال، هومر، 59 MACS و 60 و 61 SPP) العمل مع البيانات الكونية وما يليها. كما هو الحال مع الطرق الأخرى التي تعتمد على الجيل المقبل من التسلسل، بما في ذلك تسلسل الجينوم ورقاقة وما يليها، المناطق المتكررة تخلق الغموض في المحاذاة والتجمع، وبالتالي لا تزال تشكل تحديا تقنيا.
النتائج
لحساب غير موحدة تجزئة الجينوم وغيرها من المتغيرات، وينبغي دائما أن تطبيع DNA النقي ضد إشارة من الحمض النووي الإدخال. الاشعال محددة إلى موضع الاهتمام يمكن استخدامها. ومن المفيد أن نحلل أيضا مكان حيث لا يتوقع أن جزيء لربط، كوسيلة لمراقبة سلبية. ونحن نرى زيادة> 100 أضعاف ?...
Discussion
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam...
Disclosures
A.Z.A. is the sole proprietor of Vista Motif, LLC and WINStep Forward.
Acknowledgements
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
Materials
| Name | Company | Catalog Number | Comments |
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-ml conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
References
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal "supergroove". Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One - Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Biochemistry. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Biochemistry. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Biochemistry. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Biochemistry. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Biochemistry. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved